A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
אוטומציה של בדיקת מיקרוגרעין באמצעות ציטומטריית זרימת הדמיה ובינה מלאכותית
In This Article
Summary
בדיקת מיקרוגרעין (MN) היא בדיקה מבוססת היטב לכימות נזק לדנ"א. עם זאת, ניקוד הבדיקה באמצעות טכניקות קונבנציונליות כגון מיקרוסקופיה ידנית או ניתוח תמונה מבוסס תכונות הוא מייגע ומאתגר. מאמר זה מתאר את המתודולוגיה לפיתוח מודל בינה מלאכותית כדי להבקיע את מבחן MN באמצעות נתוני ציטומטריה של זרימת הדמיה.
Abstract
בדיקת המיקרוגרעין (MN) משמשת ברחבי העולם גופים רגולטוריים להערכת רעילות גנטית של כימיקלים. הבדיקה יכולה להתבצע בשתי דרכים: על ידי ניקוד MN בתאים בינוקליים חד-פעמיים מחולקים, חסומים ציטוקינזיס או תאים חד-גרעיניים מחולקים במלואם. מבחינה היסטורית, מיקרוסקופ אור היה שיטת תקן הזהב כדי להבקיע את המבחן, אבל זה מייגע וסובייקטיבי. ציטומטריית זרימה שימשה בשנים האחרונות כדי לדרג את הבדיקה, אך היא מוגבלת על ידי חוסר היכולת לאשר חזותית היבטים מרכזיים של תמונות תאיות. ציטומטריית זרימת הדמיה (IFC) משלבת לכידת תמונה בתפוקה גבוהה וניתוח תמונה אוטומטי, ויושמה בהצלחה כדי לרכוש במהירות תמונות של כל אירועי המפתח במבחן MN ולניקוד שלהם. לאחרונה, הוכח כי שיטות בינה מלאכותית (AI) המבוססות על רשתות עצביות קונבולוציוניות יכולות לשמש כדי לדרג נתוני בדיקת MN שנרכשו על ידי IFC. מאמר זה מתאר את כל השלבים לשימוש בתוכנת AI כדי ליצור מודל למידה עמוקה כדי לדרג את כל אירועי המפתח וליישם מודל זה כדי להבקיע נתונים נוספים באופן אוטומטי. תוצאות מודל הלמידה העמוקה של AI משתוות היטב למיקרוסקופיה ידנית, ולכן מאפשרות ניקוד אוטומטי לחלוטין של בדיקת MN על ידי שילוב IFC ו- AI.
Introduction
בדיקת מיקרוגרעין (MN) היא בסיסית בטוקסיקולוגיה גנטית כדי להעריך נזק לדנ"א בפיתוח קוסמטיקה, תרופות וכימיקלים לשימוש אנושי 1,2,3,4. מיקרו-גרעינים נוצרים מכרומוזומים שלמים או מקטעי כרומוזומים שאינם משתלבים בגרעין לאחר החלוקה ומתעבים לגופים קטנים ועגולים נפרדים מהגרעין. לפיכך, MN יכול לשמש כנקודת קצה לכימות נזק לדנ"א בבדיקת גנוטוקסיות1.
השיטה המועדפת לכימות MN היא בתוך תאים בינוקלים שחולקו פעם אחת (BNCs) על ידי חסימת חלוקה באמצעות Cytochalasin-B (Cyt-B). בגרסה זו של הבדיקה, ציטוטוקסיות מוערכת גם על ידי ניקוד תאים מונונוקלציה (MONO) ו polynucleated (POLY). הבדיקה יכולה להתבצע גם על ידי ניקוד MN בתאי MONO לא חסומים, שהוא מהיר וקל יותר להבקיע, כאשר ציטוטוקסיות מוערכת באמצעות ספירת תאים לפני ואחרי חשיפה כדי להעריך התפשטות 5,6.
ניקוד פיזי של הבדיקה בוצע באופן היסטורי באמצעות מיקרוסקופיה ידנית, שכן זה מאפשר אישור חזותי של כל אירועי המפתח. עם זאת, מיקרוסקופ ידני הוא מאתגר וסובייקטיבי1. כך פותחו טכניקות אוטומטיות, כולל סריקת שקופיות במיקרוסקופ וציטומטריית זרימה, כל אחת עם היתרונות והמגבלות שלה. בעוד ששיטות סריקת שקופיות מאפשרות להציג באופן חזותי אירועי מפתח, יש ליצור שקופיות בצפיפות תאים אופטימלית, דבר שעשוי להיות קשה להשגה. בנוסף, טכניקה זו לעתים קרובות חסר ויזואליזציה ציטופלזמית, אשר יכול לסכן את הניקוד של MONO ו POLY תאים 7,8. בעוד ציטומטריית זרימה מציעה לכידת נתונים בתפוקה גבוהה, התאים חייבים להיות lysed, ובכך לא לאפשר את השימוש בצורת Cyt-B של הבדיקה. בנוסף, כטכניקה שאינה הדמיה, ציטומטריית זרימה קונבנציונלית אינה מספקת אימות חזותי של אירועי מפתח 9,10.
לכן, ציטומטריית זרימת הדמיה (IFC) נחקרה לביצוע בדיקת MN. ImageStreamX Mk II משלב את המהירות והחוסן הסטטיסטי של ציטומטריית זרימה קונבנציונלית עם יכולות הדמיה ברזולוציה גבוהה של מיקרוסקופיה במערכת אחת11. הוכח כי באמצעות IFC, ניתן לצלם תמונות ברזולוציה גבוהה של כל אירועי המפתח ולהבקיע באופן אוטומטי באמצעות טכניקות מבוססות תכונות 12,13 או בינה מלאכותית (AI) 14,15. על ידי שימוש ב- IFC לביצוע בדיקת MN, הניקוד האוטומטי של תאים רבים יותר בהשוואה למיקרוסקופ בפרק זמן קצר יותר הוא בר השגה.
עבודה זו חורגת מתהליך עבודה של ניתוח תמונה16 שתואר קודם לכן ודנה בכל השלבים הנדרשים לפיתוח ואימון מודל של Random Forest (RF) ו/או רשת עצבית קונבולוציונית (CNN) באמצעות תוכנת Amnis AI (להלן "תוכנת AI"). כל השלבים הדרושים מתוארים, כולל אכלוס נתוני אמת קרקעית באמצעות כלי תיוג בסיוע AI, פרשנות של תוצאות אימון המודל, ויישום המודל לסיווג נתונים נוספים, המאפשר חישוב של גנוטוקסיות וציטוטוקסיות15.
Protocol
1. איסוף נתונים באמצעות ציטומטריית זרימת הדמיה
הערה: עיין ב- Rodrigues et al.16 עם השינויים הבאים, וציין כי ייתכן שיהיה צורך לשנות את אזורי הרכישה המשתמשים ב- IFC ללכידת תמונה מיטבית:
- בשיטה שאינה Cyt-B, בצע ספירת תאים באמצעות מונה תאים זמין מסחרית בהתאם להוראות היצרן (ראה טבלת חומרים) על כל תרבית מיד לפני התרבית ומיד לאחר תקופת ההחלמה.
- אם אתה מפעיל דגימות בציטומטר זרימת הדמיה יחיד של מצלמה, מקם את Brightfield (BF) בערוץ 4. החלף את M01 ב-M04 ואת M07 ב-M01.
הערה: "M" מתייחס לערוץ המצלמה ב- IFC. - השתמש בהגדלה של 40x במהלך הרכישה.
- בתרשים יחס גובה-רוחב של אזור BF לעומת BF במהלך הרכישה, השתמש בקואורדינטות האזור הבאות:
קואורדינטות X: 100 ו-900; קואורדינטות Y: 0.7 ו-1 (שיטת Cyt-B)
קואורדינטות X: 100 ו-600; קואורדינטות Y: 0.7 ו-1 - בתרשים עוצמת Hoechst, השתמש בקואורדינטות האזור הבאות:
קואורדינטות X: 55 ו-75; קואורדינטות Y: 9.5 ו-15 (שיטת Cyt-B)
קואורדינטות X: 55 ו-75; קואורדינטות Y: 13 ו-21 (שיטה שאינה Cyt-B) - כדי להסיר תמונות של אובייקטים אפופטוטיים ונמקיים מהנתונים, הפעל את חבילת התוכנה IDEAS 6.3 (שתיקרא מעתה "תוכנת ניתוח תמונות"; ראה טבלת חומרים).
הערה: תוכנת הבינה המלאכותית תוכננה לפעול עם קבצי .daf שעובדו באמצעות הגרסה העדכנית ביותר של תוכנת ניתוח התמונות. ודא שתוכנת ניתוח התמונות מעודכנת. - שמור עבודה זו כקובץ תבנית ( .ast).
2. יצירת קבצי .daf עבור כל קבצי .rif
- תוכנת הבינה המלאכותית מאפשרת לייבא קבצי .daf בלבד. צור קובצי .daf לכל קובצי ה- .rif בניסוי באמצעות עיבוד אצווה.
- תחת תפריט כלים , לחץ על קבצי נתוני אצווה ולאחר מכן לחץ על הוסף אצווה.
- בחלון החדש, בחר הוסף קבצים ובחר את קבצי ה- .rif שיתווספו לאצווה. תחת האפשרות בחר תבנית או קובץ ניתוח נתונים ( .ast, .daf), בחר את קובץ ה- .ast שנוצר בעבר.
- הקצה שם אצווה במידת הצורך ולחץ על אישור כדי ליצור קבצי .daf לכל קבצי ה- .rif שנטענו.
3. יצירת ניסוי בתוכנת AI
- עיין בתרשים הזרימה באיור 1 המתאר את התהליך של יצירת מודל למידה עמוקה באמצעות תוכנת AI.
- הפעל את תוכנת ה- AI וודא שהגרסה העדכנית ביותר מותקנת על ידי לחיצה על אודות בפינה השמאלית התחתונה של החלון. אם הגירסה העדכנית ביותר אינה מותקנת, פנה support@luminexcorp.com כדי להשיג אותה.
- מסך ברירת המחדל בתוכנה הוא מסך הניסוי החדש . השתמש בסמל Folder כדי לבחור היכן לשמור את הניסוי, והקלד שם לניסוי (לדוגמה, "MN model").
- תחת סוג ניסוי, לחץ על לחצן האפשרויות לצד רכבת כדי להתחיל ניסוי אימון כדי להתחיל לבנות את מודל CNN. לחץ על הבא.
- אופציונלי: אם מודל אומן בעבר, ניתן להשתמש בו כתבנית עבור מודל AI חדש, וניתן לבחור אותו כתבנית ליצירת מודל חדש מהמסך בחירת מודל תבנית . אם לא קיים מודל תבנית, פשוט דלג על שלב זה על-ידי לחיצה על הבא.
- המסך הבא הוא המסך ' הגדר דגם חדש' . תחת מודל, השם שניתן למודל בשלב 3.3 יאוכלס באופן אוטומטי.
- תחת תיאור, הקלד תיאור עבור הדגם (אופציונלי) והשאר את גודל התמונה המרבי על 150 פיקסלים.
- תחת ערוצים, לחץ על הוסף BF כדי להוסיף ערוץ brightfield לרשימה. תחת שם, לחץ פעמיים על Brightfield ושנה את שם ערוץ זה ל- BF. לחץ על הוסף FL כדי להוסיף ערוץ ניאון לרשימה. תחת שם, לחץ פעמיים על פלורסנט ושנה את שם הערוץ הזה ל- DNA.
- תחת שמות כיתות, לחץ על הוסף. בחלון הקופץ, הקלד Mononucleated ולחץ על אישור. פעולה זו מוסיפה את המחלקה החד-גרעינית לרשימת שמות המחלקות. חזור על תהליך זה כדי להבטיח ששש המחלקות הבאות מוגדרות ברשימה:
מונו-גרעינים
מונונוקל עם MN
בינוקלציה
Binucleated עם MN
פולינוקלציה
מורפולוגיה לא סדירה
לחץ על הבא.
הערה: שש מחלקות אלה של מודל אמת קרקעית ייצגו את אירועי המפתח שיש להבקיע, כמו גם תמונות עם מורפולוגיה שונה מקריטריוני הניקוד המקובלים5.- אופציונלי: אם תרצה, ניתן לכלול את תבנית הניתוח מ- 1.7 כדי להשתמש בתכונות מתוכנת ניתוח התמונות. אם ברצונך לכלול תכונות אלה במודל הבינה המלאכותית, אתר את קובץ ה- .ast ולאחר מכן מתוך התפריט הנפתח הספציפי לערוץ, בחר את ערכות המשנה של התכונות שברצונך לכלול.
- תחת Select Files, לחץ על Add Files ואתר את הקבצים הרצויים שיתווספו לתוכנת AI כדי לבנות את נתוני האמת הבסיסיים. לחץ על הבא.
הערה: חשוב להוסיף קבצי נתונים מרובים (לדוגמה, נתוני בקרה חיוביים ושליליים) המכילים מספר מספיק של כל אירועי המפתח.
- לאחר מכן, במסך בחירת אוכלוסיות בסיס , אתר את האוכלוסייה הלא-אפופטוטית מהיררכיית האוכלוסין. לחץ לחיצה ימנית על האוכלוסייה הלא אפופטוטית ובחר בחר את כל האוכלוסיות התואמות. לחץ על הבא.
הערה: חשוב להחריג אוכלוסיות שאין לסווגן (למשל, חרוזים, פסולת, כפילים וכו') - מסך זה הוא המסך ' בחר אוכלוסיות אמת' .
- אם אוכלוסיות אמת מתויגות של אירועי המפתח לא נוצרו בתוכנת ניתוח התמונות, לחץ על הבא.
- אם נוצרו אוכלוסיות אמת מתויגות בתוכנת ניתוח התמונות, הקצה אותן למחלקת המודל המתאימה.
- כדי להקצות אוכלוסיית אמת מתויגת של תאי MONO עם MN, לחץ על המחלקה Mononucleated with MN תחת Model Classes משמאל. לאחר מכן לחץ על אוכלוסיית האמת המתויגת המתאימה בצד ימין המכילה אירועים אלה.
- אם אוכלוסיות האמת המתויגות נוצרו ביותר מקובץ נתונים אחד, לחץ באמצעות לחצן העכבר הימני על אחת מאוכלוסיות האמת ובחר בחר באפשרות בחר את כל האוכלוסיות התואמות כדי להוסיף אוכלוסיות מתויגות מקבצים מרובים למחלקה המתאימה.
- לאחר שהוקצו כל אוכלוסיות האמת המתאימות, לחץ על הבא.
- במסך Select Channels , בחר את הערוצים המתאימים לניסוי. כאן, הגדר את BF לערוץ 1 ואת Hoechst לערוץ 7. לחץ לחיצה ימנית על ערוץ ובחר החל על הכל. לחץ על הבא.
- לבסוף, במסך האישור , לחץ על צור ניסוי.
- תוכנת הבינה המלאכותית טוענת תמונות מקבצי הנתונים ויוצרת את מחלקות המודל שהוגדרו בשלב 3.5.3 עם תמונות האמת הקרקעית שהוקצו בשלב 3.7. לחץ על סיום.
- לאחר יצירת הניסוי מוצגות חמש אפשרויות:
ניסוי: מספק פרטים על הניסוי, כולל קבצי נתונים שנטענו, ערוצים שנבחרו ומחלקות מוגדרות של מודלים של אמת קרקעית.
תיוג: מפעיל את כלי התיוג שבאמצעותו משתמשים יכולים לאכלס נתוני אמת קרקעית.
הדרכה: אימון מודל המבוסס על נתוני האמת בשטח.
סיווג: משתמש במודלים מאומנים כדי לסווג נתונים.
תוצאות: מספק תוצאות הן מניסוי אימון והן מניסוי סיווג.
4. אכלוס נתוני האמת בשטח באמצעות כלי תיוג בסיוע בינה מלאכותית
- לחץ על תיוג כדי להפעיל את ממשק כלי התיוג.
- לחץ על כלי הזום (סמלי זכוכית מגדלת) כדי לחתוך את התמונות לצפייה קלה יותר.
- לחץ על פס המחוון כדי להתאים את גודל התמונה כדי לשנות את מספר התמונות המוצגות בגלריה.
- לחץ על הגדרת התצוגה אפשרות ובחר Min-Max, המספקת את תמונת הניגודיות הטובה ביותר לזיהוי כל אירועי המפתח.
- לחץ על תצוגת גלריית הגדרות כדי לשנות את צבע תמונת ה- DNA לצהוב או לבן, מה שישפר את ההדמיה של אובייקטים קטנים (למשל, MN).
- לחץ על אשכול כדי להפעיל את האלגוריתם כדי לקבץ אובייקטים עם מורפולוגיה דומה יחד. לאחר השלמת קיבוץ האשכולות, האשכולות הבודדים עם מספר האובייקטים בכל אשכול מוצגים ברשימה תחת אוכלוסיות לא ידועות. בחר באשכולות בודדים כדי להציג את האובייקטים בתוך האשכול ולהקצות אובייקטים אלה למחלקות המודל המתאימות שלהם.
- לאחר שהוקצו לפחות 25 אובייקטים לכל מחלקת מודל, האלגוריתם Predict הופך לזמין. לחץ על Predict.
הערה: אובייקטים שאינם מתאימים היטב לאוכלוסייה כלשהי מסווגים כלא ידועים. ככל שמתווספים יותר עצמים לאוכלוסיות האמת, דיוק החיזוי משתפר. - המשיכו לאכלס את כיתות מודל האמת הקרקעית בדימויים מתאימים עד שיגיעו למספר מספיק של אובייקטים בכל כיתה.
- לאחר שהוקצו לפחות 100 אובייקטים לכל מחלקת דגם, לחץ על הכרטיסייה אימון בחלק העליון של המסך. לחץ על כפתור הרכבת כדי ליצור מודל באמצעות האלגוריתמים Random Forest ו- CNN.
הערה: תוכנת הבינה המלאכותית יוצרת מודלים באמצעות האלגוריתמים Random Forest ו-CNN, תיבות הסימון מאפשרות יצירת מודלים באמצעות Random Forest של אלגוריתמי CNN בלבד.
5. הערכת דיוק המודל
- לאחר השלמת אימון המודל, לחץ על הצג תוצאות.
- השתמש במסך התוצאות כדי להעריך את דיוק הדגם. השתמש בתפריט הנפתח כדי לעבור בין Random Forest ו- CNN.
הערה: ניתן לעדכן את אוכלוסיות האמת, וניתן לאמן מחדש את המודל או להשתמש בו כפי שהוא כדי לסווג נתונים נוספים.- כדי לעדכן את אוכלוסיות האמת, לחץ על תיוג בחלק העליון ופעל לפי סעיף 4.
6. סיווג נתונים באמצעות המודל
- הפעל את תוכנת ה- AI. מסך ברירת המחדל הוא מסך ניסוי חדש . השתמש בסמל תיקיה כדי לבחור היכן לשמור את הניסוי ולהקליד שם לניסוי.
- תחת סוג ניסוי, לחץ על לחצן האפשרויות לצד סיווג כדי להתחיל ניסוי סיווג. לחץ על הבא.
- לחץ על המודל שישמש לסיווג, ולאחר מכן לחץ על הבא.
- במסך Select Files, לחץ על Add Files ואתר את הקבצים שיש לסווג לפי מודל CNN. לחץ על הבא.
- לאחר מכן, במסך בחירת אוכלוסיות בסיס , לחץ על תיבת הסימון לצד האוכלוסייה הלא-אפופטוטית באחד הקבצים שנטענו. לחץ לחיצה ימנית על האוכלוסייה הלא אפופטוטית ולחץ על בחר את כל האוכלוסיות התואמות כדי לבחור אוכלוסייה זו מכל הקבצים שנטענו. לחץ על הבא.
- אופציונלי: אם הנתונים שיש לסווג מכילים אוכלוסיות אמת, ניתן להקצות אותם למחלקות המודל המתאימות במסך בחירת אוכלוסיות אמת. אחרת, לחץ על הבא כדי לדלג על שלב זה.
- במסך Select Channels , בחר Channel 1 עבור brightfield ו - Channel 7 עבור כתם DNA. לחץ לחיצה ימנית על ערוץ ולחץ על החל על הכל. לאחר מכן לחץ על הבא.
- לבסוף, במסך האישור , לחץ על צור ניסוי. תוכנת הבינה המלאכותית טוענת את המודל שנבחר ואת כל התמונות מקבצי הנתונים שנבחרו. לחץ על סיום.
- לחץ על סיווג כדי להפעיל את מסך הסיווג. לחץ על כפתור סיווג . פעולה זו מתחילה את תהליך השימוש במודל RF ו- CNN כדי לסווג נתונים נוספים ולזהות את כל האובייקטים השייכים למחלקות המודל שצוינו.
הערה: ניתן להשתמש בתיבות הסימון כדי לבחור את דגם RF ו/או את דגם CNN. - לאחר השלמת הסיווג, לחץ על הצג תוצאות.
- לחץ על לחצן עדכן DAFs כדי להציג את החלון עדכן DAFs עם תוצאות סיווג . לחץ על אישור כדי לעדכן את קבצי ה- .daf.
7. הפקת דו"ח של תוצאות הסיווג
- במסך תוצאות , לחץ על צור דוח. בחר בתיבת הסימון לצד צור דוח עבור כל דאף קלט אם נדרש דוח נפרד עבור כל דאף קלט. לחץ על אישור.
- לאחר שתסיים, פתח את התיקיה שבה נשמרו קבצי הדוח. בתוך התיקיה, יש דוח .pdf ניסוי ותיקיה משאבים .
- פתח את .pdf כדי להציג את הדוח. הדוח מכיל מידע על מודלים וניסויים, רשימת קבצי .daf קלט, ספירות המחלקות ואחוזי המחלקה בתבנית טבלאית והיסטוגרמה, ומטריצת בלבול המסכמת את הסתברות החיזוי החציונית בכל קבצי .daf הקלט.
- פתח את התיקיה משאבים ולאחר מכן את התיקיה CNN . בתוך תיקייה זו נמצאים קבצים .png של תרשימי ספירת המחלקות ועמודות האחוזים, כמו גם מטריצת הבלבול. בנוסף, ישנם קבצי .csv המכילים את ספירות המחלקות והאחוזים עבור כל קובץ קלט.
8. קביעת תדירות MN וציטוטוקסיות
- חישוב תדירות MN
- שיטה שאינה Cyt-B: כדי לקבוע תדירות MN, פתח את קובץ class_count.csv משלב 7.4. עבור כל קובץ קלט, חלק את הספירות באוכלוסיית "מונוגרעין עם MN" בספירות באוכלוסייה "מונוגרעין" והכפל ב- 100:

- שיטת Cyt-B: כדי לקבוע את תדירות MN, פתח את קובץ class_count.csv משלב 7.4. עבור כל קובץ קלט, חלק את הספירות באוכלוסיית "Binucleated with MN" בספירות באוכלוסיית "Binucleated" והכפל ב- 100:
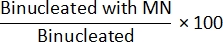
- שיטה שאינה Cyt-B: כדי לקבוע תדירות MN, פתח את קובץ class_count.csv משלב 7.4. עבור כל קובץ קלט, חלק את הספירות באוכלוסיית "מונוגרעין עם MN" בספירות באוכלוסייה "מונוגרעין" והכפל ב- 100:
- חישוב ציטוטוקסיות
- שיטת Non Cyt-B:
- באמצעות ספירת התאים הראשונית וספירת התאים שלאחר הטיפול, חשב תחילה את הכפלת האוכלוסייה (PD) עבור כל מדגם2:
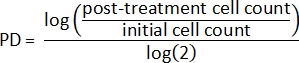
- לאחר מכן, חשב את הכפלת האוכלוסייה היחסית2:

- לבסוף, חשב את ציטוטוקסיות2 עבור כל דגימה:
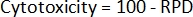
- באמצעות ספירת התאים הראשונית וספירת התאים שלאחר הטיפול, חשב תחילה את הכפלת האוכלוסייה (PD) עבור כל מדגם2:
- שיטת Cyt-B:
- כדי לחשב את Cytokinesis-Block Proliferation Index (CBPI)2, השתמש בספירות במחלקות mononucleated, binucleated, ו polynucleated עבור כל דגימה מקובץ class_count.csv :
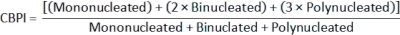
- כדי לחשב ציטוטוקסיות2, השתמש ב- CBPI מתרבויות הביקורת (C) והתרבויות החשופות (T):
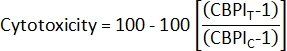
- כדי לחשב את Cytokinesis-Block Proliferation Index (CBPI)2, השתמש בספירות במחלקות mononucleated, binucleated, ו polynucleated עבור כל דגימה מקובץ class_count.csv :
- שיטת Non Cyt-B:
תוצאות
איור 1 מציג את זרימת העבודה לשימוש בתוכנת AI ליצירת מודל עבור מבחן MN. המשתמש טוען את קבצי ה- .daf הרצויים לתוכנת ה- AI, ולאחר מכן מקצה אובייקטים למחלקות מודל האמת הקרקעית באמצעות אשכול בסיוע AI (איור 2) ואלגוריתמי תיוג חיזוי (איור 3). לאחר שכל מחלקות מ...
Discussion
העבודה המוצגת כאן מתארת את השימוש באלגוריתמים של למידה עמוקה כדי להפוך את הניקוד של מבחן MN לאוטומטי. מספר פרסומים אחרונים הראו כי כלים אינטואיטיביים ואינטראקטיביים מאפשרים יצירת מודלים של למידה עמוקה לניתוח נתוני תמונה ללא צורך בידע חישובי מעמיק18,19. הפרוט...
Disclosures
המחברים מועסקים על ידי Luminex Corporation, חברת DiaSorin, יצרנית ציטומטר זרימת הדמיה ImageStream ותוכנת Amnis AI המשמשת בעבודה זו.
Acknowledgements
ללא.
Materials
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
References
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20 (2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324 (2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved