Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Görüntüleme Akış Sitometrisi ve Yapay Zeka Kullanılarak Mikronükleus Testinin Otomasyonu
Bu Makalede
Özet
Mikroçekirdek (MN) testi, DNA hasarını ölçmek için iyi kurulmuş bir testtir. Bununla birlikte, manuel mikroskopi veya özellik tabanlı görüntü analizi gibi geleneksel teknikleri kullanarak tahlilin puanlanması zahmetli ve zordur. Bu makalede, görüntüleme akışı sitometri verilerini kullanarak MN testini puanlamak için bir yapay zeka modeli geliştirme metodolojisi açıklanmaktadır.
Özet
Mikroçekirdek (MN) testi, genetik toksisite için kimyasalları değerlendirmek için dünya çapında düzenleyici kurumlar tarafından kullanılmaktadır. Tahlil iki şekilde gerçekleştirilebilir: MN'yi bir kez bölünmüş, sitokinez bloke edilmiş binüklee hücrelerde veya tamamen bölünmüş monoçekirdekli hücrelerde puanlayarak. Tarihsel olarak, ışık mikroskobu testi puanlamak için altın standart yöntem olmuştur, ancak zahmetli ve özneldir. Akış sitometrisi son yıllarda testi puanlamak için kullanılmıştır, ancak hücresel görüntülerin temel yönlerini görsel olarak doğrulayamama ile sınırlıdır. Görüntüleme akışı sitometrisi (IFC), yüksek verimli görüntü yakalama ve otomatik görüntü analizini birleştirir ve MN testindeki tüm önemli olayların görüntülerini hızlı bir şekilde elde etmek ve puanlamak için başarıyla uygulanmıştır. Son zamanlarda, IFC tarafından elde edilen MN tahlil verilerini puanlamak için evrişimli sinir ağlarına dayanan yapay zeka (AI) yöntemlerinin kullanılabileceği gösterilmiştir. Bu makalede, tüm önemli olayları puanlamak üzere bir derin öğrenme modeli oluşturmak ve ek verileri otomatik olarak puanlamak için bu modeli uygulamak üzere yapay zeka yazılımını kullanmaya yönelik tüm adımlar açıklanmaktadır. AI derin öğrenme modelinden elde edilen sonuçlar, manuel mikroskopi ile iyi bir şekilde karşılaştırılır, bu nedenle IFC ve AI'yı birleştirerek MN testinin tam otomatik puanlamasını sağlar.
Giriş
Mikronükleus (MN) testi, genetik toksikolojide, insan kullanımı için kozmetik, ilaç ve kimyasalların geliştirilmesinde DNA hasarını değerlendirmek için temeldir 1,2,3,4. Mikroçekirdekler, bölünmeyi takiben çekirdeğe dahil olmayan ve çekirdekten ayrı küçük, dairesel cisimler halinde yoğunlaşan tüm kromozomlardan veya kromozom parçalarından oluşur. Bu nedenle, MN, genotoksisite testi1'de DNA hasarını ölçmek için bir son nokta olarak kullanılabilir.
MN'yi ölçmek için tercih edilen yöntem, Sitokalasin-B (Cyt-B) kullanarak bölünmeyi bloke ederek bir kez bölünmüş binüklee hücreler (BNC'ler) içindedir. Tahlilin bu versiyonunda, sitotoksisite, monoçekirdekli (MONO) ve poliçekirdekli (POLY) hücrelerin puanlanmasıyla da değerlendirilir. Tahlil ayrıca, bloke edilmemiş MONO hücrelerinde MN'nin puanlanmasıyla da yapılabilir, bu da daha hızlı ve skorlanması daha kolaydır, sitotoksisite proliferasyonu değerlendirmek için maruziyet öncesi ve sonrası hücre sayıları kullanılarak değerlendirilir 5,6.
Tahlilin fiziksel puanlaması tarihsel olarak manuel mikroskopi ile gerçekleştirilmiştir, çünkü bu tüm önemli olayların görsel olarak doğrulanmasına izin verir. Bununla birlikte, manuel mikroskopi zorlu ve özneldir1. Böylece, mikroskop slayt taraması ve akış sitometrisi de dahil olmak üzere, her biri kendi avantajlarına ve sınırlamalarına sahip otomatik teknikler geliştirilmiştir. Slayt tarama yöntemleri önemli olayların görselleştirilmesine izin verirken, slaytlar optimum hücre yoğunluğunda oluşturulmalıdır, bu da elde edilmesi zor olabilir. Ek olarak, bu teknik genellikle MONO ve POLY hücrelerinin puanlamasını tehlikeye atabilecek sitoplazmik görselleştirmeden yoksundur 7,8. Akış sitometrisi yüksek verimli veri yakalama sunarken, hücreler lize edilmelidir, böylece tahlilin Cyt-B formunun kullanılmasına izin verilmez. Ek olarak, görüntüleme dışı bir teknik olarak, geleneksel akış sitometrisi anahtar olayların görsel doğrulamasını sağlamaz9,10.
Bu nedenle, MN testini gerçekleştirmek için görüntüleme akım sitometrisi (IFC) araştırılmıştır. ImageStreamX Mk II, geleneksel akış sitometrisinin hızını ve istatistiksel sağlamlığını mikroskopinin yüksek çözünürlüklü görüntüleme yetenekleriyle tek bir sistemde birleştirir11. IFC kullanılarak, tüm önemli olayların yüksek çözünürlüklü görüntülerinin yakalanabileceği ve özellik tabanlı 12,13 veya yapay zeka (AI) teknikleri14,15 kullanılarak otomatik olarak puanlanabileceği gösterilmiştir. MN testini gerçekleştirmek için IFC'yi kullanarak, mikroskopiye kıyasla çok daha fazla hücrenin daha kısa sürede otomatik skorlaması elde edilebilir.
Bu çalışma, daha önce açıklanan bir görüntü analizi iş akışı16'dan sapmaktadır ve Amnis AI yazılımını (bundan böyle "AI yazılımı" olarak anılacaktır) kullanarak bir Rastgele Orman (RF) ve / veya evrişimli sinir ağı (CNN) modeli geliştirmek ve eğitmek için gereken tüm adımları tartışmaktadır. AI destekli etiketleme araçlarını kullanarak zemin gerçeği verilerinin doldurulması, model eğitim sonuçlarının yorumlanması ve modelin ek verileri sınıflandırmak için uygulanması, genotoksisite ve sitotoksisitenin hesaplanmasına izin verilmesi dahil olmak üzere gerekli tüm adımlar açıklanmaktadır15.
Protokol
1. Görüntüleme akış sitometrisi kullanılarak veri toplama
NOT: Aşağıdaki değişikliklerle Rodrigues ve ark.16'ya bakın ve IFC kullanan edinme bölgelerinin optimum görüntü yakalama için değiştirilmesi gerekebileceğini unutmayın:
- Cyt-B olmayan yöntem için, kültürden hemen önce ve iyileşme döneminden hemen sonra her kültürde üreticinin talimatlarını (bkz. Malzeme Tablosu) izleyerek ticari olarak temin edilebilen bir hücre sayacı kullanarak bir hücre sayımı gerçekleştirin.
- Örnekleri tek kameralı görüntüleme akışı sitometresinde çalıştırıyorsanız, Parlak Alan'ı (BF) Kanal 4'e yerleştirin. M01'i M04 ve M07'yi M01 ile değiştirin.
NOT: "M", IFC'deki kamera kanalını ifade eder. - Edinme sırasında 40x büyütmeyi kullanın.
- Edinme sırasında BF alanı ve BF en boy oranı grafiğinde, aşağıdaki bölge koordinatlarını kullanın:
X-koordinatları: 100 ve 900; Y-koordinatları: 0.7 ve 1 (Cyt-B yöntemi)
X-koordinatları: 100 ve 600; Y-koordinatları: 0.7 ve 1 - Hoechst yoğunluk grafiğinde, aşağıdaki bölge koordinatlarını kullanın:
X-koordinatları: 55 ve 75; Y-koordinatları: 9.5 ve 15 (Cyt-B yöntemi)
X-koordinatları: 55 ve 75; Y-koordinatları: 13 ve 21 (Cyt-B olmayan yöntem) - Apoptotik ve nekrotik nesnelerin görüntülerini verilerden kaldırmak için IDEAS 6.3 yazılım paketini başlatın (bundan böyle "görüntü analiz yazılımı" olarak anılacaktır; bkz.
NOT: AI yazılımı, görüntü analiz yazılımının en son sürümü kullanılarak işlenen .daf dosyalarıyla çalışacak şekilde tasarlanmıştır. Görüntü analiz yazılımının güncel olduğundan emin olun. - Bu çalışmayı şablon (.ast) dosyası olarak kaydedin.
2. Tüm .rif dosyaları için .daf dosyaları oluşturma
- AI yazılımı yalnızca .daf dosyalarının içe aktarılmasına izin verir. Toplu işleme yoluyla denemedeki tüm .rif dosyaları için .daf dosyaları oluşturun.
- Araçlar menüsü altında, Toplu Veri Dosyaları'na ve ardından Toplu Ekle'ye tıklayın.
- Yeni pencerede, Dosya Ekle'yi seçin ve toplu işe eklenecek .rif dosyalarını seçin. Bir Şablon veya Veri Çözümleme Dosyası (.ast, .daf) Seçin seçeneğinin altında, daha önce oluşturulmuş .ast dosyasını seçin.
- Gerekirse bir toplu iş adı atayın ve yüklenen tüm .rif dosyaları için .daf dosyaları oluşturmak üzere Tamam'a tıklayın.
3. AI yazılımında bir deneme oluşturma
- AI yazılımını kullanarak derin öğrenme modeli oluşturma sürecini açıklayan Şekil 1'deki akış şemasına bakın.
- AI yazılımını başlatın ve pencerenin sol alt köşesindeki Hakkında'ya tıklayarak en son sürümün yüklendiğinden emin olun. En son sürüm yüklü değilse, edinmek için support@luminexcorp.com başvurun.
- Yazılımdaki varsayılan ekran Yeni Deneme ekranıdır. Denemenin nereye kaydedileceğini seçmek için Klasör simgesini kullanın ve deneme için bir ad yazın (ör. "MN modeli").
- Deneme Türü altında, CNN modelini oluşturmaya başlamak üzere bir eğitim denemesi başlatmak için Eğit'in yanındaki radyo düğmesini tıklatın. İleri'ye tıklayın.
- İsteğe bağlı: Bir model daha önce eğitilmişse, yeni bir AI modeli için şablon olarak kullanılabilir ve Şablon Modeli Seç ekranından yeni bir modelin oluşturulması için şablon olarak seçilebilir. Şablon modeli yoksa, İleri'yi tıklatarak bu adımı atlamanız yeterlidir.
- Bir sonraki ekran Yeni Model Tanımla ekranıdır. Model altında, adım 3.3'te modele verilen ad otomatik olarak doldurulur.
- Açıklama altında, model için bir açıklama yazın (isteğe bağlı) ve maksimum görüntü boyutunu 150 pikselde bırakın.
- Kanallar altında, listeye bir brightfield kanalı eklemek için BF Ekle'ye tıklayın. Ad altında, Brightfield'a çift tıklayın ve bu kanalı BF olarak yeniden adlandırın. Listeye floresan kanal eklemek için FL Ekle'ye tıklayın. Ad altında, Floresan'a çift tıklayın ve bu kanalı DNA olarak yeniden adlandırın.
- Sınıf Adları altında, Ekle'ye tıklayın. Açılır pencerede, Mononucleated yazın ve Tamam'a tıklayın. Bu, monoçekirdekli sınıfı sınıf adları listesine ekler. Aşağıdaki altı sınıfın listede tanımlandığından emin olmak için bu işlemi yineleyin:
Monoçekirdekli
MN ile mononüklee edilmiş
Binükleasyonlu
MN ile binüklelenmiş
Poliçekirdekli
Düzensiz morfoloji
İleri'ye tıklayın.
NOT: Bu altı temel doğruluk modeli sınıfı, puanlanacak temel olayları ve kabul edilen puanlama kriterleri5'ten farklı morfolojiye sahip görüntüleri temsil edecektir.- İsteğe bağlı: İstenirse, görüntü analiz yazılımındaki özellikleri kullanmak için 1.7'den analiz şablonu eklenebilir. Bu özellikleri yapay zeka modeline dahil etmek istiyorsanız .ast dosyasına göz atın, ardından kanala özgü açılır menülerden dahil etmek istediğiniz özellik alt kümelerini seçin.
- Dosya Seç altında, Dosya Ekle'ye tıklayın ve temel doğruluk verilerini oluşturmak için AI yazılımına eklenecek istenen dosyalara göz atın. İleri'ye tıklayın.
NOT: Tüm önemli olayların yeterli sayıda içeriğini içeren birden çok veri dosyası (örneğin, pozitif ve negatif kontrol verileri) eklemek önemlidir.
- Ardından, Temel Popülasyonları Seç ekranında, popülasyon hiyerarşisinden Apoptotik Olmayan popülasyonu bulun. Apoptotik Olmayan popülasyona sağ tıklayın ve Tüm Eşleşen Popülasyonları Seç'i seçin. İleri'ye tıklayın.
NOT: Sınıflandırılmaması gereken popülasyonları hariç tutmak önemlidir (örneğin, boncuklar, döküntüler, çiftler, vb.) - Bu ekran, Doğruluk Popülasyonlarını Seç ekranıdır.
- Önemli olayların etiketlenmiş gerçek popülasyonları görüntü analiz yazılımında oluşturulmamışsa, İleri'ye tıklayın.
- Görüntü analizi yazılımında etiketlenmiş doğruluk popülasyonları oluşturulmuşsa, bunları uygun model sınıfına atayın.
- MN ile MONO hücrelerinin etiketli bir doğruluk popülasyonunu atamak için, soldaki Model Sınıfları altındaki MN ile Monoçekirdekli sınıfına tıklayın. Ardından, bu olayları içeren sağdaki uygun etiketli gerçek popülasyonuna tıklayın.
- Etiketlenmiş doğruluk popülasyonları birden fazla veri dosyasında oluşturulmuşsa, doğruluk popülasyonlarından birine sağ tıklayın ve birden çok dosyadan etiketli popülasyonları uygun sınıfa eklemek için Tüm Eşleşen Popülasyonları Seç'i seçin.
- Tüm uygun gerçek popülasyonları atandıktan sonra, İleri'ye tıklayın.
- Kanal Seç ekranında, deneme için uygun kanalları seçin. Burada, BF'yi kanal 1'e ve Hoechst'i kanal 7'ye ayarlayın. Bir kanala sağ tıklayın ve Tümüne Uygula'yı seçin. İleri'ye tıklayın.
- Son olarak, Onay ekranında Deneme Oluştur'a tıklayın.
- AI yazılımı, veri dosyalarından görüntüleri yükler ve adım 3.7'de atanan temel doğruluk görüntüleriyle adım 3.5.3'te tanımlanan model sınıflarını oluşturur. Son'a tıklayın.
- Deneme oluşturulduktan sonra beş seçenek sunulur:
Deneme: Yüklenen veri dosyaları, seçilen kanallar ve tanımlanmış temel doğruluk modeli sınıfları dahil olmak üzere denemenin ayrıntılarını sağlar.
Etiketleme: Kullanıcıların zemin doğruluğu verilerini doldurabileceği etiketleme aracını başlatır.
Eğitim: Temel doğruluk verilerine dayalı bir modeli eğitir.
Sınıflandır: Verileri sınıflandırmak için eğitilmiş modelleri kullanır.
Sonuçlar: Hem eğitim denemesinden hem de sınıflandırma deneyinden elde edilen sonuçları sağlar.
4. AI destekli etiketleme araçlarını kullanarak temel doğruluk verilerini doldurma
- Etiketleme aracı arayüzünü başlatmak için Etiketleme'ye tıklayın.
- Daha kolay görüntüleme için görüntüleri kırpmak üzere yakınlaştırma araçlarına (büyüteç simgeleri) tıklayın.
- Galeride kaç resmin gösterileceğini değiştirmek üzere görüntü boyutunu ayarlamak için kaydırma çubuğuna tıklayın.
- Görüntü Ayarı seçeneğine tıklayın ve tüm önemli olayları tanımlamak için en iyi kontrast görüntüsünü sağlayan Min-Max'i seçin.
- DNA görüntüsünün rengini sarı veya beyaza değiştirmek için Galeri Ekranını Ayarla'ya tıklayın, bu da küçük nesnelerin (örneğin, MN) görselleştirilmesini iyileştirecektir.
- Benzer morfolojiye sahip nesneleri birlikte gruplandırmak üzere algoritmayı çalıştırmak için Küme'ye tıklayın. Kümeleme tamamlandıktan sonra, küme başına nesne sayısına sahip tek tek kümeler Bilinmeyen Popülasyonlar altındaki bir listede gösterilir. Küme içindeki nesneleri görüntülemek için tek tek kümeleri seçin ve bu nesneleri uygun model sınıflarına atayın.
- Her model sınıfına en az 25 nesne atandıktan sonra, Tahmin algoritması kullanılabilir hale gelir. Tahmin'e tıklayın.
NOT: Herhangi bir popülasyona tam olarak uymayan nesneler Bilinmeyen olarak sınıflandırılır. Gerçek popülasyonlarına daha fazla nesne eklendikçe, tahmin doğruluğu artar. - Her sınıftaki yeterli sayıda nesneye ulaşılana kadar temel doğruluk modeli sınıflarını uygun görüntülerle doldurmaya devam edin.
- Her model sınıfına en az 100 nesne atandıktan sonra, ekranın üst kısmındaki Eğitim sekmesine tıklayın. Rastgele Orman ve CNN algoritmalarını kullanarak bir model oluşturmak için Eğit düğmesine tıklayın.
NOT: AI yazılımı hem Rastgele Orman hem de CNN algoritmalarını kullanarak modeller oluşturur, onay kutuları yalnızca CNN algoritmalarının Rastgele Ormanını kullanarak modellerin oluşturulmasına izin verir.
5. Model doğruluğunu değerlendirme
- Model eğitimi tamamlandıktan sonra Sonuçları Görüntüle'ye tıklayın.
- Model doğruluğunu değerlendirmek için sonuçlar ekranını kullanın. Random Forest ve CNN arasında geçiş yapmak için açılır menüyü kullanın.
NOT: Gerçek popülasyonlar güncellenebilir ve model yeniden eğitilebilir veya ek verileri sınıflandırmak için olduğu gibi kullanılabilir.- Gerçek popülasyonlarını güncellemek için, en üstteki Etiketleme'ye tıklayın ve bölüm 4'ü izleyin.
6. Modeli kullanarak verileri sınıflandırma
- AI yazılımını başlatın. Varsayılan ekran Yeni Deneme ekranıdır. Denemenin nereye kaydedileceğini seçmek ve deneme için bir ad yazmak üzere Klasör simgesini kullanın.
- Deneme Türü altında, bir sınıflandırma denemesi başlatmak için Sınıflandır'ın yanındaki radyo düğmesini tıklayın. İleri'ye tıklayın.
- Sınıflandırma için kullanılacak modele tıklayın, ardından İleri'ye tıklayın.
- Dosya Seç ekranında, Dosya Ekle'ye tıklayın ve CNN modeline göre sınıflandırılacak dosyalara göz atın. İleri'ye tıklayın.
- Ardından, Temel Popülasyonları Seç ekranında, yüklenen dosyalardan birinde Apoptotik Olmayan popülasyonun yanındaki onay kutusuna tıklayın. Apoptotik olmayan popülasyona sağ tıklayın ve yüklenen tüm dosyalardan bu popülasyonu seçmek için Tüm Eşleşen Popülasyonları Seç'e tıklayın. İleri'ye tıklayın.
- İsteğe bağlı: Sınıflandırılacak veriler doğruluk popülasyonları içeriyorsa, Doğruluk Popülasyonlarını Seç ekranındaki uygun model sınıflarına atanabilir. Aksi takdirde, bu adımı atlamak için İleri'yi tıklatın.
- Kanal Seç ekranında, parlak alan için Kanal 1'i ve DNA lekesi için Kanal 7'yi seçin. Bir kanala sağ tıklayın ve Tümüne Uygula'ya tıklayın. Ardından İleri'ye tıklayın.
- Son olarak, Onay ekranında Deneme Oluştur'a tıklayın. AI yazılımı, seçilen modeli ve seçilen veri dosyalarından tüm görüntüleri yükler. Son'a tıklayın.
- Sınıflandırma ekranını başlatmak için Classify'a tıklayın. SınıflandırEt düğmesine tıklayın. Bu, ek verileri sınıflandırmak ve belirtilen model sınıflarına ait tüm nesneleri tanımlamak için RF ve CNN modelini kullanma işlemini başlatır.
NOT: Onay kutuları RF modelini ve/veya CNN modelini seçmek için kullanılabilir. - Sınıflandırma tamamlandıktan sonra, Sonuçları Görüntüle'ye tıklayın.
- DAF'ları Sınıflandırma Sonuçlarıyla Güncelleştir penceresini açmak için DAF'ları Güncelleştir düğmesini tıklatın. .daf dosyalarını güncellemek için Tamam'a tıklayın.
7. Sınıflandırma sonuçlarının raporunun oluşturulması
- Sonuçlar ekranında, Rapor Oluştur'a tıklayın. Her giriş daf'ı için ayrı bir rapor gerekiyorsa, Her Giriş DAF'ı için Rapor Oluştur'un yanındaki onay kutusunu seçin. Tamam'a tıklayın.
- Tamamlandığında, rapor dosyalarının kaydedildiği klasörü açın. Klasörün içinde bir deneme .pdf raporu ve bir Kaynaklar klasörü vardır.
- Raporu görüntülemek için .pdf açın. Rapor, model ve deney bilgilerini, giriş .daf dosyalarının listesini, tablo ve histogram biçimindeki sınıf sayılarını ve sınıf yüzdelerini ve tüm giriş .daf dosyalarındaki medyan tahmin olasılığını özetleyen bir karışıklık matrisini içerir.
- Kaynaklar klasörünü ve ardından CNN klasörünü açın. Bu klasörde, sınıf sayısı ve yüzde çubuk grafiklerinin yanı sıra karışıklık matrisinin .png dosyaları bulunur. Ayrıca, her giriş dosyası için sınıf sayılarını ve yüzdelerini içeren .csv dosya vardır.
8. MN sıklığının ve sitotoksisitesinin belirlenmesi
- MN frekansını hesaplama
- Non Cyt-B yöntemi: MN frekansını belirlemek için, adım 7.4'teki class_count.csv dosyasını açın. Her giriş dosyası için, "MN ile Mononükleated" popülasyonundaki sayıları "Mononucleated" popülasyonundaki sayımlara bölün ve 100 ile çarpın:

- Cyt-B yöntemi: MN frekansını belirlemek için, adım 7.4'teki class_count.csv dosyasını açın. Her giriş dosyası için, "Binükleated with MN" popülasyonundaki sayımları "Binucleated" popülasyonundaki sayımlara bölün ve 100 ile çarpın:
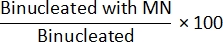
- Non Cyt-B yöntemi: MN frekansını belirlemek için, adım 7.4'teki class_count.csv dosyasını açın. Her giriş dosyası için, "MN ile Mononükleated" popülasyonundaki sayıları "Mononucleated" popülasyonundaki sayımlara bölün ve 100 ile çarpın:
- Sitotoksisitenin hesaplanması
- Non Cyt-B yöntemi:
- İlk hücre sayımlarını ve tedavi sonrası hücre sayımlarını kullanarak, önce her örnek2 için popülasyonun iki katına çıkmasını (PD) hesaplayın:
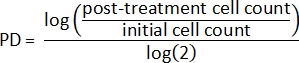
- Ardından,2'yi ikiye katlayan göreceli nüfusu hesaplayın:

- Son olarak, her numune için sitotoksisite2'yi hesaplayın:
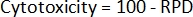
- İlk hücre sayımlarını ve tedavi sonrası hücre sayımlarını kullanarak, önce her örnek2 için popülasyonun iki katına çıkmasını (PD) hesaplayın:
- Cyt-B yöntemi:
- Sitokinezis-Blok Proliferasyon İndeksi (CBPI)2'yi hesaplamak için, class_count.csv dosyasındaki her örnek için mononüklee, binüklee ve poliçekirdekli sınıflardaki sayıları kullanın:
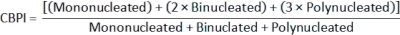
- Sitotoksisite2'yi hesaplamak için, CBPI'yi kontrol kültürlerinden (C) ve maruz kalan kültürlerden (T) kullanın:
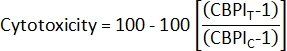
- Sitokinezis-Blok Proliferasyon İndeksi (CBPI)2'yi hesaplamak için, class_count.csv dosyasındaki her örnek için mononüklee, binüklee ve poliçekirdekli sınıflardaki sayıları kullanın:
- Non Cyt-B yöntemi:
Sonuçlar
Şekil 1, MN testi için bir model oluşturmak üzere AI yazılımını kullanmaya yönelik iş akışını göstermektedir. Kullanıcı istenen .daf dosyalarını AI yazılımına yükler, ardından AI destekli küme (Şekil 2) ve tahmin (Şekil 3) etiketleme algoritmalarını kullanarak nesneleri temel doğruluk modeli sınıflarına atar. Tüm temel doğruluk modeli sınıfları yeterli nesnelerle doldurulduktan sonra, model RF v...
Tartışmalar
Burada sunulan çalışma, MN testinin puanlamasını otomatikleştirmek için derin öğrenme algoritmalarının kullanımını açıklamaktadır. Son zamanlarda yapılan birkaç yayın, sezgisel, etkileşimli araçların, derinlemesine hesaplama bilgisine ihtiyaç duymadan görüntü verilerini analiz etmek için derin öğrenme modellerinin oluşturulmasına izin verdiğini göstermiştir18,19. Kullanıcı arayüzü güdümlü bir yazılım paketi kullanılara...
Açıklamalar
Yazarlar, bir DiaSorin Şirketi olan Luminex Corporation, ImageStream görüntüleme akışı sitometresinin üreticisi ve bu çalışmada kullanılan Amnis AI yazılımı tarafından istihdam edilmektedir.
Teşekkürler
Hiç kimse.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
Referanslar
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20 (2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324 (2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır