É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Automação do Ensaio de Micronúcleos Utilizando Imagem por Citometria de Fluxo e Inteligência Artificial
Neste Artigo
Resumo
O ensaio de micronúcleo (MN) é um teste bem estabelecido para quantificar danos ao DNA. No entanto, pontuar o ensaio usando técnicas convencionais, como microscopia manual ou análise de imagem baseada em recursos, é trabalhoso e desafiador. Este artigo descreve a metodologia para desenvolver um modelo de inteligência artificial para pontuar o ensaio de MN usando dados de citometria de fluxo de imagem.
Resumo
O ensaio de micronúcleo (MN) é usado mundialmente por órgãos reguladores para avaliar produtos químicos quanto à toxicidade genética. O ensaio pode ser realizado de duas maneiras: pontuando MN em células binucleadas bloqueadas por citocinese ou células mononucleadas totalmente divididas. Historicamente, a microscopia óptica tem sido o método padrão-ouro para pontuar o ensaio, mas é trabalhosa e subjetiva. A citometria de fluxo tem sido usada nos últimos anos para pontuar o ensaio, mas é limitada pela incapacidade de confirmar visualmente aspectos-chave da imagética celular. A citometria de fluxo por imagem (IFC) combina captura de imagem de alto rendimento e análise automatizada de imagens, e tem sido aplicada com sucesso para adquirir rapidamente imagens e pontuar todos os eventos-chave no ensaio de MN. Recentemente, foi demonstrado que métodos de inteligência artificial (IA) baseados em redes neurais convolucionais podem ser usados para pontuar dados de ensaios de MN adquiridos por IFC. Este documento descreve todas as etapas para usar o software de IA para criar um modelo de aprendizado profundo para pontuar todos os eventos-chave e aplicar esse modelo para pontuar automaticamente dados adicionais. Os resultados do modelo de aprendizagem profunda de IA se comparam bem à microscopia manual, permitindo assim a pontuação totalmente automatizada do ensaio MN combinando IFC e IA.
Introdução
O ensaio de micronúcleos (NM) é fundamental na toxicologia genética para avaliar danos ao DNA no desenvolvimento de cosméticos, fármacos e produtos químicos para uso humano 1,2,3,4. Os micronúcleos são formados a partir de cromossomos inteiros ou fragmentos cromossômicos que não se incorporam ao núcleo após a divisão e se condensam em pequenos corpos circulares separados do núcleo. Assim, o NM pode ser usado como um desfecho para quantificar danos ao DNA em testes de genotoxicidade1.
O método preferido para quantificar MN é dentro de células binucleadas (BNCs) divididas uma vez por bloqueio de divisão usando Citocalasina-B (Cyt-B). Nesta versão do ensaio, a citotoxicidade também é avaliada por meio da pontuação de células mononucleadas (MONO) e polinucleadas (POLY). O ensaio também pode ser realizado por meio da pontuação de MN em células MONO desbloqueadas, o que é mais rápido e fácil de pontuar, com a citotoxicidade sendo avaliada usando contagens de células pré e pós-exposição para avaliar a proliferação 5,6.
A pontuação física do ensaio tem sido historicamente realizada por microscopia manual, uma vez que permite a confirmação visual de todos os eventos-chave. Entretanto, a microscopia manual é desafiadora esubjetiva1. Assim, técnicas automatizadas têm sido desenvolvidas, incluindo varredura de lâminas ao microscópio e citometria de fluxo, cada uma com suas vantagens e limitações. Embora os métodos de varredura de slides permitam que eventos-chave sejam visualizados, os slides devem ser criados na densidade de célula ideal, o que pode ser difícil de alcançar. Além disso, essa técnica muitas vezes não tem visualização citoplasmática, o que pode comprometer a pontuação das células MONO e POLY 7,8. Embora a citometria de fluxo ofereça captura de dados de alto rendimento, as células devem ser lisadas, não permitindo o uso da forma Cyt-B do ensaio. Além disso, por ser uma técnica não imaginológica, a citometria de fluxo convencional não fornece validação visual de eventos-chave 9,10.
Portanto, a citometria de fluxo por imagem (CIF) tem sido investigada para realizar o ensaio de NM. O ImageStreamX Mk II combina a velocidade e a robustez estatística da citometria de fluxo convencional com os recursos de imagem de alta resolução da microscopia em um único sistema11. Foi demonstrado que, usando IFC, imagens de alta resolução de todos os eventos-chave podem ser capturadas e pontuadas automaticamente usando técnicas baseadas em recursos 12,13 ou inteligência artificial (IA) 14,15. Usando IFC para realizar o ensaio de MN, a pontuação automática de muito mais células em comparação com a microscopia em um período mais curto de tempo é alcançável.
Este trabalho se desvia de um fluxo de trabalho de análise de imagens descrito anteriormente16 e discute todas as etapas necessárias para desenvolver e treinar um modelo de Floresta Aleatória (RF) e/ou rede neural convolucional (CNN) usando o software Amnis AI (doravante referido como "software de IA"). Todas as etapas necessárias são descritas, incluindo o preenchimento de dados de verdade terrestres usando ferramentas de marcação assistidas por IA, interpretação dos resultados do treinamento do modelo e aplicação do modelo para classificar dados adicionais, permitindo o cálculo de genotoxicidade e citotoxicidade15.
Protocolo
1. Aquisição de dados por citometria de fluxo por imagem
NOTA: Consulte Rodrigues et al.16 com as seguintes modificações, observando que as regiões de aquisição usando IFC podem precisar ser modificadas para a captura ideal da imagem:
- Para o método não-Cyt-B, realizar uma contagem de células usando um contador de células disponível comercialmente seguindo as instruções do fabricante (ver Tabela de Materiais) em cada cultura imediatamente antes da cultura e imediatamente após o período de recuperação.
- Se estiver executando amostras em um citômetro de fluxo de imagem de câmera única, coloque o Brightfield (BF) no Canal 4. Substitua M01 por M04 e M07 por M01.
NOTA: "M" refere-se ao canal da câmera no IFC. - Use a ampliação de 40x durante a aquisição.
- No gráfico de proporção área BF versus BF durante a aquisição, use as seguintes coordenadas de região:
Coordenadas X: 100 e 900; Coordenadas Y: 0,7 e 1 (método Cyt-B)
Coordenadas X: 100 e 600; Coordenadas Y: 0,7 e 1 - No gráfico de intensidade Hoechst, use as seguintes coordenadas de região:
Coordenadas X: 55 e 75; Coordenadas Y: 9,5 e 15 (método Cyt-B)
Coordenadas X: 55 e 75; Coordenadas Y: 13 e 21 (método não-Cyt-B) - Para remover imagens de objetos apoptóticos e necróticos dos dados, inicie o pacote de software IDEAS 6.3 (doravante denominado "software de análise de imagens"; ver Tabela de Materiais).
NOTA: O software AI foi projetado para trabalhar com arquivos .daf que foram processados usando a versão mais recente do software de análise de imagem. Certifique-se de que o software de análise de imagens esteja atualizado. - Salve este trabalho como um arquivo de modelo (.ast).
2. Criando arquivos .daf para todos os arquivos .rif
- O software de IA só permite importar arquivos .daf. Crie arquivos .daf para todos os arquivos .rif no experimento por meio do processamento em lote.
- No menu Ferramentas , clique em Arquivos de dados em lote e, em seguida, clique em Adicionar lote.
- Na nova janela, selecione Adicionar arquivos e selecione os arquivos .rif a serem adicionados ao lote. Na opção Selecionar um modelo ou arquivo de análise de dados (.ast, .daf), selecione o arquivo .ast criado anteriormente.
- Atribua um nome de lote, se necessário, e clique em OK para criar arquivos .daf para todos os arquivos .rif carregados.
3. Criando um experimento no software de IA
- Consulte o fluxograma na Figura 1 que descreve o processo de criação de um modelo de aprendizado profundo usando o software de IA.
- Inicie o software de IA e certifique-se de que a versão mais recente está instalada clicando em Sobre no canto inferior esquerdo da janela. Se a versão mais recente não estiver instalada, entre em contato com support@luminexcorp.com para obtê-la.
- A tela padrão no software é a tela Novo Experimento . Use o ícone Pasta para escolher onde salvar o experimento e digite um nome para o experimento (por exemplo, "modelo MN").
- Em Tipo de experimento, clique no botão de opção ao lado de Treinar para iniciar um experimento de treinamento para começar a criar o modelo da CNN. Clique em Avançar.
- Opcional: se um modelo tiver sido treinado anteriormente, ele poderá ser usado como um modelo para um novo modelo de IA e poderá ser selecionado como um modelo para a criação de um novo modelo na tela Selecionar modelo de modelo. Se nenhum modelo de modelo existir, simplesmente ignore esta etapa clicando em Avançar.
- A próxima tela é a tela Definir Novo Modelo . Em Modelo, o nome que foi dado ao modelo na etapa 3.3 será preenchido automaticamente.
- Em Descrição, digite uma descrição para o modelo (opcional) e deixe o tamanho máximo da imagem em 150 pixels.
- Em Canais, clique em Adicionar BF para adicionar um canal de campo brilhante à lista. Em Nome, clique duas vezes em Brightfield e renomeie este canal para BF. Clique em Adicionar FL para adicionar um canal fluorescente à lista. Em Nome, clique duas vezes em Fluorescente e renomeie esse canal para DNA.
- Em Nomes de classe, clique em Adicionar. Na janela pop-up, digite Mononucleado e clique em OK. Isso adiciona a classe mononucleada à lista de nomes de classe. Repita esse processo para garantir que as seis classes a seguir sejam definidas na lista:
Mononucleado
Mononucleado com MN
Binucleado
Binucleado com MN
Polinucleado
Morfologia irregular
Clique em Avançar.
NOTA: Essas seis classes do modelo verdade fundamental representarão os eventos-chave a serem pontuados, bem como imagens com morfologia diferente dos critérios de pontuação aceitos5.- Opcional: Se desejar, o modelo de análise da versão 1.7 pode ser incluído para usar recursos do software de análise de imagem. Se você quiser incluir esses recursos no modelo de IA, procure o arquivo .ast e, em seguida, nas listas suspensas específicas do canal, escolha os subconjuntos de recursos que deseja incluir.
- Em Selecionar arquivos, clique em Adicionar arquivos e procure os arquivos desejados a serem adicionados ao software de IA para construir os dados de verdade de base. Clique em Avançar.
Observação : é importante adicionar vários arquivos de dados (por exemplo, dados de controle positivo e negativo) que contêm um número suficiente de todos os eventos chave.
- Em seguida, na tela Selecionar Populações Base , localize a população Não-Apoptótica da hierarquia populacional. Clique com o botão direito do mouse na população não-apoptótica e selecione Selecionar todas as populações correspondentes. Clique em Avançar.
NOTA: É importante excluir quaisquer populações que não devam ser classificadas (por exemplo, contas, detritos, duplos, etc.) - Esta tela é a tela Selecionar Populações da Verdade .
- Se as populações de verdade marcadas dos eventos-chave não tiverem sido criadas no software de análise de imagem, clique em Avançar.
- Se as populações de verdade marcadas tiverem sido criadas no software de análise de imagens, atribua-as à classe de modelo apropriada.
- Para atribuir uma população verdade marcada de células MONO com MN, clique na classe Mononucleada com MN em Classes de Modelo à esquerda. Em seguida, clique na população de verdade marcada apropriada à direita que contém esses eventos.
- Se as populações de verdade marcadas tiverem sido criadas em mais de um arquivo de dados, clique com o botão direito do mouse em uma das populações de verdade e selecione Selecionar todas as populações correspondentes para adicionar populações marcadas de vários arquivos à classe apropriada.
- Depois que todas as populações de verdade apropriadas tiverem sido atribuídas, clique em Avançar.
- Na tela Selecionar canais, escolha os canais apropriados para o experimento. Aqui, defina BF para o canal 1 e Hoechst para o canal 7. Clique com o botão direito do mouse em um canal e selecione Aplicar a Todos. Clique em Avançar.
- Por fim, na tela de confirmação , clique em Criar experimento.
- O software de IA carrega imagens dos arquivos de dados e cria as classes de modelo definidas na etapa 3.5.3 com as imagens de verdade básicas que foram atribuídas na etapa 3.7. Clique em Concluir.
- Uma vez criado o experimento, cinco opções são apresentadas:
Experimento: fornece detalhes do experimento, incluindo arquivos de dados carregados, canais escolhidos e classes de modelo verdade terrestres definidas.
Marcação: inicia a ferramenta de marcação por meio da qual os usuários podem preencher dados de verdade terrestres.
Treinamento: treina um modelo baseado nos dados da verdade terrestre.
Classificar: usa modelos treinados para classificar dados.
Resultados: fornece resultados de um experimento de treinamento e um experimento de classificação.
4. Preenchendo os dados de verdade terrestre usando ferramentas de marcação assistidas por IA
- Clique em Marcação para iniciar a interface da ferramenta de marcação.
- Clique nas ferramentas de zoom (ícones de lupa) para cortar as imagens para facilitar a visualização.
- Clique na barra deslizante para ajustar o tamanho da imagem para alterar quantas imagens são mostradas na galeria.
- Clique na opção Configuração de vídeo e escolha Min-Max, que fornece a melhor imagem de contraste para identificar todos os eventos-chave.
- Clique em Setup Gallery Display para alterar a cor da imagem de DNA para amarelo ou branco, o que melhorará a visualização de pequenos objetos (por exemplo, MN).
- Clique em Cluster para executar o algoritmo para agrupar objetos com morfologia semelhante. Quando o clustering estiver concluído, os clusters individuais com o número de objetos por cluster serão mostrados em uma lista em Populações desconhecidas. Selecione os clusters individuais para exibir os objetos dentro do cluster e atribua esses objetos às classes de modelo apropriadas.
- Depois que um mínimo de 25 objetos tiverem sido atribuídos a cada classe de modelo, o algoritmo Predict ficará disponível. Clique em Prever.
NOTA: Os objetos que não se encaixam bem em nenhuma população permanecem classificados como Desconhecidos. À medida que mais objetos são adicionados às populações de verdade, a precisão da previsão melhora. - Continue a preencher as classes de modelo verdade básicas com imagens apropriadas até que um número suficiente de objetos em cada classe seja alcançado.
- Depois que um mínimo de 100 objetos tiverem sido atribuídos a cada classe de modelo, clique na guia Treinamento na parte superior da tela. Clique no botão Treinar para criar um modelo usando os algoritmos Random Forest e CNN.
NOTA: O software de IA cria modelos usando os algoritmos Random Forest e CNN, as caixas de seleção permitem a criação de modelos usando apenas algoritmos Random Forest of CNN.
5. Avaliação da acurácia do modelo
- Quando o treinamento do modelo estiver concluído, clique em Exibir resultados.
- Use a tela de resultados para avaliar a precisão do modelo. Use o menu suspenso para alternar entre Random Forest e CNN.
NOTA: As populações de verdade podem ser atualizadas e o modelo pode ser retreinado ou usado como está para classificar dados adicionais.- Para atualizar as populações de verdade, clique em Marcação na parte superior e siga a seção 4.
6. Classificando os dados usando o modelo
- Inicie o software de IA. A tela padrão é a tela Novo Experimento . Use o ícone Pasta para escolher onde salvar o experimento e digite um nome para o experimento.
- Em Tipo de Experimento, clique no botão de opção ao lado de Classificar para iniciar um experimento de classificação. Clique em Avançar.
- Clique no modelo a ser usado para a classificação e, em seguida, clique em Avançar.
- Na tela Selecionar arquivos, clique em Adicionar arquivos e procure os arquivos a serem classificados pelo modelo CNN. Clique em Avançar.
- Em seguida, na tela Selecionar Populações Base , clique na caixa de seleção ao lado da população Não-Apoptótica em um dos arquivos carregados. Clique com o botão direito do mouse na população não-apoptótica e clique em Selecionar todas as populações correspondentes para selecionar essa população de todos os arquivos carregados. Clique em Avançar.
- Opcional: se os dados a serem classificados contiverem populações de verdade, eles poderão ser atribuídos às classes de modelo apropriadas na tela Selecionar Populações de Verdade. Caso contrário, clique em Avançar para ignorar esta etapa.
- Na tela Selecionar canais , escolha Canal 1 para campo brilhante e Canal 7 para a coloração de DNA. Clique com o botão direito do mouse em um canal e clique em Aplicar a Todos. Em seguida, clique em Avançar.
- Por fim, na tela de confirmação , clique em Criar experimento. O software de IA carrega o modelo selecionado e todas as imagens dos arquivos de dados escolhidos. Clique em Concluir.
- Clique em Classificar para iniciar a tela de classificação. Clique no botão Classificar . Isso inicia o processo de usar o modelo RF e CNN para classificar dados adicionais e identificar todos os objetos que pertencem às classes de modelo especificadas.
NOTA: As caixas de seleção podem ser usadas para selecionar o modelo de RF e/ou o modelo CNN. - Quando a classificação estiver concluída, clique em Exibir Resultados.
- Clique no botão Atualizar DAFs para abrir a janela Atualizar DAFs com Resultados da Classificação. Clique em OK para atualizar os arquivos .daf.
7. Geração de um relatório dos resultados da classificação
- Na tela Resultados, clique em Gerar Relatório. Marque a caixa de seleção ao lado de Criar relatório para cada DAF de entrada se um relatório individual para cada daf de entrada for necessário. Clique em OK.
- Depois de concluído, abra a pasta onde os arquivos de relatório foram salvos. Dentro da pasta, há um relatório de .pdf de experimento e uma pasta Recursos .
- Abra o .pdf para exibir o relatório. O relatório contém informações de modelo e experimento, a lista de arquivos .daf de entrada, as contagens de classe e porcentagens de classe em formato tabular e histograma e uma matriz de confusão resumindo a probabilidade de previsão mediana em todos os arquivos .daf de entrada.
- Abra a pasta Recursos e, em seguida, a pasta CNN . Dentro dessa pasta estão .png arquivos dos gráficos de contagem de classes e barras de porcentagem, bem como a matriz de confusão. Além disso, há .csv arquivos contendo as contagens de classe e porcentagens para cada arquivo de entrada.
8. Determinação da frequência e citotoxicidade de MN
- Calculando a frequência MN
- Método não Cyt-B: Para determinar a frequência MN, abra o arquivo class_count.csv da etapa 7.4. Para cada arquivo de entrada, divida as contagens na população "Mononucleada com MN" pelas contagens na população "Mononucleada" e multiplique por 100:

- Método Cyt-B: Para determinar a frequência MN, abra o arquivo class_count.csv da etapa 7.4. Para cada arquivo de entrada, divida as contagens na população "Binucleada com MN" pelas contagens na população "Binucleada" e multiplique por 100:
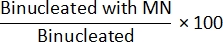
- Método não Cyt-B: Para determinar a frequência MN, abra o arquivo class_count.csv da etapa 7.4. Para cada arquivo de entrada, divida as contagens na população "Mononucleada com MN" pelas contagens na população "Mononucleada" e multiplique por 100:
- Cálculo da citotoxicidade
- Método não Cyt-B:
- Usando as contagens celulares iniciais e as contagens de células pós-tratamento, primeiro calcule a duplicação populacional (DP) para cada amostra2:
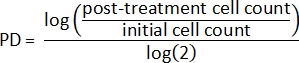
- Em seguida, calcule a duplicação relativa da população2:

- Finalmente, calcule a citotoxicidade2 para cada amostra:
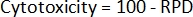
- Usando as contagens celulares iniciais e as contagens de células pós-tratamento, primeiro calcule a duplicação populacional (DP) para cada amostra2:
- Método Cyt-B:
- Para calcular o Índice de Proliferação em Bloco de Citocinese (IPCB)2, use as contagens nas classes mononucleada, binucleada e polinucleada para cada amostra do arquivo class_count.csv :
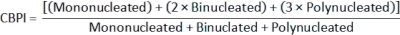
- Para calcular a citotoxicidade2, utilizar o CBPI das culturas controle (C) e expostas (T):
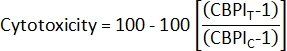
- Para calcular o Índice de Proliferação em Bloco de Citocinese (IPCB)2, use as contagens nas classes mononucleada, binucleada e polinucleada para cada amostra do arquivo class_count.csv :
- Método não Cyt-B:
Resultados
A Figura 1 mostra o fluxo de trabalho para usar o software de IA para criar um modelo para o ensaio MN. O usuário carrega os arquivos .daf desejados no software de IA e, em seguida, atribui objetos às classes de modelo verdade terrestre usando os algoritmos de marcação de cluster assistido por IA (Figura 2) e de previsão (Figura 3). Uma vez que todas as classes de modelo verdade terrestre tenham sido preenchidas com objetos suf...
Discussão
O trabalho aqui apresentado descreve o uso de algoritmos de aprendizagem profunda para automatizar a pontuação do ensaio MN. Diversas publicações recentes têm demonstrado que ferramentas intuitivas e interativas permitem a criação de modelos de deep learning para analisar dados de imagens sem a necessidade de conhecimento computacional aprofundado18,19. O protocolo descrito neste trabalho usando um pacote de software orientado por interface de usuário foi...
Divulgações
Os autores são empregados pela Luminex Corporation, uma empresa DiaSorin, fabricante do citômetro de fluxo de imagem ImageStream e do software Amnis AI usado neste trabalho.
Agradecimentos
Nenhum.
Materiais
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
Referências
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20 (2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324 (2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados