Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Автоматизация анализа микроядер с помощью визуализационной проточной цитометрии и искусственного интеллекта
В этой статье
Резюме
Анализ микроядра (MN) является хорошо зарекомендовавшим себя тестом для количественной оценки повреждения ДНК. Однако оценка анализа с использованием традиционных методов, таких как ручная микроскопия или анализ изображений на основе признаков, является трудоемкой и сложной. В этой статье описывается методология разработки модели искусственного интеллекта для оценки анализа MN с использованием данных проточной цитометрии визуализации.
Аннотация
Анализ микроядра (MN) используется регулирующими органами во всем мире для оценки химических веществ на предмет генетической токсичности. Анализ может быть выполнен двумя способами: путем оценки MN в однажды разделенных, заблокированных цитокинезом бинуклеированных клетках или полностью разделенных моноядерных клетках. Исторически сложилось так, что световая микроскопия была золотым стандартом для оценки анализа, но она трудоемка и субъективна. Проточная цитометрия использовалась в последние годы для оценки анализа, но ограничена неспособностью визуально подтвердить ключевые аспекты клеточных изображений. Визуализирующая проточная цитометрия (IFC) сочетает в себе высокопроизводительный захват изображений и автоматический анализ изображений и успешно применяется для быстрого получения изображений и оценки всех ключевых событий в анализе MN. Недавно было продемонстрировано, что методы искусственного интеллекта (ИИ), основанные на сверточных нейронных сетях, могут быть использованы для оценки данных анализа MN, полученных IFC. В этом документе описываются все шаги по использованию программного обеспечения ИИ для создания модели глубокого обучения для оценки всех ключевых событий и применения этой модели для автоматической оценки дополнительных данных. Результаты модели глубокого обучения ИИ хорошо сравниваются с ручной микроскопией, что позволяет полностью автоматизировать оценку анализа MN путем объединения IFC и AI.
Введение
Анализ микроядра (MN) имеет основополагающее значение в генетической токсикологии для оценки повреждения ДНК при разработке косметики, фармацевтических препаратов и химических веществ для использования человеком 1,2,3,4. Микроядра образуются из целых хромосом или фрагментов хромосом, которые не включаются в ядро после деления и конденсируются в небольшие круглые тела, отделенные от ядра. Таким образом, MN можно использовать в качестве конечной точки для количественной оценки повреждения ДНК при тестировании на генотоксичность1.
Предпочтительный метод количественной оценки MN заключается в некогда разделенных бинуклеированных клетках (BNC) путем блокирования деления с использованием цитохалазина-B (Cyt-B). В этой версии анализа цитотоксичность также оценивается путем оценки моноядерных (MONO) и полиядерных (POLY) клеток. Анализ также может быть выполнен путем оценки MN в незаблокированных клетках MONO, что быстрее и легче набрать, при этом цитотоксичность оценивается с использованием количества клеток до и после воздействия для оценки пролиферации 5,6.
Физический подсчет результатов анализа исторически проводился с помощью ручной микроскопии, поскольку это позволяет визуально подтверждать все ключевые события. Однако ручная микроскопия сложна и субъективна1. Таким образом, были разработаны автоматизированные методы, в том числе сканирование предметных стекол микроскопа и проточная цитометрия, каждая из которых имеет свои преимущества и ограничения. В то время как методы сканирования слайдов позволяют визуализировать ключевые события, слайды должны создаваться с оптимальной плотностью ячеек, чего может быть трудно достичь. Кроме того, в этом методе часто отсутствует цитоплазматическая визуализация, что может поставить под угрозу оценку клеток MONO и POLY 7,8. В то время как проточная цитометрия обеспечивает высокопроизводительный сбор данных, клетки должны быть лизированы, что не позволяет использовать форму анализа Cyt-B. Кроме того, традиционная проточная цитометрия, не являющаяся методом визуализации, не обеспечивает визуальной проверки ключевых событий 9,10.
Поэтому для проведения анализа MN была исследована визуализирующая проточная цитометрия (IFC). ImageStreamX Mk II сочетает в себе скорость и статистическую надежность обычной проточной цитометрии с возможностями визуализации с высоким разрешением микроскопии в одной системе11. Было показано, что с помощью IFC можно захватывать и автоматически оценивать изображения всех ключевых событий с высоким разрешением с использованием методов 12,13 на основе функций 12,13 или искусственного интеллекта (ИИ) 14,15. Используя IFC для проведения анализа MN, можно получить автоматическую оценку гораздо большего количества клеток по сравнению с микроскопией за более короткий промежуток времени.
Эта работа отличается от ранее описанного рабочего процессаанализа изображений 16 и обсуждает все шаги, необходимые для разработки и обучения модели случайного леса (RF) и/или сверточной нейронной сети (CNN) с использованием программного обеспечения Amnis AI (далее именуемого «программное обеспечение AI»). Описаны все необходимые шаги, включая заполнение наземных достоверных данных с помощью инструментов маркировки с помощью ИИ, интерпретацию результатов обучения модели и применение модели для классификации дополнительных данных, что позволяет рассчитать генотоксичность и цитотоксичность15.
протокол
1. Сбор данных с помощью визуализирующей проточной цитометрии
ПРИМЕЧАНИЕ: См. Rodrigues et al.16 со следующими изменениями, отмечая, что для оптимального захвата изображения, возможно, потребуется модифицировать области сбора данных с использованием IFC:
- Для метода, отличного от Cyt-B, выполните подсчет клеток с использованием коммерчески доступного счетчика клеток, следуя инструкциям производителя (см. Таблицу материалов) на каждой культуре непосредственно перед культурой и сразу после периода восстановления.
- При запуске образцов на проточном цитометре с одной камерой поместите Brightfield (BF) в канал 4. Замените M01 на M04, а M07 на M01.
ПРИМЕЧАНИЕ: «M» относится к каналу камеры на IFC. - Используйте 40-кратное увеличение во время съемки.
- На графике соотношения сторон области BF и BF во время съемки используйте следующие координаты региона:
X-координаты: 100 и 900; Y-координаты: 0.7 и 1 (метод Cyt-B)
X-координаты: 100 и 600; Y-координаты: 0.7 и 1 - На графике интенсивности Хёхста используйте следующие координаты региона:
X-координаты: 55 и 75; Y-координаты: 9.5 и 15 (метод Cyt-B)
X-координаты: 55 и 75; Y-координаты: 13 и 21 (метод без Cyt-B) - Чтобы удалить из данных изображения апоптотических и некротических объектов, запустите программный пакет IDEAS 6.3 (далее именуемый «программное обеспечение для анализа изображений»; см. Таблицу материалов).
ПРИМЕЧАНИЕ: Программное обеспечение AI было разработано для работы с файлами .daf, которые были обработаны с использованием последней версии программного обеспечения для анализа изображений. Убедитесь, что программное обеспечение для анализа изображений обновлено. - Сохраните эту работу в виде файла шаблона (.ast).
2. Создание DAF-файлов для всех файлов .rif
- Программное обеспечение искусственного интеллекта позволяет импортировать только файлы .daf. Создайте DAF-файлы для всех RIF-файлов в эксперименте с помощью пакетной обработки.
- В меню «Инструменты» нажмите «Пакетные файлы данных», а затем нажмите «Добавить пакет».
- В новом окне выберите «Добавить файлы» и выберите файлы .rif для добавления в пакет. В разделе Выберите шаблон или файл анализа данных (.ast, .daf) выберите AST-файл , созданный ранее.
- При необходимости назначьте имя пакета и нажмите OK, чтобы создать файлы .daf для всех загруженных файлов .rif.
3. Создание эксперимента в программном обеспечении ИИ
- На рисунке 1 показана блок-схема, описывающая процесс создания модели глубокого обучения с помощью программного обеспечения ИИ.
- Запустите программное обеспечение AI и убедитесь, что установлена самая последняя версия, нажав « О программе » в левом нижнем углу окна. Если последняя версия не установлена, свяжитесь с support@luminexcorp.com, чтобы получить ее.
- Экран по умолчанию в программном обеспечении — экран «Новый эксперимент ». С помощью значка «Папка» выберите место для сохранения эксперимента и введите имя эксперимента (например, « Модель MN»).
- В разделе " Тип эксперимента" щелкните переключатель рядом с "Обучить ", чтобы начать обучающий эксперимент и начать построение модели CNN. Нажмите « Далее».
- Необязательно: если модель была ранее обучена, ее можно использовать в качестве шаблона для новой модели ИИ, а также выбрать в качестве шаблона для создания новой модели на экране Выбор модели шаблона . Если шаблонная модель не существует, просто пропустите этот шаг, нажав кнопку Далее.
- Следующий экран — экран «Определить новую модель ». В разделе Модель будет автоматически заполнено имя, присвоенное модели на шаге 3.3.
- В разделе «Описание» введите описание модели (необязательно) и оставьте максимальный размер изображения 150 пикселов.
- В разделе « Каналы» нажмите « Добавить BF», чтобы добавить канал яркого поля в список. В разделе « Имя» дважды щелкните Brightfield и переименуйте этот канал в BF. Нажмите « Добавить FL », чтобы добавить флуоресцентный канал в список. В разделе « Имя» дважды щелкните « Флуоресцентный » и переименуйте этот канал в ДНК.
- В разделе «Имена классов» нажмите « Добавить». Во всплывающем окне введите Mononucleated и нажмите OK. При этом одноядерный класс добавляется в список имен классов. Повторите этот процесс, чтобы убедиться, что в списке определены следующие шесть классов:
Одноядерный
Моноядерный с MN
Двуядерный
Двуядерный с MN
Полиядерный
Нерегулярная морфология
Нажмите « Далее».
ПРИМЕЧАНИЕ: Эти шесть классов наземных моделей истинности будут представлять ключевые события, подлежащие оценке, а также изображения с морфологией, отличающейся от принятых критериев оценки5.- Дополнительно: При желании можно включить шаблон анализа из версии 1.7 для использования функций программного обеспечения для анализа изображений. Если вы хотите включить эти функции в модель ИИ, найдите AST-файл, а затем в раскрывающихся списках для конкретных каналов выберите подмножества функций, которые вы хотите включить.
- В разделе «Выбрать файлы» нажмите «Добавить файлы» и найдите нужные файлы , которые будут добавлены в программное обеспечение ИИ для создания наземных достоверных данных. Нажмите « Далее».
ПРИМЕЧАНИЕ: Важно добавить несколько файлов данных (например, положительные и отрицательные контрольные данные), которые содержат достаточное количество всех ключевых событий.
- Затем на экране Select Base Populations (Выбор базовых популяций) найдите неапоптотическую популяцию из иерархии популяций. Щелкните правой кнопкой мыши неапоптотическую популяцию и выберите « Выбрать все совпадающие популяции». Нажмите « Далее».
ПРИМЕЧАНИЕ: Важно исключить любые популяции, которые не должны быть классифицированы (например, бусины, мусор, дублеты и т. Д.) - Этот экран является экраном "Выбор истинных популяций".
- Если помеченные истинные популяции ключевых событий не были созданы в программном обеспечении для анализа изображений, нажмите « Далее».
- Если в программном обеспечении для анализа изображений были созданы помеченные истинные популяции, назначьте их соответствующему классу модели.
- Чтобы назначить помеченную истинную популяцию клеток MONO с MN, щелкните класс Mononucleated with MN в разделе Model Classes (Классы моделей ) слева. Затем нажмите на соответствующую помеченную правду справа, которая содержит эти события.
- Если помеченные истинные популяции были созданы в нескольких файлах данных, щелкните правой кнопкой мыши одну из истинных популяций и выберите « Выбрать все совпадающие популяции », чтобы добавить помеченные популяции из нескольких файлов в соответствующий класс.
- После того, как все соответствующие группы истины будут назначены, нажмите « Далее».
- На экране Выбор каналов выберите соответствующие каналы для эксперимента. Здесь установите BF на канал 1, а Hoechst на канал 7. Щелкните правой кнопкой мыши канал и выберите «Применить ко всем». Нажмите «Далее».
- Наконец, на экране подтверждения нажмите « Создать эксперимент».
- Программное обеспечение ИИ загружает изображения из файлов данных и создает классы моделей, определенные на шаге 3.5.3, с наземными изображениями, которые были назначены на шаге 3.7. Нажмите «Готово».
- После создания эксперимента представляются пять вариантов:
Эксперимент: предоставляет подробные сведения об эксперименте, включая загруженные файлы данных, выбранные каналы и определенные классы модели наземной истины.
Тегирование: запускает инструмент тегов, с помощью которого пользователи могут заполнять наземные достоверные данные.
Обучение: обучает модель на основе наземных достоверных данных.
Классификация: использует обученные модели для классификации данных.
Результаты: предоставляет результаты как обучающего эксперимента, так и классификационного эксперимента.
4. Заполнение наземных достоверных данных с помощью инструментов маркировки с помощью искусственного интеллекта
- Нажмите « Теги », чтобы запустить интерфейс инструмента тегов.
- Нажмите на инструменты масштабирования (значки увеличительного стекла), чтобы обрезать изображения для более удобного просмотра.
- Нажмите на ползунок, чтобы настроить размер изображения, чтобы изменить количество изображений, отображаемых в галерее.
- Нажмите на опцию « Настройка дисплея » и выберите «Мин-Макс», который обеспечивает наилучшее контрастное изображение для идентификации всех ключевых событий.
- Нажмите « Настроить отображение галереи », чтобы изменить цвет изображения ДНК на желтый или белый, что улучшит визуализацию небольших объектов (например, MN).
- Нажмите « Кластер », чтобы запустить алгоритм группировки объектов со схожей морфологией. После завершения кластеризации отдельные кластеры с количеством объектов в кластере отображаются в списке в разделе Неизвестные популяции. Выберите отдельные кластеры, чтобы просмотреть объекты в кластере, и назначьте эти объекты соответствующим классам моделей.
- После того, как каждому классу модели будет назначено не менее 25 объектов, алгоритм прогнозирования становится доступным. Нажмите « Предсказать».
ПРИМЕЧАНИЕ: Объекты, которые плохо вписываются в какую-либо популяцию, остаются неизвестными. По мере того, как все больше объектов добавляется в популяции истины, точность предсказания улучшается. - Продолжайте заполнять классы наземных моделей истинности соответствующими изображениями до тех пор, пока не будет достигнуто достаточное количество объектов в каждом классе.
- После того, как каждому классу моделей будет назначено не менее 100 объектов, нажмите на вкладку « Обучение » в верхней части экрана. Нажмите на кнопку «Обучить », чтобы создать модель с помощью алгоритмов Random Forest и CNN.
ПРИМЕЧАНИЕ: Программное обеспечение ИИ создает модели, используя алгоритмы Random Forest и CNN, флажки позволяют создавать модели с использованием только алгоритмов Random Forest of CNN.
5. Оценка точности модели
- После завершения обучения модели нажмите « Просмотреть результаты».
- Используйте экран результатов для оценки точности модели. Используйте выпадающее меню для переключения между Random Forest и CNN.
ПРИМЕЧАНИЕ: Популяции истинности могут быть обновлены, а модель может быть переобучена или использована как есть для классификации дополнительных данных.- Чтобы обновить популяции правды, нажмите « Теги » вверху и следуйте разделу 4.
6. Классификация данных с помощью модели
- Запустите программное обеспечение AI. По умолчанию используется экран «Новый эксперимент ». С помощью значка «Папка » выберите место для сохранения эксперимента и введите имя эксперимента.
- В разделе " Тип эксперимента" щелкните переключатель рядом с "Классифицировать ", чтобы начать эксперимент по классификации. Нажмите « Далее».
- Нажмите на модель, которая будет использоваться для классификации, затем нажмите « Далее».
- На экране «Выбор файлов » нажмите «Добавить файлы» и найдите файлы , которые будут классифицированы по модели CNN. Нажмите « Далее».
- Затем на экране Select Base Populations поставьте галочку напротив Non-Apoptotic population в одном из загруженных файлов. Щелкните правой кнопкой мыши неапоптотическую популяцию и выберите « Выбрать все совпадающие популяции », чтобы выбрать эту популяцию из всех загруженных файлов. Нажмите « Далее».
- Необязательно: если классифицируемые данные содержат популяции истинности, их можно назначить соответствующим классам моделей на экране Выбор популяций истинности. В противном случае нажмите кнопку Далее, чтобы пропустить этот шаг.
- На экране «Выбор каналов» выберите «Канал 1» для светлого поля и «Канал 7» для окрашивания ДНК. Щелкните правой кнопкой мыши канал и выберите «Применить ко всем». Затем нажмите «Далее».
- Наконец, на экране подтверждения нажмите «Создать эксперимент». Программное обеспечение AI загружает выбранную модель и все изображения из выбранных файлов данных. Нажмите «Готово».
- Нажмите « Классифицировать », чтобы запустить экран классификации. Нажмите на кнопку «Классифицировать ». Это запускает процесс использования модели RF и CNN для классификации дополнительных данных и идентификации всех объектов, принадлежащих к указанным классам моделей.
ПРИМЕЧАНИЕ: Флажки можно использовать для выбора модели RF и/или модели CNN. - После завершения классификации нажмите « Просмотреть результаты».
- Нажмите кнопку «Обновить DAF », чтобы открыть окно « Обновить DAF с результатами классификации ». Нажмите OK , чтобы обновить файлы .daf.
7. Формирование отчета о результатах классификации
- На экране «Результаты» нажмите «Создать отчет». Установите флажок рядом с пунктом Создать отчет для каждого входного DAF, если требуется отдельный отчет для каждого входного DAF. Нажмите на кнопку ОК.
- После завершения откройте папку, в которой были сохранены файлы отчета. В папке есть отчет об эксперименте .pdf и папка ресурсов .
- Откройте .pdf, чтобы просмотреть отчет. Отчет содержит сведения о модели и эксперименте, список входных DAF-файлов, количество классов и процентное соотношение классов в табличном формате и формате гистограммы, а также матрицу путаницы, обобщающую среднюю вероятность предсказания для всех входных DAF-файлов.
- Откройте папку «Ресурсы », а затем папку CNN . В этой папке находятся .png файлы с гистограммами количества классов и процентами, а также матрица путаницы. Кроме того, существует .csv файлов, содержащих количество классов и проценты для каждого входного файла.
8. Определение частоты МН и цитотоксичности
- Расчет частоты МН
- Метод, отличный от Cyt-B: Чтобы определить частоту MN, откройте файл class_count.csv из шага 7.4. Для каждого входного файла разделите количество в популяции «Одноядерное с MN» на количество в популяции «Одноядерное» и умножьте на 100:

- Метод Cyt-B: Чтобы определить частоту MN, откройте файл class_count.csv из шага 7.4. Для каждого входного файла разделите количество в «двуядерной» популяции на количество в «двуядерной» популяции и умножьте на 100:
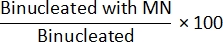
- Метод, отличный от Cyt-B: Чтобы определить частоту MN, откройте файл class_count.csv из шага 7.4. Для каждого входного файла разделите количество в популяции «Одноядерное с MN» на количество в популяции «Одноядерное» и умножьте на 100:
- Расчет цитотоксичности
- Метод Non Cyt-B:
- Используя начальное количество клеток и количество клеток после лечения, сначала рассчитайте удвоение популяции (PD) для каждой выборки2:
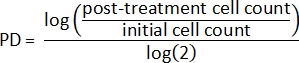
- Затем вычисляем относительное удвоение численности населения2:

- Наконец, рассчитайте цитотоксичность2 для каждого образца:
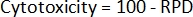
- Используя начальное количество клеток и количество клеток после лечения, сначала рассчитайте удвоение популяции (PD) для каждой выборки2:
- Метод Cyt-B:
- Чтобы рассчитать индекс пролиферации цитокинез-блоков (CBPI)2, используйте подсчет в моноядерных, бинуклеированных и полиядерных классах для каждого образца из файла class_count.csv :
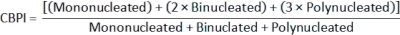
- Чтобы рассчитать цитотоксичность2, используйте CBPI из контрольных культур (C) и подвергшихся воздействию культур (T):
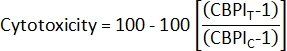
- Чтобы рассчитать индекс пролиферации цитокинез-блоков (CBPI)2, используйте подсчет в моноядерных, бинуклеированных и полиядерных классах для каждого образца из файла class_count.csv :
- Метод Non Cyt-B:
Результаты
На рисунке 1 показан рабочий процесс использования программного обеспечения ИИ для создания модели для анализа MN. Пользователь загружает нужные файлы .daf в программное обеспечение ИИ, а затем назначает объекты классам наземных моделей истинности с помощью кластера с по...
Обсуждение
В представленной здесь работе описывается использование алгоритмов глубокого обучения для автоматизации скоринга анализа MN. Несколько недавних публикаций показали, что интуитивно понятные интерактивные инструменты позволяют создавать модели глубокого обучения для анализа данных ?...
Раскрытие информации
Авторы работают в Luminex Corporation, компании DiaSorin, производителе проточного цитометра ImageStream и программного обеспечения Amnis AI, используемого в этой работе.
Благодарности
Никакой.
Материалы
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
Ссылки
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20 (2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324 (2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены