Method Article
强大的3D直接标记的探针DNA鱼
摘要
我们描述一个强大的和灵活的三维DNA鱼直接标记的荧光探针分析核架构协议。
摘要
3D DNA鱼已成为一个主要的工具,用于分析三维组织的细胞核,该技术已发表了一些变化。在这篇文章中,我们描述了协议的鲁棒性,重现性和易用性进行了优化。明亮的荧光标记的探针产生的缺口转译与氨基allyldUTP随后通过化学偶联的染料。三维DNA FISH进行使用冻融步骤,用于细胞透化和加热步骤,用于同时变性的探针和核DNA。该协议是适用于细胞类型和各种探头(BAC的质粒,fosmids,或所有的染色质油漆)的范围内,并可以为高通量的自动成像。用这种方法,我们经常探讨核本地化最多三个染色体区域。
引言
DNA荧光原位杂交(DNA FISH)技术允许单个基因位点,亚染色体域,甚至整个染色体的三维可视化在细胞周期的各个阶段。 2D鱼是用于中期研究,而3D鱼已被广泛用于探测空间组织的基因组和功能相间期间(1,2和参考文献)之间的关系。历史上,共同关联的研究进行了通过调查数十至数百个别位点通过FISH。最近强大的高通过3C 4C和Hi-C为基础的技术,例如已开发3,允许调查成千上万的不同位点的分子之间的串扰。基于3C技术和DNA鱼虽然可以互补的方法,他们不回答相同的问题。基于3C的方法提供了混合细胞群的组合读出的,从而在概率性合作协会。相比之下,尽管吞吐量低,鱼为基础的技术分析或单个细胞中的染色体位点的空间安排,根据其发育或细胞周期不同阶段提供了可能性。因此,FISH将继续是一个重要的工具,用于探测核结构与功能的关系。
有两个主要的考虑,执行成功的3D鱼实验。这些是1。获得最佳标记探针和2。选择细胞的治疗方法,包括固定,预处理和后杂交步骤,尽可能多地保存核形态,同时充分地获得的DNA探针杂交。高效的探针标记的鱼是非常重要的。传统上,已被用于引入缺口转译的半抗原或荧光标记的核苷酸4。同样,商业缺口平移套件可用于直接半抗原或荧光成立,BU吨也为两个步骤的标签使用的氨基烯丙基核苷酸和胺的反应性染料。后者呈现染料掺入DNA聚合酶一个体积不大分子,工作更有效率。最近,已开发的非酶标记的DNA的试剂盒,利用协调结合的铂的核酸。甚至可以将已经购买的标号为5的FISH探针。虽然包和商业化生产的探针毫无疑问给易用性,他们是相当昂贵的比买的各个组件和生产内部的探头。我们优化一个低成本的缺口翻译协议,以直接标示许多不同的BAC探针多种颜色。我们发现,获得高纯度的BAC DNA,是至关重要的,结果在规定只有10-20纳克的探针每FISH滑动,而到第10 - 20倍使用不纯的模板DNA时,导致主要的成本和时间积蓄。使用氨基allyldUTP的允许灵活的标签探头可用胺的活性染料( 例如的Alexa Fluor或CY-染料),或半抗原( 如生物素,地高辛)。半抗原标记的探针检测扩增并可视化的信号的荧光团标记的抗体或抗生蛋白链菌素。明亮的直接标记探针一般背景染色少,避免过度放大的信号,从而导致在一个更准确的陈述核本地化和轨迹形态。
鱼有几种不同的技术,使用不同的固定剂,前处理,杂交后洗,但这些一般分为以下类别:戊二醛固定用NaOH变性,变性与盐酸甲醛固定,热变性和甲醛固定6-9 。其中每一个都有优点和缺点。戊二醛固定核结构保存好结果,但需要与还原剂,以尽量减少治疗产生的自发荧光,和NaOH处理需要严格控制,以平衡足够的DNA变性和潜在的损害核结构6。甲醛固定少强,给人扰动核架构的可能性增加,但通常也更强大和更可靠的信号和较低的自体荧光9。 HCl处理depurinates出蛋白质的DNA和钢带的DNA探针提供良好的访问,但也可能引入DNA断裂。暖气物理上分开的两条DNA链产生良好的目标杂交和强烈的信号,但可能会导致一些扰动核结构8。在何种程度上这些技术的每一个影响蛋白的抗原表位的广泛变化6,9,从而在规定的试验方法来测定每个蛋白免疫FISH实验中使用最佳的协议。
虽然没有“完美”的DNA FISH TEchnique,他们都可以是有用的,如果得到很好的控制。我们的目标是优化稳定性和重复性DNA鱼的协议,调查在各种细胞类型10多个位点的空间定位,专注于加热的方法最可靠的信号。这个问题,以及自动成像系统的使用中,我们旨在增加通过单细胞分析的核安排。
研究方案
1。尼克翻译产生直接标记的DNA探针
注:从BAC的直接标记的探针(100-250 KB)不断产生明亮的信号。如果需要较小的探头,使用fosmids“(40-50),甚至质粒含有5-10 kb的插入。确定BAC或粘粒克隆对应使用合奏或UCSC基因组浏览器的特定基因。准备高质量的BAC DNA的反复沉淀或使用市售试剂盒。编制与细菌基因组DNA的污染,少探头每张幻灯片的需要。在两个步骤中执行标签:缺口翻译引入氨基烯丙基-dUTP和胺反应性染料的化学耦合。
1.1。尼克翻译
在缺口翻译,DNA酶I被用来创建单链断裂。 DNA聚合酶Ⅰ伸长这些“缺口”,用新的,吨取代现有的核苷酸的3'末端的特此'翻译'的缺口,从而提供了机会,将标记的核苷酸。氨基烯丙基-dUTP的选择,因为有效地掺入DNA聚合酶我和后来的化学偶联胺活性染料或半抗原的潜力。这是至关重要的DNA酶I的刻痕和DNA聚合酶I翻译之间实现适当的平衡;太多DNA酶I将导致过度的DNA消化,产量低,短的片段大小,太少,不能产生足够的刻痕聚合酶开始翻译,给予大片段大小和不良掺入氨基烯丙基-原位。下面的协议效果很好,但它可能需要滴定不同批次的或不同的制造商的DNA酶I。
- 设置标记反应在冰上孵育2个小时,在16°C。这应该会产生0.5-1微克缺口转译的DNA,并且可以扩展。的DNase I缓冲液稀释的DNase I 1:30( 即 0.3 U /微升)。
- 运行1微升2%琼脂糖凝胶上检查标记的片段的大小。当你运行的凝胶,保持冰的反应。成功的缺口translati将导致在涂抹运行150基点和700基点,与一些较大的片段〜1 KB(关键步骤:看到代表结果)之间的大量的碎片。如果需要的话,再加入1μl的新鲜1:30的DNase I稀释和孵化,在16°C 15-30分钟。孵化时间会有所不同,根据BAC DNA的数量和质量。
- 灭活DNA酶I,通过加热至75℃,持续10分钟。
- 清理胺修饰的DNA,用PCR纯化试剂盒。在100μlH 2 O洗脱
- 通过加入10微升3 M醋酸钠(pH值5.2)和275微升乙醇的乙醇沉淀DNA。在-20°C的离开至少1小时或过夜。以最大速度旋转30分钟,在4°C。用500μl70%乙醇洗涤沉淀,风干。此步骤除去微量的胺,干扰标记反应。悬浮颗粒在6微升H 2 O每1X初始反应。
- 加入1μl,以确定浓度的胺改性的DNA微量紫外光谱。
1.2。耦合的荧光染料
荧光标记的探针是通过化学偶联的染料。琥珀酰亚胺酯的Alexa Fluor染料氨基allyldUTP修饰的DNA形成共价键与胺反应,从而产生直接标记的探针。的Alexa Fluor 488,555和647染料已成功为这个过程。
- 调整-烯丙基氨基修饰的DNA的4微克至体积为5μl,热至95℃5分钟,捕捉在冰上冷却,并加入3微升的0.2M的NaHCO 3。
- 胺的反应性染料的一个等份溶于2微升无水DMSO,在室温下。新增8微升修饰过的DNA,用NaHCO 3,旋涡,脉冲自旋和室温孵育1小时在黑暗中。
注意:可以用于标记一个以上的下面的方案的基础上的探针的胺的反应性染料。由于O型1微克只有10-20纳克的探针是用来每滑标签,给人足够的探头50-100幻灯片。另外,也可以存储为将来标记反应的胺改性的DNA于-20℃。
| DNA量 | DNA量 | 0.2 M NaHCO 3中 | 染料的体积 | 总 | 够 |
| 4×1微克 | 1.25微升每个 | 0.75微升每个 | 0.5微升每个 | 2.5微升(1/4) | 每次50-100幻灯片 |
| 3×1.35微克 | 各1.67微升 | 各1μl | 0.67微升每个 | 3.34微升(1/3) | 每次65-130幻灯片 |
| 2×2微克 | 各2.5μl | 各1.5μL | 各1μl | 5微升(1/2) | 100-200每个滑动 |
| 1×4微克 | 5微升 | 3微升 | 2微升 | 10微升(全) | 200-400幻灯片 |
- 加入90微升H 2 O和净化标记探针,用PCR纯化试剂盒。 100微升10 mMTris,-CL(pH值8.5)洗脱。
- 微量全的频谱光谱( 见图2)使用比尔-朗伯方程(伴随的Alexa Fluor活性染料或可以在网上找到,说明在数据表中给出)确定探针浓度和标签效率。含有3-6染料每100个基点的探头正常工作。注册度较低的探头可能给弱鱼信号。或者,运行5微升2%琼脂糖凝胶上没有DNA染色检查掺入荧光染料使用phosphoimager。染色后的凝胶用溴化乙锭或DNA染色替代品,并比较标记的DNA总DNA。
通过以下步骤,感兴趣的细胞被固定到幻灯片和透。细胞的DNA和直接标记探针变性,然后在热板上一起,在一个不透光的湿盒杂交过夜。
2.1。 1天
- 探头降水
注:在一些点在第1天的沉淀探头。可以舒适地进行并行处理细胞,并且不具有的一天开始时完成。- 混合10-20纳克直接标记探针,6微升C 0 T-1 DNA(6微克),1微升鲑鱼睾丸(9.7微克)的单链DNA和音量调节至100微升水。
- 加入10微升3 M醋酸钠和275μL乙醇,涡旋混匀。在-20℃沉淀至少1小时
- 的自旋,在微量离心30分钟,在4℃下以最大的速度用70%乙醇洗涤沉淀,再次旋转。干颗粒和悬浮在5微升去离子甲酰胺。重悬在37℃下30分钟,同时在1000rpm下避光(这里箔在恒温)振摇。
- 加入5微升的硫酸葡聚糖混合,摇匀,再延长10分钟,于37℃再次避光。移液器上下前吹打到盖玻片。
注:对于染色体涂染结合BAC探针,全染色体涂抹探针准备使用的杂交组合所提供的是一个方便的选择。重悬沉淀BAC探针(C 0 T1 DNA单链DNA从鲑鱼睾丸)在10微升染色体调漆上下吹打。 ,而在300〜500 rpm振摇10分钟,在37℃下孵育。使用染色体油漆/ BAC探针混合物每点10微升。
- 将细胞幻灯片
注:细胞的倾向,坚持以幻灯片大大随细胞类型。对于不同的细胞类型,确定最佳的解决时间和细胞密度的经验。对于一致的结果,细胞应在单细胞悬液。
为了便于使用的圆的面积的单元格将被点样于用疏水性笔细胞重悬于PBS中,虽然这不是严格必需的。疏水屏障防止暂停幻灯片上蔓延,最多保留三个样本每张幻灯片很好地分离,允许使用极少量鱼免疫反应的抗体溶液中。另外,点样区可以保持在最低限度,如果细胞数量低。 - 小鼠胎肝细胞
- 收集每个E13.5小鼠胎肝上下吹打约1.5毫升的PBS重悬。在微量离心2分钟,以3,000 rpm的自旋。重复洗涤三次。在最后一次洗涤,应变通过70微米筛的细胞。从一个肝脏在160微升PBS重悬细胞,每点用50μ。离开细胞沉淀在室温下的2分钟。
- 中号乌斯ES细胞
1。小鼠胚胎干细胞不会容易坚持到幻灯片中。幻灯片上,圆面积的细胞会被发现疏水笔。成圆,每80-100微升准备暂停20,000-50,000细胞在正常的培养基,吸管离开附加约3小时在培养箱注意:另外,ES细胞可以培养在载玻片上,然后将形成菌落可能会阻碍自动成像。 - 固定细胞
1。有些实验需要使用固定的细胞,不重视自己的幻灯片。轻柔脱水,在300 rpm离心3分钟,使用cytocentrifuge上保留了细胞的三维结构,并确保细胞不脱落。
| 电池类型 | 悬架 | 建立时间 | 评论 |
| 鼠标胎肝 | PBS | 2分钟 | 板凳上 |
| 小鼠ES | 含补充 | 4小时 | 37℃培养箱中培养 |
| 小鼠淋巴细胞 | PBS | 2分钟 | 板凳上 |
| 鼠标P20生殖细胞 | 修复解决方案 | 离心涂片 | 板凳上 |
- 要修复细胞,轻轻地淹没在4%PFA幻灯片10分钟( 注意:在通风橱内进行)。修复细胞与滑动平托盘最好保留的细胞( 如 50毫升4%PFA在吸头盒的盖子)。 注意:这一步并不适用于细胞固定附件幻灯片前。
对于所有的后续步骤,使用50毫升或100毫升科普林氏罐子。小心不要刮到细胞移动之间滑动时罐子。
- 在0.1米的Tris-C淬火升,pH值7.4和10分钟,在RT。
- 通透细胞在0.1%saponin/0.1%的Triton X-100的PBS中,在RT下10分钟。
- 在PBS中洗两次,在RT下5分钟。
- 孵育至少20分钟,在20%甘油/ PBS中于RT。 注意:在这一点上,投影片可以被储存在-20℃下在50%甘油/ PBS中至少几个星期。已经观察到该存储至少一天的查询结果, 第9的信号强度增加。在室温20%甘油/ PBS重新校准之前跟进。 注:这是一个方便点开始沉淀探头(见2.1.1)。
- 3倍于液氮中的冷冻/解冻。淹没一张幻灯片在液氮中,使用一个小的杜瓦。退出后几秒钟和特征的爆裂声,代替纸巾除霜。等待为不透明的冷冻甘油消失,然后再次冻结。多达15个幻灯片,可以做到舒适的3个冷冻/解冻循环的旋转。
- W灰两次在PBS中5分钟,在RT。
- 孵育在0.1M HCl中,在RT下30分钟。
- 在RT下的5分钟,在PBS中清洗。
- 通透0.5%的Triton saponin/0.5%X-100/PBS在RT下30分钟。
- 在PBS中洗两次,在RT下5分钟。
- 平衡在50%formamide/2x SSC在RT下持续至少10分钟。
- 移液器探测到盖玻片。删除幻灯片从罐子和细胞周围点(次),用纸巾擦干多余的液体。由于甲酰胺干得很快,不小心干细胞。对于两个或三个细胞斑点的幻灯片,适用于10微升探头每点到22毫米x 50毫米盖玻片相应的现场在幻灯片上的位置。反转盖玻片上滑过现货(次)。水泥用橡胶密封,并允许这种完全干燥。避光所有后续阶段。
- 热幻灯片至78℃,正是一个热点板块( 如一个倒置的加热块,关键步骤:见讨论)2分钟。将一个盖(纸箱或SIM卡 ILAR)在热板保护变性过程中的光。
- 在37°C孵育过夜,在光紧加湿室。加湿光紧框,放置纸巾与水沾湿。
2.2第2天
- 准备Coplin式的解决方案,在合适的温度,罐子,随后的洗涤步骤。保护从光滑动尽可能地在所有后续的步骤。咖啡可以使一个很好的科普林氏罐轻紧盖。
- 在2X SSC橡胶水泥和地方滑动,直到揭去盖玻片松动和滑落。
- 洗净在50%formamide/2x SSC在45℃下15分钟水浴避光的地方盖。
- 在0.2X SSC冲洗15分钟,在63°C。
- 在2×SSC洗涤5分钟,在45℃下
- 在2X SSC洗5分钟,在室温。
- 在RT下的5分钟,在PBS中清洗。
- 用DAPI染色(5微克/毫升2×SSC),一个Coplin jar文件在RT中,持续2分钟。
- 在PBS中5分钟,在RT中脱色。
- 要安装盖玻片,盖玻片吸管上安装介质。每个单元当场使用10微升一个点,和22毫米×50毫米盖玻片两到三个点,用22毫米×22毫米盖玻片。 PBS周围的细胞现货尽可能擦干,但要小心不要干细胞。反转盖玻片上的幻灯片,密封指甲油。
结果
这里描述的协议概述( 图1)已被用于有很大的影响,结合DNA FISH与高通量分析的基因组组织的图像捕获。质量好的探针的产生是成功的关键(见讨论)。 图2展示了两个重要的质量控制FISH探针。缺口转译后,涂抹标本的DNA应该是可见的碎片的大部分运行在150和700之间的沸点( 图2A)。化学偶联后,掺入荧光染料,可衡量光谱法分析的探针( 图2B)。
当使用好探头,DNA FISH协议通常会导致较低的背景明亮的DNA FISH信号。我们已成功地使用此协议上的各种不同类型的细胞,并与任一落射荧光显微镜或激光共聚焦显微镜。自动成像使用一个外延fluorescencê显微镜需要锋利,容易辨认鱼小信号与背景( 图3),激光共聚焦显微镜需要更强烈的信号, 如图4A所示代表处理的图像。可以得到核坐标的FISH信号,可以计算基因组区域的空间关系(有关示例,参见图4B)。我们也使用3D建模染色体领土内鱼类信号( 电影1)通过共聚焦显微镜获得的图像栈。
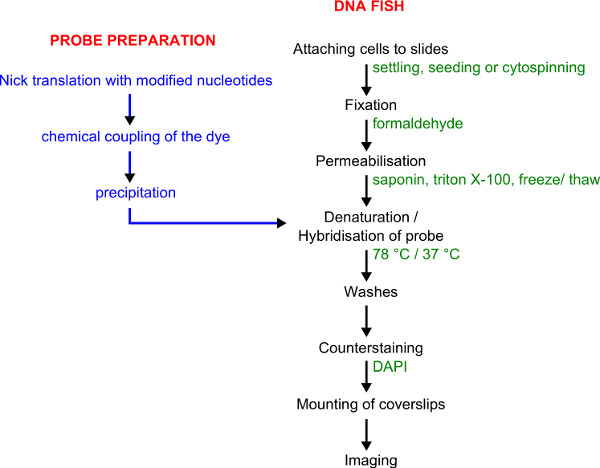
图1。探针标记和DNA FISH探针制备工作流程以蓝色显示。 DNA FISH程序显示为黑色,绿色细节。
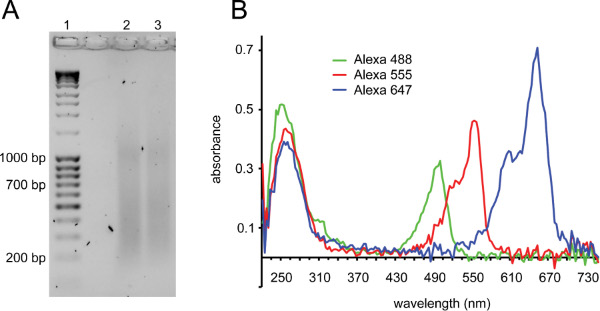
图2。通过缺口翻译和质量控制标记探针的标记探针。 A)凝胶表现出理想的涂抹前加热灭活DNA酶I(泳道1:DNA阶梯,车道2和3:BAC的缺口平移后)。片段应在150-700沸点范围。额外涂抹在大约1 kb经常观察成功的缺口翻译反应B)标记探针的微体积全频谱分析。在260nm处观察到的DNA峰与第二峰对应的探针被标记上荧光基团的吸光波长的Alexa Fluor 488:495 nm处的Alexa Fluor 555:555 nm处的Alexa Fluor 647:650纳米。三个探针具有良好的成立:荧光吸收峰是类似的高度或高于DNA峰。的Alexa Fluor 647通常具有较高的吸光比的Alexa Fluor 555,这反过来又具有较高的吸收比的Alexa Fluor 488。的Alexa Fluor 488也吸收一些光,在260 nm处,因此该探针的第一个峰高于其他。探头较差成立秀荧光吸收2〜3倍低于DNA吸光度。高2倍比DNA峰的荧光峰有超过探针通常含有未结合的染料,可能会导致在鱼的背景。
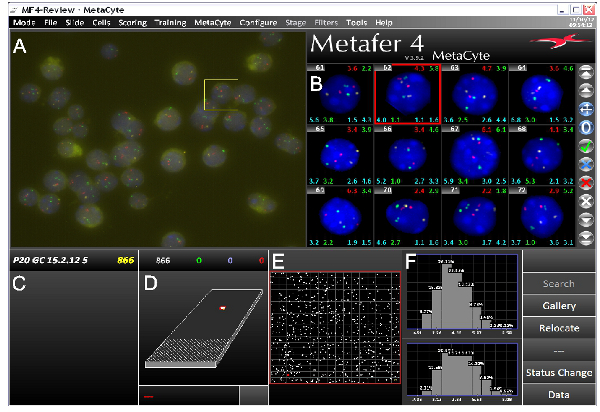
图3。屏幕截图3D鱼成像从MetaCyte的软件,呈现出典型的三色鱼实验。分成几个部分。)原场的视角(FOV)拍摄的图像Metafer /幻灯片扫描MetaCyte系统的由B)库。图像识别核有未dergone图像处理和分割鱼信号。各像册的图像上,可以显示各种类型的数据。示出在此示例中细胞数的左上角与的各种interallelic的interlocus距离右上和下面的每个图像的任一侧。C)幻灯片的名称与细胞核数确定在分析中,D)与所扫描的区域的滑动描写系统E)的扫描区域扩大。发现白色斑点代表原子核每视野F)处理的数据。在这个例子中,最大interallelic绿色信号(顶部)和红色信号(底部)之间的距离如图所示。显示的数据是从小鼠P20生殖细胞。探头位于不同染色体上,覆盖HBB和HBA基因的组蛋白集群。 点击这里查看大图 。
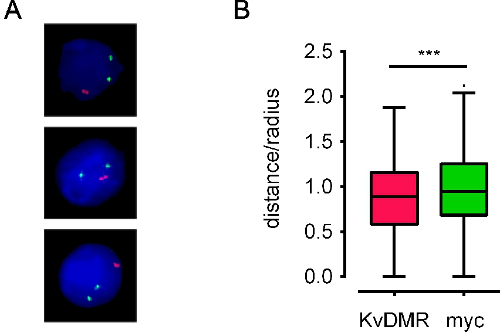
图4。处理后的图像和派生的位置数据的例子。 E13.5小鼠胎肝细胞染色与BAC探针位于不同染色体上的覆盖Myc和KCNQ1基因(称为myc和KvDMR,颜色为绿色和红色,分别)B)杜克盒须图代表interallelic距离A)处理的图像同一基因位点分布在600核。 interallelic距离为一小部分的核的半径(距离/半径),考虑到在核尺寸的变化作图。 ES细胞的核的半径约5微米。红色探针明显走得更近比绿色探头(***:P <0.01,配对t检验)。从10的数据。
动画1。 3D建模两个位点在染色体吨erritory。小鼠7号染色体的任一端靠近两个BAC探针标记(红色和远红,(伪色白)),与整个染色体漆 (绿色),小鼠ES细胞杂交在一起。 点击这里观看电影 。
讨论
3D DNA鱼已成为一种被广泛认可的工具,分析空间安排在细胞核内。而鱼提供视觉和直接的结果,每一项技术,人们需要意识到它的局限性。 DNA FISH面临的固有的问题,即相对苛刻的治疗是必要的,使染色质访问最终破坏核结构非常的事,要分析的FISH探针。几个战略已经追求玩花样无障碍和结构保存。在这里描述的协议中,染色体DNA冻融透化细胞和热变性的探针杂交前访问。 solovei, 等人,(2002)类似处理后的结构变化进行了分析,并发现,染色质域的平均位移量为300nm,这限制了分辨率的结构分析,以大约1 Mb的区域,基因组中的8。虽然这是它的分辨率不高,分析核位置的是一个合理的规模。
FISH分析是一个更实际的考虑,图像采集和核距离的测量耗时的过程,限制核的数目,可以分析。为了研究我们的目的是做高通量自动化成像的偶发事件。然后,用合适的控制措施,可以足够数量相比,允许强大的统计分析,空间关系。我们经常600原子核每个数据点,我们确定所有的鱼的三维坐标核体积内的信号进行分析。这些数据需要很少的存储空间,并允许分析间等位基因间的距离,在以后的任何点或径向位置。此外,自动化的处理是可以做到的研究员盲目的无意识的偏见的可能性大大降低。
为了使这种高通量分析产品的是,我们的目标是建立一个快速,高效的方式进行DNA鱼。我们发现,这里描述的协议是极其强大的,持续生产低背景明亮的信号。它的工作为所有类型的细胞,我们试图提供适应的步骤将细胞的幻灯片。此外,有一个例外,所有我们使用的BAC的可以变成明亮探头。我们还成功地检测到细胞核蛋白抗体染色后的DNA鱼。虽然这行之有效的一些蛋白质,我们建议比较与传统的免疫荧光法(IF),IF和DNA免疫FISH(比较11)之间的抗体染色模式,以确保一致性。
一般地,我们发现,在不同颜色的标记的探针产生了相同的结果。然而,选择标记染料时,应考虑以下几个方面。首先,需要的颜色的荧光以及检测在显微镜uSED。其次,由荧光团发射的光的波长应该有足够的不同,通过到其他信道,以避免出血。这将取决于在显微镜上的过滤器。第三,至少在我们的手中,某些颜色产生明亮的信号比其他人更一致。我们一直用DAPI(蓝色)作为染液的Alexa Fluor 555(红色),488(绿色),647(远红)不错的选择标记探针。随着我们的成像系统中,我们发现的Alexa Fluor 555(红色),产生明亮的信号,随后的Alexa Fluor 488(绿色)。的Alexa Fluor 647(远红)有一个缺点,它不能被人眼检测到,因此,没有信号,可以看出,当俯视显微镜。因此,如果做两色的鱼,我们推荐相结合的红色和绿色染料。
两个主要的问题可能产生于:洗去幻灯片和弱信号的细胞。以及细胞如何坚持到幻灯片是巨大的变量的逻辑之间EEN类型的细胞,和最好的协议来解决/种子细胞需要确定。我们已经成功地使用,也可以正电或聚-L-赖氨酸涂层幻灯片(买现成使用),但发现细胞一般坚持更好地聚-L-赖氨酸涂层幻灯片。我们还发现,当细胞剥离如纸,过于密集的整个地区将受阻,而自动成像细胞过于稀疏。因此,如果样品是不是太珍贵了,不同的手机号码,应接种以确定理想的密度。如果细胞,尤其容易洗掉,疏水性阻挡笔可以被使用,并仔细吹打直接到幻灯片上的解决方案,可以通过FISH步骤。移液也大大减少了所需的试剂量,但需要相当长的时间在做多张幻灯片。
弱信号通常是由于较差的探头,并做出好的投资时间,这是非常值得的。清洁BAC DNA应使用d为起始原料,可以通过以下方式获得反复沉淀或市售的试剂盒。细菌基因组DNA的污染造成负面影响探针的质量和期间小心处理的细胞裂解,细胞裂解物的过滤是必需的,以保持它在最低限度。然而,这是没有必要的使用核酸外切酶消化线性DNA的步骤,因为这大大降低了收率。有效的标记的探针是至关重要的。这一步是最重要的质量控制检查的片段大小缺口翻译后(见代表性的成果)。一个'好'抹黑几乎总是导致在鲜艳的探头。不过,我们已发现,偶尔目标BAC不能加工成一个很好的探针,和不同的BAC需要选择。另一个重要步骤是热变性。如果温度过低,DNA将仅部分变性的探针杂交将受到损害。如果温度过高,核结构的无线网络扰动会超过必要的。在我们手中,78℃下在一个倒置的热块了理想的折衷方案,但是这可能取决于在使用各自的热块。
我们有很好的结果使用安装介质Vectashield,但是发现,黄金SlowFade有较少的背景,并保留更长的荧光信号。硬设置安装介质,因为我们发现这影响的三维结构,并形成气泡,随着时间的推移,我们不建议使用。
最后,明亮的荧光直接标记的探针与DNA的FISH这里描述的协议提供坚固的快速分析的核的结构,适用于各种各样的生物学问题的一个有效的解决方案。
披露声明
本文的生产部分赞助由卡尔蔡司和MetaSystems的。
致谢
这项工作是由BBSRC和威康信托基金会资助。我们要感谢西蒙·沃克援助与成像和费利克斯·克鲁格与生物信息学分析的帮助。
材料
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||||||||
| Reagent/Material | |||||||||||||||||||||||||||||||||||||||
| NTB buffer | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| DTT | Invitrogen | D-1532 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| dNTPs | Bioline | BIO-39025 | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Aminoallyl-dUTP | Ambion | AM8439 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNA Polymerase I | New England Biolabs | M02095 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNase I recombinant RNase free | Roche | 4716728001 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| QIAquick PCR purification kit | Qiagen | 28104 | Step 1.1.4, 1.2.3 | ||||||||||||||||||||||||||||||||||||
| NaOAc | VWR | 27653 | Step 1.1.5, Step 2.1.1.2 | ||||||||||||||||||||||||||||||||||||
| NaHCO3 | Sigma | S5761 | Step 1.2.1 | ||||||||||||||||||||||||||||||||||||
| Alexa Fluor Reactive Dye Decapacks for Microarray Applications | Invitrogen (Molecular Probes) | A32750, A32756, A32757 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| DMSO (anhydrous) | Sigma Aldrich | 276855 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| SYBR Safe DNA gel stain | Life Technologies | S33102 | Step 1.2.4 (alternative to ethidium bromide) | ||||||||||||||||||||||||||||||||||||
| Cot-1 DNA | Invitrogen | 18440-016 | Step 2.1.1.1 (Cot-1 DNA can be home-made in large quantities and works just as well) | ||||||||||||||||||||||||||||||||||||
| Single stranded DNA from salmon testes | Sigma | D7656 | Step 2.1.1.1 | ||||||||||||||||||||||||||||||||||||
| Deionised formamide | Sigma | F-9037 | Step 2.1.1.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| Dextran sulphate | Sigma Life Sciences | D8906 | Step 2.1.1.4 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| XMP painting probes | MetaSystems | 000000-0528-837 | Step 2.1.1.4 | ||||||||||||||||||||||||||||||||||||
| Poly-L-lysine coated slides | Sigma Aldrich | P0425 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| Hydrophobic pen (ImmEdge) | Vector Laboratories | H-4000 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| 70 μm sieve (Mouse f–tal liver cells only) | BD Falcon | 352350 | Step 2.1.2.1 | ||||||||||||||||||||||||||||||||||||
| PFA | Sigma Aldrich | P6148 | Step 2.1.3 (Flammable, corrosive, acute toxicity, health hazard) (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Tris-Cl | Step 2.1.3 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Saponin from Quillaja bark | Sigma Aldrich | 47036 | Step 2.1.4, 2.1.11 (Acute toxicity), (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Triton X-100 | Sigma | T9284 | Step 2.1.4, 2.1.11 (Acute toxicity, hazardous to the environment) | ||||||||||||||||||||||||||||||||||||
| 10 x PBS | Life Technologies (GIBCO) | 70011-036 | Step 2.1.4, 2.1.5, 2.1.6, 2.1.8, 2.1.10, 2.1.11, 2.1.12, 2.2.7, 2.2.9, | ||||||||||||||||||||||||||||||||||||
| Glycerol | Fisher Scientific | G/0650 | Step 2.1.6 | ||||||||||||||||||||||||||||||||||||
| HCl | VWR | 20252 | Step 2.1.9 (Acute toxicity, corrosive) | ||||||||||||||||||||||||||||||||||||
| Formamide | Sigma Aldrich | 47670 | Step 2.1.14, 2.2.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SSC | Step 2.1.14, 2.2.2-2.2.6, 2.2.8 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 22 | Menzel-Glaser | BB022022A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 50 | Menzel-Glaser | BB022050A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Rubber cement | Marabu | 2901 (10 000) | Step 2.1.15 (Flammable, health hazard, danger to the environment) | ||||||||||||||||||||||||||||||||||||
| DAPI | Invitrogen Molecular Probes | D3571 | Step 2.2.8 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SlowFade Gold | Invitrogen | S36936 | Step 2.2.10 (mounting medium) | ||||||||||||||||||||||||||||||||||||
| Clear nail polish | Any supplier | Step 2.2.10 | |||||||||||||||||||||||||||||||||||||
| Equipment | |||||||||||||||||||||||||||||||||||||||
| Nanodrop 2000 | Thermo Scientific | Step 1.1.6, 1.2.4 (microvolume spectroscopy) | |||||||||||||||||||||||||||||||||||||
| Typhoon FLA 7000 phosphoimager | GE Life Sciences | Step 1.2.4 | |||||||||||||||||||||||||||||||||||||
| Coplin jars | Sigma-Aldrich | S6016 (6EA)/S5516 (6EA) | Step 2.1.3 onwards | ||||||||||||||||||||||||||||||||||||
| Liquid nitrogen dewar | Any supplier | Step 2.1.7 | |||||||||||||||||||||||||||||||||||||
| Base with Black Lid (light-tight chamber) | Simport | M920-2 | Step 2.1.17 | ||||||||||||||||||||||||||||||||||||
| Thermomixer comfort | Eppendorf | 5355 000.011 | Step 2.2.3 (or use shaker in a 37 °C room) | ||||||||||||||||||||||||||||||||||||
| Imaging and Image processing | |||||||||||||||||||||||||||||||||||||||
| Metafer - Imaging Automation Platform | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| MetaCyte - Automated Interphase FISH analysis | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| Axio Imager Z2 upright | Zeiss | epifluorescence microscope used with automated imaging | |||||||||||||||||||||||||||||||||||||
| IX81 confocal microscope (FV1000) | Olympus | confocal microscope | |||||||||||||||||||||||||||||||||||||
| Bitplane, version 7.3 | Imaris | image analysis and 3D modelling software | |||||||||||||||||||||||||||||||||||||
| Scientific volume Imaging, version 4.1 | Huygens | image analysis and deconvolution software | |||||||||||||||||||||||||||||||||||||
FISH buffers and reaction mixes
| |||||||||||||||||||||||||||||||||||||||
参考文献
- Chakalova, L., Debrand, E., Mitchell, J. A., Osborne, C. S., Fraser, P. Replication and transcription: shaping the landscape of the genome. Nat. Rev. Genet. 6, 669-677 (2005).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (1016).
- de Wit, E., de Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes Dev. 26, 11-24 (2012).
- Sambrook, J., Russell, D. . Molecular Cloning: A laboratory Manual. , (2000).
- Boyle, S., Rodesch, M. J., Halvensleben, H. A., Jeddeloh, J. A., Bickmore, W. A. Fluorescence in situ hybridization with high-complexity repeat-free oligonucleotide probes generated by massively parallel synthesis. Chromosome Res. 19, 901-909 (2011).
- Brown, K. Visualizing nuclear proteins together with transcribed and inactive genes in structurally preserved cells. Methods. 26, 10-18 (2002).
- Hewitt, S. L., Yin, B., et al. RAG-1 and ATM coordinate monoallelic recombination and nuclear positioning of immunoglobulin loci. Nat. Immunol. 10, 655-664 (2009).
- Solovei, I., Cavallo, A., et al. Spatial preservation of nuclear chromatin architecture during three-dimensional fluorescence in situ hybridization (3D-FISH. Experimental Cell Research. 276, 10-23 (2002).
- Solovei, I., Beatty, B., Mai, S., Squire, J. FISH on three-dimensionally preserved nuclei. FISH: A Practical Approach. , (2002).
- Krueger, C., King, M. R., et al. Pairing of homologous regions in the mouse genome is associated with transcription but not imprinting status. PLoS One. 7, e38983 (2012).
- Solovei, I., Cremer, M., Bridger, J. M., Volpi, E. V. 3D-FISH on Cultured Cells Combined with Immunostaining. Fluorescence in situ hybridization (FISH): Protocols and Applications. , (2010).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。