Method Article
Robuste FISH 3D DNA Utilisation des sondes directement marquées
Dans cet article
Résumé
Nous décrivons un protocole robuste et polyvalent pour l'analyse de l'architecture nucléaire par FISH d'ADN 3D à l'aide de sondes fluorescentes directement marqués.
Résumé
FISH 3D DNA est devenu un outil important pour analyser organisation tridimensionnelle du noyau, et plusieurs variantes de la technique ont été publiés. Dans cet article, nous décrivons un protocole qui a été optimisé pour la robustesse, la reproductibilité et la facilité d'utilisation. Vives fluorescentes sondes directement marqués sont générés par translation de coupure avec des amino-allyldUTP suivie d'un couplage chimique du colorant. FISH d'ADN 3D s'effectue à l'aide d'une étape de congélation-décongélation pour la perméabilisation de cellules et une étape de chauffage pour la dénaturation simultanée de la sonde et de l'ADN nucléaire. Le protocole est applicable à toute une gamme de types de cellules et une variété de sondes (BAC, plasmides, fosmides, ou des peintures chromosomiques entiers) et permet une imagerie automatisé à haut débit. Avec cette méthode, nous étudions systématiquement localisation nucléaire de trois régions chromosomiques.
Introduction
ADN hybridation in situ fluorescente (FISH d'ADN) permet la visualisation en trois dimensions de locus de gènes individuels, des domaines sous-chromosomique et des chromosomes entiers, même au cours de toutes les phases du cycle cellulaire. FISH 2D est utilisée pour des études métaphase tandis que les poissons 3D a été largement utilisé pour sonder la relation entre l'organisation spatiale du génome et sa fonction durant l'interphase (1,2 et références citées). Historiquement, les études de co-association ont été réalisées en enquêtant sur des dizaines à des centaines de loci individuels par les poissons. Plus récemment puissant techniques 3C basés à haut débit tels que les 4C et Salut-C ont été développés 3, permettant à l'enquête de la biologie moléculaire diaphonie entre des milliers de différents loci. Bien que les techniques 3C basés et les poissons d'ADN peuvent être des méthodes complémentaires, ils ne répondent pas nécessairement aux mêmes questions. Méthodes basées 3C offrent une lecture de l'ensemble des populations de cellules mixtes, ce qui entraîne une probabilitélité pour les co-associations. En revanche, si faible débit, les techniques de base de POISSONS offrent la possibilité d'analyser les dispositions spatiales des lieux ou des chromosomes dans les cellules individuelles en fonction de leurs différentes étapes du cycle de développement ou de la cellule. Par conséquent, FISH continuera d'être un important outil pour sonder les relations structure-fonction nucléaires.
Il ya deux considérations majeures dans la réalisation d'expériences de 3D Fish succès. Ce sont: 1. l'obtention des sondes marquées de manière optimale et 2. choix des traitements cellulaires, y compris la fixation, les étapes de pré-et de post-hybridation, pour préserver la morphologie nucléaire autant que possible tout en faisant ADN suffisamment accessible pour l'hybridation de la sonde. L'étiquetage de la sonde efficace est d'une importance cruciale pour les poissons. Traditionnellement, translation de coupure a été utilisé pour introduire soit haptène ou nucléotides fluorophore conjugué à 4. De même, les kits pseudo-traduction commerciaux sont disponibles pour haptène directe ou l'incorporation des fluorophores, but aussi pour les deux étapes en utilisant l'étiquetage nucléotides aminoallyl et des colorants réactifs aux amines. Ce dernier rend incorporation colorant plus efficace en donnant l'ADN polymérase une molécule moins encombrant pour travailler avec. Plus récemment, des kits pour l'étiquetage non enzymatique de l'ADN ont été développés qui exploitent liaison de coordination de platine aux acides nucléiques. sondes FISH peuvent même être achetés déjà marqué 5. Alors que les kits et les sondes fabriquées commercialement sans doute donner la facilité d'utilisation, ils sont nettement plus cher que d'acheter les composants individuels et la production de sondes en interne. Nous avons optimisé un protocole à faible coût nick de traduction afin de marquer directement plusieurs sondes BAC en plusieurs couleurs. Nous avons découvert que l'obtention d'ADN de BAC très pur est essentiel et se traduit par une exigence pour seulement 10-20 ng de la sonde par diapositive FISH, comparativement à 10 - 20 fois plus lors de la matrice ADN impur est utilisé, résultant en majeure coût et du temps économies. L'utilisation d'amino-allyldUTP permet étiquetage flexiblesondes à disposition colorants amine réactifs (par exemple Alexa Fluor ou Cy-colorants) ou haptènes (par exemple, la biotine, la digoxigénine). Sondes haptène marqués sont détectés par des anticorps conjugué à un fluorophore ou la streptavidine pour amplifier et visualiser le signal. Lumineux sondes directement marquées montrent généralement moins fond coloration et éviter la sur-amplification de signaux, d'où résulte une représentation plus précise de la localisation nucléaire et la morphologie du locus.
Il existe plusieurs techniques de poissons qui utilisent différents fixateurs, les pré-traitements et lavages post-hybridation mais ceux-ci tombent généralement dans les catégories suivantes: fixation de glutaraldéhyde avec NaOH dénaturation, la fixation de formaldéhyde avec HCl dénaturation, et la fixation de formaldéhyde avec dénaturation thermique 6-9 . Chacun d'entre eux présente des avantages et des inconvénients. Glutaraldehyde résultats de fixation en bon état de conservation structurelle nucléaire, mais nécessite un traitement avec des agents réducteurs de minimiserl'auto-fluorescence qui en résulte, et traitement à la soude doit être soigneusement contrôlée pour équilibrer suffisante dénaturation de l'ADN et les dommages potentiels à la structure nucléaire 6. fixation de formaldéhyde est moins forte, ce qui donne une probabilité accrue de perturbations de l'architecture nucléaire, mais aussi généralement de donner des signaux forts et plus fiables et les plus faibles autofluorescence 9. Traitement HCl depurinates ADN et supprime les protéines, offrant un bon accès à l'ADN pour les sondes, mais peut aussi introduire des cassures de l'ADN. Chauffage sépare physiquement les deux brins d'ADN résultant de bonne hybridation de la cible et des signaux forts, mais peut provoquer une certaine perturbation de la structure nucléaire 8. La mesure dans laquelle chacune de ces techniques affecte épitopes des protéines varie largement 6,9, ce qui entraîne l'obligation de déterminer expérimentalement pour chaque protéine de protocole optimal à utiliser dans les expériences d'immuno-FISH.
Bien qu'il n'existe pas «parfait» DNA FISH technique, ils peuvent tous être utile si elle est bien maîtrisée. Notre objectif était d'optimiser un protocole robuste et reproductible FISH d'ADN pour étudier le positionnement spatial de plusieurs loci dans une variété de types de cellules 10, en se concentrant sur la méthode de chauffage pour la plupart des signaux fiables. Avec cela et l'utilisation d'un système d'imagerie automatisée nous avons cherché à augmenter le débit pour l'analyse cellulaire unique des arrangements nucléaires.
Protocole
1. Générer des sondes d'ADN marquées directement par Nick Translation
Note: les sondes directement marquées fabriqués à partir de BAC (100-250 kb) produisent constamment des signaux lumineux. Si les sondes plus petites sont nécessaires, utiliser fosmides (40-50 kb) ou même des plasmides contenant 5-10 inserts ko. Identifier BAC ou clones fosmide correspondant à des gènes spécifiques à l'aide Ensemble ou navigateurs de génome UCSC. Préparer l'ADN BAC haute qualité par précipitation répétée ou utiliser des kits disponibles dans le commerce. Le moins que l'établissement est contaminé par l'ADN génomique bactérienne, moins la sonde est nécessaire par diapositive. Effectuer l'étiquetage en deux étapes: traduction nick introduction aminoallyl-dUTP et le couplage chimique d'un colorant amine-réactif.
1.1. Nick Translation
Lors de la traduction nick, DNase I est utilisé pour créer un seul brin pauses. ADN polymérase I s'allonge 'extrémités de ces «pseudos» le 3, en remplacement de nucléotides existants par de nouveaux, t présente «traduire» le nick et offrant ainsi la possibilité d'intégrer nucléotides marqués. Aminoallyl-dUTP est choisi en raison de l'incorporation efficace de l'ADN polymérase I et son potentiel de couplage chimique tard aux colorants réactifs avec les amines ou les haptènes. Il est essentiel de trouver le juste équilibre entre DNase I entaille et l'ADN polymérase de traduction I; trop DNase I se traduira par digestion de l'ADN excessive donnant un faible rendement et tailles de fragments courts, trop peu ne sera pas produire suffisamment de pseudos pour la polymérase pour initier la traduction, donnant grande taille de fragment et une mauvaise incorporation de aminoallyl-dUTP. Le protocole suivant fonctionne bien, mais il peut être nécessaire de titrer la DNAse I à partir de différents lots ou par plusieurs fabricants.
- Mettre en place l'étiquetage réaction sur glace et incuber pendant 2 heures à 16 ° C. Cela devrait produire de l'ADN translation de coupure 0,5-1 mg et peut être intensifié. Diluer DNase I 1:30 tampon DNase I (soit 0,3 U / pl).
- Exécuter 1 pi sur un gel à 2% d'agarose pour vérifier la taille des fragments marqués. Pendant que vous courez le gel, garder la réaction sur la glace. Succès nick translatisur se traduira par un frottis avec la majeure partie des fragments de fonctionnement entre 150 pb et 700 pb avec quelques gros fragments à ~ 1 kb (étape cruciale: voir résultats représentatifs). Si nécessaire, ajouter un autre 1 pi d'une fraîche 01:30 DNase I dilution et incuber à 16 ° C pendant 15-30 min. Le temps d'incubation varie en fonction de la quantité et de la qualité de l'ADN BAC.
- Inactiver la DNase I par chauffage à 75 ° C pendant 10 min.
- ADN modifié par une amine Clean-up utilisant un kit de purification PCR. Eluer dans 100 pi H 2 O.
- Ethanol ADN précipité par addition de 10 pl NaOAc 3 M (pH 5,2) et 275 pi d'éthanol. Laisser à -20 ° C pendant au moins 1 heure ou jusqu'au lendemain. Faites tourner à vitesse maximale pendant 30 min à 4 ° C. Lavez culot avec 500 ul d'éthanol à 70% et laisser sécher. Cette étape supprime amines traces qui vont interférer avec la réaction de marquage. Remettre le culot dans 6 pi H 2 O par 1x réaction initiale.
- Utilisation 1 pi à déterminer la concentration de l'ADN modifié par une aminepar spectroscopie UV micro-volume.
1.2. Le couplage du colorant fluorescent
Marquage fluorescent de la sonde est obtenue par couplage chimique du colorant. Alexa Fluor colorants succinimidyl ester réagissent avec les amines de l'ADN modifié amino-allyldUTP pour former une liaison covalente, en produisant ainsi des sondes marquées directement. Alexa Fluor 488, 555, 647 et colorants ont été couronnés de succès pour ce processus.
- Ajustez 4 pg d'ADN modifié amino-allyle à un volume de 5 pi, la chaleur à 95 ° C pendant 5 min, enclenchez refroidir sur glace et ajouter 3 ul de 0,2 M NaHCO3.
- Dissoudre une aliquote de colorant réactif amine dans 2 ul de DMSO anhydre à température ambiante. Ajouter 8 ADN modifié ul avec NaHCO 3, vortex, impulsion de spin et incuber à température ambiante dans l'obscurité pendant 1 heure.
Remarque: Les colorants réactifs amine peuvent être utilisées pour marquer plus d'une sonde sur la base du schéma suivant. Depuis oeul 10-20 ng de sonde est utilisée pour l'étiquetage des diapositives, 1 ug donne assez de sonde pour 50-100 diapositives. Il est également possible de stocker l'ADN modifié par une amine pour les réactions futures en matière d'étiquetage à -20 ° C.
| quantité d'ADN | le volume de l'ADN | 0,2 M NaHCO3 | Volume de colorant | Totale | Assez pour |
| 4 x 1 pg | 1,25 pi de chacun | 0,75 pi de chacun | 0,5 pi de chacun | 2,5 pi (1/4) | 50-100 diapositives chacun |
| 3 x 1,35 ug | 1,67 pi de chacun | 1 pl chaque | 0,67 pi de chacun | 3,34 pl (1/3) | 65-130 glisser chaque |
| 2 x 2 ug | 2,5 pi chaque | 1,5 pi de chacun | 1 pl chaque | 5 pi (1/2) | 100-200glisse chaque |
| 1 x 4 pg | 5 pl | 3 pi | 2 pl | 10 pi (complet) | 200-400 diapositives |
- Ajouter 90 ul H 2 O et sonde marquée purifier l'aide d'un kit de purification PCR. Eluer dans 100 pi 10 mMTris-Cl (pH 8,5).
- Déterminer la concentration de la sonde et l'efficacité de l'étiquetage par spectroscopie à spectre complet microvolume (voir Figure 2) en utilisant les équations de Beer-Lambert (Les instructions sont données dans la fiche technique accompagnant les Fluor colorants réactifs Alexa ou peut être trouvé en ligne). Sondes contenant des colorants 3-6 par 100 pb fonctionnent bien. Sondes à des degrés moindres de incorporation peuvent donner des signaux de poissons faibles. Vous pouvez également exécuter 5 pi sur un gel d'agarose à 2% sans tache d'ADN et vérifier l'incorporation du colorant fluorescent à l'aide d'un phosphoimager. Diffusez-tacher le gel avec du bromure d'éthidium ou alternatives de coloration de l'ADN et comparer ADN marqué à l'ADN total.
Par l'intermédiaire des étapes suivantes, les cellules d'intérêt sont fixés à des lames et perméabilisées. L'ADN cellulaire et les sondes directement marquées sont ensuite dénaturés ensemble sur une plaque chauffante et hybrides pendant la nuit dans une chambre humidifiée étanche à la lumière.
2.1. Jour 1
- Sonde Précipitations
Note: les sondes de précipiter à un certain moment durant le jour 1. Cela peut confortablement être effectuées en parallèle au traitement des cellules et ne pas avoir à faire au début de la journée.- Mélanger 10-20 ng de la sonde directement étiqueté, 6 pi 0 C t-1 DNA (6 mg), 1 pl ADN simple brin à partir de testicules de saumon (9,7 ug) et ajuster le volume à 100 pi avec de l'eau.
- Ajouter 10 ul NaOAc 3 M et 275 ul EtOH, et mélanger au vortex. Précipiter pendant au moins 1 heure à -20 ° C.
- Tournez à centrifuger à vitesse maximale pendant 30 min à 4 ° C. Lavez culot avec 70% EtOH et tourner à nouveau. Granulés secset remettre en suspension dans 5 pi formamide désionisée. Remettre en suspension pendant 30 min à 37 ° C tout en agitant à 1000 rpm abri de la lumière (lieu feuille sur le thermomixer).
- Ajouter 5 ul de sulfate de dextran mélange et agiter pendant encore 10 min à 37 ° C à nouveau abri de la lumière. Introduire à la pipette de haut en bas, juste avant le pipetage sur lamelle.
Note: Pour la peinture des chromosomes combiné avec des sondes BAC, entiers sondes de peinture chromosomiques qui sont fournis dans le prêt-à-utiliser mélange d'hybridation sont une option pratique. Remettre en suspension précipitée BAC sonde (avec C 0 t1 ADN et l'ADN simple brin à partir de testicules de saumon) dans 10 ul mélange de peinture chromosomique par aspiration et refoulement. Incuber à 37 ° C tout en agitant à 300-500 rpm pendant 10 min. Utilisez 10 pl du chromosome peinture / BAC sonde mélange par spot.
- Fixation des cellules aux diapositives
Note: Les cellules propension à adhérer aux diapositives varie considérablement selon le type de cellule. Pour chaque type de cellule, déterminer l'optimale du temps et de la densité des cellules de décantation de façon empirique. Pour obtenir des résultats cohérents, les cellules doivent être en suspension cellulaire unique.
Pour plus de convivialité cercle de la zone des cellules sera repéré sur avec un stylo hydrophobe, bien que pour les cellules remises en suspension dans PBS ce n'est pas strictement nécessaire. Une barrière hydrophobe empêche la suspension de la diffusion sur la diapositive, conserve jusqu'à trois échantillons par lame bien séparées et permet l'utilisation de très petits volumes de solution d'anticorps en immuno-FISH. En outre, la zone spotting peut être réduit au minimum si le nombre de cellules est faible. - cellules de foie foetal de souris
- Recueillir chaque foie fœtal de souris E13.5 dans environ 1,5 ml de PBS et remettre en suspension par pipetage de haut en bas. Tournez à centrifuger à 3000 rpm pendant 2 min. Répétez les lave trois fois. Lors des dernières lavage, cellules souches à travers un tamis de 70. Reprendre les cellules d'un foie dans 160 pi de PBS et utiliser 50 μ par spot. Laisser les cellules sédimenter pendant 2 min à température ambiante.
- Mouse cellules ES
1. Cellules ES de souris ne collent pas facilement à la diapositive. Dans une diapositive, encerclez le domaine des cellules sera repéré sur avec un stylo hydrophobe. Préparer une suspension de cellules par 20,000-50,000 80-100 ul dans un milieu de culture normale, pipette en cercle et laisser attacher pendant environ 3 heures dans un incubateur humidifié. Remarque: Alternativement, les cellules ES peuvent être cultivées sur des lames, mais constituera alors colonies qui pourraient entraver imagerie automatisée. - Les cellules fixes
1. Certaines expériences nécessitent l'utilisation de cellules fixes qui ne s'attachent pas à des diapositives par eux-mêmes. Filature douce à 300 rpm pendant 3 minutes en utilisant un cytocentrifugeuse préserve la structure tridimensionnelle des cellules et assure que les cellules ne tombent pas.
| Type de cellule | Suspension en | Temps de stabilisation | Commenter |
| Sourisfoie fœtal | PBS | 2 min | Sur le banc |
| Souris ES | DMEM avec des suppléments | 4 heures | 37 ° C en incubateur humidifié |
| lymphocytes de souris | PBS | 2 min | Sur le banc |
| Souris P20 cellules germinales | Correction en solution | Cytospin | Sur le banc |
- Pour fixer les cellules, doucement submerger diaporamas dans 4% PFA (ATTENTION: effectuer dans la hotte) pendant 10 min. Fixer les cellules avec la lame à plat dans un tiroir pour une meilleure rétention des cellules (par exemple 50 ml 4% PFA dans le couvercle d'une boîte de pointe). Remarque: Cette étape ne s'applique pas aux cellules fixées avant la fixation des lames.
Pour toutes les étapes ultérieures, utiliser 50 ml ou 100 ml jarres de Coplin. Prenez soin de ne pas égratigner les cellules lorsque vous déplacez des diapositives entre pots.
- Étancher dans 0,1 M Tris-Cl, pH 7,4 pendant 10 min à température ambiante.
- Perméabiliser cellules dans 0,1% saponin/0.1% de Triton X-100 dans du PBS pendant 10 min à température ambiante.
- Laver deux fois dans du PBS pendant 5 min à température ambiante.
- Incuber pendant au moins 20 min dans 20% de glycérol / PBS à température ambiante. Remarque: À ce stade, les lames peuvent être stockées dans 50% de glycérol / PBS à -20 ° C pendant au moins plusieurs semaines. Il a été observé que le stockage d'au moins une journée en résulte une augmentation de la puissance du signal 9. Recalibrer la température ambiante de 20% de glycérol / PBS avant de procéder. Note: Ceci est un point commode de commencer la précipitation des sondes (voir 2.1.1).
- 3x gel / dégel dans l'azote liquide. Immerger une diapositive à la fois dans de l'azote liquide à l'aide d'un petit vase de Dewar. Retirer au bout de quelques secondes et un claquement caractéristique, et placer sur une serviette en papier pour le dégivrage. Attendez que le glycérol congelés opaque à disparaître ensuite geler à nouveau. Diapositives jusqu'à 15 peuvent être réalisées confortablement en rotation pendant trois cycles de gel / dégel.
- WAsh deux fois dans du PBS pendant 5 min à température ambiante.
- Incuber dans HCl 0,1 M pendant 30 min à température ambiante.
- Laver dans du PBS pendant 5 min à température ambiante.
- Perméabiliser à 0,5% saponin/0.5% X-100/PBS Triton pendant 30 min à température ambiante.
- Laver deux fois dans du PBS pendant 5 min à température ambiante.
- Amener à 50% formamide/2x SSC pendant au moins 10 min à température ambiante.
- Sonde pipette sur une lamelle. Sortir les lames du pot et sécher tout excès de liquide autour de la place de la cellule (s) avec une serviette en papier. En séchant rapidement formamide, prenez soin de ne pas se dessécher cellules. Pour les diapositives avec deux ou trois points de la cellule, appliquez 10 pi de sonde par spot sur un 22 mm x 50 mm lamelle correspondant à repérer emplacement de la diapositive. Inversez lamelle sur la glissière sur place (s). Sceller avec du ciment de caoutchouc et Laisser sécher complètement. Protéger de la lumière à toutes les étapes ultérieures.
- Chauffer les lames à 78 ° C pendant exactement 2 minutes sur une plaque chauffante (par exemple un bloc de chauffage inversé, étape cruciale: voir Discussion). Placez un couvercle (boîte en carton ou sim ILAR) sur la plaque chauffante à l'abri de la lumière pendant la dénaturation.
- Incuber une nuit à 37 ° C dans une chambre humidifiée étanche à la lumière. Pour humidifier, placer des serviettes en papier dans une boîte étanche à la lumière et humidifier avec de l'eau.
2.2 Jour 2
- Préparer des jarres de Coplin avec des solutions à la bonne température pour les étapes de lavage ultérieures. Protéger les diapositives de la lumière autant que possible au cours de toutes les étapes ultérieures. Un café peut fait un bon couvercle étanche à la lumière pour les bocaux Coplin.
- Décollez la colle de caoutchouc et le lieu diapositive dans 2x SSC jusqu'à lamelles desserrer et glisser.
- Laver à 50% formamide/2x SSC pendant 15 min à 45 ° C. Placer le couvercle sur bain-marie à l'abri de la lumière.
- Laver à 0,2 x SSC pendant 15 min à 63 ° C.
- Laver à 2x SSC pendant 5 min à 45 ° C.
- Laver à 2x SSC pendant 5 min à température ambiante.
- Laver dans du PBS pendant 5 min à température ambiante.
- Colorer avec DAPI (5 ug / ml dans du SSC 2x) pendant 2 min dans un bac à coloration à température ambiante.
- Décolorer dans du PBS pendant 5 min à température ambiante.
- Pour monter la lamelle, pipette milieu de montage sur une lamelle. Utilisez 10 pi par endroit de la cellule, en utilisant 22 mm x 22 mm lamelles pour un seul endroit, et de 22 mm x 50 mm lamelles pendant deux à trois points. Séchez PBS autour de la place de la cellule, autant que possible, mais attention à ne pas assécher les cellules. Inversez lamelle sur la glissière, et sceller avec du vernis à ongles.
Résultats
Le protocole décrit ici (Figure 1 pour un aperçu) a été utilisé à bon escient pour combiner FISH d'ADN avec capture d'image haut débit pour l'analyse de l'organisation génomique. La génération de sondes de bonne qualité est essentiel à la réussite (voir la discussion). Figure 2 montre deux contrôles de qualité importants pour les sondes FISH. Après translation de coupure, un test d'ADN devrait être visible avec la majeure partie des fragments fonctionnant entre 150 et 700 pb (figure 2A). Après couplage chimique, l'incorporation du colorant fluorescent peut être mesuré par analyse spectroscopique de la sonde (figure 2B).
Lors de l'utilisation de bonnes sondes, suivant le protocole de FISH d'ADN se traduit généralement par signaux FISH d'ADN lumineux sur un fond de basses. Nous avons utilisé avec succès ce protocole sur une variété de différents types de cellules et à celles des microscopes épi-fluorescence ou confocal. Imagerie automatisée en utilisant un épi-fluorescence microscope nécessite pointus, signaux FISH facilement identifiables avec peu de fond (figure 3), tandis que la microscopie confocale nécessite des signaux plus intenses. figure 4A montre des images traitées représentatives. Coordonnées nucléaires de signaux FISH peuvent être obtenus, et les relations spatiales des régions génomiques peuvent être calculées (pour un exemple, voir la figure 4B). Nous avons également utilisé des piles d'images obtenues par microscopie confocale pour la modélisation 3D des signaux FISH au sein de leurs territoires chromosomiques (Film 1).
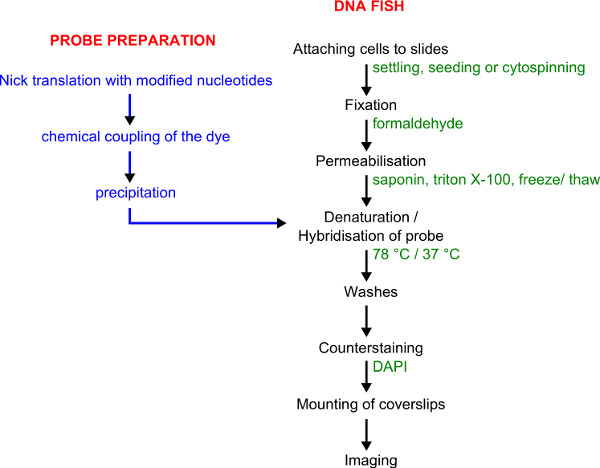
Figure 1. Flux de travail pour l'étiquetage de la sonde et les poissons d'ADN. Préparation de la sonde est représentée en bleu. procédure FISH d'ADN est représenté en noir avec détails donnés dans le vert.
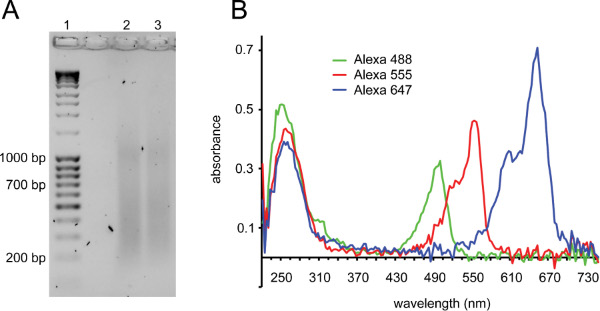
Figure 2. Marquage des sondes par translation de coupure et de contrôle de la qualité de sondes marquées. A) Gel montrant frottis idéal avant de chauffer à inactiver DNase I (piste 1: échelle d'ADN, piste 2 et 3: taux d'alcoolémie après le pseudo-traduction). Fragments devraient être dans la gamme 150-700 pb. Un frottis supplémentaire à ~ 1 kb est fréquemment observée avec succès réactions pseudo-traduction. B) l'analyse du spectre complet Microvolume de sondes marquées. Un pic d'ADN à 260 nm est observée avec un second pic correspondant à la longueur d'onde d'absorption du fluorophore de la sonde est marquée avec: Alexa Fluor 488: 495 nm, Alexa Fluor 555: 555 nm, Alexa Fluor 647: 650 nm. Indiqués sont les trois sondes ayant une bonne incorporation: pics d'absorption des fluorophores sont d'une hauteur supérieure ou les pics d'ADN similaire. Alexa Fluor 647 a généralement absorbance supérieureque Alexa Fluor 555, qui à son tour a une absorbance supérieure à Alexa Fluor 488. Alexa Fluor 488 absorbe également la lumière à 260 nm, d'où le premier pic pour cette sonde est plus élevé que les autres. Sondes à faible incorporation spectacle absorbance de fluorophore 2-3 fois plus faible que l'absorption de l'ADN. Sondes avec un plus de 2 fois pic de fluorophore supérieur ADN pic contiennent généralement colorant non conjugué qui peut causer fond dans le poisson.
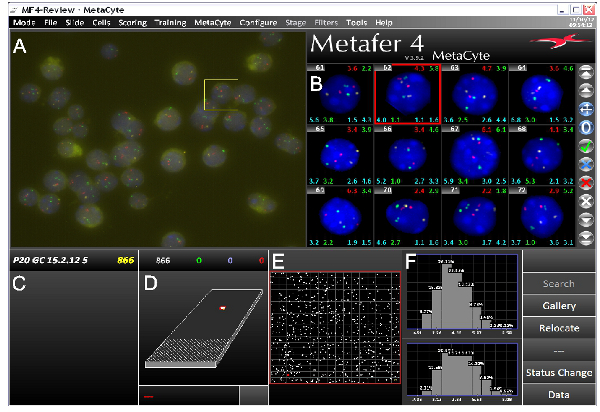
Figure 3. Capture d'écran du logiciel d'imagerie 3D Fish MetaCyte montrant une expérience typique de poisson trois couleurs. L'écran est divisé en plusieurs parties. A) champ de vision (FOV) Raw image capturée par la Metafer / système de numérisation de diapositives MetaCyte. B) Galerie des images de noyaux qui ont identifié l'ONUtraitement d'image dergone et segmentation des signaux de poissons. Différents types de données peuvent être affichées sur chaque image de la galerie. Dans cet exemple, le nombre de cellules est indiqué en haut à gauche avec divers allélique et interlocus distances en haut à droite et de chaque côté en dessous de chaque image. C) nom de la diapositive avec le nombre de noyaux identifiés dans l'analyse. D) Représentation de la lame avec la zone balayée par l' système. E) L'élargissement de la zone numérisée. Des taches blanches représentent des noyaux identifiés par FOV. F) Les données traitées. Dans cet exemple allélique distances maximales entre les signaux verts (en haut) et les signaux rouges (en bas) sont affichés. Les données présentées proviennent de souris P20 cellules germinales. Les sondes sont situés sur des chromosomes différents, couvrant Hbb et les gènes HBA et un cluster histone. Cliquez ici pour agrandir la figure .
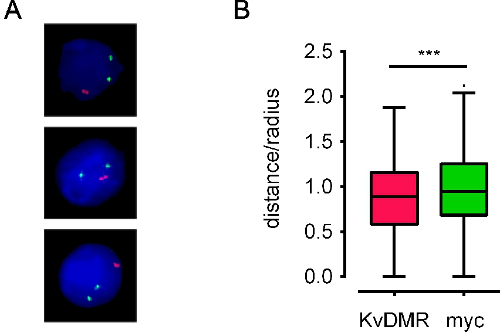
Figure 4. Exemples d'images traitées et les données de position dérivés. A images) transformés pour E13.5 souris des cellules de foie foetal colorées avec des sondes BAC situés sur différents chromosomes couvrant Myc et KCNQ1 gènes (appelées myc et KvDMR, rouge et vert, respectivement). B) Tukey parcelles boîte à moustaches représentant la distance allélique la distribution, pour les mêmes loci dans 600 noyaux. La distance allélique est tracée en une fraction du rayon du noyau (distance / rayon) pour tenir compte des variations dans la taille nucléaire. les noyaux des cellules ES ont un rayon de l'ordre de 5 um. Sondes rouges sont beaucoup plus rapprochés que les sondes vertes (***: p <0,01, test t apparié). Les données de 10.
Film 1. La modélisation en 3D de deux loci dans leur chromosome territory. Deux sondes BAC situés à proximité de chaque extrémité du chromosome de la souris 7 ont été marqués (rouge et rouge lointain, (blanc pseudo-couleur)) et hybridés avec toute une peinture chromosomique (vert) pour les cellules ES de souris. Cliquez ici pour voir le film .
Discussion
FISH 3D DNA est devenu un outil largement reconnu pour analyser les arrangements spatiaux dans le noyau. Alors que le poisson fournit des résultats visuels et direct, comme avec toutes les techniques, il faut être conscient de ses limites. FISH d'ADN est confronté au problème inhérent que les traitements relativement sévères sont nécessaires pour rendre la chromatine accessible pour les sondes FISH qui finalement perturbe nucléaire structure la chose qui doit être analysé. Plusieurs stratégies ont été poursuivis pour jongler avec l'accessibilité et la préservation de la structure. Dans le protocole décrit ici, l'ADN chromosomique est rendue accessible par gel-dégel perméabilisation des cellules et la dénaturation thermique avant l'hybridation de la sonde. Solovei, et al., (2002) a analysé les changements structurels après un traitement similaire et a constaté que le déplacement moyen de domaines chromatine était de 300 nm, ce qui limite la résolution de l'analyse structurelle à environ 1 Mb régions dans le génome 8. Si ce n'est pas haute résolution, ilest une échelle raisonnable pour analyse de la situation nucléaire.
Un examen plus pratique pour l'analyse FISH est que l'acquisition de l'image et des mesures de distances nucléaires sont des processus chronophages qui limitent le nombre de germes qui peuvent être analysés. Afin d'étudier les événements rares nous avons cherché à faire l'imagerie automatisé à haut débit. Puis, avec des contrôles appropriés en place, en nombre suffisant peuvent être comparés pour permettre une analyse statistique robuste des relations spatiales. Nous analysons régulièrement 600 noyaux par point de données pour lequel nous déterminons les coordonnées 3D de tous les signaux de pêcher dans le volume nucléaire. Ces données nécessitent très peu d'espace de stockage et permettent une analyse de distances inter-et intra-allélique, ou des positions radiales en tout point plus tard. En outre, le traitement automatisé peut être fait d'une manière aveugle des chercheurs qui réduit considérablement la probabilité de préjugés inconscients.
Pour permettre ce genre d'analys haut débitest, notre objectif était d'établir un moyen rapide et efficace d'effectuer FISH d'ADN. Nous avons trouvé le protocole décrit ici pour être extrêmement robuste, produisant régulièrement des signaux lumineux avec faible bruit de fond. Il a travaillé pour tous les types de cellules, nous avons essayé à condition que nous adaptons l'étape de fixation des cellules sur la lame. En outre, à une exception près, tous les taux d'alcoolémie, nous avons utilisé pourraient être transformés en des sondes vives. Nous avons également réussi à détecter un certain nombre de protéines nucléaires par coloration des anticorps après FISH d'ADN. Alors que pour certaines protéines Cela fonctionne bien, nous recommandons comparaison avec immunofluorescence traditionnel (IF) pour assurer la cohérence du modèle de coloration des anticorps entre SI et de l'ADN immuno FISH (cf. 11).
En règle générale, nous avons constaté que des sondes marquées de différentes couleurs produit les mêmes résultats. Toutefois, les aspects suivants devraient être considérés lors du choix du colorant de marquage. Tout d'abord, la couleur du fluorophore doit être bien détecté par le microscope used. D'autre part, la longueur d'onde de la lumière émise par les fluorophores doit être suffisamment différente pour éviter que l'encre à travers dans l'autre canal. Cela dépendra des filtres dans le microscope. Et troisièmement, au moins dans nos mains, certaines couleurs produisent des signaux lumineux plus uniforme que d'autres. Nous avons toujours utilisé DAPI (bleu) comme contre qui rend Alexa Fluor 555 (rouge), 488 (vert) et 647 (rouge lointain) de bons choix pour l'étiquetage des sondes. Grâce à notre système d'imagerie, nous avons trouvé Alexa Fluor 555 (rouge) pour produire les signaux brillants, suivis par Alexa Fluor 488 (vert). Alexa Fluor 647 (rouge lointain) présente l'inconvénient qu'il ne peut pas être détectée par l'œil humain et donc aucun signal peut être vu quand on regarde vers le bas le microscope. Ainsi, si cela FISH deux couleurs, nous recommandons la combinaison de colorants rouges et vertes.
Deux problèmes principaux peuvent se présenter: cellules de rincer les lames et les signaux faibles. Comment les cellules adhèrent bien à la diapositive est entr extrêmement variablestypes de cellules een, et le meilleur protocole pour régler / ensemencer les cellules doivent être déterminées. Nous avons utilisé avec succès soit chargé positivement ou lames recouvertes de poly-L-lysine (acheté prêt à l'emploi), mais on a découvert les cellules adhèrent globalement mieux lames recouvertes de poly-L-lysine. Nous avons également constaté que lorsque les cellules étaient des zones entières trop denses serait décoller comme une feuille, alors que l'imagerie automatisée a été entravée si les cellules étaient trop rares. Ainsi, si l'échantillon n'est pas trop précieux, des numéros de portables doivent être ensemencées pour déterminer la densité idéale. Si les cellules sont particulièrement sujettes à laver, un stylo barrière hydrophobe peut être utilisé, et pas de poisson peut être effectuée par pipetage soigneusement solutions directement sur la lame. Pipetage également réduit considérablement les volumes de réactifs nécessaires, mais prend beaucoup plus de temps lorsque vous faites plusieurs diapositives.
Les signaux faibles sont généralement dues à des sondes pauvres, et c'est bien la peine d'investir du temps dans la prise de bonnes. ADN BAC propre devrait être utiliserd comme matériau qui peut être obtenu par précipitation ou répétée des kits disponibles dans le commerce de départ. La contamination par de l'ADN génomique bactérienne affecte négativement la qualité de la sonde et une manutention prudente lors de la lyse cellulaire et de filtrage du lysat cellulaire est nécessaire pour le garder à un minimum. , Il est cependant pas nécessaire d'utiliser une étape de exonuclease à digérer l'ADN linéaire, car cela réduit considérablement le rendement. Marquage efficace de la sonde est crucial. Le contrôle de la qualité la plus importante pour cette étape est de vérifier la taille des fragments après translation de coupure (voir résultats représentatifs). Un «bon» frottis sera presque toujours dans une sonde de couleur vive. Cependant, nous avons constaté que de temps en temps un certain taux d'alcoolémie ne peut être traitée dans une bonne sonde, et un autre taux d'alcoolémie doit être choisi. Une autre étape importante est la dénaturation thermique. Si la température est trop basse, l'ADN ne sera que partiellement dénaturé et l'hybridation de la sonde sera compromise. Si la température est trop élevée, la structure nucléaire will être perturbé plus que nécessaire. Dans nos mains, 78 ° C sur un bloc chaud inversé est le compromis idéal, mais cela peut varier selon le bloc chaud respectif utilisé.
Nous avons eu de bons résultats en utilisant un milieu de montage Vectashield, mais a constaté que SlowFade or a moins d'arrière-plan et préserve le signal fluorescent pour plus longtemps. Nous ne recommandons pas l'utilisation d'un milieu de montage hard-set car nous avons trouvé que cela influe sur la structure 3D et de former des bulles d'air au fil du temps.
En conclusion, vive sondes fluorescentes directement marqués ainsi que le protocole de FISH d'ADN décrites ici offrent une solution efficace pour l'analyse robuste et rapide de l'architecture nucléaire qui est applicable à une grande variété de questions biologiques.
Déclarations de divulgation
La production de cet article a été partiellement financé par Carl Zeiss et METASYSTEMS.
Remerciements
Ce travail a été financé par le BBSRC et le Wellcome Trust. Nous tenons à remercier Simon Walker à l'aide de l'imagerie et Felix Krueger de l'aide à l'analyse bioinformatique.
matériels
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||||||||
| Reagent/Material | |||||||||||||||||||||||||||||||||||||||
| NTB buffer | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| DTT | Invitrogen | D-1532 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| dNTPs | Bioline | BIO-39025 | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Aminoallyl-dUTP | Ambion | AM8439 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNA Polymerase I | New England Biolabs | M02095 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNase I recombinant RNase free | Roche | 4716728001 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| QIAquick PCR purification kit | Qiagen | 28104 | Step 1.1.4, 1.2.3 | ||||||||||||||||||||||||||||||||||||
| NaOAc | VWR | 27653 | Step 1.1.5, Step 2.1.1.2 | ||||||||||||||||||||||||||||||||||||
| NaHCO3 | Sigma | S5761 | Step 1.2.1 | ||||||||||||||||||||||||||||||||||||
| Alexa Fluor Reactive Dye Decapacks for Microarray Applications | Invitrogen (Molecular Probes) | A32750, A32756, A32757 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| DMSO (anhydrous) | Sigma Aldrich | 276855 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| SYBR Safe DNA gel stain | Life Technologies | S33102 | Step 1.2.4 (alternative to ethidium bromide) | ||||||||||||||||||||||||||||||||||||
| Cot-1 DNA | Invitrogen | 18440-016 | Step 2.1.1.1 (Cot-1 DNA can be home-made in large quantities and works just as well) | ||||||||||||||||||||||||||||||||||||
| Single stranded DNA from salmon testes | Sigma | D7656 | Step 2.1.1.1 | ||||||||||||||||||||||||||||||||||||
| Deionised formamide | Sigma | F-9037 | Step 2.1.1.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| Dextran sulphate | Sigma Life Sciences | D8906 | Step 2.1.1.4 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| XMP painting probes | MetaSystems | 000000-0528-837 | Step 2.1.1.4 | ||||||||||||||||||||||||||||||||||||
| Poly-L-lysine coated slides | Sigma Aldrich | P0425 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| Hydrophobic pen (ImmEdge) | Vector Laboratories | H-4000 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| 70 μm sieve (Mouse f–tal liver cells only) | BD Falcon | 352350 | Step 2.1.2.1 | ||||||||||||||||||||||||||||||||||||
| PFA | Sigma Aldrich | P6148 | Step 2.1.3 (Flammable, corrosive, acute toxicity, health hazard) (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Tris-Cl | Step 2.1.3 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Saponin from Quillaja bark | Sigma Aldrich | 47036 | Step 2.1.4, 2.1.11 (Acute toxicity), (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Triton X-100 | Sigma | T9284 | Step 2.1.4, 2.1.11 (Acute toxicity, hazardous to the environment) | ||||||||||||||||||||||||||||||||||||
| 10 x PBS | Life Technologies (GIBCO) | 70011-036 | Step 2.1.4, 2.1.5, 2.1.6, 2.1.8, 2.1.10, 2.1.11, 2.1.12, 2.2.7, 2.2.9, | ||||||||||||||||||||||||||||||||||||
| Glycerol | Fisher Scientific | G/0650 | Step 2.1.6 | ||||||||||||||||||||||||||||||||||||
| HCl | VWR | 20252 | Step 2.1.9 (Acute toxicity, corrosive) | ||||||||||||||||||||||||||||||||||||
| Formamide | Sigma Aldrich | 47670 | Step 2.1.14, 2.2.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SSC | Step 2.1.14, 2.2.2-2.2.6, 2.2.8 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 22 | Menzel-Glaser | BB022022A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 50 | Menzel-Glaser | BB022050A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Rubber cement | Marabu | 2901 (10 000) | Step 2.1.15 (Flammable, health hazard, danger to the environment) | ||||||||||||||||||||||||||||||||||||
| DAPI | Invitrogen Molecular Probes | D3571 | Step 2.2.8 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SlowFade Gold | Invitrogen | S36936 | Step 2.2.10 (mounting medium) | ||||||||||||||||||||||||||||||||||||
| Clear nail polish | Any supplier | Step 2.2.10 | |||||||||||||||||||||||||||||||||||||
| Equipment | |||||||||||||||||||||||||||||||||||||||
| Nanodrop 2000 | Thermo Scientific | Step 1.1.6, 1.2.4 (microvolume spectroscopy) | |||||||||||||||||||||||||||||||||||||
| Typhoon FLA 7000 phosphoimager | GE Life Sciences | Step 1.2.4 | |||||||||||||||||||||||||||||||||||||
| Coplin jars | Sigma-Aldrich | S6016 (6EA)/S5516 (6EA) | Step 2.1.3 onwards | ||||||||||||||||||||||||||||||||||||
| Liquid nitrogen dewar | Any supplier | Step 2.1.7 | |||||||||||||||||||||||||||||||||||||
| Base with Black Lid (light-tight chamber) | Simport | M920-2 | Step 2.1.17 | ||||||||||||||||||||||||||||||||||||
| Thermomixer comfort | Eppendorf | 5355 000.011 | Step 2.2.3 (or use shaker in a 37 °C room) | ||||||||||||||||||||||||||||||||||||
| Imaging and Image processing | |||||||||||||||||||||||||||||||||||||||
| Metafer - Imaging Automation Platform | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| MetaCyte - Automated Interphase FISH analysis | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| Axio Imager Z2 upright | Zeiss | epifluorescence microscope used with automated imaging | |||||||||||||||||||||||||||||||||||||
| IX81 confocal microscope (FV1000) | Olympus | confocal microscope | |||||||||||||||||||||||||||||||||||||
| Bitplane, version 7.3 | Imaris | image analysis and 3D modelling software | |||||||||||||||||||||||||||||||||||||
| Scientific volume Imaging, version 4.1 | Huygens | image analysis and deconvolution software | |||||||||||||||||||||||||||||||||||||
FISH buffers and reaction mixes
| |||||||||||||||||||||||||||||||||||||||
Références
- Chakalova, L., Debrand, E., Mitchell, J. A., Osborne, C. S., Fraser, P. Replication and transcription: shaping the landscape of the genome. Nat. Rev. Genet. 6, 669-677 (2005).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (1016).
- de Wit, E., de Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes Dev. 26, 11-24 (2012).
- Sambrook, J., Russell, D. . Molecular Cloning: A laboratory Manual. , (2000).
- Boyle, S., Rodesch, M. J., Halvensleben, H. A., Jeddeloh, J. A., Bickmore, W. A. Fluorescence in situ hybridization with high-complexity repeat-free oligonucleotide probes generated by massively parallel synthesis. Chromosome Res. 19, 901-909 (2011).
- Brown, K. Visualizing nuclear proteins together with transcribed and inactive genes in structurally preserved cells. Methods. 26, 10-18 (2002).
- Hewitt, S. L., Yin, B., et al. RAG-1 and ATM coordinate monoallelic recombination and nuclear positioning of immunoglobulin loci. Nat. Immunol. 10, 655-664 (2009).
- Solovei, I., Cavallo, A., et al. Spatial preservation of nuclear chromatin architecture during three-dimensional fluorescence in situ hybridization (3D-FISH. Experimental Cell Research. 276, 10-23 (2002).
- Solovei, I., Beatty, B., Mai, S., Squire, J. FISH on three-dimensionally preserved nuclei. FISH: A Practical Approach. , (2002).
- Krueger, C., King, M. R., et al. Pairing of homologous regions in the mouse genome is associated with transcription but not imprinting status. PLoS One. 7, e38983 (2012).
- Solovei, I., Cremer, M., Bridger, J. M., Volpi, E. V. 3D-FISH on Cultured Cells Combined with Immunostaining. Fluorescence in situ hybridization (FISH): Protocols and Applications. , (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon