Method Article
直接標識プローブを用いた堅牢な3DのDNA FISH
要約
私たちは、直接標識蛍光プローブを用いた3次元のDNA FISHによる核のアーキテクチャを分析するための堅牢で多目的なプロトコルを記述。
要約
3DのDNA FISHは、核の三次元組織を分析するための主要なツールとなっており、技術のいくつかのバリエーションが発表されている。この記事では、堅牢性、再現性、および使いやすさのために最適化されたプロトコルを記述します。明るく蛍光直接標識プローブは、染料の化学的カップリングに続いてアミノallyldUTPたニックトランスレーションによって生成されます。 3D DNAのFISHは、細胞透過プローブと核DNAの同時変性のための加熱工程の凍結融解工程を用いて行われる。プロトコルは、細胞の種類とプローブの多様(たBAC、プラスミド、フォスミド、または全体の染色体塗料)の範囲に適用され、高スループットの自動イメージングを可能にします。この方法では、我々は日常的に3染色体領域までの核局在を調査。
概要
in situハイブリダイゼーション DNA蛍光(DNAのFISH)は、細胞周期のすべての段階で、個々の遺伝子座、subchromosomalドメインとさえ全体の染色体の三次元可視化を可能にします。 3D FISHが広く間期ゲノムおよびその機能の空間的組織(その中の1,2及び参考文献)との関係を調べるために使用されているが、2D FISHは、中期的研究のために使用される。歴史的には、共関連研究は、FISHにより、個々の遺伝子座数十〜数百を調べることにより行った。このような4CとのHi-Cのようなもっと最近強力なハイスループット3Cベースの技術は、異なる遺伝子座の何千もの間のクロストークの分子の調査を可能にし、3を開発してきた。 3Cベースの技術とDNA FISHが相補的な方法になることができますが、それらは必ずしも同じ質問に答えていない。図3Cに基づく方法はた確率で、その結果、混合細胞集団のアンサンブル読み出しを提供共同組合のための性。これとは対照的に、スループットの低い一方、FISHベースの技法は、彼らの発達または細胞周期の段階に応じて、個々の細胞における遺伝子または染色体の空間的配置を分析する可能性を提供します。したがって、FISHは核構造と機能の関係をプロービングするための重要なツールであり続けるだろう。
成功した3D FISH実験を行う2つの主要な考慮事項があります。これらは、1。最適な標識プローブおよび2を得た。プローブハイブリダイゼーションのためのDNAが十分にアクセスできるようにしながら、できるだけ核形態を維持するために固定、前後のハイブリダイゼーションステップを含む細胞治療法の選択肢。効率的なプローブラベリングFISHのために非常に重要です。伝統的に、ニックトランスレーションをハプテンまたはフルオロフォア共役ヌクレオチド4のいずれかを導入するために使用されている。同様に、商業ニックトランスレーションキットは、BU直接ハプテンまたは蛍光色素の取り込みのために用意されていtは、2つの段階の標識にアミノアリルヌクレオチドおよびアミン反応性染料を使用した。後者は、DNAポリメラーゼで動作するようにかさばら分子を与えることによって、より効率的な色素の取り込みをレンダリングします。より最近では、DNAの非酵素標識用キットは、核酸への白金配位結合を悪用が開発されている。 FISHプローブであっても既に5ラベル購入することができます。キットと市販製造プローブ疑いが使いやすさを与えるが、それらはかなり社内でプローブを、個々のコンポーネントを購入し、生産するよりも高価です。私たちは、直接、複数の色の多くの異なるBACプローブをラベルするために、低コストのニックトランスポートプロトコルを最適化しました。我々は、高純度BAC DNAを得ることが10と比較して、FISHスライドあたりのプローブの10〜20 ngのための要件で重要と結果であることを発見 - 20倍以上の不純テンプレートDNAを用いる場合には、メジャーの結果、コストと時間の貯蓄。アミノ - allyldUTPの使用は、可撓性の標識を可能にする利用可能なアミン反応性色素( 例えばアレクサフルーアまたはCY-染料)またはハプテン( 例えば 、ビオチン、ジゴキシゲニン)とプローブ。ハプテン標識プローブは、信号を増幅し、可視化するフルオロフォア標識抗体またはストレプトアビジンによって検出される。明るい直接標識プローブは、一般的により少ないバックグラウンド染色を示し、したがって、核局在と遺伝子座の形態のより正確な表現で、その結果、信号の過剰増幅を避ける。
NaOHの変性、塩酸変性とホルムアルデヒド固定とグルタルアルデヒド固定、熱変性とホルムアルデヒド固定6-9:異なる固定剤、プレトリートメント、ハイブリダイゼーション後の洗浄を使用しますが、これらは一般的に次のカテゴリに分類され、いくつかの異なるFISHのテクニックがあります。これらの各々は、利点と欠点を有する。グルタルアルデヒド固定良い核構造の保全になりますが、最小化するために還元剤を用いた治療を必要とする得られた自家蛍光、およびNaOH処理が慎重に十分なDNAの変性および核構造6が損傷する可能性のバランスをとるために制御する必要がある。ホルムアルデヒド固定、核アーキテクチャの摂動の可能性の増大を与え、より少ない強いだけでなく、一般的により強く、より信頼性の高い信号を与え、9自家蛍光低い。塩酸処理はプローブのDNAへの良好なアクセスを提供し、タンパク質うちDNAとストリップをdepurinatesだけでなく、DNA切断を生じる可能性があります。暖房は、物理的に良い標的ハイブリダイゼーションと強い信号で得られた2本のDNA鎖を分離しますが、核構造8のいくつかの摂動を引き起こす可能性があります。これらの技術のそれぞれがタンパク質エピトープに影響を与える程度は実験的に各タンパク質免疫FISH実験で使用するために最適なプロトコルのために決定するための要件で、その結果、広く6,9を変化させます。
ない '完璧' DNA FISH TEはありませんが十分に制御する場合chnique、彼らはすべての有用なことができます。私たちの目標は、最も信頼性の高い信号の加熱方法に焦点を当て、細胞型10の様々な複数の遺伝子座の空間的な位置を調査するために、堅牢で再現性のDNA FISH用のプロトコルを最適化することであった。この自動化および結像システムを用いて、核の配置の単一細胞分析のスループットの向上を目的とした。
プロトコル
1。ニック翻訳で直接標識DNAプローブの生成
注:のBACから作ら直接標識プローブ(100〜250キロバイト)は一貫して明るい信号を生成する。小さいプローブが必要な場合でも、フォスミド(40〜50キロバイト)または5〜10キロバイトインサートを含むプラスミドを使用しています。アンサンブルやUCSCゲノムブラウザを使用して、特定の遺伝子に対応するBACやフォスミドクローンを同定。繰り返し沈殿によって高品質BAC DNAを調製または市販のキットを使用しています。少ない準備が細菌のゲノムDNAで汚染され、少ないプローブは、スライドごとに必要とされる。二段階でラベル付けを実行します。ニックトランスはアミノアリル-dUTPをとアミン反応性染料の化学的カップリングを導入する。
1.1。ニック翻訳
ニックトランスレーション時に、DNアーゼIは、一本鎖切断を作成するために使用される。 DNAポリメラーゼIはT、新しいものと既存のヌクレオチドを置き換える、ニック '3'これらの両端を '伸長ここに '翻訳'ニックとこうして標識ヌクレオチドを取り込む機会を提供する。アミノアリル-dUTPをIと後で化学物質のそのポテンシャルは、アミン反応性染料またはハプテンに結合するDNAポリメラーゼであるため効率的な組み込みが選択されます。それは私が翻訳IニッキングとDNAポリメラーゼDNase処理の適切なバランスを達成するために重要であり、多すぎるのDNase私は、あまりにも少ないの翻訳を開始するポリメラーゼのための十分なニックネームを生成しません、低収率と短い断片サイズを与える過度のDNA消化になります大型フラグメントサイズとアミノアリル-dUTPをの乏しい組み込みを与える。次のプロトコルがうまく動作しますが、それは別のバッチまたは異なるメーカーのDNアーゼIを滴定する必要があるかもしれません。
- 16℃で2時間、氷とインキュベート℃の上にラベリング反応を設定しますこれは、0.5μgのニックトランスレーションDNAを生成する必要がありますし、スケールアップすることができます。 DNアーゼIバッファー( すなわち 0.3 U /μL)でのDNase I 1:30希釈します。
- ラベルされた断片のサイズを確認するには、2%アガロースゲル上で1μLを実行します。あなたがゲルを実行しながら、氷の上での反応を続ける。成功ニックtranslati150 bpのと〜1キロバイトでいくつかの大きなフラグメントを持つ700bpの間に実行されているフラグメントのバルクとスメアになります上の(重要なステップ:代表的な結果を参照してください)。必要に応じて、15〜30分間のために16で新鮮な1時30分のDNase I希釈とインキュベートの別1μlの°Cを追加します。インキュベーション時間は、BAC DNAの量と質に応じて変化するであろう。
- 10分間75℃に加熱することによってDNアーゼIを不活化する。
- PCR精製キットを用いてクリーンアップアミン変性DNA。 100μlのH 2 Oで溶出
- 10μlの3 MのNaOAc液(pH 5.2)と275μlのエタノールを追加することにより、エタノール沈殿DNA。少なくとも1時間または一晩のために-20℃のままにしておきます。 4℃で30分間、最大速度で回転℃で500μlの70%エタノールでペレットを洗浄し、空気乾燥することができます。このステップでは、標識反応を妨害するトレースアミンを除去します。 1X初期反応あたり6μlのH 2 Oに再懸ペレット。
- アミン変性DNAの濃度を決定するために、1μlを使用する微量紫外分光法による。
1.2。蛍光色素のカップリング
プローブの蛍光標識は、色素の化学的カップリングによって達成される。アレクサ弗素スクシンイミジルエステル染料は、それによって直接標識プローブを製造する、共有結合を形成するアミノallyldUTP変性DNAのアミンと反応する。アレクサフルーア488、555、及び647染料は、このプロセスのために成功している。
- 5μLの容積にアミノアリル修飾DNA4μgのを調整して、95への熱℃で5分間、氷上でクールスナップ、および0.2 MのNaHCO 3 3μLを加える。
- 室温で2μlの無水DMSO中のアミン反応性染料のいずれかのアリコートを溶解する。 NaHCO 3を 、渦、1時間、暗所で室温でパルススピンとインキュベートと8μlの改変されたDNAを追加します。
注:アミン反応性染料は、以下のスキームに基づいて、複数のプローブを標識するために使用することができる。 O以来プローブのNLY 10〜20 ngのは、スライドのラベルごとに使用される、1μgのは、50〜100のスライドのために十分なプローブを提供します。これは、-20℃で将来の標識反応のためにアミン変性DNAを格納することも可能である
| DNA量 | DNA量 | 0.2 MのNaHCO 3 | 染料の量 | 合計 | 十分なため |
| 4×1μgの | 1.25μlの各 | 0.75μlの各 | 0.5μlの各 | 2.5μlの(1/4) | 50〜100スライド各 |
| 3×1.35μgの | 1.67μlの各 | 1μlの各 | 0.67μlの各 | 3.34液(1/3) | 65から130までスライド各 |
| 2×2μgの | 2.5μlの各 | 1.5μlの各 | 1μlの各 | 5μlの(1/2) | 100-200各スライド |
| 1×4μgの | 5μlの | 3μL | 2μlの | 10μlの(フル) | 200-400スライド |
- 90μlのH 2 Oを加えて、PCR精製キットを用いて標識プローブを精製する。 100μlの10 mMTris-CL(pH8.5)中に溶出。
- ランベルト·ベールの式を(命令がアレクサフルーア反応染料に伴うデータシートに記載されているか、またはオンラインで見つけることができます)を使用します( 図2を参照)微量フルスペクトル分光法によりプローブ濃度と標識効率を決定します。 100 bpのあたり3-6色素を含むプローブはうまく機能します。定款の下度のプローブは弱いFISHシグナルを与える可能性があります。また、DNAなしで2%アガロースゲルで5μLを実行染色とホスホイメージャを用いた蛍光色素の取り込みを確認してください。エチジウムブロマイドやDNA染色の選択肢とゲルを後染色と全DNAに標識DNAを比較。
以下の工程を経て、関心のある細胞をスライドに固定され、透過処理されている。細胞のDNAを直接標識プローブは、その後、ホットプレート上で一緒に変性し、遮光加湿チャンバー内で一晩ハイブリダイズさせる。
2.1。 1日目
- プローブ降水
注:1日目の間のある時点で沈殿プローブ。これは快適な細胞の処理と並行して行うことができ、一日の開始時に行われる必要はない。- 10〜20 ngの直接標識プローブ、6μlのC 0 T-1 DNA(6μgの)、サケ精巣から1μlの一本鎖DNA(9.7μgの)を混合し、水で100μlに音量を調整します。
- ボルテックスで10μlの3 MのNaOAcと275μlのエタノールと、ミックスを追加します。 -20℃で少なくとも1時間沈殿
- 4℃で30分間、最大速度で微量のスピン℃で70%EtOHでペレットを洗浄して、もう一度スピン。乾いたペレットと5μlの脱イオン化ホルムアミドに懸濁します。光(サーモオーバー場所箔)から保護1000rpmで振とうしながら37℃で30分間のために再懸濁する。
- °C再び光から保護し、37℃で硫酸デキストランミックスを5μlを加え、さらに10分間振る。カバースリップ上にピペッティング直前上下ピペット。
注:BACプローブと組み合わせて染色体塗装の場合は、すぐに使用できるハイブリダイゼーション混合物に供給されている全体の染色体ペインティングプローブは便利なオプションです。再懸 濁し上下にピペッティングにより10μlの染色体ペイントミックスでBACプローブ(サケ精巣からC 0 t1のDNAと一本鎖DNAとの)沈殿した。 10分間、300〜500 rpmで振とうしながら37℃でインキュベートする。染色体塗料/スポットあたりのBACプローブミックスの10μlを使用する。
- スライドに細胞を取り付ける
注:スライドに付着する細胞性向が大幅細胞型によって異なります。各細胞型について決定する経験的に最適なセトリング時間と細胞密度。一貫性のある結果を得るために、細胞を単一細胞懸濁液であるべきである。
PBSに再懸濁した細胞のため、これは厳密には必要ではありませんが使用サークルを容易にするための細胞面積は、疎水性のペンで上に発見されます。疎水性のバリアがうまく分離し、免疫FISHにおける抗体溶液の非常に少量の使用を可能にするスライドごとに3つのサンプルまで続けて、スライド上に広がってから中断を防ぐことができます。細胞数が低い場合にも、スポッティング領域を最小限に抑えることができる。 - マウス胎児肝細胞
- 上下にピペッティングによりPBS再懸濁しの約1.5 mlの各E13.5マウス胎児肝臓を収集します。 2分間3,000 rpmで微量のスピン。繰り返しが3回洗う。最後の洗浄では、70ミクロンのふるいを通して細胞株。 160μlのPBS中1肝臓から細胞を再懸濁し、スポットあたり50μを使用しています。室温で2分間のために解決するために、細胞を残す。
- MウーズES細胞
1。マウスES細胞は、スライドに容易に固執しません。スライド、円領域に細胞が疎水性のペンで上で発見されます。円に通常の培地、ピペットで80-100μLあたり20,000〜50,000細胞の懸濁液を準備し、加湿インキュベーター注意で約3時間のために添付したまま:また、ES細胞は、スライド上で培養することができるが、その形成します自動化されたイメージングを妨げるかもしれないコロニー。 - 固定した細胞
1。いくつかの実験では、自分でスライドに添えない固定された細胞を使用する必要があります。細胞遠心を使って3分間300rpmで穏やかな回転は、細胞の三次元構造を保持し、細胞が脱落しないことが保証されます。
| 細胞型 | 懸濁液中の | 整定時間 | コメント |
| マウス胎児肝臓 | PBS | 2分 | ベンチで |
| マウスES | サプリメントとDMEM | 4時間 | 加湿インキュベーター中で37℃ |
| マウスリンパ | PBS | 2分 | ベンチで |
| マウスP20生殖細胞 | 溶液中で修正 | サイトスピン | ベンチで |
- 10分間:セル、4%PFA(ドラフト内で行う注意 )にそっと沈めるスライドを修正する。細胞の最高のリテンション(先端箱の蓋で例えば 50ミリリットル4%PFA)のためのトレイにフラットスライドで細胞を固定する注:この手順では、前のスライドに添付ファイルへの固定された細胞には適用されません。
後続のすべての手順については、50ミリリットルまたは100ミリリットルコプリンジャーを使用しています。ジャーの間にスライドを移動する際に、細胞を掻きないように注意してください。
- 0.1Mトリス-Cでクエンチリットル、RTで10分間、pHは7.4。
- 室温で10分間、PBS中0.1%saponin/0.1%トリトンX-100中の細胞を透過性。
- RTで5分間、PBSで2回洗浄する。
- RTで20%グリセロール/ PBS中で少なくとも20分間インキュベート注:この時点で、スライドは少なくとも数週間、-20℃で50%グリセロール/ PBSに格納することができる。これは、信号強度9の増大少なくとも1日の結果を得るために、そのストレージを観察されている。先に進む前に、室温20%グリセロール/ PBSに再キャリブレーション注:これは、(2.1.1を参照)プローブを沈殿開始するのに便利なポイントです。
- /液体窒素で凍結融解を3倍。小さなデュワーを用いた液体窒素中で一度水没つのスライド。数秒と特性破裂音の後に撤退すると、霜にペーパータオル上の場所。再びフリーズその後消えて不透明な冷凍グリセロールのを待ちます。最大15のスライドには、次の3つの凍結/融解サイクルの回転で快適に行うことができます。
- W室温で5分間、PBSで2回灰。
- 室温で30分間、0.1MのHCl中でインキュベートする。
- RTで5分間PBSで洗浄する。
- RTで30分間0.5%saponin/0.5%トリトンX-100/PBSに透過性。
- RTで5分間、PBSで2回洗浄する。
- 室温で少なくとも10分間、50%formamide/2x SSCで平衡。
- カバースリップの上にピペットプローブ。瓶からスライドを外して、ペーパータオルでセルスポット(S)の周りに余分な液体をオフに乾燥させます。ホルムアミド乾く早く、細胞を乾燥させないように注意してください。 2または3セルのスポットとスライドは、スライド上の場所を見つけることが対応する22ミリメートル×50ミリメートルカバースリップ上にスポットあたりのプローブを10μlを適用します。スポット(S)上のスライドにカバースリップを反転。ラバーセメントで密封し、これは完全に乾燥することができます。後続のすべての段階で光から保護します。
- 78への熱スライド℃のホットプレート上に正確に2分(反転加熱ブロック、重要なステップを例 :ディスカッションを参照)。カバー(段ボール箱またはシムを置き変性中に光から保護するためのホットプレート上ILAR)。
- 遮光加湿チャンバー内で37℃で一晩インキュベートする。 、加湿光タイトボックスに紙タオルを置き、水で湿らせます。
2.2 2日目
- その後の洗浄工程に適した温度でのソリューションをコプリンジャーを準備します。後続のすべてのステップの間にできるだけ多くの光からスライドを保護します。コーヒーは、コプリンジャーのための良好な光を通さないカバーを作ることができます。
- ゴムセメントと場所2×SSC中でスライドをはがしカバースリップは緩めてオフにスライドするまで。
- 45℃で15分間、50%formamide/2x SSCで洗う光から保護するために水槽に蓋を置きます。
- 63℃で15分のために0.2×SSCで洗う
- 45℃で5分間、2×SSCで洗う
- 室温で5分間、2×SSCで洗浄します。
- RTで5分間PBSで洗浄する。
- 室温でコプリンジャーで2分間DAPI(2×SSCで5μgの/ ml)で染色。
- RTで5分間PBSで脱色。
- カバースリップをマウントするには、ピペットはカバースリップ上にメディアをマウント。一つの場所、および22ミリメートル二から三のスポット用×50ミリメートルカバースリップのために22ミリメートル×22ミリメートルカバースリップを使用して、セルスポットあたり10μlを使用する。セルスポットできるだけ周りPBSを乾かすが、細胞を乾燥しないように注意する。スライド、そしてマニキュアとシールの上にカバースリップを反転。
結果
ここで説明されたプロトコル(概要について図1)は、ゲノム構成の分析のためのハイスループット画像キャプチャを有するDNA FISHを結合するために大きな効果を使用されている。良質プローブの世代は(説明を参照)成功に不可欠です。 図2は FISHプローブのための2つの重要な品質コントロールを示しています。ニックトランスレーションした後、DNAのスメアは、150と700 bpの( 図2A)との間で実行されているフラグメントのバルクと見えるはずです。化学的カップリングした後、蛍光染料の取り込みは、プローブの分光分析( 図2B)で測らすることができる。
いいプローブを使用する場合は、DNAのFISHプロトコルに従うと、通常低い背景に明るいのDNA FISHシグナルをもたらす。我々が正常に異なる細胞型の様に、このプロトコルを使用して、落射蛍光顕微鏡または共焦点のいずれかとしている。エピfluorescencを使用して自動化されたイメージング共焦点顕微鏡は、より強烈な信号を必要としながら電子顕微鏡は、少し背景( 図3)とシャープ、容易に識別FISHシグナルを必要とします。 図4Aは、代表的な処理された画像を示す。 FISHシグナルの核座標を得ることができ、ゲノム領域の空間的関係は、(例えば図4Bを参照) を算出することができる。我々はまた、染色体テリトリー( ムービー1)内のFISHシグナルの3Dモデリングのための共焦点顕微鏡によって得られた画像スタックを使用している。
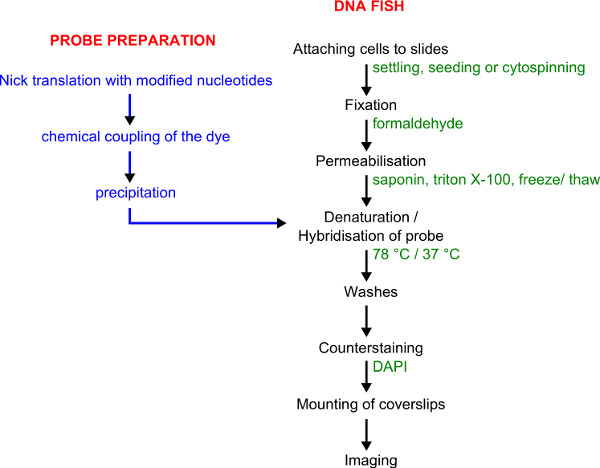
図1。プローブラベリングとのDNA FISHのワークフロー。プローブの準備は青色で示されています。のDNA FISH手順は緑色で与えられた詳細を黒で示されています。
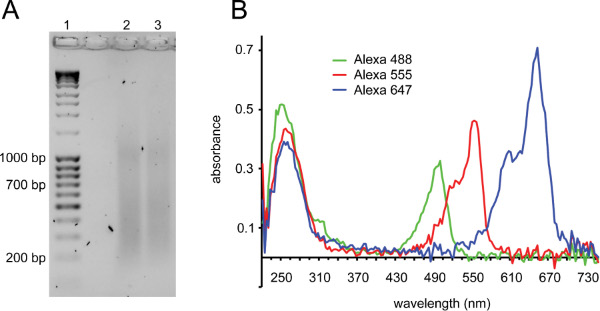
図2。ニックトランスレーションと標識プローブの品質管理によってプローブのラベリング。 DNAラダー、レーン2および3:ニックトランスレーション後のBAC)A)ゲルのDNase I(レーン1を不活性化するために加熱する前に、理想的なスミアを示す。フラグメントは150から700 bpの範囲でなければなりません。 〜1キロバイトで追加スメアは頻繁に成功したニックトランスレーション反応で観察されています。標識プローブのB)マイクロボリュームフルスペクトル分析が。アレクサフルーア488:495 nmであり、アレクサフルーア555:555 nmであり、アレクサフルーア647:650 nmの260 nmでDNAピークはプローブで標識されている蛍光体の吸収波長に対応する第2のピークが観察される。示され、良い編入を持つ3つのプローブである:蛍光体の吸収ピークは、同様の高さやDNAのピークよりも高くなっています。アレクサフルーア647は通常より高い吸光度を持っている順番にアレクサフルーア488より高い吸光度を持っているよりアレクサフルーア555、。アレクサフルーア488はまた、それ故に、このプローブの最初のピークが他より高く、260 nmでのいくつかの光を吸収する。貧しい混入ショーフルオロフォア吸光度2〜3倍のDNAの吸光度より低いとプローブ。 DNAピークより2倍以上高い蛍光体のピークとのプローブは通常、FISHで背景を引き起こす可能性があり非共役染料が含まれています。
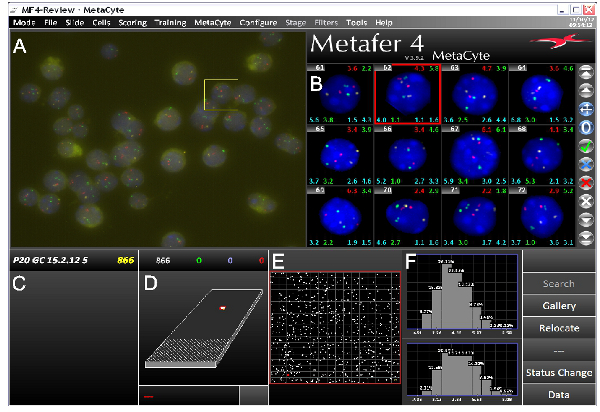
図3。典型的な三色FISH実験を示すMetaCyte 3D FISHイメージングソフトウェアからのスクリーンショット画面はいくつかの部分に分かれています。Metafer / MetaCyteスライドスキャニングシステムによって取り込ま)生の視野(FOV)の画像。B)ギャラリー国連がある特定され核の画像のFISH信号用dergone画像処理とセグメンテーション。各種のデータは、各ギャラリー画像上に表示することができる。この例では、細胞数は、様々 interallelicとinterlocus距離右上とし、各画像下のいずれかの側に左上に表示されます。C)は、スライド名が解析で同定核の数と。Dは)によってスキャンエリアとスライドの描写システムE)、スキャン領域の拡大。白い斑点はFOVあたりの核を同定した。F)が処理されたデータを表す。この例では、緑色の信号(上)と赤の信号(下)の間で最大のinterallelic距離が示されています。示されたデータは、マウスP20生殖細胞からのものである。プローブは異なる染色体、カバーHBBおよびHBA遺伝子とヒストンクラスタに配置されています。 より大きい数字を表示するには、ここをクリックしてください 。
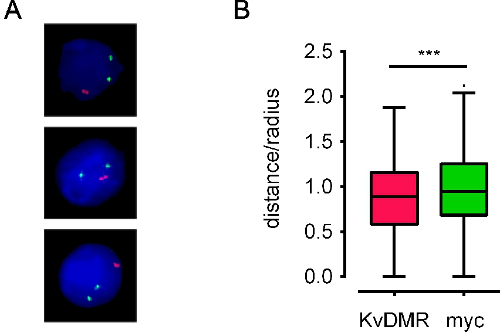
図4。処理された画像と抽出位置データの例を示す。 MycのとKCNQ1遺伝子(それぞれMYCとKvDMR、着色された緑と赤、と呼ばれる)interallelic距離を表す。B)テューキーボックスウィスカープロットをカバーする異なる染色体上にあるBACプローブで染色E13.5マウス胎児肝細胞のための)処理された画像600核の同じ遺伝子座の配布。 Interallelic距離は核の大きさの変化を考慮するために核 '半径(距離/半径)の割合としてプロットされます。 ES細胞核は約5μmの半径を有する。 (:P <0.01、対応のないt-検定***)赤プローブは緑色のプローブよりも一緒にかなり近い。 10からのデータ。
ムービー1。彼らの染色体tの内の2つの遺伝子座の3Dモデリングerritory。マウス染色体7のいずれかの端の近くに位置する2つのBACプローブは、ラベル(赤と遠赤(擬似色の白))とマウスES細胞への全体染色体塗料(グリーン)と一緒にハイブリダイズさせた。 ムービーを表示するには、ここをクリック 。
ディスカッション
3DのDNA FISHは、核内に空間配置を分析するための広く認識ツールとなっています。魚はあらゆる手法と同様に、視覚的かつ直接的な結果を提供していますが、人はその限界を認識する必要があります。 DNAのFISHは比較的厳しい治療が最終的に分析される核構造·非常にものを破壊するFISHプローブのクロマチンにアクセスできるようにするために必要とされるという固有の問題に直面している。いくつかの戦略は、アクセシビリティと構造保全を両立するために追求されています。ここで説明するプロトコルでは、染色体DNAプローブハイブリダイゼーションの前に細胞および熱変性の凍結融解透過によってアクセスされる。 Solovei ら 、(2002)同様の処理後の構造変化を分析し、クロマチンドメインの平均変位がゲノム8で約1Mbの領域に構造解析の分解能を制限300nmであることがわかった。これは、それを高解像度ではありませんが核の位置を分析するための合理的なスケールです。
FISH分析のためのより実用的な考慮事項は、その画像取得と核の距離の測定は分析することができる核の数を制限時間のかかるプロセスである。我々は、高スループット自動化された撮像を行うことを目的とした発生頻度の少ないイベントを検討するためである。その後、所定の位置に適切なコントロールを使用して、十分な数の空間的関係の堅牢な統計分析を可能にするために比較することができる。我々は、我々が日常的に核のボリューム内のすべてのFISHシグナルの3次元座標を決定するためのデータポイントにつき600核を分析する。これらのデータは、非常に少ない記憶空間を必要とし、間およびイントラ対立遺伝子距離、またはそれ以降の時点における半径位置の分析を可能にする。また、自動処理が大幅に無意識のバイアスの可能性を低減する研究ブラインドな方法で行うことができる。
ハイスループットanalysこの種のを有効にするには、私たちの目標は、DNA FISHを行うの迅速かつ効率的な方法を確立することであったされています。私たちは、プロトコルが一貫して低い背景に明るい信号を生成する、非常に堅牢であることがここで説明する発見。それは、我々はスライドに細胞を付着させる工程を適応提供しようとしたすべての種類の細胞のために働いた。また、一つの例外を除いて、我々が使用されるすべてのBACは明るいプローブに変えることができた。我々はまた、正常なDNA FISH後の抗体染色による核タンパク質の数を検出した。いくつかのタンパク質のために、これがうまく機能している間、我々は、IFとDNA免疫FISH(11を比較)との間で抗体染色パターンの一貫性を確保するために、伝統的な免疫蛍光(IF)との比較をお勧めします。
一般的に、我々は、異なる色で標識されたプローブは、同じ結果が得られたことがわかった。標識色素を選択する場合ただし、次の側面を考慮する必要がある。まず、蛍光体の色は、顕微鏡uで十分に検出可能である必要があるSED。第二に、蛍光団により放射される光の波長は、他のチャンネルに介してブリードを避けるために十分に異なるべきである。これは、顕微鏡でフィルターに依存します。そして第三に、少なくとも私たちの手の中に、いくつかの色は、より一貫して他より明るい信号を生成する。我々は常にアレクサフルーア555(赤)になります対比としてDAPI(青)、488(緑)、647を(遠赤)の標識プローブのための良い選択肢を使用している。当社のイメージング·システムでは、我々はアレクサフルーア488(緑)に続いて最も明るい信号を生成するためにアレクサフルーア555(赤)を見つけました。アレクサ弗素647(遠赤)は人間の目では検出できない、顕微鏡下を見たときに信号がないので見られないという欠点がある。二色FISHを行った場合このように、我々は赤と緑の染料の組み合わせをお勧めします。
二つの主要な問題が生じる可能性があります細胞はスライドや微弱な信号を洗浄。どれだけの細胞は、スライドに付着非常変数のbetwですEEN細胞型、及び細胞が決定される必要が/シードを解決するための最良のプロトコル。我々は、正常に使用されているいずれかの正に荷電した又はポリ-L-リジン被覆スライド(使用する準備が購入した)、しかし細胞は一般にポリ-L-リジン被覆スライドに良好な付着がわかった。細胞があまりにもまばらだったら自動撮影が妨げられていたのに対し、我々はまた、細胞があまりに密集全域だったときシートのような剥がれだろうことがわかった。試料があまりにも貴重でない場合はしたがって、異なるセル番号が理想的な濃度を決定するために播種されるべきである。細胞は洗い落とすことが特に傾向がある場合には、疎水性障壁ペンを用いることができ、FISH工程は慎重にスライド上に直接ソリューションをピペッティングすることによって行うことができる。ピはまた、大幅に必要な試薬の体積を減少させますが、複数のスライドをやったときにはかなり時間がかかります。
弱い信号は、通常、貧しいプローブによるものであり、それは良いものを作ることに時間を投資する価値は十分ある。クリーンBAC DNAが使用されるべきである繰り返し沈殿又は市販のキットにより得ることができる出発物質としてエ細菌のゲノムDNAマイナスの細胞溶解の間にプローブの品質と慎重な取り扱いに影響を与え、細胞溶解物のフィルタリングによる汚染を最小限に保つために必要とされる。これは非常に収率を低下させるように、それは、しかしながら、直鎖DNAを消化するエキソヌクレアーゼ工程を用いる必要はない。プローブの効率的なラベルは非常に重要です。このステップのための最も重要な品質管理は、ニックトランスレーション(代表的な結果を参照)した後、フラグメントサイズをチェックしています。 '良い'スミアはほとんど常に鮮やかな色のプローブになります。しかし、我々は時折、特定のBACが良好なプローブに加工し、異なるBACは、選択される必要があることはできないことを見出した。もう一つの重要なステップは、熱変性です。温度が低すぎる場合は、DNAは、部分的に変性したプローブハイブリダイゼーションは損なわれるであろう。温度が高すぎると、核構造のWiである場合必要以上に摂動ことでしょう。私たちの手で、倒立ホットブロック上78°Cは、理想的な妥協案ですが、これは使用され、それぞれのホット·ブロックによって異なる場合があります。
私たちは、封入剤封入剤を使用して良い結果を持っていたが、SlowFadeゴールドが少なく背景を持っており、より長いための蛍光信号を保持することを発見した。私たちは、これが3次元構造に影響を与えるために、時間をかけて気泡を形成するために見られるよう我々はハードセット封入剤の使用はお勧めしません。
結論として、ここで説明したDNA FISHプロトコルと一緒に明るく蛍光直接標識プローブは、生物学的な質問の多種多様に適用可能である核アーキテクチャの堅牢で迅速な分析のための効率的なソリューションを提供します。
開示事項
この記事の生産の一部がカールツァイスとメタシステムによって後援されました。
謝辞
この作品は、BBSRCとウェルカムトラストによって資金を供給された。私たちは、バイオインフォマティクス解析のヘルプのためのイメージングとフェリックスクルーガーの支援のためにサイモン·ウォーカーに感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||||||||
| Reagent/Material | |||||||||||||||||||||||||||||||||||||||
| NTB buffer | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| DTT | Invitrogen | D-1532 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| dNTPs | Bioline | BIO-39025 | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Aminoallyl-dUTP | Ambion | AM8439 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNA Polymerase I | New England Biolabs | M02095 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNase I recombinant RNase free | Roche | 4716728001 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| QIAquick PCR purification kit | Qiagen | 28104 | Step 1.1.4, 1.2.3 | ||||||||||||||||||||||||||||||||||||
| NaOAc | VWR | 27653 | Step 1.1.5, Step 2.1.1.2 | ||||||||||||||||||||||||||||||||||||
| NaHCO3 | Sigma | S5761 | Step 1.2.1 | ||||||||||||||||||||||||||||||||||||
| Alexa Fluor Reactive Dye Decapacks for Microarray Applications | Invitrogen (Molecular Probes) | A32750, A32756, A32757 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| DMSO (anhydrous) | Sigma Aldrich | 276855 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| SYBR Safe DNA gel stain | Life Technologies | S33102 | Step 1.2.4 (alternative to ethidium bromide) | ||||||||||||||||||||||||||||||||||||
| Cot-1 DNA | Invitrogen | 18440-016 | Step 2.1.1.1 (Cot-1 DNA can be home-made in large quantities and works just as well) | ||||||||||||||||||||||||||||||||||||
| Single stranded DNA from salmon testes | Sigma | D7656 | Step 2.1.1.1 | ||||||||||||||||||||||||||||||||||||
| Deionised formamide | Sigma | F-9037 | Step 2.1.1.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| Dextran sulphate | Sigma Life Sciences | D8906 | Step 2.1.1.4 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| XMP painting probes | MetaSystems | 000000-0528-837 | Step 2.1.1.4 | ||||||||||||||||||||||||||||||||||||
| Poly-L-lysine coated slides | Sigma Aldrich | P0425 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| Hydrophobic pen (ImmEdge) | Vector Laboratories | H-4000 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| 70 μm sieve (Mouse f–tal liver cells only) | BD Falcon | 352350 | Step 2.1.2.1 | ||||||||||||||||||||||||||||||||||||
| PFA | Sigma Aldrich | P6148 | Step 2.1.3 (Flammable, corrosive, acute toxicity, health hazard) (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Tris-Cl | Step 2.1.3 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Saponin from Quillaja bark | Sigma Aldrich | 47036 | Step 2.1.4, 2.1.11 (Acute toxicity), (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Triton X-100 | Sigma | T9284 | Step 2.1.4, 2.1.11 (Acute toxicity, hazardous to the environment) | ||||||||||||||||||||||||||||||||||||
| 10 x PBS | Life Technologies (GIBCO) | 70011-036 | Step 2.1.4, 2.1.5, 2.1.6, 2.1.8, 2.1.10, 2.1.11, 2.1.12, 2.2.7, 2.2.9, | ||||||||||||||||||||||||||||||||||||
| Glycerol | Fisher Scientific | G/0650 | Step 2.1.6 | ||||||||||||||||||||||||||||||||||||
| HCl | VWR | 20252 | Step 2.1.9 (Acute toxicity, corrosive) | ||||||||||||||||||||||||||||||||||||
| Formamide | Sigma Aldrich | 47670 | Step 2.1.14, 2.2.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SSC | Step 2.1.14, 2.2.2-2.2.6, 2.2.8 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 22 | Menzel-Glaser | BB022022A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 50 | Menzel-Glaser | BB022050A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Rubber cement | Marabu | 2901 (10 000) | Step 2.1.15 (Flammable, health hazard, danger to the environment) | ||||||||||||||||||||||||||||||||||||
| DAPI | Invitrogen Molecular Probes | D3571 | Step 2.2.8 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SlowFade Gold | Invitrogen | S36936 | Step 2.2.10 (mounting medium) | ||||||||||||||||||||||||||||||||||||
| Clear nail polish | Any supplier | Step 2.2.10 | |||||||||||||||||||||||||||||||||||||
| Equipment | |||||||||||||||||||||||||||||||||||||||
| Nanodrop 2000 | Thermo Scientific | Step 1.1.6, 1.2.4 (microvolume spectroscopy) | |||||||||||||||||||||||||||||||||||||
| Typhoon FLA 7000 phosphoimager | GE Life Sciences | Step 1.2.4 | |||||||||||||||||||||||||||||||||||||
| Coplin jars | Sigma-Aldrich | S6016 (6EA)/S5516 (6EA) | Step 2.1.3 onwards | ||||||||||||||||||||||||||||||||||||
| Liquid nitrogen dewar | Any supplier | Step 2.1.7 | |||||||||||||||||||||||||||||||||||||
| Base with Black Lid (light-tight chamber) | Simport | M920-2 | Step 2.1.17 | ||||||||||||||||||||||||||||||||||||
| Thermomixer comfort | Eppendorf | 5355 000.011 | Step 2.2.3 (or use shaker in a 37 °C room) | ||||||||||||||||||||||||||||||||||||
| Imaging and Image processing | |||||||||||||||||||||||||||||||||||||||
| Metafer - Imaging Automation Platform | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| MetaCyte - Automated Interphase FISH analysis | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| Axio Imager Z2 upright | Zeiss | epifluorescence microscope used with automated imaging | |||||||||||||||||||||||||||||||||||||
| IX81 confocal microscope (FV1000) | Olympus | confocal microscope | |||||||||||||||||||||||||||||||||||||
| Bitplane, version 7.3 | Imaris | image analysis and 3D modelling software | |||||||||||||||||||||||||||||||||||||
| Scientific volume Imaging, version 4.1 | Huygens | image analysis and deconvolution software | |||||||||||||||||||||||||||||||||||||
FISH buffers and reaction mixes
| |||||||||||||||||||||||||||||||||||||||
参考文献
- Chakalova, L., Debrand, E., Mitchell, J. A., Osborne, C. S., Fraser, P. Replication and transcription: shaping the landscape of the genome. Nat. Rev. Genet. 6, 669-677 (2005).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (1016).
- de Wit, E., de Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes Dev. 26, 11-24 (2012).
- Sambrook, J., Russell, D. . Molecular Cloning: A laboratory Manual. , (2000).
- Boyle, S., Rodesch, M. J., Halvensleben, H. A., Jeddeloh, J. A., Bickmore, W. A. Fluorescence in situ hybridization with high-complexity repeat-free oligonucleotide probes generated by massively parallel synthesis. Chromosome Res. 19, 901-909 (2011).
- Brown, K. Visualizing nuclear proteins together with transcribed and inactive genes in structurally preserved cells. Methods. 26, 10-18 (2002).
- Hewitt, S. L., Yin, B., et al. RAG-1 and ATM coordinate monoallelic recombination and nuclear positioning of immunoglobulin loci. Nat. Immunol. 10, 655-664 (2009).
- Solovei, I., Cavallo, A., et al. Spatial preservation of nuclear chromatin architecture during three-dimensional fluorescence in situ hybridization (3D-FISH. Experimental Cell Research. 276, 10-23 (2002).
- Solovei, I., Beatty, B., Mai, S., Squire, J. FISH on three-dimensionally preserved nuclei. FISH: A Practical Approach. , (2002).
- Krueger, C., King, M. R., et al. Pairing of homologous regions in the mouse genome is associated with transcription but not imprinting status. PLoS One. 7, e38983 (2012).
- Solovei, I., Cremer, M., Bridger, J. M., Volpi, E. V. 3D-FISH on Cultured Cells Combined with Immunostaining. Fluorescence in situ hybridization (FISH): Protocols and Applications. , (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved