Method Article
Robusto PESCADO ADN 3D utilizando sondas marcadas directamente
En este artículo
Resumen
Se describe un protocolo robusto y versátil para el análisis de la arquitectura nuclear por FISH 3D DNA usando sondas fluorescentes marcados directamente.
Resumen
3D PEZ ADN se ha convertido en una herramienta importante para el análisis de la organización tridimensional del núcleo, y varias variaciones de la técnica se han publicado. En este artículo se describe un protocolo que ha sido optimizado para la robustez, reproducibilidad y facilidad de uso. Brillantemente fluorescentes sondas directamente marcadas son generados por la mella con amino-allyldUTP seguido de acoplamiento químico del colorante. 3D PEZ ADN se lleva a cabo utilizando una etapa de congelación-descongelación para la permeabilización celular y una etapa de calentamiento para la desnaturalización simultánea de la sonda y el ADN nuclear. El protocolo es aplicable a una gama de tipos de células y una variedad de sondas (BAC, plásmidos, fosmids, o pinturas de todo el cromosoma) y permite la formación de imágenes automatizado de alto rendimiento. Con este método de manera rutinaria investigamos localización nuclear de hasta tres regiones cromosómicas.
Introducción
ADN hibridación in situ fluorescente (FISH ADN) permite la visualización tridimensional de loci de genes individuales, dominios subchromosomal y cromosomas enteros incluso durante todas las etapas del ciclo celular. PEZ 2D se utiliza para los estudios en metafase mientras que los peces 3D se ha utilizado ampliamente para investigar la relación entre la organización espacial del genoma y su función durante la interfase (1,2 y las referencias en él). Históricamente, los estudios de co-asociación se realizaron mediante la investigación de decenas a cientos de loci individuales por FISH. Técnicas más recientemente potente de alto rendimiento basados en 3C como 4C y Hi-C se han desarrollado 3, lo que permite la investigación de cross-talk molecular entre muchos miles de diferentes loci. Mientras que las técnicas basadas 3C y FISH de ADN pueden ser métodos complementarios, que no necesariamente responden a las mismas preguntas. Métodos basados en 3C proporcionan una lectura de conjunto de poblaciones de células mixtas, lo que resulta en una probabilidaddad para los compañeros de las asociaciones. En contraste, mientras que la baja en el rendimiento, basados en técnicas de FISH ofrecen la posibilidad de analizar disposiciones espaciales de los loci o los cromosomas en las células individuales de acuerdo a sus etapas del ciclo de desarrollo o celular. Por lo tanto, PEZ, seguirá siendo una importante herramienta para sondear las relaciones estructura-función nucleares.
Hay dos consideraciones importantes en la realización de experimentos de FISH 3D exitosas. Estos son: 1. obtención de sondas marcadas de manera óptima y 2. elección de los tratamientos celulares, incluyendo la fijación, pre-y pasos posteriores a la hibridación, para preservar la morfología nuclear tanto como sea posible al tiempo que ADN suficientemente accesible para la hibridación de la sonda. Sonda de etiquetado eficiente es muy importante para los peces. Tradicionalmente, la mella se ha utilizado para introducir ya sea hapteno o nucleótidos fluoróforo conjugados con 4. Del mismo modo, los kits de la mella comerciales están disponibles para hapteno o incorporación directa fluoróforo, but también para dos pasos etiquetado utilizando nucleótidos aminoallyl y colorantes reactivos con amina. Este último hace que la incorporación de tinte más eficiente, dando una molécula de ADN polimerasa menos voluminoso que trabajar. Más recientemente, los kits para el etiquetado no enzimática de ADN se han desarrollado que explotan unión coordinativa de platino a los ácidos nucleicos. Sondas FISH incluso se pueden comprar ya marcado 5. Mientras que los kits y sondas fabricadas comercialmente sin duda le dan la facilidad de uso, son considerablemente más caro que comprar los componentes individuales y la producción de sondas en el local. Hemos optimizado un protocolo de traslado de cortes de bajo coste con el fin de etiquetar directamente a muchas diferentes sondas de alcoholemia en varios colores. Hemos descubierto que la obtención de ADN altamente puro BAC es crítico y los resultados en un requisito para sólo el 10-20 ng de sonda por diapositiva PEZ, en comparación con 10 - 20 veces más cuando se utiliza la plantilla de ADN impuro, lo que resulta en mayor costo y tiempo ahorros. El uso de amino-allyldUTP permite etiquetado flexible de lossondas con colorantes reactivos con amina disponibles (por ejemplo, Cy-colorantes Alexa Fluor o) o haptenos (por ejemplo, biotina, digoxigenina). Sondas de hapteno-etiquetados son detectados por anticuerpos conjugados con fluoróforo o estreptavidina para amplificar y visualizar la señal. Bright sondas directamente marcadas generalmente muestran menos tinción de fondo y evitar la sobre-amplificación de las señales, por lo tanto, lo que resulta en una representación más precisa de la localización nuclear y la morfología locus.
Hay varias técnicas de FISH diferentes que utilizan diferentes fijadores, pre-tratamientos, y lavados post-hibridación pero éstos generalmente se clasifican en las siguientes categorías: fijación de glutaraldehído con NaOH desnaturalización, fijación de formaldehído con HCl desnaturalización, y la fijación de formaldehído con desnaturalización por calor 6-9 . Cada uno de ellos tiene sus ventajas y desventajas. Glutaraldehído resultados fijación en buen estado de conservación estructural nuclear, pero requiere tratamiento con agentes reductores para reducir al mínimola autofluorescencia resultante, y tratamiento con NaOH necesita ser cuidadosamente controlado para equilibrar suficiente desnaturalización del ADN y la posibilidad de daños en la estructura nuclear 6. La fijación de formaldehído es menos fuerte, dando un aumento de la probabilidad de perturbaciones de la arquitectura nuclear, pero también suelen dar señales más bajas y autofluorescencia 9 más fuertes y más fiable. Tratamiento HCl depurinates ADN y tiras fuera proteínas, proporcionando un buen acceso al ADN de las sondas, pero también puede presentar roturas en el ADN. Calefacción separa físicamente las dos cadenas de ADN que resultan en buen objetivo de hibridación y las señales fuertes, pero puede causar alguna perturbación de la estructura nuclear 8. El grado en que cada una de estas técnicas afecta a epítopos de proteínas varía ampliamente 6,9, lo que resulta en el requisito para determinar experimentalmente para cada proteína el protocolo óptimo para utilizar en experimentos de inmuno-FISH.
Aunque no existe una "perfecta" ADN PESCADO technique, que todo puede ser útil si está bien controlado. Nuestro objetivo era optimizar un protocolo para robusto y reproducible PEZ ADN para investigar posicionamiento espacial de múltiples loci en una variedad de tipos de células 10, centrándose en el método de calentamiento para la mayoría de señales fiables. Con esto y el uso de un sistema automatizado de imágenes apuntamos a incrementar el rendimiento para el análisis de células individuales de los acuerdos nucleares.
Protocolo
1. La generación de sondas de ADN marcado directamente por Nick Translation
Nota: las sondas de marcado directamente elaborados a partir de BAC (100-250 kb) consistentemente producen señales luminosas. Si se requieren sondas más pequeñas, use fosmids (40-50 kb) o incluso plásmidos que contenían insertos 5-10 kb. Identificar BAC o fosmid clones correspondientes a genes específicos utilizando Conjunto o navegadores genoma UCSC. Preparar ADN de BAC de alta calidad por precipitación repetida o utilizar kits disponibles comercialmente. Cuanto menos la preparación está contaminado con ADN genómico bacteriano, se requiere menos sonda por diapositiva. Lleve a cabo el etiquetado en dos etapas: traducción nick introducir aminoallyl-dUTP y el acoplamiento químico de un colorante reactivo con amina.
1.1. Nick Translation
Durante el desplazamiento de la mella, DNasa I se utiliza para crear un solo capítulo rompe. ADN polimerasa I alarga 'extremos de estos "el 3 nicks', en sustitución de nucleótidos existentes por otras nuevas, t presente 'traducir' el nick y proporcionando así la oportunidad de incorporar nucleótidos marcados. Aminoalil-dUTP se elige debido a la incorporación eficiente por la polimerasa I de ADN y su potencial para el acoplamiento químico más tarde a los colorantes reactivos con amina o haptenos. Es crítico para lograr el equilibrio adecuado entre DNasa I formación de muescas y ADN polimerasa I de traducción; demasiado DNasa I se traducirá en la digestión de ADN excesiva dando bajo rendimiento y tamaños de los fragmentos cortos, demasiado poco no producirá suficientes mellas para la polimerasa para iniciar la traducción, dando gran tamaño del fragmento y la mala incorporación de aminoalil-dUTP. El siguiente protocolo funciona bien, pero puede ser necesario para titular la DNAsa I a partir de diferentes lotes o de diferentes fabricantes.
- Configurar el etiquetado de reacción sobre hielo y se incuba durante 2 horas a 16 ° C. Esto debería producir 0,5-1 mg ADN mella y puede ser ampliado. Diluir DNasa I 01:30 en tampón de DNasa I (es decir, 0,3 U / l).
- Ejecutar 1 l en un gel de agarosa al 2% para comprobar el tamaño de los fragmentos marcados. Mientras se ejecuta el gel, mantener la reacción en hielo. Exitosa nick translatien se traducirá en una prueba con la mayor parte de los fragmentos que funcionan entre 150 pb y 700 pb con algunos fragmentos más grandes en ~ 1 kb (PASO CRÍTICO: ver resultados representativos). Si es necesario, añadir otro 1 l de una dilución fresca 01:30 DNasa I y se incuba a 16 ° C durante 15-30 min. El tiempo de incubación variará en función de la cantidad y calidad de ADN de BAC.
- Se inactiva la DNasa I por calentamiento a 75 ° C durante 10 min.
- ADN modificado con amina de limpieza utilizando un kit de purificación de PCR. Se eluye en 100 l H 2 O.
- El etanol precipitado de ADN mediante la adición de 10 l de NaOAc 3 M (pH 5,2) y 275 l de etanol. Agregar a -20 ° C durante al menos 1 hora o toda la noche. Girar a velocidad máxima durante 30 min a 4 ° C. Lave pellet con 500 l de etanol al 70% y dejar secar al aire. Este paso elimina las trazas de aminas que puedan interferir con la reacción de marcaje. Resuspender el pellet en 6 l H 2 O por 1x reacción inicial.
- Usar 1 l para determinar la concentración del ADN modificado con aminapor espectroscopía UV microvolumen.
1.2. El acoplamiento del colorante fluorescente
Etiquetado fluorescente de la sonda se consigue mediante acoplamiento químico del colorante. Alexa Fluor succinimidil éster colorantes reaccionan con las aminas de la ADN modificado con amino-allyldUTP para formar un enlace covalente, produciendo de este modo sondas marcadas directamente. Alexa Fluor 488, 555, y 647 colorantes han tenido éxito para este proceso.
- Ajuste 4 g de ADN alilo amino-modificado a un volumen de 5 l, calentar a 95 ° C durante 5 min, encaje fresco en hielo, y añadir 3 l de 0,2 M de NaHCO3.
- Se disuelve una alícuota de colorante reactivo de amina en 2 l de DMSO anhidro a temperatura ambiente. Añadir 8 l de ADN modificado con NaHCO3, vórtice, el pulso giro y se incuba a temperatura ambiente en la oscuridad durante 1 hora.
Nota: Los colorantes reactivos de amina se pueden utilizar para etiquetar más de una sonda basada en el siguiente esquema. Desde oólo 10-20 ng de la sonda se utiliza por etiquetado de diapositivas, 1 g da suficiente sonda para 50-100 diapositivas. También es posible almacenar ADN modificado con amina para futuras reacciones de marcaje a -20 ° C.
| Cantidad de ADN | Volumen de ADN | 0,2 M NaHCO 3 | Volumen de tinte | Total | Suficiente para |
| 4 x 1 g | 1,25 l cada uno | 0,75 l cada uno | 0,5 l cada uno | 2,5 l (1/4) | 50-100 diapositivas cada |
| 3 x 1,35 g | 1,67 l cada uno | 1 l cada uno | 0,67 l cada uno | 3,34 l (1/3) | 65-130 cada diapositiva |
| 2 x 2 g | 2,5 l cada uno | 1,5 l cada uno | 1 l cada uno | 5 l (1/2) | 100-200desliza cada |
| 1 x 4 mg | 5 l | 3 l | 2 l | 10 l (completo) | 200-400 diapositivas |
- Añadir 90 l H 2 O y sonda marcada purificar utilizando un kit de purificación de PCR. Eluir en 100 l 10 mMTris-Cl (pH 8,5).
- Determinar la concentración de la sonda y el etiquetado de eficiencia mediante espectroscopia de espectro completo microvolumen (ver Figura 2) usando ecuaciones Beer-Lambert (Las instrucciones se dan en la ficha técnica que acompaña a los colorantes reactivos Alexa Fluor o se pueden encontrar en línea). Las sondas que contienen 3-6 colorantes por 100 pb funcionan bien. Sondas con menores grados de incorporación pueden dar señales de FISH débiles. Alternativamente, ejecute 5 l en un gel de agarosa al 2% de ADN sin mancha y comprobar la incorporación del colorante fluorescente usando un phosphoimager. Post-tinción del gel con bromuro de etidio o alternativas de tinción de ADN y comparar ADN marcado con el ADN total.
A través de los siguientes pasos, las células de interés se fijan a portaobjetos y se permeabilizaron. El ADN celular y sondas directamente marcadas se desnaturalizaron a continuación, juntos en una placa caliente y se hibridaron durante la noche en una cámara humidificada hermética a la luz.
2.1. Día 1
- Sonda Precipitación
Nota: las sondas Precipitado en algún momento durante el día 1. Esto puede ser realizado cómodamente en paralelo con el tratamiento de las células y no tiene que ser hecho en el comienzo del día.- Mezclar 10-20 ng de sonda marcado directamente, 6 l C 0 t-1 DNA (6 g), 1 l de ADN de cadena sencilla a partir de testículos de salmón (9,7 g) y ajustar el volumen de 100 l con agua.
- Añadir 10 l 3 M NaOAc y 275 l EtOH y mezclar mediante agitación. Precipitar durante al menos 1 hora a -20 ° C.
- Girar en microcentrífuga a velocidad máxima durante 30 min a 4 ° C. Lave pellet con EtOH al 70% y girar de nuevo. Dry pellety volver a suspender en 5 l de formamida desionizada. Volver a suspender durante 30 minutos a 37 ° C con agitación a 1000 rpm protegidos de la luz (coloque papel de aluminio en el termomezclador).
- Añadir 5 l de sulfato de dextrano mezcla y se agita durante 10 min a 37 º C de nuevo protegido de la luz. Pipetear hacia arriba y abajo justo antes de pipetear en cubreobjetos.
Nota: Para la pintura cromosoma combinado con sondas BAC, enteros sondas de cromosoma pintura que se suministran en su forma lista para usar mezcla de hibridación son una opción conveniente. Resuspender el precipitado BAC sonda (con C 0 t1 ADN y ADN de una sola hebra a partir de testículos de salmón) en 10 l de la mezcla de pintura cromosoma mediante pipeteo hacia arriba y hacia abajo. Incubar a 37 ° C con agitación a 300-500 rpm durante 10 min. Utilice 10 l de cromosoma pintura / BAC sonda de mezcla por punto.
- Colocación de las células a las diapositivas
Nota: La propensión de las células para adherirse a las diapositivas varía en gran medida con el tipo de célula. Para cada tipo de célula, determinar laóptima el tiempo de establecimiento y la densidad celular empíricamente. Para obtener resultados consistentes, las células deben estar en una suspensión de células individuales.
Para facilidad de uso círculo del área las células va a ser detectado con una pluma hidrófobo, aunque para las células se resuspendieron en PBS esto no es estrictamente necesario. Una barrera hidrofóbica impide que la suspensión se extienda en la diapositiva, mantiene hasta tres muestras por lámina muy bien separados y permite el uso de volúmenes muy pequeños de solución de anticuerpo en inmuno-FISH. Además, la zona manchado se puede mantener a un mínimo si el número de células son bajos. - Células de hígado fetal Ratón
- Recoger cada ratón E13.5 hígado fetal en aproximadamente 1,5 ml de PBS y se resuspenden por pipeteo hacia arriba y hacia abajo. Girar en microcentrífuga a 3000 rpm durante 2 min. Repetir lava tres veces. En los últimos lavado, las células de la cepa a través de un tamiz de 70 micras. Resuspender las células de un hígado en 160 l de PBS y el uso de 50 μ por mancha. Deja células que conformarse con 2 minutos a temperatura ambiente.
- Mouse ES Cells
1. Mouse células madre embrionarias no se adhieren fácilmente a la diapositiva. En una diapositiva, círculo del área las células se pueden observar con un lápiz hidrofóbico. Preparar una suspensión de 20.000-50.000 células por 80-100 l en medio de cultivo normal, pipeta en círculo y se van a adjuntar durante aproximadamente 3 horas en un incubador humidificado Nota:. Alternativamente, las células ES pueden ser cultivadas en portaobjetos, pero luego formar colonias que pudiera entorpecer el automatizado de imágenes. - Las células fijadas
1. Algunos experimentos requieren el uso de células fijas que no se adhieren a las diapositivas por sí mismos. Hilado suave a 300 rpm durante 3 min usando una citocentrífuga conserva la estructura tridimensional de las células y se asegura de que las células no se caen.
| Tipo de la célula | Suspensión en | Tiempo de estabilización | Comentario |
| Ratónhígado fetal | PBS | 2 min | El banco de |
| Ratón ES | DMEM con suplementos | 4 hr | 37 ° C en incubador humidificado |
| Linfocitos de ratón | PBS | 2 min | El banco de |
| Ratón P20 células germinales | Fijar en solución | Cytospin | El banco de |
- Para fijar las células, deslice suavemente sumergirse en el 4% PFA (PRECAUCIÓN: realice en campana) durante 10 min. Fijar las células con la tapa plana en una bandeja para el mejor mantenimiento de las células (por ejemplo, 50 ml de PFA al 4% en la tapa de una caja de propina) Nota:. Este paso no se aplica a las células fijadas antes de la unión a las diapositivas.
Para todos los pasos subsiguientes, utilice 50 ml o 100 ml jarras Coplin. Tenga cuidado de no raspar las células de al mover diapositivas entre frascos.
- Se detiene en 0,1 M Tris-Cl, pH 7,4 durante 10 min a TA.
- Permeabilizar las células en 0,1% saponin/0.1% de Triton X-100 en PBS durante 10 min a TA.
- Lavar dos veces en PBS durante 5 min a TA.
- Incubar durante al menos 20 min en 20% de glicerol / PBS a RT Nota:. En este punto, las diapositivas se pueden almacenar en 50% de glicerol / PBS a -20 ° C durante al menos varias semanas. Se ha observado que el almacenamiento durante al menos unos días resulta en un aumento de la intensidad de la señal 9. Vuelva a calibrar la temperatura ambiente en 20% de glicerol / PBS antes de proceder Nota:. Este es un punto conveniente para iniciar la precipitación de las sondas (véase 2.1.1).
- 3x congelación / descongelación en nitrógeno líquido. Sumerja una diapositiva a la vez en nitrógeno líquido utilizando un pequeño termo. Retirar al cabo de unos segundos y un sonido que hace estallar característico, y el lugar en una toalla de papel para descongelar. Espere a que el glicerol congelado opaca a desaparecer luego congelar de nuevo. Hasta 15 diapositivas se pueden hacer cómodamente en la rotación durante tres ciclos de congelación / descongelación.
- WASH dos veces en PBS durante 5 min a TA.
- Incubar en HCl 0,1 M durante 30 min a TA.
- Lavar en PBS durante 5 min a TA.
- Permeabilizar en 0,5% Triton X-100/PBS saponin/0.5% durante 30 min a TA.
- Lavar dos veces en PBS durante 5 min a TA.
- Equilibrar en 50% formamide/2x SSC durante al menos 10 min a TA.
- Sonda de pipeta en un cubreobjetos. Saque los portaobjetos del frasco y secarse cualquier exceso de líquido alrededor de la mancha célula (s) con una toalla de papel. Al secarse formamida rápidamente, tenga cuidado de no secar las células. Para diapositivas con dos o tres puntos de células, aplique 10 l de la sonda por mancha en un 22 mm x 50 mm cubreobjetos correspondiente a manchar ubicación en la diapositiva. Invertir en cubreobjetos se deslizan sobre terreno (s). Sellar con cemento de caucho y permita que este se seque por completo. Proteger de la luz en todas las etapas posteriores.
- Diapositivas de calor a 78 ° C durante exactamente 2 minutos en un plato caliente (por ejemplo, un bloque de calentamiento invertida, PASO CRÍTICO: ver Discusión). Coloque una cubierta (caja de cartón o sim ilar) sobre la placa caliente para protegerlo de la luz durante la desnaturalización.
- Incubar toda la noche a 37 ° C en una cámara húmeda a prueba de luz. Para humedecer, colocar toallas de papel en una caja hermética a la luz y apagar con agua.
2.2 Día 2
- Prepare jarras Coplin con soluciones a la temperatura adecuada para posteriores etapas de lavado. Proteja las diapositivas de la luz tanto como sea posible en todas las fases posteriores. Un café se hace una buena tapa hermética a la luz de jarras Coplin.
- Retire el cemento de goma y lugar diapositiva en 2x SSC hasta cubreobjetos aflojar y deslice.
- Lave en 50% formamide/2x SSC durante 15 minutos a 45 ° C. Coloque la tapa de tanque de agua para protegerla de la luz.
- Lave en 0,2 x SSC durante 15 minutos a 63 ° C.
- Lave en 2x SSC durante 5 min a 45 ° C.
- Lave en 2x SSC durante 5 minutos a temperatura ambiente.
- Lavar en PBS durante 5 min a TA.
- Teñir con DAPI (5 g / ml en 2x SSC) durante 2 min en una jarra Coplin a TA.
- Destain en PBS durante 5 min a TA.
- Para montar el cubreobjetos, pipeta medio de montaje sobre un cubreobjetos. Utilice 10 l por lugar de células, utilizando 22 mm x 22 mm cubreobjetos de un punto, y 22 mm x 50 mm cubreobjetos durante dos o tres puntos. Seque PBS alrededor de la mancha de células tanto como sea posible, pero tenga cuidado de no secar las células. Invertir cubreobjetos en portaobjetos, y selle con barniz de uñas.
Resultados
El protocolo se describe aquí (Figura 1 para una descripción) se ha utilizado con gran efecto de combinar PEZ ADN con la captura de imágenes de alto rendimiento para el análisis de la organización genómica. La generación de sondas de buena calidad es fundamental para el éxito (ver Discusión). Figura 2 muestra dos controles de calidad importantes para sondas FISH. Después de la mella, una mancha de ADN debe ser visible con la mayor parte de los fragmentos de funcionamiento entre 150 y 700 pb (Figura 2A). Después de acoplamiento químico, la incorporación del colorante fluorescente se puede medir por análisis espectroscópico de la sonda (Figura 2B).
Al utilizar buenas sondas, siguiendo el protocolo PESCADO ADN suele dar lugar a señales de FISH de ADN brillantes sobre fondo bajo. Hemos utilizado con éxito este protocolo en una variedad de diferentes tipos de células, y ya sea con microscopios de epifluorescencia o confocal. Imágenes automatizada utilizando un epi-fluorescencmicroscopio electrónico requiere afilados señales de FISH, fácilmente identificables con poco fondo (Figura 3), mientras que la microscopía confocal requiere señales más intensas. Figura 4A muestra imágenes procesadas representativas. Coordenadas nucleares de señales de FISH se pueden obtener, y las relaciones espaciales de regiones genómicas pueden ser calculadas (por ejemplo, véase la Figura 4B). También hemos utilizado pilas de imágenes obtenidas por microscopía confocal para el modelado 3D de señales de FISH en sus territorios cromosómicos (Película 1).
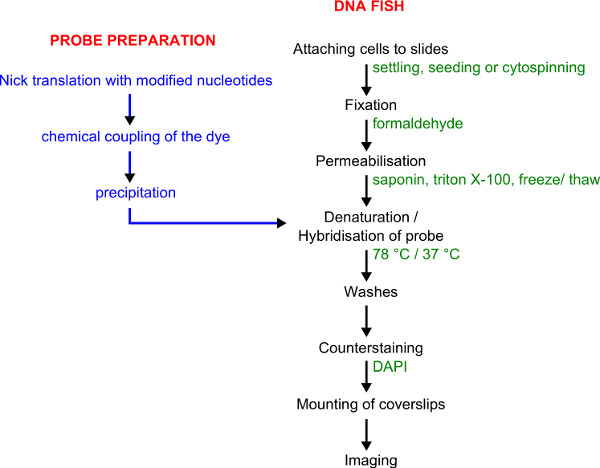
Figura 1. Flujo de trabajo para el etiquetado y FISH sonda de ADN. Preparación de la sonda se muestra en azul. Procedimiento PESCADO ADN se muestra en color negro con detalles dados en verde.
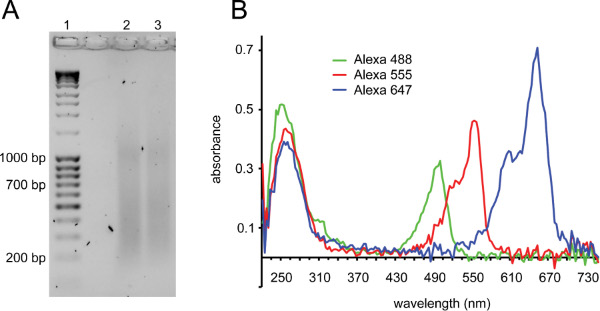
Figura 2. Etiquetado de sondas de la mella y el control de calidad de las sondas marcadas. A) gel que muestra frotis ideal antes de calentamiento para inactivar la DNasa I (carril 1: escalera de ADN, carril 2 y 3: BAC después de la mella). Los fragmentos deben estar en el rango de 150-700 pb. Una prueba adicional de ~ 1 kb se observa frecuentemente con reacciones nick de traducción con éxito. B) microvolumen análisis completo espectro de sondas marcadas. Un pico de ADN a 260 nm se observa con un segundo pico correspondiente a la longitud de onda de absorbancia del fluoróforo de la sonda se marca con: Alexa Fluor 488: 495 nm, Alexa Fluor 555: 555 nm, Alexa Fluor 647: 650 nm. Se muestra son tres sondas con buena constitución: picos de absorción fluoróforos son una altura o más alto que los picos de ADN similar. Alexa Fluor 647 típicamente tiene una mayor absorbanciade Alexa Fluor 555, que a su vez tiene mayor absorbancia de Alexa Fluor 488. Alexa Fluor 488 también absorbe algo de luz a 260 nm, por lo tanto, el primer pico de esta sonda es más alto que los demás. Sondas con mala incorporación espectáculo absorbancia fluoróforo 2-3 veces menor que la absorción de ADN. Las sondas con una más de 2 veces mayor pico fluoróforo que el ADN pico típicamente contienen colorante no conjugado que puede causar fondo en el FISH.
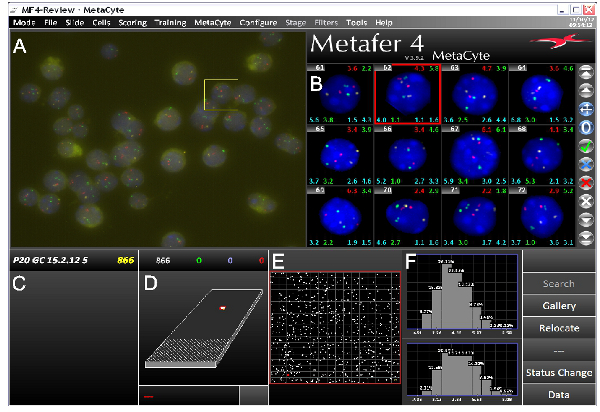
Figura 3. Captura de pantalla del software de imágenes 3D peces MetaCyte muestra un experimento típico pez de tres colores. La pantalla se divide en varias partes. A) Imagen de campo de visión (FOV) Raw capturado por el Metafer / sistema de escaneo de diapositivas MetaCyte. B) Galería de imágenes de núcleos identificado que tienen las Naciones Unidasprocesamiento de imágenes dergone y la segmentación de las señales de FISH. Varios tipos de datos se pueden mostrar en cada imagen de la galería. En este ejemplo, el número de células se muestra arriba a la izquierda con diversos interallelic y distancias interlocus arriba a la derecha y a cada lado por debajo de cada imagen. C) Nombre de diapositivas con el número de núcleos identificado en el análisis. D) Representación de la corredera con el área escaneada por el sistema. E) Ampliación de la zona explorada. Los puntos blancos representan núcleos identificados por FOV. F) Los datos procesados. En este ejemplo se muestran las distancias máximas entre interallelic señales verdes (arriba) y las señales de color rojo (inferior). Los datos que se muestran son de ratón P20 células germinales. Las sondas se encuentran en cromosomas diferentes, que abarcan Hbb y genes HBA y un grupo de histonas. Haz clic aquí para ver más grande la figura .
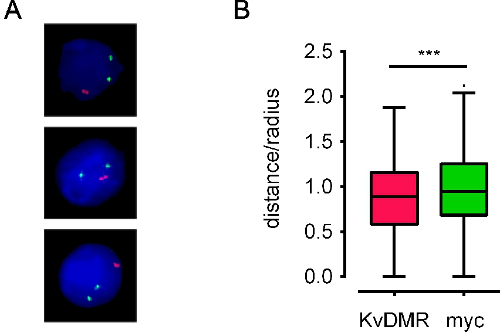
La Figura 4. Los ejemplos de imágenes procesadas y datos posicionales derivados. A las imágenes) Elaboradas para E13.5 células de hígado fetal de ratones teñidos con sondas BAC situados en diferentes cromosomas que cubren Myc y Kcnq1 genes (llamados myc y KvDMR, de color verde y rojo, respectivamente). B) Tukey parcelas box-whisker representan la distancia interallelic distribución de los mismos loci en 600 núcleos. Distancia alélica se representará gráficamente como una fracción del radio del núcleo '(distancia / radio) para dar cuenta de las variaciones en el tamaño nuclear. Núcleos de células ES tienen un radio de alrededor de 5 micras. Sondas rojas son mucho más juntos que sondas verdes (***: p <0,01, prueba t no pareada). Los datos de 10.
Movie 1. Modelado 3D de dos loci dentro de su cromosoma territorio. Dos sondas BAC ubicados cerca de los extremos del cromosoma 7 del ratón fueron etiquetados (rojo y rojo lejano, (blanco pseudo-color)) y se hibridan con una pintura de todo el cromosoma (verde) a las células madre embrionarias de ratón. Haz clic aquí para ver la película .
Discusión
3D PEZ ADN se ha convertido en una herramienta ampliamente reconocido para analizar disposiciones espaciales en el núcleo. Mientras que el pescado proporciona resultados visuales y directa, como con todas las técnicas, hay que ser conscientes de sus limitaciones. PEZ ADN se enfrenta el problema inherente de que se necesitan tratamientos relativamente duras para hacer accesible la cromatina para sondas FISH que en última instancia interrumpe la estructura-la misma cosa nuclear que se va a analizar. Varias estrategias se han seguido para hacer malabares con la accesibilidad y la conservación de la estructura. En el protocolo descrito aquí, el ADN cromosómico se hace accesible por congelación-descongelación permeabilización de las células y la desnaturalización por calor antes de la hibridación de la sonda. Solovei, et al., (2002) analiza los cambios estructurales después de un tratamiento similar y se encontró que el desplazamiento medio de dominios de la cromatina era 300 nm, lo que limita la resolución del análisis estructural a aproximadamente 1 Mb regiones en el genoma 8. Si bien esto no es de alta resolución, sees una escala razonable para el análisis de la posición nuclear.
Una consideración más práctica para el análisis de FISH es que la adquisición y mediciones de distancias nucleares imagen son procesos que consumen tiempo que limitan el número de núcleos que se puede analizar. Para el estudio de los eventos poco frecuentes que nos propusimos hacer imágenes de alto rendimiento automatizado. Entonces, con los controles adecuados en su lugar, un número suficiente se pueden comparar para permitir un análisis estadístico robusto de las relaciones espaciales. Se analiza rutinariamente 600 núcleos por punto de datos para la que se determina las coordenadas 3D de todas las señales de FISH en el volumen nuclear. Estos datos requieren muy poco espacio de almacenamiento y permitir el análisis de las distancias inter-e intra-alélica, o posiciones radiales en cualquier momento posterior. Por otra parte, el procesamiento automatizado se puede hacer de una manera ciega investigador que reduce en gran medida la probabilidad de sesgo inconsciente.
Para habilitar este tipo de analys de alto rendimientoes que nuestro objetivo era establecer una forma rápida y eficiente de realizar PESCADO ADN. Encontramos el protocolo descrito aquí para ser extremadamente robusto, produciendo constantemente señales luminosas con bajo fondo. Se trabajó para todos los tipos de células que probamos siempre y cuando adaptó la etapa de fijar las células a la diapositiva. Por otra parte, con una excepción, todos los BACs que utilizamos podrían convertirse en sondas luminosas. También hemos detectado con éxito una serie de proteínas nucleares mediante la tinción de anticuerpos después PESCADO ADN. Mientras que para algunas proteínas que esto funciona así, se recomienda la comparación con inmunofluorescencia tradicional (IF) para asegurar la consistencia del patrón de tinción de anticuerpos entre IF y el ADN inmuno FISH (comparación 11).
En general, se encontró que las sondas marcadas en diferentes colores producen los mismos resultados. Sin embargo, los siguientes aspectos deben ser considerados al momento de elegir el tinte de etiquetado. En primer lugar, el color del fluoróforo tiene que ser así detectable por el microscopio used. En segundo lugar, la longitud de onda de la luz emitida por los fluoróforos debe ser suficientemente diferente para evitar sangrado a través en el otro canal. Esto dependerá de los filtros en el microscopio. Y en tercer lugar, al menos en nuestras manos, algunos colores producen señales luminosas más consistente que los demás. Siempre hemos utilizado DAPI (azul) como de contraste que hace Alexa Fluor 555 (rojo), 488 (verde) y 647 (rojo lejano) buenas opciones para marcar sondas. Con nuestro sistema de formación de imágenes, encontramos Alexa Fluor 555 (rojo) para producir las señales más brillantes, seguido por Alexa Fluor 488 (verde). Alexa Fluor 647 (rojo lejano) tiene la desventaja de que no puede ser detectada por el ojo humano y por lo tanto no hay señales puede ser visto cuando mirando hacia abajo el microscopio. Por lo tanto, si al hacerlo PESCADO dos colores, se recomienda la combinación de tintes rojos y verdes.
Pueden surgir dos problemas principales: las células de lavado de las diapositivas y las señales débiles. Cómo las células y se adhieren a la diapositiva es entr enormemente variablestipos de células een, y el mejor protocolo para resolver / semillas de las células tiene que ser determinado. Hemos utilizado con éxito ya sea con carga positiva o portaobjetos recubiertos con poli-L-lisina (comprado listo para usar), pero encontramos células generalmente se adhirieron mejor a portaobjetos recubiertos de poli-L-lisina. También se encontró que cuando las células eran zonas enteras demasiado densos haría pelar como una hoja, mientras que las imágenes automatizado se vio obstaculizado si las células eran demasiado escasa. Por lo tanto, si la muestra no es demasiado precioso, diferentes números de células se sembraron para determinar la densidad ideal. Si las células son particularmente propensos a lavar, una pluma barrera hidrófoba se puede utilizar, y pasos de FISH se puede realizar por pipeteo cuidadosamente soluciones directamente sobre el portaobjetos. Pipetear también reduce drásticamente los volúmenes de reactivos necesarios, pero lleva mucho más tiempo al hacer varias diapositivas.
Señales débiles son por lo general debido a las sondas pobres, y es bien vale la pena invertir tiempo en hacer buenos. Clean BAC ADN debe utilizard como material que puede ser obtenido por precipitación repetida o kits disponibles comercialmente de partida. La contaminación con ADN genómico bacteriano afecta negativamente a la calidad de la sonda y el manejo cuidadoso durante la lisis celular y la filtración del lisado celular se requiere para mantener a un mínimo. Es, es, sin embargo, no es necesario utilizar un paso exonucleasa para digerir ADN lineal, ya que esto reduce en gran medida el rendimiento. Etiquetado de la eficiencia de la sonda es crucial. El control de calidad más importante de este paso es la comprobación del tamaño de los fragmentos después de la traducción nick (ver resultados representativos). Una "buena" desprestigio casi siempre como resultado una sonda de colores brillantes. Sin embargo, hemos encontrado que de vez en cuando una cierta BAC no se puede procesar en una buena sonda, y un BAC diferente debe ser elegido. Otro paso importante es la desnaturalización por calor. Si la temperatura es demasiado baja, el ADN será sólo parcialmente desnaturalizado y la hibridación de la sonda se verá perjudicada. Si la temperatura es demasiado alta, la estructura wi nuclearll ser perturbado más de lo necesario. En nuestras manos, 78 ° C en un bloque caliente invertida es el compromiso ideal, pero esto puede variar dependiendo del bloque caliente respectivos usado.
Hemos tenido buenos resultados con medio de montaje Vectashield, pero encontró que SlowFade oro tiene menos fondo y preserva la señal fluorescente durante más tiempo. No se recomienda el uso de medio de montaje dura-set, ya que encontramos que esto afecte la estructura 3D y formar burbujas de aire en el tiempo.
En conclusión, brillantemente fluorescentes sondas marcadas directamente junto con el protocolo de PEZ ADN descrito aquí ofrecen una solución eficiente para el análisis de forma fiable y rápida de la arquitectura nuclear, que es aplicable a una amplia variedad de cuestiones biológicas.
Divulgaciones
La producción de este artículo fue parcialmente patrocinado por Carl Zeiss y MetaSystems.
Agradecimientos
Este trabajo fue financiado por el BBSRC y el Wellcome Trust. Nos gustaría dar las gracias a Simon Walker para obtener ayuda con imágenes y Felix Krueger para ayudar con el análisis bioinformático.
Materiales
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||||||||
| Reagent/Material | |||||||||||||||||||||||||||||||||||||||
| NTB buffer | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| DTT | Invitrogen | D-1532 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| dNTPs | Bioline | BIO-39025 | Step 1.1.1 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Aminoallyl-dUTP | Ambion | AM8439 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNA Polymerase I | New England Biolabs | M02095 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| DNase I recombinant RNase free | Roche | 4716728001 | Step 1.1.1 | ||||||||||||||||||||||||||||||||||||
| QIAquick PCR purification kit | Qiagen | 28104 | Step 1.1.4, 1.2.3 | ||||||||||||||||||||||||||||||||||||
| NaOAc | VWR | 27653 | Step 1.1.5, Step 2.1.1.2 | ||||||||||||||||||||||||||||||||||||
| NaHCO3 | Sigma | S5761 | Step 1.2.1 | ||||||||||||||||||||||||||||||||||||
| Alexa Fluor Reactive Dye Decapacks for Microarray Applications | Invitrogen (Molecular Probes) | A32750, A32756, A32757 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| DMSO (anhydrous) | Sigma Aldrich | 276855 | Step 1.2.2 | ||||||||||||||||||||||||||||||||||||
| SYBR Safe DNA gel stain | Life Technologies | S33102 | Step 1.2.4 (alternative to ethidium bromide) | ||||||||||||||||||||||||||||||||||||
| Cot-1 DNA | Invitrogen | 18440-016 | Step 2.1.1.1 (Cot-1 DNA can be home-made in large quantities and works just as well) | ||||||||||||||||||||||||||||||||||||
| Single stranded DNA from salmon testes | Sigma | D7656 | Step 2.1.1.1 | ||||||||||||||||||||||||||||||||||||
| Deionised formamide | Sigma | F-9037 | Step 2.1.1.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| Dextran sulphate | Sigma Life Sciences | D8906 | Step 2.1.1.4 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| XMP painting probes | MetaSystems | 000000-0528-837 | Step 2.1.1.4 | ||||||||||||||||||||||||||||||||||||
| Poly-L-lysine coated slides | Sigma Aldrich | P0425 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| Hydrophobic pen (ImmEdge) | Vector Laboratories | H-4000 | Step 2.1.2 | ||||||||||||||||||||||||||||||||||||
| 70 μm sieve (Mouse f–tal liver cells only) | BD Falcon | 352350 | Step 2.1.2.1 | ||||||||||||||||||||||||||||||||||||
| PFA | Sigma Aldrich | P6148 | Step 2.1.3 (Flammable, corrosive, acute toxicity, health hazard) (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Tris-Cl | Step 2.1.3 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Saponin from Quillaja bark | Sigma Aldrich | 47036 | Step 2.1.4, 2.1.11 (Acute toxicity), (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||
| Triton X-100 | Sigma | T9284 | Step 2.1.4, 2.1.11 (Acute toxicity, hazardous to the environment) | ||||||||||||||||||||||||||||||||||||
| 10 x PBS | Life Technologies (GIBCO) | 70011-036 | Step 2.1.4, 2.1.5, 2.1.6, 2.1.8, 2.1.10, 2.1.11, 2.1.12, 2.2.7, 2.2.9, | ||||||||||||||||||||||||||||||||||||
| Glycerol | Fisher Scientific | G/0650 | Step 2.1.6 | ||||||||||||||||||||||||||||||||||||
| HCl | VWR | 20252 | Step 2.1.9 (Acute toxicity, corrosive) | ||||||||||||||||||||||||||||||||||||
| Formamide | Sigma Aldrich | 47670 | Step 2.1.14, 2.2.3 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SSC | Step 2.1.14, 2.2.2-2.2.6, 2.2.8 (See 'FISH buffers and reaction mixes' list below) | ||||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 22 | Menzel-Glaser | BB022022A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Coverslips 22 x 50 | Menzel-Glaser | BB022050A1 | Step 2.1.15 | ||||||||||||||||||||||||||||||||||||
| Rubber cement | Marabu | 2901 (10 000) | Step 2.1.15 (Flammable, health hazard, danger to the environment) | ||||||||||||||||||||||||||||||||||||
| DAPI | Invitrogen Molecular Probes | D3571 | Step 2.2.8 (Health hazard) | ||||||||||||||||||||||||||||||||||||
| SlowFade Gold | Invitrogen | S36936 | Step 2.2.10 (mounting medium) | ||||||||||||||||||||||||||||||||||||
| Clear nail polish | Any supplier | Step 2.2.10 | |||||||||||||||||||||||||||||||||||||
| Equipment | |||||||||||||||||||||||||||||||||||||||
| Nanodrop 2000 | Thermo Scientific | Step 1.1.6, 1.2.4 (microvolume spectroscopy) | |||||||||||||||||||||||||||||||||||||
| Typhoon FLA 7000 phosphoimager | GE Life Sciences | Step 1.2.4 | |||||||||||||||||||||||||||||||||||||
| Coplin jars | Sigma-Aldrich | S6016 (6EA)/S5516 (6EA) | Step 2.1.3 onwards | ||||||||||||||||||||||||||||||||||||
| Liquid nitrogen dewar | Any supplier | Step 2.1.7 | |||||||||||||||||||||||||||||||||||||
| Base with Black Lid (light-tight chamber) | Simport | M920-2 | Step 2.1.17 | ||||||||||||||||||||||||||||||||||||
| Thermomixer comfort | Eppendorf | 5355 000.011 | Step 2.2.3 (or use shaker in a 37 °C room) | ||||||||||||||||||||||||||||||||||||
| Imaging and Image processing | |||||||||||||||||||||||||||||||||||||||
| Metafer - Imaging Automation Platform | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| MetaCyte - Automated Interphase FISH analysis | MetaSystems | automated imaging software | |||||||||||||||||||||||||||||||||||||
| Axio Imager Z2 upright | Zeiss | epifluorescence microscope used with automated imaging | |||||||||||||||||||||||||||||||||||||
| IX81 confocal microscope (FV1000) | Olympus | confocal microscope | |||||||||||||||||||||||||||||||||||||
| Bitplane, version 7.3 | Imaris | image analysis and 3D modelling software | |||||||||||||||||||||||||||||||||||||
| Scientific volume Imaging, version 4.1 | Huygens | image analysis and deconvolution software | |||||||||||||||||||||||||||||||||||||
FISH buffers and reaction mixes
| |||||||||||||||||||||||||||||||||||||||
Referencias
- Chakalova, L., Debrand, E., Mitchell, J. A., Osborne, C. S., Fraser, P. Replication and transcription: shaping the landscape of the genome. Nat. Rev. Genet. 6, 669-677 (2005).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (1016).
- de Wit, E., de Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes Dev. 26, 11-24 (2012).
- Sambrook, J., Russell, D. . Molecular Cloning: A laboratory Manual. , (2000).
- Boyle, S., Rodesch, M. J., Halvensleben, H. A., Jeddeloh, J. A., Bickmore, W. A. Fluorescence in situ hybridization with high-complexity repeat-free oligonucleotide probes generated by massively parallel synthesis. Chromosome Res. 19, 901-909 (2011).
- Brown, K. Visualizing nuclear proteins together with transcribed and inactive genes in structurally preserved cells. Methods. 26, 10-18 (2002).
- Hewitt, S. L., Yin, B., et al. RAG-1 and ATM coordinate monoallelic recombination and nuclear positioning of immunoglobulin loci. Nat. Immunol. 10, 655-664 (2009).
- Solovei, I., Cavallo, A., et al. Spatial preservation of nuclear chromatin architecture during three-dimensional fluorescence in situ hybridization (3D-FISH. Experimental Cell Research. 276, 10-23 (2002).
- Solovei, I., Beatty, B., Mai, S., Squire, J. FISH on three-dimensionally preserved nuclei. FISH: A Practical Approach. , (2002).
- Krueger, C., King, M. R., et al. Pairing of homologous regions in the mouse genome is associated with transcription but not imprinting status. PLoS One. 7, e38983 (2012).
- Solovei, I., Cremer, M., Bridger, J. M., Volpi, E. V. 3D-FISH on Cultured Cells Combined with Immunostaining. Fluorescence in situ hybridization (FISH): Protocols and Applications. , (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados