Method Article
CRISPR/Cas12a 多路复用基因组编辑糖母细胞和酵母像素艺术的创作
摘要
CRISPR/Cas12a 系统与单个 crRNA 阵列相结合,可同时在多个位点对S. cerevisae基因组进行高效的多路复用编辑。这通过构建产生酵母菌株的类胡萝卜素来证明,这些菌株随后用于创建酵母像素艺术。
摘要
高效率、易用性和多功能性的集群定期间隔短纤维重复/CRISPR相关蛋白9(CRISPR/Cas9)系统促进了高级基因改造的糖霉素,一个模型工业生物技术中的有机体和主力。CRISPR相关蛋白12a(Cas12a),一种RNA引导的内切酶,其特征与Cas9不同,因此进一步扩展了用于基因组编辑的分子工具箱。CRISPR/Cas12a系统的一个优点是,它可用于多路基因组编辑,从单个转录单元(单个CRISPRRNA(crRNA)阵列表达的多个导导RNA。我们使用CRISPR/Cas12a系统,用单个crRNA阵列构造表达的多个crRNA表达的CRISPR/Cas12a系统,提出了一种将多个异构基因多路整合到S.cerevisae基因组独立位点的方案。该方法利用S.cerevisae在体内对DNA片段进行重组的能力,将单个crRNA阵列组装成一个质粒,可用于转化剂选择,以及供体DNA的组装以预期位置集成到基因组中的序列。Cas12a 是预先表达的构成,在单crRNA阵列的表达时,促进S.cerevisae基因组在预期位置的裂解。该协议包括设计和构建单个crRNA阵列和供体DNA表达盒,并利用独特的50bpDNA连接器序列和单独的集成侧翼DNA序列的集成方法,简化了实验设计通过标准化和模块化,并扩展了应用范围。最后,我们演示了一种简单的技术,即使用声学液体处理程序创建酵母像素艺术,使用不同颜色的类胡萝卜素生成构建的酵母菌株。
引言
CRISPR/Cas酶无疑已经彻底改变了分子生物学,并被广泛地用作工程基因组的工具,其速度以前是不可行的。DiCarlo等人报告了CRISPR/Cas9基因组编辑系统对糖霉菌基因组的首次修改。2、使用外部引入的寡核苷酸证明成功的基因敲除和进行点突变。进一步的酵母CRISPR工具箱发展包括:通过融合催化非活性死亡Cas9(dCas9)与转录效应器域的转录调节,使转录3的激活和沉默,适用于两者基因组编辑和调节功能代谢通路工程通过同时激活,抑制和删除4,从S.cerevisae基因组5删除大片段,和多染色体融合6.
CRISPR/Cas基因组编辑系统在细菌和古生物的适应性免疫系统中找到了它们的起源,这些系统已被分子生物学家用于基因组编辑。其功能基于聚类定期间隔短单体重复 (CRISPR) DNA 区域编码 RNA 负责识别外来 DNA 或 RNA 和 CRISPR 相关基因 (Cas), 编码RNA引导内西西莱西1,7,8,9 。根据最近对CRISPR/Cas系统的基因组分析,建议将CRISPR/Cas系统划分为两个类、5个类型和16个亚型10。这两个类根据目标裂解中涉及的效应复合物的组织进行区分。通常,具有多子单元组织的 CRISPR/Cas 系统被归类为类 1,而单个子单元效应器复合体属于类 210,11。在本文中,我们将探讨 2 类 V Cas12a,以前称为 Cpf110,12,这是 2 类 II Cas9 的替代方法。虽然Cas9具有很好的特征,并广泛用于研究,Cas12a提供了附加功能12。首先,Cas12a与CRISPRRNA(crRNA)形成42至44个核苷酸的复合物,无需额外的转活CRISPRRNA(tracrRNA)。因此,与CRISPR/Cas9相比,CRISPR/Cas12a系统可用于基因组编辑,较短的导引RNA。其次,Cas12a独特的内西酸酶和内分酶活性使其在crRNA13前成熟。此 RNase 活性允许在单个 CRISPR crRNA 阵列上编码多个 crRNA,而 Cas9 要求每个所谓的单导RNA (sgRNA) 的单独表达,或者例如附加内切酶的表达(例如,Csy4)结合识别图案的Csy4围绕每个sgRNA14,15。第三,Cas12a 目标站点识别需要从目标位于 5' 端的原型空间相邻图案 (PAM),并在 PAM 的 ±18/+23 位置后从其 PAM 上分离,导致带有粘性端的 DNA 被分离,而 Cas9 要求 PAM 位于 3' 端从目标并切割后-3位置创建钝端削减在DNA12。第四,PAM的共识核苷酸序列在Cas12a(T)TTV和Cas9(NGG)之间有所不同,这使得Cas12a成为瞄准T-富启动子和终结者序列16的有希望的候选者。最后,最近的一项研究报告,Cas12a的目标特异性比原生Cas917更大。
我们提出了使用CRISPR/Cas12a系统进行S.cerevisae基因组编辑的协议,特别注重将多个DNA表达盒同时引入独立的基因组位点(多路基因组编辑)。单个 crRNA 阵列。协议的关键步骤如图1所示。作为概念的证明,CRISPR/Cas12a系统应用于将三个表达盒引入S.cerevisae的基因组,从而能够生产如图2所示的β-胡萝卜素18。β-胡萝卜素的产生影响S.cerevisae的表型:即,在成功引入类胡萝卜素生物合成所需的所有三个异源基因后,白色S.cerevisae细胞变成黄色或橙色,取决于每个基因的启动子的表达强度。由于该通路的视觉读出简单,已引入开发先进的基于CRISPR的系统和方法,用于基因组编辑19,20。在这项工作中,编码类胡萝卜素基因crtE、crtYB和crtI的表达盒使用金门克隆(GGC)方法21与异源启动子和同源终止器用于驱动基因的表达。表达盒周围有独特的 50 碱基对 (bp) 序列,称为连接器,允许具有相同 50 bp 序列的集成侧翼 DNA 序列(侧翼区域)的体内组装,以及后续集成在侧翼区域确定的位置进入酵母的基因组DNA。通过使用不同的促进剂强度,获得不同水平的类胡萝卜素生产菌株,导致细胞颜色的变化。这些菌株-灵感来自"酵母艺术项目"22 - 用于一个斑点设置与声学液体处理程序,以创建罗莎琳德富兰克林的4色高分辨率"酵母照片"。富兰克林(1920-1958)是英国化学家和X射线晶体学家,以她对第5123、24、25号DNA结构的发现做出了贡献而闻名。
研究方案
1. Cas12a质粒的制备
注:含有Lachnospiracea细菌ND2006 Cas12a(LbCpf1,pCSN067)的质粒,在S.cerevisiae中针对表达进行了优化,先前构造了19个,沉积在质粒储存库(见表) 材料)。这是一个单拷贝表皮S.cerevisae/大肠杆菌穿梭质粒,含有KanMX抗标记基因,允许在遗传基因(G418)上选择S.cereviae转化剂。
- 获取 pCSN067 质粒(参见材料表)。
- 放大pCSN067质粒,获得高量。
- 根据制造商的协议,用质粒 pCSN067 将购买的具有化学能力的大肠杆菌细胞的 25 μL 进行转化。在 2x 肽酵母 (PY) 中稀释转化混合物 10 和 50 次。在含有阿霉素(0.1 g/L)的2x PY琼脂板上将10倍和50倍稀释板,并在37°C下孵育过夜。
- 选择2至3个菌落,在3 mL的2x PY中接种,并在180 rpm的摇动培养箱中以37°C在37°C生长过夜。
- 根据制造商的说明,使用质粒纯化试剂盒纯化质粒。
2. 制备单crRNA阵列表达盒
- 准备单个 crRNA 阵列。
注:单crRNA阵列包括S.cerevisiae2的SNR52RNA聚合酶III启动子,对LbCas12a和间隔器(基因组靶序列)的直接重复,每个靶点19和末端一起重复与SUP4终结器从S.cerevisae2.单crRNA阵列通过体内重组组合成线性质粒pRN1120来生成圆形质粒,因此在单个crRNA阵列的开始和结束时必须存在与质粒同源区pRN1120的区域(见图2A).建议提前评估一些设计crRNA的功能分别19。此信息随后用于选择大多数功能的 crRNA,将这些组合到直接重复序列和间隔序列中,以创建用于多路复用目的的单个 crRNA 数组。- 将单crRNA阵列作为合成DNA订购为多路基因组编辑实验(参见补充表1中单crRNA阵列的DNA序列)。
- 放大有序的单 crRNA 阵列(例如,使用引注 KC-101 和 KC-102(补充表2)。制备含有:0.5 μL的DNA聚合酶,DNA聚合酶所需的10μL5x缓冲液,1μL的10mM dNTPs,2.5μL的10μM正向底漆,2.5μL的10μM反向底漆,2μL的DNA模板,浓度为5纳克/μL和超纯H2O 的总体积为 50 μL。
- 使用以下程序在热循环器中执行反应:(i) 98 °C 3 分钟,(ii) 98 °C 10 秒,(iii) 60 °C 20 秒,(iv) 72 °C 15 s = 重复步骤 (ii) 到 (iv) 30 次;(v) 72 °C 在 12°C 保持 5 分钟 (vi) 直到进一步分析。
- 使用DNA加载染料和DNA阶梯,在100至10,000 bp范围内使用DNA加载染料和DNA阶梯,在5V/cm的0.8%agarose凝胶上运行样品,通过电泳分析PCR产物。
- 根据制造商的说明,使用 PCR 纯化套件纯化 PCR 产品。
- 准备单crRNA阵列受体质粒。
注: 单 crRNA 阵列由S. cerevisae/大肠杆菌穿梭质粒 pRN112019表示(参见材料表)。这种多拷贝质粒含有NatMX抗标记基因,允许在诺西辛(NTC)上选择S.cerevisae转化剂。- 获得pRN1120质粒。
- 放大pRN1120质粒,获得高量。
- 根据制造商的协议,用质粒 pRN1120 将购买的具有化学能力的大肠杆菌细胞的 25 μL 进行转化。在 2x PY 中稀释转换组合 10 和 50 次。在含有阿霉素(0.1 g/L)的2x PY琼脂板上将10倍和50倍稀释板,并在37°C下孵育过夜。
- 选择2至3个菌落,在3 mL的2x PY中接种,并在180 rpm的摇动培养箱中以37°C在37°C生长过夜。
- 根据制造商的说明,使用质粒纯化试剂盒纯化质粒。
- 使用EcoRI-HF 和XhoI 线性化质粒 pRN1120。为此,准备一个消化混合物,由1μg的pRN1120,5μL的10倍缓冲液(1x缓冲液含有50mM醋酸钾,20mM三酸酯,10mM醋酸镁,100μg/mL牛血清白蛋白[BSA];pH 7.9),1μL的生态RI-HF(20 U),Xho I (20 U) 的 1 μL 和超纯 H2O 的总体积为 50 μL。在 37°C 孵育消化混合物 2 小时,在 65°C 下灭活 20 分钟。
- 使用DNA加载染料和DNA阶梯,在100至10,000 bp范围内,通过电泳在胶原蛋白(0.8%、40分钟、5 V/cm)上通过电泳分析线性质粒。作为对照,在分析中包括圆形质粒。
- 根据制造商的说明,使用 PCR 纯化试剂盒纯化线性质粒。
3. 制备促进者-ORF-终结器(POT)供体DNA结构
- 订购一组不同强度的启动子 (P),开放读取帧 (O) 和终结器 (T) 序列作为合成 DNA,使每个元素包含由BsaI 站点两侧的标准化 4 bp 识别序列,以使金门克隆 ()GGC) 装配体 26(参见补充表 3中的详细设计和补充表 4中的序列 )。
- 使用 GGC 反应21将由启动子、打开读取帧、终结器和连接器序列组成的 POT 表达盒组装到已包含预先指定的 50 bp 连接器序列的目标向量中(见补充表4和参考文献26,27。
- 使用分光光度计测量DNA部件的浓度。将超纯H2 O中的每个DNA部分稀释至15 fmol/μL的最终浓度。
- 准备由DNA片段组成的反应混合物:2 μL的启动子,2μL的开放阅读框架,2μL的终结者和2μL主干(26中所述的1级目标载体),4μL的5xT4DNA连带缓冲液,2.5μL的1U/μL T4维加他命,1.5 μL 20 U/μL BsaI-HF 和超纯 H2O,总体积高达 20 μL。
- 使用以下程序在热循环器中执行 GGC 反应:(i) 37 °C 2 分钟,(ii) 16 °C 5 分钟 = 重复步骤 (i) 和 (ii) 50 次;(iii) 50°C 60 分钟,(iv) 80 °C 45 分钟,(v) 在 12°C 保持,直到进一步分析。
- 根据制造商的协议,将购买的具有化学能力的大肠杆菌28细胞的 25 μL 转化为 3 μL 的 GGC 反应混合物。在 2x PY 中稀释转换组合 10 和 50 次。在含有阿霉素(0.1 g/L)的2x PY琼脂板上将10倍和50倍稀释板,并在37°C下孵育过夜。
- 选择2至3个菌落,在3 mL的2x PY中接种,并在180 rpm的摇动培养箱中以37°C在37°C生长过夜。
- 根据制造商的说明,使用质粒纯化试剂盒纯化质粒。
- 检查 PCR 在 GGC 反应中是否正确组装 POT 表达盒。
- 设计引体,以补充每个表达盒的开头和结尾的连接器序列(参见图 2B)。对于该协议中选择的连接器,请使用引体 KC-103 到 KC-108(参见补充表 2)。
- 为每个含有的质粒制备PCR扩增混合物:0.5 μL校对DNA聚合酶,DNA聚合酶所需的10μL5x缓冲液,1μL的10mM dNTPs,2.5 μL的10μM正向底漆,2.5μL的10μM反向底漆,2μL的DNA模板与浓缩剂5 纳克/μL 的 ion 和超纯 H2O 的总体积为 50 μL。
- 使用以下程序在热循环器中执行 PCR 反应:(i) 98 °C 3 分钟,(ii) 98 °C 10 秒,(iii) 60 °C 20 秒,(iv) 72 °C 2 分钟 30 秒 = 重复步骤 (ii) 至 (iv) 30 次,(v) 72°C 为 5 分钟,(vi)保持在12°C,直到进一步分析。
注:生成的 PCR 产品由 5' 连接器、启动器、打开读取框、终结器和 50 bp 的 3' 连接器组成。
- 使用DNA加载染料和DNA阶梯,在100至10,000 bp范围内使用DNA加载染料和DNA阶梯,在5V/cm的0.8%agarose凝胶上运行样品,通过电泳分析PCR产物。
4. 制备包含连接器序列的集成侧翼DNA序列
- 从野生类型S.cerevisiae CEN.PK113-7D29中纯化基因组DNA。
- 在 500 mL 摇瓶中生长应变,其中填充了 100 mL 的酵母提取物肽葡萄糖 (YEPD, 2% 葡萄糖) 培养基,在 30°C 下在 250 rpm 下摇动 48 小时。
- 在16,000 x g下,通过2mL的汤离心来收获细胞1分钟,然后丢弃上清液。
- 用RNase(10μL,10mg/mL)和酵母溶酶(4μL)将细胞重新悬浮在生理盐中(200μL;0.85%NaCl溶液)。在37°C下孵育细胞悬浮液15分钟。
- 加入300μL的细胞解液(见材料表)和涡流。
- 加入168μL的蛋白质沉淀溶液(见材料表)和涡旋大力20s。
- 在16,000 x g和4°C下离心分离蛋白质分数10分钟。在新管中收集600μL的上清液,并很快与600μL的异丙醇和涡旋混合。
- 在室温下以16,000 x g旋转10分钟,回收DNA。
- 用200μL的乙醇(70%)清洗颗粒。在室温下在16,000 x g下离心10分钟,并去除上清液。在室温下孵育管子10分钟,打开盖子,蒸发乙醇。
注:如果管中的液体仍然可见,请重复步骤 4.1.8。不要将颗粒干燥超过 10 分钟,以防止 DNA 的溶解度降低。 - 将DNA溶解在50μL的TE缓冲液中。将纯化DNA储存在4°C。
- 对于每个整合位点,设计集成侧翼DNA序列(约500 bp),这样在引入供体DNA后,将去除大约1000 bp的基因组DNA(参见图2B中的原理图设计和补充中的序列表 4)
- 设计引体,以 PCR 生成齿面区域。
- 对于左侧侧翼区域,设计正向和反向引体,以放大位于感兴趣积分位点 5'(左)的基因组 DNA 区域约 500 bp。
注:正向引基包括20 bp的同源与预定的侧翼区域。反向引基包括 20 bp 与预期齿面区域同源性,并包含所需的 50 bp 连接器序列,以便在以后的基因组 Cas12a 编辑中实现体内组装。 - 对于右侧侧翼区域,设计正向和反向引体,以放大位于感兴趣积分位点 3'(右)的基因组 DNA 区域约 500 bp。
注: 正向引基包括 20 bp 与预期齿面区域同源,并包含所需的 50 bp 连接器序列,以便在稍后的 Cas12a 基因组编辑中启用体内组装。反向引基包括20 bp的同源与预定的侧翼区域。
- 对于左侧侧翼区域,设计正向和反向引体,以放大位于感兴趣积分位点 5'(左)的基因组 DNA 区域约 500 bp。
- 使用设计引脚剂放大侧翼区域(例如,补充表 2中随附的引注 KC-109 到 KC-120)。
- 测量作为PCR模板的纯化基因组DNA的浓度。将DNA浓度调整至50纳克/μL。
- 制备 PCR 扩增混合物,由基因组 DNA (1 + 4 μL 的 50 纳克/μL 基因组 DNA 稀释) 中纯化步骤 4.1, 正向和反向底漆 (10 μM 每个), 1 μL 10 mM dNTP, 10 μL 5x 缓冲液需要的 DNA 聚合酶, 0.5 μL DNA 聚合酶 (1.0 U)和超纯H2O,总体积高达50μL。
- 使用以下程序在热循环器中执行 PCR:(i) 98 °C 3 分钟,(ii) 98 °C 20 秒,(iii) 60 °C 20 秒,(iv) 72 °C 15 秒,重复步骤 (ii) 到 (iv) 30 次,(v) 72 °C 5 分钟,(vi)保持在12°C,直到进一步分析。
- 使用DNA加载染料和DNA阶梯,在5V/cm的0.8%甘蔗凝胶上通过电泳分析PCR产物,DNA片段范围为100至10,000 bp。
- 根据制造商的说明,使用 PCR 纯化套件纯化正确的 PCR 产品。
5.向S.cerevisae的转型
注:使用基于Gietz等人开发的方法的协议执行转换。(1995)30和希尔等人。31,可用于S.cerevisae的各种菌株。下面描述的协议足以进行 1 转换。
- 准备转型所需的解决方案。
- 准备以下库存溶液和过滤器消毒:10x TE缓冲液,含有100 mM Tris-HCl (pH 7.5),10 mM EDTA,总体积为 50 mL;1 M LiAc,pH 7.5,总体积为 50 mL;50% PEG 4000,总容量为 100 mL。
注:始终检查 PEG 4000 库存是否为 pH 5。此库存的存储时间不应超过一个月。 - 使用库存准备以下解决方案:准备包含 0.1 M LiAc、10 mM Tris-HCl、1 mM EDTA、总体积 0.5 mL 的 LiAc-TE 溶液。制备 PEG-LiAc-TE 溶液,包含 40% PEG 4000、0.1 M LiAc、10 mM Tris-HCl、1 mM EDTA,总容积为 1 mL。
注:PEG-LiAc-TE 和 LiAc-TE 解决方案是新准备的,对于成功转型至关重要。
- 准备以下库存溶液和过滤器消毒:10x TE缓冲液,含有100 mM Tris-HCl (pH 7.5),10 mM EDTA,总体积为 50 mL;1 M LiAc,pH 7.5,总体积为 50 mL;50% PEG 4000,总容量为 100 mL。
- 第一轮转换(准备应变预表达Cas12a)。
注:在所有转换步骤中,使用 pH 高于 5 的水。建议在转化的所有步骤中使用脱矿水。- 通过生长应变 CEN 准备预培养。PK113-7D 在含有 20 mL YEPD(2% 葡萄糖)介质的 100 mL 摇瓶中,在 30°C 下孵育过夜,在 250 rpm 下摇动。
- 测量预培养的 OD600 (ODpc)。计算预培养量与制备在转化(转化培养)中使用的细胞预表达Cas12a所需的新鲜介质体积之间的稀释系数(df)。在计算中假设转换培养 (OD tc) 的光密度在 5.2.3 (ti ) 中描述的孵化步骤后为 1.0。

其中ti和 * 分别是孵育时间和倍增时间。- 根据稀释因子计算接种转化培养 (Vtc) 所需的预培养 (Vi) 的体积.
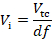
- 根据稀释因子计算接种转化培养 (Vtc) 所需的预培养 (Vi) 的体积.
- 通过接种 20 mL 的 YEPD (2% 葡萄糖) (Vtc) 来制备转化培养,用上一步(Vi) 中确定的预培养量。在 30°C 下孵育,在 250 rpm 下摇动。
- 测量转换区域性的 OD600,直到达到 1.0 的 OD600。
- 在2,500 x g的20mL汤的离心下收集细胞5分钟。丢弃上清液,在室温脱矿水中清洗细胞。重复离心步骤并保持细胞颗粒。
- 在LiAc-TE溶液的100μL中重新悬浮细胞,并转移到微离心管中。
- 加入5μL的单链载体DNA(10mg/mL鲑鱼精子DNA),并通过移液混合。
- 移液器 1 μg 质粒 pCSN067 到微离心管。
注:DNA混合物的总体积不应超过100μL,以防止转化效率降低。 - 加入 600 μL 的 PEG-LiAc-TE 溶液,并通过移液混合。在30°C下孵育30分钟,在台顶热块中以450rpm摇动。
- 加入 70 μL DMSO (100%)到转化混合物,并通过移液混合。在水浴中,在42°C下孵育转化混合物15分钟,从而进行热休克。
- 通过将混合物转移到15 mL圆形底管,并将 10 mL YEPD(2% 葡萄糖)添加到管中,以恢复细胞。在 30°C 下孵育过夜,在 250 rpm 下摇动。
- 在2,500 x g下将转化混合物离心5分钟,丢弃上清液,并在剩余溶液的大约200μL中重新悬浮细胞颗粒。
- 在YEPD(2%葡萄糖)琼脂片上,在YEPD(2%葡萄糖)的转化混合物中,将150μL的转化混合物和20倍稀释,辅以0.2 g/L G418。在30°C下孵育板48~72小时。
- 在YEPD(2%葡萄糖)琼脂板上选择单个转化剂并重新条纹,辅以0.2g/L G418,以获得单菌落。
- 第二轮转换(使用CRISPR/Cas12a执行多路复用基因组编辑)。
- 通过在第一个转化轮(步骤 5.2)中创建的菌株预表达 Cas12a,在含有 20 mL YEPD(2% 葡萄糖)介质的 100 mL 摇摇瓶中,辅以 0.2 g/L G418,从而培养预培养剂。在 30°C 下孵育过夜,摇动 250 rpm。
注: 对于多个转换,调整预区域性的体积。 - 按照步骤 5.2.2 到 5.2.7 进行第一轮转换。
注: 对于多个转换,调整所需解的体积和应变预表达 Cas12a 的培养。 - 移液器 1 μg 的单个 crRNA 阵列,1 μg 的线性受体质粒用于 crRNA 阵列,1 μg 的供体 DNA 和 1 μg 的每个侧翼区域(步骤 4.3)在微离心管中。
注:DNA混合物的总体积不应超过100μL,以防止转化效率降低。 - 为转换准备以下控件:负控制(超纯 H2O);确定转换效率的正控制(1 μg循环pRN1120);控制验证是否通过CRISPR编辑(1 μg的圆形pRN1120,1μg的所有供体DNA表达盒和1μg的侧翼区域,但没有单crRNA阵列);控制验证供体DNA是否可以在靶外集成(1 μg线性化pRN1120,1μg供体DNA表达盒和1μg单crRNA阵列,但没有侧翼区域);一个控制,验证pRN1120(1μg线性化pRN1120)的完全线性化。
- 按照步骤 5.2.9 到 5.2.12 进行第一轮转换。
- 在YEPD(2%葡萄糖)琼脂(2%葡萄糖)的转化混合物中,将150μL的转化混合物和20倍稀释,辅以0.2 g/L G418和0.2 g/L NTC。在 YEPD(2% 葡萄糖)琼脂上进行板出控制,辅以适当的选择(G418 和/或 NTC 或无选择)。在30°C下孵育板48~72小时。
- 在 YEPD(2% 葡萄糖)琼脂板上选择单色转化剂并重新条纹,以获得单色菌落。
- 通过在第一个转化轮(步骤 5.2)中创建的菌株预表达 Cas12a,在含有 20 mL YEPD(2% 葡萄糖)介质的 100 mL 摇摇瓶中,辅以 0.2 g/L G418,从而培养预培养剂。在 30°C 下孵育过夜,摇动 250 rpm。
6. 基因组编辑效率评估
- 计算转化板上的彩色殖民地和白色菌落的数量。
- 计算基因组编辑效率,方法是将彩色菌落数除以菌落总数(白色和彩色),如表1所示。
7. 确认在预定位点内整合供体DNA
- 在没有G418和NTC选择的情况下,从YEPD(2%葡萄糖)琼脂板上的转化板上重新分离一个彩色单菌群,并在30°C下孵育48小时。
- 选择单个菌落,接种充满 100 mL YEPD(2% 葡萄糖)介质的 500 mL 摇瓶。在 30°C 下孵育 48 小时,在 250 rpm 下摇动。
- 如第 4.1 节所述,分离基因组 DNA。
注:或者,使用一种协议来制备酵母的菌群PCR之前由Looke等人提出的。32. 在这种情况下,可以跳过液体介质的增长(第 7.2 节)。 - 通过每个集成表达盒放大两个片段来验证正确集成。
- 设计引源,在转化的侧翼区域和感兴趣的基因之外对基因组DNA进行发火(参见补充表2、KC-121到KC-132中的示例)。使用引体 KC-121 到 KC-132 时,将 PCR 程序中的退火温度设置为 62°C。
- 如第 4.4.2 节所述,放大感兴趣的区域。调整 PCR 程序,根据模板的长度和制造商对 DNA 聚合酶的建议,具体调整 PCR 中延长步骤的时间。
- 使用DNA加载染料和DNA阶梯,在100至10,000 bp范围内使用DNA加载染料和DNA阶梯,通过电泳在胶合糖凝胶(0.8%、40分钟、5 V/cm)上检查PCR产品的大小。
8. 使用声学液体处理程序创建酵母像素艺术
- 为酵母像素图准备图片模板。
- 调整原始 RGB 图片的大小(220 × 280 像素,查看代表性结果),例如,使用 ImageJ 创建最终 64 × 96 像素(宽度 + 高度)灰度图像,以预期颜色(代表性结果)显示。
- 使用以下公式将 RGB 图片转换为灰度:

其中,我,我,我g,我b是灰色,红色,绿色和蓝色的强度,分别。 - 为了对像素进行分类,开发一个 ImageJ 插件,应用以下规则:(a) 如果Igr = 64,则对此像素使用深橙色酵母(应变 1,补充表3)。(b) 如果 64 < Igr = 128,则使用此像素使用橙色酵母(应变 2,补充表3)。(c) 如果 128 < Igr = 192,则使用此像素使用黄色酵母(应变 3,补充表3)。(d) 如果igr > 192,请使用白色酵母 (CEN.此像素的 PK113-7D)。
- 点出酵母细胞以创建酵母像素艺术。
- 接种500 mL摇瓶含有100 mL的YEPD(2%葡萄糖)介质与三种不同颜色的类胡萝卜素产生S.cerevisiae菌株和野生类型CEN。PK113-7D。在 30°C 下孵育培养物过夜,在 250 rpm 下摇动。
- 将0.5 mL的过夜培养物转移到充满0.5 mL无菌非离子密度梯度介质的管中(参见材料表)。通过短暂涡旋混合。
- 将电池悬架转移到合格的储液罐,2 x 3 孔。使用从合格的储液源板到含有 50 mL YEPD (2% 葡萄糖)琼脂的微板(参见材料表)的声学液体处理仪器进行检测。为了简化电镀,请定义板上的孔,例如。使用微孔板作为 6144 孔板 (64 × 96)。
- 使用带有流体校准设置6RES_AQ_GPSA2的 .csv 文件,将 2x 3 井储液源板中每个S. cerevisae应变的 25 nL 点到目标微板上。在这 25 个 nL 液滴中,每个液滴都定义为 64 x 96 网格中的像素,该像素可转换为井位(A01、B01、C01等)。
- 在30°C下孵育微孔板48小时。为了加强菌株的颜色,将琼脂板储存在4°C下至少72小时。
结果
使用CRISRP/Cas12a进行多路基因组编辑的协议通过构建三个类胡萝卜素产生S.cerevisiae菌株,表达crtE、crtYB和crtI基因, 高、中、低强度:应变1、2和3(补充表3)。这些菌株的构造需要产生三个供体DNA表达盒和六个侧翼区域每个菌株,以靶向基因组DNA中的三个不同的位点(如图2 B所示)。如本文所述,启动子、开放读取帧、终结器和两个连续的 50 bp 连接器序列通过金门克隆反应组装成表达盒,并通过 PCR 验证该组件(图3A)。单crRNA阵列被作为合成DNA片段排序,由PCR扩增(图3B)。单crRNA阵列的受体质粒(质粒pRN1120)采用Eco RI-HF和XhoI进行线性化,线性化通过电泳得到确认(图3C)。引入的供体DNA表达盒和侧翼区域的设计和核苷酸序列列于补充表3和补充表4。单crRNA阵列表达盒的序列在补充表1中提供。单crRNA阵列中包含的垫片功能事先通过单组基因组编辑与单个crRNA19进行测试。
首先根据转化后获得的彩色菌落数量对Cas12a的基因组编辑效率进行了评价(表1,图4)。三种构造菌株的编辑效率从50%到94%不等。值得注意的是,用于生成应变1的表达盒的引入显示最低编辑效率,可能是由于供体DNA的性质(即,这些表达盒编码crtE,crtYB和crtI从三个高强度促进器)。其次,PCR(图5)确认了基因组DNA上预期位点的三个供体DNA表达盒的正确整合。引源的设计方式是,当在预定位点正确整合供体DNA时,PCR产品得到。对于每个转化实验,从变换板中选取了8个菌落并进行测试(请注意,图5中只给出了三个殖民地)。一般来说,在按供体DNA测试的8个菌落中,在INT1位点、INT2位点的crtYB和INT3位点的crtI中,在+90%的转化剂中确认crtE供体DNA的正确整合。这些结果证明CRISPR/Cas12a系统与单个crRNA阵列相结合,可同时在多个位点对S.cereviae基因组进行有效的多路复用编辑。
此外,我们演示了使用三种类胡萝卜素产生菌株的"酵母像素艺术"的创作,这些菌株与非彩色野生型菌株一起构造。从罗莎琳德·富兰克林的黑白图片(图6A)开始,创建了一张4色图片(图6B)和斑点列表,然后用一种声液处理器,导致罗莎琳德富兰克林的高分辨率"酵母画"(图6C,D,E)。

图 1:CRISPR/Cas12a多路复用基因组编辑协议的工作流程。 工作流包括所呈现方法的关键步骤。有关详细信息,请参阅《议定书》。请点击此处查看此图的较大版本。

图 2:使用单个crRNA阵列进行CRISPR/Cas12a多路复用基因组编辑方案。(A) 单个 crRNA 阵列由三个成熟形式的 crRNA 单位组成,这是一个 20 bp 直接重复,特定于 LbCas12a(灰色方块),具有 23 bp 引导序列(彩色钻石)。crRNA阵列的表达由SNR52启动子和SUP4终止器启用。具有线性化pRN1120的S.cerevisiae的转化和单crRNA阵列表达盒,含有与pRN1120(对角条纹)的同源性,允许在细胞预表达中重组成圆形质粒LbCas12a.单crRNA阵列随后由Cas12a处理。(B) Cas12a 被定向到预期的INT1、INT2和INT3基因组靶点,并产生双重搁浅断裂。在转化混合物中,包括侧翼区域和类胡萝卜素基因表达盒的供体DNA。由于存在50bp同源连接器序列,在体内重组,供体DNA组被定位在INT1(crtE)、INT2(crtYB)和INT3(crtI)位点周围的基因组DNA中的一段DNA,指示为 5、A、B、C、D 或 E. P1_P3,不同的启动子;T1_T3,不同的终结器。这个数字已被修改从Verwaal等人201819。 使用合成生物学开放语言(SBOL)视觉符号40显示的遗传构造。请点击此处查看此图的较大版本。
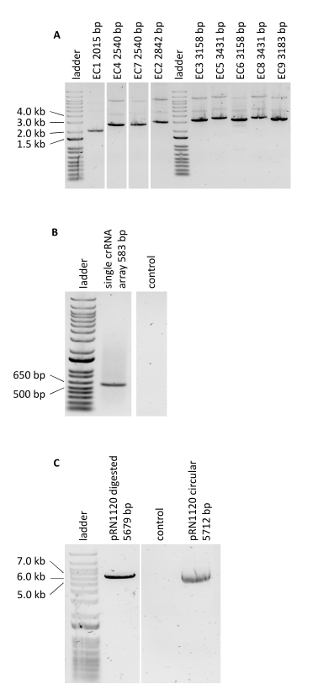
图 3:PCR验证基因组编辑实验。(A) 验证金门克隆反应组装的供体DNA盒。获得的结果与预期长度一致。(B) 单crRNA阵列的PCR.(C) 质粒pRN1120的线性化.请点击此处查看此图的较大版本。

图 4:使用多路复用基因组编辑方法的S.cerevisae变换板。(A) 应变 1 表示crtE、 crtYB和crtI从三个强大的促进器 (深橙色殖民地).(B) 从三个中等强度促进器(橙色菌落)中表达crtE、crtYB和crtI的应变2表示crtE、crtYB和crtI。(C) 应变 3 表示crtE、 crtYB和crtI从三个低强度促进器 (黄色殖民地).请点击此处查看此图的较大版本。

图 5:PCR验证在基因组DNA中预期位点的供体DNA表达盒的整合。(A) 验证菌株1的三个菌落.(B) 验证菌株2的三个菌落。(C) 验证菌株3的三个菌落。请点击此处查看此图的较大版本。

图 6:罗莎琳德·富兰克林的酵母像素艺术。(A) 用作模板的罗莎琳德·富兰克林的 220 × 280 像素的黑白 RGB 照片。(B) 计算机将罗莎琳德·富兰克林的黑白照片转换为 4 色 64 × 96 像素列表。(C) 具有 64 × 96 酵母菌落的酵母像素艺术照片,带有放大部分。(D) 具有两个完整生长板的声学液体处理机的照片。(E) 具有 64 × 96 酵母菌落的完全生长微孔板的照片。请点击此处查看此图的较大版本。
| 应变 1 | 应变 2 | 应变 3 | |
| 彩色殖民地 | 16 | 279 | 220 |
| 白色殖民地 | 16 | 18 | 18 |
| 殖民地总数 | 32 | 297 | 238 |
| 效率 | 50% | 94% | 92% |
表1:多路复用基因组编辑方法的编辑效率。
| crRNA数组序列a,b,c,d,e,f |
|
CATGTATCAGCATATATATATATATATATATATGGGGGCTCAATGGGGGGGGCCCCGGCGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGTCTTGAAAA 加塔加特加特加特加特塔塔塔塔加特特加特塔格塔塔塔塔塔塔塔格格 塔加特加特加特加特加特加特加特加特加特格格格格格格格格格格塔格格格格格格格格格格格GCA TTTTCTACCCATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATGATAAGAGAGAGAAGAAGATGGC ATGTCTGGCTATCTCTCTCTCTCTCTCTCTCTCTGATAATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATAT CTGGGGGAAGAGATATGAAATCTATATATATATATATATATATATAtAtAtAtAtAtGTGGGGGTACTAC GCCGAGCCGACGAATCTATATATATATATATATAtAtTTGCCCCTATATATATT TTTTTTTTATATATCTGGGGGGGGCCCTTTTTTTTTTTTTTTTTTTGGG 格塔加特加特格格格格格加特加特塔加特克特克特克特克特克特格格 |
| a. 同源性到 pRN1120(粗体)。 b. SNR52 启动器(斜体)。 c. 基因组靶序列(带下划线)。 d. 指导针对 LbCas12a 的定向重复(斜体,粗体)。 e. SUP4 终结器(斜体)。 f. 同源性到 pRN1120(粗体)。 |
补充表1:含有质粒pRN1120的同源性同源性LbCas12a的单crRNA阵列。
| 名字 | 序列a | 描述b | 用于点 |
| KC-101 | 卡特加特加加加特卡特克特茨克 | 用于扩增单crRNA阵列的FW引种 | 2.1.4 |
| KC-102 | 卡卡加阿卡阿卡阿卡达格加特加理事会 | 用于扩增单crRNA阵列的RV引热剂 | 2.1.4 |
| KC-103 | AAGCGACTCTATCTCTCTGC | 用于用连接器5扩增供体DNA的FW引体 | 3.6.1 |
| KC-104 | AAAGAAGGAAGAGAGAAC | 用于用连接器 A 扩增供体 DNA 的 RV 引热剂 | 3.6.1 |
| KC-105 | CGGATATATGACACAACCG | 用于用连接器 B 扩增供体 DNA 的 FW 引因 | 3.6.1 |
| KC-106 | 卡塔格格格格加加塔塔茨 | 用于用连接器 C 扩增供体 DNA 的 RV 引基 | 3.6.1 |
| KC-107 | AACGTGTAGTTTTTCC | 用于用连接器 D 扩增供体 DNA 的 FW 引因 | 3.6.1 |
| KC-108 | 阿加塔卡加加格格格 | 用于用连接器 E 扩增供体 DNA 的 RV 引热剂 | 3.6.1 |
| KC-109 | 中航协 | 用于放大 INT1 5' 的 FW 底漆,带连接器 5 | 4.4 |
| KC-110 |
AAACGCCGTGTGTGGTAC TGGATATGCAAGGGGAA GTCGCTTGACTCCTCCTCC ATTCC | 用于放大 INT1 5' 的 RV 底漆,带连接器 5 | 4.4 |
| KC-111 |
特海加茨塔加塔格股份公司 TACTCCTCTCTCTCTCTCTCTCTCTTT TGCTTAAGCGAGTTTCTCTC TTTG | 用于放大 INT1 3' 的 FW 底漆,带连接器 A | 4.4 |
| KC-112 | TGTCAACTGGAGAGCATATCG | 用于放大 INT1 3' 的 RV 底漆,带连接器 A | 4.4 |
| KC-113 | AGAA加特克特克特克特茨 | 用于放大 INT2 5' 的 FW 底漆,带连接器 B | 4.4 |
| KC-114 |
TGCAA加特GTGTGTGTGTTT TGGGTGGTGTGTGTGTGTTCAT CGATCCGCCCTATCAAGGATACC TGGTTG | 用于放大 INT2 5' 的 RV 底漆,带连接器 B | 4.4 |
| KC-115 |
中国反恐委员会 GACCACATATCCCCCCCCC CCTGTTGGGCGATTACACAGCG GTGG | 用于放大 INT2 3' 的 FW 底漆,带连接器 C | 4.4 |
| KC-116 | TCTCTCGATGGGG | 用于放大 INT2 3' 的 RV 底漆,带连接器 C | 4.4 |
| KC-117 | GGTCTTTTTGGAGATATG | 用于放大 INT3 5' 的 FW 底漆,带连接器 D | 4.4 |
| KC-118 |
GCGGATATATATGGGGGGGGGGG 阿卡卡塔卡卡阿加特格 GACAACGTTTTCCAAGGG AAGAACG | 用于放大 INT3 5' 的 RV 底漆,带连接器 D | 4.4 |
| KC-119 |
AAATAACAAAATATATAT CCCATATGCTCGGTGTTT 格加特加特加格格格茨卡加卡茨ACTACT GTAC | 用于放大 INT3 3' 的 FW 底漆,带连接器 E | 4.4 |
| KC-120 | 加加特CTATATATATATATTTTTTTTTTTTTTTTTTT | 用于放大 INT3 3' 的 RV 底漆,带连接器 E | 4.4 |
| KC-121 | GTTACTAACTAACTGG | 用于验证 con5-crtE-conA 集成到 INT1 5' 的 FW 引结 | 7.4.1 |
| KC-122 | CACTGCACACTTTTTC | 用于验证 con5-crtE-conA 集成到 INT1 3' 的 FW 引结 | 7.4.1 |
| KC-123 | CACTGGAACT加特加特加特加特加格 | 用于验证 conB-crtYB-conC 到 INT2 5' 的集成的 FW 引基 | 7.4.1 |
| KC-124 | GTCTCGCTATTGGTCC | 用于验证 conB-crtYB-conC 到 INT2 3' 的集成的 FW 引基 | 7.4.1 |
| KC-125 | CTCTATGAAGGTCAAGTC | 用于验证 conD-crtI-conE 集成到 INT3 5' 的 FW 引基 | 7.4.1 |
| KC-126 | GATCGATATATATAGAG | 用于验证 conD-crtI-conE 集成到 INT3 3' 的 FW 引基 | 7.4.1 |
| KC-127 | CCTGTCCAGGTCC | 用于验证 con5-crtE-conA 集成到 INT1 5' 的 RV 引热器 | 7.4.1 |
| KC-128 | GCTATATATATATATATAAC | 用于验证 con5-crtE-conA 与 INT1 3' 集成的 RV 引热 | 7.4.1 |
| KC-129 | CTGGCAATTGACCAATGC | 用于验证 conB-crtYB-conC 与 INT2 5' 集成的 RV 引基 | 7.4.1 |
| KC-130 | 中加协的塔亚特克特理事会 | 用于验证 conB-crtYB-conC 与 INT2 3' 集成的 RV 引基 | 7.4.1 |
| KC-131 | 中塔委员会,中国 | 用于验证 conD-crtI-conE 集成到 INT3 5' 的 RV 引基 | 7.4.1 |
| KC-132 | CTGGTACTCTCTCTTAAGAGG | 用于验证 conD-crtI-conE 与 INT3 3' 集成的 RV 引基 | 7.4.1 |
| a. 粗体序列表示连接器序列。 b. 正向和反向引引分别指定为 FW 和 RV。 |
补充表2:引性序列。
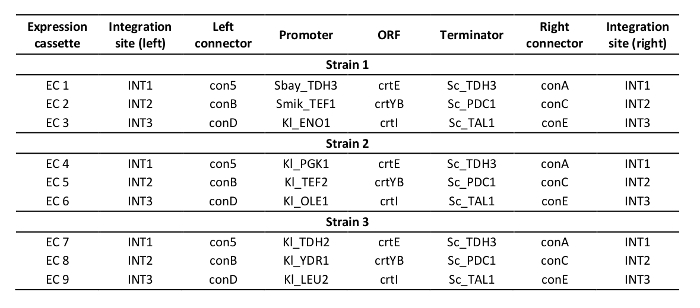
补充表3:结构菌株的设计。

补充表4:供体DNA表达盒和剥落区域的序列。请点击此处下载此文件。
讨论
所提供的协议描述了使用来自Lachnospiracea细菌的Cas12a与单个crRNA阵列和供体DNA相结合的S.cerevisae的多路基因组编辑。详细介绍了单crRNA阵列和供体DNA的设计。与成熟的CRISPR/Cas9系统相比,CRISPR/Cas12a具有独特的额外能力,能够处理从单个crRNA阵列13、33中表达的多个crRNA。由于此功能,同时编辑多个目标更易于设置,并且可以在单个转换中实现。Zetsche等人之前已经演示了这种单一crRNA阵列方法。34人使用AsCas12a和Swiat等人同时编辑了哺乳动物细胞中的多达4个基因。35人使用FnCas12a将4个DNA片段引入酵母基因组。据我们所知,使用 Cas12a 系统同时进行的基因组修饰数量更多,并且 Cas12a 每个阵列的目标最大限值尚未确定。进一步的研究利用单crRNA阵列结合Cas12a包括多路转录调节在广泛的生物33,36,37。
在所呈现的协议中有一些关键步骤。仔细设计Cas12a基因组编辑实验中涉及的所有DNA序列,特别是在引入新型DNA序列的情况下。确定crRNA中新的间隔序列部分的功能,例如通过Verwaal等人描述的单丛基因组编辑实验。19之前将它们组合到单个 crRNA 阵列中。按照 Cas12a 编辑实验中使用的转换缓冲液制备的建议,实现酵母的良好转化效率。
技术有一些可选的修改。建议在转化中使用每个供体DNA的1μg、线性化pRN1120或单crRNA阵列表达盒,尽管使用较低的DNA量也有望产生令人满意的转化效率。执行测试转换以确定是否可以使用较低的 DNA 量。S. cerevisae的转换可能使用与该协议中描述的方法不同的方法执行,例如 Gietz等人描述的协议。(2007)38.导引RNA受体质粒pRN1120适用于不同Cas12a变异的单个crRNA和单crRNA阵列的表达(例如,来自酸米诺卡丘spp)。BV3L6或方济各会诺维西达U112),以及表达sgRNA结合Cas919。供体DNA不需要局限于类胡萝卜素基因表达盒和侧翼区域,目标为基因组DNA中所述的INT1、INT2和INT3位点。任何感兴趣的DNA都可以通过该协议中描述的设计原则以多路复用的方式引入宿主的基因组DNA,或者供体DNA可用于从宿主基因组中删除DNA。单crRNA阵列的模块化结构便于调整间隔和直接重复序列。间隔序列的修改允许改变预定的集成位点,可以通过一个用于识别基因组目标位点的工具来设计,例如GuideScan软件1.0 39。通过将这些 50 bp 齿面区域序列纳入 PCR 中使用的引体中,可以将 50 bp 的齿面区域序列纳入供体 DNA 序列,而不是使用包含连接器序列的大型齿面序列。在这种情况下,一个成功的多路复用基因组编辑实验总共只需要三个,而不是九个供体DNA片段。
总之,该协议提供了分步方向,使用Cas12a与单个crRNA阵列方法相结合,在S.cerevisae中执行多路复用基因组编辑。该协议通过使用9个供体DNA片段和3个gRNA的单crRNA阵列编码进行多路基因组编辑。对于此处报告的三种应变设计,我们显示高达 50% 到 94% 的总体编辑频率。最后,Cas12a 的独特功能是能够将单个 crRNA 阵列处理成细胞中的单个 crRNA,这使得 Cas12a 成为实现多路复用基因组编辑和开发针对多个表达盒在一去。最后,获得三种菌株,在不同水平和黄色和橙色之间的色调下生产类胡萝卜素。有了这些菌株和野生型菌株,我们展示了如何直接使用声学液体处理机制作酵母像素艺术 - 这是为了纪念罗莎琳德·富兰克林,她65年前通过她著名的照片51 23.为发现DNA结构做出了贡献。/c1°.
披露声明
提交人声明存在利益冲突。作者已提交与提交方法相关的知识产权。
致谢
该项目根据第686070号(DD-DeCaf)和764591(SynCrop)赠款协议,从欧洲联盟的Horizon 2020研究和创新方案获得资金,以及项目号为737.016.005(SynCrop)的研究方案"生命组成部分",项目号为737.016.005荷兰科学研究组织(NWO)。T.E.G.得到了英国皇家学会(授予UF160357)和BBSRC/EPSRC合成生物学研究中心(资助BB/L01386X/1)的支持。我们感谢齐迪和杰弗里·范·维克克为酵母发现实验所做出的贡献,他们创造了酵母像素艺术。
材料
| Name | Company | Catalog Number | Comments |
| Chemicals specific for the protocol | |||
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | Electrophoresis |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | Selection of E. coli transformants |
| BsaI-HF (20 U/µl) | New England BioLabs | R353L | Golden Gate Cloning |
| Cell Lysis Solution (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| CutSmart Buffer | New England BioLabs | B7204S | Linearization of pRN1120 |
| Deoxyribonucleic acid sodium salt from salmon testes | Sigma Aldrich | D1626 | Transfromation of S. cerevisiae (carrier DNA) |
| dNTPs | Invitrogen | 10297018 | PCRs |
| EcoRI-HF | New England BioLabs | R3101S | Linearization of pRN1120 |
| Ethanol absolute for analysis | Merck | 100983 | Isolation of genomic DNA from S. cerevisiae |
| Ethylenediamine-tetraacetic acid | Sigma Aldrich | ED | Transformation of S. cerevisiae |
| G418 disulfate salt | Sigma Aldrich | A1720 | Selection of S. cerevisiae transformants |
| Histodenz | Sigma Aldrich | D2158 | Yeast pixel art |
| Isopropanol | Merck | 100993 | Isolation of genomic DNA from S. cerevisiae |
| Lithium acetate dihydrate | Sigma Aldrich | L6883 | Transformation of S. cerevisiae |
| Nancy-520 DNA Gel Stain | Sigma Aldrich | 1494 | Electrophoresis |
| NEB10 competent E. coli cells | New England BioLabs | C3019H | Transformation of E. coli: dx.doi.org/10.17504/protocols.io.nkvdcw6 |
| Nourseothricin | Jena Bioscience | AB102 | Selection of S. cerevisiae transformants |
| Phusion buffer | New England BioLabs | M0530L | PCRs |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530L | PCRs |
| Polyethylene glycol 4000 | Merck | 7490 | Transformation of S. cerevisiae |
| Protein Precipitation Solution (10 M NH4AC) (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| Purple loading dye | New England BioLabs | B7024S | Electrophoresis |
| QIAprep Spin Miniprep Kit | QIAGEN | 27106 | Purification of plasmids |
| RNase coctail enzyme mix | Thermo Fisher Scientific | AM2286 | Isolation of genomic DNA from S. cerevisiae |
| T4 DNA ligase buffer | Invitrogen | 46300-018 | Golden Gate Cloning |
| T4 DNA Ligase (1 U/µl) | Invitrogen | 1705218 | Golden Gate Cloning |
| UltraPure Agarose | Invitrogen | 16500500 | Electrophoresis |
| Wizard SV Gel and PCR Clean-Up System Kit | Promega | A9282 | Purification of PCR products and linearized pRN1120 |
| Xhol | New England BioLabs | R0146S | Linearization of pRN1120 |
| Zymolyase 50 mg/ml (5 units/µL) | Zymo Research | E1006 | Isolation of genomic DNA from S. cerevisiae (yeast lysis enzyme) |
| Zymolyase storage buffer | Zymo Research | E1004-B | Isolation of genomic DNA from S. cerevisiae (necessary for the preparation of yeast lysis enzyme) |
| Chemicals of general use | |||
| 2*Peptone-Yeast extract (PY) agar | Plate growth of E. coli | ||
| 2*PY medium | Cultivation of E. coli | ||
| Demineralized water | Transformation of S. cerevisiae | ||
| ELFO buffer | Electrophoresis | ||
| MQ | Multiple steps | ||
| Physiological salt solution | Transformation of S. cerevisiae | ||
| TE buffer | Storage of DNA, transformation of S. cerevisiae | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | Cultivation of S. cerevisiae | ||
| YEPD (2% glucose) agar | Plate growth of S. cerevisiae | ||
| Consumables | |||
| Eppendorf tubes | |||
| Falcon tubes (50 mL) | |||
| Microplate 96 wells | |||
| Petri dishes | |||
| Pipette tips 0.5 - 10 µL | |||
| Pipette tips 10 - 200 µL | |||
| Pipette tips 100 - 1000 µL | |||
| Shake flasks (500 mL) | |||
| Sterile filters | |||
| Equipment | |||
| Centrifuge (Falcon tubes) | |||
| Echo 525 acoustic liquid handler | |||
| Incubator | |||
| NanoDrop | |||
| Set for eletrophoresis | |||
| Spectrophotometer | |||
| Table centrifuge (Eppendorfs tubes) | |||
| Thermocycler | |||
| Plasmids | |||
| pCSN067 | Addgene | ID 101748 | https://www.addgene.org/ |
| pRN1120 | Addgene | ID 101750 | https://www.addgene.org/ |
| Strains | |||
| S. cerevisiae strain CEN.PK113-7D | EUROSCARF collection | http://www.euroscarf.de |
参考文献
- Knott, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361 (6405), 866-869 (2018).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Lian, J., HamediRad, M., Hu, S., Zhao, H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications. 8 (1), 1688 (2017).
- Li, Z. -. H., Liu, M., Lyu, X. -. M., Wang, F. -. Q., Wei, D. -. Z. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology. 58 (12), 1100-1104 (2018).
- Shao, Y., Lu, N., Qin, Z., Xue, X. CRISPR-Cas9 facilitated multiple-chromosome fusion in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (11), 2706-2708 (2018).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology. 13 (11), 722-736 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), (2016).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovič, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Lian, J., HamediRad, M., Zhao, H. Advancing metabolic engineering of Saccharomyces cerevisiae. using the CRISPR/Cas System. Biotechnology Journal. 13 (9), 1700601 (2018).
- Ferreira, R., et al. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (1), 10-15 (2018).
- Swarts, D. C., Martin, J. Cas9 versus Cas12a/Cpf1: Structure–function comparisons and implications for genome editing. Wiley Interdisciplinary Reviews: RNA. 9 (5), 1481 (2018).
- Strohkendl, I., Saifuddin, F. A., Rybarski, J. R., Finkelstein, I. J., Russell, R. Kinetic Basis for DNA Target Specificity of CRISPR-Cas12a. Molecular Cell. 71 (5), 816-824 (2018).
- Verwaal, R., et al. High-level production of beta-carotene in Saccharomyces cerevisiae. by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous. Applied and Environmental Microbiology. 73 (13), 4342-4350 (2007).
- Verwaal, R., Buiting-Wiessenhaan, N., Dalhuijsen, S., Roubos, J. A. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast. 35 (2), 201-211 (2018).
- Jakociunas, T., Jensen, M. K., Keasling, J. D. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering. 34, 44-59 (2016).
- Engler, C., Romy, K., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PloS One. 3 (11), 3647 (2008).
- Franklin, R. E., Gosling, R. G. Molecular configuration in sodium thymonucleate. Nature. 171, 740-741 (1953).
- Watson, J. D., Crick, F. H. A structure for deoxyribose nucleic acid. Nature. 171, 737-738 (1953).
- Wilkins, M. H. F., Stokes, A. R., Wilson, H. R. Molecular structure of deoxypentose nucleic acids. Nature. 171, 738-740 (1953).
- Young, E. M., et al. Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast. Metabolic Engineering. 48, 33-43 (2018).
- Roubos, J. A., Pel, H. J., Meijrink, B. . Cloning Method. , (2013).
- Mandel, M., Higa, A. Calcium-dependent bacteriophage DNA infection. Journal of Molecular Biology. 53 (1), 159-162 (1970).
- Van Dijken, J. P., et al. An interlaboratory comparison of physiological and genetic properties of four Saccharomyces cerevisiae strains. Enzyme and Microbial Technology. 26 (9-10), 706-714 (2000).
- Gietz, R. D., Schiestl, R. H., Willems, A. R., Woods, R. A. Studies on the transformation of intact yeast cells by the LiAc/SS-DNA/PEG procedure. Yeast. 11 (4), 355-360 (1995).
- Hill, J., Donald, K. A., Griffiths, D. E., Donald, G. DMSO-enhanced whole cell yeast transformation. Acids Research. 19 (20), 5791 (1991).
- Looke, M., Kristjuhan, K., Kristjuhan, A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques. 50 (5), 325-328 (2011).
- Tak, Y. E., et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nature Methods. 14 (12), 1163-1166 (2017).
- Zetsche, B., et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature biotechnology. 35 (1), 31-34 (2017).
- Swiat, M. A., et al. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research. 45 (21), 12585-12598 (2017).
- Li, L., et al. CRISPR-Cpf1-Assisted Multiplex Genome Editing and Transcriptional Repression in Streptomyces. Applied Environmental Microbiology. 84 (18), 00827-00918 (2018).
- Zhang, X., et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery. 3, 17018 (2017).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 1-4 (2007).
- Perez, A. R., et al. GuideScan software for improved single and paired CRISPR guide RNA design. Nature Biotechnology. 35 (4), 347-349 (2017).
- Cox, R. S., et al. Synthetic Biology Open Language Visual (SBOL Visual) Version 2.0. Journal of Integrative Bioinformatics. 15 (1), 1613-4516 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。