Method Article
Edição do genoma de CRISPR/Cas12a multiplex de Saccharomyces cerevisiae e a criação da arte do pixel do fermento
Neste Artigo
Resumo
O sistema CRISPR/Cas12a em combinação com uma única matriz crRNA permite a edição multiplex eficiente do genoma S. cerevisiae em vários locos simultaneamente. Isto é demonstrado pela construção de carotenóides produzindo cepas de levedura que são subsequentemente utilizados para criar a arte do pixel de levedura.
Resumo
Alta eficiência, facilidade de uso e versatilidade das repetições palindrômicas curtas interespaçadas agrupadas/proteína 9 (CRISPR/Cas9) associada ao sistema CRISPR-Associated tem facilitado a modificação genética avançada de Saccharomyces cerevisiae, um modelo organismo e laborioso em biotecnologia industrial. Proteína 12A associada a CRISPR (Cas12a), uma endonuclease guiada por RNA com características distinguíveis de Cas9 é aplicada neste trabalho, estendendo ainda mais a caixa de ferramentas molecular para fins de edição de genoma. Um benefício do sistema CRISPR/Cas12a é que ele pode ser usado na edição do genoma multiplex com várias RNAs de guia expressas a partir de uma única unidade transcricional (única matriz de RNA CRISPR (crRNA)). Nós apresentamos um protocolo para a integração multiplex de genes heterólogo múltiplos no locus independente do genoma de S. cerevisiae usando o sistema de crispr/Cas12a com os crrnas múltiplos expressados de uma única construção da disposição do crrna. O método proposto explora a capacidade de S. cerevisiae para realizar a recombinação in vivo de fragmentos de DNA para montar a matriz crrna única em um plasmídeo que pode ser usado para a seleção transformante, bem como a montagem de DNA doador sequências que se integram no genoma em posições pretendidas. Cas12a é pré-expresso constitutivamente, facilitando a clivagem do genoma de S. cerevisiae nas posições pretendidas após a expressão do único array crrna. O protocolo inclui o projeto e a construção de uma única matriz de crRNA e de gavetas fornecedoras da expressão do ADN, e explora uma aproximação da integração que faz o uso de seqüências originais dos conectores do ADN de 50-BP e de seqüências separadas do ADN do flanco da integração, que simplifica projeto experimental através de padronização e modularização e amplia a gama de aplicações. Finalmente, Nós demonstramos uma técnica direta para criar a arte do pixel do fermento com um alimentador líquido acústico usando o carotenóide diferentemente colorido produzindo as tensões do fermento que foram construídas.
Introdução
As enzimas CRISPR/CAS revolucionaram inquestionavelmente a biologia molecular e foram amplamente adotadas como ferramentas para genomas de engenharia a uma velocidade que antes era inviável1. A primeira modificação de um genoma de Saccharomyces cerevisiae pelo sistema de edição de GENOMA crispr/Cas9 foi relatada por DiCarlo et al. 2, demonstrando sucesso gene knock-out e fazendo mutações ponto usando externamente introduzido oligonucleotides. Promova os desenvolvimentos da caixa de ferramentas de crispr do fermento incluíram: Regulamento do transcricional pela fusão de Cas9 inoperantes catalisador inoperante (dCas9) com os domínios do efetoras do transcricional para permitir a ativação e o silenciamento da transcrição3, aplicação para ambos edição do genoma e funções regulatórias para a engenharia da via metabólica por ativação simultânea, repressão e deleção4, deleção de grandes fragmentos do genoma S. cerevisiae 5e fusões de cromossomo múltiplos6 .
Os sistemas de edição de genoma CRISPR/CAS encontram sua origem em sistemas imunes adaptativos de bactérias e Archaea e esses sistemas foram adaptados por biólogos moleculares para edição de genoma. A sua funcionalidade baseia-se nas regiões de DNA das repetições Palindromicas curtas agrupadas regularmente Interespaçadas (CRISPR) que codificam o RNA responsável pelo reconhecimento do ADN ou RNA estrangeiro e dos genes associados CRISPR (CAS) que codifica a endonucleases1,7,8,9. Com base na recente análise do genoma dos sistemas CRISPR/CAS, foi proposto dividir os sistemas CRISPR/CAS em duas classes, cinco tipos e 16 subtipos10. As duas classes são distinguidas com base na organização de complexos efetores envolvidos na clivagem alvo. Tipicamente, os sistemas CRISPR/CAS com uma organização de várias subunidades são categorizados como classe 1, enquanto os complexos efetores de subunidades individuais pertencem à classe 210,11. Neste artigo, exploramos a classe 2 tipo V Cas12a, anteriormente denominada Cpf1,10,12, que é uma alternativa à classe 2 tipo II Cas9. Embora Cas9 seja bem caracterizado e amplamente utilizado na pesquisa, o Cas12a oferece recursos adicionais12. Em primeiro lugar, Cas12a forma um complexo com RNA CRISPR (crRNA) de 42 a 44 nucleotídeos sem a necessidade de um RNA CRISPR trans-ativador adicional (tracrRNA). Portanto, um RNA guia mais curto pode ser utilizado na edição do genoma com sistemas CRISPR/Cas12a em comparação com CRISPR/Cas9. Em segundo lugar, a atividade única de endonuclease e endorribonuclease de Cas12a possibilita a maturação de seu pré-crRNA13. Esta atividade de RNase permite a codificação de crRNAs múltiplo em uma única disposição do crRNA de CRISPR, visto que Cas9 exige a expressão separada de cada RNA so-called do único-guia (sgRNA) ou alternativamente para a expressão do exemplo de um endonuclease adicional ( por exemplo, Csy4) em combinação com motivos de reconhecimento para Csy4 em torno de cada sgrna14,15. Em terceiro lugar, Cas12a reconhecimento do local de destino requer um motivo adjacente de protoespaçador (PAM) na extremidade 5 ' do alvo e Cleaves após a posição + 18/+ 23 de seu PAM resultando em DNA clivada com extremidades pegajosas, enquanto que Cas9 requer um PAM localizado na extremidade 3 ' do alvo e Cleaves após a posição-3 que cria cortes sem corte da extremidade no ADN12. Em quarto lugar, a seqüência de nucleotídeo consensual do PAM difere entre Cas12a ((T) TTV) e Cas9 (NGG), o que torna Cas12a um candidato promissor para o direcionamento de T-Rich promotor e terminador seqüências16. Finalmente, um estudo recente relatou maior especificidade do alvo para Cas12a do que para o nativo Cas917.
Nós apresentamos um protocolo para usar o sistema de CRISPR/Cas12a para a edição do genoma de S. cerevisiae com um foco particular na introdução de gavetas múltiplas da expressão do ADN em Locus genomic independentes simultaneamente (edição multiplex do genoma) usando uma única matriz crRNA. As etapas-chave do protocolo são representadas na Figura 1. Como prova de conceito, o sistema CRISPR/Cas12a foi aplicado para introdução de três fitas de expressão no genoma de S. cerevisiae que possibilitam a produção de β-caroteno18 como esquematicamente mostrado na Figura 2. A produção de β-caroteno afeta o fenótipo de s. cerevisiae: ou seja, após a introdução bem-sucedida de todos os três genes heterólogos necessários para a biossíntese de carotenóides, as células brancas de s. cerevisiae se transformam em amarelo ou laranja, dependendo da força de expressão do promotor de cada gene. Devido à simples leitura visual desta via, foi introduzido para desenvolver sistemas avançados baseados em crispr e métodos para edição de genoma19,20. Neste trabalho, as gavetas da expressão que codificam os genes carotenóides CRTe, crtyb e crti foram construídas usando uma aproximação de Golden Gate clonagem (GGC)21 com promotores heterólogo e terminadores homólogos usado para conduzir a expressão dos genes. As fitas de expressão são rodeadas por sequências de pares 50-base (BP) exclusivas, chamadas de conectores, que permitem a montagem in vivo com sequências de DNA de flanco de integração (regiões flanqueadas) com as mesmas sequências 50-BP, e subsequente integração no ADN genomic do fermento na posição determinada pelas regiões flanqueando. Usando diferentes pontos fortes do promotor, foram obtidas cepas com diferentes níveis de produção de carotenóides, resultando em variação na cor das células. Estas estirpes-inspirado pelo "fermento Art Project"22 -foram usados em uma configuração spotting com um manipulador de líquido acústico para criar um 4-cor de alta resolução "levedura fotografia" de Rosalind Franklin. Franklin (1920-1958) foi um químico inglês e cristalografo de raios-X bem conhecido por sua contribuição para a descoberta da estrutura do DNA por foto 5123,24,25.
Protocolo
1. preparação dos plasmís Cas12a
Nota: o plasmídeo contendo o Codon Lachnospiraceae bactéria ND2006 Cas12a (LbCpf1, pCSN067) otimizado para expressão em S. cerevisiae, foi previamente construído19, depositado em um repositório de plasmídeo (ver a tabela de Materiais). Esta é uma única cópia epissomal s. cerevisiae/E. coli Shuttle plasmídeo contendo um gene de marcador de resistência kanmx para permitir a seleção de transformantes de s. cerevisiae na geneticina (G418).
- Obter o plasmídeo pCSN067 (veja a tabela de materiais).
- Amplificar o plasmídeo pCSN067 para obter uma quantidade elevada.
- Transforme 25 μL de células de E. coli quimicamente competentes adquiridas com o plasmídeo pCSN067 de acordo com o protocolo do fabricante. Diluir a mistura de transformação 10 e 50 vezes em 2x peptona-levedura (PY). Placa para fora diluições 10x e 50x em placas de agar 2x PY contendo ampicilina (0,1 g/L) e incubar durante a noite a 37 ° c.
- Escolha 2 a 3 colônias e inocular cada colônia em 3 ml de 2x py e cresça durante a noite em 37 ° c em uma incubadora de agitação em 180 rpm.
- Purify o plasmídeo usando um kit de purificação de plasmídeo de acordo com as instruções do fabricante.
2. preparação da única gaveta da expressão da matriz de crRNA
- Prepare a única matriz crRNA.
Nota: o único array crRNA compreende um promotor SNR52 RNA POLIMERASE III de S. cerevisiae2, uma repetição direta específica para LbCas12a e um espaçador (sequência alvo genômica), repetida em conjunto para cada alvo19 e termina com um terminador SUP4 de S. cerevisiae2. A única matriz de crRNA é montada pela recombinação in vivo no plasmídeo linearizado pRN1120 para gerar um plasmídeo circular, assim regiões homíparas para plasmídeo pRN1120 devem estar presentes no início e no fim da matriz crrna única (ver Figura 2a ). Recomenda-se com antecedência avaliar a funcionalidade de um número de crRNAs projetado separadamente19. Esta informação é usada subseqüentemente para selecionar a maioria de crRNAs funcionais para combiná-los nas seqüências diretas da repetição e do espaçador para criar uma única disposição do crRNA para a finalidade de multiplexação.- Encomende a matriz crRNA única para experimentos de edição de genoma multiplex como DNA sintético (veja a sequência de DNA do array crRNA único na tabela complementar 1).
- Amplifique a matriz de crRNA única ordenada (por exemplo, usando os primers kc-101 e kc-102 (tabela suplementar 2)). Prepare a mistura de amplificação de PCR contendo: 0,5 μL de DNA polimerase, 10 μL de tampão 5x necessários para a polimerase de ADN, 1 μL de dNTPs de 10 mM, 2,5 μL de primer para a frente de 10 μM, 2,5 μL de primer reverso de 10 μM, 2 μL de modelo de ADN a uma concentração de 5 ng/μL e ultrapura H 2. º O até um volume total de 50 μL.
- Realize a reação em um termocicer usando o seguinte programa: (i) 98 ° c por 3 min, (II) 98 ° c por 10 s, (III) 60 ° c por 20 s, (IV) 72 ° c por 15 s – repita os passos (II) a (IV) 30 vezes, (v) 72 ° c por 5 min (vi) em espera a 12 ° c até análise posterior.
- Analise os produtos do PCR pela electroforese executando as amostras em um gel do agarose de 0,8% em 5 V/cm por 40 minutos usando uma tintura do carregamento do ADN e uma escada do ADN com fragmentos do ADN em uma escala de 100 a 10.000 BP.
- Purify os produtos do PCR usando um jogo da purificação do PCR de acordo com as instruções do fabricante.
- Prepare o único plasmídeo de receptor de matriz crRNA.
Nota: a única matriz crRNA é expressa a partir do plasmídeo de transporte S. cerevisiae/E. coli pRN112019 (veja a tabela de materiais). Este plasmídeo de múltiplas cópias contém um gene de marcador de resistência NatMX para permitir a seleção de transformantes de S. cerevisiae em nourseothricin (NTC).- Obter o plasmídeo pRN1120.
- Amplificar o plasmídeo pRN1120 para obter uma quantidade elevada.
- Transforme 25 μL de células de E. coli quimicamente competentes adquiridas com plasmídeo pRN1120 de acordo com o protocolo do fabricante. Diluir a mistura de transformação 10 e 50 vezes em 2x PY. Placa para fora diluições 10x e 50x em placas de agar 2x PY contendo ampicilina (0,1 g/L) e incubar durante a noite a 37 ° c.
- Escolha 2 a 3 colônias e inocular cada colônia em 3 ml de 2x py e cresça durante a noite em 37 ° c em uma incubadora de agitação em 180 rpm.
- Purify o plasmídeo usando um kit de purificação de plasmídeo de acordo com as instruções do fabricante.
- Linearize o plasmídeo pRN1120 com ecori-HF e xhoI. Para isso, prepare uma mistura de digestão composta por 1 μg de pRN1120, 5 μL de tampão 10x (tampão 1x contém 50 mM de acetato de potássio, 20 mM de Tris-Acetato, 10 mM de acetato de magnésio, 100 μg/mL de albumina sérica bovina [BSA]; pH 7,9), 1 μL de ecori-HF (20 U) , 1 μL de xhoI (20 U) e ultrapura H2o até um volume total de 50 μL. Incubar a mistura de digestão a 37 ° c por 2 H e inativar a 65 ° c durante 20 min.
- Analise o plasmídeo linearizado por eletroforese em gel de agarose (0,8%, 40 min, 5 V/cm) usando um corante de carregamento de DNA e uma escada de DNA com fragmentos de DNA em uma faixa de 100 a 10.000 BP. Como um controle incluem um plasmídeo circular na análise.
- Purify o plasmídeo linearizada usando um jogo da purificação do PCR de acordo com as instruções do fabricante.
3. preparação de construtores de DNA do doador promotor-ORF-Terminator (POT)
- Encomende um conjunto de promotor (P) de diferentes forças, abrir quadro de leitura (O) e terminador (T) seqüências como DNA sintético de tal forma que cada elemento contém padronizado 4-BP seqüências de reconhecimento que são flanqueados por BSAI sites para permitir Golden Gate clonagem ( GGC) montagem26 (Ver os desenhos detalhados na tabela complementar 3 e sequências na tabela complementar 4).
- Monte as gavetas da expressão do potenciômetro compostas de um promotor, de um frame de leitura aberto, de umas seqüências do terminador e dos conectores através de um conjunto de 4 partes usando uma reação de GGC21, em um vetor do destino que já contenha as seqüências pré-especificadas dos conectores 50-BP ( Ver tabela complementar 4 e referências26,27).
- Meça a concentração de peças de DNA usando um espectrofotômetro. Diluir cada parte do DNA em H2o ultrapura a uma concentração final de 15 fmol/μl.
- Prepare uma mistura de reacção composta por fragmentos de ADN: 2 μL de promotor, 2 μL de moldura de leitura aberta, 2 μL de terminador e 2 μL de espinha dorsal (vectores de destino de nível 1, conforme descrito em 26), 4 μL de tampão de LIGASE de ADN de 5x t4, 2,5 μL de 1 U/μl T4 de ADN ligase , 1,5 μL de 20 U/μL BSAI-HF e ultrapura H2o até um volume total de 20 μl.
- Realize a reação de GGC em um termociclador usando o seguinte programa: (i) 37 ° c para 2 minutos, (II) 16 ° c por 5 minutos-repita etapas (i) e (II) 50 vezes, (III) 50 ° c para 60 minutos, (IV) 80 ° c para 45 minutos, (v) preensão em 12 °
- Transforme 25 μL de células de E. coli28 quimicamente adquiridas com 3 μL de mistura de reação de GGC de acordo com o protocolo do fabricante. Diluir a mistura de transformação 10 e 50 vezes em 2x PY. Placa para fora diluições 10x e 50x em placas de agar 2x PY contendo ampicilina (0,1 g/L) e incubar durante a noite a 37 ° c.
- Escolha 2 a 3 colônias e inocular cada colônia em 3 ml de 2x py e cresça durante a noite em 37 ° c em uma incubadora de agitação em 180 rpm.
- Purify os plasmídeos usando um kit de purificação de plasmídeo de acordo com as instruções do fabricante.
- Verifique se as gavetas da expressão POT foram montadas corretamente na reação de GGC por PCR.
- Projete iniciadores complementares à seqüência do conector presente no início e no final de cada gaveta de expressão (vide Figura 2b). Para os conectores escolhidos neste protocolo, use os primers KC-103 para KC-108 (ver tabela complementar 2).
- Prepare misturas de amplificação de PCR para cada plasmídeo contendo: 0,5 μL de ADN polimerase de revisão, 10 μL de tampão 5x necessários para a polimerase de ADN, 1 μL de dNTPs de 10 mM, 2,5 μL de primer para a frente de 10 μM, 2,5 μL de primer reverso de 10 μM, 2 μL de modelo de ADN com um concentrador íon de 5 ng/μL, e ultrapura H2o até um volume total de 50 μL.
- Realize a reação do PCR em um termociclador usando o seguinte programa: (i) 98 ° c 3 minuto, (II) 98 ° c para 10 s, (III) 60 ° c para 20 s, (IV) 72 ° c para 2 minutos 30 s – repita etapas (II) a (IV) 30 vezes, (v) 72 ° c por 5 minutos , (vi) Segure a 12 ° c até uma análise mais aprofundada.
Nota: os produtos resultantes do PCR consistem em 50-BP do conector 5 ', promotor, frame de leitura aberto, terminador e 50-BP do conector 3 '.
- Analise os produtos do PCR pela electroforese executando amostras em um gel do agarose de 0,8% em 5 V/cm por 40 minutos usando uma tintura do carregamento do ADN e uma escada do ADN com fragmentos do ADN em uma escala de 100 a 10.000 BP.
4. preparação de sequências de DNA de flanco de integração contendo sequências de conectores
- Purify o ADN genomic do tipo selvagem S. cerevisiae CEN. PK113-7D29.
- Aumente a tensão em um balão de agitação de 500 mL preenchido com 100 mL de extrato de levedura peptona dextrose (YEPD, 2% glicose) médio a 30 ° c e agitando a 250 rpm por 48 horas.
- Colher as células por centrifugação de 2 mL de caldo a 16.000 x g por 1 min e descartar o sobrenadante.
- Ressuscita as células em sal fisiológico (200 μL; 0,85% solução de NaCl) com RNase (10 μL, 10 mg/mL) e enzima lítica de levedura (4 μL). Incubar a suspensão celular a 37 ° c por 15 min.
- Adicionar 300 μL de solução de lise celular (ver tabela de materiais) e Vortex em breve.
- Adicionar 168 μL de solução de precipitação proteica (ver tabela de materiais) e vórtice vigorosamente durante 20 s.
- Separe a fração protéica por centrifugação a 16.000 x g e 4 ° c por 10 min. colete 600 μl de sobrenadante em um novo tubo e misture com 600 μl de isopropanol e Vortex em breve.
- Recupere o DNA girando para baixo em 16.000 x g na temperatura ambiente por 10 min. descarte o sobrenadante e mantenha o pellet.
- Lave o pellet com 200 μL de etanol (70%). Centrifugue a 16.000 x g à temperatura ambiente durante 10 min e retire o sobrenadante. Evate o etanol incubando o tubo à temperatura ambiente durante 10 min com a tampa aberta.
Nota: se o líquido no tubo ainda estiver visível, repita o passo 4.1.8. Não seque o pellet por mais de 10 min para evitar a diminuição da solubilidade do DNA. - Dissolver o DNA em 50 μL de tampão TE. Armazenar DNA purificado a 4 ° c.
- Para cada local de integração, a integração do projeto flanqueiam seqüências do ADN (aproximado. 500 BP) tais que aproximadamente 1000 BP do ADN genomic serão removidos em cima da introdução do ADN fornecedor (Veja o projeto esquemático na Figura 2b e as seqüências em suplementar Tabela 4).
- Projete primers para gerar as regiões flanqueando pelo PCR.
- Para a região flanqueando esquerda, projete para a frente e os primers reversos para amplificar aproximadamente 500 BP da região genomic do ADN posicionada 5 ' (esquerdo) do local da integração do interesse.
Nota: o primer dianteiro inclui 20 BP de homologia com a região de flanquear pretendida. A cartilha reversa inclui 20 BP com homologia com a região de flanquear pretendida e contém a sequência de conectores 50-BP desejada para permitir a montagem in vivo na edição Cas12a no genoma mais tarde. - Para a região de flanquear direita, projete primers para diante e reverso para amplificar aproximadamente 500 BP da região genomic do ADN posicionada 3 ' (direita) do local da integração do interesse.
Nota: a cartilha de avanço inclui 20 BP com homologia com a região de flanquear pretendida e contém a sequência de conectores 50-BP desejada para permitir a montagem in vivo na edição Cas12a no genoma mais tarde. A cartilha reversa inclui 20 BP de homologia com a região de flanquear pretendida.
- Para a região flanqueando esquerda, projete para a frente e os primers reversos para amplificar aproximadamente 500 BP da região genomic do ADN posicionada 5 ' (esquerdo) do local da integração do interesse.
- Amplificar as regiões flanqueadas com os primers projetados (por exemplo, os primers kc-109 para kc-120 incluídos na tabela complementar 2).
- Meça a concentração do ADN genomic purified que servirá como o molde no PCR. Ajuste a concentração de DNA para 50 ng/μL.
- Prepare misturas da amplificação do PCR compostas do ADN genomic (1 – 4 μL da diluição genomic do ADN de 50 ng/μL) purified na etapa 4,1, para diante e a primeira demão reversa (10 μM cada), 1 μL de dNTPs de 10 milímetros, 10 μL do amortecedor 5x exigido para o polymerase do ADN, 0,5 μL do polymerase do ADN (1,0 U) e ultrapura H2o até volume total de 50 μL.
- Executar PCRs em um termociclador usando o seguinte programa: (i) 98 ° c para 3 min, (II) 98 ° c para 20 s, (III) 60 ° c para 20 s, (IV) 72 ° c para 15 s, repita as etapas (II) a (IV) 30 vezes, (v) 72 ° c por 5 min , (vi) Segure a 12 ° c até uma análise mais aprofundada.
- Analise os produtos do PCR pela electroforese em um gel do agarose de 0,8% em 5 V/cm por 40 minutos usando uma tintura do carregamento do ADN e uma escada do ADN com fragmentos do ADN em uma escala de 100 a 10.000 BP.
- Purify os produtos corretos do PCR usando um jogo da purificação do PCR concordando as instruções do fabricante.
5. transformação para S. cerevisiae
Nota: Realize a transformação utilizando um protocolo baseado nos métodos desenvolvidos por Gietz et al. (1995) 30 e Hill et al. 31 que pode ser usado para várias cepas de S. cerevisiae. O protocolo descrito abaixo é suficiente para 1 transformação.
- Prepare soluções necessárias para a transformação.
- Prepare as seguintes soluções de ações e esterilize-filtro: 10x TE tampão contendo 100 mM Tris-HCl (pH 7,5), 10 mM EDTA, volume total de 50 mL; 1 M LiAc a pH 7,5, volume total de 50 mL; 50% PEG 4000, volume total de 100 mL.
Nota: Verifique sempre se o estoque PEG 4000 está em pH 5. Este estoque não deve ser armazenado por mais de um mês. - Prepare as seguintes soluções usando estoques: Prepare solução LiAc-TE contendo 0,1 M LiAc, 10 mM Tris-HCl, 1 mM EDTA, volume total de 0,5 mL. Prepare a solução PEG-LiAc-TE contendo 40% de PEG 4000, 0,1 M LiAc, 10 mM de Tris-HCl, 1 mM de EDTA, volume total de 1 mL.
Nota: é crucial para a transformação bem-sucedida que as soluções PEG-LiAc-TE e LiAc-TE estão preparadas na hora.
- Prepare as seguintes soluções de ações e esterilize-filtro: 10x TE tampão contendo 100 mM Tris-HCl (pH 7,5), 10 mM EDTA, volume total de 50 mL; 1 M LiAc a pH 7,5, volume total de 50 mL; 50% PEG 4000, volume total de 100 mL.
- Primeira rodada de transformação (Prepare a estirpe pré-expressando Cas12a).
Nota: em todas as etapas de transformação, use água com um pH superior a 5. Recomenda-se a utilização de água desmineralizada em todas as etapas da transformação.- Prepare uma pré-cultura por crescente cepa CEN. PK113-7D em um balão da agitação de 100 mL que contem 20 mL do meio de YEPD (glicose 2%) e incubar durante a noite em 30 ° c com agitação em 250 rpm.
- Meça o OD600 do pre-Culture (PCdo OD). Calcule o fator de diluição (DF) entre o volume de pré-cultura e o volume de meio fresco necessário para a preparação das células pré-expressando Cas12a a serem utilizadas na transformação (cultura de transformação). Nos cálculos supor a densidade ótica da cultura da transformação (TCdo OD) para ser 1,0 após a etapa da incubação descrita em 5.2.3 (ti).

onde ti e τ são o tempo de incubação e o tempo de duplicação, respectivamente.- Calcule o volume da pré-cultura (vi) necessária para a inoculação da cultura de transformação (vTC) com base no fator de diluição.
- Calcule o volume da pré-cultura (vi) necessária para a inoculação da cultura de transformação (vTC) com base no fator de diluição.
- Preparar a cultura de transformação por inoculação de 20 mL de YEPD (2% glicose) (vTC) com o volume de pré-cultura determinado na etapa anterior (vi). Incubar a 30 ° c com agitação a 250 rpm.
- Meça o OD600 da cultura da transformação até que um OD600 de 1,0 esteja alcangado.
- Colha as células por centrifugação do caldo de 20 mL a 2.500 x g durante 5 min. descarte o sobrenadante e lave as células em 20 ml de água desmineralizada a temperatura ambiente. Repita a etapa de centrifugação e mantenha o pellet celular.
- Ressuscitar as células em 100 μL de solução LiAc-TE e transferir para um tubo de microcentrífuga.
- Adicione 5 μL de DNA portador de uma única fita (10 mg/mL de DNA de esperma de salmão) e misture com pipetagem.
- Pipete 1 μg de plasmídeo pCSN067 para o tubo de microcentrífuga.
Nota: o volume total da mistura de DNA não deve exceder 100 μL para evitar uma menor eficiência de transformação. - Adicionar 600 μL de solução de PEG-LiAc-TE e misturar com pipetagem. Incubar por 30 min a 30 ° c, enquanto agitando a 450 rpm em um bloco de calor de mesa.
- Adicionar 70 μL de DMSO (100%) para a mistura de transformação e misture por pipetagem. Realize choque térmico incubando a mistura da transformação em 42 ° c por 15 minutos em um banho de água.
- Recupere as células transferindo a mistura para um tubo inferior redondo de 15 mL e adicione 10 mL de YEPD (2% de glicose) ao tubo. Incubar durante a noite a 30 ° c com agitação em 250 rpm.
- Centrifugue a mistura da transformação em 2.500 x g por 5 min. descarte o sobrenadante e resuma a pelota da pilha em aproximadamente 200 μl da solução restante.
- Placa para fora 150 μL da mistura da transformação e uma diluição 20x no YEPD (glicose de 2%) da mistura da transformação em placas do agar de YEPD (glicose 2%) suplementadas com 0,2 g/L G418. Incubar as placas a 30 ° c por 48 – 72 horas.
- Escolha um único transformante e re-Streak em uma placa de agar YEPD (2% glicose) suplementada com 0,2 g/L G418 para obter colônias únicas.
- Segunda rodada de transformação (realize a edição do genoma multiplex com CRISPR/Cas12a).
- Prepare uma pré-cultura, crescendo a cepa pré-expressando Cas12a, criada na primeira rodada de transformação (etapa 5,2), em um balão de agitação de 100 mL contendo 20 mL de meio de YEPD (2% de glicose) suplementado com 0,2 g/L G418. Incubar durante a noite a 30 ° c com agitação 250 rpm.
Nota: para várias transformações, adapte o volume da pré-cultura. - Siga as etapas 5.2.2 a 5.2.7 para a primeira rodada de transformação.
Nota: para várias transformações, adaptar os volumes de soluções necessárias e cultura da estirpe pre-expressando Cas12a. - Pipetar 1 μg da matriz crRNA única, 1 μg do plasmídeo receptor linearizado para a matriz crRNA, 1 μg de DNA do doador e 1 μg de cada região flanqueadora (passo 4,3) no tubo de microcentrífuga.
Nota: o volume total da mistura de DNA não deve exceder 100 μL para evitar uma menor eficiência de transformação. - Prepare os seguintes controles para a transformação: controle negativo (ultrapura H2O); controle positivo para determinação da eficiência de transformação (1 μg de pRN1120 circular); um controle verificando se a introdução do DNA do doador é conduzida por meio da edição CRISPR (1 μg de pRN1120 circular, 1 μg de todas as gavetas de expressão de DNA do doador e 1 μg de regiões flanqueantes, mas nenhuma matriz crRNA única); controle verificando se o DNA do doador pode ser integrado fora do alvo (1 μg de pRN1120 linearizados, 1 μg de gavetas de expressão de DNA do doador e 1 μg do array crRNA único, mas sem regiões flanqueadas); um controle que verifica a linearização completa de pRN1120 (1 μg de pRN1120 linearizado).
- Siga as etapas 5.2.9 para 5.2.12 para a primeira rodada de transformação.
- Placa para fora 150 μL da mistura da transformação e da diluição 20x no YEPD (glicose de 2%) da mistura da transformação no agar de YEPD (glicose 2%) suplementado com 0,2 g/L G418 e 0,2 g/L NTC. Controles de placa para fora em agar YEPD (2% glicose) suplementado com a seleção apropriada (G418 e/ou NTC ou nenhuma seleção). Incubar as placas a 30 ° c por 48 – 72 horas.
- Escolha um único transformante colorido e re-Streak em um YEPD (2% glicose) placa de agar para obter únicas colônias coloridas.
- Prepare uma pré-cultura, crescendo a cepa pré-expressando Cas12a, criada na primeira rodada de transformação (etapa 5,2), em um balão de agitação de 100 mL contendo 20 mL de meio de YEPD (2% de glicose) suplementado com 0,2 g/L G418. Incubar durante a noite a 30 ° c com agitação 250 rpm.
6. avaliação da eficiência de edição do genoma
- Conte o número de colônias coloridas e colônias brancas nas placas de transformação.
- Calcule a eficiência de edição do genoma dividindo o número de colônias coloridas pelo número total de colônias (brancas e coloridas), como mostrado na tabela 1.
7. confirmação da integração do DNA do doador no loci pretendido
- Re-Streak uma única colônia colorida a partir de uma placa de transformação em uma placa de agar YEPD (2% glicose) sem G418 e seleção NTC e incubar por 48 horas a 30 ° c.
- Escolha uma única colônia e inocular um balão de agitação de 500 mL preenchido com 100 mL de meio de YEPD (2% de glicose). Incubar por 48 horas a 30 ° c e agitação em 250 rpm.
- Isolar o DNA genómico como descrito na seção 4,1.
Nota: Alternativamente, use um protocolo para a preparação do fermento para a PCR da colônia propor previamente por looke et al. 32. neste caso, o crescimento no meio líquido (secção 7,2) pode ser ignorado. - Verifique a integração correta por amplificação de dois fragmentos por gaveta de expressão integrada.
- Projete primers que recoze ao ADN genomic fora das regiões flanqueando transformadas e do gene do interesse (veja exemplos na tabela complementar 2, KC-121 a KC-132). Ao usar os primers KC-121 para KC-132, defina a temperatura de recozimento no programa de PCR para 62 ° c.
- Amplificar a região de interesse, conforme descrito na seção 4.4.2. Adapte o programa do PCR, ajusta especificamente a época da etapa da extensão no PCR de acordo com o comprimento do molde e das recomendações do fabricante para o polymerase do ADN.
- Verific o tamanho dos produtos do PCR pela electroforese em um gel do agarose (0,8%, 40 minutos, 5 V/cm) usando uma tintura do carregamento do ADN e uma escada do ADN com fragmentos do ADN em uma escala de 100 a 10.000 BP.
8. criação da arte do pixel do fermento usando um alimentador líquido acústico
- Prepare um modelo de imagem para a arte de pixel de levedura.
- Redimensione a imagem RGB original (220 × 280 pixels, veja os resultados representativos), por exemplo , usando o ImageJ para criar um final 64 × 96 pixels (largura × altura) em escala de cinza visualizado em cores pretendidas (resultados representativos).
- Converta a imagem RGB em escala de cinza usando esta fórmula:

onde eugr, ir, ig, ib são as intensidades cinza, vermelho, verde e azul, respectivamente. - A fim de categorizar os pixels, desenvolver um plugin ImageJ aplicando as seguintes regras: (a) se eugr is ≤ 64, use o fermento laranja escuro (cepa 1, tabela complementar 3) para este pixel. (b) se 64 < Igr ≤ 128, use o fermento laranja (estirpe 2, tabela complementar 3) para este pixel. (c) se 128 < Igr ≤ 192, use o fermento amarelo (estirpe 3, tabela complementar 3) para este pixel. d) se eugr > 192, use o fermento branco (CEN. PK113-7D) para este pixel.
- Spot células de levedura para criar a arte de pixel de levedura.
- Inocular 500 mL agitar frascos contendo 100 mL de YEPD (2% glicose) médio com três diferentes de cor carotenóide produzindo S. cerevisiae estirpe e selvagem tipo CEN. PK113-7D. Incubar culturas durante a noite a 30 ° c com agitação em 250 rpm.
- Transfira 0,5 mL da cultura overnight para um tubo preenchido com 0,5 mL de meio de gradiente de densidade não-iônico estéril (veja a tabela de materiais). Misture por vortexing brevemente.
- Transfira a suspensão celular para um reservatório qualificado, 2 x 3 bem. Realize a mancha usando um instrumento acústico do alimentador líquido de uma placa qualificada da fonte do reservatório a um microplate (veja a tabela dos materiais) que contem 50 ml do agar de yepd (glicose 2%). Para simplificar o chapeamento, definir poços na placa, por exemplo. Use uma microplaca como uma placa de poço 6144 (64 × 96).
- Spot 25 nL de cada cepa S. cerevisiae da placa de fonte de reservatório de 2x 3 bem usando um arquivo. csv com a configuração de calibração de fluido 6Res_aq_gpsa2 na microplaca de destino. Defina cada uma dessas 25 gotas de nL como um pixel na grade 64 x 96 que é traduzida para as posições de poço (A01, B01, C01 etc.).
- Incubar a microplaca a 30 ° c durante 48 horas. Para intensificar as cores das cepas armazenar a placa de agar a 4 ° c por pelo menos 72 horas.
Resultados
O protocolo para a edição multiplex do genoma usando crisrp/Cas12a foi demonstrado construindo três estirpes de S. cerevisiae de produção do carotenóide que expressam os genes de CRTe, de crtyb e de crti usando promotores heterólogo de alta, média e baixa resistência: estirpe 1, 2 e,3 respectivamente (tabela complementar 3). A construção destas estirpes exigiu a geração de três gavetas da expressão do ADN do doador e de seis regiões flanqueando por a tensão para alvejar a três Locus diferentes no ADN genomic (mostrado em Figura 2B). Como descrito nisto, o promotor, o frame de leitura aberto, o terminador e as duas seqüências contíguas dos conectores 50-BP foram montados em uma gaveta da expressão através de uma reação dourada da clonagem da porta e o conjunto foi verificado pelo PCR (Figura 3a). A única matriz de crRNA foi ordenada como fragmento de DNA sintético e foi amplificada por PCR (Figura 3B). O plasmídeo receptor para a única matriz de crRNA (plasmídeo pRN1120) foi linearizado com ecori-HF e xhoI e a linearização foi confirmada por eletroforese (Figura 3C). As sequências de desenho e nucleotídeo das gavetas de expressão de DNA do doador introduzido e das regiões de flanqueamento são mostradas na tabela complementar 3 e na tabela complementar 4. A sequência de gavetas de expressão de matriz crRNA única é fornecida na tabela complementar 1. A funcionalidade dos espaçadores incluídos no único array crRNA foi testada com antecedência pela edição do genoma singleplex com crRNAs individuais19.
A eficiência da edição do genoma utilizando o Cas12a foi primeiramente avaliada com base no número de colônias coloridas obtidas após a transformação (tabela 1, Figura 4). A eficiência de edição das três cepas construídas variou de 50% a 94%. Notavelmente, a introdução de fitas de expressão usadas para gerar estirpe 1 exibiu a menor eficiência de edição, possivelmente causada pela natureza do DNA do doador (i.e., essas fitas de expressão codificam CRTe, Crtyb e crti de três promotores de alta resistência). Em segundo lugar, a correta integração das três gavetas de expressão de DNA dos doadores nos Locos pretendidos no DNA genómico foi confirmada por PCR (Figura 5). Os primers foram projetados de tal maneira que os produtos do PCR foram obtidos quando a integração correta do ADN fornecedor no locus pretendido ocorreu. Para cada experimento de transformação, oito colônias foram colhidas da placa de transformação e testadas (Note-se que apenas três são apresentadas na Figura 5). Em geral, das 8 colônias testadas por DNA doador, foi confirmada a correta integração do DNA do doador CRTe no locus INT1, CRTYB no locus Int2 e crti no locus Int3, em > 90% dos transformantes. Estes resultados demonstram o sistema de CRISPR/Cas12a em combinação com uma única disposição do crRNA permite a edição multiplex eficiente do genoma de S. cerevisiae em loci múltiplo simultaneamente.
Além disso, demonstramos a criação de "arte de pixel de levedura" usando as três cepas produtoras de carotenóides que foram construídas em conjunto com uma cepa de tipo selvagem não colorida. A partir de uma imagem em preto e branco de Rosalind Franklin (Figura 6a), um quadro de 4 cores (Figura 6B) e lista de spotting foi criado, que foi então usado para detectar as quatro diferentes cepas de levedura em uma microplaca de agar usando um manipulador de líquido acústico, resultando em uma "pintura de levedura" de alta resolução de Rosalind Franklin (Figura 6C, D, E).

Figura 1 : Fluxo de trabalho do protocolo para edição de genoma MULTIPLEX crispr/Cas12a em S. cerevisiae. O fluxo de trabalho inclui etapas cruciais do método apresentado. Para obter detalhes, consulte o protocolo. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2 : Esquema de edição de genoma multiplex CRISPR/Cas12a usando uma única matriz crRNA. (A) a única matriz de crrna é compor de três unidades de crrnas em sua forma madura, uma repetição direta de 20 BP específica para LbCas12a (quadrados cinzentos) com uma seqüência do guia 23-BP (diamantes coloridos). A expressão da matriz crRNA é habilitada pelo SNR52 promoter e SUP4 Terminator. A transformação de S. cerevisiae com um pRN1120 linearizado e a única gaveta da expressão da disposição do crrna que contem o homologia com pRN1120 (listras diagonais) permite in vivo o recombinação em um plasmídeo circular nas pilhas que pre-expressam LbCas12a. A única matriz crRNA é processada subseqüentemente por Cas12a. (B) Cas12a é dirigido aos locais pretendidos do INT1, Int2 e Int3 genomic e cria rupturas encalhados dobro. Na mistura da transformação, o ADN fornecedor que consiste em regiões flanqueando e na gaveta do gene da expressão do carotenóide foi incluído. Os conjuntos do ADN do doador foram alvejados a um estiramento do ADN no ADN genomic em torno do INT1 (CRTe), Int2 (crtyb) e Int3 (crti) loci pela recombinação in vivo devido à presença de seqüências homólogo dos conectores 50-BP, indicado como 5, A, B, C, D ou E. P1 – P3, diferentes promotores; T1 – T3, terminadores diferentes. Este valor foi modificado de Verwaal et al. 201819. Construções genéticas mostradas usando símbolos visuais da linguagem aberta de biologia sintética (SBOL)40. Por favor clique aqui para ver uma versão maior desta figura.
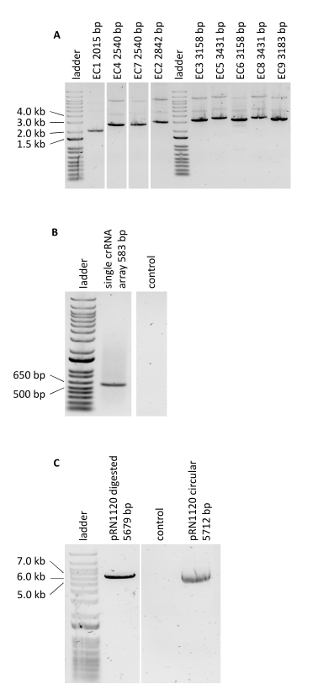
Figura 3 : PCR verificando os experimentos de edição do genoma. (A) verificação das reações de clonagem de portão dourado de fitas de DNA de doadores montadas. Os resultados obtidos estão de acordo com os comprimentos esperados. (B) PCR do único array crrna. (C) linearização do plasmídeo pRN1120. Por favor clique aqui para ver uma versão maior desta figura.

Figura 4 : Placas de transformações de S. cerevisiae usando a abordagem de edição de genoma multiplex. (A) estirpe 1 expressando CRTe, Crtyb e crti de três fortes promotores (colônias de laranja escura). (B) estirpe 2 expressando CRTe, Crtyb e crti de três promotores de força média (colônias de laranjeira). (C) estirpe 3 expressando CRTe, Crtyb e crti de três promotores de baixa resistência (colônias amarelas). Por favor clique aqui para ver uma versão maior desta figura.

Figura 5 : PCR que verific a integração das gavetas fornecedoras da expressão do ADN no loci pretendido dentro do ADN genomic. (A) verificação de três colônias da cepa 1. B) verificação de três colónias da estirpe 2. (C) verificação de três colônias da cepa 3. Por favor clique aqui para ver uma versão maior desta figura.

Figura 6 : Arte do pixel do fermento de Rosalind Franklin. (A) preto e branco foto RGB de 220 × 280 pixels de Rosalind Franklin que foi usado como um modelo. (B) conversão de computador da foto em preto e branco de Rosalind Franklin em uma lista de pixel de 4 cores 64 × 96. (C) foto da arte do pixel do fermento com 64 × 96 colônias do fermento com uma seção ampliada. (D) foto de um manipulador de líquido acústico com duas placas cultivadas completas. (E) foto de um microplate crescido cheio com 64 × 96 colônias do fermento. Por favor clique aqui para ver uma versão maior desta figura.
| Estirpe 1 | Estirpe 2 | Estirpe 3 | |
| Colônias coloridas | 16 | 279 | 220 |
| Colônias brancas | 16 | 18 | 18 |
| Colônias totais | 32 | 297 | 238 |
| Eficiência | 50% de | 94% de | 92% de |
Tabela 1: editando a eficiência da abordagem de edição de genoma multiplex.
| seqüência de matriz de crRNAa, b, c, d, e, f |
|
Catgtttgacagcttatcatcgataatccggagctagcatgcggccgctctagaactagtggatcccccgggctgcag O Tctttgaaaa GATAATGTATGATTATGCTTTCACTCATATTTATACAGAAACTTGATGTTTTCTTTCGAGTATATACAAGG TGATTACATGTACGTTTGAAGTACAACTCTAGATTTTGTAGTGCCCTCTTGGGCTAGCGGTAAAGGTGCGCA TTTTTTCACACCCTACAATGTTCTGTTCAAAAGATTTTGGTCAAACGCTGTAGAAGTGAAAGTTGGTGCGC ATGTTTCGGCGTTCGAAACTTCTCCGCAGTGAAAGATAAATGATCAatttctactaagtgtagat CTGGTGGGAGAGAAAGCTTATGAAatttctactaagtgtagatgtgccgtac GCCGGAGCCGACGGAatttctactaagtgtagattgcccctcttatacgattatattTT TTTTTGTTTTTTATGTCTggggggcccggtacccagcttttgttccctttagtgagg GTTAATTCCGAGCTTGGCGTAATCATGGTCATAGCTGTTTCCTGTGTG |
| a. homologia para pRN1120 (negrito). promotor b. SNR52 (itálico). c. sequências de alvos genómicas (sublinhadas). d. guia de repetições diretas específicas para LbCas12a (itálico, negrito). e. SUP4 terminador (itálico). f. homologia para pRN1120 (negrito). |
Tabela complementar 1: matriz crRNA única para LbCas12a contendo homologia com pRN1120 plasmídeo.
| Nome | Sequênciaa | Descrição ob | Usado no ponto |
| KC-101 | CATGTTTGACAGCTTATCATC | Primer FW para amplificação de matriz crRNA única | 2.1.4 |
| KC-102 | CACACAGGAAACAGCTATGAC | Primer RV para amplificação de matriz crRNA única | 2.1.4 |
| KC-103 | AAGCGACTTCCAATCGCTTTGC | Primer FW para amplificação do DNA do doador com conector 5 | 3.6.1 |
| KC-104 | AAGCAAGGAAGGAGAGAAC | Primer RV para amplificação de DNA doador com conector A | 3.6.1 |
| KC-105 | CGGATCGATGTACACAACCG | Primer FW para amplificação do DNA do doador com conector B | 3.6.1 |
| KC-106 | CAACAGGAGGCGGATGGATATAC | Primer RV para amplificação de DNA doador com conector C | 3.6.1 |
| KC-107 | AACGTTGTCCAGGTTTGTATCC | Primer FW para amplificação do DNA do doador com conector D | 3.6.1 |
| KC-108 | AGGTACAACAAGCACGACCG | Primer RV para amplificação do DNA do doador com conector E | 3.6.1 |
| KC-109 | CACTATAGCAATCTGGCTATATG | Primer FW para amplificação de INT1 5 ' com conector 5 | 4,4 |
| KC-110 |
AAACGCCTGTGGGTGTGGTAC TGGATATGCAAAGCGATTGGAA gactcctctgccgtc GTCGCTT O ATTCC | Primer RV para amplificação de INT1 5 ' com conector 5 | 4,4 |
| KC-111 |
TTGCCCATCGAACGTACAAG TACTCCTCTGTTCTCTCCTTCCTT TGCTTTaagcgttgaagtttcctc TAGT | Primer FW para amplificação de INT1 3 ' com conector A | 4,4 |
| KC-112 | TGTCAACTGGAGAGCTATCG | Primer RV para amplificação de INT1 3 ' com conector A | 4,4 |
| KC-113 | AGAAGATTTCTCTTCAATCTC | Primer FW para amplificação de INT2 5 ' com conector B | 4,4 |
| KC-114 |
TGCTAAGATTTGTGTTCGTT TGGGTGCAGTCGGTTGTGTACAT CGATCCGcccttatcaaggatacc TGGTTG | Primer RV para amplificação de INT2 5 ' com conector B | 4,4 |
| KC-115 |
ACGCTTTCCGGCATCTTCCA GACCACAGTATATCCATCCGCCT (Araceli) O GTGG | Primer FW para amplificação de INT2 3 ' com conector C | 4,4 |
| KC-116 | TCTCCTCTTCGATGACCGGG | Primer RV para amplificação de INT2 3 ' com conector C | 4,4 |
| KC-117 | GGTCGTTTTTGTGCAGCATATTG | Primer FW para amplificação de INT3 5 ' com conector D | 4,4 |
| KC-118 |
GCGGAATATTGGCGGAACGG ACACACGTGGATACAAACCTG (araacgtt) AAGAACG | Primer RV para amplificação de INT3 5 ' com conector D | 4,4 |
| KC-119 |
AAATAACCACAAACATCCTT CCCATATGCTCGGTCGTGCTTGTT o GTACCTgatgggacgtcagcact GTAC | Primer FW para amplificação de INT3 3 ' com conector E | 4,4 |
| KC-120 | GAGCTTACTCTATATATTCATTC | Primer RV para amplificação de INT3 3 ' com conector E | 4,4 |
| KC-121 | GTTACTAAACTGGAACTGTCCG | Primer FW para verificação da integração de CON5-crtE-INT1 5 ' | 7.4.1 |
| KC-122 | CACTGCTAACTACGTTTACTTC | Primer FW para verificação da integração de CON5-crtE-INT1 3 ' | 7.4.1 |
| KC-123 | CACTGGAACTTGAGCTTGAG | FW primer para verificação da integração de conB-crtYB-conC a INT2 5 ' | 7.4.1 |
| KC-124 | GTCTCCAGCTGAATTGGTCC | FW primer para verificação da integração de conB-crtYB-conC a INT2 3 ' | 7.4.1 |
| KC-125 | CTCTCATGAAGCAGTCAAGTC | Primer FW para verificação da integração de conD-crtI-conE a INT3 5 ' | 7.4.1 |
| KC-126 | GATCGGTCAATTAGGTGAAG | Primer FW para verificação da integração de conD-crtI-conE a INT3 3 ' | 7.4.1 |
| KC-127 | CCTTGTCCAAGTAGGTGTCC | Primer RV para verificação da integração de CON5-crtE-INT1 5 ' | 7.4.1 |
| KC-128 | GCTGTCATGATCTGTGATAAC | Primer RV para verificação da integração de CON5-crtE-INT1 3 ' | 7.4.1 |
| KC-129 | O CTGGCAATGTTGACCAATTGC | Primer RV para verificação da integração de conB-crtYB-conC a INT2 5 ' | 7.4.1 |
| KC-130 | CCAACGTGCCTTAAAGTCTG | Primer RV para verificação da integração de conB-crtYB-conC a INT2 3 ' | 7.4.1 |
| KC-131 | CCTTACCTTCTGGAGCAGCAG | Primer RV para verificação da integração de conD-crtI-conE a INT3 5 ' | 7.4.1 |
| KC-132 | O CTGGTTACTTCCCTAAGACTG | Primer RV para verificação da integração de conD-crtI-conE a INT3 3 ' | 7.4.1 |
| a. seqüências bold (realce) denotam seqüências do conector. b. os primers dianteiro e reverso são designados como FW e RV, respectivamente. |
Tabela complementar 2: sequências de primer.
Tabela complementar 3: desenho de cepas construídas.

Tabela complementar 4: seqüências de gavetas de expressão de DNA de doadores e regiões de descamação. Por favor, clique aqui para baixar este arquivo.
Discussão
O protocolo fornecido descreve a edição multiplex do genoma de S. cerevisiae usando Cas12a de Lachnospiraceae bactéria ND2006 em combinação com uma única disposição do crrna e ADN do doador. O projeto do único array do crRNA e do ADN do doador é explicado em detalhe. Em contraste com o sistema crispr/Cas9 bem estabelecido, o crispr/Cas12a tem a capacidade adicional única de processar crrnas múltiplas expressas a partir de uma única matriz de crrna13,33. Devido a esse recurso, a edição simultânea de vários alvos é mais fácil de configurar e pode ser alcançada em uma única transformação. Esta única aproximação da disposição do crRNA foi demonstrada antes por Zetsche et al. 34 que, simultaneamente, editou até quatro genes em células de mamíferos usando AsCas12a, e por Swiat et al. 35 que introduziu quatro fragmentos de DNA em um genoma de levedura usando FnCas12a. A nosso conhecimento, um número mais elevado de modificações genomic simultâneas usando um sistema Cas12a não foi relatado e o limite máximo dos alvos por a única disposição para Cas12a é ainda ser determinado. Uma pesquisa mais adicional que utiliza únicas matrizes do crrna em combinação com o Cas12a inclui o Regulamento transcricional multiplex em uma escala larga dos organismos33,36,37.
Existem algumas etapas críticas no protocolo apresentado. Projete cuidadosamente todas as sequências de DNA envolvidas no experimento de edição do genoma Cas12a, especialmente no caso de novas sequências de DNA serem introduzidas. Determine a funcionalidade de novas sequências de espaçador parte de um crRNA, por exemplo, por um experimento de edição de genoma singleplex, conforme descrito por Verwaal et al. 19 antes de combiná-los em uma única matriz crrna. Siga as recomendações para a preparação de soluções de buffer de transformação usadas no experimento de edição Cas12a para alcançar uma boa eficiência de transformação do fermento.
Existem algumas modificações opcionais da técnica. Recomenda-se usar 1 μg de cada DNA de doador, pRN1120 linearizado ou única gaveta de expressão de matriz crRNA na transformação, embora o uso de uma menor quantidade de DNA também seja esperado para resultar em uma eficiência de transformação satisfatória. Realize uma transformação de teste para determinar se os valores de DNA inferiores podem ser usados. A transformação de S. cerevisiae pode ser realizada por meio de um método diferente daquele descrito neste protocolo, por exemplo, o protocolo descrito por gietz et al. (2007) 38. o plasmídeo receptor de RNA guia pRN1120 é adequado para a expressão de uma única matriz crrna e crrna única de diferentes variantes de Cas12a (por exemplo, de acidaminococcus spp. BV3L6 ou Francisella novicida U112) bem como para a expressão de sgrna em combinação com Cas919. O ADN do doador não precisa de ser limitado às gavetas da expressão de gene do carotenóide e às regiões flanqueando que visam o ADN do doador aos locais descritos do INT1, do INT2 e do INT3 no ADN genomic. Todo o ADN do interesse pode ser introduzido, em uma maneira multiplex, no ADN genomic do anfitrião pelos princípios de projeto descritos neste protocolo, ou alternativamente o ADN fornecedor pode ser usado para suprimir do ADN de um genoma do anfitrião. A estrutura modular do único array crRNA facilita o ajuste fácil do espaçador e sequências de repetição diretas. A modificação das sequências espaçadoras permite uma alteração do locus de integração pretendido que pode ser desenhado por uma das ferramentas para a identificação de um local de destino genómico, por exemplo , software guidescan 1,039. Em vez de usar grandes sequências de flanquear que contêm seqüências de conectores, 50-BP da região de flanqueamento pode ser incluído nas seqüências de DNA do doador incorporando essas seqüências de região de flanqueamento 50-BP nos primers usados na PCR. Neste caso, no total apenas três em vez de nove fragmentos de DNA de doadores são necessários para um experimento de edição de genoma multiplex bem-sucedido.
Em resumo, este protocolo fornece instruções passo a passo para executar a edição multiplex do genoma em S. cerevisiae usando Cas12a em combinação com uma única abordagem de matriz crrna. O protocolo foi demonstrado pela edição multiplex do genoma usando 9 fragmentos do ADN do doador e a única codificação da disposição do crRNA para três gRNAs. Mostramos altas frequências de edição global entre 50% e 94% para os três designs de estirpe relatados aqui. Concluindo, a característica única do Cas12a é a capacidade de processar uma única matriz crRNA em crRNAs individuais em uma célula, o que torna Cas12a uma excelente ferramenta para permitir a edição de genoma multiplex e desenvolver módulos de regulação transcricional visando vários fitas de expressão de uma só vez. No final, três cepas foram obtidas produzindo carotenóides em um nível diferente e cores em tons entre amarelo e laranja. Com essas cepas e uma estirpe de tipo selvagem, mostramos como um manipulador de líquido acústico pode ser usado de forma direta para fazer a arte de pixel de levedura – isso em homenagem a Rosalind Franklin que contribuiu para a descoberta da estrutura do DNA 65 anos atrás por sua famosa foto 5123 < /C1 >.
Divulgações
Os autores declaram que existe um conflito de interesses. Os autores preencheram o IP relacionado aos métodos apresentados.
Agradecimentos
Este projecto recebeu financiamento do programa de investigação e inovação Horizon 2020 da União Europeia, a Convenção de subvenção n. º 686070 (DD-DeCaf) e 764591 (SynCrop), e do programa de investigação Building Blocks of Life com o número de projecto 737.016.005 por da organização neerlandesa de investigação científica (NWO). T foi apoiado pela sociedade real (Grant UF160357) e BrisSynBio, um centro de pesquisa de biologia sintética BBSRC/EPSRC (Grant BB/L01386X/1). Agradecemos Zi di e Jeffrey Van Wijk por sua contribuição para as experiências de manchas de levedura para criar a arte de pixel de levedura.
Materiais
| Name | Company | Catalog Number | Comments |
| Chemicals specific for the protocol | |||
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | Electrophoresis |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | Selection of E. coli transformants |
| BsaI-HF (20 U/µl) | New England BioLabs | R353L | Golden Gate Cloning |
| Cell Lysis Solution (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| CutSmart Buffer | New England BioLabs | B7204S | Linearization of pRN1120 |
| Deoxyribonucleic acid sodium salt from salmon testes | Sigma Aldrich | D1626 | Transfromation of S. cerevisiae (carrier DNA) |
| dNTPs | Invitrogen | 10297018 | PCRs |
| EcoRI-HF | New England BioLabs | R3101S | Linearization of pRN1120 |
| Ethanol absolute for analysis | Merck | 100983 | Isolation of genomic DNA from S. cerevisiae |
| Ethylenediamine-tetraacetic acid | Sigma Aldrich | ED | Transformation of S. cerevisiae |
| G418 disulfate salt | Sigma Aldrich | A1720 | Selection of S. cerevisiae transformants |
| Histodenz | Sigma Aldrich | D2158 | Yeast pixel art |
| Isopropanol | Merck | 100993 | Isolation of genomic DNA from S. cerevisiae |
| Lithium acetate dihydrate | Sigma Aldrich | L6883 | Transformation of S. cerevisiae |
| Nancy-520 DNA Gel Stain | Sigma Aldrich | 1494 | Electrophoresis |
| NEB10 competent E. coli cells | New England BioLabs | C3019H | Transformation of E. coli: dx.doi.org/10.17504/protocols.io.nkvdcw6 |
| Nourseothricin | Jena Bioscience | AB102 | Selection of S. cerevisiae transformants |
| Phusion buffer | New England BioLabs | M0530L | PCRs |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530L | PCRs |
| Polyethylene glycol 4000 | Merck | 7490 | Transformation of S. cerevisiae |
| Protein Precipitation Solution (10 M NH4AC) (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| Purple loading dye | New England BioLabs | B7024S | Electrophoresis |
| QIAprep Spin Miniprep Kit | QIAGEN | 27106 | Purification of plasmids |
| RNase coctail enzyme mix | Thermo Fisher Scientific | AM2286 | Isolation of genomic DNA from S. cerevisiae |
| T4 DNA ligase buffer | Invitrogen | 46300-018 | Golden Gate Cloning |
| T4 DNA Ligase (1 U/µl) | Invitrogen | 1705218 | Golden Gate Cloning |
| UltraPure Agarose | Invitrogen | 16500500 | Electrophoresis |
| Wizard SV Gel and PCR Clean-Up System Kit | Promega | A9282 | Purification of PCR products and linearized pRN1120 |
| Xhol | New England BioLabs | R0146S | Linearization of pRN1120 |
| Zymolyase 50 mg/ml (5 units/µL) | Zymo Research | E1006 | Isolation of genomic DNA from S. cerevisiae (yeast lysis enzyme) |
| Zymolyase storage buffer | Zymo Research | E1004-B | Isolation of genomic DNA from S. cerevisiae (necessary for the preparation of yeast lysis enzyme) |
| Chemicals of general use | |||
| 2*Peptone-Yeast extract (PY) agar | Plate growth of E. coli | ||
| 2*PY medium | Cultivation of E. coli | ||
| Demineralized water | Transformation of S. cerevisiae | ||
| ELFO buffer | Electrophoresis | ||
| MQ | Multiple steps | ||
| Physiological salt solution | Transformation of S. cerevisiae | ||
| TE buffer | Storage of DNA, transformation of S. cerevisiae | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | Cultivation of S. cerevisiae | ||
| YEPD (2% glucose) agar | Plate growth of S. cerevisiae | ||
| Consumables | |||
| Eppendorf tubes | |||
| Falcon tubes (50 mL) | |||
| Microplate 96 wells | |||
| Petri dishes | |||
| Pipette tips 0.5 - 10 µL | |||
| Pipette tips 10 - 200 µL | |||
| Pipette tips 100 - 1000 µL | |||
| Shake flasks (500 mL) | |||
| Sterile filters | |||
| Equipment | |||
| Centrifuge (Falcon tubes) | |||
| Echo 525 acoustic liquid handler | |||
| Incubator | |||
| NanoDrop | |||
| Set for eletrophoresis | |||
| Spectrophotometer | |||
| Table centrifuge (Eppendorfs tubes) | |||
| Thermocycler | |||
| Plasmids | |||
| pCSN067 | Addgene | ID 101748 | https://www.addgene.org/ |
| pRN1120 | Addgene | ID 101750 | https://www.addgene.org/ |
| Strains | |||
| S. cerevisiae strain CEN.PK113-7D | EUROSCARF collection | http://www.euroscarf.de |
Referências
- Knott, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361 (6405), 866-869 (2018).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Lian, J., HamediRad, M., Hu, S., Zhao, H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications. 8 (1), 1688(2017).
- Li, Z. -H., Liu, M., Lyu, X. -M., Wang, F. -Q., Wei, D. -Z. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology. 58 (12), 1100-1104 (2018).
- Shao, Y., Lu, N., Qin, Z., Xue, X. CRISPR-Cas9 facilitated multiple-chromosome fusion in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (11), 2706-2708 (2018).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology. 13 (11), 722-736 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), (2016).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovič, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Lian, J., HamediRad, M., Zhao, H. Advancing metabolic engineering of Saccharomyces cerevisiae. using the CRISPR/Cas System. Biotechnology Journal. 13 (9), 1700601(2018).
- Ferreira, R., et al. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (1), 10-15 (2018).
- Swarts, D. C., Martin, J. Cas9 versus Cas12a/Cpf1: Structure–function comparisons and implications for genome editing. Wiley Interdisciplinary Reviews: RNA. 9 (5), 1481(2018).
- Strohkendl, I., Saifuddin, F. A., Rybarski, J. R., Finkelstein, I. J., Russell, R. Kinetic Basis for DNA Target Specificity of CRISPR-Cas12a. Molecular Cell. 71 (5), 816-824 (2018).
- Verwaal, R., et al. High-level production of beta-carotene in Saccharomyces cerevisiae. by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous. Applied and Environmental Microbiology. 73 (13), 4342-4350 (2007).
- Verwaal, R., Buiting-Wiessenhaan, N., Dalhuijsen, S., Roubos, J. A. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast. 35 (2), 201-211 (2018).
- Jakociunas, T., Jensen, M. K., Keasling, J. D. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering. 34, 44-59 (2016).
- Engler, C., Romy, K., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PloS One. 3 (11), 3647(2008).
- Yeast Art project. , Available from: http://www.yeastart.org (2018).
- Franklin, R. E., Gosling, R. G. Molecular configuration in sodium thymonucleate. Nature. 171, 740-741 (1953).
- Watson, J. D., Crick, F. H. A structure for deoxyribose nucleic acid. Nature. 171, 737-738 (1953).
- Wilkins, M. H. F., Stokes, A. R., Wilson, H. R. Molecular structure of deoxypentose nucleic acids. Nature. 171, 738-740 (1953).
- Young, E. M., et al. Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast. Metabolic Engineering. 48, 33-43 (2018).
- Roubos, J. A., Pel, H. J., Meijrink, B. Cloning Method. , WO2013144257 (2013).
- Mandel, M., Higa, A. Calcium-dependent bacteriophage DNA infection. Journal of Molecular Biology. 53 (1), 159-162 (1970).
- Van Dijken, J. P., et al. An interlaboratory comparison of physiological and genetic properties of four Saccharomyces cerevisiae strains. Enzyme and Microbial Technology. 26 (9-10), 706-714 (2000).
- Gietz, R. D., Schiestl, R. H., Willems, A. R., Woods, R. A. Studies on the transformation of intact yeast cells by the LiAc/SS-DNA/PEG procedure. Yeast. 11 (4), 355-360 (1995).
- Hill, J., Donald, K. A., Griffiths, D. E., Donald, G. DMSO-enhanced whole cell yeast transformation. Acids Research. 19 (20), 5791(1991).
- Looke, M., Kristjuhan, K., Kristjuhan, A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques. 50 (5), 325-328 (2011).
- Tak, Y. E., et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nature Methods. 14 (12), 1163-1166 (2017).
- Zetsche, B., et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature biotechnology. 35 (1), 31-34 (2017).
- Swiat, M. A., et al. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research. 45 (21), 12585-12598 (2017).
- Li, L., et al. CRISPR-Cpf1-Assisted Multiplex Genome Editing and Transcriptional Repression in Streptomyces. Applied Environmental Microbiology. 84 (18), 00827-00918 (2018).
- Zhang, X., et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery. 3, 17018(2017).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 1-4 (2007).
- Perez, A. R., et al. GuideScan software for improved single and paired CRISPR guide RNA design. Nature Biotechnology. 35 (4), 347-349 (2017).
- Cox, R. S., et al. Synthetic Biology Open Language Visual (SBOL Visual) Version 2.0. Journal of Integrative Bioinformatics. 15 (1), 1613-4516 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados