Method Article
CRISPR/Cas12a Multiplex Genom Bearbeitung von Saccharomyces cerevisiae und die Schaffung von Hefe Pixel Kunst
In diesem Artikel
Zusammenfassung
Das CRISPR/Cas12a-System in Kombination mit einem einzigen crRNA-Array ermöglicht eine effiziente Multiplex-Bearbeitung des S. cerevisiae Genoms an mehreren Loci gleichzeitig. Dies wird durch die Konstruktion von Carotinoid produzierenden Hefestämmen demonstriert, die anschließend zur Erzeugung von Hefepixelkunst verwendet werden.
Zusammenfassung
Hohe Effizienz, Benutzerfreundlichkeit und Vielseitigkeit des gruppierten, regelmäßig interspaceierten kurzen palindromischen Wiederholungs-/CRISPR-assoziierten Proteins 9 (CRISPR/Cas9) System hat eine fortgeschrittene genetische Modifikation von Saccharomyces cerevisiaeermöglicht, einem Modell Und Arbeitspferd in der industriellen Biotechnologie. CRISPR-assoziiertes Protein 12a (Cas12a), eine RNA-geführte Endonuklease mit von Cas9 unterscheidbaren Merkmalen, wird in dieser Arbeit angewendet, wodurch die molekulare Toolbox für die Genombearbeitung weiter erweitert wird. Ein Vorteil des CRISPR/Cas12a-Systems ist, dass es in der Multiplex-Genombearbeitung mit mehreren Führungs-RNAs verwendet werden kann, die aus einer einzigen Transkriptionseinheit (einzelnes CRISPR-RNA-Array (crRNA)-Array) exprimiert werden. Wir präsentieren ein Protokoll zur Multiplex-Integration mehrerer heterologer Gene in unabhängige Loci des S. cerevisiae-Genoms mit dem CRISPR/Cas12a-System mit mehreren crRNAs, die aus einem einzigen crRNA-Arraykonstrukt exprimiert werden. Die vorgeschlagene Methode nutzt die Fähigkeit von S. cerevisiae, in vivo Rekombination von DNA-Fragmenten durchzuführen, um das einzelne crRNA-Array zu einem Plasmid zusammenzubauen, das für die transformative Selektion sowie die Montage von Spender-DNA verwendet werden kann. Sequenzen, die sich an beabsichtigten Positionen in das Genom integrieren. Cas12a ist konstitutiv vorab exprimiert, was die Spaltung des S. cerevisiae Genoms an den vorgesehenen Positionen bei Expression des einzelnen crRNA-Arrays erleichtert. Das Protokoll umfasst das Design und die Konstruktion eines einzelnen crRNA-Arrays und einer Spender-DNA-Expressionskassette und nutzt einen Integrationsansatz, der einzigartige 50-bp-DNA-Konnektorensequenzen und separate Integrationsflanken-DNA-Sequenzen nutzt, was die experimentelles Design durch Standardisierung und Modularisierung und erweitert das Anwendungsspektrum. Schließlich zeigen wir eine einfache Technik zur Erstellung von Hefepixelkunst mit einem akustischen Flüssigkeitshandler mit verschiedenfarbigem Carotinoid, das Hefestämme produziert, die konstruiert wurden.
Einleitung
CRISPR/Cas-Enzyme haben zweifellos die Molekularbiologie revolutioniert und wurden weithin als Werkzeuge für die Entwicklung von Genomen in einer Geschwindigkeit angenommen, die bisher nicht machbar war1. Die erste Modifikation eines Saccharomyces cerevisiae Genoms durch das CRISPR/Cas9 Genom-Editing-System wurde von DiCarlo et al. berichtet. 2, um erfolgreicheS Gen-Knock-out und Punktmutationen mit extern eingeführten Oligonukleotiden zu demonstrieren. Weitere Entwicklungen in der Hefe-CRISPR-Toolbox umfassten: Transkriptionsregulierung durch Fusion von katalytisch inaktiven toten Cas9 (dCas9) mit Transkriptionseffektordomänen, um die Aktivierung und das Silencing von Transkription3zu ermöglichen, Anwendung für beide Genom-Editing und regulatorische Funktionen für Metabolische Signalweg-Engineering durch gleichzeitige Aktivierung, Unterdrückung und Löschung4, Löschung von großen Fragmenten aus dem S. cerevisiae Genom5und Multiple-Chromosom-Fusionen6 .
CRISPR/Cas Genom-Editing-Systeme finden ihren Ursprung in adaptiven Immunsystemen von Bakterien und Archaeen und diese Systeme wurden von Molekularbiologen für die Genombearbeitung angepasst. Ihre Funktionalität basiert auf den Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR) DNA-Regionen, die RNA kodieren, die für die Erkennung der fremden DNA oder RNA und der CRISPR-assoziierten Gene (Cas) verantwortlich ist, die RNA-geführte endonukleases1,7,8,9. Basierend auf der jüngsten Genomanalyse von CRISPR/Cas-Systemen wurde vorgeschlagen, die CRISPR/Cas-Systeme in zwei Klassen, fünf Typen und 16 Subtypen10zu unterteilen. Die beiden Klassen werden anhand der Organisation von Effektorkomplexen unterschieden, die an der Zielspaltung beteiligt sind. In der Regel werden CRISPR/Cas-Systeme mit einer Organisation mit mehreren Untereinheiten als Klasse 1 kategorisiert, während Effektorkomplexe mit einer Untereinheit zur Klasse 210,11gehören. In diesem Artikel untersuchen wir die Klasse 2 Typ V Cas12a, früher Cpf110,12genannt, die eine Alternative zur Klasse 2 Typ II Cas9 darstellt. Obwohl Cas9 gut charakterisiert und weit verbreitet in der Forschung verwendet wird, bietet Cas12a zusätzliche Funktionen12. Erstens bildet Cas12a einen Komplex mit CRISPR-RNA (crRNA) von 42 bis 44 Nukleotiden, ohne dass eine zusätzliche transaktivierende CRISPR-RNA (tracrRNA) erforderlich ist. Daher kann eine kürzere Führungs-RNA in der Genombearbeitung mit CRISPR/Cas12a-Systemen im Vergleich zu CRISPR/Cas9 verwendet werden. Zweitens ermöglicht die einzigartige Endonuklease- und Endoribonuklease-Aktivität von Cas12a die Reifung seiner Pre-crRNA13. Diese RNase-Aktivität ermöglicht die Codierung mehrerer crRNAs auf einem einzelnen CRISPR crRNA-Array, während Cas9 die separate Expression jeder sogenannten Single-Guide-RNA (sgRNA) oder alternativ z.B. expression einer zusätzlichen Endonuklease ( erfordert. z.B., Csy4) in Kombination mit Erkennungsmotiven für Csy4, die jede sgRNA14,15umgeben. Drittens erfordert die Cas12a-Zielstandorterkennung ein protospacer-benachbartes Motiv (PAM) am 5'-Ende des Ziels und spaltet nach der +18/+23-Position von seinem PAM, was zu einer geballten DNA mit klebrigen Enden führt, während Cas9 ein PAM benötigt, das sich am 3'-Ende Ziel und spaltet nach der -3 Position und erzeugt stumpfe Endschnitte in der DNA12. Viertens unterscheidet sich die Konsensnukleotidsequenz des PAM zwischen Cas12a ((T)TTV) und Cas9 (NGG), was Cas12a zu einem vielversprechenden Kandidaten für die Ausrichtung auf T-reiche Promoter- und Terminator-Sequenzenmacht 16. Schließlich berichtete eine aktuelle Studie über eine größere Zielspezifität für Cas12a als für die einheimische Cas917.
Wir präsentieren ein Protokoll zur Verwendung des CRISPR/Cas12a-Systems zur Genombearbeitung von S. cerevisiae mit besonderem Schwerpunkt auf der gleichzeitigen Einführung mehrerer DNA-Expressionskassetten in unabhängige genomische Loci (Multiplex-Genombearbeitung) ein einzelnes crRNA-Array. Die wichtigsten Schritte des Protokolls sind in Abbildung 1dargestellt. Als Proof of Concept wurde das CRISPR/Cas12a-System zur Einführung von drei Expressionskassetten in das Genom von S. cerevisiae eingesetzt, die die Herstellung von .-Carotin18 ermöglichen, wie schematisch in Abbildung 2dargestellt. Die Produktion von C-Carotin wirkt sich auf den Phänotyp von S. cerevisiaeaus: d.h.bei erfolgreicher Einführung aller drei heterologen Gene, die für die Carotinoide Biosynthese benötigt werden, werden die weißen S. cerevisiae Zellen gelb oder orange, abhängig von der Expressionsstärke des Promotors jedes Gens. Aufgrund der einfachen visuellen Auslesung dieses Weges wurde es eingeführt, um fortschrittliche CRISPR-basierte Systeme und Methoden für die Genombearbeitung zu entwickeln19,20. In dieser Arbeit wurden Expressionskassetten, die die Carotinoidgene crtE, crtYB und crtI kodieren, mit einem Golden Gate Kloning (GGC) Ansatz21 mit heterologen Promotoren und homologen Terminatoren konstruiert. verwendet, um die Expression der Gene zu fördern. Die Expressionskassetten sind von eindeutigen 50-Base-Paaren (bp) Sequenzen umgeben, die als Konnektoren bezeichnet werden und eine In-vivo-Montage mit Integrationsflanken-DNA-Sequenzen (flankierende Bereiche) mit den gleichen 50-bp-Sequenzen und der anschließenden Integration ermöglichen. in die genomische DNA der Hefe an der Position, die durch die flankierenden Regionen bestimmt wird. Durch die Verwendung unterschiedlicher Promotorstärken wurden Stämme mit unterschiedlichen Carotinoiden-Produktionen erhalten, was zu einer Variation der Farbe der Zellen führte. Diese Stämme - inspiriert vom "Yeast Art Project"22 - wurden in einem Spotting-Setup mit einem akustischen Flüssigkeitshandler verwendet, um ein 4-farbiges hochauflösendes "Hefefoto" von Rosalind Franklin zu erstellen. Franklin (1920-1958) war eine englische Chemikerin und Röntgenkristallographin, die für ihren Beitrag zur Entdeckung der DNA-Struktur durch Photo 5123,24,25bekannt ist.
Protokoll
1. Herstellung der Cas12a-Plasmide
ANMERKUNG: Das Plasmid, das das Lachnospiraceae-Bakterium ND2006 Cas12a (LbCpf1, pCSN067) enthält, wurde zuvor 19 konstruiert, in einem Plasmid-Repository abgelagert (siehe Tabelle Materialien). Dies ist ein einkopiertes episomaler S. cerevisiae/E. coli Shuttle-Plasmid, das ein KanMX-Resistenzmarker-Gen enthält, um die S. cerevisiae-Transformationsmittel auf Geneticin (G418) zu selektionieren.
- Erhalten Sie das pCSN067-Plasmid (siehe Materialtabelle).
- Verstärken Sie das pCSN067-Plasmid, um eine hohe Menge zu erhalten.
- Transformieren Sie 25 l der gekauften chemisch kompetenten E. coli-Zellen mit dem Plasmid pCSN067 gemäß dem Herstellerprotokoll. Verdünnen Sie die Transformationsmischung 10 und 50 Mal in 2x Pepton-Hefe (PY). 10x und 50x Verdünnungen auf 2x PY Agarplatten mit Ampicillin (0,1 g/L) versiebenen und über Nacht bei 37 °C inkubieren.
- Wählen Sie 2 bis 3 Kolonien und impfen Sie jede Kolonie in 3 ml 2x PY und wachsen Sie über Nacht bei 37 °C in einem Schüttel-Inkubator bei 180 U/min.
- Reinigen Sie das Plasmid mit einem Plasmid-Reinigungskit gemäß den Anweisungen des Herstellers.
2. Vorbereitung der einzelnen crRNA-Array-Expressionskassette
- Bereiten Sie das einzelne crRNA-Array vor.
ANMERKUNG: Das einzelne crRNA-Array besteht aus einemSNR52-RNA-Polymerase-III-Promotor aus S. cerevisiae2 , einer direkten Wiederholung spezifisch für LbCas12a und einem Abstandsmesser (genomische Zielsequenz), die zusammen für jedes Ziel19 und mit einem SUP4-Terminator von S. cerevisiae2. Das einzelne crRNA-Array wird durch In-vivo-Rekombination in das linearisierte Plasmid pRN1120 zusammengesetzt, um ein kreisförmiges Plasmid zu erzeugen, daher müssen regionen homologe bis plasmid pRN1120 am Anfang und Ende des einzelnen crRNA-Arrays vorhanden sein (siehe Abbildung 2A ). Es wird empfohlen, die Funktionalität einer Reihe von entworfenen crRNAs separat zu bewerten19. Diese Informationen werden anschließend verwendet, um die meisten funktionalen crRNAs auszuwählen, um diese in den direkten Wiederholungs- und Abstandsreihen zu kombinieren, um ein einzelnes crRNA-Array für den Multiplexing-Zweck zu erstellen.- Bestellen Sie das einzelne crRNA-Array für Multiplex-Genom-Editing-Experimente als synthetische DNA (siehe die DNA-Sequenz des einzelnen crRNA-Arrays in Supplementary Table 1).
- Verstärken Sie das bestellte einzelne crRNA-Array(z.B.mit primer KC-101 und KC-102 (Supplemental Table 2)). Bereiten Sie den PCR-Amplifikationsmix vor, der folgende Gehalt an DNA-Polymerase, 10 l 5x Puffer für die DNA-Polymerase, 1 l mit 10 mM dNTPs, 2,5 l mit 10 -M Vorwärtsgrundierung, 2,5 l mit 10 -M Reverseprimer, 2 ,L DNA-Vorlage bei einer Konzentration von 5 ng/l und 2 O bis zu einem Gesamtvolumen von 50 l.
- Führen Sie die Reaktion in einem Thermocycler mit folgendem Programm durch: (i) 98 °C für 3 min, (ii) 98 °C für 10 s, (iii) 60 °C für 20 s, (iv) 72 °C für 15 s – Wiederholungsschritte (ii) bis (iv) 30 mal, (v) 72 °C für 5 min (vi) halten bei 12 °C bis zur weiteren Analyse.
- Analysieren Sie die PCR-Produkte durch Elektrophorese, indem Sie die Proben auf einem 0,8% Agarose-Gel bei 5 V/cm für 40 min mit einem DNA-Ladefarbstoff und einer DNA-Leiter mit DNA-Fragmenten im Bereich von 100 bis 10.000 bp ausführen.
- Reinigen Sie die PCR-Produkte mit einem PCR-Reinigungskit gemäß den Anweisungen des Herstellers.
- Bereiten Sie das einzelne crRNA-Array-Empfänger-Plasmid vor.
ANMERKUNG: Das einzelne crRNA-Array wird aus dem S. cerevisiae/E. coli Shuttle Plasmid pRN112019 (siehe Tabelle der Materialien) exprimiert. Dieses Multi-Copy-Plasmid enthält ein NatMX-Resistenzmarker-Gen, um die Auswahl von S. cerevisiae-Transformationsmitteln auf Nourseothricin (NTC) zu ermöglichen.- Erhalten Sie das pRN1120 Plasmid.
- Verstärken Sie das pRN1120 Plasmid, um eine hohe Menge zu erhalten.
- Transformieren Sie 25 l der gekauften chemisch kompetenten E. coli-Zellen mit Plasmid pRN1120 gemäß dem Herstellerprotokoll. Verdünnen Sie den Transformationsmix 10 und 50 Mal in 2x PY. 10x und 50x Verdünnungen auf 2x PY Agarplatten mit Ampicillin (0,1 g/L) versiebenen und über Nacht bei 37 °C inkubieren.
- Wählen Sie 2 bis 3 Kolonien und impfen Sie jede Kolonie in 3 ml 2x PY und wachsen Sie über Nacht bei 37 °C in einem Schüttel-Inkubator bei 180 U/min.
- Reinigen Sie das Plasmid mit einem Plasmid-Reinigungssatz gemäß den Anweisungen des Herstellers.
- Linearisieren Sie das Plasmid pRN1120 mit EcoRI-HF und XhoI. Bereiten Sie dazu eine Verdauungsmischung vor, die aus 1 g pRN1120, 5 l 10x Puffer (1x Puffer enthält 50 mM Kaliumacetat, 20 mM Trisacetat, 10 mM Magnesiumacetat, 100 g/mL Rinderserumalbumin [BSA]; pH 7,9), 1 ml EcoRI-HF (20 U) , 1 l XhoI (20 U) und Ultrapure H2O bis zu einem Gesamtvolumen von 50 l. Inkubieren Sie den Verdauungsmix bei 37 °C für 2 h und inaktivieren bei 65 °C für 20 min.
- Analysieren Sie das linearisierte Plasmid durch Elektrophorese auf einem Agarose-Gel (0,8%, 40 min, 5 V/cm) mit einem DNA-Ladefarbstoff und einer DNA-Leiter mit DNA-Fragmenten im Bereich von 100 bis 10.000 bp. Als Kontrolle ein kreisförmiges Plasmid in die Analyse einbeziehen.
- Reinigen Sie das linearisierte Plasmid mit einem PCR-Reinigungskit gemäß den Anweisungen des Herstellers.
3. Vorbereitung der Spender-DNA-Konstrukte des Promoter-ORF-Terminators (POT)
- Ordnen Sie einen Satz von Promotor (P) mit unterschiedlicher Stärke, offenem Leserahmen (O) und Terminator (T) Sequenzen als synthetische DNA so an, dass jedes Element standardisierte 4-bp-Erkennungssequenzen enthält, die von BsaI-Sites flankiert werden, um Golden Gate Cloning ( zu ermöglichen ( GGC) Baugruppe26 (siehe die detaillierten Entwürfe in Ergänzender Tabelle 3 und Sequenzen in Ergänzender Tabelle 4).
- Montieren Sie POT-Expressionskassetten, die aus einem Promotor, einem offenen Leserahmen, einem Terminator und Anschlüssen bestehen, über eine 4-teilige Baugruppe mithilfe einer GGC-Reaktion21in einen Zielvektor, der bereits vorgegebene 50-bp-Steckverbindersequenzen enthält ( siehe Ergänzende Tabelle 4 und Referenzen26,27).
- Messen Sie die Konzentration von DNA-Teilen mit einem Spektralphotometer. Verdünnen Sie jedes DNA-Teil in reinemH2O auf eine Endkonzentration von 15 fmol/l.
- Bereiten Sie einen Reaktionsmix vor, der aus DNA-Fragmenten besteht: 2 l Promotor, 2 l offener Leserahmen, 2 l Terminator und 2 L-Rückgrat (Level 1-Zielvektoren wie in 26beschrieben), 4 l 5x T4-DNA-Ligase-Puffer, 2,5 l von 1 U/L T4-DNA-Ligase , 1,5 l mit 20 U/L BsaI-HF und reinemH2O bis zu einem Gesamtvolumen von 20 l.
- Führen Sie die GGC-Reaktion in einem Thermocycler mit folgendem Programm durch: (i) 37 °C für 2 min, (ii) 16 °C für 5 min – Wiederholungsschritte (i) und (ii) 50-mal, (iii) 50 °C für 60 min, (iv) 80 °C für 45 min, (v) bis zur weiteren Analyse bei 12 °C halten.
- Transformieren Sie 25 l der gekauften chemisch kompetenten E. coli28-Zellen mit 3 l ggC-Reaktionsmix gemäß Herstellerprotokoll. Verdünnen Sie den Transformationsmix 10 und 50 Mal in 2x PY. 10x und 50x Verdünnungen auf 2x PY Agarplatten mit Ampicillin (0,1 g/L) versiebenen und über Nacht bei 37 °C inkubieren.
- Wählen Sie 2 bis 3 Kolonien und impfen Sie jede Kolonie in 3 ml 2x PY und wachsen Sie über Nacht bei 37 °C in einem Schüttel-Inkubator bei 180 U/min.
- Reinigen Sie die Plasmide mit einem Plasmid-Reinigungskit gemäß den Anweisungen des Herstellers.
- Prüfen Sie, ob POT-Expressionskassetten in der GGC-Reaktion per PCR korrekt montiert wurden.
- Designprimer, die die Zusendesequenz am Anfang und am Ende jeder Ausdruckskassette ergänzen (siehe Abbildung 2B). Verwenden Sie für in diesem Protokoll ausgewählte Anschlüsse die Primer KC-103 bis KC-108 (siehe Zusatztabelle 2).
- Bereiten Sie PCR-Amplifikationsmischungen für jedes Plasmid vor, das folgende Werte enthält: 0,5 l Korrektur-DNA-Polymerase, 10 l 5x Puffer für die DNA-Polymerase, 1 l mit 10 mM dNTPs, 2,5 l von 10 'M Vorwärtsprimer, 2,5 'L von 10 'M Reverse Primer, 2 'L DNA-Vorlage mit einem Konzentrat und ultrareines H2Obis zu einem Gesamtvolumen von 50 l.
- Führen Sie die PCR-Reaktion in einem Thermocycler mit folgendem Programm durch: (i) 98 °C 3 min, (ii) 98 °C für 10 s, (iii) 60 °C für 20 s, (iv) 72 °C für 2 min 30 s – Wiederholungsschritte (ii) bis (iv) 30 mal, (v) 72 °C für 5 min , (vi) bis zur weiteren Analyse bei 12 °C halten.
HINWEIS: Die resultierenden PCR-Produkte bestehen aus 50-bp des 5-Bp-Steckverbinders, des Promoters, des offenen Leserahmens, des Terminators und 50-bp des 3-K-Steckers.
- Analysieren Sie die PCR-Produkte durch Elektrophorese, indem Sie Proben auf einem 0,8% Agarose-Gel bei 5 V/cm für 40 min mit einem DNA-Ladefarbstoff und einer DNA-Leiter mit DNA-Fragmenten im Bereich von 100 bis 10.000 bp ausführen.
4. Erstellung von Integrationsflanken-DNA-Sequenzen mit Konnektorensequenzen
- Reinigen Sie genomische DNA vom wilden Typ S. cerevisiae CEN.PK113-7D29.
- Wachsen Sie die Sorte in einem 500 ml Schüttelkolben gefüllt mit 100 ml Hefeextrakt Peptondextrose (YEPD, 2% Glukose) Medium bei 30 °C und schütteln bei 250 U/min für 48 Stunden.
- Ernten Sie die Zellen durch Zentrifugation von 2 ml Brühe bei 16.000 x g für 1 min und entsorgen Sie den Überstand.
- Setzen Sie die Zellen in physiologischem Salz (200 l; 0,85 % NaCl-Lösung) mit RNase (10 l, 10 mg/ml) und Hefelytusenzym (4 l) wieder auf. Inkubieren Sie die Zellsuspension bei 37 °C für 15 min.
- Fügen Sie 300 L Zelllyselösung (siehe Materialtabelle)und Wirbel in Kürze hinzu.
- Fügen Sie 168 L Protein-Fälllösung (siehe Materialtabelle)und Wirbel kräftig für 20 s.
- Trennen Sie die Proteinfraktion durch Zentrifugation bei 16.000 x g und 4 °C für 10 min. Sammeln Sie 600 l Überstand in einem neuen Röhrchen und mischen Sie in Kürze 600 l Isopropanol und Wirbel.
- Dna wiederherstellen, indem Sie bei 16.000 x g bei Raumtemperatur 10 min. den Überstand entsorgen und das Pellet behalten.
- Waschen Sie das Pellet mit 200 l Ethanol (70%). Zentrifuge bei 16.000 x g bei Raumtemperatur für 10 min und entfernen Sie den Überstand. Verdampfen Sie das Ethanol, indem Sie das Rohr bei Raumtemperatur für 10 min mit geöffnetem Deckel inkubieren.
HINWEIS: Wenn Flüssigkeit im Rohr noch sichtbar ist, wiederholen Sie den Schritt 4.1.8. Trocknen Sie das Pellet nicht länger als 10 min, um eine verminderte Löslichkeit der DNA zu verhindern. - Lösen Sie die DNA in 50 L TE-Puffer auf. gereinigte DNA bei 4 °C aufbewahren.
- Design-Integrations-Flanken-DNA-Sequenzen (ca. 500 bp) werden für jede Integrationsstelle so gestaltet, dass bei Einführung der Spender-DNA ca. 1000 bp genomische DNA entfernt werden (siehe Schaltplan in Abbildung 2B und Sequenzen in Supplementary Tabelle 4).
- Design-Primer, um die flankierenden Bereiche durch PCR zu generieren.
- Für den linken flankierenden Bereichentwerfen Sie Vorwärts- und Rückwärtsgrundierungen, um etwa 500 bp der genomischen DNA-Region zu verstärken, die 5' (links) der Integrationsstelle von Interesse positioniert ist.
HINWEIS: Die Vorwärtsgrundierung enthält 20 bp Homologie mit dem beabsichtigten flankierenden Bereich. Der Reverse Primer enthält 20 bp mit Homologie mit dem vorgesehenen flankierenden Bereich und enthält die gewünschte 50-bp-Steckverbindersequenz, um später die In-vivo-Montage in der Cas12a-Bearbeitung am Genom zu ermöglichen. - Für den rechten flankierenden Bereichentwerfen Sie Vorwärts- und Rückwärtsgrundierungen, um etwa 500 bp der genomischen DNA-Region zu verstärken, die 3' (rechts) der Integrationsstelle von Interesse positioniert ist.
HINWEIS: Der Vorwärtsprimer enthält 20 bp mit Homologie mit dem vorgesehenen flankierenden Bereich und enthält die gewünschte 50-bp-Steckverbindersequenz, um die In-vivo-Montage in der Cas12a-Bearbeitung des Genoms später zu ermöglichen. Die umgekehrte Grundierung umfasst 20 bp Homologie mit dem beabsichtigten flankierenden Bereich.
- Für den linken flankierenden Bereichentwerfen Sie Vorwärts- und Rückwärtsgrundierungen, um etwa 500 bp der genomischen DNA-Region zu verstärken, die 5' (links) der Integrationsstelle von Interesse positioniert ist.
- Verstärken Sie die flankierenden Bereiche mit den entworfenen Primern(z.B.Primer KC-109 bis KC-120, die in Zusatztabelle 2eingeschlossen sind).
- Messen Sie die Konzentration der gereinigten genomischen DNA, die als Vorlage in der PCR dienen wird. Stellen Sie die DNA-Konzentration auf 50 ng/L ein.
- Herstellung von PCR-Amplifikationsmischungen, die aus genomischer DNA bestehen (1 – 4 l mit 50 ng/l genomischer DNA-Verdünnung), gereinigt in Schritt 4.1, Vorwärts- und Rückwärtsgrundierung (je 10 M), 1 l von 10 mM dNTPs, 10 l 5x Puffer für die DNA-Polymerase, 0,5 l DNA-Polymerase (1,0 U) und ultrareine H2O bis zu einem Gesamtvolumen von 50 l.
- PCRs in einem Thermocycler mit folgendem Programm durchführen: (i) 98 °C für 3 min, (ii) 98 °C für 20 s, (iii) 60 °C für 20 s, (iv) 72 °C für 15 s, Wiederholungsschritte (ii) bis (iv) 30 mal, (v) 72 °C für 5 min , (vi) bis zur weiteren Analyse bei 12 °C halten.
- Analysieren Sie die PCR-Produkte durch Elektrophorese auf einem 0,8% Agarose-Gel bei 5 V/cm für 40 min mit einem DNA-Ladefarbstoff und einer DNA-Leiter mit DNA-Fragmenten im Bereich von 100 bis 10.000 bp.
- Reinigen Sie die richtigen PCR-Produkte mit einem PCR-Reinigungskit gemäß den Anweisungen des Herstellers.
5. Umwandlung nach S. cerevisiae
HINWEIS: Führen Sie die Transformation mit einem Protokoll auf der Grundlage der von Gietz et al. entwickelten Methoden durch. 1995: 30 und Hill et al. 31 die für verschiedene Stämme von S. cerevisiaeverwendet werden können. Das unten beschriebene Protokoll ist für eine Transformation ausreichend.
- Bereiten Sie Lösungen vor, die für die Transformation erforderlich sind.
- Bereiten Sie folgende Lagerlösungen und Filtersterilisieren vor: 10x TE Puffer mit 100 mM Tris-HCl (pH 7.5), 10 mM EDTA, Gesamtvolumen von 50 ml; 1 M LiAc bei pH 7,5, Gesamtvolumen von 50 ml; 50% PEG 4000, Gesamtvolumen von 100 ml.
HINWEIS: Überprüfen Sie immer, ob der PEG 4000-Bestand bei pH 5 liegt. Dieser Bestand sollte nicht länger als einen Monat gelagert werden. - Bereiten Sie die folgenden Lösungen mit Vorräten vor: Bereiten Sie die LiAc-TE-Lösung mit 0,1 M LiAc, 10 mM Tris-HCl, 1 mM EDTA, Gesamtvolumen von 0,5 ml vor. Bereiten Sie PEG-LiAc-TE Lösung mit 40% PEG 4000, 0,1 M LiAc, 10 mM Tris-HCl, 1 mM EDTA, Gesamtvolumen von 1 ml vor.
HINWEIS: Für eine erfolgreiche Transformation ist es entscheidend, dass DIE Lösungen von PEG-LiAc-TE und LiAc-TE frisch vorbereitet werden.
- Bereiten Sie folgende Lagerlösungen und Filtersterilisieren vor: 10x TE Puffer mit 100 mM Tris-HCl (pH 7.5), 10 mM EDTA, Gesamtvolumen von 50 ml; 1 M LiAc bei pH 7,5, Gesamtvolumen von 50 ml; 50% PEG 4000, Gesamtvolumen von 100 ml.
- Erste Transformationsrunde (bereiten Sie die Spannung vor dem Ausdrücken von Cas12a vor).
HINWEIS: Verwenden Sie in allen Transformationsschritten Wasser mit einem pH-Wert von mehr als 5. Es wird empfohlen, demineralisiertes Wasser in allen Schritten der Transformation zu verwenden.- Bereiten Sie eine Vorkultur durch wachsende Belastung CEN. PK113-7D in einem 100 ml Schüttel-Shakekolben mit 20 ml YEPD-Medium (2% Glukose) und über Nacht bei 30 °C mit Schütteln bei 250 U/min inkubieren.
- Messen Sie die OD600 der Vorkultur (ODpc). Berechnen Sie den Verdünnungsfaktor (df) zwischen dem Volumen der Vorkultur und dem Volumen des frischen Mediums, das für die Vorbereitung der Zellen erforderlich ist, die Cas12a vorexmitenieren, um in der Transformation verwendet zu werden (Transformationskultur). In den Berechnungen wird davon ausgegangen, dass die optische Dichte der Transformationskultur (ODtc) nach dem in 5.2.3 (ti) beschriebenen Inkubationsschritt 1,0 betragen wird.

wobei ti und n die Inkubationszeit bzw. die Verdoppelungszeit sind.- Berechnen Sie das Volumen der Vorkultur (Vi) die für die Impfung der Transformationskultur (Vtc) erforderlich ist, basierend auf dem Verdünnungsfaktor.
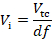
- Berechnen Sie das Volumen der Vorkultur (Vi) die für die Impfung der Transformationskultur (Vtc) erforderlich ist, basierend auf dem Verdünnungsfaktor.
- Bereiten Sie die Transformationskultur durch Impfung von 20 ml YEPD (2% Glukose) (Vtc) mit dem im vorherigen Schritt festgelegten Vorkulturvolumen vor (Vi). Bei 30 °C mit Schütteln bei 250 Umdrehungen bei 250 Umdrehungen/min bebrüten.
- Messen Sie die OD600 der Transformationskultur, bis eine OD600 von 1,0 erreicht ist.
- Ernte der Zellen durch Zentrifugation der 20 ml Brühe bei 2.500 x g für 5 min. Entsorgen Sie den Überstand und waschen Sie die Zellen in 20 ml Raumtemperatur demineralisiertes Wasser. Wiederholen Sie den Zentrifugationsschritt und bewahren Sie das Zellpellet auf.
- Setzen Sie die Zellen in 100 l LiAc-TE-Lösung wieder aus und übertragen Sie sie in ein Mikrozentrifugenrohr.
- Fügen Sie 5 l einsträngige Träger-DNA (10 mg/ml Lachssperma-DNA) hinzu und mischen Sie sie durch Pipettieren.
- Pipette 1 g Plasmid pCSN067 in das Mikrozentrifugenrohr.
HINWEIS: Das Gesamtvolumen des DNA-Gemischs sollte 100 l nicht überschreiten, um eine geringere Transformationseffizienz zu verhindern. - Fügen Sie 600 L PEG-LiAc-TE Lösung und mischen durch Pipettieren. 30 min bei 30 °C inkubieren, bei 450 Umdrehungen pro Minute in einem Tischwärmer blockieren.
- 70 L DMSO hinzufügen (100%) Transformationsmischung und Durchmischung durch Pipettieren. Führen Sie einen Hitzeschock durch Inkubieren der Transformationsmischung bei 42 °C für 15 Minuten in einem Wasserbad durch.
- Stellen Sie die Zellen wieder her, indem Sie das Gemisch in ein 15 ml rundes Unterrohr übertragen und 10 ml YEPD (2% Glukose) in das Rohr geben. Über Nacht bei 30 °C mit Schütteln bei 250 Umdrehungen von 150 Rpm inkubieren.
- Zentrifugieren Sie den Transformationsmix bei 2.500 x g für 5 min. Entsorgen Sie den Überstand und setzen Sie das Zellpellet in ca. 200 l der verbleibenden Lösung wieder auf.
- Plate out 150 l des Transformationsmixes und eine 20-fache Verdünnung in YEPD (2% Glukose) des Transformationsmixes auf YEPD (2% Glukose) Agarplatten, ergänzt mit 0,2 g/L G418. Die Platten 48 – 72 Stunden bei 30 °C bebrüten.
- Wählen Sie eine einzelne transformierte und re-streak auf einem YEPD (2% Glukose) Agar-Platte mit 0,2 g/L G418 ergänzt, um einzelne Kolonien zu erhalten.
- Zweite Transformationsrunde (Multiplex-Genombearbeitung mit CRISPR/Cas12a durchführen).
- Bereiten Sie eine Vorkultur vor, indem Sie den Stamm vor dem Ausdruck von Cas12a, der in der ersten Transformationsrunde (Schritt 5.2) erstellt wurde, in einem 100 ml-Shakekolben mit 20 ml YEPD (2% Glukose) Medium mit 0,2 g/L G418 ergänzen. Über Nacht bei 30 °C mit Schütteln 250 Rpm inkubieren.
HINWEIS: Passen Sie bei mehreren Transformationen das Volumen der Vorkultur an. - Befolgen Sie die Schritte 5.2.2 bis 5.2.7 für die erste Transformationsrunde.
HINWEIS: Passen Sie für mehrere Transformationen die Volumina der erforderlichen Lösungen und die Kultur der Dehnung vor dem Ausdruck von Cas12a an. - Pipette 1 g des einzelnen crRNA-Arrays, 1 g des linearisierten Empfängerplasmids für das crRNA-Array, 1 g Spender-DNA und 1 g jeder flankierenden Region (Schritt 4.3) im Mikrozentrifugenrohr.
HINWEIS: Das Gesamtvolumen des DNA-Gemischs sollte 100 l nicht überschreiten, um eine geringere Transformationseffizienz zu verhindern. - Bereiten Sie die folgenden Steuerelemente für die Transformation vor: Negativkontrolle (Ultrapure H2O); Positive Kontrolle zur Bestimmung der Transformationseffizienz (1 g kreisförmig pRN1120); eine Kontrolle, ob die Einführung der Spender-DNA über DIE CRISPR-Bearbeitung erfolgt (1 g zirkuläre pRN1120, 1 g aller Spender-DNA-Expressionskassetten und 1 g flankierende Regionen, aber kein einziges crRNA-Array); Kontrolle, ob Spender-DNA außerhalb des Ziels integriert werden kann (1 g linearisierte pRN1120, 1 g Spender-DNA-Expressionskassetten und 1 g des einzelnen crRNA-Arrays, aber keine flankierenden Regionen); ein Steuerelement zur Überprüfung der vollständigen Linearisierung von pRN1120 (1 g linearisierte pRN1120).
- Befolgen Sie die Schritte 5.2.9 bis 5.2.12 für die erste Transformationsrunde.
- Platte aus 150 l des Transformationsmixes und 20x Verdünnung in YEPD (2% Glukose) des Transformationsmixes auf YEPD (2% Glukose) Agar, ergänzt mit 0,2 g/L G418 und 0,2 g/L NTC. Platten-Out-Kontrollen auf YEPD (2% Glukose) Agar mit der entsprechenden Auswahl ergänzt (G418 und/oder NTC oder keine Auswahl). Die Platten 48 – 72 Stunden bei 30 °C bebrüten.
- Wählen Sie ein einfarbiges Transformant und re-streak auf einer YEPD (2% Glukose) Agarplatte, um einzelne farbige Kolonien zu erhalten.
- Bereiten Sie eine Vorkultur vor, indem Sie den Stamm vor dem Ausdruck von Cas12a, der in der ersten Transformationsrunde (Schritt 5.2) erstellt wurde, in einem 100 ml-Shakekolben mit 20 ml YEPD (2% Glukose) Medium mit 0,2 g/L G418 ergänzen. Über Nacht bei 30 °C mit Schütteln 250 Rpm inkubieren.
6. Bewertung der Genom-Editing-Effizienz
- Zählen Sie die Anzahl der farbigen Kolonien und weißen Kolonien auf den Transformationsplatten.
- Berechnen Sie die Effizienz der Genombearbeitung, indem Sie die Anzahl der farbigen Kolonien durch die Gesamtzahl der Kolonien (sowohl weiß als auch farbig) dividieren, wie in Tabelle 1dargestellt.
7. Bestätigung der Integration der Spender-DNA am vorgesehenen Ort
- Eine farbige Einzelkolonie von einer Transformationsplatte auf einer YEPD (2% Glukose) Agarplatte ohne G418- und NTC-Auswahl neu streichen und 48 Stunden lang bei 30 °C brüten.
- Wählen Sie eine einzelne Kolonie und impfen Sie einen 500 ml Schüttelkolben, der mit 100 ml YEPD (2% Glukose) Medium gefüllt ist. 48 Stunden bei 30 °C inkubieren und bei 250 Umdrehungen pro Minute schütteln.
- Isolieren Sie die genomische DNA, wie in Abschnitt 4.1 beschrieben.
HINWEIS: Alternativ verwenden Sie ein Protokoll zur Vorbereitung von Hefe für Kolonie PCR zuvor von Looke et alvorgeschlagen . 32. In diesem Fall kann das Wachstum des flüssigen Mediums (Abschnitt 7.2) übersprungen werden. - Überprüfen Sie die korrekte Integration durch Verstärkung von zwei Fragmenten pro integrierter Ausdruckskassette.
- Entwerfen Sie Primer, die außerhalb der transformierten flankierenden Regionen und des Gens von Interesse zu genomischer DNA führen (siehe Beispiele in Ergänzender Tabelle 2, KC-121 bis KC-132). Wenn Sie die Primer KC-121 auf KC-132 verwenden, stellen Sie die Glühtemperatur im PCR-Programm auf 62 °C ein.
- Verstärkung der Interessenregion, wie in Abschnitt 4.4.2 beschrieben. Passen Sie das PCR-Programm an, passen Sie die Zeit des Verlängerungsschritts in PCR speziell an die Länge der Schablone und die Herstellerempfehlungen für die DNA-Polymerase an.
- Überprüfen Sie die Größe der PCR-Produkte durch Elektrophorese auf einem Agarosegel (0,8 %, 40 min, 5 V/cm) mit einem DNA-Ladefarbstoff und einer DNA-Leiter mit DNA-Fragmenten im Bereich von 100 bis 10.000 bp.
8. Erstellung von Hefe-Pixel-Kunst mit einem akustischen Flüssigkeitshandler
- Bereiten Sie eine Bildvorlage für die Hefepixelgrafik vor.
- Ändern Sie die Größe des ursprünglichen RGB-Bildes (220 x 280 Pixel, siehe die repräsentativen Ergebnisse), z. B. mithilfe von ImageJ, um ein endgültiges Graubild mit 64 x 96 Pixeln (Breite x Höhe) zu erstellen, das in den gewünschten Farben visualisiert wird (repräsentative Ergebnisse).
- Konvertieren Sie das RGB-Bild mit dieser Formel in Graustufen:

wobei ichgr, Ir, Ig, Ib sind die grauen, roten, grünen und blauen Intensitäten, beziehungsweise. - Um die Pixel zu kategorisieren, entwickeln Sie ein ImageJ-Plugin, das die folgenden Regeln anwendet: (a) Wenn ichgr 64 ist, verwenden Sie die dunkelorange Hefe (Stamm 1, Zusatztabelle 3) für dieses Pixel. b) Wenn 64 < Igr 128, verwenden Sie die orange Hefe (Stamm 2, Zusatztabelle 3) für dieses Pixel. (c) Verwenden Sie für dieses Pixel die gelbe Hefe (Stamm 3, Zusatztabelle 3). (d) Wenn ichgr > 192, verwenden Sie die weiße Hefe (CEN. PK113-7D) für dieses Pixel.
- Erkennen Sie Hefezellen, um die Hefepixel-Grafik zu erstellen.
- 500 ml Schüttelkolben mit 100 ml YEPD (2% Glukose) Medium mit drei verschieden farbigen Carotinoiden, die S. cerevisiae Stamm und wildes Typ CEN produzieren. PK113-7D. Inkubieren Sie Kulturen über Nacht bei 30 °C mit Schütteln bei 250 Rpm.
- Übertragen Sie 0,5 ml der Nachtkultur auf ein Rohr, das mit 0,5 ml sterilem nichtionischen Dichtegradientenmedium gefüllt ist (siehe Materialtabelle). Mischen Sie durch Kurzwirbeln.
- Übertragen Sie die Zellsuspension in ein qualifiziertes Reservoir, 2 x 3 gut. Führen Sie Spotting mit einem akustischen Flüssigkeitshandler-Instrument von einer qualifizierten Reservoir-Quellplatte zu einer Mikroplatte (siehe Materialtabelle) mit 50 ml YEPD (2% Glukose) Agar. Um die Beschichtung zu vereinfachen, definieren Sie Bohrungen auf der Platte, z.B.. eine Mikroplatte als 6144-Wellplatte (64 x 96) verwenden.
- Spot 25 nL jeder S. cerevisiae Stamm von der 2x 3 Well Reservoir Quellplatte mit einer .csv Datei mit der Flüssigkeitskalibrierung Einstellung 6RES_AQ_GPSA2 auf die Ziel-Mikroplatte. Definieren Sie jedes dieser 25 nL Tröpfchen als Pixel im 64 x 96 Raster, das in die Brunnenpositionen (A01, B01, C01 usw.) übersetzt wird.
- Inkubieren Sie die Mikroplatte 48 Stunden lang bei 30 °C. Um die Farben der Stämme zu intensivieren, lagern Sie die Agarplatte bei 4 °C für mindestens 72 Stunden.
Ergebnisse
Das Protokoll zur Multiplex-Genombearbeitung mit CRISRP/Cas12a wurde durch die Konstruktion von drei Carotinoid-Produktionsstämmen von S. cerevisiae demonstriert, die die Gene crtE, crtYB und crtI mit hilfe heterologen Promotoren von hohe, mittlere und niedrige Festigkeit: Dehnung 1, 2 bzw. 3 (Zusatztabelle 3). Der Aufbau dieser Stämme erforderte die Erzeugung von drei Spender-DNA-Expressionskassetten und sechs flankierenden Regionen pro Stamm für das Targeting auf drei verschiedene Loci in der genomischen DNA (siehe Abbildung 2B). Wie hierbeschrieben, wurden Promotor, offener Leserahmen, Terminator und zwei zusammenhängende 50-bp-Steckverbindersequenzen über eine Golden Gate Cloning-Reaktion zu einer Ausdruckskassette zusammengebaut und die Baugruppe durch PCR überprüft (Abbildung 3A). Das einzelne crRNA-Array wurde als synthetisches DNA-Fragment geordnet und durch PCR verstärkt (Abbildung 3B). Das Empfängerplasmid für das einzelne crRNA-Array (Plasmid pRN1120) wurde mit EcoRI-HF und XhoI linearisiert und die Linearisierung wurde durch Elektrophorese bestätigt (Abbildung 3C). Die Design- und Nukleotidsequenzen der eingeführten Spender-DNA-Expressionskassetten und flankierenden Bereiche sind in der ergänzenden Tabelle 3 und der Ergänzenden Tabelle 4dargestellt. Die Sequenz einzelner crRNA-Arrayexpressionskassetten ist in Der Ergänzenden Tabelle 1angegeben. Die Funktionalität der im einzelnen crRNA-Array enthaltenen Abstandshalter wurde zuvor durch Singleplex-Genombearbeitung mit einzelnen crRNAs19getestet.
Die Effizienz der Genombearbeitung mit Cas12a wurde zunächst anhand der Anzahl der farbigen Kolonien bewertet, die nach der Transformation erhalten wurden (Tabelle 1, Abbildung 4). Die Bearbeitungseffizienz der drei konstruierten Stämme schwankte zwischen 50 % und 94 %. Insbesondere die Einführung von Expressionskassetten, die zur Erzeugung von Stamm 1 verwendet werden, zeigte die geringste Bearbeitungseffizienz, die möglicherweise durch die Art der Spender-DNA verursacht wurde(d. h.diese Expressionskassetten kodieren crtE, crtYB und crtI von drei hochfesten Promotoren). Zweitens wurde die korrekte Integration der drei Spender-DNA-Expressionskassetten an der vorgesehenen Loci in die genomische DNA durch PCR bestätigt (Abbildung 5). Primer wurden so konzipiert, dass PCR-Produkte erhalten wurden, wenn die korrekte Integration der Spender-DNA am vorgesehenen Ort erfolgte. Für jedes Transformationsexperiment wurden acht Kolonien aus der Transformationsplatte entnommen und getestet (beachten Sie, dass nur drei in Abbildung 5dargestellt sind). Im Allgemeinen wurde von 8 Kolonien, die pro Spender-DNA getestet wurden, die korrekte Integration der crtE-Spender-DNA am INT1-Heuschreck, crtYB am INT2-Lokus und crtI am INT3-Lokus in >90% der Transformanten bestätigt. Diese Ergebnisse zeigen das CRISPR/Cas12a-System in Kombination mit einem einzigen crRNA-Array, das eine effiziente Multiplex-Bearbeitung des S. cerevisiae Genoms an mehreren Loci gleichzeitig ermöglicht.
Darüber hinaus zeigen wir die Erstellung von "Hefe-Pixel-Kunst" mit den drei Carotinoid-produzierenden Stämmen, die zusammen mit einer nicht farbigen Wild-Typ-Sorte konstruiert wurden. Ausgehend von einem Schwarz-Weiß-Bild von Rosalind Franklin (Abbildung 6A) wurde ein 4-Farben-Bild (Abbildung 6B) und eine Spotting-Liste erstellt, die dann verwendet wurde, um die vier verschiedenen Hefestämme auf einer Agar-Mikroplatte mit einem akustischen Flüssigkeitshandler, was zu einer hochauflösenden "Hefemalerei" von Rosalind Franklin führt (Abbildung 6C,D,E).

Abbildung 1 : Workflow des Protokolls für CRISPR/Cas12a Multiplex-Genombearbeitung in S. cerevisiae. Der Workflow enthält entscheidende Schritte der vorgestellten Methode. Weitere Informationen finden Sie im Protokoll. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2 : Schema der CRISPR/Cas12a Multiplex-Genombearbeitung mit einem einzigen crRNA-Array. (A) Das einzelne crRNA-Array besteht aus drei crRNAs-Einheiten in ihrer ausgereiften Form, einer 20-bp-Direktwiederholung spezifisch für LbCas12a (graue Quadrate) mit einer 23-bp-Führungssequenz (farbige Diamanten). Die Expression des crRNA-Arrays wird durch den SNR52-Promotor und den SUP4-Terminator aktiviert. Die Transformation von S. cerevisiae mit einem linearisierten pRN1120 und der einzelnen crRNA-Arrayexpressionskassette, die Homologie mit pRN1120 (diagonale Streifen) enthält, ermöglicht eine In-vivo-Rekombination in ein kreisförmiges Plasmid in Zellen, das LbCas12a. Das einzelne crRNA-Array wird anschließend von Cas12a verarbeitet. (B) Cas12a ist auf die vorgesehenen genomischen Zielstellen INT1, INT2 und INT3 gerichtet und erzeugt doppelsträngige Brüche. In der Transformationsmischung wurden Spender-DNA, bestehend aus flankierenden Regionen und der Carotinoid-Genexpressionskassette, aufgenommen. Spender-DNA-Assemblys wurden auf einen DNA-Abschnitt in der genomischen DNA um die INT1 (crtE), INT2 (crtYB) und INT3 (crtI) loci durch In-vivo-Rekombination aufgrund des Vorhandenseins von 50-bp homologen Konnektorensequenzen, angegeben als 5, A, B, C, D oder E. P1-P3, verschiedene Promotoren; T1–T3, verschiedene Terminatoren. Diese Zahl wurde von Verwaal et al. 201819geändert. Genetische Konstrukte, die mit Synthetischen Biologie Open Language (SBOL) Visuelle Symbole40gezeigt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
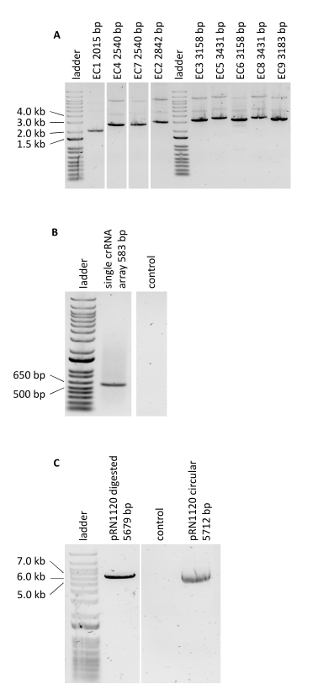
Abbildung 3 : PCR-Überprüfung der Genom-Editing-Experimente. (A) Überprüfung der Golden Gate Cloning Reaktionen von montierten Spender-DNA-Kassetten. Die erzielten Ergebnisse sind mit den erwarteten Längen übereinstimmend. (B) PCR des einzelnen crRNA-Arrays. (C) Linearisierung des Plasmids pRN1120. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4 : Platten von S. cerevisiae Transformationen mit dem Multiplex-Genom-Editing-Ansatz. (A) Stamm 1, der crtE, crtYB und crtI von drei starken Promotoren (dunkelorange Kolonien) ausdrückt. (B) Stamm 2, der crtE, crtYB und crtI aus drei mittelstarken Promotoren (orange Kolonien) ausdrückt. (C) Stamm 3, der crtE, crtYB und crtI aus drei niederfesten Promotoren (gelbe Kolonien) ausdrückt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5 : PCR-Überprüfung der Integration der Spender-DNA-Expressionskassetten an der vorgesehenen Loci in die genomische DNA. (A) Überprüfung von drei Kolonien des Stammes 1. (B) Überprüfung von drei Kolonien des Stammes 2. (C) Überprüfung von drei Kolonien des Stammes 3. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6 : Hefe-Pixel-Kunst von Rosalind Franklin. (A) Schwarz-Weiß RGB-Foto von 220 x 280 Pixel von Rosalind Franklin, die als Vorlage verwendet wurde. (B) Computerkonvertierung des Schwarz-Weiß-Fotos von Rosalind Franklin in eine 4-Farben-Liste mit 64 x 96 Pixeln. (C) Foto von Hefe-Pixel-Kunst mit 64 x 96 Hefekolonien mit einem vergrößerten Abschnitt. (D) Foto eines akustischen Flüssigkeitshandlers mit zwei ausgewachsenen Platten. (E) Foto einer ausgewachsenen Mikroplatte mit 64 x 96 Hefekolonien. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| last | Dehnung 2 | Dehnung 3 | |
| Farbige Kolonien | 16 | 279 | 220 |
| Weiße Kolonien | 16 | 18 | 18 |
| Kolonien insgesamt | 32 | 297 | 238 |
| fähigkeit | 50% | 94% | 92% |
Tabelle 1: Bearbeitungseffizienz des Multiplex-Genom-Editing-Ansatzes.
| crRNA-Arraysequenza,b,c,d,e,f |
|
CATGTTTGACAGCTTATCATCGATAATCCGGAGCTAGCATGCGGCCGCTCTAGAACTAGTGGATCCCCCGGGCTGCAG TCTTTGAAAA GATAATGTATGATTATGCTTTCACTATTTATTTATTTATACAGAAACTTGATGTTTTCTTTCTATTATATACAAGG TGATTACATGTACGTTTGAAGTACAACTCTAGATTTTGTAGTGCCCTCTTGGGCTAGCGGTAAAGGTGCGCA TTTTTTCACACCCTACAATGTGTTCAAAAAGATTTTGGTCAAACGCTGTAGAAGTGAAAGTTGGTGCGC ATGTTTCGGCGGAAACTTCTCCGCAGTGAAAGATAAATGATCAATTTCTACTAAGTGTAGAT CTGGTGGGAGAGAAAGCTTATGAAATTTCTACTAAGTGTAGATGTGCCGTAC GCCGGAGCCGACGGAATTTCTACTAAGTGTAGATTGCCCCTCTTATACGATTATTTT TTTTTTTTTTTTATGTCTGGGGGGCCCGGTACCCAGCTTTTGTTCCCTTTAGTGAGG GTTAATTCCGAGCTTGGCGTAATCATGGTCATAGCTGTCTCTGTGTG |
| a. Homologie zu pRN1120 (fett). b. SNR52-Promotor (italics). c) Genomische Zielsequenzen (unterstrichen). d. Leitlinie direkte Wiederholungen spezifisch für LbCas12a (italics, fett). e. SUP4-Terminator (italics). f. Homologie zu pRN1120 (fett). |
Ergänzende Tabelle 1: Einzelnes crRNA-Array für LbCas12a mit Homologie mit Plasmid pRN1120.
| name | Sequenza | Beschreibungb | Verwendet in Punkt |
| KC-101 | CATGTTTGACAGCTTATCATC | FW-Primer zur Verstärkung einzelner crRNA-Arrays | 2.1.4 |
| KC-102 | CACACAGGAAACAGCTATGAC | RV-Primer zur Verstärkung einzelner crRNA-Arrays | 2.1.4 |
| KC-103 | AAGCGACTTCCAATCGCTTTGC | FW-Grundierung zur Verstärkung der Spender-DNA mit Stecker 5 | 3.6.1 |
| KC-104 | AAAGCAAAGGAAGGAGAGAAC | RV-Grundierung zur Verstärkung der Spender-DNA mit Stecker A | 3.6.1 |
| KC-105 | CGGATCGATGTACACAACCG | FW-Grundierung zur Verstärkung der Spender-DNA mit Stecker B | 3.6.1 |
| KC-106 | CAACAGGAGGCGGATGGATATAC | RV-Grundierung zur Verstärkung der Spender-DNA mit Stecker C | 3.6.1 |
| KC-107 | AACGTTGTCCAGGTTTGTATCC | FW-Grundierung zur Verstärkung der Spender-DNA mit Stecker D | 3.6.1 |
| KC-108 | AGGTACAACAAGCACGACCG | RV-Grundierung zur Verstärkung der Spender-DNA mit Stecker E | 3.6.1 |
| KC-109 | CACTATAGCAATCTCTCTATATG | FW-Grundierung zur Verstärkung von INT1 5' mit Stecker 5 | 4.4 |
| KC-110 |
AAACGCCTGTGGGTGGTAC TGGATATGCAAAGCGATTGGAA GTCGCTTGACTCCTCTGCCGTC ATTCC | RV-Grundierung zur Verstärkung von INT1 5' mit Stecker 5 | 4.4 |
| KC-111 |
TTGCCCATCGAACGTACAAG TACTCCTCTGTTCTCTCTCTCTT TGCTTTAAGCGTTGAAGTTTCCTC TTTG | FW-Grundierung zur Verstärkung von INT1 3' mit Stecker A | 4.4 |
| KC-112 | TGTCAACTGGAGAGCTATCG | RV-Grundierung zur Verstärkung von INT1 3' mit Stecker A | 4.4 |
| KC-113 | AGAAGATTTCTCTTCAATCTC | FW-Grundierung zur Verstärkung von INT2 5' mit Stecker B | 4.4 |
| KC-114 |
TGCTAAGATTTGTGTTCGTT TGGGTGCAGGGTTGTACAT CGATCCGCCCTTATCAAGGATACC TGGTTG | RV-Grundierung zur Verstärkung von INT2 5' mit Stecker B | 4.4 |
| KC-115 |
ACGCTTTCCGGCATCTTCCA GACCACAGTATATCCATCCGCCT CCTGTTGGGCGATTACACAAGCG GTGG | FW-Grundierung zur Verstärkung von INT2 3' mit Stecker C | 4.4 |
| KC-116 | TCTCTTCTCGATGACCGGG | RV-Grundierung zur Verstärkung von INT2 3' mit Stecker C | 4.4 |
| KC-117 | GGTCGTTTTTGTGCAGCATATTG | FW-Grundierung zur Verstärkung von INT3 5' mit Stecker D | 4.4 |
| KC-118 |
GCGGAATATTGGCGGAACGG ACACACGTGGATACAAACCTG GACAACGTTTTCCAAGGAGGTG AAGAACG | RV-Grundierung zur Verstärkung von INT3 5' mit Stecker D | 4.4 |
| KC-119 |
AAATAACCACAAAACATCCTT CCCATATGCTCTCTCGCGCTTGTT GTACCTGATGGGACGTCAGCACT GTAC | FW-Grundierung zur Verstärkung von INT3 3' mit Stecker E | 4.4 |
| KC-120 | GAGCTTACTCTATATATTCATTC | RV-Grundierung zur Verstärkung von INT3 3' mit Stecker E | 4.4 |
| KC-121 | GTTACTAAACTGGAACTGTCCG | FW-Primer zur Überprüfung der Integration von con5-crtE-conA in INT1 5' | 7.4.1 |
| KC-122 | CACTGCTAACTACGTTTACTTC | FW-Primer zur Überprüfung der Integration von con5-crtE-conA in INT1 3' | 7.4.1 |
| KC-123 | CACTGGAACTTGAGCTTGAG | FW-Primer zur Überprüfung der Integration von conB-crtYB-conC in INT2 5' | 7.4.1 |
| KC-124 | GTCTCCAGCTGAATTGGTCC | FW-Primer zur Überprüfung der Integration von conB-crtYB-conC in INT2 3' | 7.4.1 |
| KC-125 | CTCTCATGAAGCAGTCAAGTC | FW-Primer zur Überprüfung der Integration von conD-crtI-conE in INT3 5' | 7.4.1 |
| KC-126 | GATCGGTCAATTAGGTGAAG | FW-Primer zur Überprüfung der Integration von conD-crtI-conE in INT3 3' | 7.4.1 |
| KC-127 | CCTTGTCCAAGTAGGTCC | RV-Primer zur Überprüfung der Integration von con5-crtE-conA in INT1 5' | 7.4.1 |
| KC-128 | GCTGTCATGATCTGTGATAAC | RV-Primer zur Überprüfung der Integration von con5-crtE-conA in INT1 3' | 7.4.1 |
| KC-129 | CTGGCAATGTTGACCAATTGC | RV-Primer zur Überprüfung der Integration von conB-crtYB-conC in INT2 5' | 7.4.1 |
| KC-130 | CCAACGTGCCTTAAAGTCTG | RV-Primer zur Überprüfung der Integration von conB-crtYB-conC in INT2 3' | 7.4.1 |
| KC-131 | CCTTACCTTCTGGAGCAGCAG | RV-Grundierung zur Überprüfung der Integration von conD-crtI-conE in INT3 5' | 7.4.1 |
| KC-132 | CTGGTTACTTCCCTAAGACTG | RV-Primer zur Überprüfung der Integration von conD-crtI-conE in INT3 3' | 7.4.1 |
| a. Fette Sequenzen bezeichnen Verbindersequenzen. b. Vorwärts- und Rückwärtsprimer werden als FW bzw. RV bezeichnet. |
Ergänzende Tabelle 2: Primersequenzen.
Ergänzende Tabelle 3: Konstruktion konstruierter Stämme.

Ergänzende Tabelle 4: Sequenzen von Spender-DNA-Expressionskassetten und Abplatzungsbereichen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das bereitgestellte Protokoll beschreibt die Multiplex-Genombearbeitung von S. cerevisiae mit Cas12a aus Lachnospiraceae-Bakterium ND2006 in Kombination mit einem einzigen crRNA-Array und Spender-DNA. Das Design des einzelnen crRNA-Arrays und der Spender-DNA wird ausführlich erläutert. Im Gegensatz zum bewährten CRISPR/Cas9-System verfügt das CRISPR/Cas12a über die einzigartige zusätzliche Fähigkeit, mehrere crRNAs aus einem einzigen crRNA-Array13,33zu verarbeiten. Aufgrund dieser Funktion ist die gleichzeitige Bearbeitung mehrerer Ziele einfacher einzurichten und kann in einer einzigen Transformation erreicht werden. Dieser einzelne crRNA-Array-Ansatz wurde zuvor von Zetsche etal. demonstriert. 34, die gleichzeitig bis zu vier Gene in Säugetierzellen mit AsCas12a und von Swiat et al. 35, die mit FnCas12a vier DNA-Fragmente in ein Hefegenom einführten. Unserer Kenntnis nach wurde eine höhere Anzahl simultaner genomischer Modifikationen mit einem Cas12a-System nicht gemeldet, und die maximale Zielgrenze pro einzelnem Array für Cas12a steht noch nicht fest. Weitere Forschung mit einzelnen crRNA-Arrays in Kombination mit Cas12a umfasst Multiplex-Transkriptionsregulation in einer Vielzahl von Organismen33,36,37.
Es gibt einige wichtige Schritte im vorgestellten Protokoll. Entwerfen Sie sorgfältig alle DNA-Sequenzen, die am Cas12a-Genombearbeitungsexperiment beteiligt sind, insbesondere im Falle der Einführung neuartiger DNA-Sequenzen. Bestimmen Sie die Funktionalität neuer Spacer-Sequenzen, die Teil einer crRNA sind, z. B. durch ein Singleplex-Genombearbeitungsexperiment, wie von Verwaal et albeschrieben. 19, bevor sie zu einem einzigen crRNA-Array kombiniert werden. Befolgen Sie die Empfehlungen für die Vorbereitung von Transformationspufferlösungen, die im Cas12a-Bearbeitungsexperiment verwendet werden, um eine gute Transformationseffizienz von Hefe zu erreichen.
Es gibt einige optionale Änderungen der Technik. Es wird empfohlen, bei der Transformation 1 g jeder Spender-DNA, linearisierte pRN1120 oder eine einzelne crRNA-Array-Expressionskassette zu verwenden, obwohl die Verwendung einer geringeren DNA-Menge ebenfalls zu einer zufriedenstellenden Transformationseffizienz führen dürfte. Führen Sie eine Testtransformation durch, um festzustellen, ob niedrigere DNA-Mengen verwendet werden können. Die Transformation von S. cerevisiae kann mit einer anderen Methode als der in diesem Protokoll beschriebenen durchgeführt werden, zum Beispiel das von Gietz et al. beschriebene Protokoll. 2007: 38. Das Guide-RNA-Empfänger-Plasmid pRN1120 eignet sich für die Expression eines einzelnen crRNA- und einzelnen crRNA-Arrays verschiedener Cas12a-Varianten(z.B.aus Acidaminococcus spp. BV3L6 oder Francisella novicida U112) sowie zur Expression von sgRNA in Kombination mit Cas919. Die Spender-DNA muss sich nicht auf Carotinoid-Genexpressionskassetten und flankierende Regionen beschränken, die die Spender-DNA auf die beschriebenen INT1-, INT2- und INT3-Standorte in der genomischen DNA zielen. Jede DNA von Interesse kann in Multiplex-Manier durch die in diesem Protokoll beschriebenen Designprinzipien in die genomische DNA des Wirts eingeführt werden, oder alternativ kann Spender-DNA verwendet werden, um DNA aus einem Wirtsgenom zu löschen. Der modulare Aufbau eines einzelnen crRNA-Arrays ermöglicht eine einfache Einstellung von Abstands- und direkten Wiederholungssequenzen. Die Modifikation von Abstandsreihen ermöglicht eine Änderung des vorgesehenen Integrationsortes, der durch eines der Werkzeuge zur Identifizierung einer genomischen Zielstelle entworfen werden kann, z.B. GuideScan Software 1.039. Anstatt große Flankensequenzen zu verwenden, die Konnektorensequenzen enthalten, können 50-bp des flankierenden Bereichs in die Spender-DNA-Sequenzen einbezogen werden, indem diese 50-bp-Flankenbereichssequenzen in die in der PCR verwendeten Primer integriert werden. In diesem Fall sind insgesamt nur drei statt neun Spender-DNA-Fragmente für ein erfolgreiches Multiplex-Genom-Editing-Experiment erforderlich.
Zusammenfassend bietet dieses Protokoll Schritt-für-Schritt-Anleitungen zur Multiplex-Genombearbeitung in S. cerevisiae mit Cas12a in Kombination mit einem einzigen crRNA-Array-Ansatz. Das Protokoll wurde durch Multiplex-Genombearbeitung mit 9 Spender-DNA-Fragmenten und einer einzigen crRNA-Array-Codierung für drei gRNAs demonstriert. Wir zeigen hohe Gesamtbearbeitungsfrequenzen zwischen 50% und 94% für die drei hier gemeldeten Dehnungsdesigns. Das einzigartige Merkmal von Cas12a ist die Fähigkeit, ein einzelnes crRNA-Array in einzelne crRNAs in einer Zelle zu verarbeiten, was Cas12a zu einem ausgezeichneten Werkzeug macht, um multiple Xx-Genombearbeitung zu ermöglichen und Transkriptions-Regulierungsmodule zu entwickeln, die auf mehrere Ausdruckskassetten in einem Rutsch. Am Ende wurden drei Stämme erhalten, die Carotinoide auf einer anderen Ebene und Farben in Schattierungen zwischen Gelb und Orange produzieren. Mit diesen Stämmen und einer wilden Sorte zeigten wir, wie ein akustischer Flüssigkeitshandler einfach eingesetzt werden kann, um Hefe-Pixel-Kunst zu machen – dies zu Ehren von Rosalind Franklin, die vor 65 Jahren durch ihr berühmtes Foto 51 23 zur Entdeckung der DNA-Struktur beitrug. /c1>.
Offenlegungen
Die Autoren erklären, dass es einen Interessenkonflikt gibt. Die Autoren haben IP in Bezug auf präsentierte Methoden eingereicht.
Danksagungen
Dieses Projekt wurde aus dem Forschungs- und Innovationsprogramm Horizont 2020 der Europäischen Union im Rahmen der Fördervereinbarung Nr. 686070 (DD-DeCaf) und 764591 (SynCrop) sowie aus dem Forschungsprogramm Bausteine des Lebens mit der Projektnummer 737.016.005 von der Niederländischen Organisation für wissenschaftliche Forschung (NWO). T.E.G. wurde von der Royal Society (Grant UF160357) und BrisSynBio, einem BBSRC/EPSRC Synthetic Biology Research Centre (Grant BB/L01386X/1), unterstützt. Wir danken Zi Di und Jeffrey van Wijk für ihren Beitrag zu den Hefe-Spotting-Experimenten zur Entstehung der Hefe-Pixel-Kunst.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals specific for the protocol | |||
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | Electrophoresis |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | Selection of E. coli transformants |
| BsaI-HF (20 U/µl) | New England BioLabs | R353L | Golden Gate Cloning |
| Cell Lysis Solution (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| CutSmart Buffer | New England BioLabs | B7204S | Linearization of pRN1120 |
| Deoxyribonucleic acid sodium salt from salmon testes | Sigma Aldrich | D1626 | Transfromation of S. cerevisiae (carrier DNA) |
| dNTPs | Invitrogen | 10297018 | PCRs |
| EcoRI-HF | New England BioLabs | R3101S | Linearization of pRN1120 |
| Ethanol absolute for analysis | Merck | 100983 | Isolation of genomic DNA from S. cerevisiae |
| Ethylenediamine-tetraacetic acid | Sigma Aldrich | ED | Transformation of S. cerevisiae |
| G418 disulfate salt | Sigma Aldrich | A1720 | Selection of S. cerevisiae transformants |
| Histodenz | Sigma Aldrich | D2158 | Yeast pixel art |
| Isopropanol | Merck | 100993 | Isolation of genomic DNA from S. cerevisiae |
| Lithium acetate dihydrate | Sigma Aldrich | L6883 | Transformation of S. cerevisiae |
| Nancy-520 DNA Gel Stain | Sigma Aldrich | 1494 | Electrophoresis |
| NEB10 competent E. coli cells | New England BioLabs | C3019H | Transformation of E. coli: dx.doi.org/10.17504/protocols.io.nkvdcw6 |
| Nourseothricin | Jena Bioscience | AB102 | Selection of S. cerevisiae transformants |
| Phusion buffer | New England BioLabs | M0530L | PCRs |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530L | PCRs |
| Polyethylene glycol 4000 | Merck | 7490 | Transformation of S. cerevisiae |
| Protein Precipitation Solution (10 M NH4AC) (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| Purple loading dye | New England BioLabs | B7024S | Electrophoresis |
| QIAprep Spin Miniprep Kit | QIAGEN | 27106 | Purification of plasmids |
| RNase coctail enzyme mix | Thermo Fisher Scientific | AM2286 | Isolation of genomic DNA from S. cerevisiae |
| T4 DNA ligase buffer | Invitrogen | 46300-018 | Golden Gate Cloning |
| T4 DNA Ligase (1 U/µl) | Invitrogen | 1705218 | Golden Gate Cloning |
| UltraPure Agarose | Invitrogen | 16500500 | Electrophoresis |
| Wizard SV Gel and PCR Clean-Up System Kit | Promega | A9282 | Purification of PCR products and linearized pRN1120 |
| Xhol | New England BioLabs | R0146S | Linearization of pRN1120 |
| Zymolyase 50 mg/ml (5 units/µL) | Zymo Research | E1006 | Isolation of genomic DNA from S. cerevisiae (yeast lysis enzyme) |
| Zymolyase storage buffer | Zymo Research | E1004-B | Isolation of genomic DNA from S. cerevisiae (necessary for the preparation of yeast lysis enzyme) |
| Chemicals of general use | |||
| 2*Peptone-Yeast extract (PY) agar | Plate growth of E. coli | ||
| 2*PY medium | Cultivation of E. coli | ||
| Demineralized water | Transformation of S. cerevisiae | ||
| ELFO buffer | Electrophoresis | ||
| MQ | Multiple steps | ||
| Physiological salt solution | Transformation of S. cerevisiae | ||
| TE buffer | Storage of DNA, transformation of S. cerevisiae | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | Cultivation of S. cerevisiae | ||
| YEPD (2% glucose) agar | Plate growth of S. cerevisiae | ||
| Consumables | |||
| Eppendorf tubes | |||
| Falcon tubes (50 mL) | |||
| Microplate 96 wells | |||
| Petri dishes | |||
| Pipette tips 0.5 - 10 µL | |||
| Pipette tips 10 - 200 µL | |||
| Pipette tips 100 - 1000 µL | |||
| Shake flasks (500 mL) | |||
| Sterile filters | |||
| Equipment | |||
| Centrifuge (Falcon tubes) | |||
| Echo 525 acoustic liquid handler | |||
| Incubator | |||
| NanoDrop | |||
| Set for eletrophoresis | |||
| Spectrophotometer | |||
| Table centrifuge (Eppendorfs tubes) | |||
| Thermocycler | |||
| Plasmids | |||
| pCSN067 | Addgene | ID 101748 | https://www.addgene.org/ |
| pRN1120 | Addgene | ID 101750 | https://www.addgene.org/ |
| Strains | |||
| S. cerevisiae strain CEN.PK113-7D | EUROSCARF collection | http://www.euroscarf.de |
Referenzen
- Knott, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361 (6405), 866-869 (2018).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Lian, J., HamediRad, M., Hu, S., Zhao, H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications. 8 (1), 1688 (2017).
- Li, Z. -. H., Liu, M., Lyu, X. -. M., Wang, F. -. Q., Wei, D. -. Z. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology. 58 (12), 1100-1104 (2018).
- Shao, Y., Lu, N., Qin, Z., Xue, X. CRISPR-Cas9 facilitated multiple-chromosome fusion in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (11), 2706-2708 (2018).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology. 13 (11), 722-736 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), (2016).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovič, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Lian, J., HamediRad, M., Zhao, H. Advancing metabolic engineering of Saccharomyces cerevisiae. using the CRISPR/Cas System. Biotechnology Journal. 13 (9), 1700601 (2018).
- Ferreira, R., et al. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (1), 10-15 (2018).
- Swarts, D. C., Martin, J. Cas9 versus Cas12a/Cpf1: Structure–function comparisons and implications for genome editing. Wiley Interdisciplinary Reviews: RNA. 9 (5), 1481 (2018).
- Strohkendl, I., Saifuddin, F. A., Rybarski, J. R., Finkelstein, I. J., Russell, R. Kinetic Basis for DNA Target Specificity of CRISPR-Cas12a. Molecular Cell. 71 (5), 816-824 (2018).
- Verwaal, R., et al. High-level production of beta-carotene in Saccharomyces cerevisiae. by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous. Applied and Environmental Microbiology. 73 (13), 4342-4350 (2007).
- Verwaal, R., Buiting-Wiessenhaan, N., Dalhuijsen, S., Roubos, J. A. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast. 35 (2), 201-211 (2018).
- Jakociunas, T., Jensen, M. K., Keasling, J. D. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering. 34, 44-59 (2016).
- Engler, C., Romy, K., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PloS One. 3 (11), 3647 (2008).
- Franklin, R. E., Gosling, R. G. Molecular configuration in sodium thymonucleate. Nature. 171, 740-741 (1953).
- Watson, J. D., Crick, F. H. A structure for deoxyribose nucleic acid. Nature. 171, 737-738 (1953).
- Wilkins, M. H. F., Stokes, A. R., Wilson, H. R. Molecular structure of deoxypentose nucleic acids. Nature. 171, 738-740 (1953).
- Young, E. M., et al. Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast. Metabolic Engineering. 48, 33-43 (2018).
- Roubos, J. A., Pel, H. J., Meijrink, B. . Cloning Method. , (2013).
- Mandel, M., Higa, A. Calcium-dependent bacteriophage DNA infection. Journal of Molecular Biology. 53 (1), 159-162 (1970).
- Van Dijken, J. P., et al. An interlaboratory comparison of physiological and genetic properties of four Saccharomyces cerevisiae strains. Enzyme and Microbial Technology. 26 (9-10), 706-714 (2000).
- Gietz, R. D., Schiestl, R. H., Willems, A. R., Woods, R. A. Studies on the transformation of intact yeast cells by the LiAc/SS-DNA/PEG procedure. Yeast. 11 (4), 355-360 (1995).
- Hill, J., Donald, K. A., Griffiths, D. E., Donald, G. DMSO-enhanced whole cell yeast transformation. Acids Research. 19 (20), 5791 (1991).
- Looke, M., Kristjuhan, K., Kristjuhan, A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques. 50 (5), 325-328 (2011).
- Tak, Y. E., et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nature Methods. 14 (12), 1163-1166 (2017).
- Zetsche, B., et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature biotechnology. 35 (1), 31-34 (2017).
- Swiat, M. A., et al. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research. 45 (21), 12585-12598 (2017).
- Li, L., et al. CRISPR-Cpf1-Assisted Multiplex Genome Editing and Transcriptional Repression in Streptomyces. Applied Environmental Microbiology. 84 (18), 00827-00918 (2018).
- Zhang, X., et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery. 3, 17018 (2017).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 1-4 (2007).
- Perez, A. R., et al. GuideScan software for improved single and paired CRISPR guide RNA design. Nature Biotechnology. 35 (4), 347-349 (2017).
- Cox, R. S., et al. Synthetic Biology Open Language Visual (SBOL Visual) Version 2.0. Journal of Integrative Bioinformatics. 15 (1), 1613-4516 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten