Method Article
CRISPR/Cas12a Multiplex Genoma Edición de Saccharomyces cerevisiae y la creación de Arte de píxeles de levadura
En este artículo
Resumen
El sistema CRISPR/Cas12a en combinación con una sola matriz crRNA permite la edición multiplexación eficiente del genoma de S. cerevisiae a múltiples loci simultáneamente. Esto se demuestra mediante la construcción de carotenoides que producen cepas de levadura que posteriormente se utilizan para crear arte de píxeles de levadura.
Resumen
Alta eficiencia, facilidad de uso y versatilidad del sistema agrupado regularmente interespacilado de repeticiones palindrómicas cortas/proteína asociada a CRISPR 9 (CRISPR/Cas9) ha facilitado la modificación genética avanzada de Saccharomyces cerevisiae,un modelo organismo y caballo de batalla en biotecnología industrial. En este trabajo se aplica la proteína 12a asociada a CRISPR (Cas12a), una endonucleasa guiada por ARN con características distinguibles de Cas9, extendiendo aún más la caja de herramientas molecular es para fines de edición del genoma. Una ventaja del sistema CRISPR/Cas12a es que se puede utilizar en la edición del genoma multiplex con múltiples ARN guía expresados a partir de una sola unidad transcripcional (matriz de ARN CRISPR único (crRNA). Presentamos un protocolo para la integración múltiple de múltiples genes hetólogos en loci independientes del genoma de S. cerevisiae utilizando el sistema CRISPR/Cas12a con múltiples crRNAs expresados a partir de una sola construcción de matriz crRNA. El método propuesto aprovecha la capacidad de S. cerevisiae para realizar la recombinación in vivo de fragmentos de ADN para ensamblar la matriz de crRNA única en un plásmido que se puede utilizar para la selección transformadora, así como el montaje de ADN de donante secuencias que se integran en el genoma en las posiciones previstas. Cas12a se expresa de forma pre-expresada, facilitando el escisión del genoma de S. cerevisiae en las posiciones previstas tras la expresión de la matriz de crRNA única. El protocolo incluye el diseño y la construcción de una única matriz de crRNA y casetes de expresión de ADN de donantes, y aprovecha un enfoque de integración que hace uso de secuencias únicas de conectores de ADN de 50 bp y secuencias de ADN de flanco de integración separadas, lo que simplifica diseño experimental a través de la estandarización y modularización y amplía la gama de aplicaciones. Por último, demostramos una técnica sencilla para crear arte de píxeles de levadura con un controlador de líquido acústico que utiliza cepas de levadura de producción de carotenoides de diferentes colores que se construyeron.
Introducción
Las enzimas CRISPR/Cas han revolucionado indudablemente la biología molecular y han sido ampliamente adoptadas como herramientas para los genomas de ingeniería a una velocidad que antes era inviable1. La primera modificación de un genoma de Saccharomyces cerevisiae por el sistema de edición del genoma CRISPR/Cas9 fue reportada por DiCarlo et al. 2, demostrando el éxito del knock-out genético y la fabricación de mutaciones puntuales utilizando oligonucleótidos introducidos externamente. Otros desarrollos de la caja de herramientas CRISPR de levadura incluidos: regulación transcripcional por fusión de Cas9 muerto catalíticamente inactivo (dCas9) con dominios efectores transcripcionales para permitir la activación y silenciamiento de la transcripción3, aplicación para ambos la edición del genoma y las funciones reguladoras para la ingeniería de vías metabólicas mediante activación simultánea, represión y eliminación4, eliminación de fragmentos grandes del genomade S. cerevisiae 5, y fusiones de cromosomas múltiples6 .
Los sistemas de edición del genoma CRISPR/Cas encuentran su origen en sistemas inmunes adaptativos de bacterias y arqueas y estos sistemas han sido adaptados por biólogos moleculares para la edición del genoma. Su funcionalidad se basa en las regiones de ADN agrupadas regularmente interespaciales (CRISPR) que codifican el ARN responsable del reconocimiento del ADN o ARN extraño y los genes asociados a CRISPR (Cas) que codifican los genes guiados por ARN endonucleasas1,7,8,9. Sobre la base del reciente análisis del genoma de los sistemas CRISPR/Cas se propuso dividir los sistemas CRISPR/Cas en dos clases, cinco tipos y 16 subtipos10. Las dos clases se distinguen en función de la organización de los complejos de efectos implicados en el escote objetivo. Normalmente, los sistemas CRISPR/Cas con una organización multisubunidad se clasifican como clase 1, mientras que los complejos de efector de subunidad única pertenecen a la clase 210,11. En este artículo, exploramos la clase 2 tipo V Cas12a, anteriormente llamada Cpf110,12, que es una alternativa a la clase 2 tipo II Cas9. Aunque Cas9 está bien caracterizado y ampliamente utilizado en la investigación, Cas12a ofrece características adicionales12. En primer lugar, Cas12a forma un complejo con ARN CRISPR (crRNA) de 42 a 44 nucleótidos sin necesidad de un ARN CRISPR activador adicional (tracrRNA). Por lo tanto, se puede utilizar un ARN guía más corto en la edición del genoma con sistemas CRISPR/Cas12a en comparación con CRISPR/Cas9. En segundo lugar, la actividad única de endonucleasa y endoribonucleasa de Cas12a permite la maduración de su pre-crRNA13. Esta actividad De RNase permite la codificación de múltiples crRNAs en una sola matriz crRNA CRISPR, mientras que Cas9 requiere la expresión separada de cada ARN de guía único (sgRNA) o, alternativamente, por ejemplo, la expresión de una endonucleasa adicional ( porejemplo, Csy4) en combinación con motivos de reconocimiento para Csy4 que rodean cada sgRNA14,15. En tercer lugar, el reconocimiento del sitio objetivo Cas12a requiere un motivo adyacente protoespacial (PAM) en el extremo de 5' del objetivo y se corta después de la posición +18/+23 de su PAM, lo que resulta en ADN de hojalata con extremos pegajosos, mientras que Cas9 requiere un PAM situado en el extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' del extremo 3' objetivo y cortes después de la posición -3 creando cortes finales contundentes en el ADN12. En cuarto lugar, la secuencia de nucleótidos de consenso del PAM difiere entre Cas12a ((T)TTV) y Cas9 (NGG), lo que convierte a Cas12a en un candidato prometedor para apuntar a la secuencia16de promotores y terminadores ricos en T. Finalmente, un estudio reciente reportó una mayor especificidad de destino para Cas12a que para el Cas917nativo.
Presentamos un protocolo para utilizar el sistema CRISPR/Cas12a para la edición del genoma de S. cerevisiae con un enfoque particular en la introducción de múltiples casetes de expresión de ADN en loci genómicos independientes simultáneamente (edición del genoma multiplex) utilizando una sola matriz de crRNA. Los pasos clave del protocolo se describen en la Figura1. Como prueba de concepto, se aplicó el sistema CRISPR/Cas12a para la introducción de tres casetes de expresión en el genoma de S. cerevisiae que permiten la producción de caroteno18 como se muestra esquemáticamente en la Figura2. La producción de caroteno afecta al fenotipo de S. cerevisiae: esdecir, tras la introducción exitosa de los tres genes hetrólogos necesarios para la biosíntesis de carotenoides, las células blancas de S. cerevisiae se vuelven amarillas o anaranjadas, dependiendo de la fuerza de expresión del promotor de cada gen. Debido a la simple lectura visual de esta vía, se ha introducido para desarrollar sistemas avanzados basados en CRISPR y métodos para la edición del genoma19,20. En este trabajo, los casetes de expresión que codifican los genes carotenoides crtE, crtYB y crtI se han construido utilizando un enfoque de clonación Golden Gate (GGC)21 con promotores heterólogos y terminadores homólogos para impulsar la expresión de los genes. Los casetes de expresión están rodeados por secuencias únicas de pares de 50 bases (bp), llamadas conectores, que permiten el montaje in vivo con secuencias de ADN de flanco de integración (regiones de flanqueo) con las mismas secuencias de 50 bp, y la posterior integración en el ADN genómico de la levadura en la posición determinada por las regiones flanqueantes. Mediante el uso de diferentes fortalezas promotoras, se obtuvieron cepas con diferentes niveles de producción de carotenoides dando como resultado la variación en el color de las células. Estas cepas - inspiradas en el "Proyecto de Arte de levadura"22 - se utilizaron en una configuración de detección con un manipulador de líquido acústico para crear una "fotografía de levadura" de alta resolución de 4 colores de Rosalind Franklin. Franklin (1920-1958) fue una química inglesa y cristalógrafa de rayos X muy conocida por su contribución al descubrimiento de la estructura de ADN por Photo 5123,24,25.
Protocolo
1. Preparación de los plásmidos Cas12a
NOTA: El plásmido que contiene la bacteria Lachnospiraceae ND2006 Cas12a (LbCpf1, pCSN067) codón optimizado para la expresión en S. cerevisiae, fue construido previamente19, depositado en un repositorio plásmido (véase la Tabla de Materiales). Se trata de un plásmido de lanzadera episómico de una sola copia S. cerevisiae/E. coli que contiene un gen marcador de resistencia KanMX para permitir la selección de transformadores de S. cerevisiae en la genética (G418).
- Obtenga el plásmido pCSN067 (consulte la Tabla de materiales).
- Amplificar el plásmido pCSN067 para obtener una gran cantidad.
- Transformar 25 l de células de E. coli químicamente competentes con el plásmido pCSN067 de acuerdo con el protocolo del fabricante. Diluir la mezcla de transformación 10 y 50 veces en 2x peptona-levadura (PY). Placa de diluciones de 10x y 50x en 2 placas de agar PY que contienen ampicilina (0,1 g/L) e incubación durante la noche a 37 oC.
- Escoge de 2 a 3 colonias e inocula cada colonia en 3 mL de 2x PY y crece durante la noche a 37oC en una incubadora de temblores a 180 rpm.
- Purificar el plásmido utilizando un kit de purificación de plásmido de acuerdo con las instrucciones del fabricante.
2. Preparación del casete de expresión de matriz crRNA único
- Prepare la matriz de crRNA única.
NOTA: La matriz de crRNA única comprende un promotor de arn polimerasa III SNR52 de S. cerevisiae2, una repetición directa específica para LbCas12a y un espaciador (secuencia de diana genómica), repetidas juntas para cada objetivo19 y extremos con un terminador SUP4 de S. cerevisiae2. La matriz de crRNA única se ensambla mediante la recombinación in vivo en el plásmido linealizado pRN1120 para generar un plásmido circular, por lo que las regiones homólogas a plásmidas pRN1120 deben estar presentes al principio y al final de la matriz de crRNA única (ver Figura 2A ). Se recomienda evaluar de antemano la funcionalidad de una serie de crRNAs diseñados por separado19. Esta información se utiliza posteriormente para seleccionar la mayoría de los CRRA funcionales para combinarlos en las secuencias de repetición directa y espaciador para crear una sola matriz crRNA para el propósito de multiplexación.- Ordene la matriz de crRNA única para experimentos de edición del genoma multiplex como ADN sintético (consulte la secuencia de ADN de la matriz de crRNA única en la Tabla Suplementaria 1).
- Amplifique la matriz decrRNA única ordenada (por ejemplo, utilizando los imprimadores KC-101 y KC-102 (Tablasuplementaria 2)). Preparar la mezcla de amplificación de PCR que contiene: 0,5 l de polimerasa de ADN, 10 ml de tampón de ADN necesario para la polimerasa de ADN, 1 l de dNTPs de 10 mM, 2,5 ml de imprimación delantera de 10 oM, 2,5 ml de imprimación inversa de 10 oM, 2 ml de plantilla de ADN a una concentración de 5 ng/L y hpuro ultrapuro 2 O hasta un volumen total de 50 l.
- Realizar la reacción en un termociclador utilizando el siguiente programa: (i) 98 oC durante 3 min, (ii) 98 oC durante 10 s, (iii) 60 oC durante 20 s, (iv) 72 oC para 15 s – repita los pasos (ii) a (iv) 30 veces, (v) 72 oC durante 5 min (vi) mantenga a 12 oC hasta un análisis posterior.
- Analizar los productos de PCR por electroforesis ejecutando las muestras en un gel de agarosa del 0,8% a 5 V/cm durante 40 minutos utilizando un tinte de carga de ADN y una escalera de ADN con fragmentos de ADN en un rango de 100 a 10.000 bp.
- Purifique los productos PCR utilizando un kit de purificación de PCR de acuerdo con las instrucciones del fabricante.
- Prepare el plásmido receptor de la matriz crRNA única.
NOTA: La matriz de crRNA única se expresa a partir del plásmido de lanzadera S. cerevisiae/E. coli pRN112019 (véase la Tabla de Materiales). Este plásmido multicopia contiene un gen marcador de resistencia NatMX para permitir la selección de transformadores de S. cerevisiae en nourseotracina (NTC).- Obtenga el plásmido pRN1120.
- Amplificar el plásmido pRN1120 para obtener una gran cantidad.
- Transformar 25 l de células de E. coli químicamente competentes con plásmido pRN1120 según el protocolo del fabricante. Diluir la mezcla de transformación 10 y 50 veces en 2x PY. Placa de diluciones de 10x y 50x en 2 placas de agar PY que contienen ampicilina (0,1 g/L) e incubación durante la noche a 37 oC.
- Escoge de 2 a 3 colonias e inocula cada colonia en 3 mL de 2x PY y crece durante la noche a 37oC en una incubadora de temblores a 180 rpm.
- Purificar el plásmido utilizando un kit de purificación de plásmido de acuerdo con las instrucciones del fabricante.
- Linealice el plásmido pRN1120 con EcoRI-HF y XhoI. Para ello, prepare una mezcla de digestión compuesta por 1 g de pRN1120, 5 l de tampón 10x (1x tampón contiene acetato de potasio de 50 mM, 20 mM de acetato de Tris-acetato, acetato de magnesio de 10 mM, albúmina sérica bovina de 100 g/ml [BSA]; pH 7,9), 1 l de EcoRI-HF (20 U) , 1 l de XhoI (20 U) y Ultrapuro H2O hasta un volumen total de 50 ol. Incubar la mezcla de digestión a 37 oC durante 2 h e inactivar a 65 oC durante 20 min.
- Analizar el plásmido linealizado por electroforesis en un gel de agarosa (0,8%, 40 min, 5 V/cm) utilizando un tinte de carga de ADN y una escalera de ADN con fragmentos de ADN en un rango de 100 a 10.000 bp. Como control incluir un plásmido circular en el análisis.
- Purificar el plásmido linealizado utilizando un kit de purificación de PCR de acuerdo con las instrucciones del fabricante.
3. Preparación de construcciones de ADN de donante Promotor-ORF-Terminator (POT)
- Ordene un conjunto de secuencias de promotor (P) de diferente fuerza, marco de lectura abierto (O) y terminador (T) como ADN sintético de tal manera que cada elemento contenga secuencias de reconocimiento estandarizadas de 4 bp que están flanqueadas por sitios BsaI para habilitar la clonación de Golden Gate ( GGC)26 (véanse los diseños detallados en el Cuadro Suplementario 3 y las secuencias en el Cuadro Suplementario 4).
- Ensamblar casetes de expresión POT compuestos por un promotor, fotograma de lectura abierto, terminador y secuencias de conectores a través de un ensamblaje de 4 partes utilizando una reacción GGC21, en un vector de destino que ya contiene secuencias de conectores de 50 bp preespecificadas ( véase el Cuadro Suplementario 4 y las referencias26,27).
- Mida la concentración de las partes de ADN utilizando un espectrofotómetro. Diluir cada parte del ADN en H2O ultrapuro a una concentración final de 15 fmol/L.
- Preparar una mezcla de reacción compuesta de fragmentos de ADN: 2 l de promotor, 2 l de marco de lectura abierto, 2 l de terminador y columna vertebral de 2 l (vectores de destino de nivel 1, tal como se describe en 26), 4 ml de tampón de ligasa de ADN 5x T4, 2,5 ml de 1 ligados a ADN U/L T4 , 1,5 l de 20 U/L BsaI-HF y Ultrapure H2O hasta un volumen total de 20 sL.
- Realizar la reacción GGC en un termociclador utilizando el siguiente programa: (i) 37 oC durante 2 min, (ii) 16 oC durante 5 min – repetir los pasos (i) y (ii) 50 veces, (iii) 50 oC para 60 min, (iv) 80 oC para 45 min, (v) mantener a 12 oC hasta realizar un análisis posterior.
- Transformar 25 ml de células compradas de E. coli28 químicamente competentes con 3 l de la mezcla de reacción GGC de acuerdo con el protocolo del fabricante. Diluir la mezcla de transformación 10 y 50 veces en 2x PY. Placa de diluciones de 10x y 50x en 2 placas de agar PY que contienen ampicilina (0,1 g/L) e incubación durante la noche a 37 oC.
- Escoge de 2 a 3 colonias e inocula cada colonia en 3 mL de 2x PY y crece durante la noche a 37oC en una incubadora de temblores a 180 rpm.
- Purificar los plásmidos usando un kit de purificación de plásmido de acuerdo con las instrucciones del fabricante.
- Compruebe si los casetes de expresión POT se montaron correctamente en la reacción GGC por PCR.
- Diseñe imprimaciones complementarias a la secuencia de conectores presente al principio y al final de cada casete de expresión (consulte la figura 2B). Para los conectores elegidos en este protocolo utilice los imprimadores KC-103 a KC-108 (véase la Tabla Suplementaria2).
- Preparar las mezclas de amplificación de PCR para cada plásmido que contenga: 0,5 l de mestizaje de ADN polimerasa, 10 ml de tampón de ADN necesario para la polimerasa de ADN, 1 l de dNTPs de 10 mM, 2,5 ml de imprimación directa de 10 oM, 2,5 ml de inversa de 10 m, 2 ml de plantilla de ADN con una imprimación de concentrado ión de 5 ng/L, y Ultrapuro H2O hasta un volumen total de 50 sL.
- Realizar la reacción de PCR en un termociclador utilizando el siguiente programa: (i) 98 oC 3 min, (ii) 98 oC durante 10 s, (iii) 60 oC para 20 s, (iv) 72 oC para 2 min 30 s – repetir los pasos (ii) a (iv) 30 veces, (v) 72 oC durante 5 min , vi) mantener a 12 oC hasta que se pueda seguir analizando.
NOTA: Los productos PCR resultantes consisten en 50-bp del conector de 5', promotor, marco de lectura abierto, terminador y 50-bp del conector de 3'.
- Analizar los productos de PCR por electroforesis ejecutando muestras en un gel de agarosa del 0,8% a 5 V/cm durante 40 minutos utilizando un tinte de carga de ADN y una escalera de ADN con fragmentos de ADN en un rango de 100 a 10.000 bp.
4. Preparación de secuencias de ADN de flanco de integración que contienen secuencias de conectores
- Purificar el ADN genómico del tipo silvestre S. cerevisiae CEN.PK113-7D29.
- Cultivar la cepa en un matraz de batido de 500 ml lleno de 100 ml de dextrosa de peptona de extracto de levadura (YEPD, 2% glucosa) a 30 oC y agitar a 250 rpm durante 48 horas.
- Cosecha las células por centrifugación de 2 ml de caldo a 16.000 x g durante 1 min y deseche el sobrenadante.
- Resuspender las células en sal fisiológica (200 l; 0,85% solución de NaCl) con RNase (10 l, 10 mg/ml) y enzima lítica de levadura (4 l). Incubar la suspensión celular a 37oC durante 15 min.
- Añadir 300 l de solución de lisis celular (ver Tabla de materiales)y vórtice en breve.
- Añadir 168 ml de solución de precipitación proteica (ver Tabla de Materiales)y vórtice vigorosamente durante 20 s.
- Separar la fracción proteica por centrifugación a 16.000 x g y 4 oC durante 10 min. Recoger 600 ml de sobrenadante en un tubo nuevo y mezclar con 600 ml de isopropanol y vórtice en breve.
- Recuperar el ADN girando hacia abajo a 16,000 x g a temperatura ambiente durante 10 min. Deseche el sobrenadante y mantenga el pellet.
- Lavar el pellet con 200 ml de etanol (70%). Centrifugar a 16.000 x g a temperatura ambiente durante 10 min y retirar el sobrenadante. Evaporar el etanol incubando el tubo a temperatura ambiente durante 10 minutos con la tapa abierta.
NOTA: Si el líquido en el tubo sigue siendo visible, repita el paso 4.1.8. No seque el pellet durante más de 10 minutos para evitar una disminución de la solubilidad del ADN. - Disolver el ADN en 50 s de tampón te. Almacene el ADN purificado a 4oC.
- Para cada sitio de integración, la integración del diseño flanquea secuencias de ADN (aprox. 500 bp) de modo que se eliminarán aproximadamente 1000 bp de ADN genómico al presentar ADN de donante (véase el diseño esquemático en la Figura 2B y las secuencias en Tabla 4).
- Diseñar imprimaciones para generar las regiones de flanqueo por PCR.
- Para la región de flanqueoizquierdo, diseñe imprimadores hacia adelante y hacia atrás para amplificar aproximadamente 500 bp de la región de ADN genómico posicionado 5' (izquierda) del sitio de integración de interés.
NOTA: La imprimación delantera incluye 20 bp de homología con la región de flanqueo prevista. La imprimación inversa incluye 20 bp con homología con la región de flanqueo prevista y contiene la secuencia de conectores de 50 bp deseada para permitir el montaje in vivo en la edición Cas12a en el genoma más adelante. - Para la región de flanqueoderecho, diseñe imprimadores hacia adelante y hacia atrás para amplificar aproximadamente 500 bp de la región de ADN genómico posicionado 3' (derecha) del sitio de integración de interés.
NOTA: La imprimación delantera incluye 20 bp con homología con la región de flanqueo prevista y contiene la secuencia de conectores de 50 bp deseada para permitir el montaje in vivo en la edición Cas12a en el genoma más adelante. La imprimación inversa incluye 20 bp de homología con la región de flanqueo prevista.
- Para la región de flanqueoizquierdo, diseñe imprimadores hacia adelante y hacia atrás para amplificar aproximadamente 500 bp de la región de ADN genómico posicionado 5' (izquierda) del sitio de integración de interés.
- Amplifique las regiones de flanqueo con las imprimaciones diseñadas (por ejemplo, los imprimadores KC-109 a KC-120 incluidos en la Tabla Suplementaria 2).
- Mida la concentración de ADN genómico purificado que servirá como plantilla en el PCR. Ajustar la concentración de ADN a 50 ng /L.
- Preparar mezclas de amplificación de PCR compuestas de ADN genómico (1 – 4 l de dilución de ADN genómico de 50 ng/L) purificados en el paso 4.1, imprimación delantera y inversa (10 m cada una), 1 l de dNTP de 10 mM, 10 ml de 5 x tampón necesario para la polimerasa de ADN, 0,5 l de polimerasa de ADN (1,0 U) , y ultrapuro H2O hasta un volumen total de 50 l.
- Realizar PCRs en un termociclador utilizando el siguiente programa: (i) 98 oC durante 3 min, (ii) 98 oC durante 20 s, (iii) 60 oC para 20 s, (iv) 72 oC para 15 s, repita los pasos (ii) a (iv) 30 veces, (v) 72 oC durante 5 min, , vi) mantener a 12 oC hasta que se pueda seguir analizando.
- Analizar los productos de PCR por electroforesis en un gel de agarosa del 0,8% a 5 V/cm durante 40 minutos utilizando un tinte de carga de ADN y una escalera de ADN con fragmentos de ADN en un rango de 100 a 10.000 bp.
- Purifique los productos pcR correctos utilizando un kit de purificación de PCR de acuerdo con las instrucciones del fabricante.
5. Transformación a S. cerevisiae
NOTA: Realice la transformación utilizando un protocolo basado en los métodos desarrollados por Gietz et al. (1995) 30 y Hill et al. 31 que se puede utilizar para diversas cepas de S. cerevisiae. El protocolo descrito a continuación es suficiente para la transformación 1.
- Preparar las soluciones necesarias para la transformación.
- Preparar las siguientes soluciones de stock y filtrar-esterilizar: 10x tampón TE que contiene 100 mM Tris-HCl (pH 7.5), 10 mM EDTA, volumen total de 50 mL; 1 M LiAc a pH 7,5, volumen total de 50 ml; 50% PEG 4000, volumen total de 100 mL.
NOTA: Compruebe siempre que el stock de PEG 4000 esté en pH 5. Este stock no debe almacenarse más de un mes. - Prepare las siguientes soluciones utilizando stocks: Preparar la solución LiAc-TE que contenga 0,1 M LiAc, 10 mM Tris-HCl, EDTA de 1 mM, volumen total de 0,5 ml. Preparar la solución PEG-LiAc-TE que contiene 40% PEG 4000, 0.1 M LiAc, 10 mM Tris-HCl, EDTA de 1 mM, volumen total de 1 mL.
NOTA: Es crucial para una transformación exitosa que las soluciones PEG-LiAc-TE y LiAc-TE estén recién preparadas.
- Preparar las siguientes soluciones de stock y filtrar-esterilizar: 10x tampón TE que contiene 100 mM Tris-HCl (pH 7.5), 10 mM EDTA, volumen total de 50 mL; 1 M LiAc a pH 7,5, volumen total de 50 ml; 50% PEG 4000, volumen total de 100 mL.
- Primera ronda de transformación (preparar la cepa pre-expresando Cas12a).
NOTA: En todos los pasos de transformación, utilice agua con un pH superior a 5. Se recomienda utilizar agua desmineralizada en todos los pasos de la transformación.- Preparar un precultivo mediante el cultivo de la cepa CEN. PK113-7D en un matraz de batido de 100 ml que contiene 20 ml de medio YEPD (2% glucosa) e incubadurante durante la noche a 30oC con agitación a 250 rpm.
- Mida la OD600 de la precultura (ODpc). Calcular el factor de dilución (df) entre el volumen de precultivo y el volumen de medio fresco necesario para la preparación de las células pre-expresando Cas12a para ser utilizado en la transformación (cultivo de transformación). En los cálculos, supongamos que la densidad óptica del cultivo de transformación (ODtc) es 1,0 después del paso de incubación descrito en 5.2.3 (ti).

donde ti y é son el tiempo de incubación y el tiempo de duplicación, respectivamente.- Calcular el volumen de la precultura (Vi) necesaria para la inoculación de la referencia cultural de transformación (Vtc) en función del factor de dilución.
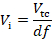
- Calcular el volumen de la precultura (Vi) necesaria para la inoculación de la referencia cultural de transformación (Vtc) en función del factor de dilución.
- Preparar el cultivo de transformación por inoculación de 20 ml de YEPD (2% glucosa) (Vtc) con el volumen de precultivo determinado en el paso anterior (Vi). Incubar a 30oC con agitación a 250 rpm.
- Mida la OD600 de la referencia cultural de transformación hasta que se alcance una OD600 de 1.0.
- Cosecha las células por centrifugación del caldo de 20 ml a 2.500 x g durante 5 min. Deseche el sobrenadante y lave las células en 20 ml de agua desmineralizada a temperatura ambiente. Repita el paso de centrifugación y mantenga el pellet celular.
- Resuspenda las células en 100 ml de solución LiAc-TE y transfiera a un tubo de microcentrífuga.
- Añadir 5 l de ADN portador de una sola cadena (10 mg/ml de ADN esperal del salmón) y mezclar mediante pipeteo.
- Pipetear 1 g de plásmido pCSN067 al tubo de microcentrífuga.
NOTA: El volumen total de la mezcla de ADN no debe exceder los 100 ml para evitar una menor eficiencia de transformación. - Añadir 600 l de solución PEG-LiAc-TE y mezclar mediante pipeteo. Incubar durante 30 min a 30 oC mientras agita a 450 rpm en un bloque de calor de la mesa.
- Añadir 70 l de DMSO (100%) a la mezcla de transformación y mezclar mediante pipeteo. Realizar el choque térmico incubando la mezcla de transformación a 42 oC durante 15 minutos en un baño de agua.
- Recuperar las células transfiriendo la mezcla a un tubo inferior redondo de 15 ml y añadir 10 ml de YEPD (2% glucosa) al tubo. Incubar durante la noche a 30oC con agitación a 250 rpm.
- Centrifugar la mezcla de transformación a 2.500 x g durante 5 min. Deseche el sobrenadante y vuelva a suspender el gránulos celulares en aproximadamente 200 ml de la solución restante.
- Placa de 150 oL de la mezcla de transformación y una dilución de 20x en YEPD (2% glucosa) de mezcla de transformación en placas de agar YEPD (2% glucosa) complementadas con 0,2 g/L G418. Incubar las placas a 30oC durante 48 – 72 horas.
- Escoge una sola placa de agar YEPD (2% glucosa) suplementada con 0,2 g/L G418 para obtener colonias individuales.
- Segunda ronda de transformación (realizar edición del genoma multiplex con CRISPR/Cas12a).
- Preparar un precultivo mediante el cultivo de la cepa pre-expresando Cas12a, creado en la primera ronda de transformación (paso 5.2), en un matraz de batido de 100 ml que contiene 20 mL de medio YEPD (2% glucosa) complementado con 0,2 g/L G418. Incubar durante la noche a 30oC con 250 rpm.
NOTA: Para múltiples transformaciones, adapte el volumen de la precultura. - Siga los pasos 5.2.2 a 5.2.7 para la primera ronda de transformación.
NOTA: Para múltiples transformaciones, adapte los volúmenes de soluciones y cultivos requeridos de la cepa pre-expresando Cas12a. - Pipeta 1 g de la matriz de crRNA única, 1 g del plásmido del receptor linealizado para la matriz de crRNA, 1 g de ADN del donante y 1 g de cada región de flanco (paso 4.3) en el tubo de microcentrífuga.
NOTA: El volumen total de la mezcla de ADN no debe exceder los 100 ml para evitar una menor eficiencia de transformación. - Preparar los siguientes controles para la transformación: control negativo (ultrapure H2O); control positivo para la determinación de la eficiencia de transformación (1 g de pRN1120 circular); un control que verifique si la introducción del ADN del donante se lleva a cabo a través de la edición CRISPR (1 g de pRN1120 circular, 1 g de todos los casetes de expresión de ADN de los donantes y 1 g de regiones de flanqueo, pero sin una sola matriz de CRRNA); controlar la verificación si el ADN del donante puede integrarse fuera del objetivo (1 g de pRN1120 linealizado, 1 g de casetes de expresión de ADN de donante y 1 g de la matriz de crRNA única, pero sin regiones de flanqueo); un control que verifica la linealización completa de pRN1120 (1 g de pRN1120 linealizado).
- Siga los pasos 5.2.9 a 5.2.12 para la primera ronda de transformación.
- Placa de 150 oL de la mezcla de transformación y 20x de dilución en YEPD (2% glucosa) de mezcla de transformación en agar YEPD (2% glucosa) complementado con 0,2 g/L G418 y 0,2 g/L NTC. Controles de placa en el agar YEPD (2% glucosa) complementados con la selección adecuada (G418 y/o NTC o sin selección). Incubar las placas a 30oC durante 48 – 72 horas.
- Escoge un solo transformador de color y vuelve a rayar en una placa de agar YEPD (2% glucosa) para obtener colonias de un solo color.
- Preparar un precultivo mediante el cultivo de la cepa pre-expresando Cas12a, creado en la primera ronda de transformación (paso 5.2), en un matraz de batido de 100 ml que contiene 20 mL de medio YEPD (2% glucosa) complementado con 0,2 g/L G418. Incubar durante la noche a 30oC con 250 rpm.
6. Evaluación de la eficiencia de edición del genoma
- Cuente el número de colonias de colores y colonias blancas en las placas de transformación.
- Calcule la eficiencia de edición del genoma dividiendo el número de colonias de colores por el número total de colonias (tanto blancas como de color), como se muestra en la Tabla1.
7. Confirmación de la integración del ADN de los donantes en los loci previstos
- Re-streak una colonia de color a partir de una placa de transformación en una placa de agar YEPD (2% glucosa) sin selección G418 y NTC e incubar durante 48 horas a 30 oC.
- Escoge una sola colonia e inocula un matraz de batido de 500 ml lleno de 100 ml de medio DeSPD (2% glucosa). Incubar durante 48 horas a 30oC y agitar a 250 rpm.
- Aísle el ADN genómico como se describe en la Sección 4.1.
NOTA: Alternativamente, utilice un protocolo para la preparación de levadura para la PCR de colonia previamente propuesto por Looke et al. 32. En este caso, se puede omitir el crecimiento en medio líquido (sección 7.2). - Verifique la integración correcta mediante la amplificación de dos fragmentos por casete de expresión integrado.
- Diseño de imprimadores que reanneal a ADN genómico fuera de las regiones de flanqueo transformadas y el gen de interés (ver ejemplos en el Cuadro Suplementario 2, KC-121 a KC-132). Cuando utilice los imprimadores KC-121 a KC-132, ajuste la temperatura de recocido en el programa PCR a 62 oC.
- Amplifique la región de interés como se describe en la Sección 4.4.2. Adaptar el programa PCR, ajustar específicamente el tiempo del paso de extensión en PCR de acuerdo con la longitud de la plantilla y las recomendaciones del fabricante para la adn polimerasa.
- Compruebe el tamaño de los productos pcR mediante electroforesis en un gel de agarosa (0,8%, 40 min, 5 V/cm) utilizando un tinte de carga de ADN y una escalera de ADN con fragmentos de ADN en un rango de 100 a 10.000 bp.
8. Creación de arte de píxeles de levadura utilizando un manipulador de líquidos acústicos
- Prepare una plantilla de imagen para el arte de píxeles de levadura.
- Cambie el tamaño de la imagen RGB original (220 x 280 píxeles, consulte los resultados representativos), por ejemplo, utilizando ImageJ para crear una imagen final en escala de grises de 64 a 96 píxeles (ancho a alto) visualizada en colores deseados (resultados representativos).
- Convierta la imagen RGB en escala de grises utilizando esta fórmula:

donde gr, Ir, Ig, Ib son las intensidades gris, rojo, verde y azul, respectivamente. - Para categorizar los píxeles, desarrolle un plugin ImageJ aplicando las siguientes reglas: (a) Si gr es 64, utilice la levadura naranja oscura (strain 1, Supplementary Table 3) para este píxel. (b) Si 64 < Igr á 128, utilice la levadura naranja (strain 2, Supplementary Table 3) para este píxel. (c) Si 128 < Igr á 192, utilice la levadura amarilla (strain 3, Supplementary Table 3) para este píxel. (d) Sigr > 192, utilice la levadura blanca (CEN. PK113-7D) para este píxel.
- Detecte las células de levadura para crear el arte de píxeles de levadura.
- Inocular frascos de batido de 500 ml que contienen 100 ml de medio YEPD (2% glucosa) con tres carotenoides de color diferente que producen cepa S. cerevisiae y tipo salvaje CEN. PK113-7D. Incubar cultivos durante la noche a 30oC con agitación a 250 rpm.
- Transfiera 0,5 ml del cultivo nocturno a un tubo lleno de 0,5 ml de medio de gradiente de densidad no iónica estéril (véase la Tabla de Materiales). Mezclar por vórtice brevemente.
- Transfiera la suspensión celular a un depósito calificado, pozo de 2 x 3. Realice el manchado utilizando un instrumento de manipulador de líquido acústico desde una placa de origen de depósito calificada hasta una microplaca (ver la Tabla de Materiales)que contenga 50 ml de agar YEPD (2% glucosa). Para simplificar el chapado, defina los pozos en la placa, porejemplo. utilizar una microplaca como placa de pozo 6144 (64 x 96).
- Detecte 25 nL de cada cepa de S. cerevisiae de la placa de origen del depósito de 2x 3 pocillos utilizando un archivo .csv con el ajuste de calibración de fluidos 6RES_AQ_GPSA2 en la placa de microplaca de destino. Defina cada una de estas gotas de 25 nL como un píxel en la cuadrícula de 64 x 96 que se traduce a las posiciones del pozo (A01, B01, C01, etc.).
- Incubar la microplaca a 30oC durante 48 horas. Para intensificar los colores de las cepas, guarde la placa de agar a 4 oC durante al menos 72 horas.
Resultados
El protocolo para la edición del genoma multiplex utilizando CRISRP/Cas12a se demostró mediante la construcción de tres cepas de carotenoides que producen S. cerevisiae que expresan los genes crtE, crtYB y crtI utilizando promotores heterólogos de alta, media y baja resistencia: cepa 1, 2 y, 3 respectivamente (Tablacomplementaria 3). La construcción de estas cepas requirió la generación de tres casetes de expresión de ADN de donantes y seis regiones flanqueantes por cepa para apuntar a tres loci diferentes en el ADN genómico (mostrado en la Figura 2B). Como se describe en el presente documento, el promotor, el marco de lectura abierto, el terminador y dos secuencias de conectores contiguas de 50 bps se ensamblaron en un casete de expresión a través de una reacción de clonación de Golden Gate y el montaje fue verificado por PCR (Figura3A). La matriz de crRNA única fue ordenada como un fragmento de ADN sintético y fue amplificada por PCR (Figura3B). El plásmido receptor para la matriz de crRNA única (plasmio pRN1120) se erigió con EcoRI-HF y XhoI y la linealización se confirmó mediante electroforesis (Figura3C). El diseño y las secuencias de nucleótidos de los casetes de expresión de ADN de donantes introducidos y las regiones flanqueantes se muestran en la Tabla Suplementaria 3 yen la Tabla Suplementaria 4. La secuencia de casetes de expresión de matriz crRNA únicos se proporciona en la Tabla Suplementaria 1. La funcionalidad de los espaciadores incluidos en la matriz de crRNA única fue probada previamente por la edición del genoma de un solo plex con crRNAsindividuales 19.
La eficiencia de la edición del genoma utilizando Cas12a se evaluó en primer lugar en función del número de colonias de colores obtenidas después de la transformación (Tabla1, Figura 4). La eficiencia de edición de las tres cepas construidas varió del 50% al 94%. En particular, la introducción de casetes de expresión utilizados para generar la tensión 1 mostró la menor eficiencia de edición, posiblemente causada por la naturaleza del ADN del donante (esdecir, estos casetes de expresión codifican crtE, crtYB y crtI de tres promotores de alta resistencia). En segundo lugar, la PCR (Figura 5) confirmó la correcta integración de los tres casetes de expresión de ADN de los donantes en los loci previstos en el ADN genómico (Figura 5). Los imprimadores se diseñaron de tal manera que se obtuvieron productos de PCR cuando se produjo la correcta integración del ADN del donante en el locus previsto. Para cada experimento de transformación, se recogieron ocho colonias de la placa de transformación y se probaron (tenga en cuenta que solo tres se presentan en la Figura5). En general, de las 8 colonias probadas por ADN de donante, se confirmó la correcta integración del ADN del donante crtE en el locus INT1, crtYB en el locus INT2 y crtI en el locus INT3 en >90% de los transformadores. Estos resultados demuestran que el sistema CRISPR/Cas12a en combinación con una sola matriz crRNA permite la edición multiplexación eficiente del genoma de S. cerevisiae a múltiples loci simultáneamente.
Además, demostramos la creación de "arte de píxeles de levadura" utilizando las tres cepas de producción de carotenoides que fueron construidas junto con una cepa de tipo salvaje no coloreado. A partir de una imagen en blanco y negro de Rosalind Franklin (Figura6A), se creó una imagen de 4 colores (Figura6B) y una lista de manchas que luego se utilizó para detectar las cuatro cepas de levadura diferentes en una microplaca de agar usando un manipulador de líquidos acústicos, lo que resulta en una "pintura de levadura" de alta resolución de Rosalind Franklin (Figura6C,D,E).

Figura 1 : Flujo de trabajo del protocolo para la edición del genoma multiplex CRISPR/Cas12a en S. cerevisiae. El flujo de trabajo incluye pasos cruciales del método presentado. Para obtener más información, consulte el Protocolo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 : Esquema de edición del genoma multiplex CRISPR/Cas12a utilizando una sola matriz crRNA. (A) La matriz de crRNA única se compone de tres unidades crRNAs en su forma madura, una repetición directa de 20 bp específica para LbCas12a (cuadrados grises) con una secuencia guía de 23 bp (diamantes de color). La expresión de la matriz crRNA está habilitada por el promotor SNR52 y el terminador SUP4. Transformación de S. cerevisiae con un pRN1120 linealizado y el casete de expresión de matriz de crRNA único que contiene homología con pRN1120 (rayas diagonales) permite la recombinación in vivo en un plásmido circular en células que pre-expresan LbCas12a. La matriz de crRNA única es procesada posteriormente por Cas12a. (B) Cas12a se dirige a los sitios de destino genómicos INT1, INT2 e INT3 previstos y crea roturas de doble cadena. En la mezcla de transformación, se incluyó el ADN de los donantes que consiste en regiones flanqueantes y el casete de expresión génica carotenoides. Los conjuntos de ADN de donantes se dirigió a un tramo de ADN en ADN genómico alrededor del INT1 (crtE), INT2 (crtYB) e INT3 (crtI) loci por recombinación in vivo debido a la presencia de secuencias de conectores homólogos de 50 bp, 5, A, B, C, D o E. P1–P3, diferentes promotores; T1–T3, diferentes terminadores. Esta cifra ha sido modificada de Verwaal et al. 201819. Construcciones genéticas mostradas utilizando símbolos visuales de biología sintética (SBOL)40. Haga clic aquí para ver una versión más grande de esta figura.
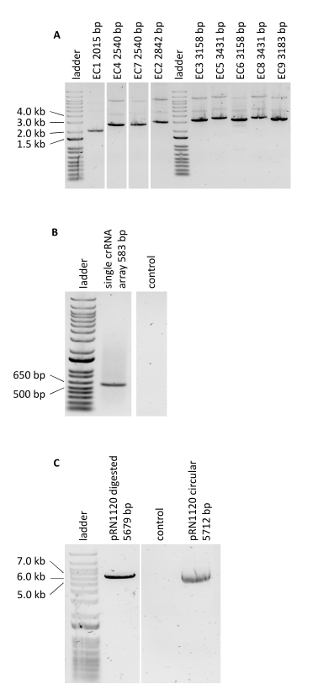
Figura 3 : PCR verificando los experimentos de edición del genoma. (A) Verificación de las reacciones de clonación de Golden Gate de los casetes de ADN de donantes ensamblados. Los resultados obtenidos están de acuerdo con las longitudes esperadas. (B) PCR de la matriz crRNA única. (C) Linealización del plásmido pRN1120. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Placas de transformaciones de S. cerevisiae utilizando el enfoque de edición del genoma multiplex. (A) Cepa 1 que expresa crtE, crtYB y crtI de tres promotores fuertes (colonias de naranja oscura). (B) Cepa 2 que expresa crtE, crtYB y crtI de tres promotores de resistencia media (colonias naranjas). (C) Cepa 3 que expresa crtE, crtYB y crtI de tres promotores de baja resistencia (colonias amarillas). Haga clic aquí para ver una versión más grande de esta figura.

Figura 5 : PCR verificando la integración de los casetes de expresión de ADN del donante en los loci previstos dentro del ADN genómico. (A) Verificación de tres colonias de la cepa 1. (B) Verificación de tres colonias de la cepa 2. (C) Verificación de tres colonias de la cepa 3. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6 : Arte de píxeles de levadura de Rosalind Franklin. (A) Foto RGB en blanco y negro de 220 x 280 píxeles de Rosalind Franklin que se utilizó como plantilla. (B) Conversión por ordenador de la foto en blanco y negro de Rosalind Franklin en una lista de 4 colores de 64 x 96 píxeles. (C) Foto del arte de píxeles de levadura con 64 colonias de levadura de 96 x con una sección ampliada. (D) Foto de un manipulador de líquidos acústicos con dos placas completamente cultivadas. (E) Foto de una microplaca completamente cultivada con 64 colonias de levadura de 96. Haga clic aquí para ver una versión más grande de esta figura.
| Tensión 1 | Tensión 2 | Tensión 3 | |
| Colonias de colores | 16 | 279 | 220 |
| Colonias blancas | 16 | 18 | 18 |
| Colonias totales | 32 | 297 | 238 |
| Eficiencia | 50% | 94% | 92% |
Tabla 1: Eficiencia de edición del enfoque de edición del genoma multiplex.
| secuencia de matriz crRNAa,b,c,d,e,f |
|
CATGTTTGACAGCTTATCATCATCATATCCGGAGCTAGCATGCCCCCTCTCTAGAACTAGTAGATCCCCGCGGCAG TCTTTGAAAA GATAATGTATATGCTTTCACTCATATTATACAGAACTTGATGTTTTCTTTCATGTATATACAAGG TGATTACATGTACGTGTTTGAAGTACAACTCTAGATTTTGTAGTGCCCTTGGGCTAGCGGTAAAGGTGCA TTTTTTTCCCTACTACAATGTTCTGTTCAAAAGATTTTTTTTTGT-AAACGCTGTAAAGTAT ATGTTTCGGCTCTCTCTCGAAACTTCCCCAGTAAAGATAAATGATCAATTTCTAAGTGTAGAT CTGGTGGGAGAGAAAGCTTATGAAATTTCTAAGTGTAGATGTGCCGTAC GCCGGAGCCGACGGAATTTCTAAGTGTAGATTGCCCCTCTTATACGATTATATTTT TTTTTTGTTTTTTATGTCTGGGGGGCCCGGTACCCAGCTTTTTTCCCTTTAGTGAGG GTTAATTCCGAGCTTGGCGTAATCATGGTCATAGCTTCCTGTGTG |
| a. Homología a pRN1120 (valiente). b. Promotor SNR52 (en cursiva). c. Secuencias de destino genómicas (subrayadas). d. Guía de repeticiones directas específicas para LbCas12a (en cursiva, negrita). e. Terminador SUP4 (en cursiva). f. Homología a pRN1120 (negrita). |
Tabla Suplementaria 1: Matriz de crRNA única para LbCas12a que contiene homología con plásmido pRN1120.
| Nombre | Secuenciaa | Descripciónb | Utilizado en el punto |
| KC-101 | CATGTTTGACAGCTTATCATC | Imprimación FW para amplificación de matriz de crRNA única | 2.1.4 |
| KC-102 | CACACAGGAAACAGCTATGAC | Imprimación RV para amplificación de una sola matriz crRNA | 2.1.4 |
| KC-103 | AAGCGACTTCCAATCGCTTTGC | Imprimación FW para amplificación del ADN del donante con conector 5 | 3.6.1 |
| KC-104 | AAAGCAAAGGAAGGAGAGAAC | Imprimación RV para amplificación del ADN del donante con conector A | 3.6.1 |
| KC-105 | CGGATCGATGTACACAACCG | Imprimación FW para amplificación del ADN del donante con conector B | 3.6.1 |
| KC-106 | CAACAGGAGGCGGATGGATATAC | Imprimación RV para amplificación del ADN del donante con conector C | 3.6.1 |
| KC-107 | AACGTTGTCCAGGTTTGTATCC | Imprimación FW para amplificación del ADN del donante con conector D | 3.6.1 |
| KC-108 | AGGTACAACAAGCACGACCG | Imprimación RV para amplificación del ADN del donante con conector E | 3.6.1 |
| KC-109 | CACTATAGCAATCTGGCTATATG | Imprimación FW para amplificación de INT1 5' con conector 5 | 4.4 |
| KC-110 |
AAACGCCTGTGGGTGGTAC TGGATATGCAAAGCGATTGGAA GTCGCTTGACTCCTCTGCCGTC ATTCC | Imprimación RV para amplificación de INT1 5' con conector 5 | 4.4 |
| KC-111 |
TTGCCCATCGAACGTACAAG TACTCCTCTGTCTCTCCTTCCTCCTTTT TGCTTTAAGCGTTGAAGTTCCTC TTTG | Imprimación FW para amplificación de INT1 3' con conector A | 4.4 |
| KC-112 | TGTCAACTGGAGAGCTATCG | Imprimación RV para amplificación de INT1 3' con conector A | 4.4 |
| KC-113 | AGAAGATTTCTCTCTCAATCTC | Imprimación FW para amplificación de INT2 5' con conector B | 4.4 |
| KC-114 |
TGCTAAGATTTGTGTGTGTT TGGGTGCAGTCGGTTGTACAT CGATCCGCCCTTATCAAGGATACC TGGTTG | Imprimación RV para amplificación de INT2 5' con conector B | 4.4 |
| KC-115 |
ACGCTTTCCGGCATCTTCCA GACCACAGTATATCCATCCGCCT CCTGTTGGGCGATTACACAAGCG GTGG | Imprimación FW para amplificación de INT2 3' con conector C | 4.4 |
| KC-116 | TCTCCTCTTCGATCGCGGG | Imprimación RV para amplificación de INT2 3' con conector C | 4.4 |
| KC-117 | GGTCGTTTTTGTGCAGCATATTG | Imprimación FW para amplificación de INT3 5' con conector D | 4.4 |
| KC-118 |
GCGGAATATTGGCGGAACGG ACACACGTGGATACAAACCTG GACAACGTTTTCCAAGGAGGTG AAGAACG | Imprimación RV para amplificación de INT3 5' con conector D | 4.4 |
| KC-119 |
AAATAACCACAAACCTT CCCATATGCTCGGTCGTGCTTGTT GTACCTGATGGGACGTCAGCACT GTAC | Imprimación FW para amplificación de INT3 3' con conector E | 4.4 |
| KC-120 | GAGCTTACTCTATATTCATTC | Imprimación RV para amplificación de INT3 3' con conector E | 4.4 |
| KC-121 | GTTACTAAACTGGAACTCCCCG | Imprimación FW para la verificación de la integración de con5-crtE-conA a INT1 5' | 7.4.1 |
| KC-122 | CACTGCTAACTACGTTACTTC | Imprimación FW para la verificación de la integración de con5-crtE-conA a INT1 3' | 7.4.1 |
| KC-123 | CACTGGAACTTGAGCTTGAG | Imprimación FW para la verificación de la integración de conB-crtYB-conC a INT2 5' | 7.4.1 |
| KC-124 | GTCTCCAGCTGAATTGGTCC | Imprimación FW para la verificación de la integración de conB-crtYB-conC a INT2 3' | 7.4.1 |
| KC-125 | CTCTCATGAAGCAGTCAAGTC | Imprimación FW para la verificación de la integración de conD-crtI-conE a INT3 5' | 7.4.1 |
| KC-126 | GATCGGTCAATTAGGTGAAG | Imprimación FW para la verificación de la integración de conD-crtI-conE a INT3 3' | 7.4.1 |
| KC-127 | CCTTGTCCAAGTAGGTCC | Imprimación RV para la verificación de la integración de con5-crtE-conA a INT1 5' | 7.4.1 |
| KC-128 | GCTGTCATGATCTGTGATAAC | Imprimación RV para la verificación de la integración de con5-crtE-conA a INT1 3' | 7.4.1 |
| KC-129 | CTGGCAATGTTGACCAATTGC | Imprimación RV para la verificación de la integración de conB-crtYB-conC a INT2 5' | 7.4.1 |
| KC-130 | CCAACGTGCCTTAAAGTCTG | Imprimación RV para la verificación de la integración de conB-crtYB-conC a INT2 3' | 7.4.1 |
| KC-131 | CCTTACCTTCTGGAGCAGCAG | Imprimación RV para la verificación de la integración de conD-crtI-conE a INT3 5' | 7.4.1 |
| KC-132 | CTGGTTACTTCCCTAAGACTG | Imprimación RV para la verificación de la integración de conD-crtI-conE a INT3 3' | 7.4.1 |
| a. Las secuencias en negrita denotan secuencias de conectores. b. Los imprimadores delanteros y inversos se designan como FW y RV, respectivamente. |
Tabla Suplementaria 2: Secuencias de imprimación.
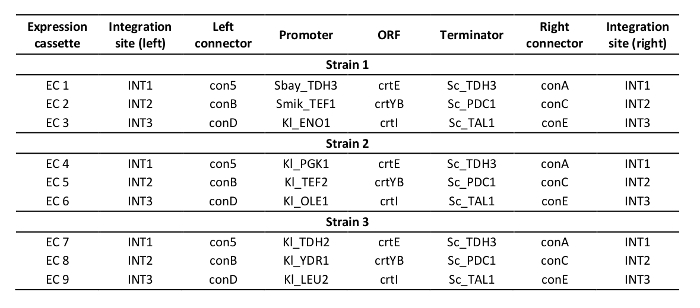
Tabla Complementaria 3: Diseño de cepas construidas.

Tabla Suplementaria 4: Secuencias de casetes de expresión de ADN de donantes y regiones de descamación. Haga clic aquí para descargar este archivo.
Discusión
El protocolo proporcionado describe la edición del genoma multiplex de S. cerevisiae utilizando Cas12a de la bacteria Lachnospiraceae ND2006 en combinación con una sola matriz de CRRNA y ADN de donante. El diseño de la matriz de crRNA única y el ADN de los donantes se explica en detalle. A diferencia del bien establecido sistema CRISPR/Cas9, el CRISPR/Cas12a tiene la capacidad adicional única de procesar múltiples crRNAs expresados desde una única matriz de crRNA13,33. Debido a esta característica, la edición simultánea de varios destinos es más fácil de configurar y se puede lograr en una sola transformación. Este enfoque de matriz de crRNA único fue demostrado antes por Zetsche et al. 34 que editaron simultáneamente hasta cuatro genes en células de mamíferos usando AsCas12a, y por Swiat et al. 35 que introdujeron cuatro fragmentos de ADN en un genoma de levadura usando FnCas12a. Hasta nuestro punto, no se ha informado de un mayor número de modificaciones genómicas simultáneas utilizando un sistema Cas12a y el límite máximo de objetivos por matriz única para Cas12a está aún por determinar. Investigaciones adicionales utilizando matrices de crRNA individuales en combinación con Cas12a incluye regulación transcripcional multiplex en una amplia gama de organismos33,36,37.
Hay algunos pasos críticos en el protocolo presentado. Diseñe cuidadosamente todas las secuencias de ADN que intervienen en el experimento de edición del genoma Cas12a, especialmente en el caso de que se introduzcan secuencias de ADN novedosas. Determinar la funcionalidad de las nuevas secuencias espaciadoras que forman parte de un crRNA, por ejemplo mediante un experimento de edición del genoma de un solo plex como se describe en Verwaal et al. 19 antes de combinarlos en una sola matriz crRNA. Siga las recomendaciones para la preparación de soluciones de búfer de transformación utilizadas en el experimento de edición Cas12a para lograr una buena eficiencia de transformación de la levadura.
Hay algunas modificaciones opcionales de la técnica. Se recomienda utilizar 1 g de ADN de cada donante, pRN1120 linealizado o casete de expresión de matriz de crRNA único en la transformación, aunque también se espera que el uso de una cantidad de ADN menor resulte en una eficiencia de transformación satisfactoria. Realice una transformación de prueba para determinar si se pueden utilizar cantidades de ADN más bajas. La transformación de S. cerevisiae podría realizarse utilizando un método diferente al descrito en este protocolo, por ejemplo, el protocolo descrito por Gietz et al. (2007) 38. El plásmido receptor de ARN guía pRN1120 es adecuado para la expresión deun solo crRNA y una matriz de crRNA único de diferentes variantes Cas12a (por ejemplo, , de Acidaminococcus spp. BV3L6 o Francisella novicida U112) así como para la expresión de sgRNA en combinación con Cas919. No es necesario limitar el ADN de los donantes a los casetes de expresión génica carotenoides y a las regiones flanqueantes que dirigen el ADN de los donantes a los sitios INT1, INT2 e INT3 descritos en el ADN genómico. Cualquier ADN de interés puede introducirse, de manera multiplexado, en el ADN genómico del huésped mediante los principios de diseño descritos en este protocolo, o alternativamente el ADN del donante se puede utilizar para eliminar el ADN de un genoma del huésped. La estructura modular de la matriz de crRNA simple facilita el ajuste de las secuencias de repetición directa y espaciadora. La modificación de las secuencias de espaciadores permite un cambio del locus de integración previsto que puede ser diseñado por una de las herramientas para la identificación de un sitio de destino genómico, por ejemplo, el software GuideScan 1.039. En lugar de utilizar grandes secuencias de flanqueo que contienen secuencias de conectores, 50-bp de la región de flanqueo se pueden incluir en las secuencias de ADN del donante mediante la incorporación de estas secuencias de región de flanqueo de 50 bp en las imprimaciones utilizadas en el PCR. En este caso, en total sólo se requieren tres en lugar de nueve fragmentos de ADN de donantes para un experimento exitoso de edición del genoma multiplex.
En resumen, este protocolo proporciona instrucciones paso a paso para realizar la edición del genoma multiplex en S. cerevisiae utilizando Cas12a en combinación con un enfoque de matriz crRNA único. El protocolo fue demostrado mediante la edición del genoma multiplex utilizando 9 fragmentos de ADN de donantes y codificación de matriz de crRNA única para tres gRNAs. Mostramos altas frecuencias de edición general entre 50% y 94% para los tres diseños de tensión reportados aquí. Concluyendo, la característica única de Cas12a es la capacidad de procesar una sola matriz crRNA en crRNA individuales en una celda, lo que hace de Cas12a una excelente herramienta para permitir la edición del genoma multiplex y desarrollar módulos de regulación transcripcional dirigidos a múltiples expresión casetes de una sola vez. Al final, se obtuvieron tres cepas produciendo carotenoides a un nivel diferente y colores en tonos entre amarillo y naranja. Con esas cepas y una cepa de tipo salvaje, mostramos cómo un manipulador de líquido acústico se puede utilizar directamente para hacer arte de píxeles de levadura - esto en honor de Rosalind Franklin que contribuyó al descubrimiento de la estructura de ADN hace 65 años por su famosa foto 5123< /c1>.
Divulgaciones
Los autores declaran que hay un conflicto de intereses. Los autores han presentado una IP relacionada con los métodos presentados.
Agradecimientos
Este proyecto recibió financiación del programa de investigación e innovación Horizonte 2020 de la Unión Europea en virtud del acuerdo de subvención no 686070 (DD-DeCaf) y 764591 (SynCrop), y del programa de investigación Building Blocks of Life con el número de proyecto 737.016.005 por organización neerlandesa de investigación científica (NWO). T.E.G. contó con el apoyo de la Royal Society (concesión UF160357) y BrisSynBio, un Centro de Investigación en Biología Sintética BBSRC/EPSRC (subvención BB/L01386X/1). Agradecemos a Zi Di y Jeffrey van Wijk por su contribución a los experimentos de detección de levadura para crear el arte de píxeles de levadura.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals specific for the protocol | |||
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | Electrophoresis |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | Selection of E. coli transformants |
| BsaI-HF (20 U/µl) | New England BioLabs | R353L | Golden Gate Cloning |
| Cell Lysis Solution (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| CutSmart Buffer | New England BioLabs | B7204S | Linearization of pRN1120 |
| Deoxyribonucleic acid sodium salt from salmon testes | Sigma Aldrich | D1626 | Transfromation of S. cerevisiae (carrier DNA) |
| dNTPs | Invitrogen | 10297018 | PCRs |
| EcoRI-HF | New England BioLabs | R3101S | Linearization of pRN1120 |
| Ethanol absolute for analysis | Merck | 100983 | Isolation of genomic DNA from S. cerevisiae |
| Ethylenediamine-tetraacetic acid | Sigma Aldrich | ED | Transformation of S. cerevisiae |
| G418 disulfate salt | Sigma Aldrich | A1720 | Selection of S. cerevisiae transformants |
| Histodenz | Sigma Aldrich | D2158 | Yeast pixel art |
| Isopropanol | Merck | 100993 | Isolation of genomic DNA from S. cerevisiae |
| Lithium acetate dihydrate | Sigma Aldrich | L6883 | Transformation of S. cerevisiae |
| Nancy-520 DNA Gel Stain | Sigma Aldrich | 1494 | Electrophoresis |
| NEB10 competent E. coli cells | New England BioLabs | C3019H | Transformation of E. coli: dx.doi.org/10.17504/protocols.io.nkvdcw6 |
| Nourseothricin | Jena Bioscience | AB102 | Selection of S. cerevisiae transformants |
| Phusion buffer | New England BioLabs | M0530L | PCRs |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530L | PCRs |
| Polyethylene glycol 4000 | Merck | 7490 | Transformation of S. cerevisiae |
| Protein Precipitation Solution (10 M NH4AC) (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| Purple loading dye | New England BioLabs | B7024S | Electrophoresis |
| QIAprep Spin Miniprep Kit | QIAGEN | 27106 | Purification of plasmids |
| RNase coctail enzyme mix | Thermo Fisher Scientific | AM2286 | Isolation of genomic DNA from S. cerevisiae |
| T4 DNA ligase buffer | Invitrogen | 46300-018 | Golden Gate Cloning |
| T4 DNA Ligase (1 U/µl) | Invitrogen | 1705218 | Golden Gate Cloning |
| UltraPure Agarose | Invitrogen | 16500500 | Electrophoresis |
| Wizard SV Gel and PCR Clean-Up System Kit | Promega | A9282 | Purification of PCR products and linearized pRN1120 |
| Xhol | New England BioLabs | R0146S | Linearization of pRN1120 |
| Zymolyase 50 mg/ml (5 units/µL) | Zymo Research | E1006 | Isolation of genomic DNA from S. cerevisiae (yeast lysis enzyme) |
| Zymolyase storage buffer | Zymo Research | E1004-B | Isolation of genomic DNA from S. cerevisiae (necessary for the preparation of yeast lysis enzyme) |
| Chemicals of general use | |||
| 2*Peptone-Yeast extract (PY) agar | Plate growth of E. coli | ||
| 2*PY medium | Cultivation of E. coli | ||
| Demineralized water | Transformation of S. cerevisiae | ||
| ELFO buffer | Electrophoresis | ||
| MQ | Multiple steps | ||
| Physiological salt solution | Transformation of S. cerevisiae | ||
| TE buffer | Storage of DNA, transformation of S. cerevisiae | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | Cultivation of S. cerevisiae | ||
| YEPD (2% glucose) agar | Plate growth of S. cerevisiae | ||
| Consumables | |||
| Eppendorf tubes | |||
| Falcon tubes (50 mL) | |||
| Microplate 96 wells | |||
| Petri dishes | |||
| Pipette tips 0.5 - 10 µL | |||
| Pipette tips 10 - 200 µL | |||
| Pipette tips 100 - 1000 µL | |||
| Shake flasks (500 mL) | |||
| Sterile filters | |||
| Equipment | |||
| Centrifuge (Falcon tubes) | |||
| Echo 525 acoustic liquid handler | |||
| Incubator | |||
| NanoDrop | |||
| Set for eletrophoresis | |||
| Spectrophotometer | |||
| Table centrifuge (Eppendorfs tubes) | |||
| Thermocycler | |||
| Plasmids | |||
| pCSN067 | Addgene | ID 101748 | https://www.addgene.org/ |
| pRN1120 | Addgene | ID 101750 | https://www.addgene.org/ |
| Strains | |||
| S. cerevisiae strain CEN.PK113-7D | EUROSCARF collection | http://www.euroscarf.de |
Referencias
- Knott, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361 (6405), 866-869 (2018).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Lian, J., HamediRad, M., Hu, S., Zhao, H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications. 8 (1), 1688 (2017).
- Li, Z. -. H., Liu, M., Lyu, X. -. M., Wang, F. -. Q., Wei, D. -. Z. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology. 58 (12), 1100-1104 (2018).
- Shao, Y., Lu, N., Qin, Z., Xue, X. CRISPR-Cas9 facilitated multiple-chromosome fusion in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (11), 2706-2708 (2018).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology. 13 (11), 722-736 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), (2016).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovič, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Lian, J., HamediRad, M., Zhao, H. Advancing metabolic engineering of Saccharomyces cerevisiae. using the CRISPR/Cas System. Biotechnology Journal. 13 (9), 1700601 (2018).
- Ferreira, R., et al. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (1), 10-15 (2018).
- Swarts, D. C., Martin, J. Cas9 versus Cas12a/Cpf1: Structure–function comparisons and implications for genome editing. Wiley Interdisciplinary Reviews: RNA. 9 (5), 1481 (2018).
- Strohkendl, I., Saifuddin, F. A., Rybarski, J. R., Finkelstein, I. J., Russell, R. Kinetic Basis for DNA Target Specificity of CRISPR-Cas12a. Molecular Cell. 71 (5), 816-824 (2018).
- Verwaal, R., et al. High-level production of beta-carotene in Saccharomyces cerevisiae. by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous. Applied and Environmental Microbiology. 73 (13), 4342-4350 (2007).
- Verwaal, R., Buiting-Wiessenhaan, N., Dalhuijsen, S., Roubos, J. A. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast. 35 (2), 201-211 (2018).
- Jakociunas, T., Jensen, M. K., Keasling, J. D. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering. 34, 44-59 (2016).
- Engler, C., Romy, K., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PloS One. 3 (11), 3647 (2008).
- Franklin, R. E., Gosling, R. G. Molecular configuration in sodium thymonucleate. Nature. 171, 740-741 (1953).
- Watson, J. D., Crick, F. H. A structure for deoxyribose nucleic acid. Nature. 171, 737-738 (1953).
- Wilkins, M. H. F., Stokes, A. R., Wilson, H. R. Molecular structure of deoxypentose nucleic acids. Nature. 171, 738-740 (1953).
- Young, E. M., et al. Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast. Metabolic Engineering. 48, 33-43 (2018).
- Roubos, J. A., Pel, H. J., Meijrink, B. . Cloning Method. , (2013).
- Mandel, M., Higa, A. Calcium-dependent bacteriophage DNA infection. Journal of Molecular Biology. 53 (1), 159-162 (1970).
- Van Dijken, J. P., et al. An interlaboratory comparison of physiological and genetic properties of four Saccharomyces cerevisiae strains. Enzyme and Microbial Technology. 26 (9-10), 706-714 (2000).
- Gietz, R. D., Schiestl, R. H., Willems, A. R., Woods, R. A. Studies on the transformation of intact yeast cells by the LiAc/SS-DNA/PEG procedure. Yeast. 11 (4), 355-360 (1995).
- Hill, J., Donald, K. A., Griffiths, D. E., Donald, G. DMSO-enhanced whole cell yeast transformation. Acids Research. 19 (20), 5791 (1991).
- Looke, M., Kristjuhan, K., Kristjuhan, A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques. 50 (5), 325-328 (2011).
- Tak, Y. E., et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nature Methods. 14 (12), 1163-1166 (2017).
- Zetsche, B., et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature biotechnology. 35 (1), 31-34 (2017).
- Swiat, M. A., et al. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research. 45 (21), 12585-12598 (2017).
- Li, L., et al. CRISPR-Cpf1-Assisted Multiplex Genome Editing and Transcriptional Repression in Streptomyces. Applied Environmental Microbiology. 84 (18), 00827-00918 (2018).
- Zhang, X., et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery. 3, 17018 (2017).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 1-4 (2007).
- Perez, A. R., et al. GuideScan software for improved single and paired CRISPR guide RNA design. Nature Biotechnology. 35 (4), 347-349 (2017).
- Cox, R. S., et al. Synthetic Biology Open Language Visual (SBOL Visual) Version 2.0. Journal of Integrative Bioinformatics. 15 (1), 1613-4516 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados