Method Article
CRISPR/Cas12a Multiplex Genome Editing of Saccharomyces cerevisiae and the Creation of Yeast Pixel Art CRISPR/Cas12a Multiplex Genome Editing of Saccharomyces cerevisiae and the Creation of Yeast Pixel Art CRISPR/Cas12a Multiplex Genome Editing of Saccharomyces cerevisiae and the Creation of Yeast Pixel Art CRISPR
Dans cet article
Résumé
Le système CRISPR/Cas12a, combiné à un seul réseau de crRNA, permet une édition multiplexe efficace du génome de S. cerevisiae à plusieurs loci simultanément. Ceci est démontré par la construction de caroténoïdes produisant des souches de levure qui sont ensuite utilisés pour créer de l'art pixel levure.
Résumé
L'efficacité élevée, la facilité d'utilisation et la polyvalence du système de répétitions palindromic courtes et de courte durée interespacement interespacement interspaced/CRISPR-associated protein 9 (CRISPR/Cas9) a facilité la modification génétique avancée de Saccharomyces cerevisiae, un modèle l'organisme et le cheval de bataille de la biotechnologie industrielle. La protéine 12a (Cas12a), une endodoucane guidée par l'ARN dont les caractéristiques se distinguent de Cas9 est appliquée dans ce travail, élargissant davantage la boîte à outils moléculaire à des fins d'édition du génome. Un avantage du système CRISPR/Cas12a est qu'il peut être utilisé dans l'édition du génome multiplexe avec plusieurs ARN guides exprimés à partir d'une seule unité transcriptionnelle (single CRISPR RNA (crRNA) tableau). Nous présentons un protocole pour l'intégration multiplexe des gènes hétérologues multiples dans les loci indépendants du génome de S. cerevisiae utilisant le système CRISPR/Cas12a avec les crRNAs multiples exprimés à partir d'une construction unique de tableau de CRRNA. La méthode proposée exploite la capacité de S. cerevisiae à effectuer une recombinaison in vivo de fragments d'ADN pour assembler le tableau de crRNA unique en un plasmide qui peut être utilisé pour la sélection des transformateurs, ainsi que l'assemblage de l'ADN du donneur séquences qui s'intègrent dans le génome aux positions prévues. Cas12a est pré-exprimé constitutivement, facilitant le clivage du génome de S. cerevisiae aux positions prévues sur l'expression du tableau unique de crRNA. Le protocole comprend la conception et la construction d'un tableau unique de crRNA et de cassettes d'expression d'ADN des donneurs, et exploite une approche d'intégration faisant usage de séquences uniques de connecteurs d'ADN de 50 pb et de séquences d'ADN séparées de flanc d'intégration, ce qui simplifie conception expérimentale par la normalisation et la modularisation et élargit la gamme d'applications. Enfin, nous démontrons une technique simple pour créer l'art de pixel de levure avec un gestionnaire acoustique liquide utilisant le caroténoïde différemment coloré produisant des souches de levure qui ont été construites.
Introduction
Les enzymes CRISPR/Cas ont incontestablement révolutionné la biologie moléculaire et ont été largement adoptées comme outils pour l'ingénierie des génomes à une vitesse qui était auparavant irréalisable1. La première modification d'un génome de Saccharomyces cerevisiae par le système d'édition du génome CRISPR/Cas9 a été rapportée par DiCarlo et al. 2, démontrant le knock-out réussi de gène et faisant des mutations de point utilisant des oligonucléotides introduits externement. D'autres développements de boîte à outils de la levure CRISPR inclus: régulation transcriptionnelle par fusion de Catalyically inactif mort Cas9 (dCas9) avec des domaines effecteurs transcriptionnels pour permettre l'activation et le silence de la transcription3, application pour les deux l'édition du génome et les fonctions réglementaires pour l'ingénierie des voies métaboliques par activation simultanée, répression et suppression4, suppression de gros fragments du génome de S. cerevisiae 5, et fusions multichromosome6 .
Les systèmes d'édition du génome CRISPR/Cas trouvent leur origine dans les systèmes immunitaires adaptatifs des bactéries et des archées et ces systèmes ont été adaptés par des biologistes moléculaires pour l'édition du génome. Leur fonctionnalité est basée sur les régions d'ADN de répétitions palindromic courtes (CRISPR) encodant l'ARN de l'ADN ou de l'ARN étranger et les gènes associés au CRISPR (Cas) qui codent l'ARN-guidé endonucleases1,7,8,9. Sur la base de l'analyse récente du génome des systèmes CRISPR/Cas, il a été proposé de diviser les systèmes CRISPR/Cas en deux classes, cinq types et 16 sous-types10. Les deux classes sont distinguées en fonction de l'organisation de complexes effecteurs impliqués dans le clivage cible. En règle générale, les systèmes CRISPR/Cas dotés d'une organisation multi-sous-unitit sont classés en classe 1, tandis que les complexes d'effecteurs de sous-unité unique appartiennent à la classe 210,11. Dans cet article, nous explorons la classe 2 de type V Cas12a, anciennement appelé Cpf110,12, qui est une alternative à la classe 2 type II Cas9. Bien que Cas9 est bien caractérisé et largement utilisé dans la recherche, Cas12a offre des fonctionnalités supplémentaires12. Tout d'abord, Cas12a forme un complexe avec DE l'ARN CRISPR (ARN) de 42 à 44 nucléotides sans avoir besoin d'un ARN CRISPR transactivant supplémentaire (tracrRNA). Par conséquent, un ARN guide plus court peut être utilisé dans l'édition du génome avec des systèmes CRISPR/Cas12a par rapport à CRISPR/Cas9. Deuxièmement, l'activité unique d'endouclease et d'endoribonuclease de Cas12a permet la maturation de son pré-crRNA13. Cette activité RNase permet l'encodage de plusieurs CRRNAs sur un seul tableau DE CRRNA CRISPR, tandis que Cas9 nécessite l'expression séparée de chaque ARN dit unique (sgRNA) ou alternativement par exemple l'expression d'une endonuca supplémentaire ( p. ex., Csy4) en combinaison avec des motifs de reconnaissance pour Csy4 entourant chaque sgRNA14,15. Troisièmement, la reconnaissance du site cible Cas12a nécessite un motif adjacent protospacer (PAM) à l'extrémité 5' de la cible et s'enprend après la position de 18/23 de son PAM résultant en l'ADN clivé avec des extrémités collantes, tandis que Cas9 nécessite un PAM situé sur l'extrémité 3' de la cible et s'enlisse après la position -3 créant des coupes d'extrémité émoussées dans l'ADN12. Quatrièmement, la séquence de nucléotide de consensus de la PAM diffère entre Cas12a ((T)TTV) et Cas9 (NGG), ce qui fait cas12a un candidat prometteur pour cibler les séquences de promoteur et de terminaison T-rich16. Enfin, une étude récente a fait état d'une plus grande spécificité cible pour Cas12a que pour le Cas917natif .
Nous présentons un protocole pour l'utilisation du système CRISPR/Cas12a pour l'édition du génome de S. cerevisiae avec un accent particulier sur l'introduction de cassettes d'expression d'ADN multiples dans des loci génomiques indépendants simultanément (édition du génome multiplex) en utilisant un tableau crRNA unique. Les étapes clés du protocole sont représentées à la figure 1. Comme preuve de concept, le système CRISPR/Cas12a a été appliqué pour l'introduction de trois cassettes d'expression dans le génome de S. cerevisiae qui permettent la production de carotène18 comme le montre schématiquement la figure 2. La production de carotène affecte le phénotype de S. cerevisiae: c.-à-d., lors de l'introduction réussie des trois gènes hétérologues requis pour la biosynthèse des caroténoïdes, les cellules blanches de S. cerevisiae deviennent jaunes ou oranges, en fonction de la force d'expression du promoteur de chaque gène. En raison de la simple lecture visuelle de cette voie, il a été introduit pour développer des systèmes et des méthodes avancés à base de CRISPR pour l'édition du génome19,20. Dans ce travail, cassettes d'expression codant les gènes caroténoïdes crtE, crtYB et crtI ont été construits à l'aide d'un clonage Golden Gate (GGC) approche21 avec des promoteurs hétérologues et terminators homologues utilisé pour stimuler l'expression des gènes. Les cassettes d'expression sont entourées de séquences uniques de paires de 50 bases (bp), appelées connecteurs, qui permettent un assemblage in vivo avec des séquences d'ADN de flanc d'intégration (régions de flanc) avec les mêmes séquences de 50 pb, et l'intégration ultérieure dans l'ADN génomique de levure à la position déterminée par les régions de flanc. En utilisant différentes forces de promoteur, des souches avec différents niveaux de production de caroténoïdes ont été obtenues résultant en une variation de la couleur des cellules. Ces souches - inspirées par le "Yeast Art Project"22 - ont été utilisées dans une installation de repérage avec un gestionnaire de liquide acoustique pour créer une 4 couleurs haute résolution "photographie de levure" de Rosalind Franklin. Franklin (1920-1958) était une chimiste anglaise et cristallographe à rayons X bien connue pour sa contribution à la découverte de la structure de l'ADN par la photo 5123,24,25.
Protocole
1. Préparation des plasmides Cas12a
REMARQUE: Le plasmide contenant la bactérie Lachnospiraceae ND2006 Cas12a (LbCpf1, pCSN067) codon optimisé pour l'expression dans S. cerevisiae, a été précédemment construit19, déposé à un dépôt plasmide (voir le tableau de Matériaux). Il s'agit d'un plasmide épisomal de s. cerevisiae/E. coli de navette de S. cerevisiae / E. coli contenant un gène de marqueur de résistance de KanMX pour permettre la sélection des transformateurs de S. cerevisiae sur la génétique (G418).
- Obtenir le plasmide pCSN067 (voir le Tableau des matériaux).
- Amplifiez le plasmide pCSN067 pour obtenir une quantité élevée.
- Transformez 25 L de cellules E. coli chimiquement compétentes achetées avec le plasmide pCSN067 selon le protocole du fabricant. Diluer le mélange de transformation 10 et 50 fois dans 2x peptone-levure (PY). Déposer les dilutions 10x et 50x sur les plaques d'agar PY 2x contenant de l'ampicilline (0,1 g/L) et couver toute la nuit à 37 oC.
- Choisissez 2 à 3 colonies et inoculez chaque colonie dans 3 ml de 2 x PY et poussez pendant la nuit à 37 oC dans un incubateur secouant à 180 tr/min.
- Purifez le plasmide à l'aide d'un kit de purification plasmide selon les instructions du fabricant.
2. Préparation de la cassette d'expression de tableau de crRNA simple
- Préparer le tableau crRNA unique.
REMARQUE : Le tableau de crRNA simple comprend un promoteur de polymérase III d'ARN SNR52 de S. cerevisiae2, une répétition directe spécifique pour LbCas12a et un espaceur (séquence de cible génomique), répétés pour chaque cible19 et se termine avec un terminateur SUP4 de S. cerevisiae2. Le tableau de crRNA unique est assemblé par recombinaison in vivo dans le plasmide linéaire pRN1120 pour générer un plasmide circulaire, donc les régions homologues au plasmide pRN1120 doivent être présentes au début et à la fin du tableau de crRNA unique (voir figure 2A ). Il est recommandé d'évaluer à l'avance la fonctionnalité d'un certain nombre de CRRNAs conçus séparément19. Ces informations sont ensuite utilisées pour sélectionner la plupart des crARN fonctionnels pour les combiner dans les séquences de répétition directe et d'espaceur pour créer un tableau de crRNA unique pour le but de multipler.- Commandez le tableau de crRNA unique pour les expériences d'édition du génome multiplex sous forme d'ADN synthétique (voir la séquence d'ADN du tableau unique de l'ARN dans le tableau supplémentaire 1).
- Amplifiez le tableau crRNA unique commandé(p. ex., à l'aide des amorces KC-101 et KC-102 (tableau supplémentaire 2)). Préparer le mix d'amplification PCR contenant : 0,5 L de polymérase d'ADN, 10 l de tampon de 5x requis pour la polymérase d'ADN, 1 L de 10 mL de dNTP, 2,5 L d'amorce avant de 10 M, 2,5 L de 10 m d'amorce inverse, 2 l de modèle d'ADN à une concentration de 5 ng/L et ultrapure H 2 (en) O jusqu'à un volume total de 50 L.
- Effectuer la réaction dans un thermocycleur en utilisant le programme suivant : (i) 98 oC pour 3 min, (ii) 98 oC pour 10 s, (iii) 60 oC pour 20 s, (iv) 72 oC pour 15 s - étapes répétées (ii) à (iv) 30 fois, (v) 72 oC pour 5 min (vi) tenir à 12 oC jusqu'à une analyse plus poussée.
- Analyser les produits PCR par électrophorèse en exécutant les échantillons sur un gel d'agarose de 0,8 % à 5 V/cm pendant 40 min à l'aide d'un colorant de chargement d'ADN et d'une échelle d'ADN avec des fragments d'ADN dans une fourchette de 100 à 10 000 pb.
- Purifie les produits PCR à l'aide d'un kit de purification PCR selon les instructions du fabricant.
- Préparer le plasmide récepteur du tableau crRNA unique.
REMARQUE : Le tableau de l'ARRC unique est exprimé à partir du plasmique plasmique plasmique plasmide pLAsmid pRN112019 (voir le Tableau des matériaux). Ce plasmide multi-copie contient un gène marqueur de résistance NatMX pour permettre la sélection des transformateurs de S. cerevisiae sur la nourseothricine (NTC).- Obtenir le plasmide pRN1120.
- Amplifiez le plasmide pRN1120 pour obtenir une quantité élevée.
- Transformer 25 l de cellules E. coli achetées chimiquement compétentes avec plasmide pRN1120 selon le protocole du fabricant. Diluer le mélange de transformation 10 et 50 fois en 2x PY. Déposer les dilutions 10x et 50x sur les plaques d'agar PY 2x contenant de l'ampicilline (0,1 g/L) et couver toute la nuit à 37 oC.
- Choisissez 2 à 3 colonies et inoculez chaque colonie dans 3 ml de 2 x PY et poussez pendant la nuit à 37 oC dans un incubateur secouant à 180 tr/min.
- Purifez le plasmide à l'aide d'un kit de purification plasmide selon les instructions du fabricant.
- Linéarisez plasmid pRN1120 avec EcoRI-HF et XhoI. Pour cela, préparer un mélange de digestion composé de 1 g de pRN1120, 5 'L de tampon 10x (1x tampon contient 50 mM d'acétate de potassium, 20 mM d'acétate tris, 10 mM d'atate de magnésium, 100 g/mL d'albumine de sérum bovin [BSA]; pH 7,9), 1 'L d'Eco RI-HF (20 U) , 1 oL de XhoI (20 U) et ultrapure H2O jusqu'à un volume total de 50 l. Incuber le mélange de digestion à 37 oC pendant 2 h et inactiver à 65 oC pendant 20 min.
- Analyser le plasmide linéaire par électrophorèse sur un gel d'agarose (0,8 %, 40 min, 5 V/cm) à l'aide d'un colorant de chargement d'ADN et d'une échelle d'ADN avec des fragments d'ADN dans une fourchette de 100 à 10 000 pb. Comme un contrôle inclure un plasmide circulaire dans l'analyse.
- Purifie le plasmide linéaire à l'aide d'un kit de purification PCR selon les instructions du fabricant.
3. Préparation des constructions d'ADN de donneur de Promoteur-ORF-Terminator (POT)
- Commandez un ensemble de séquences de promoteur (P) de force différente, cadre de lecture ouverte (O) et terminator (T) comme ADN synthétique de sorte que chaque élément contient des séquences de reconnaissance standardisées 4 pb qui sont flanquées de sites BsaI pour activer Golden Gate Cloning ( GGC) assemblage26 (voir les dessins détaillés dans le tableau 3 et les séquences dans le tableau supplémentaire 4).
- Assembler des cassettes d'expression POT composées d'un promoteur, cadre de lecture ouvert, terminaison et connecteurs séquences via un assemblage en 4 parties à l'aide d'une réaction GGC21, dans un vecteur de destination qui contient déjà pré-spécifié 50-bp séquences de connecteurs ( voir Tableau supplémentaire 4 et références26,27).
- Mesurer la concentration des parties d'ADN à l'aide d'un spectrophotomètre. Diluer chaque partie d'ADN dans l'ultrapure H2O à une concentration finale de 15 fmol/L.
- Préparer un mélange de réaction composé de fragments d'ADN : 2 l de promoteur, 2 l de cadre de lecture ouvert, 2 l de terminaison et 2 colonnes vertébrales L (vecteurs de destination de niveau 1 tels que décrits dans 26), 4 oL de tampon de ligase d'ADN T4 de 5x, 2,5 l de 1 U/L T4 DNA Ligase , 1,5 L de 20 U/L BsaI-HF et ultrapure H2O jusqu'à un volume total de 20 l.
- Effectuer la réaction GGC dans un thermocycleur en utilisant le programme suivant : (i) 37 oC pour 2 min, (ii) 16 oC pendant 5 min - répéter les étapes (i) et (ii) 50 fois, (iii) 50 oC pendant 60 min, (iv) 80 oC pour 45 min, (v) tenir à 12 oC jusqu'à une analyse plus approfondie.
- Transformer 25 L de cellules E. coli28 achetées chimiquement compétentes avec 3 l l du mélange de réaction GGC selon le protocole du fabricant. Diluer le mélange de transformation 10 et 50 fois en 2x PY. Déposer les dilutions 10x et 50x sur les plaques d'agar PY 2x contenant de l'ampicilline (0,1 g/L) et couver toute la nuit à 37 oC.
- Choisissez 2 à 3 colonies et inoculez chaque colonie dans 3 ml de 2 x PY et poussez pendant la nuit à 37 oC dans un incubateur secouant à 180 tr/min.
- Purifie les plasmides à l'aide d'un kit de purification plasmide selon les instructions du fabricant.
- Vérifiez si les cassettes d'expression POT ont été assemblées correctement dans la réaction GGC par PCR.
- Amorces de conception complémentaires à la séquence de connecteur présente au début et à la fin de chaque cassette d'expression (voir Figure 2B). Pour les connecteurs choisis dans ce protocole, utilisez les amorces KC-103 à KC-108 (voir tableau supplémentaire 2).
- Préparer des mélanges d'amplification PCR pour chaque plasmide contenant : 0,5 L de polymérase d'ADN de relecture, 10 l de tampon 5x requis pour la polymérase d'ADN, 1 L de 10 mL de dNTP, 2,5 L de 10 'M amorce avant, 2,5 'L de 10 'M amorce inversée, 2 'L de modèle d'ADN avec une concentratorie ion de 5 ng/L, et ultrapure H2O jusqu'à un volume total de 50 l.
- Effectuer la réaction PCR dans un thermocycleur en utilisant le programme suivant : (i) 98 oC 3 min, (ii) 98 oC pour 10 s, (iii) 60 oC pour 20 s, (iv) 72 oC pour 2 min 30 s - étapes répétées (ii) à (iv) 30 fois, (v) 72 oC pour 5 min , (vi) tenir à 12 oC jusqu'à une analyse plus approfondie.
REMARQUE : Les produits PCR qui en résultent se composent de 50 pb du connecteur 5', promoteur, cadre de lecture ouvert, terminateur et 50-bp du connecteur 3'.
- Analyser les produits PCR par électrophorèse en exécutant des échantillons sur un gel d'agarose de 0,8 % à 5 V/cm pendant 40 min à l'aide d'un colorant de chargement d'ADN et d'une échelle d'ADN avec des fragments d'ADN dans une fourchette de 100 à 10 000 pb.
4. Préparation des séquences d'ADN de flanc d'intégration contenant des séquences de connecteurs
- Purifier l'ADN génomique du type sauvage S. cerevisiae CEN.PK113-7D29.
- Cultivez la souche dans un flacon de 500 ml rempli de 100 ml de peptone d'extrait de levure dextrose (YEPD, 2 % de glucose) moyen à 30 oC et secouant à 250 tr/min pendant 48 heures.
- Récolter les cellules par centrifugation de 2 ml de bouillon à 16 000 x g pendant 1 min et jeter le supernatant.
- Resuspendre les cellules dans du sel physiologique (200 l; 0,85% solution NaCl) avec rNase (10 l, 10 mg/mL) et l'enzyme lytique de levure (4 l). Incuber la suspension de la cellule à 37 oC pendant 15 min.
- Ajouter 300 l de solution de lyse cellulaire (voir Tableau des matériaux) et vortex sous peu.
- Ajouter 168 l de solution de précipitation de protéines (voir Tableau des matériaux) et vortex vigoureusement pour 20 s.
- Séparer la fraction de protéines par centrifugation à 16 000 x g et 4 oC pendant 10 min. Recueillir 600 l de supernatant dans un nouveau tube et mélanger avec 600 L d'isopropanol et de vortex sous peu.
- Récupérer l'ADN en tournant vers le bas à 16 000 x g à température ambiante pendant 10 min. Jetez le supernatant et gardez le granule.
- Laver les granulés avec 200 l'éthanol (70%). Centrifugeàeur à 16 000 x g à température ambiante pendant 10 min et retirer le supernatant. Évaporez l'éthanol en couvant le tube à température ambiante pendant 10 min avec le couvercle ouvert.
REMARQUE : Si le liquide dans le tube est encore visible, répétez l'étape 4.1.8. Ne pas sécher la pastille pendant plus de 10 min pour éviter une diminution de la solubilité de l'ADN. - Dissoudre l'ADN dans 50 l de tampon TE. Conserver l'ADN purifié à 4 oC.
- Pour chaque site d'intégration, l'intégration de la conception des séquences d'ADN de flanc (environ 500 pb) de sorte qu'environ 1000 pb d'ADN génomique seront prélevés lors de l'introduction de l'ADN des donneurs (voir la conception schématique de la figure 2B et les séquences en Tableau 4).
- Concevoir des amorces pour générer les régions de flanc par PCR.
- Pour la région de flancgauche, les amorces avant et arrière pour amplifier environ 500 pb de la région d'ADN génomique positionnée 5' (à gauche) du site d'intégration d'intérêt.
REMARQUE : L'apprêt avant comprend 20 pb d'homologie avec la région de flanc prévue. L'amorce inverse comprend 20 bp avec homologie avec la région de flanc prévue et contient la séquence de connecteur souhaitée de 50 pb pour permettre l'assemblage in vivo dans l'édition Cas12a sur le génome plus tard. - Pour la région de flancdroit, les amorces avant et arrière pour amplifier environ 500 pb de la région d'ADN génomique positionnée 3' (à droite) du site d'intégration d'intérêt.
REMARQUE : L'apprêt avant comprend 20 pb avec homologie avec la région de flanc prévue et contient la séquence de connecteur de 50 pb souhaitée pour permettre l'assemblage in vivo dans l'édition Cas12a sur le génome plus tard. L'amorce inverse comprend 20 bp d'homologie avec la région de flanc prévue.
- Pour la région de flancgauche, les amorces avant et arrière pour amplifier environ 500 pb de la région d'ADN génomique positionnée 5' (à gauche) du site d'intégration d'intérêt.
- Amplifiez les régions de flanc avec les amorces conçues(p. ex., amorces KC-109 à KC-120 incluses dans le tableau supplémentaire 2).
- Mesurer la concentration d'ADN génomique purifié qui servira de modèle dans le PCR. Ajuster la concentration d'ADN à 50 ng/L.
- Préparer des mélanges d'amplification PCR composés d'ADN génomique (1 à 4 l de dilution de l'ADN génomique de 50 ng/L) purifiés à l'étape 4.1, amorce avant et inverse (10 M chacun), 1 L de 10 mL dNTP, 10 oL de tampon de 5x requis pour la polymérase d'ADN, 0,5 l de polymériase d'ADN (1,0 U) , et ultrapure H2O jusqu'à un volume total de 50 L.
- Effectuer des PCR dans un thermocycleur en utilisant le programme suivant : (i) 98 oC pour 3 min, (ii) 98 oC pour 20 s, (iii) 60 oC pour 20 s, (iv) 72 oC pour 15 s, étapes répétées (ii) à (iv) 30 fois, (v) 72 oC pour 5 min , (vi) tenir à 12 oC jusqu'à une analyse plus approfondie.
- Analyser les produits PCR par électrophorèse sur un gel agarose de 0,8% à 5 V/cm pendant 40 min à l'aide d'un colorant de chargement d'ADN et d'une échelle d'ADN avec des fragments d'ADN dans une fourchette de 100 à 10 000 pb.
- Purifier les bons produits PCR à l'aide d'un kit de purification PCR selon les instructions du fabricant.
5. Transformation en S. cerevisiae
REMARQUE : Effectuer la transformation à l'aide d'un protocole basé sur les méthodes développées par Gietz et al. (1995) 30 et Hill et coll. 31 qui peut être utilisé pour diverses souches de S. cerevisiae. Le protocole décrit ci-dessous est suffisant pour 1 transformation.
- Préparer les solutions nécessaires à la transformation.
- Préparer les solutions de stock suivantes et la stérilisation des filtres : tampon TE 10x contenant 100 mM Tris-HCl (pH 7,5), 10 mM EDTA, volume total de 50 mL; 1 M LiAc au pH 7,5, volume total de 50 ml; 50% PEG 4000, volume total de 100 ml.
REMARQUE: Vérifiez toujours que le stock PEG 4000 est au pH 5. Ce stock ne doit pas être stocké plus d'un mois. - Préparer les solutions suivantes à l'aide de stocks : Préparer la solution LiAc-TE contenant 0,1 M LiAc, 10 mM Tris-HCl, 1 mM EDTA, volume total de 0,5 mL. Préparer la solution PEG-LiAc-TE contenant 40% PEG 4000, 0.1 M LiAc, 10 mM Tris-HCl, 1 mM EDTA, volume total de 1 mL.
REMARQUE : Il est crucial pour une transformation réussie que les solutions PEG-LiAc-TE et LiAc-TE soient fraîchement préparées.
- Préparer les solutions de stock suivantes et la stérilisation des filtres : tampon TE 10x contenant 100 mM Tris-HCl (pH 7,5), 10 mM EDTA, volume total de 50 mL; 1 M LiAc au pH 7,5, volume total de 50 ml; 50% PEG 4000, volume total de 100 ml.
- Premier tour de transformation (préparer la souche pré-exprimant Cas12a).
REMARQUE : Dans toutes les étapes de transformation, utilisez de l'eau avec un pH supérieur à 5. Il est recommandé d'utiliser de l'eau déminéralisée dans toutes les étapes de la transformation.- Préparer une pré-culture en cultivant la souche CEN. PK113-7D dans un flacon de secousse de 100 ml contenant 20 ml de YEPD (2 % de glucose) moyen et incuber pendant la nuit à 30 oC en secouant à 250 tr/min.
- Mesurer l'OD600 de la pré-culture (ODpc). Calculer le facteur de dilution (df) entre le volume de pré-culture et le volume de milieu frais nécessaire à la préparation des cellules pré-exprimant Cas12a à utiliser dans la transformation (culture de transformation). Dans les calculs supposent que la densité optique de la culture de transformation (ODtc) est de 1,0 après l'étape d'incubation décrite dans 5.2.3 (ti).

où ti et - sont le temps d'incubation et le temps de doublement, respectivement.- Calculer le volume de la pré-culture (Vi) nécessaire à l'inoculation de la culture de transformation (Vtc) en fonction du facteur de dilution.
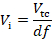
- Calculer le volume de la pré-culture (Vi) nécessaire à l'inoculation de la culture de transformation (Vtc) en fonction du facteur de dilution.
- Préparer la culture de transformation par inoculation de 20 ml de YEPD (2% de glucose) (Vtc) avec le volume de pré-culture déterminé dans l'étape précédente (Vi). Incuber à 30 oC en secouant à 250 tr/min.
- Mesurer l'OD600 de la culture de transformation jusqu'à ce qu'un OD600 de 1.0 soit atteint.
- Récolter les cellules par centrifugation du bouillon de 20 ml à 2 500 x g pendant 5 min. Jeter le supernatant et laver les cellules dans 20 ml d'eau déminéralisée à température ambiante. Répétez l'étape de centrifugation et gardez le granule de cellules.
- Resuspendre les cellules dans 100 l de la solution LiAc-TE et transférer dans un tube de microcentrifuge.
- Ajouter 5 ll d'ADN porteur à brin unique (10 mg/mL d'ADN de sperme de saumon) et mélanger par pipetting.
- Pipette 1 g de plasmien pCSN067 au tube de microcentrifuge.
REMARQUE : Le volume total du mélange d'ADN ne doit pas dépasser 100 L pour éviter une efficacité de transformation plus faible. - Ajouter 600 l de solution PEG-LiAc-TE et mélanger par pipetting. Incuber pendant 30 min à 30 oC tout en secouant à 450 tr/min dans un bloc de chaleur de table.
- Ajouter 70 l de DMSO (100%) au mélange de transformation et mélanger par pipetting. Effectuer le choc thermique en couvant le mélange de transformation à 42 oC pendant 15 minutes dans un bain d'eau.
- Récupérer les cellules en transférant le mélange à un tube de fond rond de 15 ml et ajouter 10 ml de YEPD (2 % de glucose) au tube. Incuber toute la nuit à 30 oC en secouant à 250 tr/min.
- Centrifuger le mélange de transformation à 2 500 x g pendant 5 min. Jeter le supernatant et resuspendre le granule cellulaire dans environ 200 l l de la solution restante.
- Plaquer 150 L du mélange de transformation et une dilution de 20x dans le YEPD (2% de glucose) du mélange de transformation sur les plaques d'agar YEPD (2% de glucose) complétées par 0,2 g/L G418. Incuber les assiettes à 30 oC pendant 48 à 72 heures.
- Choisissez un seul transformateur et re-streak sur une plaque d'agar YEPD (2% de glucose) complétée de 0,2 g/L G418 pour obtenir des colonies simples.
- Deuxième tour de transformation (réaliser l'édition du génome multiplex avec CRISPR/Cas12a).
- Préparer une pré-culture en augmentant la souche pré-exprimant Cas12a, créé dans le premier cycle de transformation (étape 5.2), dans un flacon de secousse de 100 ml contenant 20 ml de YEPD (2% de glucose) moyen complété par 0,2 g/L G418. Incuber toute la nuit à 30 oC en secouant 250 tr/min.
REMARQUE : Pour de multiples transformations, adaptez le volume de la pré-culture. - Suivez les étapes 5.2.2 à 5.2.7 pour le premier tour de transformation.
REMARQUE : Pour de multiples transformations, adaptez les volumes de solutions requises et la culture de la souche pré-exprimant Cas12a. - Pipette 1 g du tableau de crRNA unique, 1 g du plasmide du receveur linéaire pour le tableau de l'ARNc, 1 g d'ADN de donneur et 1 g de chaque région de flanc (étape 4.3) dans le tube de microcentrifuge.
REMARQUE : Le volume total du mélange d'ADN ne doit pas dépasser 100 L pour éviter une efficacité de transformation plus faible. - Préparer les contrôles suivants pour la transformation : contrôle négatif (ultrapure H2O); contrôle positif pour la détermination de l'efficacité de la transformation (1 g de pRN1120 circulaire); un contrôle vérifiant si l'introduction de l'ADN du donneur est effectuée par l'intermédiaire de l'édition CRISPR (1 g de pRN1120 circulaire, 1 g de toutes les cassettes d'expression d'ADN des donneurs et 1 g de régions flanquées, mais aucun tableau de crRNA unique); contrôle de la vérification de la façon dont l'ADN du donneur peut être intégré à l'extérieur de la cible (1 g de pRN1120 linéaire, 1 g de cassettes d'expression d'ADN des donneurs et 1 g du tableau de l'ARNc unique, mais pas de régions de flanc); un contrôle vérifiant la linéarisation complète de pRN1120 (1 g de pRN1120 linéaire).
- Suivez les étapes 5.2.9 à 5.2.12 pour le premier tour de transformation.
- Plaquer 150 L du mélange de transformation et 20x dilution dans YEPD (2% glucose) de mélange de transformation sur YEPD (2% glucose) agar complété avec 0.2 g/L G418 et 0.2 g/L NTC. Plaquer les contrôles sur l'agar YEPD (2% de glucose) complété par la sélection appropriée (G418 et/ou NTC ou pas de sélection). Incuber les assiettes à 30 oC pendant 48 à 72 heures.
- Choisissez un seul transformateur de couleur et re-streak sur une plaque d'agar YEPD (2% de glucose) pour obtenir des colonies de couleur unique.
- Préparer une pré-culture en augmentant la souche pré-exprimant Cas12a, créé dans le premier cycle de transformation (étape 5.2), dans un flacon de secousse de 100 ml contenant 20 ml de YEPD (2% de glucose) moyen complété par 0,2 g/L G418. Incuber toute la nuit à 30 oC en secouant 250 tr/min.
6. Évaluation de l'efficacité de l'édition du génome
- Comptez le nombre de colonies colorées et de colonies blanches sur les plaques de transformation.
- Calculer l'efficacité de l'édition du génome en divisant le nombre de colonies colorées par le nombre total de colonies (blanches et colorées), comme le montre le tableau 1.
7. Confirmation de l'intégration de l'ADN des donneurs au loci prévu
- Re-streak une colonie unique colorée à partir d'une plaque de transformation sur une plaque d'agar YEPD (2% de glucose) sans sélection G418 et NTC et couver pendant 48 heures à 30 oC.
- Choisissez une seule colonie et inoculez un flacon de 500 ml rempli de 100 ml de yEPD (2 % de glucose) moyen. Incuber pendant 48 heures à 30 oC et secouer à 250 tr/min.
- Isoler l'ADN génomique tel que décrit dans la section 4.1.
REMARQUE: Alternativement, utilisez un protocole pour la préparation de la levure pour la colonie PCR précédemment proposé par Looke et al. 32. Dans ce cas, la croissance du milieu liquide (article 7.2) peut être ignorée. - Vérifier l'intégration correcte par amplification de deux fragments par cassette d'expression intégrée.
- Concevoir des amorces qui annexent à l'ADN génomique à l'extérieur des régions de flanc transformées et du gène d'intérêt (voir des exemples dans le tableau supplémentaire 2, KC-121 à KC-132). Lors de l'utilisation des amorces KC-121 à KC-132, définir la température d'annealing dans le programme PCR à 62 oC.
- Amplifier la région d'intérêt décrite à la section 4.4.2. Adapter le programme PCR, ajuster spécifiquement le temps de l'étape d'extension dans PCR en fonction de la longueur du modèle et des recommandations du fabricant pour la polymérase d'ADN.
- Vérifier la taille des produits PCR par électrophorèse sur un gel d'agarose (0,8%, 40 min, 5 V/cm) à l'aide d'un colorant de chargement d'ADN et d'une échelle d'ADN avec des fragments d'ADN dans une gamme de 100 à 10 000 pb.
8. Création d'art pixel de levure à l'aide d'un gestionnaire de liquide acoustique
- Préparer un modèle d'image pour l'art pixel levure.
- Redimensionnez l'image RGB originale (220 à 280 pixels, voir les résultats représentatifs), par exemple en utilisant ImageJ pour créer une image finale de 64 à 96 pixels (largeur et hauteur) à l'échelle grise visualisée dans les couleurs prévues (Résultats représentatifs).
- Convertissez l'image RGB en gris à l'aide de cette formule :

où jegr, ir, ig, ib sont les intensités grises, rouges, vertes et bleues, respectivement. - Afin de catégoriser les pixels, développer un plugin ImageJ en appliquant les règles suivantes: (a) Si jegr est de 64, utilisez la levure orange foncé (souche 1, tableau supplémentaire 3) pour ce pixel. (b) Si 64 'lt; Igr '128, utilisez la levure orange (souche 2, tableau supplémentaire 3) pour ce pixel. (c) Si 128 lt; Igr 192, utilisez la levure jaune (souche 3, tableau supplémentaire 3) pour ce pixel. (d) Si jegr 'gt; 192, utiliser la levure blanche (CEN. PK113-7D) pour ce pixel.
- Repérez les cellules de levure pour créer l'art de pixel de levure.
- Inoculer 500 ml de flacons de secousse contenant 100 ml de yEPD (2 % de glucose) moyen avec trois caroténoïdes de couleur différente produisant la souche S. cerevisiae et le type sauvage CEN. PK113-7D. Incuber les cultures pendant la nuit à 30 oC avec des secousses à 250 tr/min.
- Transférer 0,5 ml de la culture de la nuit dans un tube rempli de 0,5 ml de milieu de gradient de densité stérile non ionique (voir le Tableau des matériaux). Mélanger en tourbillonnant brièvement.
- Transférer la suspension cellulaire dans un réservoir qualifié, 2 x 3 puits. Effectuer le repérage à l'aide d'un instrument acoustique de manutention de liquide d'une plaque de source de réservoir qualifiée à une microplaque (voir le tableau des matériaux) contenant 50 ml d'agar YEPD (2 % de glucose). Pour simplifier le placage, définissez les puits sur la plaque, par exemple. utiliser une microplaque comme plaque de puits 6144 (64 à 96).
- Spot 25 nL de chaque souche S. cerevisiae de la plaque de source du réservoir de puits 2x 3 à l'aide d'un fichier .csv avec le réglage d'étalonnage fluide 6RES-AQ-GPSA2 sur la microplaque de destination. Définissez chacune de ces gouttelettes de 25 nL comme un pixel dans la grille 64 x 96 qui est traduite aux positions de puits (A01, B01, C01 etc.).
- Incuber la microplaque à 30 oC pendant 48 heures. Pour intensifier les couleurs des souches, entreposez la plaque d'agar à 4 oC pendant au moins 72 heures.
Résultats
Le protocole pour l'édition du génome multiplex à l'aide de CRISRP/Cas12a a été démontré en construisant trois souches caroténoïdes produisant S. cerevisiae exprimant les gènes crtE, crtYB et crtI en utilisant des promoteurs hétérologues de résistance élevée, moyenne et faible : souche 1, 2 et 3 respectivement (tableau supplémentaire 3). La construction de ces souches a nécessité la production de trois cassettes d'expression d'ADN des donneurs et de six régions de flanc par souche pour cibler trois loci différents dans l'ADN génomique (indiqué à la figure 2B). Comme décrit ci-dessus, le promoteur, le cadre de lecture ouvert, le terminateur et deux séquences de connecteurs contigus de 50 pb ont été assemblés dans une cassette d'expression par l'intermédiaire d'une réaction de clonage golden Gate et l'assemblage a été vérifié par PCR (Figure 3A). Le tableau de crRNA unique a été commandé comme fragment d'ADN synthétique et a été amplifié par PCR (Figure 3B). Le plasmide receveur pour le tableau de crRNA unique (plasmid pRN1120) a été linéaire avec EcoRI-HF et XhoI et la linéarisation a été confirmée par électrophorèse (figure 3C). La conception et les séquences de nucléotides des cassettes d'expression d'ADN des donneurs introduits et des régions de flanc sont présentées dans le tableau 3 et le tableau supplémentaire 4. La séquence des cassettes d'expression de tableau de crRNA simple est fournie dans le tableau supplémentaire 1. La fonctionnalité des espaceurs inclus dans le tableau crRNA unique a été testée à l'avance par l'édition du génome singleplex avec des crRNAs individuels19.
L'efficacité de l'édition du génome à l'aide de Cas12a a d'abord été évaluée en fonction du nombre de colonies colorées obtenues après la transformation (tableau 1, figure 4). L'efficacité d'édition des trois souches construites variait de 50 % à 94 %. Notamment, l'introduction de cassettes d'expression utilisées pour générer la souche 1 a montré la plus faible efficacité d'édition, peut-être causée par la nature de l'ADN du donneur(c.-à-d., ces cassettes d'expression codecr crtE, crtYB et crtI de trois promoteurs de haute résistance). Deuxièmement, l'intégration correcte des trois cassettes d'expression d'ADN des donneurs au loci prévu sur l'ADN génomique a été confirmée par PCR (figure 5). Les amorces ont été conçues de telle sorte que les produits PCR ont été obtenus lorsque l'intégration correcte de l'ADN du donneur au lieu prévu s'est produite. Pour chaque expérience de transformation, huit colonies ont été cueillies à partir de la plaque de transformation et testées (à noter que seulement trois sont présentées à la figure 5). En général, sur 8 colonies testées par ADN de donneur, l'intégration correcte de l'ADN du donneur crtE au cri usin1, le crtYB au locus INT2 et le crtI au locus INT3 ont été confirmés dans les transformateurs. Ces résultats démontrent que le système CRISPR/Cas12a en combinaison avec un seul tableau de crRNA permet une édition multiplexe efficace du génome de S. cerevisiae à plusieurs loci simultanément.
En outre, nous démontrons la création de "yeast pixel art" en utilisant les trois souches de production de caroténoïdes qui ont été construits avec une souche de type sauvage non-coloré. À partir d'une image en noir et blanc de Rosalind Franklin (Figure 6A), une image en 4 couleurs (Figure 6B) et la liste de repérage a été créé qui a ensuite été utilisé pour repérer les quatre souches de levure différentes sur une microplaque agar à l'aide d'un gestionnaire de liquide acoustique, résultant en une haute résolution "peinture de levure" de Rosalind Franklin (Figure 6C,D,E).

Figure 1 : Flux de travail du protocole pour l'édition du génome du multiplexe CRISPR/Cas12a en S. cerevisiae. Le flux de travail comprend des étapes cruciales de la méthode présentée. Pour plus de détails, consultez le Protocole. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Schéma d'édition du génome du multiplexe CRISPR/Cas12a à l'aide d'un tableau crRNA unique. (A) Le tableau de crRNA unique est composé de trois unités crRNAs dans leur forme mature, une répétition directe de 20 pb spécifique pour LbCas12a (carrés gris) avec une séquence de guide de 23 pb (diamants de couleur). L'expression du tableau crRNA est activée par le promoteur SNR52 et le terminateur SUP4. La transformation de S. cerevisiae avec un pRN1120 linéaire et la cassette d'expression de tableau de crRNA simple contenant l'homologie avec pRN1120 (bandes diagonales) permet la recombinaison in vivo dans un plasmide circulaire dans les cellules pré-exprimant LbCas12a. Le tableau crRNA unique est ensuite traité par Cas12a. (B) Cas12a est dirigé vers les sites cibles génomiques INT1, INT2 et INT3 prévus et crée des ruptures doubles. Dans le mélange de transformation, l'ADN de distributeur se composant des régions de flanc et de la cassette d'expression de gène caroténoïde ont été inclus. Les assemblages d'ADN des donneurs ont été ciblés sur un tronçon d'ADN dans l'ADN génomique autour de l'INT1 (crtE), INT2 (crtYB) et INT3 (crtI) loci par recombinaison in vivo en raison de la présence de séquences de connecteurs homologues de 50 pb, indiqués comme 5, A, B, C, D ou E. P1-P3, différents promoteurs; T1-T3, différents terminateurs. Ce chiffre a été modifié à partir de Verwaal et al. 201819. Constructions génétiques montrées à l'aide de la biologie synthétique Open Language (SBOL) Symboles visuels40. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
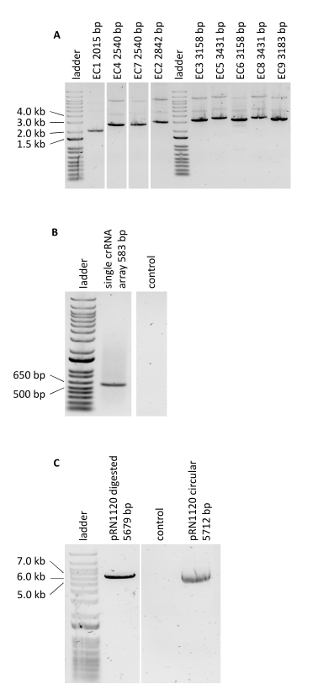
Figure 3 : PCR vérifiant les expériences d'édition du génome. (A) Vérification des réactions de clonage de la porte d'or des cassettes d'ADN des donneurs assemblés. Les résultats obtenus sont en accord avec les longueurs attendues. (B) PCR du tableau crRNA unique. (C) Linearisation du plasmide pRN1120. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Plaques de transformations S. cerevisiae en utilisant l'approche d'édition du génome multiplex. (A) Souche 1 exprimant crtE, crtYB et crtI de trois promoteurs forts (colonies orange foncé). (B) Souche 2 exprimant crtE, crtYB et crtI de trois promoteurs de force moyenne (colonies orange). (C) Souche 3 exprimant crtE, crtYB et crtI de trois promoteurs de faible résistance (colonies jaunes). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 5 : PCR vérifiant l'intégration des cassettes d'expression d'ADN de distributeur aux loci prévus dans l'ADN génomique. (A) Vérification de trois colonies de la souche 1. (B) Vérification de trois colonies de la souche 2. (C) Vérification de trois colonies de la souche 3. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 6 : Art de pixel de levure de Rosalind Franklin. (A) Photo RGB en noir et blanc de 220 à 280 pixels de Rosalind Franklin qui a été utilisé comme modèle. (B) Conversion informatique de la photo en noir et blanc de Rosalind Franklin en une liste de 4 couleurs 64 à 96 pixels. (C) Photo de l'art de pixel de levure avec 64 -96 colonies de levure avec une section zoomée-dans. (D) Photo d'un manutentionnaire de liquide acoustique avec deux plaques pleines de croissance. (E) Photo d'une microplaque adulte avec 64 à 96 colonies de levures. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| La souche 1 | Souche 2 | Souche 3 | |
| Colonies colorées | 16 Annonces | 279 Annonces | 220 Ans, états-unis |
| Colonies blanches | 16 Annonces | 18 ans, états-unis qui | 18 ans, états-unis qui |
| Colonies totales | 32 Ans, états-unis ( | 297 Annonces | 238 Annonces |
| efficacité | 50% | 94 % | 92% |
Tableau 1 : Efficacité de montage de l'approche d'édition du génome multiplex.
| crRNA tableau séquencea,b,c,d,e,f |
|
CATGTTTGACAGCTTATCATCATCGATATCCGGAGCTAGCATGCGGCCGCCTAGAACTAGTGGATCCCMGCTGCAG TCTTTGAAAAAA TCTTGAAAAA GATAATGTATGATTATGCTTTCACTCATATTTATACAACTTGATTTTTTTTTTTTTTTGagTATATACAAGG TGATTACATGTACGTTTGAAGTACAACTCTAGATTTTTTTagTGCCCTTTTTGgGCTAGCGCGGTAAAGGTGCGCA TTTTTTTCACACCCTACAATGTTTGTTGTTCAAAGATTTTGGTCAACGCTGTAGAGTGAAGtTGGTGCGC ATGTTTTCGGCGTTCGAAACTTCTCCGCAGTGAAAGATAATGATCAATTTCTACTAGTGTAGAT CTGGTGGGAGAGAAAGCTTATGAAATTTTActAGTGTAGATGTGCCGTAC GCCGGAGCCGACGGAATTTTTACTAAGTGTAGATTGCCCCTCTTATACGATTATATTTT TTTTTTGTTTTTATGTCTGGGGCCCGGTACCACGCTTTTGTTCCCT GTTAATTCCGAGCTTGGCGTAATCATTCATAGCTGTTTCCTGTGTGGTGGG |
| a. Homologie à pRN1120 (bold). b. Promoteur SNR52 (italiques). c. Séquences cibles génomiques (soulignées). d. Guide rit en direct répète spécifique pour LbCas12a (italiques, gras). e. SUP4 terminator (italiques). f. Homologie à pRN1120 (bold). |
Tableau supplémentaire 1 : tableau de crRNA simple pour LbCas12a contenant l'homologie avec plasmide pRN1120.
| nom | Séquencea | Descriptionb | Utilisé en point |
| KC-101 | CATGTTTGACAGCTTATCATC | Apprêt FW pour l'amplification du tableau crRNA unique | 2.1.4 |
| KC-102 | CACACAGGAAACAGCTATGAC CACACAGGAAACAGCTATGAC | Amorce RV pour l'amplification du tableau crRNA unique | 2.1.4 |
| KC-103 | AAGCGACTTCCAATCGCTTTTGC AAGCGACTTCCAATCGCTTTTGC AAGAGCGACTTCCAATCGCTTT | Apprêt FW pour l'amplification de l'ADN du donneur avec connecteur 5 | 3.6.1 Annonces |
| KC-104 | AAAGCAAAGGAGGAGAGAAC AAAGCAAAGGAAA | Amorce de RV pour l'amplification de l'ADN du donneur avec le connecteur A | 3.6.1 Annonces |
| KC-105 | CGGATCGATGTACACAACCG | Apprêt FW pour l'amplification de l'ADN du donneur avec connecteur B | 3.6.1 Annonces |
| KC-106 | CAACAGGAGGCGGATATATAC | Amorce de RV pour l'amplification de l'ADN de donneur avec le connecteur C | 3.6.1 Annonces |
| KC-107 | AACGTTGTCCAGGTTTGTATCC | Apprêt FW pour l'amplification de l'ADN du donneur avec connecteur D | 3.6.1 Annonces |
| KC-108 | AGGTACAAACACACGACCG | Amorce DE RV pour l'amplification de l'ADN de donneur avec le connecteur E | 3.6.1 Annonces |
| KC-109 | CACTATAGCAATCTGGCTATATTG | Amorce FW pour amplification de L'INT1 5' avec connecteur 5 | 4,4 |
| KC-110 |
AAACGCCTGTGGGTGTGGTAC TGGATATGCAAAGCGATTGGAA GTCGCTTGACTCTTGCCGTC ATTCC (en anglais seulement) | Amorce RV pour amplification de L'INT1 5' avec connecteur 5 | 4,4 |
| KC-111 |
TTGCCCATCGAACGTACAAG TACTCCTCTGTTCTCTCTCTCTCTCTCTCTCTTTTTTTTTTTTTTTTTTTTTTTT TGCTTTAAGCGTTGAAGTTTCCTC TTTG (en) | Amorce FW pour amplification de L'INT1 3' avec connecteur A | 4,4 |
| KC-112 | TGTCAACTGGAGAGCTATCG TGTCAACTGGAGAGCTATCG | Amorce RV pour amplification de L'INT1 3' avec connecteur A | 4,4 |
| KC-113 | AGAAGATTTCTTCAATCTCTCC | Amorce FW pour amplification de l'INT2 5' avec connecteur B | 4,4 |
| KC-114 |
TGCTAAGATTTGTGTTCGTT TGGGTGCAGTGTGTGTGTGTGTGTACAT CGATCCGCCCTTATCAAGGATACC TGGTTG TGGTTG | Amorce RV pour amplification de L'INT2 5' avec connecteur B | 4,4 |
| KC-115 |
ACGCTTTCCGGCATCTTCCA GACCACAGTATATCCATCCGCCT GACCACAGTGTATCCATCCGCCT CCTGTTGGGCGATTACACAAGCG GTGG (EN anglais seulement) | Amorce FW pour amplification de l'INT2 3' avec connecteur C | 4,4 |
| KC-116 | TCTCCTTCGATGACCGGGGG | Amorce RV pour amplification de l'INT2 3' avec connecteur C | 4,4 |
| KC-117 | GGTCGTTTTTGTGCAGCATATTTG GGTCGTTTTTGTGCAGCATATTGGGGG | Amorce FW pour amplification de l'INT3 5' avec connecteur D | 4,4 |
| KC-118 |
GCGGAATTGGCGGAACGG ACACACGTGGATACAAACCTG GACAACGTTTTCCAAGGAGGTG AAGAACG (En) | Amorce RV pour amplification de L'INT3 5' avec connecteur D | 4,4 |
| KC-119 |
AAATAAAACAACATCCTT CCCATATGCTCGGTCGTGCTTGTTGTTGTT GTACCTGATGGGACGTCAGCACT GTAC (En) | Amorce FW pour amplification de l'INT3 3' avec connecteur E | 4,4 |
| KC-120 | GAGCTTACTCTATATTCATTC | Amorce RV pour amplification de L'INT3 3' avec connecteur E | 4,4 |
| KC-121 | GTTACTAAACTGGAACTGTCCG | Apprêt FW pour la vérification de l'intégration de con5-crtE-conA à INT1 5' | 7,4,1 |
| KC-122 | CACTGCTAACTACGTTACTTC | Apprêt FW pour la vérification de l'intégration de con5-crtE-conA à INT1 3' | 7,4,1 |
| KC-123 | CACTGGAACTTGAGCTTGAG | Apprêt FW pour la vérification de l'intégration de conB-crtYB-conC à INT2 5' | 7,4,1 |
| KC-124 | GTCTCCAGCTGATTGGTCC | Apprêt FW pour la vérification de l'intégration de conB-crtYB-conC à INT2 3' | 7,4,1 |
| KC-125 | CTCTCATGAAGCAGTCAAGTC | Apprêt FW pour la vérification de l'intégration de conD-crtI-conE à INT3 5' | 7,4,1 |
| KC-126 | GATCGGTCAATTAGGTGAAG GATCGGTCAATTAGGTGAAG | Apprêt FW pour la vérification de l'intégration de conD-crtI-conE à INT3 3' | 7,4,1 |
| KC-127 | CCTTGTCCAAGTAGGTGTCC | Amorce DE RV pour la vérification de l'intégration de con5-crtE-conA à INT1 5' | 7,4,1 |
| KC-128 | GCTGTCATCATGATCTGTGATAAC | Amorce DE RV pour la vérification de l'intégration de con5-crtE-conA à INT1 3' | 7,4,1 |
| KC-129 | CTGGCAATGTTGACCAATTGC | Amorce de RV pour la vérification de l'intégration du conB-crtYB-conC à l'INT2 5' | 7,4,1 |
| KC-130 | CCAACGTGCCTTAAGTCTG | Amorce de RV pour la vérification de l'intégration du conB-crtYB-conC à l'INT2 3' | 7,4,1 |
| KC-131 | CCTTACCTTCTGGAGCAGCAG | Amorce DE RV pour la vérification de l'intégration du conD-crtI-conE à l'INT3 5' | 7,4,1 |
| KC-132 | CTGGTACTTCCCTAAGACTG | Amorce DE RV pour la vérification de l'intégration du conD-crtI-conE à inT3 3' | 7,4,1 |
| a. Les séquences audacieuses dénotent les séquences de connecteurs. b. Les amorces avant et arrière sont désignées comme FW et RV, respectivement. |
Tableau supplémentaire 2 : Séquences d'apprêt.
Tableau supplémentaire 3 : Conception des souches construites.

Tableau supplémentaire 4 : Séquences de cassettes d'expression d'ADN des donneurs et régions écaillantes. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole fourni décrit l'édition du génome multiplex de S. cerevisiae à l'aide de Cas12a de la bactérie Lachnospiraceae ND2006 en combinaison avec un tableau unique de crRNA et l'ADN du donneur. La conception du tableau de crRNA unique et de l'ADN de donneur est expliquée en détail. Contrairement au système CRISPR/Cas9 bien établi, le CRISPR/Cas12a a la capacité supplémentaire unique de traiter plusieurs crARN exprimés à partir d'un seul tableau crRNA13,33. Grâce à cette fonctionnalité, l'édition simultanée de plusieurs cibles est plus facile à configurer et peut être réalisée en une seule transformation. Cette approche de tableau de crRNA simple a été démontrée avant par Zetsche etautres. 34 qui ont simultanément édité jusqu'à quatre gènes dans les cellules de mammifères en utilisant AsCas12a, et par Swiat et al. 35 qui ont introduit quatre fragments d'ADN dans un génome de levure en utilisant FnCas12a. À notre connaissance, un plus grand nombre de modifications génomiques simultanées utilisant un système Cas12a n'a pas été signalée et la limite maximale de cibles par tableau unique pour Cas12a n'a pas encore été déterminée. D'autres recherches utilisant des tableaux de crRNA simples en combinaison avec Cas12a inclut la régulation transcriptionnelle multiplex dans un large éventail d'organismes33,36,37.
Il y a quelques étapes critiques dans le protocole présenté. Concevez soigneusement toutes les séquences d'ADN qui sont impliquées dans l'expérience d'édition du génome Cas12a, en particulier dans le cas où de nouvelles séquences d'ADN sont introduites. Déterminer la fonctionnalité des nouvelles séquences d'espaceurs faisant partie d'un crRNA, par exemple par une expérience d'édition du génome singleplex telle que décrite par Verwaal et al. 19 avant de les combiner en un seul tableau crRNA. Suivez les recommandations pour la préparation des solutions tampon de transformation utilisées dans l'expérience d'édition Cas12a pour atteindre une bonne efficacité de transformation de la levure.
Il y a quelques modifications facultatives de la technique. Il est recommandé d'utiliser 1 g de chaque ADN de donneur, pRN1120 linéaire ou cassette d'expression de tableau de crRNA unique dans la transformation, bien que l'utilisation d'une quantité inférieure d'ADN devrait également avoir comme conséquence une efficacité satisfaisante de transformation. Effectuer une transformation de test pour déterminer si des quantités d'ADN plus faibles peuvent être utilisées. La transformation de S. cerevisiae pourrait être effectuée en utilisant une méthode différente de celle décrite dans ce protocole, par exemple le protocole décrit par Gietz et al. (2007) 38. Le plasmine plasmide pRN1120 du récepteur d'ARN guide convient à l'expression d'un seul crRNA et d'un tableau unique de crRNA de différentes variantes De12a(p. ex., d'Acidaminococcus spp. BV3L6 ou Francisella novicida U112) ainsi que pour l'expression de sgRNA en combinaison avec Cas919. L'ADN du donneur n'a pas besoin d'être limité aux cassettes d'expression génique caroténoïdes et aux régions de flanc qui ciblent l'ADN des donneurs aux sites INT1, INT2 et INT3 décrits dans l'ADN génomique. Tout ADN d'intérêt peut être introduit, de manière multiplexe, dans l'ADN génomique de l'hôte par les principes de conception décrits dans ce protocole, ou alternativement l'ADN de donneur peut être utilisé pour supprimer l'ADN d'un génome hôte. La structure modulaire du tableau de crRNA simple facilite l'ajustement des séquences d'espaceur et de répétition directe. La modification des séquences d'espaceur permet de modifier le lieu d'intégration prévu qui peut être conçu par l'un des outils d'identification d'un site cible génomique, par exemple le logiciel GuideScan 1.039. Au lieu d'utiliser de grandes séquences de flanc qui contiennent des séquences de connecteurs, 50 pb de la région de flanc peuvent être inclus dans les séquences d'ADN des donneurs en incorporant ces séquences de région de 50 pb dans les amorces utilisées dans le PCR. Dans ce cas, au total seulement trois au lieu de neuf fragments d'ADN de donneur sont nécessaires pour une expérience réussie d'édition de génome de multiplexe.
En résumé, ce protocole fournit des instructions étape par étape pour effectuer l'édition du génome multiplex dans S. cerevisiae en utilisant Cas12a en combinaison avec une approche de tableau crRNA unique. Le protocole a été démontré par l'édition du génome multiplexe à l'aide de 9 fragments d'ADN de donneur et d'un seul codage de tableau de crRNA pour trois gRNAs. Nous montrons des fréquences d'édition globales élevées entre 50% et 94% pour les trois conceptions de contrainte rapportées ici. En conclusion, la caractéristique unique de Cas12a est la possibilité de traiter un seul tableau crRNA en CRRNAs individuels dans une cellule, ce qui fait cas12a un excellent outil pour permettre l'édition du génome multiplex et de développer des modules de régulation transcriptionnelle ciblant plusieurs cassettes d'expression en une seule fois. En fin de compte, trois souches ont été obtenues produisant des caroténoïdes à un niveau différent et des couleurs dans des tons entre le jaune et l'orange. Avec ces souches et une souche de type sauvage, nous avons montré comment un gestionnaire de liquide acoustique peut être utilisé directement pour faire de l'art pixel levure - ce en l'honneur de Rosalind Franklin qui a contribué à la découverte de la structure de l'ADN il ya 65 ans par sa célèbre photo 5123 /c1.
Déclarations de divulgation
Les auteurs déclarent qu'il y a conflit d'intérêts. Les auteurs ont déposé la propriété intellectuelle liée aux méthodes présentées.
Remerciements
Ce projet a reçu un financement du programme de recherche et d'innovation Horizon 2020 de l'Union européenne dans le cadre de l'accord de subvention no 686070 (DD-DeCaf) et 764591 (SynCrop), et du programme de recherche Building Blocks of Life avec le numéro de projet 737.016.005 par l'Organisation néerlandaise pour la recherche scientifique (NWO). T.E.G. a reçu l'appui de la Royal Society (grant UF160357) et de BrisSynBio, un CENTRE de recherche en biologie synthétique BBSRC/EPSRC (subvention BB/L01386X/1). Nous remercions Zi Di et Jeffrey van Wijk pour leur contribution aux expériences de repérage de levurepour la création de l'art pixel de levure.
matériels
| Name | Company | Catalog Number | Comments |
| Chemicals specific for the protocol | |||
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | Electrophoresis |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | Selection of E. coli transformants |
| BsaI-HF (20 U/µl) | New England BioLabs | R353L | Golden Gate Cloning |
| Cell Lysis Solution (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| CutSmart Buffer | New England BioLabs | B7204S | Linearization of pRN1120 |
| Deoxyribonucleic acid sodium salt from salmon testes | Sigma Aldrich | D1626 | Transfromation of S. cerevisiae (carrier DNA) |
| dNTPs | Invitrogen | 10297018 | PCRs |
| EcoRI-HF | New England BioLabs | R3101S | Linearization of pRN1120 |
| Ethanol absolute for analysis | Merck | 100983 | Isolation of genomic DNA from S. cerevisiae |
| Ethylenediamine-tetraacetic acid | Sigma Aldrich | ED | Transformation of S. cerevisiae |
| G418 disulfate salt | Sigma Aldrich | A1720 | Selection of S. cerevisiae transformants |
| Histodenz | Sigma Aldrich | D2158 | Yeast pixel art |
| Isopropanol | Merck | 100993 | Isolation of genomic DNA from S. cerevisiae |
| Lithium acetate dihydrate | Sigma Aldrich | L6883 | Transformation of S. cerevisiae |
| Nancy-520 DNA Gel Stain | Sigma Aldrich | 1494 | Electrophoresis |
| NEB10 competent E. coli cells | New England BioLabs | C3019H | Transformation of E. coli: dx.doi.org/10.17504/protocols.io.nkvdcw6 |
| Nourseothricin | Jena Bioscience | AB102 | Selection of S. cerevisiae transformants |
| Phusion buffer | New England BioLabs | M0530L | PCRs |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530L | PCRs |
| Polyethylene glycol 4000 | Merck | 7490 | Transformation of S. cerevisiae |
| Protein Precipitation Solution (10 M NH4AC) (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| Purple loading dye | New England BioLabs | B7024S | Electrophoresis |
| QIAprep Spin Miniprep Kit | QIAGEN | 27106 | Purification of plasmids |
| RNase coctail enzyme mix | Thermo Fisher Scientific | AM2286 | Isolation of genomic DNA from S. cerevisiae |
| T4 DNA ligase buffer | Invitrogen | 46300-018 | Golden Gate Cloning |
| T4 DNA Ligase (1 U/µl) | Invitrogen | 1705218 | Golden Gate Cloning |
| UltraPure Agarose | Invitrogen | 16500500 | Electrophoresis |
| Wizard SV Gel and PCR Clean-Up System Kit | Promega | A9282 | Purification of PCR products and linearized pRN1120 |
| Xhol | New England BioLabs | R0146S | Linearization of pRN1120 |
| Zymolyase 50 mg/ml (5 units/µL) | Zymo Research | E1006 | Isolation of genomic DNA from S. cerevisiae (yeast lysis enzyme) |
| Zymolyase storage buffer | Zymo Research | E1004-B | Isolation of genomic DNA from S. cerevisiae (necessary for the preparation of yeast lysis enzyme) |
| Chemicals of general use | |||
| 2*Peptone-Yeast extract (PY) agar | Plate growth of E. coli | ||
| 2*PY medium | Cultivation of E. coli | ||
| Demineralized water | Transformation of S. cerevisiae | ||
| ELFO buffer | Electrophoresis | ||
| MQ | Multiple steps | ||
| Physiological salt solution | Transformation of S. cerevisiae | ||
| TE buffer | Storage of DNA, transformation of S. cerevisiae | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | Cultivation of S. cerevisiae | ||
| YEPD (2% glucose) agar | Plate growth of S. cerevisiae | ||
| Consumables | |||
| Eppendorf tubes | |||
| Falcon tubes (50 mL) | |||
| Microplate 96 wells | |||
| Petri dishes | |||
| Pipette tips 0.5 - 10 µL | |||
| Pipette tips 10 - 200 µL | |||
| Pipette tips 100 - 1000 µL | |||
| Shake flasks (500 mL) | |||
| Sterile filters | |||
| Equipment | |||
| Centrifuge (Falcon tubes) | |||
| Echo 525 acoustic liquid handler | |||
| Incubator | |||
| NanoDrop | |||
| Set for eletrophoresis | |||
| Spectrophotometer | |||
| Table centrifuge (Eppendorfs tubes) | |||
| Thermocycler | |||
| Plasmids | |||
| pCSN067 | Addgene | ID 101748 | https://www.addgene.org/ |
| pRN1120 | Addgene | ID 101750 | https://www.addgene.org/ |
| Strains | |||
| S. cerevisiae strain CEN.PK113-7D | EUROSCARF collection | http://www.euroscarf.de |
Références
- Knott, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361 (6405), 866-869 (2018).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Lian, J., HamediRad, M., Hu, S., Zhao, H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications. 8 (1), 1688 (2017).
- Li, Z. -. H., Liu, M., Lyu, X. -. M., Wang, F. -. Q., Wei, D. -. Z. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology. 58 (12), 1100-1104 (2018).
- Shao, Y., Lu, N., Qin, Z., Xue, X. CRISPR-Cas9 facilitated multiple-chromosome fusion in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (11), 2706-2708 (2018).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology. 13 (11), 722-736 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), (2016).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovič, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Lian, J., HamediRad, M., Zhao, H. Advancing metabolic engineering of Saccharomyces cerevisiae. using the CRISPR/Cas System. Biotechnology Journal. 13 (9), 1700601 (2018).
- Ferreira, R., et al. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (1), 10-15 (2018).
- Swarts, D. C., Martin, J. Cas9 versus Cas12a/Cpf1: Structure–function comparisons and implications for genome editing. Wiley Interdisciplinary Reviews: RNA. 9 (5), 1481 (2018).
- Strohkendl, I., Saifuddin, F. A., Rybarski, J. R., Finkelstein, I. J., Russell, R. Kinetic Basis for DNA Target Specificity of CRISPR-Cas12a. Molecular Cell. 71 (5), 816-824 (2018).
- Verwaal, R., et al. High-level production of beta-carotene in Saccharomyces cerevisiae. by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous. Applied and Environmental Microbiology. 73 (13), 4342-4350 (2007).
- Verwaal, R., Buiting-Wiessenhaan, N., Dalhuijsen, S., Roubos, J. A. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast. 35 (2), 201-211 (2018).
- Jakociunas, T., Jensen, M. K., Keasling, J. D. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering. 34, 44-59 (2016).
- Engler, C., Romy, K., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PloS One. 3 (11), 3647 (2008).
- Franklin, R. E., Gosling, R. G. Molecular configuration in sodium thymonucleate. Nature. 171, 740-741 (1953).
- Watson, J. D., Crick, F. H. A structure for deoxyribose nucleic acid. Nature. 171, 737-738 (1953).
- Wilkins, M. H. F., Stokes, A. R., Wilson, H. R. Molecular structure of deoxypentose nucleic acids. Nature. 171, 738-740 (1953).
- Young, E. M., et al. Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast. Metabolic Engineering. 48, 33-43 (2018).
- Roubos, J. A., Pel, H. J., Meijrink, B. . Cloning Method. , (2013).
- Mandel, M., Higa, A. Calcium-dependent bacteriophage DNA infection. Journal of Molecular Biology. 53 (1), 159-162 (1970).
- Van Dijken, J. P., et al. An interlaboratory comparison of physiological and genetic properties of four Saccharomyces cerevisiae strains. Enzyme and Microbial Technology. 26 (9-10), 706-714 (2000).
- Gietz, R. D., Schiestl, R. H., Willems, A. R., Woods, R. A. Studies on the transformation of intact yeast cells by the LiAc/SS-DNA/PEG procedure. Yeast. 11 (4), 355-360 (1995).
- Hill, J., Donald, K. A., Griffiths, D. E., Donald, G. DMSO-enhanced whole cell yeast transformation. Acids Research. 19 (20), 5791 (1991).
- Looke, M., Kristjuhan, K., Kristjuhan, A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques. 50 (5), 325-328 (2011).
- Tak, Y. E., et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nature Methods. 14 (12), 1163-1166 (2017).
- Zetsche, B., et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature biotechnology. 35 (1), 31-34 (2017).
- Swiat, M. A., et al. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research. 45 (21), 12585-12598 (2017).
- Li, L., et al. CRISPR-Cpf1-Assisted Multiplex Genome Editing and Transcriptional Repression in Streptomyces. Applied Environmental Microbiology. 84 (18), 00827-00918 (2018).
- Zhang, X., et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery. 3, 17018 (2017).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 1-4 (2007).
- Perez, A. R., et al. GuideScan software for improved single and paired CRISPR guide RNA design. Nature Biotechnology. 35 (4), 347-349 (2017).
- Cox, R. S., et al. Synthetic Biology Open Language Visual (SBOL Visual) Version 2.0. Journal of Integrative Bioinformatics. 15 (1), 1613-4516 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon