Method Article
CRISPR/Cas12a מולטיפלקס עריכת הגנום של סכביקוע cerevisiae ס ויצירת שמרים אמנות פיקסל
In This Article
Summary
מערכת CRISPR/Cas12a בשילוב עם מערך crRNA יחיד מאפשר עריכה משולבת יעילה של הגנום S. cerevisiae ס בבית מרובים בו זמנית. זה הפגינו על ידי בניית קרוטנאיד הפקת זנים שמרים אשר משמשים לאחר מכן כדי ליצור פיקסל ארט שמרים.
Abstract
יעילות גבוהה, קלות השימוש צדדיות של האשכולות באופן קבוע במרווחים של התאמה מלאה של palindromic מלאה/CRISPR-חלבון משויך 9 (CRISPR/Cas9) מערכת הקלה שינוי גנטי מתקדם של סכביסיסים cerevisiae ס, מודל אורגניזם וסוס עבודה בביוטכנולוגיה תעשייתית. CRISPR הקשורים חלבון 12a (Cas12a), מונחה RNA endonuclease עם תכונות להבדיל מ Cas9 מוחל בעבודה זו, עוד הרחבת ארגז הכלים המולקולרי למטרות עריכת הגנום. יתרון של מערכת CRISPR/Cas12a היא כי ניתן להשתמש בו ב מולטיפלקס עריכת הגנום עם מדריך מרובים RNAs מבוטא יחידה transcript יחיד (מערך CRISPR בודדים (crRNA) המערך). אנו מציגים פרוטוקול לאינטגרציה של מולטיפלקס של גנים מרובים הטרוולוגי לתוך הבית העצמאי של הגנום S. cerevisiae ס באמצעות מערכת crispr/Cas12a עם מספר crRNAs מבוטא מערך crrna בודד. השיטה המוצעת מנצלת את היכולת של S. cerevisiae ס לבצע ב vivo רקומבינציה של שברי DNA כדי להרכיב את מערך crrna יחיד לתוך פלבאמצע שיכול לשמש עבור בחירת טרנספוראנט, כמו גם הרכבה של ה-DNA תורם רצפים המשולבים לתוך הגנום בעמדות מיועדות. Cas12a מתבטא מראש באופן מכוון, הקלה על המחשוף של ה-S. cerevisiae ס הגנום על העמדה המיועדת על פי ביטוי של מערך crrna יחיד. הפרוטוקול כולל את העיצוב והבנייה של מערך crRNA יחיד וביטוי DNA התורם קלטות, ומנצל גישה שילוב עושה שימוש ייחודי 50 bp רצפי מחברי DNA רצפים ושילוב נפרד האגף DNA רצפים, אשר מפשט את תכנון ניסיוני באמצעות סטנדרטיזציה ומודולליזציה ומרחיב את מגוון היישומים. לבסוף, אנו מדגימים טכניקה פשוטה ליצירת פיקסל אמנות שמרים עם מטפל נוזלי אקוסטי באמצעות שונה בצבע קרוטנאיד הפקת זנים שמרים שנבנו.
Introduction
האנזימים crispr/Cas יש ללא ספק מהפכה בביולוגיה מולקולרית והיה אימץ באופן נרחב כלים עבור גנום הנדסה במהירות שהיתה בעבר לא ריאלי1. השינוי הראשון של מערכת הגנום הסרביסים על ידי CRISPR/Cas9 הגנום המערכת העריכה דווחה על ידי dicarlo ואח '. 2, הוכחת גן מוצלח מעלף וביצוע מוטציות הנקודות באמצעות הציג מבחוץ oligונו, מו, הזרמים. עוד שמרים ההתפתחויות בארגז הכלים של CRISPR כללו: רגולציה בתקנה על ידי היתוך של מCas9 בלתי פעיל לזרז מתים (dCas9) עם הטרנססקריפט התחומים כדי לאפשר הפעלה והשתקה של שעתוק3, בקשה עבור שניהם עריכת הגנום ופונקציות רגולציה עבור הנדסת מסלול מטבולית על ידי הפעלה בו זמנית, דיכוי ומחיקה4, מחיקה של שברי גדול מ -S. cerevisiae ס הגנום5, וכרומוזום מרובה fusions6 .
CRISPR/Cas מערכות עריכת הגנום למצוא את המקור שלהם במערכות החיסון אדפטיבית של חיידקים ומערכות אלה הותאמו על ידי ביולוגים מולקולריים לעריכת הגנום. הפונקציונליות שלהם מבוססת על באשכולות קבועים מקובצים באופן סדיר בטווח הקצר (CRISPR) אזורי DNA קידוד RNA אחראי על ההכרה של ה-DNA הזר או RNA ו CRISPR הקשורים גנים (Ca) אשר מקודד RNA-מונחה .שבעה, שמונה,9 בהתבסס על ניתוח הגנום האחרונות של מערכות CRISPR/Cas זה הוצע לחלק את מערכות CRISPR/Cas לשני מחלקות, חמישה סוגים 16 תת-סוגי10. שני הכיתות נבדלים על בסיס ארגון של מכלולי אפקטור המעורבים במחשוף מטרה. בדרך כלל, מערכות CRISPR/Cas עם ארגון רב-ממדי מסווגים כמחלקה 1, ואילו מכלולי משנה אחת של מתחמי שייכות לכיתה 210,11. במאמר זה, אנו לחקור את class 2 סוג V Cas12a, שנקרא בעבר Cpf110,12, שהיא חלופה לכיתה 2 סוג II Cas9. למרות שCas9 מאופיין היטב ונעשה בו שימוש נרחב במחקר, Cas12a מציעה תכונות נוספות12. ראשית, Cas12a צורות מורכב עם CRISPR RNA (crRNA) של 42 כדי 44 נוקלאוטידים מבלי לדרוש עוד מפעיל הפעלה-RNA CRISPR (tracrRNA). לכן, a מדריך קצר RNA יכול להיות מנוצל עריכת הגנום עם CRISPR/Cas12a מערכות לעומת CRISPR/Cas9. שנית, endonuclease ייחודי ו endoribonucחכירה הפעילות של Cas12a מאפשר התבגרות של pre-crRNA שלה13. פעילות RNase זו מאפשרת קידוד של crRNAs מרובים על מערך CRISPR יחיד, בעוד Cas9 דורש את הביטוי הנפרד של כל מה שנקרא מדריך יחיד RNA (sgRNA) או לחילופין ביטוי לדוגמה של endonuclease נוספת ( לדוגמה, Csy4) בשילוב עם מוטיבים הוקרה עבור Csy4 המקיפים כל sgrna14,15. שלישית, Cas12a היעד הכרה באתר דורש מוטיב הסמוך פרוטומי (פאם) בסוף 5 ' היעד ואת דבק לאחר + 18/+ 23 מיקום של פאם שלה וכתוצאה מכך ביקתה DNA עם קצוות דביק, בעוד Cas9 דורש פם ממוקם על 3 ' הקצה מן ה יעד ו-דבק לאחר מיקום -3 ליצור חתכים בקצה קהה ב-DNA12. רביעית, הקונצנזוס ההסכמה הרצף של פאם שונה בין Cas12a ((T) TTV) ו Cas9 (NGG), מה שהופך Cas12a מועמד מבטיח עבור התמקדות מקדם המקדם T-עשיר ושליחות קטלנית רצפים16. לבסוף, מחקר שנערך לאחרונה דיווח ספציפיות יותר היעד עבור Cas12a מאשר יליד Cas917.
אנו מציגים פרוטוקול לשימוש במערכת CRISPR/Cas12a לעריכת הגנום של S. cerevisiae ס עם מיקוד מסוים על המבוא של ביטוי DNA מרובים לתוך הקורה גנומית עצמאית בו זמנית (עריכת הגנום מולטיפלקס) באמצעות מערך crRNA יחיד. השלבים המרכזיים של הפרוטוקול מתוארים באיור 1. כהוכחה למושג, מערכת CRISPR/Cas12a הוחל על הקדמה של שלוש קלטות הביטוי לתוך הגנום של S. cerevisiae ס המאפשרים ייצור של β-קרוטן18 כפי שמוצג באיור 2. ייצור של β-קרוטן משפיע על הפנוטיפ של S. cerevisiae ס: כלומר, על הקדמה מוצלחת של כל שלושת הגנים הטרוולוגיים הדרושים עבור biotenoid הביוסינתזה, התאים הלבנים של cerevisiae ס להפוך צהוב או כתום, בהתאם לחוזק הביטוי של כל אחד מהמקדם של הגן. בשל הקריאה הפשוטה ויזואלית של מסלול זה, הוא הוצג לפתח מערכות מבוססות crispr מבוסס ושיטות לעריכת הגנום19,20. בעבודה זו, קלטות ביטויים הקידוד של הגנים קרוטאיד Crte, crtyb ו crte נבנו באמצעות שיבוט שער הזהב (ggc) גישה21 עם הטרוולוגיים היזמים ומדבירי הומוולוגי משמש כדי לנהוג בביטוי של הגנים. קלטות הביטויים מוקפות ברצפים ייחודיים של 50-בסיס (bp), המכונים מחברים, המאפשרים בהרכבה vivo עם מאגף שילוב רצפי DNA (אזורים מאגפים) עם רצפים בעלי אותו 50 bp, ואינטגרציה עוקבת לתוך ה-DNA גנומית של שמרים בעמדה שנקבעת על ידי האזורים האגפים. באמצעות החוזקות המקדם שונים, זנים עם רמות שונות של ייצור קרוטנואידים הושגו וכתוצאה מכך וריאציה בצבע של התאים. אלה זנים בהשראת "פרויקט שמרים אמנות"22 -שימשו בכיוונון האיתור עם מטפל נוזלי אקוסטי כדי ליצור 4 צבעים ברזולוציה גבוהה "צילום שמרים" של רוזלינד פרנקלין. פרנקלין (1920-1958) היה כימאי אנגלי ו-X-ray קריסטלוגרף ידוע בשל תרומתו לגילוי של מבנה ה-DNA על ידי צילום 5123,24,25.
Protocol
1. הכנת הפלמידים Cas12a
הערה: הפלסמיד המכיל את החיידק הND2006 Cas12a (LbCpf1, pCSN067) קודון אופטימיזציה לביטוי ב- S. cerevisiae ס, נבנה בעבר19, הופקד במאגר פלאמצע (לראות את השולחן של חומרים). זהו עותק יחיד של epiזומיום S. cerevisiae ס/E. coli המעבורת באמצע המכיל גן התנגדות kanmx הסמן כדי לאפשר בחירה של S. cerevisiae ס Transformants על גנטיסין (G418).
- השג את pCSN067 פלמיד (ראה את טבלת החומרים).
- להגביר את הפלpCSN067. כדי להשיג סכום גבוה
- לשנות את 25 μL של נרכש תאים כימית E. coli המוסמכת עם pCSN067 באמצע לפי פרוטוקול של היצרן. לדלל את ערבוב שינוי 10 ו 50 פעמים ב-2x peptone-שמרים (PY). צלחת החוצה 10x ו 50x דילול על לוחות 2x PY אגר המכיל אמפיצילין (0.1 g/L) ו מודקות לילה ב 37 ° c.
- בחר 2 כדי 3 מושבות ומחסן כל מושבה ב 3 מ ל של 2x PY ולגדול לילה ב 37 ° c בחממה רועדת ב 180 rpm.
- לטהר את הפלביניים באמצעות ערכת טיהור פלמיד על פי הוראות היצרן.
2. הכנת הקסטה היחידה של ביטוי מערך crRNA
- הכן את מערך ה-crRNA היחיד.
הערה: מערך crRNA יחיד כולל מקדם SNR52 RNA פולימראז III מ -S. cerevisiae ס2, לחזור ישיר ספציפי עבור LbCas12a ומרווח (רצף היעד גנומית), יחד חוזרים על כל יעד19 ומסתיים עם SUP4 המחסל מ- S. cerevisiae ס2. מערך crrna יחיד מורכב על ידי בvivo רקומבינציה לתוך הפלסטיק להפוך pRN1120 כדי ליצור פלבאמצע מעגלי, ולכן אזורים הומולוגיים כדי פלאמיד pRN1120 חייב להיות נוכח בתחילתו ובסופו של מערך crrna יחיד (ראה איור 2a ). מומלץ מראש להעריך את הפונקציונליות של מספר crRNAs מעוצב בנפרד19. מידע זה משמש לאחר מכן כדי לבחור ב-crRNAs הפונקציונליים ביותר כדי לשלב אותם ברצפי החזרה והרווח הישירים כדי ליצור מערך crRNA יחיד עבור מטרת ריבוב.- הזמנת מערך crRNA יחיד לניסויים בהגנום מולטיפלקס כמו דנ א סינתטי (ראה רצף ה-DNA של מערך crRNA יחיד בטבלה המשלים 1).
- ההגביר את מערך crRNA המסודרת בודד (לדוגמה, באמצעות התחל KC-101 ו-KC-102 (טבלה משלימה 2)). הכינו את שילוב הגברה PCR המכיל: 0.5 μL של דנ א פולימראז, 10 μL של מאגר 5x הנדרש DNA פולימראז, 1 μL של 10 מ"מ dNTPs, 2.5 μL של 10 μM קדימה פריימר, 2.5 μL של 10 μM פריימר הפוכה, 2 μL של תבנית DNA בריכוז של 5 ng/μL ו מיכל השני O עד נפח כולל של 50 μL.
- בצע את התגובה באמצעות התוכנית הבאה: (i) 98 ° צ' עבור 3 דקות, (ii) 98 ° צ' עבור 10, (iii) 60 ° צ' עבור 20, (iv) 72 ° צ' עבור 15 s-לחזור על הצעדים (ii) 30 פעמים, (v) 72 ° c עבור 5 דקות (vi) להחזיק ב 12 ° c עד לניתוח נוסף.
- לנתח את מוצרי ה-PCR על ידי אלקטרופורזה על ידי הפעלת דגימות על 0.8% agarose ג'ל ב 5 V/cm עבור 40 דקות באמצעות צבע הטעינה DNA ו-DNA סולם עם שברי DNA במגוון של 100 כדי 10,000 bp.
- טיהור מוצרי ה-PCR באמצעות ערכת טיהור PCR לפי הוראות היצרן.
- הכן את הנמען היחיד של מערך crRNA.
הערה: מערך ה-crRNA היחיד מתבטא מ -S. cerevisiae ס/E. Coli המעבורת pRN112019 (ראה את שולחן החומרים). זה multi-העתק פלמיד מכיל גן התנגדות NatMX סמן כדי לאפשר בחירה של S. cerevisiae ס transformants על nourse, המנתצין (ntc).- השג את pRN1120 פלמיד.
- להגביר את הפלpRN1120. כדי להשיג סכום גבוה
- לשנות את 25 μL של נרכש תאים כימית E. coli המוסמכים עם pRN1120 בהתאם לפרוטוקול של היצרן. לדלל את ערבוב שינוי 10 ו 50 פעמים ב 2x PY. צלחת החוצה 10x ו 50x דילול על לוחות 2x PY אגר המכיל אמפיצילין (0.1 g/L) ו מודקות לילה ב 37 ° c.
- בחר 2 כדי 3 מושבות ומחסן כל מושבה ב 3 מ ל של 2x PY ולגדול לילה ב 37 ° c בחממה רועדת ב 180 rpm.
- לטהר את הפלסטלינה באמצעות ערכת טיהור פלמיד על פי הוראות היצרן.
- לינאריזה פלמיד pRN1120 עם EcoRI-HF ו xhoI. בשביל זה, להכין שילוב העיכול מורכב 1 μg של pRN1120, 5 μL של מאגר 10x (מאגר 1x מכיל 50 מילימטר אשלגן אצטט, 20 mM טריס-אצטט, 10 מ"מ אצטט מגנזיום, 100 μg/mL סרום הפרה אלבומין [BSA]; pH 7.9), 1 μL של אקוRI-HF (20 U , 1 μl של xhoI (20 U) ו-H באלקטרופורזה2O עד נפח כולל של 50 μl. דגירה את תערובת העיכול ב 37 ° צ' עבור 2 H ו להשבית ב 65 ° c עבור 20 דקות.
- לנתח את הפלסטיק לינבית על ידי אלקטרופורזה על ג'ל agarose (0.8%, 40 דקות, 5 V/cm) באמצעות צבע הטעינה DNA ו-DNA סולם עם שברי דנ א במגוון של 100 כדי 10,000 bp. כפקד כולל פלסמיד עגול בניתוח.
- לטהר את הפלבאמצע באמצעות ערכת טיהור PCR בהתאם להוראות של היצרן.
3. הכנת היזם-ORF-שליחות קטלנית (POT) תורם DNA בנייה
- להזמין קבוצה של היזם (P) של כוח שונה, פתח מסגרת קריאה (O) ו שליחות קטלנית (T) רצפים כמו דנ א סינתטי, כך שכל אלמנט מכיל רצפי זיהוי 4-bp מתוקננת כי הם מקיפים על ידי אתרי BsaI כדי לאפשר שיבוט שער הזהב ( GGC) מכלול26 (ראה את העיצובים המפורטים בטבלה 3 ורצפים משלימים בטבלה השלמה 4).
- להרכיב ביטוי POT קלטות המורכב מיזם, פתח מסגרת קריאה, שליחות קטלנית ורצפי מחברים באמצעות הרכבה 4 חלקים באמצעות תגובה GGC21, לתוך וקטור יעד שכבר מכיל מראש שצוין 50-bp רצפי מחברים ( ראה טבלה משלימה 4 והפניות26,27).
- למדוד את הריכוז של חלקי DNA באמצעות ספקטרוסקופיה. לדלל כל חלק DNA ב-H באלקטרופורזה2O לריכוז הסופי של 15 fmol/μl.
- להכין מיקס תגובה מורכב של שברי דנ א: 2 μL של יזם, 2 μL של מסגרת קריאה פתוחה, 2 μL של שליחות קטלנית 2 μL השדרה (רמה 1 וקטורים היעד כפי שמתואר 26), 4 μl של מאגר ה-dna של 5 x t4, 2.5 μl של 1 U/μl של t4 , 1.5 μL של 20 U/μL BsaI-HF ו-H באולטרסאונד למעלה2O עד נפח כולל של 20 μl.
- בצע את תגובת GGC בציקלטרנר באמצעות התוכנית הבאה: (i) 37 ° צ' עבור 2 דקות, (ii) 16 ° צ' עבור 5 דקות – חזור על שלבים (i) ו-(ii) 50 פעמים, (iii) 50 ° צ' עבור 60 דקות, (iv) 80 ° צ' עבור 45 דקות, (v) להמתין 12 ° צ' עד
- לשנות את 25 μL של נרכש כימית E. coli28 תאים עם 3 μl של התגובה ggc לערבב על פי פרוטוקול של היצרן. לדלל את ערבוב שינוי 10 ו 50 פעמים ב 2x PY. צלחת החוצה 10x ו 50x דילול על לוחות 2x PY אגר המכיל אמפיצילין (0.1 g/L) ו מודקות לילה ב 37 ° c.
- בחר 2 כדי 3 מושבות ומחסן כל מושבה ב 3 מ ל של 2x PY ולגדול לילה ב 37 ° c בחממה רועדת ב 180 rpm.
- לטהר את הפלמידים באמצעות ערכת טיהור פלמיד על פי הוראות היצרן.
- בדוק אם קלטות ביטוי חשיש התאספו כראוי בתגובה GGC על ידי ה-PCR.
- עיצוב משלים את רצף המחברים שנמצא בתחילתו ובסופו של כל קלטת ביטוי (ראה איור 2B). עבור מחברים שנבחרו בפרוטוקול זה להשתמש בלוח התחל KC-103 כדי KC-108 (ראה טבלה משלימים 2).
- הכנת תערובות PCR הגברה עבור כל פלסטלינה המכיל: 0.5 μL של הגהה DNA פולימראז, 10 μL של מאגר 5x נדרש עבור ה-DNA פולימראז, 1 μL של 10 מ"מ dNTPs, 2.5 μL של 10 μM קדימה פריימר, 2.5 μL של 10 μM הפוכה פריימר, 2 μL של תבנית DNA עם מרכז יון של 5 ng/μl, ו-H באלקטרופורזה2O עד נפח כולל של 50 μl.
- לבצע את תגובת ה-PCR באמצעות התוכנית הבאה: (i) 98 ° c 3 דקות, (ii) 98 ° צ' עבור 10, (iii) 60 ° צ' עבור 20, (iv) 72 ° צ' עבור 2 דקות 30-שלבים חוזרים (ii) ל-(iv) 30 פעמים, (v) 72 ° c עבור 5 דקות (vi) החזיקו ב-12 ° צ' עד לניתוח נוסף.
הערה: מוצרי ה-PCR כתוצאה מורכב 50-bp של 5 ' מחבר, יזם, לפתוח את מסגרת הקריאה, שליחות קטלנית ו 50-bp של מחבר 3 '.
- לנתח את מוצרי ה-PCR על ידי אלקטרופורזה על ידי הפעלת דגימות על 0.8% agarose ג'ל ב 5 V/cm עבור 40 דקות באמצעות צבע הטעינה DNA ו-DNA סולם עם שברי DNA במגוון של 100 כדי 10,000 bp.
4. הכנת רצפי דנ א מאגף המכיל רצפי מחברים
- לטהר דנ א גנומית מסוג פראי S. cerevisiae ס PK113-7d29.
- לגדל את המתח בבקבוקון 500 mL מילא עם 100 mL של תמצית שמרים peptone דקסטרוז (YEPD, 2% גלוקוז) בינוני ב 30 ° צ' ורועד ב 250 rpm עבור 48 שעות.
- לקצור את התאים על ידי צנטריפוגה של 2 מ ל של ציר ב 16,000 x g עבור 1 דקות ולהיפטר supernatant.
- השהה מחדש את התאים במלח הפיזיולוגי (200 μL; 0.85% הפתרון הנכון) עם RNase (10 μL, 10 מ"ג/mL) ו אנזים lytic שמרים (4 μL). מכבית את השעיית התא ב 37 ° c עבור 15 דקות.
- הוסף 300 μL של פתרון לפירוק תאים (ראה טבלת חומרים) ומערבולת בקרוב.
- הוסף 168 μL של פתרון משקעים בחלבון (ראה טבלת חומרים) ומערבולת במרץ עבור 20 s.
- הפרד את שבר החלבון על ידי צנטריפוגה ב 16,000 x g ו 4 ° c עבור 10 דקות. לאסוף 600 μl של supernatant בצינור חדש ולערבב עם 600 μl של איזופנול ומערבולת בקרוב.
- לשחזר את ה-DNA על ידי ספינינג ב 16,000 x g בטמפרטורת החדר עבור 10 דקות. להיפטר הסופרנטאנט ולשמור את הגלולה.
- לשטוף את הגלולה עם 200 μL של אתנול (70%). צנטריפוגה ב 16,000 x g בטמפרטורת החדר עבור 10 דקות ולהסיר את סופרנטאנט. להתנדף את האתנול על ידי דגירה הצינור בטמפרטורת החדר עבור 10 דקות עם המכסה נפתח.
הערה: אם הנוזל בצינורית עדיין גלוי, חזור על השלב 4.1.8. לא לייבש את הגלולה עבור יותר מ 10 דקות כדי למנוע מסיסות ירידה של ה-DNA. - התמוססות ה-DNA ב 50 μL של מאגר TE. החנות DNA מטוהרים ב 4 ° c.
- עבור כל אתר שילוב, שילוב עיצוב האגף רצפי DNA (כ. 500 bp) כזה כ 1000 bp של דנ א גנומית יהיה להסיר על הקדמה של ה-DNA תורם (לראות את העיצוב הסכמטי באיור 2B ורצפים משלימים שולחן 4).
- עיצוב התחל לייצר את האזורים האגפים על ידי ה-PCR.
- עבור האזור השמאלי האגף, לעצב קדימה והפוך התחל להגביר כ 500 bp של אזור ה-DNA גנומית ממוקם 5 ' (משמאל) של אתר האינטגרציה של עניין.
הערה: התחל הקדמי כולל 20 bp של הומולוגיה עם האזור המיועד לאגפים. פריימר הפוכה כוללת 20 bp עם הומולוגיה עם האזור המיועד האגף ומכיל את הרצוי 50-bp רצף מחבר כדי לאפשר את ההרכבה vivo בעריכה Cas12a על הגנום בהמשך. - עבור אזור מימין, לעצב קדימה הפוך התחל להגביר כ 500 bp של אזור ה-DNA גנומית ממוקם 3 ' (מימין) של אתר האינטגרציה של עניין.
הערה: התחל הקדמי כולל 20 bp עם הומולוגיה עם האזור המיועד לאגף ומכיל את הרצף הרצוי 50-bp מחבר כדי לאפשר את ההרכבה vivo בעריכה Cas12a על הגנום בהמשך. ההילוך ההפוך כולל 20 bp של הומולוגיה עם האזור המיועד האגף.
- עבור האזור השמאלי האגף, לעצב קדימה והפוך התחל להגביר כ 500 bp של אזור ה-DNA גנומית ממוקם 5 ' (משמאל) של אתר האינטגרציה של עניין.
- הימבו את האזורים האגפים בצבעי היסוד המעוצבים (למשל, מ109, לדוגמא, לכיוון kc-120 המוקפים בטבלה השלמה 2).
- למדוד את הריכוז של דנ א מטוהרים גנטי שישמש כתבנית ב-PCR. כוונן את ריכוז ה-DNA כדי 50 ng/μL.
- הכנת תערובות PCR הגברה המורכבת של דנ א גנומית (1 – 4 μL של 50 ng/μL DNA דילול) מטוהרים בשלב 4.1, קדימה והפוך פריימר (10 μM כל אחד), 1 μL של 10 מ"מ dNTPs, 10 μL של מאגר 5x נדרש DNA פולימראז, 0.5 μL של DNA פולימראז (1.0 U , ו-H באולטרסאונד למעלה2O עד נפח הכולל של 50 μl.
- בצע בדיקות באמצעות התוכנית הבאה: (i) 98 ° c עבור 3 דקות, (ii) 98 ° צ' עבור 20 s, (iii) 60 ° צ' עבור 20, (iv) 72 ° צ' עבור 15 s, חזור על הצעדים (ii) ל (iv) 30 פעמים, (v) 72 ° c עבור 5 דקות (vi) החזיקו ב-12 ° צ' עד לניתוח נוסף.
- לנתח את מוצרי ה-PCR על ידי אלקטרופורזה על 0.8% agarose ג'ל ב 5 V/cm עבור 40 דקות באמצעות צבע הטעינה DNA ו-DNA סולם עם שברי DNA במגוון של 100 כדי 10,000 bp.
- טיהור מוצרי ה-PCR הנכונים באמצעות ערכת טיהור ה-PCR בהתאם להוראות היצרן.
5. טרנספורמציה ל- S. cerevisiae ס
הערה: בצע טרנספורמציה באמצעות פרוטוקול המבוסס על השיטות שפותחו על ידי Gietz ואח '. (1995) 30 וגבעה ואח '. 31 אשר ניתן להשתמש עבור זנים שונים של S. cerevisiae ס. הפרוטוקול המתואר להלן מספיק עבור המרה 1.
- הכינו פתרונות הדרושים לשינוי.
- הכן את פתרונות המניה הבאים וסנן-חטא: 10x TE מאגר המכיל 100 mM טריס-HCl (pH 7.5), 10 מ"מ EDTA, נפח כולל של 50 mL; 1 M LiAc ב-pH 7.5, נפח כולל של 50 mL; 50% פג 4000, נפח כולל של 100 mL.
הערה: תמיד לבדוק כי יתד 4000 מניות הוא ב-pH 5. אין לאחסן מלאי זה יותר מחודש אחד. - הכינו את הפתרונות הבאים באמצעות מניות: להכין את הפתרון LiAc-TE המכיל 0.1 M LiAc, 10 מ"מ טריס-HCl, 1 מ"מ EDTA, נפח כולל של 0.5 mL. הכנת יתד-LiAc-TE פתרון המכיל 40% פג 4000, 0.1 M LiAc, 10 מ"מ טריס-HCl, 1 מ"מ EDTA, נפח כולל של 1 mL.
הערה: חיוני לשינוי מוצלח כי הפתרונות של פג-LiAc-TE ו-LiAc-TE מוכנים טרי.
- הכן את פתרונות המניה הבאים וסנן-חטא: 10x TE מאגר המכיל 100 mM טריס-HCl (pH 7.5), 10 מ"מ EDTA, נפח כולל של 50 mL; 1 M LiAc ב-pH 7.5, נפח כולל של 50 mL; 50% פג 4000, נפח כולל של 100 mL.
- השינוי הראשון בסיבוב (להכין את המתח מראש מביע Cas12a).
הערה: בכל שלבי הטרנספורמציה, השתמש במים עם pH גבוה מ -5. מומלץ להשתמש במים מינרלים בכל שלבי הטרנספורמציה.- להכין טרום תרבות על ידי מאמץ גדל מרכז. PK113-7D בבקבוקון לנער 100 mL המכיל 20 מ ל של YEPD (2% גלוקוז) בינוני ו-דגירה לילה ב 30 ° צ' עם טלטול ב 250 סל ד.
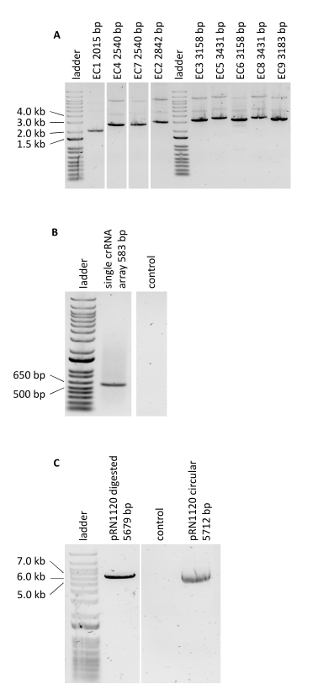
- מדוד את ה-OD600 של הקדם-תרבות (odpc). לחשב את מקדם הדילול (df) בין נפח של טרום תרבות לבין נפח של בינונית טרייה הנדרשת להכנת התאים הCas12a מראש כדי לשמש את הטרנספורמציה (תרבות טרנספורמציה). בחישובים להניח את הצפיפות האופטית של תרבות הטרנספורמציה (ODtc) להיות 1.0 לאחר שלב הדגירה המתואר 5.2.3 (ti).

כאשר ti ו-τ הם זמן הדגירה וזמן ההכפלה, בהתאמה.- חשב את עוצמת הקול של הקדם-תרבות (vi) הדרוש לחיסונים של תרבות הטרנספורמציה (vtc) המבוססת על פקטור הדילול.
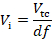
- חשב את עוצמת הקול של הקדם-תרבות (vi) הדרוש לחיסונים של תרבות הטרנספורמציה (vtc) המבוססת על פקטור הדילול.
- להכין את תרבות הטרנספורמציה על ידי החיסון 20 מ ל של YEPD (2% גלוקוז) (Vtc) עם נפח של טרום תרבות נקבע בשלב הקודם (vi). מודטה ב 30 ° צ' עם טלטול ב 250 סל ד.
- מדוד את ה-OD600 של תרבות הטרנספורמציה עד שתגיע ל-od600 של 1.0.
- לקצור את התאים על ידי צנטריפוגה של 20 מ ל ציר ב 2,500 x g עבור 5 דקות. לבטל את הסופרנטאנט ולשטוף את התאים ב 20 מ ל של טמפרטורת החדר מים מינרלים. חזור על הצעד הצנטריפוגה ושמור את הגלולה הסלולרית.
- השהה מחדש את התאים ב-100 μL של פתרון LiAc-TE והעבר לשפופרת מיקרוצנטריפוגה.
- הוסף 5 μL של ה-DNA המוביל יחיד-תקוע (10 מ"ג/mL סלמון DNA זרע) ולערבב על ידי ליטוף.
- פיפטה 1 μg של פלסמיד pCSN067 לצינור המיקרוצנטריפוגה.
הערה: הנפח הכולל של תערובת ה-DNA לא יעלה על 100 μL כדי למנוע יעילות שינוי נמוכה יותר. - הוסף 600 μL של יתד-LiAc-TE פתרון ומערבבים על ידי ליטוף. מודטה עבור 30 דקות ב 30 ° צ' בעוד רועד ב 450 סל ד בבלוק חום העליון שולחן.
- הוסף 70 μL של DMSO (100%) לתערובת הטרנספורמציה ומערבבים באמצעות ליטוף. לבצע זעזוע חום על ידי הדגירה של תערובת השינוי ב 42 ° c עבור 15 דקות באמבט מים.
- לשחזר את התאים על ידי העברת התערובת ל 15 מ"ל בתחתית הצינור התחתון ולהוסיף 10 מ ל של YEPD (2% גלוקוז) לצינור. דגירה הלילה ב 30 ° צ' עם טלטול ב 250 סל ד.
- צנטריפוגה את ערבוב שינוי ב 2,500 x g עבור 5 דקות. למחוק את supernatant ולהשעות מחדש את הגלולה תא ב כ 200 μl של הפתרון הנותר.
- צלחת החוצה 150 μL של תערובת שינוי ו-20x דילול ב YEPD (2% גלוקוז) של ערבוב שינוי ב YEPD (2% גלוקוז) אגר צלחות בתוספת עם 0.2 g/L G418. מודיית את לוחיות ב 30 ° צ' עבור 48 – 72 שעות.
- בחר טרנספורנט בודד ו-רצף מחדש על YEPD (2% גלוקוז) צלחת אגר בתוספת עם 0.2 g/L G418 כדי להשיג מושבות יחיד.
- השינוי השני בסיבוב (לבצע עריכת הגנום מולטיפלקס עם CRISPR/Cas12a).
- להכין טרום תרבות על ידי הגדלת מאמץ מראש Cas12a, נוצר בסיבוב השינוי הראשון (שלב 5.2), בבקבוקון לנער 100 mL המכיל 20 מ ל של YEPD (2% גלוקוז) בינוני בתוספת 0.2 g/L G418. דגירה הלילה ב 30 ° צ' עם טלטול 250 סל ד.
הערה: עבור שינויי צורה מרובים, התאם את הנפח של הקדם-תרבות. - בצע את השלבים 5.2.2 כדי ל5.2.7 לסיבוב השינוי הראשון.
הערה: עבור שינויי צורה מרובים, התאם את אמצעי האחסון של פתרונות נדרשים והתרבות של המאמץ לבטא מראש את Cas12a. - פיפטה 1 μg של מערך crrna יחיד, 1 μg של הנמען המקבל לליניארית אמצע עבור מערך crrna, 1 μg של ה-DNA של התורם ו-1 μg של כל אזור האגפים (שלב 4.3) בצינורית המיקרוצנטריפוגה.
הערה: הנפח הכולל של תערובת ה-DNA לא יעלה על 100 μL כדי למנוע יעילות שינוי נמוכה יותר. - הכן את הפקדים הבאים עבור השינוי: פקד שלילי (H2בדיקת אולטרה-סאונד); בקרה חיובית לקביעת יעילות השינוי (1 μg של מעגלי pRN1120); פקד אימות אם מבוא של ה-DNA התורם מתנהל באמצעות עריכת CRISPR (1 μg של pRN1120 עגול, 1 μg של כל הביטוי ה-DNA התורמים קלטות ו 1 μg של אזורים האגפים, אבל לא מערך crRNA יחיד); שליטה אימות אם ה-DNA התורם יכול להיות משולב מחוץ ליעד (1 μg של לינpRN1120, 1 μg של הביטוי DNA התורם קלטות ו 1 μg של מערך crRNA יחיד אבל לא אזורים האגפים); פקד המאשר שורה מלאה של pRN1120 (1 μg של לינבית pRN1120).
- בצע את השלבים 5.2.9 כדי ל5.2.12 לסיבוב השינוי הראשון.
- צלחת החוצה 150 μL של ערבוב שינוי ו 20x דילול ב YEPD (2% גלוקוז) של ערבוב שינוי ב YEPD (2% גלוקוז) אגר בתוספת עם 0.2 g/L G418 ו 0.2 g/L NTC. הצלחת להוציא את הלוח על YEPD (2% גלוקוזה) אגר בתוספת עם הבחירה המתאימה (G418 ו/או NTC או לא בחירה). מודיית את לוחיות ב 30 ° צ' עבור 48 – 72 שעות.
- בחר טרנספורנט בצבע אחד ומחדש רצף על YEPD (2% גלוקוז) צלחת אגר להשיג מושבות צבעוניות אחת.
- להכין טרום תרבות על ידי הגדלת מאמץ מראש Cas12a, נוצר בסיבוב השינוי הראשון (שלב 5.2), בבקבוקון לנער 100 mL המכיל 20 מ ל של YEPD (2% גלוקוז) בינוני בתוספת 0.2 g/L G418. דגירה הלילה ב 30 ° צ' עם טלטול 250 סל ד.
6. הערכה של יעילות עריכת הגנום
- ספרו את מספר המושבות הצבעוניות והמושבות הלבנות על לוחיות השינוי.
- חישוב יעילות של עריכת הגנום על-ידי חלוקת מספר המושבות הצבעוניות במספר הכולל של מושבות (לבנים וצבעוניים), כפי שמוצג בטבלה 1.
7. אישור שילוב דנ א של תורם בבית המיועד
- Re-פס מושבה אחת צבעונית מצלחת טרנספורמציה על YEPD (2% גלוקוז) צלחת אגר ללא G418 ו NTC בחירה ו מודקת עבור 48 שעות ב 30 ° c.
- בחר מושבה אחת והאיחסן 500 mL לנער בקבוקון ממולא 100 mL של YEPD (2% גלוקוז) בינוני. מודטה עבור 48 שעות ב 30 ° צ' ורועד ב 250 סל ד.
- לבודד את ה-DNA גנומית כפי שמתואר בסעיף 4.1.
הערה: לחלופין, השתמש בפרוטוקול להכנת שמרים למושבת ה-PCR שהוצע בעבר על-ידי לינקי et al. 32. במקרה זה ניתן לדלג על גידול במדיום הנוזלי (סעיף 7.2). - ודא שילוב נכון על-ידי הגברה של שני קטעים לכל קלטת ביטוי משולבת.
- עיצוב התחל אשר מספח ל-DNA גנומית מחוץ לאזורים שהפכו שינוי גן העניין (ראה דוגמאות בטבלה משלימים 2, kc-121 כדי KC-132). כאשר משתמשים ב-KC-121 ל-KC-132, הגדר את טמפרטורת הריפוי בתוכנית ה-PCR ל-62 ° c.
- ההגביר את אזור העניין כמתואר בסעיף 4.4.2. להתאים את התוכנית ה-PCR, במיוחד להתאים את הזמן של שלב ההארכה ב-PCR בהתאם לאורך ההמלצות של התבנית והיצרן עבור ה-DNA פולימראז.
- בדוק את הגודל של מוצרי ה-PCR על ידי אלקטרופורזה ב-agarose ג'ל (0.8%, 40 דקות, 5 V/ס מ) באמצעות צבע הטעינה DNA וסולם DNA עם שברי DNA במגוון של 100 כדי 10,000 bp.
8. יצירה של פיקסל שמרים אמנות באמצעות מטפל נוזלי אקוסטי
- הכינו תבנית תמונה לאמנות הפיקסלים המרים.
- שנה את גודל תמונת ה-RGB המקורית (220 × 280 פיקסלים, עיין בתוצאות הנציגים), לדוגמה , באמצעות imagej כדי ליצור את התמונה הסופית 64 × 96 פיקסלים (רוחב × גובה) תמונות בקנה מידה אפור דמיינו בצבעים מיועדים (תוצאות מייצגות).
- המירו את תמונת ה-RGB לקנה מידה אפור באמצעות נוסחה זו:

היכן שאני, אני, אני, אני , אניב' , הם כוונות אפורות, אדומות, ירוקות וכחולות, בהתאמה. - כדי לסווג את הפיקסלים, לפתח התוסף ImageJ החלת הכללים הבאים: (א) אם אניgr הוא ≤ 64, השתמש בשמרים כתום כהה (זן 1, הטבלה המשלים 3) עבור פיקסל זה. (ב) אם 64 < Igr ≤ 128, השתמש שמרים כתום (זן 2, המשלים טבלה 3) עבור פיקסל זה. (ג) אם 128 < Igr ≤ 192, השתמש בשמרים הצהובים (זן 3, שולחן משלים 3) עבור פיקסל זה. (ד) אם אניgr > 192, השתמש בשמרים הלבנים (מרכז PK113-7D) עבור פיקסל זה.
- ספוט תאים שמרים ליצור אמנות פיקסל שמרים.
- האיחסן 500 mL מבחנות לנער המכיל 100 mL של YEPD (2% גלוקוז) בינוני עם שלושה קרוטאיד בצבע שונה לייצר S. cerevisiae ס זן מסוג פראי. . PK113-7 ד תרביות התרבות הלילה ב 30 ° צ' עם טלטול ב 250 סל ד.
- העברת 0.5 mL של התרבות לילה לצינור ממולא 0.5 mL של בינונית ללא מעבר הצפיפות הבלתי יונית של בינוני (לראות את הטבלה של חומרים). מערבבים על ידי הורטקנג בקצרה.
- העבר את ההשעיה התא למאגר מוסמך, 2 x 3 היטב. לבצע האיתור באמצעות מכשיר אקוסטי נוזלי מטפל מלוח מקור מוסמך מאגר מיקרופלייט (לראות את הטבלה של חומרים) המכיל 50 ML של yepd (2% גלוקוז) אגר. כדי לפשט את הציפוי, להגדיר בארות על צלחת, למשל. להשתמש במיקרופלייט כמו צלחת 6144 הבאר (64 × 96).
- ספוט 25 nL של מאמץ של כל S. cerevisiae ס מ 2x 3 הצלחת מאגר המאגר היטב באמצעות קובץ. csv עם הגדרת כיול הנוזלים 6RES_AQ_GPSA2 אל מיקרופלייט היעד. להגדיר כל אלה 25 nL טיפות כפיקסל ב 64 x 96 הרשת אשר מתורגמת היטב עמדות (A01, B01, C01 וכו ').
- מודקת את המיקרוצלחת ב 30 ° c עבור 48 שעות. כדי להעצים את הצבעים של זנים לאחסן את הצלחת אגר ב 4 ° c עבור לפחות 72 שעות.
תוצאות
הפרוטוקול לעריכת הגנום מולטיפלקס באמצעות CRISRP/Cas12a הפגינו על ידי בניית שלושה קרוטאיד הפקת זנים S. cerevisiae ס ביטוי את crte, Crtyb ו crte גנים באמצעות הטרוולוגי היזמים של גבוה, בינוני ונמוך חוזק: זן 1, 2 ו, 3 בהתאמה (שולחן משלים 3). הבנייה של זנים אלה נדרש הדור של שלושה ביטוי DNA התורם קלטות ושישה אזורים האגפים לכל זן עבור התמקדות שלושה שונים היכן ב-DNA גנומית (המוצג באיור 2ב). כפי שמתואר לעיל, יזם, מסגרת קריאה פתוחה, שליחות קטלנית ושני רצפים רציפים 50-bp התאספו לתוך קלטת ביטוי באמצעות תגובת שיבוט שער הזהב וההרכבה אומתה על ידי ה-PCR (איור 3א). מערך ה-crRNA היחיד הוזמן כרסיס DNA סינתטי והוגדל על ידי ה-PCR (איור 3ב). הנמען הדוק עבור מערך crRNA יחיד (פלמיד pRN1120) היה לינארי עם EcoRI-HF ו xhoI ו לינלון אושרה על ידי אלקטרופורזה (איור 3ג). העיצוב והנוקלאוטידים של ביטוי ה-DNA של התורמים המוצגים והאזורים האגפים מוצגים בטבלה השלמה 3 ובטבלה משלימה 4. הרצף של ביטוי מערך crRNA בודד מסופק בטבלה משלימה 1. פונקציונליות של הרווחים כלולים במערך crRNA יחיד נבדק מראש על ידי עריכת הגנום בודד עם crRNAs בודדים19.
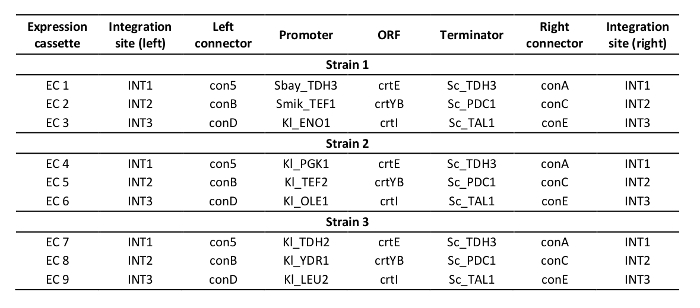
היעילות של עריכת הגנום באמצעות Cas12a היה ראשית הוערך מבוסס על מספר מושבות צבעוניות שהתקבלו לאחר שינוי (שולחן 1, איור 4). יעילות העריכה של שלושה זנים שנבנו מגוון מ 50% כדי 94%. בעיקר, המבוא של קלטות הבעה המשמש להפקת זן 1 הציג את יעילות העריכה הנמוכה ביותר, שנגרמה כתוצאה מאופיו של ה-DNA התורם (כלומר, הקלטות האלה מקודד crte, Crtyb ו crte משלושה מיזמים בעלי עוצמה גבוהה). שנית, השילוב הנכון של שלושה ביטוי DNA התרומה הקלטות בבית המיועד על ה-DNA גנומית היה אישר ב-PCR (איור 5). התחל תוכנן בצורה כזאת שמוצרי PCR הושגו כאשר השילוב הנכון של הדנ א של התורם במצב המיועד התרחש. עבור כל ניסוי שינוי, שמונה מושבות נאספו מלוחית השינוי ונבדקו (שימו לב שרק שלושה מוצגים באיור 5). באופן כללי, מתוך 8 מושבות נבדק ל-DNA התורם, שילוב נכון של ה-DNA של תורם Crte בINT1 לוקוס, crtyb ב INT2 לוקוס ו crte ב INT3 לוקוס אושרה ב > 90% מtransformants. תוצאות אלה מדגימים את מערכת CRISPR/Cas12a בשילוב עם מערך crRNA יחיד מאפשר עריכה משולבת יעילה של הגנום S. cerevisiae ס בבית מרובים בו זמנית.
בנוסף, אנו להדגים את היצירה של "פיקסל שמרים אמנות" באמצעות שלושה מייצרים קרוטנאיד זנים שנבנו יחד עם זן פראי בצבע בר. החל מתמונה שחור-לבן של רוזלינד פרנקלין (איור 6א), תמונה 4 צבעים (איור 6B) ורשימת האיתור נוצר אשר השתמשו לזהות את ארבעת זנים שמרים שונים על מיקרולוח אגר באמצעות מטפל נוזלי אקוסטי, וכתוצאה מכך "ציור שמרים" ברזולוציה גבוהה של רוזלינד פרנקלין (איור 6C, D, E).

איור 1 : זרימת העבודה של פרוטוקול עבור CRISPR/Cas12a הגנום מערכת עריכה ב- S. cerevisiae ס. זרימת העבודה כוללת צעדים מכריעים של השיטה המוצגת. לקבלת פרטים, עיין בפרוטוקול. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2 : ערכה של CRISPR/Cas12a הגנום מולטיפלקס באמצעות מערך crRNA יחיד. (א) מערך ה-crrna היחיד מורכב משלוש יחידות crRNAs בצורה בוגרת שלהם, מערכת חוזרת ישירה של 20 Bp לLbCas12a (ריבועים אפורים) עם רצף מדריך 23-bp (יהלומים צבעוניים). ביטוי של מערך crRNA מופעל על ידי המקדם SNR52 ו SUP4 שליחות קטלנית. טרנספורמציה של ס' cerevisiae ס עם pRN1120 להמרה ואת מערך crrna ביטוי הקלטת המכילה הומולוגיה עם pRN1120 (פסים אלכסוניים) מאפשר בvivo שילוב מעגלי לתוך פלבאמצע בתאים מראש הביטוי LbCas12a. מערך ה-crRNA היחיד מעובד לאחר מכן על-ידי Cas12a. (ב) Cas12a מכוונת לINT1 המיועדים, INT2 ו INT3 גנומית היעד ויוצרת הפסקות תקועות כפולות. בתערובת הטרנספורמציה, דנ א של תורם המורכב אזורים מאגפים וקלטת הביטוי הגן קרוטנאיד נכללו. הרכבות dna תורם היו ממוקדות למתוח אחד של dna ב-dna גנומית סביב INT1 (crte), INT2 (crtyb) ו INT3 (crte) המקום באמצעות vivo רקומבינציה בשל הנוכחות של 50 bp-הומוולוגי מחברים רצפים, המצוין כ 5, A, B, C, D או E. P1 – P3, יזמים שונים; . T1-T3, מחסלים שונים דמות זו השתנתה מתוך Verwaal ואח '. 201819. מבנים גנטיים המוצגים באמצעות השפה הפתוחה בביולוגיה סינתטית (SBOL) סמלים חזותיים40. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
איור 3 : ה-PCR מאמת את ניסויי עריכת הגנום. (א) אימות שיבוט של שער הזהב תגובות של קלטות DNA התאספו של התורמים. התוצאות המתקבלות מתקבלות בהסכם באורכים צפויים. (ב) PCR של מערך ה-crrna היחיד. (ג) לינריזציה של פלסמיד pRN1120. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4 : צלחות של S. cerevisiae ס העתקות באמצעות הגישה הגנום מולטיפלקס. (א) זן 1 המבטא crte, Crtyb ו crte מתוך שלושה יזמים חזקים (כתום כהה מושבות). (ב) להתאמץ 2 ביטוי crte, Crtyb ו crte מתוך שלושה היזמים חוזק בינוני (כתום מושבות). (ג) להתאמץ 3 הבעת crte, Crtyb ו crte מתוך שלושה היזמים כוח נמוך (מושבות צהובות). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 5 : ה-PCR מאמת שילוב של קלטות הדנ א של התורם בתוך הבית המיועד של ה-dna גנומית. (א) אימות של שלוש מושבות של מאמץ 1. (ב) אימות של שלוש מושבות של מאמץ 2. (ג) אימות של שלוש מושבות של הנבג 3. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 6 : פיקסל שמרים אמנות של רוזלינד פרנקלין. (א) צילום RGB שחור-לבן של 220 × 280 פיקסלים של רוזלינד פרנקלין ששימש כתבנית. (ב) המרת מחשב של התמונה שחור לבן של רוזלינד פרנקלין לתוך 4-צבע 64 × 96 פיקסל רשימה. (ג) תמונה של פיקסל שמרים אמנות עם 64 × 96 מושבות שמרים עם מקטע מוגדלת. (ד) תמונה של מטפל נוזלי אקוסטי עם שני צלחות מבוגרות מלא. (ה) תמונה של מיקרוצלחת בוגרת מלאה עם 64 × 96 מושבות שמרים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| מסננים 1 | מסננים 2 | מסננים 3 | |
| מושבות צבעוניות | 16 | 279 | 220 |
| מושבות לבנות | 16 | 18 | 18 |
| סה כ מושבות | 32 | 297 | 238 |
| יעילות | 50% | 94% | 92% |
טבלה 1: עריכת יעילות של גישה לתוך הגנום מולטיפלקס.
| מערך crRNA רצףa, b, c, d, e, f |
|
בדיקת מלאי בדיקת מלאי בעלי ה, הגנה מפני בעלי-ה, בדיקת מגנציה (מחלקה שוגית) מיכאל לטנוב GATAATGTATGATTATGCTTTCACTCATATTTATACAGAAACTTGATGTTTTCTTTCGAGTATATACAAGG TGATTACATGTACGTTTGAAGTACAACTCTAGATTTTGTAGTGCCCTCTTGGGCTAGCGGTAAAGGTGCGCA כיצד לעשות זאת לאחר מכן, הינך מאוד מאוד מאוד מאוד מאוד מאוד מאוד מאוד מצער ATGTTTCGGCGTTCGAAACTTCTCCGCAGTGAAAGATAAATGATCהכהן מלון לגונה שלמהבעלי הגנה עלמיזוג אוויר הTGCCCCTCTTATACGATTATATT TT מדריך כיצדלעשות מGGGGGGCCCGGTACCCAGCTTTTGTTCCCTTTAGTGAGG הגנה GTTAATTCCGAGCTTGGCGTAATCATGGTCATAGCTGTTTCCTGTGTG |
| א. הומולוגיה לpRN1120 (מודגש). b. SNR52 יזם (נטוי). ג. רצפי יעד גנומית (מסומנים בקו תחתון). ד. המדריך חוזר באופן ישיר לLbCas12a (נטוי, מודגש). e. SUP4 שליחות קטלנית (נטוי). ו. הומולוגיה לpRN1120 (מודגש). |
טבלה משלימה 1: מערך crRNA יחיד עבור LbCas12a המכיל הומולוגיה עם פלמיד pRN1120.
| שם | רצףa | תיאורb | משמש לנקודה |
| KC-101 | כמוסות מגנט | FW פריימר עבור הגברה של מערך crRNA יחיד | 2.1.4 |
| KC-102 | מיכל הכהן | קרוואן פריימר עבור הגברה של מערך crRNA יחיד | 2.1.4 |
| KC-103 | AAGCGACTTCCAATCGCTTTGC | כרטיס FW עבור הגברה של דנ א תורם עם מחבר 5 | 3.6.1 |
| KC-104 | ליאת כהן | קרוואן פריימר עבור הגברה של ה-DNA תורם עם מחבר A | 3.6.1 |
| KC-105 | מיכל ברקת | כרטיס FW עבור הגברה של דנ א תורם עם מחבר B | 3.6.1 |
| KC-106 | מוזיאון האוויר הגותי | קרוואן פריימר עבור הגברה של ה-DNA התורם עם מחבר C | 3.6.1 |
| KC-107 | ליאת מאור | FW פריימר עבור הגברה של ה-DNA תורם עם מחבר D | 3.6.1 |
| KC-108 | מיכל שלמה | קרוואן פריימר עבור הגברה של דנ א תורם עם מחבר E | 3.6.1 |
| KC-109 | מיכל ברקת | FW פריימר עבור הגברה של INT1 5 עם מחבר 5 | 4.4 |
| KC-110 |
מיכל הלוי TGGATATGCAAAGCGATTGGAA GTCGCTTגלקטקצ הובלה מרבית | קרוואן פריימר עבור הגברה של INT1 5 עם מחבר 5 | 4.4 |
| KC-111 |
לינה והגנה מיכל שטטין TGCTTTAAGCGTTGAAGTTTCCTC מיכל הגנה | FW פריימר עבור הגברה של INT1 3 ' עם מחבר A | 4.4 |
| KC-112 | מיכל ברקת | קרוואן פריימר עבור הגברה של INT1 3 ' עם מחבר A | 4.4 |
| KC-113 | AGAAGATTTCTCTTCAATCTC | FW פריימר עבור הגברה של INT2 5 עם מחבר B | 4.4 |
| KC-114 |
כמוסות מגנט מוזיאון הבית מיכל האור הגנה מפני משחק | קרוואן פריימר עבור הגברה של INT2 5 ' עם מחבר B | 4.4 |
| KC-115 |
ACGCTTTCCGGCATCTTCCA מיכל הפקות CCTGTTGGGCGATTACACAAGCG GTGG | FW פריימר עבור הגברה של INT2 3 ' עם מחבר C | 4.4 |
| KC-116 | מיכל ברקת | קרוואן פריימר עבור הגברה של INT2 3 ' עם מחבר C | 4.4 |
| KC-117 | GGTCGTTTTTGTGCAGCATATTG | FW פריימר עבור הגברה של INT3 5 עם מחבר D | 4.4 |
| KC-118 |
GCGGAATATTGGCGGAACGG ACACACGTGGATACAAACCTG מוטי שגיא מיכל כהן | קרוואן פריימר עבור הגברה של INT3 5 ' עם מחבר D | 4.4 |
| KC-119 |
ליאת שגיא CCCATATGCTCGGTCGTGCTTGTT בדיקתמשחק GTAC | FW פריימר עבור הגברה של INT3 3 ' עם מחבר E | 4.4 |
| KC-120 | מיכל הפקות | קרוואן פריימר עבור הגברה של INT3 3 ' עם מחבר E | 4.4 |
| KC-121 | משחק החלקה | FW לאימות האינטגרציה של con5-crtE-קונה כדי INT1 5 ' | 7.4.1 |
| KC-122 | כמוסות מגנט | FW לאימות האינטגרציה של con5-crtE-קונה כדי INT1 3 ' | 7.4.1 |
| KC-123 | מחסום הגנה | FW פריימר לאימות האינטגרציה של conB-crtYB-Conb ל INT2 5 ' | 7.4.1 |
| KC-124 | מרכז התיירות | FW פריימר לאימות האינטגרציה של conB-crtYB-Conb ל INT2 3 ' | 7.4.1 |
| KC-125 | בעלי מניה | הוראות FW לאימות האינטגרציה של מנצח-crtI-קונוס ל INT3 5 ' | 7.4.1 |
| KC-126 | GATCGGTCAATTAGGTGAAG | הוראות FW לאימות האינטגרציה של מנצח-crtI-קונוס ל INT3 3 ' | 7.4.1 |
| KC-127 | מרכז הגנה | קרוואן פריימר לאימות של אינטגרציה של con5-crtE-קונה כדי INT1 5 ' | 7.4.1 |
| KC-128 | מיכל שונוב | קרוואן פריימר לאימות של אינטגרציה של con5-crtE-קונה כדי INT1 3 ' | 7.4.1 |
| KC-129 | מוזיאון הגנה | קרוואן פריימר לאימות של אינטגרציה של conB-crtYB-Conb ל INT2 5 ' | 7.4.1 |
| KC-130 | מיכל הלוי | קרוואן פריימר לאימות של אינטגרציה של conB-crtYB-Conb ל INT2 3 ' | 7.4.1 |
| KC-131 | מדריך | קרוואן פריימר לאימות של אינטגרציה של מנצח crtI-קונוס ל INT3 5 ' | 7.4.1 |
| KC-132 | מיכל האור | קרוואן פריימר לאימות של אינטגרציה של מנצח crtI-קונוס ל INT3 3 ' | 7.4.1 |
| a. רצפים מודגשים מציינות רצפי מחברים. ב. התחל והפוך מיועדים כFW ו-RV, בהתאמה. |
שולחן משלים 2: רצפים פריימר.
שולחן משלים 3: עיצוב זנים שנבנו.

שולחן משלים 4: רצפי דנ א של התורמים ואזורי הביטוי. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
הפרוטוקול המסופק מתאר את עריכת הגנום מולטיפלקס של ס. cerevisiae ס באמצעות Cas12a מחיידק הND2006 בשילוב עם מערך crrna יחיד ו-DNA תורם. עיצוב של מערך crRNA יחיד ו-DNA התורם מוסבר בפרוטרוט. בניגוד היטב מערכת crispr/Cas9, crispr/Cas12a יש את היכולת הייחודית הנוספת של עיבוד crrnas מרובים ביטא מערך crrna אחד13,33. הודות לתכונה זו, ניתן לבצע עריכה בו של מספר מטרות מרובות וניתנת להשגה בטרנספורמציה אחת. זו גישה מערך crRNA יחיד הוכח בעבר על ידי זצ'ה ואח'. 34 אשר בו העריכה עד ארבעה גנים בתאי היונקים באמצעות AsCas12a, ועל ידי swiat ואח'. 35 מי הציג ארבעה שברי DNA לתוך הגנום שמרים באמצעות FnCas12a. לידיעתך, מספר גבוה יותר של שינויים גנוביים בו באמצעות מערכת Cas12a לא דווחו ואת המגבלה המקסימלית של מטרות לכל מערך בודד עבור Cas12a עדיין נקבע. מחקר נוסף ניצול מערכים crrna יחיד בשילוב עם Cas12a כולל בקרת מולטיטרסקריפט במגוון רחב של אורגניזמים33,36,37.
קיימים מספר שלבים קריטיים בפרוטוקול המוצג. עיצוב בקפידה את כל רצפי ה-DNA כי הם מעורבים בניסוי Cas12a הגנום עריכת, במיוחד במקרה כאשר רצפי DNA הרומן מוצגים. לקבוע את הפונקציונליות של רצפים הרווח החדש חלק של crRNA, למשל על ידי ניסוי הגנום בלבד לערוך כמתואר על ידי Verwaal ואח'. 19 לפני שילוב אותם לתוך מערך crrna יחיד. בצע את ההמלצות להכנת פתרונות מאגר טרנספורמציה בשימוש בניסוי עריכת Cas12a כדי להשיג יעילות שינוי טוב של שמרים.
ישנם כמה שינויים אופציונליים של הטכניקה. מומלץ להשתמש 1 μg של כל ה-DNA של התורם, pRN1120 או יחיד crRNA מערך ביטוי הקלטת בטרנספורמציה, למרות השימוש בסכום ה-DNA נמוך צפוי גם לגרום ליעילות שינוי משביע רצון. בצע שינוי בדיקה כדי לקבוע אם ניתן להשתמש בכמויות DNA נמוכות. הטרנספורמציה של ה-S. cerevisiae ס עשויה להתבצע בשיטה שונה מזו המתוארת בפרוטוקול זה, למשל הפרוטוקול המתואר באמצעות gietz et al. (2007) 38. the מדריך RNA הנמען פלמיד pRN1120 מתאים לביטוי של crrna בודד ומערך crrna יחיד של משתניםCas12a שונים (למשל, מ-"מוחמץ" . BV3L6 או פרנסיז'לה novicida U112) כמו גם לביטוי של sgRNA בשילוב עם Cas919. ה-DNA התורם אינו צריך להיות מוגבל לתוך הביטוי גנים קרוטנאיד בתוך האזורים האגפים היעד ה-DNA התורם המתואר INT1, INT2 ו INT3 אתרים ב-DNA גנומית. כל ה-DNA של עניין יכול להיות הציג, באופן מדומה, ל-DNA גנומית של המארח על ידי עקרונות העיצוב המתוארים בפרוטוקול זה, או לחילופין DNA תורם יכול לשמש כדי למחוק DNA מן הגנום המארח. המבנה המודולרי של מערך crRNA יחיד מקל על התאמה קלה של מרווח ורצפים חוזרים ישירים. שינוי רצפי מרווח מאפשר שינוי של לוקוס שילוב מיועד אשר יכול להיות מעוצב על ידי אחד הכלים לזיהוי של אתר יעד גנומית, לדוגמה תוכנת הנחיה 1.039. במקום להשתמש ברצפי אגפים גדולים המכילים רצפי מחברים, 50-bp של האזור האגף ניתן לכלול ברצפי ה-DNA של התורם על ידי שילוב רצפי האזורים האלה 50 bp האגפים בצבעי היסוד בשימוש ב-PCR. במקרה זה, בסך הכל רק שלושה במקום תשעה שברי דנ א התורמים נדרשים לניסוי מוצלח של הגנום בקולנוע.
לסיכום, פרוטוקול זה מספק הוראות צעד אחר צעד כדי לבצע עריכת הגנום מולטיפלקס ב- S. cerevisiae ס באמצעות Cas12a בשילוב עם גישה אחת מערך crrna. הפרוטוקול הוכח על ידי עריכת הגנום מולטיפלקס באמצעות 9 שברי ה-DNA תורם וקוד מערך crRNA יחיד עבור שלוש gRNAs. אנו מציגים תדרים גבוהים עריכה הכוללת בין 50% ו 94% עבור שלושת העיצובים הזנים שדווחו כאן. מסכם, התכונה הייחודית של Cas12a היא היכולת לעבד מערך crRNA יחיד לתוך crRNAs בודדים בתא, מה שהופך Cas12a כלי מצוין כדי לאפשר הגנום מולטיפלקס עריכת ולפתח מודולים רגולציה מיקוד מרובים קלטות ביטוי באחד הולכים. בסופו של דבר, שלושה זנים הושגו הפקת קרוטנואידים ברמה שונה וצבעים בגוונים בין צהוב וכתום. עם אלה זנים וזן פראי, הראנו כיצד מטפל נוזלי אקוסטי יכול לשמש ישר לעשות אמנות פיקסל שמרים – זה לכבוד רוזלינד פרנקלין שתרמו לגילוי של מבנה ה-DNA 65 לפני שנים על ידי צילום המפורסם שלה 5123 < /c1 >.
Disclosures
המחברים מצהירים שקיים ניגוד אינטרסים. המחברים הגישו IP הקשורים שיטות שהוצגו.
Acknowledgements
פרויקט זה קיבל מימון של אופק 2020 האיחוד האירופי תוכנית מחקר וחדשנות תחת הסכם מענק no. 686070 (DD-נטול) ו 764591 (SynCrop), ומתוך תוכנית המחקר בניית אבני החיים עם מספר הפרויקט 737.016.005 על ידי ארגון הולנד למחקר מדעי (NWO). T.E.G. היה נתמך על ידי החברה המלכותית (גרנט UF160357) ו BrisSynBio, BBSRC/EPSRC מחקר ביולוגיה סינתטי מרכז (הענק BB/L01386X/1). אנו מודים Zi Di וג ואן וייק עבור תרומתם שמרים האיתור ניסויים ליצירת אמנות פיקסל שמרים.
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals specific for the protocol | |||
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | Electrophoresis |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | Selection of E. coli transformants |
| BsaI-HF (20 U/µl) | New England BioLabs | R353L | Golden Gate Cloning |
| Cell Lysis Solution (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| CutSmart Buffer | New England BioLabs | B7204S | Linearization of pRN1120 |
| Deoxyribonucleic acid sodium salt from salmon testes | Sigma Aldrich | D1626 | Transfromation of S. cerevisiae (carrier DNA) |
| dNTPs | Invitrogen | 10297018 | PCRs |
| EcoRI-HF | New England BioLabs | R3101S | Linearization of pRN1120 |
| Ethanol absolute for analysis | Merck | 100983 | Isolation of genomic DNA from S. cerevisiae |
| Ethylenediamine-tetraacetic acid | Sigma Aldrich | ED | Transformation of S. cerevisiae |
| G418 disulfate salt | Sigma Aldrich | A1720 | Selection of S. cerevisiae transformants |
| Histodenz | Sigma Aldrich | D2158 | Yeast pixel art |
| Isopropanol | Merck | 100993 | Isolation of genomic DNA from S. cerevisiae |
| Lithium acetate dihydrate | Sigma Aldrich | L6883 | Transformation of S. cerevisiae |
| Nancy-520 DNA Gel Stain | Sigma Aldrich | 1494 | Electrophoresis |
| NEB10 competent E. coli cells | New England BioLabs | C3019H | Transformation of E. coli: dx.doi.org/10.17504/protocols.io.nkvdcw6 |
| Nourseothricin | Jena Bioscience | AB102 | Selection of S. cerevisiae transformants |
| Phusion buffer | New England BioLabs | M0530L | PCRs |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530L | PCRs |
| Polyethylene glycol 4000 | Merck | 7490 | Transformation of S. cerevisiae |
| Protein Precipitation Solution (10 M NH4AC) (from kit Puregene Yeast/Bact. Kit B) | QIAGEN | 854016 | Isolation of genomic DNA from S. cerevisiae |
| Purple loading dye | New England BioLabs | B7024S | Electrophoresis |
| QIAprep Spin Miniprep Kit | QIAGEN | 27106 | Purification of plasmids |
| RNase coctail enzyme mix | Thermo Fisher Scientific | AM2286 | Isolation of genomic DNA from S. cerevisiae |
| T4 DNA ligase buffer | Invitrogen | 46300-018 | Golden Gate Cloning |
| T4 DNA Ligase (1 U/µl) | Invitrogen | 1705218 | Golden Gate Cloning |
| UltraPure Agarose | Invitrogen | 16500500 | Electrophoresis |
| Wizard SV Gel and PCR Clean-Up System Kit | Promega | A9282 | Purification of PCR products and linearized pRN1120 |
| Xhol | New England BioLabs | R0146S | Linearization of pRN1120 |
| Zymolyase 50 mg/ml (5 units/µL) | Zymo Research | E1006 | Isolation of genomic DNA from S. cerevisiae (yeast lysis enzyme) |
| Zymolyase storage buffer | Zymo Research | E1004-B | Isolation of genomic DNA from S. cerevisiae (necessary for the preparation of yeast lysis enzyme) |
| Chemicals of general use | |||
| 2*Peptone-Yeast extract (PY) agar | Plate growth of E. coli | ||
| 2*PY medium | Cultivation of E. coli | ||
| Demineralized water | Transformation of S. cerevisiae | ||
| ELFO buffer | Electrophoresis | ||
| MQ | Multiple steps | ||
| Physiological salt solution | Transformation of S. cerevisiae | ||
| TE buffer | Storage of DNA, transformation of S. cerevisiae | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | Cultivation of S. cerevisiae | ||
| YEPD (2% glucose) agar | Plate growth of S. cerevisiae | ||
| Consumables | |||
| Eppendorf tubes | |||
| Falcon tubes (50 mL) | |||
| Microplate 96 wells | |||
| Petri dishes | |||
| Pipette tips 0.5 - 10 µL | |||
| Pipette tips 10 - 200 µL | |||
| Pipette tips 100 - 1000 µL | |||
| Shake flasks (500 mL) | |||
| Sterile filters | |||
| Equipment | |||
| Centrifuge (Falcon tubes) | |||
| Echo 525 acoustic liquid handler | |||
| Incubator | |||
| NanoDrop | |||
| Set for eletrophoresis | |||
| Spectrophotometer | |||
| Table centrifuge (Eppendorfs tubes) | |||
| Thermocycler | |||
| Plasmids | |||
| pCSN067 | Addgene | ID 101748 | https://www.addgene.org/ |
| pRN1120 | Addgene | ID 101750 | https://www.addgene.org/ |
| Strains | |||
| S. cerevisiae strain CEN.PK113-7D | EUROSCARF collection | http://www.euroscarf.de |
References
- Knott, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361 (6405), 866-869 (2018).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Lian, J., HamediRad, M., Hu, S., Zhao, H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications. 8 (1), 1688 (2017).
- Li, Z. -. H., Liu, M., Lyu, X. -. M., Wang, F. -. Q., Wei, D. -. Z. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology. 58 (12), 1100-1104 (2018).
- Shao, Y., Lu, N., Qin, Z., Xue, X. CRISPR-Cas9 facilitated multiple-chromosome fusion in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (11), 2706-2708 (2018).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology. 13 (11), 722-736 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), (2016).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovič, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Lian, J., HamediRad, M., Zhao, H. Advancing metabolic engineering of Saccharomyces cerevisiae. using the CRISPR/Cas System. Biotechnology Journal. 13 (9), 1700601 (2018).
- Ferreira, R., et al. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. ACS Synthetic Biology. 7 (1), 10-15 (2018).
- Swarts, D. C., Martin, J. Cas9 versus Cas12a/Cpf1: Structure–function comparisons and implications for genome editing. Wiley Interdisciplinary Reviews: RNA. 9 (5), 1481 (2018).
- Strohkendl, I., Saifuddin, F. A., Rybarski, J. R., Finkelstein, I. J., Russell, R. Kinetic Basis for DNA Target Specificity of CRISPR-Cas12a. Molecular Cell. 71 (5), 816-824 (2018).
- Verwaal, R., et al. High-level production of beta-carotene in Saccharomyces cerevisiae. by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous. Applied and Environmental Microbiology. 73 (13), 4342-4350 (2007).
- Verwaal, R., Buiting-Wiessenhaan, N., Dalhuijsen, S., Roubos, J. A. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast. 35 (2), 201-211 (2018).
- Jakociunas, T., Jensen, M. K., Keasling, J. D. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering. 34, 44-59 (2016).
- Engler, C., Romy, K., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PloS One. 3 (11), 3647 (2008).
- Franklin, R. E., Gosling, R. G. Molecular configuration in sodium thymonucleate. Nature. 171, 740-741 (1953).
- Watson, J. D., Crick, F. H. A structure for deoxyribose nucleic acid. Nature. 171, 737-738 (1953).
- Wilkins, M. H. F., Stokes, A. R., Wilson, H. R. Molecular structure of deoxypentose nucleic acids. Nature. 171, 738-740 (1953).
- Young, E. M., et al. Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast. Metabolic Engineering. 48, 33-43 (2018).
- Roubos, J. A., Pel, H. J., Meijrink, B. . Cloning Method. , (2013).
- Mandel, M., Higa, A. Calcium-dependent bacteriophage DNA infection. Journal of Molecular Biology. 53 (1), 159-162 (1970).
- Van Dijken, J. P., et al. An interlaboratory comparison of physiological and genetic properties of four Saccharomyces cerevisiae strains. Enzyme and Microbial Technology. 26 (9-10), 706-714 (2000).
- Gietz, R. D., Schiestl, R. H., Willems, A. R., Woods, R. A. Studies on the transformation of intact yeast cells by the LiAc/SS-DNA/PEG procedure. Yeast. 11 (4), 355-360 (1995).
- Hill, J., Donald, K. A., Griffiths, D. E., Donald, G. DMSO-enhanced whole cell yeast transformation. Acids Research. 19 (20), 5791 (1991).
- Looke, M., Kristjuhan, K., Kristjuhan, A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques. 50 (5), 325-328 (2011).
- Tak, Y. E., et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nature Methods. 14 (12), 1163-1166 (2017).
- Zetsche, B., et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature biotechnology. 35 (1), 31-34 (2017).
- Swiat, M. A., et al. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research. 45 (21), 12585-12598 (2017).
- Li, L., et al. CRISPR-Cpf1-Assisted Multiplex Genome Editing and Transcriptional Repression in Streptomyces. Applied Environmental Microbiology. 84 (18), 00827-00918 (2018).
- Zhang, X., et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery. 3, 17018 (2017).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 1-4 (2007).
- Perez, A. R., et al. GuideScan software for improved single and paired CRISPR guide RNA design. Nature Biotechnology. 35 (4), 347-349 (2017).
- Cox, R. S., et al. Synthetic Biology Open Language Visual (SBOL Visual) Version 2.0. Journal of Integrative Bioinformatics. 15 (1), 1613-4516 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved