需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
果蝇、果蝇细胞免疫应答的评估
摘要
该方案描述了一种在成虫体内吞噬试验的方法, 以量化吞噬细胞的识别和微生物感染的清除。
摘要
在所有动物中, 先天免疫提供了对广泛的病原体的即时和强大的防御。体液和细胞免疫反应是先天免疫的主要分支, 许多调节这些反应的因素在无脊椎动物和哺乳动物之间在进化上是保守的。吞噬作用是细胞先天免疫的核心成分, 由免疫系统的专门血细胞进行。果蝇果蝇已成为研究整个动物吞噬的分子机制和生理影响的有力遗传模型。在这里, 我们演示了一种基于注射的体内吞噬试验, 以量化亲精细胞、血细胞对颗粒的吸收和破坏。该程序允许研究人员精确控制颗粒浓度和剂量, 从而能够在短时间内获得高度可重复的结果。该实验是定量的, 易于执行, 并可用于筛选影响病原体识别、吸收和清除的宿主因子。
引言
天生的免疫防御是抵御病原微生物的第一道防线。这些反应可以在功能上分为体液和细胞先天免疫, 这两个反应都是由细菌线编码模式识别受体 (Prr) 介导的, 感知病原体相关的分子模式 (Pampp)1。许多先天免疫的信号通路和效应机制在哺乳动物和无脊椎动物中得到了保护, 如线虫、线虫和果蝇、果蝇2。果蝇已成为研究宿主防御传染性微生物的有力系统 3.五味子在基因上是可追踪的, 在实验室里容易和廉价饲养, 并且有很短的世代时间。此外, 果蝇对一系列微生物表现出高效的防御能力, 从而能够检查宿主对病毒、细菌、真菌或寄生虫病原体的免疫力。
果蝇免疫学家历来利用前瞻性基因筛选、基因组范围 rna 介导的昆虫细胞系干扰 (rnai) 筛选以及先前存在的突变苍蝇株来检查先天免疫--从而导致识别和表征几种进化保守的体液免疫途径4,5,6,7,8。可以说, 体液先天免疫反应是果蝇免疫防御的最佳特征。感染后, 体液反应导致产生和系统释放的抗菌肽 (AMP) 分子进入血淋巴, 在昆虫的血液当量。Amp 是由高度保守的收费和 Imd 信号通路产生的。Toll 通路与哺乳动物 TLRXIL-1R 受体信号转导同源, Imd 通路与哺乳动物肿瘤坏死因子α信号转相同源。在食人鱼中,恐惧症是由革兰氏杆菌诱导的, 霉菌、真菌和食子蛋白 x viru6、9、10和 imd 信号是由革兰氏菌诱导的. ,12岁。
细胞免疫, 由侵入性病原体的包封、黑色素化和吞噬组成, 由称为血细胞13的专门血细胞进行。果蝇血细胞有三类: 晶体细胞、层状细胞和浆细胞13。晶体细胞占幼虫循环血细胞的 5%, 它释放异丙氧酶 (proPO) 酶, 导致病原体和宿主组织在伤口部位的黑色素化。在健康的胚胎或幼虫中通常找不到的细胞是包封异物的粘附细胞。这些细胞是在产后或寄生黄蜂卵沉积在幼虫体内时诱导的。吞噬性浆细胞占幼虫循环血流细胞的 95%, 在成人所有剩余血细胞中发挥着作用, 在发育过程中对组织重塑起着重要作用, 值得注意的是, 吞噬细胞是嗜铬菌细胞免疫的主要效应细胞。
吞噬作用是先天免疫防御的一条关键线;破坏宿主上皮屏障的微生物很快被吞噬血细胞吞没和消灭 (关于吞噬细胞生物学的综合回顾见参考14)。当血细胞上的种细胞编码模式识别受体 (Prr) 识别微生物的病原体相关分子模式 (Pampp) 时, 就开始了这一过程。一旦绑定到他们的目标, Prr 启动信号级联, 导致通过肌动蛋白细胞骨架重塑形成假足类。伪足类动物围绕着微生物, 微生物随后被吞没并内化为一个新生的细胞器--噬虫体。当噬虫体被贩运到血细胞内部并通过与溶酶体的一系列相互作用而酸化时, 当噬虫体经历噬虫体成熟的过程时, 微生物就会被破坏。在哺乳动物原代细胞的体外和细胞生物学研究中, 已经有助于识别和表征调节吞噬的因素, 如哺乳动物 fc-γ受体和 c3b 受体15,16。然而, 在哺乳动物系统中, 执行大规模屏幕或体内研究的能力有限。
在这里, 我们提出了一个体内检测在成年果蝇的吞噬, 这是基于一个程序首次推出的大卫·施奈德实验室在 2000年17。施耐德实验室显示, 沿着腹背血管聚集的无柄血细胞很容易产生吞噬聚苯乙烯珠和细菌。为了想象吞噬, 苍蝇被注入荧光标记的颗粒 (如贴有荧光素异硫氰酸酯 (e. colie-fitc) 标签的大肠杆菌), 孵育 30分钟, 让血细胞的时间吞没这些颗粒, 然后再注射色氨酸蓝, 在潜伏期内抑制不吞噬的颗粒的荧光。然后使用倒置荧光显微镜对飞行背侧血管进行成像。这篇开创性的论文, 使用一个相对简单的实验, 证明血细胞吞噬细菌和乳胶珠, 细菌吞噬可以通过预注射苍蝇与乳胶珠, 以及苍蝇没有细胞和体液免疫反应是易受感染的, 甚至大肠杆菌。本报告中介绍的检测方法以施耐德实验室的工作为基础, 通过测量与背血管相关的血细胞所吞没的颗粒的荧光强度来量化体内吞噬作用。
与哺乳动物系统中采用的方法类似,果蝇遗传学家最初使用全基因组的体外 rnai 屏幕来识别细胞免疫反应所需的基因18, 19,20 ,21,22,23。然而, 成人体内吞噬试验的发展使后续实验能够在整个动物中轻松进行, 从而使研究人员能够验证在体外研究中确定的因素的生物学作用。跨膜受体食客的情况就是如此, 它最初被确定为使用 S2 细胞24的 RNAi 筛查中的细菌受体, 后来被证明是介导大肠杆菌(大肠杆菌)、肠球菌粪便和金黄色葡萄球菌(金黄色葡萄球菌) 在成人中的吞噬作用25。
我们的实验室在前瞻性基因屏幕和全基因组关联研究中使用了体内吞噬检测方法 (使用果蝇遗传参考小组 (dgrp)) 来识别调节成人血细胞吞噬功能的新基因。这些研究导致了受体 PGRP-SC1A 和 PGRP-SA26、细胞内囊泡贩运蛋白 rab1427、谷氨酸转运体 polyphemus28和 rna 结合蛋白 fisu-129的表征。
我们预计, 未来的屏幕纳入体内吞噬可能会导致识别额外的基因, 这对果蝇的细胞免疫反应很重要。使用完全测序自交系的屏幕, 如 DGRP 或食子体合成种群资源 (dspr), 可以识别影响吞噬或血细胞发育的自然变异。此外, 这种技术可用于其他果蝇物种, 也可用于筛选新的社区资源, 例如收集国家果蝇物种储存中心 (ndsssc) 维持的 250个果蝇物种) 在康奈尔大学。这些实验可以使用荧光标记的细菌或真菌壁生物制品进行, 这些生物物品可在商业上买到, 也可以使用任意数量的细菌或真菌物种进行--前提是微生物表达荧光标记.
研究方案
1. 准备荧光素颗粒进行注射
- 通过添加990μl 无菌 1x PBS 和 10μl 50 mm 氮化钠, 将商用中可获得的热杀灭细菌颗粒重新添加到 10 Mg/ml 的库存浓度 (见材料表)。漩涡混合。
- 在 0.2 mL 管中分为一次性使用的 8μl aliquots, 并在4°c 的黑暗盒中存储, 以最大限度地降低与光相关的灵敏度。
注: 氮化钠防腐剂是可选的, 如果用1毫升无菌 1x PBS 制成 10 mgml 库存, 该防腐剂的报价, 并储存在-20°c。
- 在 0.2 mL 管中分为一次性使用的 8μl aliquots, 并在4°c 的黑暗盒中存储, 以最大限度地降低与光相关的灵敏度。
- 通过混合500Μl 注射器过滤绿色食品着色和 9.5 mL 无菌 1x PBS, 在 1x pbs 中制造5% 食品着色的10毫升溶液。
- 注射前清洗颗粒, 去除氮化钠。将42μl 无菌 1x PBS 和8μl 的 10Mg/ml 混合在 1.7 mL 管中。在室温下以最大速度离心2.5 分钟。
- 取出上清液, 加入 50Μl 1x PBS, 并以最高速度离心, 在室温下为2.5分钟。
- 重复步骤1.3 和 1.3.1 2x, 共洗3次。
- 最后清洗后, 在 1x PBS 中, 将上清液和悬浮颗粒重新悬浮到 1.6 mgml, 在 50μl 5% 的食品着色中。
- 用铝箔包裹管子, 以防止光线照射。存放在 4°c, 1周后丢弃。
2. 准备注射站和苍蝇
- 准备注射垫。要同时注入多达4个苍蝇基因型, 请使用实验室胶带将矩形 CO2 飞气垫分成4个部分.在显微镜附近的长凳上, 指定区域放置小瓶, 一旦苍蝇在垫子上排成一行 (垫的每个角落一个)。
- 准备与年龄相仿的小瓶, 4-7, 苍蝇注射。对于每个要测试的菌株, 将5名男性和5名女性转移到一个新鲜的, 标记小瓶准备的苍蝇食物, 并保持在25°c。
- 准备气动喷射器 (见材料表), 方法是将仪器设置为100毫秒 (将气体压力短暂爆发以排出液体, 从而允许提供亚纳米管体积)定时模式。
- 准备显微镜幻灯片。切割1.5 英寸的胶带, 折叠成一个循环, 将粘合剂面伸出来, 然后放在贴有标签的显微镜幻灯片上。
3. 准备玻璃毛细管
- 使用拉针器拉玻璃针 (薄壁玻璃毛细血管)。
- 用千分尺将针头固定在显微镜下, 并使用 #5 细点不锈钢推拿器将针头弄断。100μm 的尖端足以刺穿苍蝇的角质层, 同时最大限度地减少伤害。
- 测量将注入到每只苍蝇中的液体体积。用无菌5% 的食品着色在 1x PBS 中装入针头, 并将液体排出 0.01 mm 级千分尺上的一滴矿物油上。
注: 如果液滴是球形的, 则以皮花性计计算出的体积为 3/1910(尺寸)。30直径为100μm 的针头将在100毫秒内弹出 ~ 2 nl。
4. 注射苍蝇
- 将 1.6 mg/mL 颗粒的移液器10μl 放在一个小正方形的副体上。
- 将液体放入针头, 然后安装在喷油器喷嘴中 (参见材料表)。
- 用二氧化碳对苍蝇进行麻醉, 并将它们排在平板板上的指定区域, 腹侧向上, 头部朝向垫的前面。将小瓶放在长凳上的相应区域。
- 在腹部的上角注入 5, 100 毫秒的液体泵 (总约 10 nL)。
- 将注射的苍蝇转移到适当的小瓶, 注意小瓶上的时间。保持在25°c。
- 加载一个新的针与 0.4% Trypan 蓝色解决方案。
- 将气动喷射器设置为关控系统, 它允许恒定的气流将液体从针头中挤出。
- 在飞行休息30分钟后, 对苍蝇进行麻醉, 并注入 Trypan Blue, 直到腹部丰满和扩张。
注: 当检测带有 ph 敏感染料标记的颗粒的噬虫体成熟时, 允许苍蝇休息 1小时, 在安装苍蝇之前不要注入色氨酸蓝色。 - 在显微镜下的滑梯上安装有胶带, 腹侧向下。将翅膀推到苍蝇的一侧, 并将其固定在磁带上。此外, 轻轻地将头部推入磁带, 以确保苍蝇不会移动。
- 立即移动到步骤5。
5. 成像苍蝇
- 图像飞行, 一次一个, 在 25 x 或32x 放大, 使用倒置荧光显微镜连接到数码相机和计算机 (见材料表)。利用数码相机的计算机软件聚焦苍蝇的背侧容器。
- 记录实验之间的曝光时间和放大倍率。
注: 在一次会议上拍摄多个菌株时, 失去基因型的跟踪是一个潜在的错误来源。为避免给苍蝇贴标签错误, 请记下每个基因型的第一张和最后一张苍蝇照片的图像编号。
6. 对荧光进行定量和归一化
- 打开软件, 一次打开一个图像。
- 测量背侧血管的荧光强度。在背侧血管周围画一个多边形。选择"测量"并记录多边形内的荧光强度。
- 确定背景荧光强度。复制第一个多边形, 并将其移动到与苍蝇背侧容器相邻的区域。选择"测量"并记录背景区域的荧光强度。
- 通过背景荧光使背侧血管荧光正常化:
背侧容器. 背景。 - 计算毒株中所有苍蝇的平均归一化背侧血管荧光强度。
- 再重复实验2次。
- 使用学生的未配对t检验来比较三个实验中控制苍蝇和试验苍蝇的平均相对荧光强度。使用公式计算效果大小: Cohen ' s d = (m1-m2)-Sd 集合, 其中M 1是基因型1的平均值, m2 是基因型2的平均值 &
Sd集合=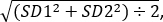
Sd1是基因型1的标准偏差, sd2是基因型2的标准偏差。
结果
图 1a 显示了使用荧光素标记颗粒进行体内吞噬试验的示意图。苍蝇安装在腹侧的胶带上, 腹部的前两个部分, 背血管所在的地方, 是清晰可见的 (图 1b)。实验误差的主要来源出现在过程的注射和成像步骤 (图 1c)。使用相同的针头注入多只苍蝇可能会导致它被苍蝇组织或颗粒堵塞 (图 1C?...
讨论
商业上可用的荧光标记颗粒用于评估一般的吞噬性 (0.2 微米羧改性后期微球) 或微生物的吞噬作用 (荧光标记的热或化学杀生细菌或酵母)。为了评估噬体体组的成熟, 研究人员可以选择带有 ph 敏感染料标记的颗粒, 当 ph 值从中性变低到酸性时, 当噬亚耳溶于酸性时, 这种染料会发光。或者, 为了检查吞噬、病原体识别和吸收的初始步骤, 研究人员应该选择带有荧光标记的粒子, 这些标记在细...
披露声明
作者没有什么可透露的。
致谢
作者感谢贝丝·冈萨雷斯博士和阿普拉伊塔·加格博士在进行体内吞噬实验方面提供的支持。NSF UMD 发展种子赠款和 UMD NIH T32 培训赠款、细胞和分子生物学 (CMB) 和宿主病原体相互作用 (HPI) 为这项工作提供了资金。
材料
| Name | Company | Catalog Number | Comments |
| 0.2μm Red Fluorescent Carboxylate Modified FluoSpheres | Invitrogen | F8810 | Fluorescently-labeled latex beads to test general phagocytic capacity of phagocytes. (~580/~605 nm) Inject a 1:20 dilution in PBS with 5% dye. |
| 5430-10 PicoNozzle Kit | World Precision Instruments | 5430-10 | Holder for 1.0mm pipette |
| E. coli (K-12 Strain) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | E13231 | Killed E. coli labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~495/~519 nm) |
| E. coli (K-12 Strain) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | E23370 | Killed E. coli labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~590/~617 nm) |
| E. coli (K-12 Strain) BioParticles, Fluorescein conjugate | Invitrogen | E2861 | Killed E. coli labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~494/~518 nm) |
| E. coli (K-12 Strain) BioParticles, Texas Red conjugate | Invitrogen | E2863 | Killed E. coli labeled with Texas Red. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~595/~615 nm) |
| E. coli (K-12 Strain) BioParticles, Texas Red conjugate | Invitrogen | E2863 | Killed E. coli labeled with Texas Red. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~595/~615 nm) |
| Needle Pipette Puller | David Kopf Instruments | Model 725 | |
| pHrodo Red E. coli BioParticles Conjugate for Phagocytosis | Invitrogen | P35361 | Killed E. coli labeled with pHrodo Red. Use to test phagocyte reconition, uptake, and phagosome maturation of gram-negative bacteria. (~560/~585 nm). No need to quench with Trypan Blue. |
| pHrodo Red S. aureus BioParticles Conjugate for Phagocytosis | Invitrogen | A10010 | Killed S. aureus labeled with pHrodo Red. Use to test phagocyte reconition, uptake, and phagosome maturation of gram-positve bacteria. (~560/~585 nm). No need to quench with Trypan Blue. |
| Pneumatic PicoPump PV820 | World Precision Instruments | SYS-PV820 | The World Precision Instruments Pneumatic PicoPump PV820 uses differential pressures to hold liquid in the glass needle between injections. The user manually controls short bursts of gas pressure to expel the liquid – allowing delivery of sub-nanoliter volumes. The amount of liquid delivered depends on two main variables – the size of the glass needle opening and the amount of time injection pressure is applied. set the instrument to 100 ms “TIMED” mode. |
| S. aureus (Wood Strain without protein A) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | S23371 | Killed S. aureus labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~495/~519 nm) |
| S. aureus (Wood Strain without protein A) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | S23372 | Killed S. aureus labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~590/~617 nm) |
| S. aureus (Wood Strain without protein A) BioParticles, Fluorescein conjugate | Invitrogen | E2851 | Killed S. aureus labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~494/~518 nm) |
| Thin Wall Glass Capillaries | World Precision Instruments | TW100F-3 | Needles for injection. OD = 1.0 mm |
| Trypan Blue Solution (0.4%) | Sigma | T8154 | Used to quench extracellular fluorescence of Fluorescein, Alexa Fluor, or Texas Red labeled particles. |
| ZEISS SteREO Microscope (Discovery.V8) | Zeiss | SteREO Discovery.V8 | Inverted fluorescence microscope for imaging flies. Use a digital camera (example: AxioCam HC camera) and the accompanying software (example: AxioVision 4.7 software) to take pictures. |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | Z23373 | Killed labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of yeast. (~495/~519 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | Z23374 | Killed labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of yeast. (~590/~617 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Fluorescein conjugate | Invitrogen | Z2841 | Killed labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of yeast. (~494/~518 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Texas Red | Invitrogen | Z2843 | Killed labeled with Texas Red. Use to test phagocyte recogntion and uptake of yeast. (~595/~615 nm) |
参考文献
- Akira, S., Uematsu, S., Takeuchi, O. Pathogen recognition and innate immunity. Cell. 124 (4), 783-801 (2006).
- Kim, D. Studying host-pathogen interactions and innate immunity in Caenorhabditis elegans. Disease Models & Mechanisms. 1 (4-5), 205-208 (2008).
- Lemaitre, B., Hoffmann, J. The host defense of Drosophila melanogaster. Annual Review Immunology. 25, 697-743 (2007).
- Wu, L. P., Choe, K. M., Lu, Y., Anderson, K. V. Drosophila Immunity: Genes on the Third Chromosome Required for the Response to Bacterial Infection. Genetics. 159 (1), 189-199 (2001).
- De Gregorio, E., Spellman, P. T., Tzou, P., Rubin, G. M., Lemaitre, B. The Toll and Imd pathways are the major regulators of the immune response in Drosophila. EMBO Journal. 21 (11), 2568-2579 (2002).
- Michel, T., Reichhart, J. M., Hoffmann, J. A., Royet, J. Drosophila Toll is activated by Gram-positive bacteria through a circulating peptidoglycan recognition protein. Nature. 414 (6865), 756-759 (2001).
- Choe, K. M., Werner, T., Stoven, S., Hultmark, D., Anderson, K. V. Requirement for a peptidoglycan recognition protein (PGRP) in Relish activation and antibacterial immune responses in Drosophila. Science. 296 (5566), 359-362 (2002).
- Wu, J., Randle, K. E., Wu, L. P. ird1 is a Vps15 homologue important for antibacterial immune responses in Drosophila. Cellular Microbiology. 9 (4), 1073-1085 (2007).
- Lemaitre, B., Nicolas, E., Michaut, L., Reichhart, J. M., Hoffmann, J. A. The dorsoventral regulatory gene cassette spatzle/Toll/cactus controls the potent antifungal response in Drosophila adults. Cell. 86 (6), 973-983 (1996).
- Zambon, R. A., Nandakumar, M., Vakharia, V. N., Wu, L. P. The Toll pathway is important for an antiviral response in Drosophila. Proceedings of the National Academy of Science of the United States of America. 102 (20), 7257-7262 (2005).
- Lemaitre, B., et al. A recessive mutation, immune deficiency (imd), defines two distinct control pathways in the Drosophila host defense. Proceedings of the National Academy of Science of the United States of America. 92 (21), 9465-9469 (1995).
- Leulier, F., Rodriguez, A., Khush, R. S., Abrams, J. M., Lemaitre, B. The Drosophila caspase Dredd is required to resist gram-negative bacterial infection. EMBO Reports. 1 (4), 353-358 (2000).
- Meister, M., Lagueux, M. Drosophilablood cells. Cellular Microbiology. 5 (9), 573-580 (2003).
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annual Review Pathology. 7, 61-98 (2012).
- Anderson, R. A., Sando, G. N. Cloning and expression of cDNA encoding human lysosomal acid lipase/cholesteryl ester hydrolase. Similarities to gastric and lingual lipases. Journal of Biological Chemistry. 266 (33), 22479-22484 (1991).
- Ross, G. D., Reed, W., Dalzell, J. G., Becker, S. E., Hogg, N. Macrophage cytoskeleton association with CR3 and CR4 regulates receptor mobility and phagocytosis of iC3b-opsonized erythrocytes. Journal of Leukocyte Biology. 51 (2), 109-117 (1992).
- Elrod-Erickson, M., Mishra, S., Schneider, D. S. Interactions between the cellular and humoral immune responses in Drosophila. Current Biology. 10, 781-784 (2000).
- Ramet, M., Pearson, A. M., Manfruelli, P., Li, X., Koziel, H., Gobel, V. Drosophila Scavenger Receptor CI Is a Pattern Recognition Receptor for Bacteria. Immunity. 15 (6), 1027-1038 (2001).
- Ramet, M., Manfruelli, P., Pearson, A. M., Mathey-Prevot, B., Ezekowitz, R. A. Functional genomic analysis of phagocytosis and identification of a Drosophila receptor for E. coli. Nature. 416 (6881), 644-648 (2002).
- Philips, J. A., Rubin, E. J., Perrimon, N. Drosophila RNAi screen reveals CD36 family member required for mycobacterial infection. Science. 309, 1251-1253 (2005).
- Agaisse, H., Burrack, L. S., Philips, J. A., Rubin, E. J., Perrimon, N., Higgins, D. E. Genome-wide RNAi screen for host factors required for intracellular bacterial infection. Science. 309 (5738), 1248-1251 (2005).
- Stuart, L. M., et al. Response to Staphylococcus aureus requires CD36-mediated phagocytosis triggered by the COOH-terminal cytoplasmic domain. Journal of Cell Biology. 170 (3), 477-485 (2005).
- Stroschein-Stevenson, S. L., Foley, E., O’Farrell, P. H., Johnson, A. D. Identification of Drosophila gene products required for phagocytosis of Candida albicans. PLoS Biology. 4 (1), e4 (2006).
- Kocks, C., et al. Eater, a transmembrane protein mediating phagocytosis of bacterial pathogens in Drosophila. Cell. 123 (2), 335-346 (2005).
- Nehme, N. T., et al. Relative roles of the cellular and humoral responses in the Drosophila host defense against three gram-positive bacterial infections. PLoS One. 6 (3), e14743 (2011).
- Garver, L. S., Wu, J., Wu, L. P. The peptidoglycan recognition protein PGRP-SC1a is essential for Toll signaling and phagocytosis of Staphylococcus aureus in Drosophila. Proceedings of the National Academy of Science of the United States of America. 103 (3), 660-665 (2006).
- Garg, A., Wu, L. P. Drosophila Rab14 mediates phagocytosis in the immune response to Staphylococcus aureus. Cellular Microbiology. 16 (2), 296-310 (2014).
- Gonzalez, E. A., Garg, A., Tang, J., Nazario-Toole, A. E., Wu, L. P. A glutamate-dependent redox system in blood cells is integral for phagocytosis in Drosophila melanogaster. Current Biology. 23 (22), 2319-2324 (2013).
- Nazario-Toole, A. E., Robalino, J., Okrah, K., Corrada-Bravo, H., Mount, S. M., Wu, L. P. The Splicing Factor RNA-Binding Fox Protein 1 Mediates the Cellular Immune Response in Drosophila melanogaster. Journal of Immunology (Baltimore, Md: 1950). 201 (4), 1154-1164 (2018).
- Guille, M. . Molecular Methods in Developmental Biology: Xenopus and Zebrafish. , (1999).
- Koundakjian, E. J., Cowan, D. M., Hardy, R. W., Becker, A. H. The Zuker collection: a resource for the analysis of autosomal gene function in Drosophila melanogaster. Genetics. 167 (1), 203-206 (2004).
- Horn, L., Leips, J., Starz-Gaiano, M. Phagocytic ability declines with age in adult Drosophila hemocytes. Aging Cell. 13 (4), 719-728 (2014).
- Brennan, C. A., Delaney, J. R., Schneider, D. S., Anderson, K. V. Psidin is required in Drosophila blood cells for both phagocytic degradation and immune activation of the fat body. Current Biology. 17 (1), 67-72 (2007).
- Akbar, M. A., Tracy, C., Kahr, W. H., Kramer, H. The full-of-bacteria gene is required for phagosome maturation during immune defense in Drosophila. Journal of Cell Biology. 192 (3), 383-390 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。