È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutare la risposta immunitaria cellulare del moscerino della frutta, Drosophila melanogaster, utilizzando un test di fagocitosi In Vivo
In questo articolo
Riepilogo
Questo protocollo descrive un test di fagocitosi in vivo in adulto Drosophila melanogaster quantificare fagocita riconoscimento e liquidazione delle infezioni microbiche.
Abstract
In tutti gli animali, l'immunità innata fornisce un immediato e robusta difesa contro un ampio spettro di agenti patogeni. Le risposte immunitarie umorali e cellulari sono i rami principali dell'immunità innata, e molti dei fattori che regolano queste risposte sono evolutivamente conservate tra gli invertebrati e mammiferi. Fagocitosi, il componente centrale di immunità innata cellulare, viene effettuata da cellule di sangue specializzate del sistema immunitario. Moscerino della frutta, Drosophila melanogaster, è emerso come un potente modello genetico per studiare i meccanismi molecolari e fisiologici degli impatti della fagocitosi in animali interi. Qui dimostriamo un test di fagocitosi in vivo basati su iniezione per quantificare l'assorbimento delle particelle e la distruzione di cellule del sangue di Drosophila , emociti. La procedura permette ai ricercatori di controllare con precisione la concentrazione di particelle e la dose, che permette di ottenere risultati altamente riproducibili in un breve lasso di tempo. L'esperimento è quantitativa, facile da eseguire e può essere applicato per schermo per fattori ospite che influenza patogeno riconoscimento, assorbimento e liquidazione.
Introduzione
Difese immunitarie innate formano la prima linea di difesa contro i microbi patogeni. Queste risposte possono essere diviso funzionalmente in immunità innata umorale e cellulare, entrambi i quali sono mediati da recettori di riconoscimento modello germinale-codificato (PRRs) che senso pathogen-associated molecular pattern (PAMP)1. Molte delle vie di segnalazione e meccanismi effettori dell'immunità innata sono conservati nei mammiferi e invertebrati, come il nematode, elegans di Caenorhabditis e la Mosca della frutta, Drosophila melanogaster2. Moscerino della frutta è emerso come un potente sistema di difesa dell'ospite contro microrganismi infettivi3di studiare. Drosophila è geneticamente trattabile, facilmente ed a buon mercato allevati nei laboratori e ha un tempo di generazione breve. Inoltre, la Mosca della frutta esibisce altamente efficienti difese contro una matrice di microbi, che consente l'esame dell'immunità ospite contro gli agenti patogeni virali, batterici, fungini o parassitari.
Drosophila immunologi hanno storicamente utilizzato avanti schermi genetici, genoma mediata da RNA interferenza (RNAi) proiezione di linee cellulari di insetto e pre-esistente ceppi mutanti volare per esaminare l'immunità innata – che conduce la identificazione e caratterizzazione di diverse vie immuni umorali evolutivamente conservati4,5,6,7,8. La risposta immunitaria innata umorale è, senza dubbio, il migliore caratterizzata difesa immunitaria nei moscerini della frutta. A seguito di infezione, la risposta umorale conduce alla produzione e rilascio sistemico di peptide antimicrobico molecole (AMP) nell'emolinfa, il sangue equivalente negli insetti. Amplificatori sono prodotte da pedaggio altamente conservata e Imd vie di segnalazione. La via di pedaggio è omologo al mammifero segnalazione del ricevitore TLR/IL-1R e pathway Imd è omologo alla segnalazione di fattore di necrosi tumorale-alfa dei mammiferi. In Drosophila, pedaggio segnalazione è indotto da batteri Gram-positivi e funghi Drosophila X virus6,9,10 e Imd segnalazione è indotta da batteri gram-negativi11 ,12.
Immunità cellulare, composto di incapsulamento, melanizzazione e fagocitosi dei patogeni invasivi, eseguiti da cellule specializzate del sangue chiamate emociti13. Ci sono tre classi di emociti nel moscerino della frutta: cristallo celle, lamellocytes e plasmatocytes13. Crystal cellule, che costituiscono il 5% degli emociti circolanti nelle larve, rilasciano enzimi proPhenoloxidase (proPO) che conduce la melanizzazione degli agenti patogeni e tessuti dell'ospite presso siti ferita. Lamellocytes, che non si trovano in embrioni sani o larve, sono cellule aderenti che incapsulano oggetti estranei. Queste cellule sono indotte su pupariation o quando parassitanti Vespa uova sono depositate nelle larve. Plasmatocytes fagocitaria, che compongono il 95% del circolante emociti nelle larve e tutti i rimanenti emociti negli adulti, gioca un ruolo nel rimodellamento durante lo sviluppo del tessuto e, in particolare, servire come cellula effettore principale della drosofila l'immunità cellulare.
Fagocitosi un'immediato e fondamentale linea di difesa immunitaria innata; i microbi che violano la barriera epiteliale di host sono rapidamente inghiottiti ed eliminati dalle cellule fagocitiche del sangue (per una rassegna completa di biologia delle cellule della fagocitosi vedere riferimento 14). Questo processo è iniziato quando il riconoscimento di pattern con codifica germline recettori (PRRs) su emociti riconoscono patogeni associati pattern molecolari (PAMPs) di microbi. Una volta legato ai loro target, PRRs avviare cascate di segnalazione che portano alla formazione di pseudopodi attraverso actina citoscheletrica rimodellamento. Pseudopodi circondano il microbo, che successivamente viene inghiottito e interiorizzato in un organello nascente phagosome. I microbi sono distrutti come phagosome subisce il processo di maturazione del fagosoma quando phagosome è trafficata verso l'interno degli emociti e acidifica attraverso una serie di interazioni con i lisosomi. In vitro e cella studi di biologia in cellule di mammifero primario sono stati strumentali nell'identificazione e caratterizzazione di fattori che regolano la fagocitosi, come il ricevitore di Fc-gamma dei mammiferi e C3b recettori15,16. Tuttavia, la possibilità di eseguire grandi schermi o studi in vivo sono limitati nei sistemi mammiferi.
Qui presentiamo un test in vivo per la fagocitosi in adulti mosche della frutta, che si basa su una procedura introdotta dal laboratorio di David Schneider nel 200017. Il laboratorio di Schneider ha mostrato che sessile emociti cluster lungo il vaso dorsale addominale prontamente fagocitare batteri e perle di polistirolo. Per visualizzare la fagocitosi, mosche sono iniettati con fluorescente contrassegnate particelle (come e. coli etichettati con isotiocianato di fluorescina (e. coli -FITC)), incubate per 30 minuti permettere emociti di fagocitare le particelle e poi iniettato con trypan blu, che disseta la fluorescenza delle particelle non phagocytosed durante il periodo di incubazione. Volare navi dorsale sono quindi Imaging utilizzando un microscopio invertito a fluorescenza. Questa carta seminale, utilizzando un esperimento relativamente semplice, ha dimostrato che emociti fagocitare batteri e granuli di lattice, che fagocitosi batterica possono essere inibite iniettando pre-Vola con granuli di lattice, e che vola senza sia cellulare ed umorale le risposte immunitarie sono suscettibili anche di e. coli. L'analisi presentata in questo rapporto si basa sul lavoro del laboratorio Schneider per quantificare in vivo fagocitosi misurando l'intensità della fluorescenza delle particelle inghiottito da emociti vaso dorsale associato.
Simile all'approccio adottato nei sistemi mammiferi, Drosophila genetisti inizialmente utilizzato genoma schermi RNAi in vitro per identificare i geni richiesti per la risposta immunitaria cellulare18,19,20 ,21,22,23. Tuttavia, lo sviluppo del dosaggio adulto fagocitosi in vivo attivato follow-up esperimenti per essere prontamente effettuati in animali interi, consentendo ai ricercatori di verificare il biologico il ruolo dei fattori individuati negli studi in vitro. Tale era il caso con il recettore transmembrana Eater, che è stato prima identificato come un recettore batterico in un schermo di RNAi utilizzando S2 cellule24 e poi più tardi mostrato a mediare Escherichia coli (Escherichia coli),, Enterococcus faecalis, e fagocitosi di Staphylococcus aureus (Staphylococcus aureus) in adulti25.
Il nostro laboratorio impiegato il test di fagocitosi in vivo in avanti schermi genetici e studi di associazione genome-wide (usando il pannello di riferimento genetico drosofila (DGRP)) per identificare nuovi geni che regolano la fagocitosi in adulti emociti. Questi studi hanno portato alla caratterizzazione i recettori PGRP-SC1A e PGRP-SA26, la vescicola intracellulare traffico proteina Rab1427, il trasportatore del glutammato Polifemo28e RNA-proteina Fox-129.
Anticipiamo che futuri schermi che incorporano la fagocitosi in vivo potrebbero portare all'identificazione di ulteriori geni che sono importanti per la risposta immunitaria cellulare in drosofila. Schermate mediante linee inbred completamente sequenziato, ad esempio la DGRP o la drosofila sintetico popolazione risorsa (DSPR), possono identificare varianti naturali che influenzano lo sviluppo di fagocitosi o emociti. Inoltre, la tecnica potrebbe essere adottata in altre specie di Drosophila o utilizzata per nuove risorse comunitarie di schermo, ad esempio la raccolta di 250 specie di Drosophila mantenuto dal National Drosophila specie Stock Center (NDSSC ) alla Cornell. Questi esperimenti possono essere effettuati utilizzando fluorescente etichettati batteriche o fungine-parete bioparticelle che sono commercialmente disponibili o possono essere eseguiti utilizzando qualsiasi numero di specie batteriche o fungine – fornito che il microbo esprime marcatori fluorescenti .
Protocollo
1. preparare le particelle della fluorescina per iniezione
- Ricostituire 10mg di commercialmente disponibili, calore-uccisi batteri particelle etichettate con fluoresceina (Vedi Tabella materiali) ad una concentrazione stock di 10 mg/mL aggiungendo 990 µ l sterile 1X PBS e 10 µ l 50 mM sodio azide. Vortice di mescolare.
- Dividere in aliquote di µ l 8 monouso in provette da 0,2 mL e conservare in una scatola scura a 4 ° C per ridurre al minimo la sensibilità alla luce associata.
Nota: Conservante sodio azide è facoltativa e può essere omesso se gli stock di 10 mg/mL sono realizzati con 1 mL di PBS 1X sterile, aliquotati e conservati a-20 ° C.
- Dividere in aliquote di µ l 8 monouso in provette da 0,2 mL e conservare in una scatola scura a 4 ° C per ridurre al minimo la sensibilità alla luce associata.
- Fare un 10 mL di soluzione di 5% colorante alimentare in PBS 1X mescolando 500 µ l siringa filtrata cibo verde colorazione e 9,5 mL sterile 1X PBS.
- Lavare le particelle prima dell'iniezione per rimuovere sodio azide. Sterile 1 µ l di mix 42 x PBS e 8 µ l di 10 mg/mL in una provetta da 1,7 mL. Centrifugare a massima velocità per 2,5 min a temperatura ambiente.
- Eliminare il surnatante, aggiungere 50 µ l 1 x PBS e centrifuga a velocità max per 2,5 min a temperatura ambiente.
- Ripetere i passaggi da 1.3 e 1.3.1 x 2, per un totale di 3 lavaggi.
- Dopo il lavaggio finale, scartare il surnatante e risospendere le particelle a 1,6 mg/mL in 50 µ l di 5% colorante alimentare in PBS 1X.
- Avvolgere il tubo in alluminio per proteggerlo dalla luce. Conservare a 4 ° C, gettare dopo 1 settimana.
2. preparare la stazione di iniezione e mosche
- Preparare il tampone di iniezione. Per iniettare fino a 4 genotipi di mosche allo stesso tempo, uso laboratorio nastro per dividere una rettangolare CO2 Mosca pad in 4 sezioni. Sulla panchina vicino al microscopio, designare le zone per posizionare le fiale una volta mosche sono stati allineati sul pad (uno per ogni angolo del pad).
- Preparare i flaconcini di pari età, 4-7 giorni-vecchie, mosche per iniezione. Per ogni sforzo essere testato, trasferire 5 maschi e 5 femmine in una fiala di fresca, con etichetta di cibo preparato Vola e mantenere a 25 ° C.
- Preparare l'iniettore pneumatico (Vedi Tabella materiali) impostando lo strumento su una modalità temporizzata di 100 ms (brevi raffiche di pressione del gas per espellere il liquido – che consentono la fornitura di sub-nanolitro volumi).
- Preparare i vetrini di microscopio. Tagliare le strisce di 1,5 pollici di nastro isolante, piegare in un ciclo con il lato adesivo fuori e posto su un vetrino da microscopio con etichetta.
3. preparare gli aghi capillare di vetro
- Tirare gli aghi di vetro (tubi capillari in vetro parete sottile) utilizzando un estrattore di ago.
- Tenere l'ago sotto il microscopio con un micrometro e rompere la punta con una pinzetta #5 punta fine in acciaio inox. 100 µm suggerimenti sono sufficienti per perforare la cuticola della Mosca mentre minimizza il ferimento.
- Misurare il volume di liquido che sarà iniettato in ogni volo. Caricare l'ago con colorante in 1X PBS sterile 5% alimentare ed espellere il liquido su una goccia di olio minerale su un micrometro di 0,01 mm.
Nota: Se la goccia di liquido è sferica, il volume in picolitri è calcolato come (dimensione)3/1910. 30 un ago con un diametro di 100 µm espellerà ~ 2 nL a 100 ms.
4. iniettare mosche
- Pipettare 10 µ l di particelle di 1,6 mg/mL su una piccola piazza di parafilm.
- Tirare il liquido nell'ago e montare l'ugello iniettore (Vedi Tabella materiali).
- Anestetizzare mosche con CO2 e metterli in fila nella loro area designata su flypad, con il lato ventrale verso l'alto e le teste orientate verso la parte anteriore del pad. Posizionare fiale nelle aree corrispondenti in panchina.
- Iniettare mosche nell'angolo superiore dell'addome con 5, ms 100 pompe di liquido (totale di ~ 10 nL).
- Trasferire le mosche iniettate alle fiale appropriate, il tempo di notare sul flacone. Conservare a 25 ° C.
- Caricare un nuovo ago con 0,4% soluzione di blu di Trypan.
- Impostare l'iniettore pneumatico GATED, che permette un flusso costante di aria per spingere il liquido fuori l'ago.
- Anestetizzare mosche dopo che essi hanno riposato per 30 min e iniettare con Trypan Blue, fino a quando l'addome è completo e dilatato.
Nota: Quando si esamina la maturazione del fagosoma con particelle marcate con un colorante sensibile al pH, consente mosche a riposare per 1 h e non iniettare con trypan blu prima di montaggio mosche. - Montare mosche su vetrini da microscopio con del nastro isolante, lato ventrale verso il basso. Spingere le ali al lato della Mosca e fissarli al nastro. Inoltre, spingere delicatamente la testa nel nastro per garantire che non si muoverà al volo.
- Passare immediatamente alla fase 5.
5. imaging mosche
- Immagine Vola, uno alla volta, a x 25 o 32 x ingrandimento, utilizzando un microscopio di fluorescenza invertito collegato a una fotocamera digitale e computer (Vedi Tabella materiali). Concentrarsi sul vaso dorsale della Mosca utilizzando software per computer per la fotocamera digitale.
- Registrare il tempo di esposizione e l'ingrandimento tra esperimenti.
Nota: Perdere traccia dei genotipi è una potenziale fonte di errore quando si fotografano ceppi multipli in una sola seduta. Per evitare un'etichettatura erronea mosche, annotare il numero dell'immagine della prima e l'ultima fotografia Vola per ciascun genotipo.
6. quantificare e normalizzare la fluorescenza
- Aprire il software e aprire una sola immagine alla volta.
- Misurare l'intensità di fluorescenza del vaso dorsale. Disegnare un poligono attorno al vaso dorsale. Selezionare misura e registra l'intensità della fluorescenza all'interno del poligono.
- Determinare l'intensità di fluorescenza di fondo. Copiare il primo poligono e spostarlo in un'area adiacente al vaso dorsale della Mosca. Selezionare la misura e l'intensità di fluorescenza record della zona di sfondo.
- Normalizzare la fluorescenza del vaso dorsale di fluorescenza di fondo:
Priorità bassa di ÷ vaso dorsale. - Calcolare la che media normalizzati intensità di fluorescenza del vaso dorsale di tutte le mosche in un ceppo.
- Ripetere l'esperimento 2 volte.
- Uso uno studente di spaiati t-test per confrontare le intensità media di fluorescenza relativa di controllo mosche e testare Vola da tre esperimenti. Calcolare la dimensione dell'effetto utilizzando la formula: Cohen d = (M1- M2) ÷ SDpool, dove M1 è la media di genotipo 1 e M2 è la media del genotipo 2 &
SD in pool =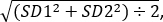
dove SD1 è la deviazione standard di genotipo 1 e SD2 è la deviazione standard di genotipo 2.
Risultati
Un disegno schematico del dosaggio fagocitosi in vivo utilizzando particelle della fluorescina-labeled è mostrato in Figura 1A. Le mosche sono montati sul lato ventrale verso il basso su un pezzo di nastro isolante e i primi due segmenti dell'addome, dove il vaso dorsale si trova, è chiaramente visibile (Figura 1B). Principali fonti di errore sperimentale derivano alle fasi della procedura (Figura 1
Discussione
Commercialmente disponibile, fluorescente contrassegnate particelle sono utilizzate per valutare la fagocitosi in generale (0,2 µm per volta carbossilato microsfere) o fagocitosi dei microbi (calore - fluorescente etichettati o chimicamente uccisi batteri o lieviti). Per valutare la maturazione del fagosoma, i ricercatori possono selezionare particelle marcate con un colorante di pH-sensibile che reagisce in quando il pH si abbassa da neutro ad acido, come il phagolysosome. In alternativa, per esaminare i p...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori ringraziano il Dr. Beth Gonzalez e Dr. Aprajita Garg per supporto nello svolgimento degli esperimenti in vivo fagocitosi. Una sovvenzione di seme di NSF UMD ADVANCE e UMD NIH T32 borse per la formazione, cellula e biologia molecolare (CMB) e le interazioni ospite-patogeno (HPI), finanziato quest'opera.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2μm Red Fluorescent Carboxylate Modified FluoSpheres | Invitrogen | F8810 | Fluorescently-labeled latex beads to test general phagocytic capacity of phagocytes. (~580/~605 nm) Inject a 1:20 dilution in PBS with 5% dye. |
| 5430-10 PicoNozzle Kit | World Precision Instruments | 5430-10 | Holder for 1.0mm pipette |
| E. coli (K-12 Strain) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | E13231 | Killed E. coli labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~495/~519 nm) |
| E. coli (K-12 Strain) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | E23370 | Killed E. coli labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~590/~617 nm) |
| E. coli (K-12 Strain) BioParticles, Fluorescein conjugate | Invitrogen | E2861 | Killed E. coli labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~494/~518 nm) |
| E. coli (K-12 Strain) BioParticles, Texas Red conjugate | Invitrogen | E2863 | Killed E. coli labeled with Texas Red. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~595/~615 nm) |
| E. coli (K-12 Strain) BioParticles, Texas Red conjugate | Invitrogen | E2863 | Killed E. coli labeled with Texas Red. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~595/~615 nm) |
| Needle Pipette Puller | David Kopf Instruments | Model 725 | |
| pHrodo Red E. coli BioParticles Conjugate for Phagocytosis | Invitrogen | P35361 | Killed E. coli labeled with pHrodo Red. Use to test phagocyte reconition, uptake, and phagosome maturation of gram-negative bacteria. (~560/~585 nm). No need to quench with Trypan Blue. |
| pHrodo Red S. aureus BioParticles Conjugate for Phagocytosis | Invitrogen | A10010 | Killed S. aureus labeled with pHrodo Red. Use to test phagocyte reconition, uptake, and phagosome maturation of gram-positve bacteria. (~560/~585 nm). No need to quench with Trypan Blue. |
| Pneumatic PicoPump PV820 | World Precision Instruments | SYS-PV820 | The World Precision Instruments Pneumatic PicoPump PV820 uses differential pressures to hold liquid in the glass needle between injections. The user manually controls short bursts of gas pressure to expel the liquid – allowing delivery of sub-nanoliter volumes. The amount of liquid delivered depends on two main variables – the size of the glass needle opening and the amount of time injection pressure is applied. set the instrument to 100 ms “TIMED” mode. |
| S. aureus (Wood Strain without protein A) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | S23371 | Killed S. aureus labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~495/~519 nm) |
| S. aureus (Wood Strain without protein A) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | S23372 | Killed S. aureus labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~590/~617 nm) |
| S. aureus (Wood Strain without protein A) BioParticles, Fluorescein conjugate | Invitrogen | E2851 | Killed S. aureus labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~494/~518 nm) |
| Thin Wall Glass Capillaries | World Precision Instruments | TW100F-3 | Needles for injection. OD = 1.0 mm |
| Trypan Blue Solution (0.4%) | Sigma | T8154 | Used to quench extracellular fluorescence of Fluorescein, Alexa Fluor, or Texas Red labeled particles. |
| ZEISS SteREO Microscope (Discovery.V8) | Zeiss | SteREO Discovery.V8 | Inverted fluorescence microscope for imaging flies. Use a digital camera (example: AxioCam HC camera) and the accompanying software (example: AxioVision 4.7 software) to take pictures. |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | Z23373 | Killed labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of yeast. (~495/~519 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | Z23374 | Killed labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of yeast. (~590/~617 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Fluorescein conjugate | Invitrogen | Z2841 | Killed labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of yeast. (~494/~518 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Texas Red | Invitrogen | Z2843 | Killed labeled with Texas Red. Use to test phagocyte recogntion and uptake of yeast. (~595/~615 nm) |
Riferimenti
- Akira, S., Uematsu, S., Takeuchi, O. Pathogen recognition and innate immunity. Cell. 124 (4), 783-801 (2006).
- Kim, D. Studying host-pathogen interactions and innate immunity in Caenorhabditis elegans. Disease Models & Mechanisms. 1 (4-5), 205-208 (2008).
- Lemaitre, B., Hoffmann, J. The host defense of Drosophila melanogaster. Annual Review Immunology. 25, 697-743 (2007).
- Wu, L. P., Choe, K. M., Lu, Y., Anderson, K. V. Drosophila Immunity: Genes on the Third Chromosome Required for the Response to Bacterial Infection. Genetics. 159 (1), 189-199 (2001).
- De Gregorio, E., Spellman, P. T., Tzou, P., Rubin, G. M., Lemaitre, B. The Toll and Imd pathways are the major regulators of the immune response in Drosophila. EMBO Journal. 21 (11), 2568-2579 (2002).
- Michel, T., Reichhart, J. M., Hoffmann, J. A., Royet, J. Drosophila Toll is activated by Gram-positive bacteria through a circulating peptidoglycan recognition protein. Nature. 414 (6865), 756-759 (2001).
- Choe, K. M., Werner, T., Stoven, S., Hultmark, D., Anderson, K. V. Requirement for a peptidoglycan recognition protein (PGRP) in Relish activation and antibacterial immune responses in Drosophila. Science. 296 (5566), 359-362 (2002).
- Wu, J., Randle, K. E., Wu, L. P. ird1 is a Vps15 homologue important for antibacterial immune responses in Drosophila. Cellular Microbiology. 9 (4), 1073-1085 (2007).
- Lemaitre, B., Nicolas, E., Michaut, L., Reichhart, J. M., Hoffmann, J. A. The dorsoventral regulatory gene cassette spatzle/Toll/cactus controls the potent antifungal response in Drosophila adults. Cell. 86 (6), 973-983 (1996).
- Zambon, R. A., Nandakumar, M., Vakharia, V. N., Wu, L. P. The Toll pathway is important for an antiviral response in Drosophila. Proceedings of the National Academy of Science of the United States of America. 102 (20), 7257-7262 (2005).
- Lemaitre, B., et al. A recessive mutation, immune deficiency (imd), defines two distinct control pathways in the Drosophila host defense. Proceedings of the National Academy of Science of the United States of America. 92 (21), 9465-9469 (1995).
- Leulier, F., Rodriguez, A., Khush, R. S., Abrams, J. M., Lemaitre, B. The Drosophila caspase Dredd is required to resist gram-negative bacterial infection. EMBO Reports. 1 (4), 353-358 (2000).
- Meister, M., Lagueux, M. Drosophilablood cells. Cellular Microbiology. 5 (9), 573-580 (2003).
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annual Review Pathology. 7, 61-98 (2012).
- Anderson, R. A., Sando, G. N. Cloning and expression of cDNA encoding human lysosomal acid lipase/cholesteryl ester hydrolase. Similarities to gastric and lingual lipases. Journal of Biological Chemistry. 266 (33), 22479-22484 (1991).
- Ross, G. D., Reed, W., Dalzell, J. G., Becker, S. E., Hogg, N. Macrophage cytoskeleton association with CR3 and CR4 regulates receptor mobility and phagocytosis of iC3b-opsonized erythrocytes. Journal of Leukocyte Biology. 51 (2), 109-117 (1992).
- Elrod-Erickson, M., Mishra, S., Schneider, D. S. Interactions between the cellular and humoral immune responses in Drosophila. Current Biology. 10, 781-784 (2000).
- Ramet, M., Pearson, A. M., Manfruelli, P., Li, X., Koziel, H., Gobel, V. Drosophila Scavenger Receptor CI Is a Pattern Recognition Receptor for Bacteria. Immunity. 15 (6), 1027-1038 (2001).
- Ramet, M., Manfruelli, P., Pearson, A. M., Mathey-Prevot, B., Ezekowitz, R. A. Functional genomic analysis of phagocytosis and identification of a Drosophila receptor for E. coli. Nature. 416 (6881), 644-648 (2002).
- Philips, J. A., Rubin, E. J., Perrimon, N. Drosophila RNAi screen reveals CD36 family member required for mycobacterial infection. Science. 309, 1251-1253 (2005).
- Agaisse, H., Burrack, L. S., Philips, J. A., Rubin, E. J., Perrimon, N., Higgins, D. E. Genome-wide RNAi screen for host factors required for intracellular bacterial infection. Science. 309 (5738), 1248-1251 (2005).
- Stuart, L. M., et al. Response to Staphylococcus aureus requires CD36-mediated phagocytosis triggered by the COOH-terminal cytoplasmic domain. Journal of Cell Biology. 170 (3), 477-485 (2005).
- Stroschein-Stevenson, S. L., Foley, E., O’Farrell, P. H., Johnson, A. D. Identification of Drosophila gene products required for phagocytosis of Candida albicans. PLoS Biology. 4 (1), e4 (2006).
- Kocks, C., et al. Eater, a transmembrane protein mediating phagocytosis of bacterial pathogens in Drosophila. Cell. 123 (2), 335-346 (2005).
- Nehme, N. T., et al. Relative roles of the cellular and humoral responses in the Drosophila host defense against three gram-positive bacterial infections. PLoS One. 6 (3), e14743 (2011).
- Garver, L. S., Wu, J., Wu, L. P. The peptidoglycan recognition protein PGRP-SC1a is essential for Toll signaling and phagocytosis of Staphylococcus aureus in Drosophila. Proceedings of the National Academy of Science of the United States of America. 103 (3), 660-665 (2006).
- Garg, A., Wu, L. P. Drosophila Rab14 mediates phagocytosis in the immune response to Staphylococcus aureus. Cellular Microbiology. 16 (2), 296-310 (2014).
- Gonzalez, E. A., Garg, A., Tang, J., Nazario-Toole, A. E., Wu, L. P. A glutamate-dependent redox system in blood cells is integral for phagocytosis in Drosophila melanogaster. Current Biology. 23 (22), 2319-2324 (2013).
- Nazario-Toole, A. E., Robalino, J., Okrah, K., Corrada-Bravo, H., Mount, S. M., Wu, L. P. The Splicing Factor RNA-Binding Fox Protein 1 Mediates the Cellular Immune Response in Drosophila melanogaster. Journal of Immunology (Baltimore, Md: 1950). 201 (4), 1154-1164 (2018).
- Guille, M. . Molecular Methods in Developmental Biology: Xenopus and Zebrafish. , (1999).
- Koundakjian, E. J., Cowan, D. M., Hardy, R. W., Becker, A. H. The Zuker collection: a resource for the analysis of autosomal gene function in Drosophila melanogaster. Genetics. 167 (1), 203-206 (2004).
- Horn, L., Leips, J., Starz-Gaiano, M. Phagocytic ability declines with age in adult Drosophila hemocytes. Aging Cell. 13 (4), 719-728 (2014).
- Brennan, C. A., Delaney, J. R., Schneider, D. S., Anderson, K. V. Psidin is required in Drosophila blood cells for both phagocytic degradation and immune activation of the fat body. Current Biology. 17 (1), 67-72 (2007).
- Akbar, M. A., Tracy, C., Kahr, W. H., Kramer, H. The full-of-bacteria gene is required for phagosome maturation during immune defense in Drosophila. Journal of Cell Biology. 192 (3), 383-390 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon