Method Article
使用超低输入载波保持架进行转录启动站点映射
摘要
基因表达(CAGE)帽分析是一种对mRNA 5'ends进行全基因组定量映射的方法,以单核苷酸分辨率捕获RNA聚合酶II转录起始位点。本作品描述了一种低输入(SLIC-CAGE)协议,用于使用纳米图总RNA生成高质量库。
摘要
基因表达(CAGE)帽分析是一种用于RNA聚合酶II转录起始位点(TSSs)的单核苷酸分辨率检测方法。准确检测 TSS 可增强核心启动子的识别和发现。此外,可以通过双向转录启动的签名来检测有源增强剂。此处介绍用于执行超低输入载波 CAGE(SLIC-CAGE)的协议。CAGE协议的这种SLIC适应通过人工增加RNA量,通过使用在感兴趣的样品中加入的体外转录RNA载体混合物,从而从纳米图总量中制备,从而最大限度地减少了RNA损失RNA(即数千个细胞)。载体模拟预期的DNA库片段长度分布,从而消除由同质载体丰度引起的偏差。在协议的最后阶段,载体通过带宿主内分酶的降解被移除,目标库被放大。目标样本库受到保护,防止降解,因为宿主内分酶识别位点很长(在18至27bp之间),使得它们在真核基因组中存在的概率非常低。最终结果是DNA库为下一代测序做好准备。协议中的所有步骤,至顺序,可在 6 天内完成。承运人准备需要一个完整的工作日;然而,它可以大量制备,并保持在-80°C冷冻。一旦测序,读取可以处理获得全基因组的单核苷酸分辨率TSS.TSS可用于核心促进剂或增强剂发现,提供对基因调控的洞察。聚合到启动器后,数据还可用于 5' 为中心的表达式分析。
引言
基因表达(CAGE)的帽分析是一种用于单核苷酸解析全基因组图谱的RNA聚合酶II转录起始位点(TSSs)1。其定量性质还允许 5' 端为中心的表达式分析。围绕TSS(约40 bp上游和下游)的区域是核心启动子,代表RNA聚合酶II和一般转录因子结合的物理位置(前2、3)。有关 TSS 的确切位置的信息可用于核心启动器发现和监测启动子动力学。此外,由于活性增强剂表现出双向转录的特征,CAGE数据也可用于增强剂发现和监测增强剂动力学4。CAGE方法最近越来越受欢迎,因为它在诸如ENCODE 5、modENCODE6和FANTOM项目7等备受瞩目的研究项目中有着广泛的应用和用途。此外,TSS 信息也被证明是重要的区分健康和疾病组织,因为疾病特异性的TSS可用于诊断目的8。
尽管有几种 TSS 映射方法可用(CAGE、RAMPAGE、STRT、纳米笼、纳米CAGE-XL、寡核封盖),但我们和其他人最近已经表明,CAGE 是最无偏见的方法来捕获假阳性数最少的真实 TSS9,10.最近的CAGE协议,nAnT-iCAGE11,是TSS分析最公正的协议,因为它避免使用限制性酶将片段切割成短标记,并且不使用PCR扩增。nAnT-iCAGE 协议的局限性是需要大量起始材料(例如,每个样品的总 RNA 量为 5 μg)。为了回答具体、与生物学相关的问题,通常无法获得如此大量的起始材料(例如,对于FACS排序的细胞或早期胚胎阶段)。最后,如果nAnT-iCAGE成功,每个样本只能获得1-2 ng的DNA库材料,从而限制了可实现的测序深度。
为了仅使用总RNA的纳米图进行TSS分析,我们最近开发了超低输入载波-CAGE 10(SLIC-CAGE,图1)。SLIC-CAGE 只需要总 RNA 的 10 纳克才能获得高复杂度库。我们的协议依赖于精心设计的合成RNA载体添加到感兴趣的RNA,实现总共5μg的RNA材料。合成载体在长度分布上模仿目标DNA库,以避免由过量的均质分子引起的潜在偏差。载体的序列基于大肠杆菌-tRNA合成酶基因(表1)的序列,原因有二。首先,最终库中载体的任何剩余,即使已测序,也不会映射到真核基因组。其次,由于大肠杆菌是一种嗜血菌物种,其内务管理基因针对适用于SLIC-CAGE的温度范围进行了优化。载体序列还嵌入了宿主内源内切酶识别位点,允许从载体RNA分子中提取的DNA进行特定降解。目标样本派生库保持不变,因为宿主内分酶识别站点很长(I-CeuI = 27 bp;I-SceI = 18 bp),在真核基因组中不太可能发现。在载体具体降解和通过大小排除去除碎片后,目标库被扩增,为下一代测序做好准备。根据起始RNA量(1-100纳克),预计在13-18PCR扩增周期之间。每个样本的最终DNA量在5-50 ng之间,产生足够的材料进行深度测序。当只使用总RNA的1-2纳克时,可以检测到真正的TSS;但是,这些库的复杂性预计会较低。最后,由于SLIC-CAGE基于nAnT-iCAGE协议11,因此在测序之前,它能对多达8个样本进行多路复用。
研究方案
1. 准备承运商
-
制备体外转录的DNA模板
- 通过组合41μL水、20μL的5xHF缓冲液、8μL的2.5mM dNTP、10μL的10μM独特正向底漆(PCR_GN5_f1,表2;引热器在水中溶解和稀释),为每个PCR模板制备PCR混合物。,10 μL 的 2 纳克/μL 模板质粒,包含合成载体基因和 1 μL Phusion 聚合酶。通过移液混合 PCR 混合物。可以同时准备所有 10 个模板的主组合(为 11 个反应做好准备)。
- 将 PCR 混合物的 90 μL 添加到每 10 μM 反向底漆的 10 μL (PCR_N6_r1-r10,表 2)。通过移液混合。
- PCR 使用以下程序放大模板:98 °C 表示 60 秒,(98 °C 表示 10 秒,50 °C 表示 30 秒),35 周期为 72°C,72°C 为 10 分钟,在 4°C 保持。
-
PCR扩增DNA模板的凝胶纯化
- 准备1%的甘蔗凝胶(建议低熔胶)。
- 为了减小体积,在低温(30-40°C)下,使用真空集中器将PCR反应混合物的总体积从100μL浓缩至20μL总体积。
- 加入6μL的6x加载染料,混合良好,并加载在凝胶上。在适用于所用电泳罐(5~10 V/cm)的电压下,在 1x TAE 缓冲液中运行电泳 30 分钟。并行运行 100 bp 或 1,000 bp DNA 阶梯。
- 使用干净的手术刀,切除含有目标PCR产品的凝胶片。避免过量的阿加玫瑰凝胶。使用凝胶萃取套件净化 PCR 产品(根据制造商的说明)。
注:从胶凝糖凝胶中分离出的A260/A230DNA比例通常较低(0.1~0.3)。预期目标产品和侧面产品如图2A所示。100 μL PCR 反应的预期收率为 1.2±3 μg。反应可以放大以获得更高的收率。
-
载体分子体外转录
- 根据制造商的说明,使用T7 RNA聚合酶在体外抄录载体RNA。设置 10~20 μL 反应(推荐的试剂盒在材料表中)。
- 使用RNA纯化试剂盒纯化体外转录RNA。按照制造商的标准说明,使用DNase I在溶液中建立DNA消化,并在50μL水中释放RNA。要提高洗脱产量,在离心前将水留在柱中 5 分钟。
注:请注意不要超过列的最大装订能力(在材料表中提及的套件中,容量高达 100 μg)。PCR 模板 1~10 (长度为 1 kbp 至 200 bp)的预期收率为 10 μL 体外转录反应的 25–50 μg。反应可以放大,以获得更多的载体分子。
-
体外转录载体RNA分子的封顶
- 通过组合 2 μL 的 10x 封盖缓冲液、1 μL 的 10 mM GTP、1 μL 的 2 mM SAM(新鲜稀释)和 1 μL 的 Vaccinia 封盖酶(每个载体 RNA)来制备封盖混合物。
- 将每个载体分子的10μg混合在总体积的15μL中,在65°C下变性10分钟。立即放置在冰上,以防止二级结构形成。
- 将变性载体RNA与5μL的封顶混合物混合,在37°C下孵育1小时。
- 使用RNA纯化试剂盒纯化封顶RNA分子 – 遵循制造商的清理方案。在30μL水中的ELute RNA。要提高洗脱产量,在离心前将水留在柱中 5 分钟。
注:使用微体积分光光度计测量浓度。预期 A260/A280 比率为 >2,A260/A230 为 >2。请注意,对于某些 RNA 样本,A260/A230 可能介于 1.3⁄2 之间。使用10μg未封顶RNA时的预期收率为9~10μg的封顶RNA。
- 通过组合表 3中描述的金额,准备封顶和未封顶车厢的组合。通过轻拂管子和使用微体积分光光度计测量浓度,将混合良好。
注:如果需要较高的载体浓度以适合逆转录反应(见下文),则载波混合物可在中低温(30-35 °C)下使用真空集中器浓缩,直到达到所需的最终浓度。步骤 2_14 从村田等人报告的标准 nAnT-iCAGE 协议修改,11
2. 反向转录
- 将1 μL的RT引物(2.5mM TCT-N6溶解在水中,对于序列见补充表1),10 ng的总RNA感兴趣和4,990纳克的载体混合物(表3)在10μL的总体积在低结合PCR板。通过轻拂管子进行混合。
注:如果样品RNA稀释过,无法进行逆转录(见下文),将其与适当量的载体结合,使用真空浓缩器浓缩至9μL总体积,并添加1μL的RT底漆。加入载体耳,达到5μg的RNA总可防止样品流失。 - 在 65°C 下将混合物从步骤 2.1 加热 5 分钟,并立即放在冰上,以防止补放。
-
准备逆转录 (RT) 混合。
- 对于每个样品,结合6.1 μL的水(无RNase和DNase),7.6 μL的5x第一链缓冲液,1.9μL的0.1M DTT,1μL的10mM dNTP,7.6 μL的沙洛糖/山梨醇混合物(见村田等人11的配方)和3.8μL的推荐逆转录酶(见[见]c)6、材料表)。轻拂管子,将管很好地混合。
- 将28μL的RT混合物加入PCR管中,加入10μL的RNA、载体和RT底漆(总体积38μL)。通过移液将混合良好。
注:由于沙沙洛/山梨醇,该混合物具有高度粘性。混合,直到明显同质。 - 使用以下程序在热循环器中孵育:25 °C 30 秒,50°C 60 分钟,并在 4°C 保持。
-
使用SPRI磁珠纯化cDNA:RNA杂交
- 将68.4 μL的推荐RNA酶和无DNase SPRI磁珠(见材料表)添加到RT混合物的38μL(珠与样品比1.8:1)。在室温 (RT) 下,通过移液和孵育 5 分钟,将混合良好。
- 将磁力支架上的珠子分离5分钟,丢弃上清液,用200μL的70%乙醇(新鲜制备)清洗珠子两次。
注:乙醇在不混合的情况下,当管子位于磁性支架上时,将乙醇添加到磁珠中。添加的乙醇被立即去除。在进行处理时,应小心不要丢失任何珠子,因为它可能导致样品丢失。 - 当管子仍然位于磁性支架上时,去除所有乙醇痕迹。使用 P10 移液器可以取出乙醇液滴并将其推出管中。不要让珠子干燥。
- 在 37°C 下预热 42 μL 的水,通过上下移液 60 倍来洗取样品。
注:小心不要通过移液引起发泡,因为它可能导致泡沫中的珠子(即带着样品)损失。 - 在37°C下孵育5分钟,不盖,允许微量乙醇蒸发。
- 将磁架上的磁珠分开 5 分钟,并将上清液转移到新板。
注:尝试检索所有上清液,以防止样品丢失,同时避免珠子结转。使用 P10 移液器获取最后一个样品液滴。
3. 氧化
- 在纯化RT反应中加入2 μL的1M NaOAc(pH 4.5)。通过移液混合,加入 2 μL 的 250 mM NaIO 4,然后再次混合。
- 在冰上孵育45分钟,用铝箔盖住盘子,避免光线照射。
- 在氧化混合物中加入16μL的Tris-HCl(pH 8.5),以中和pH值。
- 使用 SPRI 磁珠纯化氧化 cDNA:RNA 杂交。将 108 μL 的 SPRI 磁珠添加到 60 μL 的氧化混合物中(1.8:1 珠与样品比)。重复步骤 2.6.1~2.6.6 中所述的纯化。使用42μL的水预热至37°C。
注:通过每 1 毫克 NaIO4加入 18.7 μL 水,新鲜制备 250 mM NaIO4 。NaIO4是光敏的;因此,将溶液保存在铝箔覆盖的管中或耐光管中。
4. 生物异化
- 在含有纯化氧化样品的管中加入4μL的1M NaOAc(pH 6.0),并通过移液混合。
- 加入4μL的10 mM生物素溶液,通过移液混合,在23°C下在热循环器中孵育2小时,以避免光线。
注:通过将 50 mg 生物锡与 13.5 mL DMSO 混合制备生物锡溶液。制作一次性等分,在-80°C冷冻。 - 使用 SPRI 磁珠纯化生物素化 cDNA:RNA 杂交。加入12μL的2丙醇,并通过移液混合。加入 108 μL 的 SPRI 珠(1.8:1 珠与样品比),然后重复步骤 2.6.1~2.6.6 中所述的纯化。使用42 μL的水在37°C预热。
注:协议可以在这里暂停,样品冷冻在-80°C。
5. RNase I 消化不良
- 将 4.5 μL 的 10x RNase I 缓冲液与每个样品 0.5 μL 的 RNase I (10 U/μL) 混合制备 RNase I。通过移液混合。
- 在每个纯化样品中加入5μL的混合物(共45μL)。通过移液混合,在37°C下孵育30分钟。
6. 准备链球菌珠
- 对于每个样品,将30μL的链球菌珠浆与0.38μL的20mg/mL tRNA混合。在冰上孵育30分钟,每5分钟通过轻拂管子混合。
注:在移液之前,通过轻拂瓶子,重新悬浮链球菌珠浆。tRNA溶液应根据村田等人11 - 将磁性支架上的珠子分开 2⁄3 分钟,取出上清液。
- 通过重新悬浮在15 μL的缓冲液A中清洗珠子。将磁性支架上的珠子分离2⁄3分钟,并去除上清液。重复洗涤并取出上清液。
- 在105μL的缓冲A中重新悬浮珠子,并加入0.19 μL的20mg/mL的tRNA。通过移液将混合良好。
注:使用前应新鲜准备珠子。在RNase I消化期间开始准备珠子。对于多个样品,将珠子放在一个管中。
7. 捕获盖
-
示例绑定
- 将105 μL的制备链球菌珠加入45μL的RNase I处理样品中。通过移液混合,在37°C下孵育30分钟。每10分钟移液混合一次。
- 将磁性支架上的珠子分开 2⁄3 分钟,取出上清液。
-
洗涤珠
- 加入150 μL的洗涤缓冲液A,并通过移液重新悬浮珠子。将磁性支架上的磁珠分开 2⁄3 分钟,然后取出上清液。
- 加入150 μL的洗涤缓冲液B,并通过移液重新悬浮珠子。将磁性支架上的磁珠分开 2⁄3 分钟,然后取出上清液。
- 加入150 μL的洗涤缓冲液C,并通过移液重新悬浮珠子。将磁性支架上的磁珠分开 2⁄3 分钟,然后取出上清液。
注:缓冲液 B 和 C 应预热至 37°C。洗涤缓冲液A、B和C的配方如村田等人11所述
-
cDNA 释放
- 通过将 58.5 μL 的水与 6.5 μL 的 10x RNase I 缓冲液混合,制备 1x RNase I 缓冲液。
- 在 35 μL 的 1x RNase I 缓冲液中重新悬浮珠子。在95°C孵育5分钟,直接在冰上转移2分钟,以防止cDNA重新结合。在转移到冰上时握住盖子,因为它们可能会因压力积聚而弹出。
- 在磁性支架上分离珠子 2⁄3 分钟,并将上清液转移到新板。
- 在 30 μL 的 1x RNase I 缓冲液中重新悬浮珠子。将磁性支架上的珠子分离 2⁄3 分钟,并将上清液转移到先前收集的上清液(洗脱 cDNA 的总体积应约为 65 μL)。
8. RNA去除由RNase H和RNase I消化
- 每样品,结合2.4 μL的水,0.5 μL的10xRNase I缓冲液,0.1 μL的RNase H和2μL的RNase I。
- 将5μL的混合物加入释放的cDNA样品的65μL,并通过移液混合。在37°C孵育15分钟,在4°C保持。
- 使用 SPRI 磁珠从 RNase 消化混合物中纯化 cDNA。将 126 μL 的 SPRI 珠添加到 70 μL 的降解反应中,并通过移液混合。按照 2.6.1~2.6.6 中 SPRI 珠子纯化所述的纯化步骤进行操作。如所述,在37°C下使用42 μL的水预热。
- 通过组合 4.5 μL 10x RNase I 缓冲液和 0.5 μL RNase I 混合制备 RNase I。
- 将5μL的RNase混合物加入纯化cDNA样品的40μL。通过移液混合,在37°C下孵育30分钟,在4°C下保持。
- 使用 SPRI 磁珠净化样品。将 81 μL 的 SPRI 珠添加到 45 μL 的降解反应中,并通过移液混合。按照 2.6.1~2.6.6 中 SPRI 珠子纯化所述的纯化步骤进行操作。如上所述,使用42 μL的水。
9. 5' 链接器的关联
- 使用真空浓缩器将纯化的 cDNA 样品浓缩到 4 μL。保持温度在30~35 °C。使用移液器测试卷。如果样品干燥至完整,则加入4μL水溶解。
注:最好避免干燥,防止样品流失。 - 在95°C孵育浓缩样品5分钟,并立即在冰上放置2分钟,以防止补放。在转移管时握住盖子,因为盖子可能会因压力积聚而弹出。
- 在55°C下孵育2.5μM 5'连结器的4μL,5分钟,并立即在冰上放置2分钟,以防止肾上当。
- 将 2.5 μM 5' 链接器的 4 μL 与 4 μL 样品混合。
注:5'链接者应按照补充表2、补充表3、补充表4和补充表5编制。使用前使用 100 mM NaCl 将 10 μM 5' 连结器稀释至 2.5 μM 浓度。 - 将16μL的结扎预混合(见材料表)加入混合5'链接器和样品中,并通过移液很好地混合。在16°C孵育16小时。
- 使用 SPRI 磁珠净化结扎混合物。添加 43.2 μL 的 SPRI 珠,然后按照步骤 2.6.1~2.6.6 进行操作。使用42 μL的水在37°C预热。
- 在步骤 9.6 中重复纯化,将 72 μL 的 SPRI 磁珠添加到转移的上清液(1.8:1 珠与样品比)。
注:5'链接器包含条形码,允许在测序前汇集多达 8 个样本(如村田等人11和补充表 1中所述,提供 8 个三核苷酸条形码)。
10. 3' 链接器的关联
- 使用步骤 9.1 中所述的真空集中器将纯化样品浓缩到 4 μL。
- 在95°C孵育浓缩样品5分钟,并立即在冰上放置2分钟,以防止补放。在转移管时握住盖子,因为盖子可能会因压力积聚而弹出。
- 在 65°C 下孵育 2.5 μM 3' 连结器的 4 μL,5 分钟,并立即在冰上放置 2 分钟,以防止肾兴奋。
- 将2.5 μM 3' 链接器的4 μL添加到浓缩样品的4μL中。
- 加入16μL的结扎预混合,并通过移液混合良好。在16°C孵育16小时。
- 使用 SPRI 磁珠净化结扎混合物。添加 43.2 μL 的 SPRI 珠,然后按照步骤 2.6.1~2.6.6 进行操作。使用42 μL的水预热至37°C。
注:3' 链接器应根据补充表 6和补充表 7进行准备。使用 100 mM NaCl 将 10 μM 3' 连结器稀释至 2.5 μM 浓度。
11. 脱磷化
- 通过组合 4 μL 的水、5 μL 的 10x SAP 缓冲液和 1 μL SAP 酶来制备 SAP 混合物。
- 将 10 μL 的 SAP 混合物加入纯化的结扎样品(总体积 50 μL),并使用以下程序在热循环器中孵育:37°C 30 分钟,65°C 15 分钟,在 4°C 保持。
12. 使用乌拉西尔特异性切除酶降解 3' 链接器上绞线
- 在脱磷酸化样品中加入2μL的尿素特异性切除酶(见材料表),使用以下程序在热循环器中通过移液和孵育混合:37°C30分钟,95°C5分钟,并立即在冰上放置2分钟至防止支离破碎的上股的重新绞合。
- 在 52μl 混合物中加入 93.6 μL 的 SPRI 磁珠,并通过移液混合均匀地净化反应混合物。重复纯化步骤 2.6.1~2.6.6。如所述,在37°C下预热42μL水。
13. 第二股合成
- 通过组合 5 μL 的 10x DNA 聚合酶反应缓冲液、2 μL 水、1 μL 的 10 mM dNTP、1 μL 的 50 μM nAnT-iCAGE 第二链底漆(序列在补充表 1中)和 1 μL DNA 外糖缺乏聚合酶酶(参见材料表中推荐的聚合酶)。
- 将10μL的混合物加入纯化样品中,通过移液混合(总体积为50μL)。使用以下程序在热循环器中孵育:95°C 5分钟,55°C 5分钟,72°C 30分钟,保持4°C。
14. 使用外糖酶I降解第二股合成引物
- 将1μL的外糖酶I加入第二股合成混合物中。通过移液混合,在37°C孵育30分钟,然后保持在4°C。
- 将 91.8 μL 的 SPRI 磁珠加入 51 μL 的 Exonuclease I 处理样品,以纯化双绞合 DNA。重复 2.6.1~2.6.6 中所述的纯化步骤。并随42μL的水预热至37°C,如所述。
- 如步骤 9.1 所述,使用真空集中器将样品浓缩到 15 μL。
15. 质量和数量控制
- 使用1μL的浓缩样品,并在DNA质量分析仪上运行高灵敏度DNA芯片。预期配置文件/数量如图3所示。
16. 第一轮承运人退化
- 通过组合2μL的水、2μL的10x限制性酶缓冲液、1μL的I-SceI和1μL的I-CeuI来制备降解混合物。
- 将6μL的降解混合物加入浓缩样品的14μL,并通过移液混合。在37°C孵育3小时,随后在65°C下进行20分钟失活,并在4°C下保持。
- 使用 SPRI 磁珠净化降解混合物。加入5μL的水以增加降解混合物的体积,并加入45μL的SPRI珠(1.8:1珠与样品比)。重复纯化,如步骤 2.6.1~2.6.6 中所述。并用42μL的水预热至37°C。
- 如步骤 9.1 所述,将洗脱样品从总体积的 42 μL 浓缩到 20 μL。
17. 控制降解水平并确定PCR放大周期数
- 准备 qPCR 组合,用于放大整个库(适配器组合)。将 3.8 μL 的水、5 μL 的 qPCR 预混合 (2x)、0.1 μL 的 10 μM 适配器_f1 引物(5'-AATGATACAGACCACCA-3')和 0.1 μL 的 10 μM 适配器_r1 引物(5' - CAAGAGAGACACACGA-3') (参见推荐材料表)。
- 将 9 μL 的 qPCR 适配器组合与步骤 16.4 中的 1 μL 样品混合,并通过移液很好地混合。
- 准备qPCR混合物,用于扩增从载体(载体混合)中提取的DNA。结合 3.8 μL 的水,5 μL 的 qPCR 预混合 (2x), 0.1 μl 10 μM 载波_f1 引基 (5' -GCGGGCGCGCGCTAC-3'), 和 0.1 μL 的 10 μM 适配器_r1 引油每个样品
- 将 9 μL 的 qPCR 载体混合物与步骤 16.4 中的 1 μL 样品混合,并通过移液很好地混合。
- 设置 qPCR 程序:95 °C 3 分钟(95 °C 20 秒,60 °C 20 秒,72 °C 2 分钟)重复 40x,然后是仪器特异性变性曲线 (65-95 °C),并保持在 4 °C。
注:用水替换样品,准备负片控制。
18. 目标库的PCR扩增
- 通过组合 6 μL 水、0.5 μL 的 10 μM 适配器_f1 底漆、0.5 μL 的 10 μM 适配器_r1 引底和 25 μL 的 PCR 预混合(2x),制备 PCR 扩增混合物。通过移液混合(参见推荐的PCR预混料的材料表)。
- 从步骤16.4将PCR混合物的32μL添加到样品的18μL。通过移液彻底混合。
- 设置 PCR 放大:95 °C 3 分钟(98 °C 表示 20 秒,60°C 表示 15 秒,72°C 为 2 分钟)12-18 个周期,72°C 为 2 分钟,在 4°C 下保持。
注:PCR 循环的确切数量由 qPCR 结果确定,对应于使用适配器引物组合获得的 Ct 值(PCR 周期数等于 Ct 值)。 - 将 90 μL 的 SPRI 磁珠加入 50 μL 的放大样品中,并通过移液彻底混合,从而净化放大的样品。重复步骤 2.6.1~2.6.6 中描述的纯化步骤。并使用 42 μL 的水对样品进行洗取,如上所述。
19. 第二轮承运人退化
- 重复步骤 16.1~16.3。
- 使用 SPRI 磁珠净化降解混合物。向样品中加入10μL的水以增加体积,并与30μL的SPRI珠(1:1珠与样品比)混合。重复纯化,如步骤 2.6.1~2.6.6 中所述。并随42μL的水在37°C下预热,如所述。
- 将洗脱样品从总体积的42μL浓缩至30μL。
20. 库大小选择
- 将 24 μL 的 SPRI 磁珠与步骤 19.3 中的 30 μL 样品混合。(0.8:1 珠子与样品比)。重复步骤 2.6.1~2.6.6 中所述的纯化步骤。并在42μL的水中洗取样品,如上所述。
- 如步骤 9.1 所述,将样品浓缩到大约 14 μL。
21. 质量控制
-
规模分布评估
- 在高灵敏度DNA芯片上运行1μL的样品。预期结果如图4所示。
注:如果小于 200 bp 的片段可见(参见图 4A,C所示),则应重复大小选择(步骤 20.1–20.2),直到删除短片段(图 4B,D)。通常,额外的一轮大小选择就足够了。如果短片段的量很严重(如图4C所示),珠子与样本比应降至0.6:1。
- 在高灵敏度DNA芯片上运行1μL的样品。预期结果如图4所示。
-
车厢降解质量控制
- 重复步骤 17.1~17.5。
注:根据HS DNA芯片运行(区域分析)中估计的库的浓度,样品需要在qPCR之前稀释。使用0.5 μL的样品,以避免样品损失,并稀释100~500倍的水(稀释至1~20 pg/μL最终浓度)。使用适配器和载波组合获得的 Ct 值之间的预期差值为 5–10。
- 重复步骤 17.1~17.5。
-
库量化
- 通过将 20 μL 的 100 mg/mL lambda DNA 标准与 980 μL 的 1x TE 混合(通过稀释 DNA 定量试剂盒中提供的 20x TE 进行制备),制备 lambda DNA 标准的工作稀释。羔羊DNA的稀释可以储存在-20°C。
- 根据补充表8,通过混合稀释的lambda标准和1x TE制备lambda DNA标准连续稀释剂。
注:为获得更高的精度,建议在所有管中添加 100 μL 的 1x TE 缓冲液,并根据需要根据要添加的稀释的 lambda 体积去除 1x TE 体积。不要使用超过 1 μL 的库;建议使用 384 孔板进行此测量。
结果
本报告描述了从起始总RNA材料的纳米图中获取测序就绪库的完整SLIC-CAGE协议(图1)。为了获得合成RNA载体混合物,首先,PCR载体模板需要制备和凝胶纯化,以消除PCR侧产物(图2A)。每个PCR模板(共10个)是使用共同的正向,但不同的反向引物(表2),导致PCR模板的不同长度,使合成RNA载体的大小变异。一旦纯化,PCR模板用于载体分子的体外转录。如果模板经过凝胶纯化,则预期会提供单个RNA载体产品(参见图2B中的代表性凝胶分析)。载体的制备可根据需要进行升级,在制备时,混合和冷冻在-80°C下供将来使用。
使用推荐的最小样品总RNA(10 ng)结合16-18周期的PCR扩增,可以达到高复杂性SLIC-CAGE库。扩增最终库所需的PCR周期数在很大程度上取决于总输入RNA的数量(预期周期数在表4中)。
在第一轮降级后,在 qPCR 结果(步骤 17)中,使用适配器_f1 或载波_f1 引物获得的 Ct 值之间的预期差值为 1-2,使用适配器_f1 比载波_f1 低获得 Ct 值。
最终库中片段长度的分布在 200-2,000 bp 之间,平均片段大小为 700-900 bp(基于使用生物分析仪软件的区域分析,图 4B,D)。如图4A,C所示,较短的片段必须通过额外的大小排除(步骤20-21)来移除。这些短片段是 PCR 扩增人工制品,而不是目标库。请注意,较短的片段在测序流单元上更好地聚类,并可能导致排序问题。
每个样本获得库材料的预期量在 5-50 纳克之间。显著降低数量表明在协议期间样品损失。如果获得的低数量足以进行测序(需要 2-3 ng 的池库),则库的复杂性可能较低(见下文)。
根据测序机的不同,可能需要优化加载到流单元的库数量。使用 Illumina HiSeq 2500,加载 8-12 pM SLIC-CAGE 库平均提供 1.5 亿-2 亿次读取,其中 80% 的读取通过质量分数 Q30 作为阈值。
然后,获取的读取映射到参考基因组 [对于 50 bp 读取,Bowtie212可与允许每个种子序列(22 bp)零不匹配的默认参数一起使用]。预期的映射效率取决于总RNA输入量,如表5。然后,可以将唯一映射的读取加载到 R 图形和统计计算环境13中,并使用 CAGEr(生物导体封装14)进行处理。包晕影易于理解,并详细介绍了映射数据的工作流和处理。库复杂性的一个简单可视化控制是启动子宽度的分布,因为低复杂度库将人为地缩小启动子(图5A,SLIC-CAGE库派生自总RNA的1 ng,详情请见前文出版物10。然而,即使是低复杂度SLIC-CAGE库也能识别真正的CTSS,其精度比低/中位TSS映射的替代方法更精确(图5B,C)。
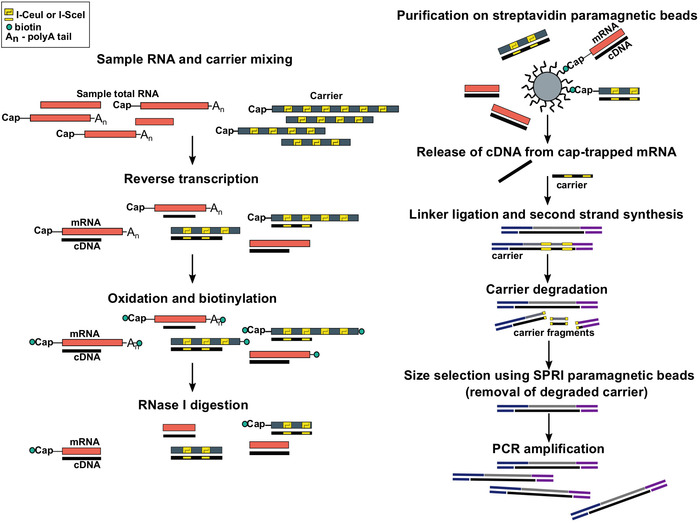
图 1:SLIC-CAGE 协议中的步骤。样品RNA与RNA载体混合物混合,达到总RNA材料的5μg。cDNA通过逆转录合成,盖使用酸钠氧化。氧化允许使用生物素氢化物将生物素附着在帽子上。生物蛋白附着在mRNA的3°端,因为它也使用酸钠被氧化。为了从mRNA:cDNA杂交物中消除生物素,从未完全合成的cDNA和mRNA的3°端,用RNase I.cDNA处理,然后通过链球菌素磁珠的亲亲纯化选择达到mRNA5°末端的rNase I.cDNA(捕获帽)。释放cDNA后,5+和3+链接器被连接。源自载体的库分子使用I-SceI和I-CeuI致内分酶降解,并使用SPRI磁珠去除片段。然后,库被放大。请点击此处查看此图的较大版本。
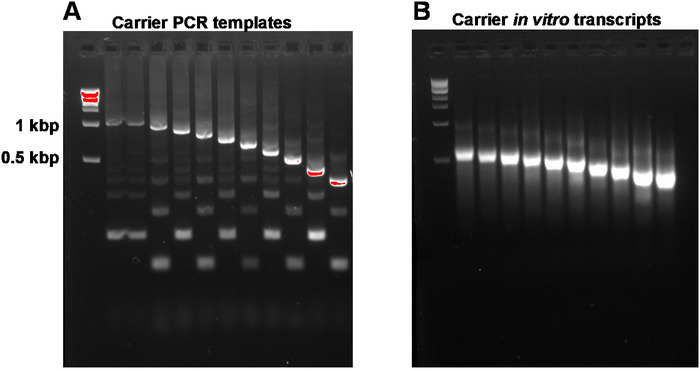
图2:载体PCR模板和载体体外转录本的代表性凝胶分析。(A)凝胶纯化前的载体PCR模板:第一口井包含1 kbp标记,然后是载体PCR模板1,1-10。(B)载体体外转录本:第一口井包含1 kbp标记,后跟载体转录1-10。在装车前,在95°C下加热5分钟,使载波转录。请点击此处查看此图的较大版本。
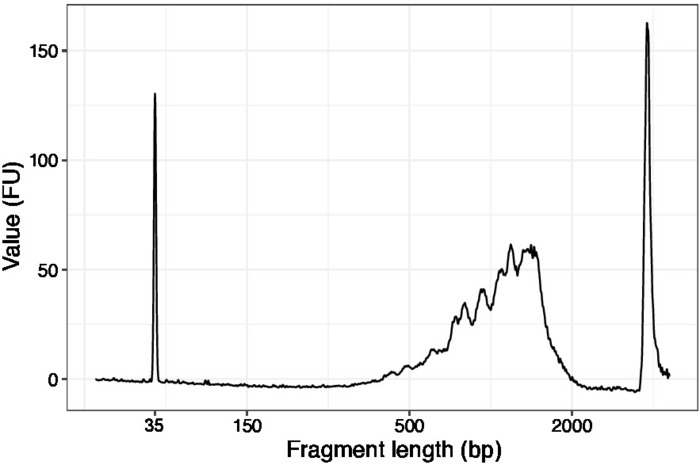
图3:代表性DNA质量(高灵敏度DNA芯片)的SLIC-CAGE在第一轮载体降解前的痕迹。请点击此处查看此图的较大版本。
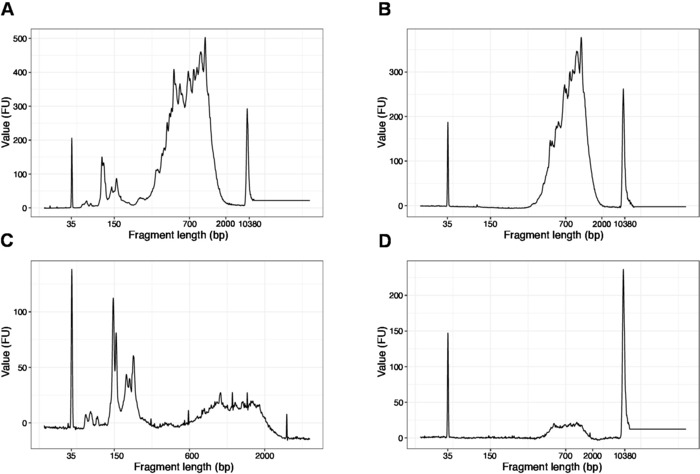
图4:PCR扩增后的SLIC-CAGE库的代表性DNA质量(高灵敏度DNA芯片)痕迹。(A) SLIC-CAGE 库需要额外的大小选择来删除短片段。(B) SLIC-CAGE 库在尺寸选择后使用 0.6x SPRI 磁珠与采样比。(C) 输出量较低的 SLIC-CAGE 库,需要选择大小以去除短片段。(D) SLIC-CAGE 库在选择尺寸后输出量较低,使用 0.6:1 SPRI 磁珠与采样比。请点击此处查看此图的较大版本。
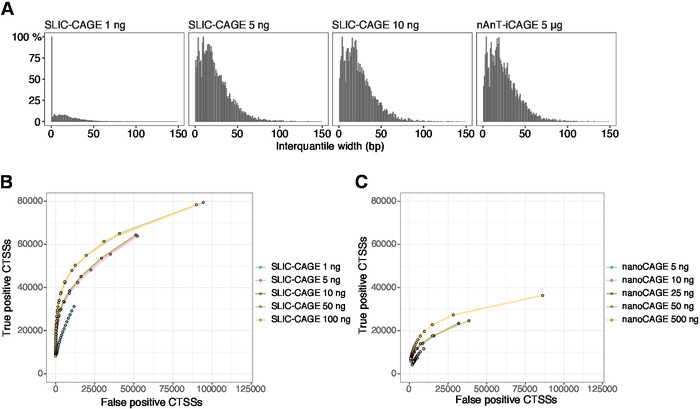
图 5:SLIC-CAGE 库的验证。(A)在 SLIC-CAGE 库中标记簇间宽度的分布,这些宽度从S.cerevisae总 RNA 的 1、5 或 10 ng 制备,并在 nAnT-iCAGE 库中从 5 μg 的S. cerevisae总 RNA 制备。1 ng SLIC-CAGE 库中的大量窄标记群集表示其低复杂性。(B) 在S. s. SLIC-CAGE 库中用于 CTSS 标识的 ROC 曲线。所有S. cerevisae nAnT-iCAGE CTSS 都用作真正的集。(C) 在S. cerevisae纳米库中用于 CTSS 识别的 ROC 曲线.所有S. cerevisae nAnT-iCAGE CTSS 都用作真正的集。ROC曲线的比较表明,SLIC-CAGE在CTSS识别中优于纳米凯奇。使用了阵列快递E-MTAB-6519的数据。请点击此处查看此图的较大版本。
表1:载体合成基因序列。I-SceI 网站为粗体,斜体为紫色,I-CeuI 识别站点为绿色。请点击此处下载此文件。
| 载体 | 反向引漆 5'-3' | PCR 产品长度 / bp | |
| 1 | PCR_N6_r1: NNNNCTACGTCGCGAATT | 1034 | |
| 2 | PCR_N6_r2: NNNNATATATATATATATATATGGGGGGGGGGGG | 966 | |
| 3 | PCR_N6_r3: NNNNCACTGGGATCTCTTCG | 889 | |
| 4 | PCR_N6_r4: NNNNNGCCGTCGATATATTTTCGT | 821 | |
| 5 | PCR_N6_r5: NNNNNNNATGCAAGTCTTC | 744 | |
| 6 | PCR_N6_r6: NNNNNGTGAATATCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCT | 676 | |
| 7 | PCR_N6_r7: NNNNNCGCGCTCCCTCATAC | 599 | |
| 8 | PCR_N6_r8: NNNNNNTATACGTTTTCGTAC | 531 | |
| 9 | PCR_N6_r9: NNNNNACCGCCGCCCCCGCGG | 454 | |
| 10 | PCR_N6_r10: NNNNNCAGGACATTTTGCCCCACAA | 386 | |
| • 所有载体模板的正向底漆都相同。下划线是 T7 启动子序列。PCR_GN5_f1: 塔塔塔CTCTCTA | |||
表2:载体模板放大的引信。对于所有载波模板,正向底漆都相同。下划线是 T7 启动子序列。PCR_GN5_f1: TAATACGACTCACTATAGNNNNCAGCGTTCTA.使用不同的反向引漆,产生PCR模板,从而产生不同长度的载波RNA。
| 载体 | 长度 | 未封顶/μg | 盖/μg |
| 1 | 1034 | 3.96 | 0.45 |
| 2 | 966 | 8.36 | 0.95 |
| 3 | 889 | 4.4 | 0.5 |
| 4 | 821 | 6.6 | 0.75 |
| 5 | 744 | 4.4 | 0.5 |
| 6 | 676 | 3.08 | 0.35 |
| 7 | 599 | 4.4 | 0.5 |
| 8 | 531 | 3.96 | 0.45 |
| 9 | 454 | 2.64 | 0.3 |
| 10 | 386 | 2.2 | 0.25 |
表3:RNA载体混合物。载波混合物共 49 μg 0.3-1 kbp:未封顶 = 44 μg,上限 = 5 μg。
| 总RNA输入/ng | PCR 周期 |
| 1 纳克 | 18 |
| 2 纳克 | 17 |
| 5 纳克 | 16 |
| 10 纳克 | 15-16 |
| 25 纳克 | 14-15 |
| 50 纳克 | 13-15 |
| 100 纳克 | 12-14 |
表 4:样品总RNA输入依赖性中PCR周期的预期数量。近似周期数基于使用糖精菌、果蝇黑色素和肌肉总RNA进行的实验。
| 总RNA输入/纳克 | 总体映射百分比 | % 唯一映射 | 百分比载波 |
| 1 纳克 | 30 | 20-30 | 30 |
| 2 纳克 | 60 | 20-50 | 10 |
| 5 纳克 | 60-70 | 40-60 | 5-10 |
| 10 纳克 | 60-70 | 40-60 | 5-10 |
| 25 纳克 | 65-80 | 40-70 | 0-5 |
| 50 纳克 | 65-80 | 40-70 | 0-3 |
| 100 纳克 | 70-85 | 40-70 | 0-2 |
表5:预期映射效率和RNA总输入量的依赖程度。根据使用糖精和肌肉总RNA进行的实验,给出了近似数字。
讨论
要成功进行 SLIC-CAGE 库准备,使用低结合提示和管来防止样品吸附导致样品损失至关重要。在涉及检索上清液的所有步骤中,建议恢复整个样本体积。由于该协议具有多个步骤,连续样本丢失将导致库失败。
如果 CAGE (nAnT-iCAGE) 没有正常执行,最好用相同总RNA样本的不同输入量(10 ng、20 ng、50 ng、100 ng、200 ng)测试SLIC-CAGE,并与使用5μg总RNA制备的nAnT-iCAGE库进行比较。如果 nAnT-iCAGE 库不成功(小于每个样本获得的 DNA 库的 0.5-1 ng),SLIC-CAGE 不太可能工作,并且需要将样本损失降至最低。
确保没有未封顶的降解RNA或rRNA的高质量库是关键步骤,第7节所述的封顶。非常重要的是,链球菌素珠子在洗涤缓冲液中彻底悬浮,在继续下一步洗涤步骤或 cDNA 洗脱之前,清除洗涤缓冲液。
如果第一轮载波降级后qPCR的结果显示适配器_f1和载波_f1引能的使用没有区别,仍建议继续使用该协议。如果在第二轮载波降级后,Ct 值的差异小于 5,则建议进行第三轮载波降级。我们从来没有发现第三轮降解是必要的,如果发生,建议取代宿主内分糖种群。
如果获得的库的最终量不足以进行测序,则可在协议中添加其他多轮 PCR 扩增。然后,PCR 扩增可以设置所需的最小扩增周期,以产生足够的材料进行测序,同时考虑到在尺寸选择中无法避免的样品损失。然后,应使用 SPRI 磁珠进行纯化或尺寸选择,直到去除所有小 (<200 bp) 片段(如果需要,使用 0.6:1 珠与采样比),并且库应使用 Picogreen 进行量化。
库可以在单端或成对端模式下排序。使用成对端测序,可以获得有关转录等形的信息。此外,由于使用随机引物(TCT-N 6,N6是随机六项机)进行逆转录,因此,来自测序3'端的信息可用作唯一分子标识符(UMI),以折叠PCR重复项。由于使用适量的PCR扩增周期(最多18个),以前发现使用UMIs是不必要的。
由于协议的核心依赖于nAnT-iCAGE1,SLIC-CAGE使用8个条形码。因此,当前不支持多路复用超过 8 个示例。此外,SLIC-CAGE 和 nAnT-iCAGE 都不适合捕获小于 200 bp 的 RNA,因为协议旨在通过使用 AMPure XP 磁珠排除尺寸来去除链接器和 PCR 人工制品。
SLIC-CAGE 是唯一一种使用总RNA材料的纳米图绘制转录起始位点的无偏低输入单核苷酸分辨率方法。替代方法依赖于逆转录酶的模板切换活性,以条形码封盖RNA,而不是捕获上限(例如,NanoCAGE15和 NanoPARE16)。由于模板切换,这些方法在TSS检测中表现出序列特定的偏差,导致误报TS的数量减少和真TSS9、10的数量减少。
披露声明
可降解载体RNA/DNA的专利已经填补。
致谢
这项工作得到了威尔康信托赠款(106954)的支持,该赠款授予了B.L.和医学研究委员会(MRC)核心基金(MC-A652-5QA10)。N.C.得到EMBO长期研究金的支持(EMBO ALTF 1279-2016);E. P. 得到了英国医学研究理事会的支持;B. L. 得到了英国医学研究理事会(MC UP 1102/1)的支持。
材料
| Name | Company | Catalog Number | Comments |
| 2-propanol, Bioultra, for molecular biology, ≥99.5% | Sigma-Aldrich | 59304-100ML-F | Used in RNAclean XP purification. |
| 3' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 3'linkers is described in the supplementary of this protocol. | ||
| 5' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 5'linkers is described in the supplementary of this protocol. | ||
| Agencourt AMPure XP, 60 mL | Beckman Coulter | A63881 | Purification of DNA |
| Agencourt RNAClean XP Kit | Beckman Coulter | A63987 | Purification of RNA and RNA:cDNA hybrids in CAGE steps. |
| Axygen 0.2 mL Polypropylene PCR Tube Strips and Domed Cap Strips | Axygen (available through Corning) | PCR-0208-CP-C | Or any 8-tube PCR strips (used only for water and mixes). |
| Axygen 1 x 8 strip domed PCR caps | Axygen (available through Corning) | PCR-02CP-C | Caps for PCR plates. |
| Axygen 1.5 mL Maxymum Recovery Snaplock Microcentrifuge Tube | Axygen (available through Corning) | MCT-150-L-C | Low-binding 1.5 mL tubes, used for enzyme mixes or sample concentration. |
| Axygen 96 well no skirt PCR microplate | Axygen (available through Corning) | PCR-96-C | Low-binding PCR plates - have to be used for all steps in the protocol. Note that plates should be cut to contain 2 x 8 wells for easier visibility of the samples |
| Bioanalyzer (or Tapestation): RNA nano and HS DNA kits | Agilent | To determine quality of RNA, efficient size selection and final quality of the library (Tapestation can also be used) | |
| Biotin (Long Arm) Hydrazide | Vector laboratories | SP-1100 | Biotinylation/tagging |
| Cutsmart buffer | NEB | Restriction enzyme buffer | |
| Deep Vent (exo-) DNA Polymerase | NEB | M0259S | Second strand synthesis |
| DNA Ligation Kit, Mighty Mix | Takara | 6023 | Used for 5' and 3'-linker ligation |
| dNTP mix (10 mM each) | ThermoFisher Scientific | 18427013 | dNTP mix for production of carrier templates (or any dNTPs suitable for PCR) |
| Dynabeads M-270 Streptavidin | Invitrogen | 65305 | Cap-trapping. Do not use other beads as these are optimised with the buffers used. |
| DynaMag-2 Magnet | ThermoFisher Scientific | 12321D | Magnetic stand for 1.5 mL tubes - used to prepare Streptavidin beads. |
| DynaMag-96 Side Skirted Magnet | ThermoFisher Scientific | 12027 | Magnetic stand for PCR plates (96 well-plates) - used with cut plates to contain 2 x 8 wells. |
| Ethanol, BioUltra, for molecular biology, ≥99.8% | Sigma-Aldrich | 51976-500ML-F | Used in AMPure washes. Any molecular biology suitable ethanol can be used. |
| Exonuclease I (E. coli) | NEB | M0293S | Leftover primer degradation |
| Gel Loading Dye, Purple (6x), no SDS | NEB | B7025S | agarose gel loading dye |
| HiScribe T7 High Yield RNA Synthesis Kit | New England Biolabs | E2040S | Kit for carrier in vitro transcription |
| Horizontal electrophoresis apparatus | purification of carrier DNA templates from agarose gels | ||
| I-Ceu | NEB | R0699S | Homing endonuclease used for carrier degradation. |
| I-SceI | NEB | R0694S | Homing endonuclease used for carrier degradation. |
| KAPA HiFi HS ReadyMix (2x) | Kapa Biosystems (Supplied by Roche) | KK2601 | PCR mix for target library amplification |
| KAPA SYBR FAST qPCR kit (Universal) 2x | Kapa Biosystems (Supplied by Roche) | KK4600 | qPCR mix to assess degradation efficiency and requiered number of PCR amplification cycles |
| Micropipettes and multichannel micropipettes (0.1-10 µL, 1-20 µL, 20-200 µL) | Gilson | Use of Gilson with the low-binding Sorenson tips is recommended. Other micropippetes might not be compatible. Different brand low-binding tips may not be of equal quality and may increase sample loss. | |
| Microplate reader | For Picogreen concentration measurement of the final library. Microplates are used to allow small volume measurement and reduce sample waste. | ||
| nuclease free water | ThermoFisher Scientific | AM9937 | Or any nuclease (DNase and RNase) free water |
| PCR thermal cycler | incubation steps and PCR amplficication | ||
| Phusion High-Fidelity DNA Polymerase | ThermoFisher Scientific | F530S | DNA polymerase for amplification of carrier templates (or any high fidelity polymerase) |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Purification of carrier PCR templates from agarose gels. |
| qPCR machine | determining PCR amplification cyle number and degree of carrier degradation | ||
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher Scientific | P11495 | Used to measure final library concentration - recommended as, in our hands, it is more accurate and reproducible than Qubit. |
| Quick-Load Purple 100 bp DNA Ladder | NEB | N0551S | DNA ladder |
| Quick-Load Purple 1 kb Plus DNA Ladder | NEB | N0550S | DNA ladder |
| Ribonuclease H | Takara | 2150A | Digestion of RNA after cap-trapping. |
| RNase ONE Ribonuclease | Promega | M4261 | Degradation of single stranded RNA not protected by cDNA. |
| RNase-Free DNase Set | Qiagen | 79254 | Removal of carrier DNA templates after in vitro transcription. |
| RNeasy Mini Kit | Qiagen | 74104 | For cleanup of carrier RNA from in vitro transcription or capping |
| Sodium acetate, 1 M, aq.soln, pH 4.5 RNAse free | VWR | AAJ63669-AK | Or any nuclease (DNase and RNase) free solution |
| Sodium acetate, 1 M, aq.soln, pH 6.0 RNAse free | Or any nuclease (DNase and RNase) free solution | ||
| Sodium periodate | Sigma-Aldrich | 311448-100G | Oxidation of vicinal diols |
| Sorenson low binding aerosol barrier tips, MicroReach Guard, volume range 10 μL, Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719390-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 1,000 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719463-1000EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 20 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719412-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 200 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719447-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| SpeedVac Vacuum Concentrator | concentrating samples in various steps to lower volume | ||
| SuperScript III Reverse Transcriptase | ThermoFisher Scientific | 18080044 | Used for reverse transcription (1st CAGE step) |
| Trehalose/sorbitol solution | Preparation is described in Murata et al. 2014. | ||
| Tris-HCl, 1 M aq.soln, pH 8.5 | 1 M solution, DNase and RNase free | ||
| tRNA (20 mg/mL) | tRNA solution. Preparation is described in Murata et al. 2014. | ||
| UltraPure Low Melting Point Agarose | ThermoFisher Scientific | 16520050 | Or any suitable pure low-melt agarose. |
| USB Shrimp Alkaline Phosphatase (SAP) | Applied Biosystems (Provided by ThermoFisher Scientific) | 78390500UN | |
| USER Enzyme | NEB | M5505S | Degradation of 3'linker's upper strand, Uracil Specific Excision Reagent/Enzyme |
| Vaccinia Capping System | NEB | M2080S | Enzymatic kit for in vitro capping of carrier molecules |
| Wash buffer A | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer B | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer C | Cap trapping washes. Preparation is described in Murata et al. 2014. |
参考文献
- Shiraki, T., et al. Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proceedings of the National Academy of Sciences of the United States of America. 100 (26), 15776-15781 (2003).
- Haberle, V., Lenhard, B. Promoter architectures and developmental gene regulation. Seminars in Cell and Developmental Biology. 57, 11-23 (2016).
- Haberle, V., Stark, A. Eukaryotic core promoters and the functional basis of transcription initiation. Nature Reviews Molecular Cell Biology. 19 (10), 621-637 (2018).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Celniker, S. E., et al. Unlocking the secrets of the genome. Nature. 459 (7249), 927-930 (2009).
- Consortium, F., et al. A promoter-level mammalian expression atlas. Nature. 507 (7493), 462-470 (2014).
- Boyd, M., et al. Characterization of the enhancer and promoter landscape of inflammatory bowel disease from human colon biopsies. Nature Communications. 9 (1), 1661(2018).
- Adiconis, X., et al. Comprehensive comparative analysis of 5'-end RNA-sequencing methods. Nature Methods. , (2018).
- Cvetesic, N., et al. SLIC-CAGE: high-resolution transcription start site mapping using nanogram-levels of total RNA. Genome Research. 28 (12), 1943-1956 (2018).
- Murata, M., et al. Detecting expressed genes using CAGE. Methods in Molecular Biology. 1164, 67-85 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9, 357(2012).
- R Core Team. A language and environment for statistical computing. , Available from: https://www.R-project.org/ (2017).
- Haberle, V., Forrest, A. R., Hayashizaki, Y., Carninci, P., Lenhard, B. CAGEr: precise TSS data retrieval and high-resolution promoterome mining for integrative analyses. Nucleic Acids Research. 43 (8), e51(2015).
- Poulain, S., et al. NanoCAGE: A Method for the Analysis of Coding and Noncoding 5'-Capped Transcriptomes. Methods in Molecular Biology. 1543, 57-109 (2017).
- Schon, M. A., Kellner, M. J., Plotnikova, A., Hofmann, F., Nodine, M. D. NanoPARE: parallel analysis of RNA 5' ends from low-input RNA. Genome Research. 28 (12), 1931-1942 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。