Method Article
超低入力キャリア-CAGEを使用した転写開始サイトマッピング
要約
遺伝子発現のキャップ解析(CAGE)は、単一ヌクレオチド分解能でRNAポリメラーゼII転写開始部位を捕捉するためにmRNA5'端部のゲノム全体の定量マッピングのための方法である。本研究では、総RNAのナノグラム量を用いて高品質なライブラリを生成するための低入力(SLIC-CAGE)プロトコルについて述べている。
要約
遺伝子発現のキャップ解析(CAGE)は、RNAポリメラーゼII転写開始部位(TSS)の単一ヌクレオチド分解能検出に用いられる方法である。TSSの正確な検出は、コアプロモーターの同定と発見を強化します。さらに、活性エンハンサーは、双方向転写開始のシグネチャを介して検出することができる。ここでは、超低入力キャリアケージ(SLIC-CAGE)を実行するためのプロトコルです。CAGEプロトコルのこのSLIC適応は、目的のサンプルに追加されたインビトロ転写RNAキャリアミックスの使用を通じてRNA量を人工的に増加させることによりRNA損失を最小限に抑え、合計ナノグラム量からのライブラリー調製を可能にします。RNA(すなわち、何千もの細胞)。キャリアは、予想されるDNAライブラリー断片長分布を模倣し、それによって均質なキャリアの豊富さによって引き起こされる可能性のあるバイアスを排除する。プロトコルの最後の段階では、ホーミングエンドヌクレアーゼでキャリアが分解され、ターゲットライブラリが増幅されます。標的サンプルライブラリーは、ホーミングエンドヌクレアーゼ認識部位が長く(18~27bpの間)、真核ゲノム中に存在する確率が非常に低いため、劣化から保護されています。最終的な結果は、次世代シーケンシングの準備ができているDNAライブラリです。プロトコル内のすべてのステップは、シーケンスまで、6日以内に完了できます。キャリアの準備は、完全な営業日を必要とします。しかし、それは大量に調製することができ、-80 °Cで凍結保存することができます。配列決定後、読み取りを処理してゲノム全体の単一ヌクレオチド分解能TSSsを得ることができます。プロモーターに集約されると、データは 5'中心の式プロファイリングにも使用できます。
概要
遺伝子発現のキャップ解析(CAGE)は、RNAポリメラーゼII転写開始部位(TSSS)1の単一ヌクレオチド分解能ゲノムワイドマッピングに用いられる方法である。その定量的な性質により、5'エンド中心の式プロファイリングも可能になります。TSSを取り巻く領域(上流および下流約40bp)はコアプロモーターであり、RNAポリメラーゼIIと一般的な転写因子が結合する物理的位置を表す(前述の2、3を検討)。TSSの正確な位置に関する情報は、コアプロモーターの発見およびプロモーターダイナミクスの監視に使用できます。さらに、アクティブエンハンサーは双方向転写のシグネチャを示すので、CAGEデータはエンハンサーダイナミクス4のエンハンサー検出およびモニタリングにも使用できます。CAGEの方法論は、最近、ENCODE 5、modENCODE 6、およびFANTOMプロジェクト7などの注目の研究プロジェクトでの幅広い応用と使用のために人気が高まっています。さらに、TSS情報はまた、疾患特異的TSSが診断目的8に使用することができるので、健康な組織および疾患組織を区別するために重要であることが証明されている。
TSSマッピングのためのいくつかの方法(CAGE、RAMPAGE、STRT、ナノケージ、ナノケージ-XL、オリゴキャッピング)が利用可能ですが、私たちと他の人は最近、CAGEが偽陽性の数が最も少ない真のTSSをキャプチャする最も公平な方法であることを示しました9,10.最近のCAGEプロトコルnAnT-iCAGE11は、制限酵素を使用してフラグメントを短いタグに切断することを回避し、PCR増幅を使用しないため、TSSプロファイリングのための最も公平なプロトコルです。nAnT-iCAGEプロトコルの制限は、大量の出発物質(例えば、各サンプルに対する総RNAの5μg)の要件である。特定の、生物学的に関連する質問に答えるために、多くの場合、そのような大量の出発物質(例えば、FACS選別細胞または初期胚期)を得ることは不可能である。最後に、nAnT-iCAGEが成功した場合、各サンプルから入手できるDNAライブラリー材料は1~2ngのみで、達成可能なシーケンシング深さを制限します。
総RNAのナノグラムのみを用いてTSSプロファイリングを可能にするために、最近、超低入力キャリアCAGE 10(SLIC-CAGE、図1)を開発しました。SLIC-CAGEは、高複雑性ライブラリを得るために合計RNAのわずか10 ngしか必要としならない。当社のプロトコルは、目的のRNAに追加された慎重に設計された合成RNAキャリアに依存し、合計5μgのRNA材料を達成します。合成キャリアは、過剰に均質な分子によって引き起こされる可能性のある潜在的なバイアスを回避するために、長さの分布の標的DNAライブラリーを模倣します。担体の配列は、2つの理由から、大腸菌ロイチルtRNA合成酵素遺伝子(表1)の配列に基づいている。第一に、最終ライブラリー内のキャリアの残りは、たとえ配列されたとしても、真核生物ゲノムにマッピングされない。第二に、大腸菌は中親和種であるため、そのハウスキーピング遺伝子はSLIC-CAGEに適した温度範囲に最適化されています。キャリア配列はまた、キャリアRNA分子に由来するDNAの特異的な分解を可能にするためにホーミングエンドヌクレアーゼ認識部位と組み込まれています。ホーミングエンドヌクレアーゼ認識部位が長いため、標的、サンプル由来ライブラリーはそのまま残ります(I-CeuI = 27 bp;I-SceI = 18 bp)および真核生物ゲノムに統計的に見られる可能性は低い。キャリアの特定の劣化とサイズ除外によるフラグメントの除去後、ターゲットライブラリはPCR増幅され、次世代シーケンシングの準備が整います。開始RNA量(1-100 ng)に応じて、13〜18 PCR増幅サイクルが必要とされる。各サンプルあたりのDNAの最終的な量は5〜50 ngの間の範囲で、非常に深いシーケンシングのための十分な材料を得る。総RNAの1-2 ngのみを使用する場合、真のTSSを検出することができます。ただし、ライブラリの複雑さは低くなるものが予想されます。最後に、SLIC-CAGEはnAnT-iCAGEプロトコル11に基づいているため、シーケンス前に最大8つのサンプルの多重化が可能です。
プロトコル
1. キャリアの準備
-
インビトロ転写のためのDNAテンプレートの調製
- 水の41 μL、5x HFバッファーの20 μL、2.5 mM dNTPの8 μL、10 μLの10 μLの10 μLのユニークなフォワードプライマー(PCR_GN5_f1、表2;プライマーを水に溶解し、希釈する)を組み合わせて、各PCRテンプレートのPCRミックスを準備します。、合成担体遺伝子および1μLプシオンポリメラーゼを含有する2ng/μLテンプレートプラスミドの10μL。ピペッティングでPCRミックスを混ぜます。すべての10のテンプレートのためのマスターミックスは、一度に準備することができます(11の反応のために準備)。
- 各10 μM リバースプライマーの 10 μL に PCR ミックスの 90 μL を追加します (PCR_N6_r1-r10,表2)。ピペッティングで混ぜます。
- PCRは、次のプログラムを使用してテンプレートを増幅します:60sのための98 °C、(10sのための98 °C、30sのための50 °C、30sのための72 °C)35サイクル、10分間の72 °C、4 °Cで保持します。
-
PCR増幅DNAテンプレートのゲル精製
- 1%のアガロースゲルを準備します(低溶融アガロースが推奨されます)。
- 体積を下げるには、低中温(30~40°C)で真空濃縮器を使用して、PCR反応混合物を100μLから20μLの総体積に濃縮します。
- 6倍の荷重染料の6 μLを加え、よく混ぜ、ゲルに負荷を加えます。使用済みの電気泳動タンク(5~10V/cm)に適した電圧で、1x TAEバッファで30分間電気泳動を実行します。並行して100 bpまたは1,000 bp DNAはしごを実行します。
- きれいなメスを使用して、ターゲットPCR製品を含むゲルスライスを切除する。余分なアガロースゲルを避けてください。(製造元の指示に従って)ゲル抽出キットを使用してPCR製品を浄化します。
注:アガロースゲルから単離されたDNAのA260/A230比は、通常低い(0.1~0.3)。期待される対象製品および副産物を図 2Aに示します。100 μL PCR反応からの期待降伏は1.2~3 μgです。
-
キャリア分子のインビトロ転写
- 製造業者の指示に従ってT7 RNAポリメラーゼを用いてインビトロでキャリアRNAを転写する。10~20 μLの反応を設定します(推奨キットは材料の表にあります)。
- RNA精製キットを使用してインビトロ転写RNAを精製します。DNase Iを使用して溶液中にDNA消化を設定し、メーカーの標準的な指示に従い、50μLの水でRNAを溶出します。溶出収率を上げるには、遠心分離の前に5分間カラムに水を残します。
注:カラムの最大結合容量を超えないように注意してください(材料の表に記載されているキットでは、容量は最大100 μgです)。PCRテンプレート1~10(長さ1kbp~200bp)からの期待収量は、インビトロ転写反応で10μLから25~50μgです。反応は、キャリア分子のより大きなストックを得るためにスケールアップすることができます。
-
インビトロ転写キャリアRNA分子のキャッピング
- キャッピングミックスを10倍キャッピングバッファーの2μL、10mM GTPの1μL、2mM SAMの1μL(新規希釈)、およびキャリアRNA当たりワクシナキャッピング酵素の1μLを組み合わせてキャッピングミックスを調製します。
- 各キャリア分子の最大10 μgを総体積の15μLに混合し、65°Cで10分間変性する。二次構造の形成を防ぐために、すぐに氷の上に置きます。
- 変性キャリアRNAをキャッピングミックスの5μLと混合し、37°Cで1時間インキュベートします。
- RNA精製キットを使用してキャップされたRNA分子を精製する - 製造元のクリーンアッププロトコルに従ってください。水の30 μLで溶出RNA。溶出収率を上げるには、遠心分離の前に5分間カラムに水を残します。
注:マイクロボリューム分光光度計を用いて濃度を測定します。期待される A260/A280 の比率は >2、A260/A230 は >2 です。一部のRNAサンプルでは、A260/A230が1.3~2の間であってもよいことに注意してください。上限なしRNAの10 μgを使用した場合の期待収率は、キャップされたRNAの9~10 μgです。
- 表 3に記載されている金額を組み合わせて、上限と上限なしのキャリアの組み合わせを準備します。チューブをフリックしてよく混ぜ、マイクロボリューム分光光度計を使用して濃度を測定します。
注:キャリアのより高い濃度が逆転写反応に収まるように必要な場合(下記参照)、キャリアミックスは、所望の最終濃度に達するまで低中温(30〜35°C)で真空濃縮器を使用して濃縮することができる。ステップ2-14は、村田ら11によって報告された標準的なnAnT-iCAGEプロトコルから変更される
2. 逆転写
- RTプライマーの1 μL(水に溶解した2.5mM TCT-N6、配列については補足表1を参照)、目的の総RNAの10ng、および低結合PCRプレート内の総体積の10μLのキャリアミックス(表3)の4,990ngngを組み合わせます。チューブをフリックして混ぜます。
注:サンプルRNAが逆転転写のために希釈しすぎる場合(下記参照)、それをキャリアの適切な量と組み合わせ、真空濃縮器を使用して9μL総容積に濃縮し、RTプライマーの1 μLを追加する。キャリアアールを添加し、合計で5μgのRNAに達すると、サンプル損失を防ぎます。 - ステップ2.1から65°Cで5分間加熱し、すぐに氷の上に置き、発色を防ぎます。
-
逆転写(RT)ミックスを調記する。
- 各サンプルは、水の6.1 μL(RNase-およびDNaseフリー)、5xファーストストランドバッファの7.6 μL、0.1 M DTTの1.9 μL、10mM dNtPの1μL、トレハロース/ソルビトールミックスの7.6 μL(ムラタら11のレシピを参照)、および3.8μLを組み合わせたもの(ムラタら11のレシピを参照)。6>材料のテーブル)チューブをフリックしてよく混ぜます。
- RNA、キャリア、RTプライマーの10 μL(総体積38μL)を持つPCRチューブにRTミックスの28 μLを追加します。ピペッティングでよく混ぜます。
注:トレハロース/ソルビトールによるミックスは粘性が高い。目に見えて均質になるまで混ぜます。 - 次のプログラムを使用して熱サイクラーでインキュベート:30sのための25 °C、60分間50 °C、4 °Cで保持します。
-
CDNAの精製:SPRI磁気ビーズを用いたRNAハイブリッド
- 推奨されるRNAseおよびDNaseフリーSPRIビーズの68.4 μLをRTミックスの38 μLに加えます(ビーズ対サンプル比1.8:1)。ピペッティングでよく混ぜ、室温(RT)で5分間インキュベートします。
- 5分間磁気スタンドのビーズを分離し、上清を捨て、70%エタノールの200 μLでビーズを2回洗います(作りたて)。
注:エタノールは混合せずにビーズに添加され、チューブが磁気スタンド上にある間。添加したエタノールは直ちに除去される。それはサンプル損失につながる可能性があるため、洗い出し中にビーズを失わないように注意する必要があります。 - チューブがまだ磁気スタンドにある間、エタノールの痕跡をすべて取り除きます。エタノールの液滴は、P10ピペットを使用してチューブから除去し、押し出すことができます。ビーズを乾燥させないようにしてください。
- ビーズに37°Cで予熱した水の42 μLをビーズに加え、60倍上下にピペッティングしてサンプルを溶出させます。
注:発泡体中のビーズ(結合サンプル)の損失を引き起こす可能性があるため、ピペッティングによって発泡を引き起こさないように注意してください。 - 蓋なしで37°Cで5分間インキュベートし、微量のエタノールを蒸発させてください。
- 磁気スタンドのビーズを5分間分離し、上清を新しいプレートに移します。
注:ビーズの持ち越しを避けながら、サンプルの損失を防ぐためにすべての上清を取得してみてください。P10ピペットを使用して、最後のサンプル液滴を取得します。
3. 酸化
- 精製されたRT反応に1 M NaOAc(pH 4.5)の2 μLを加えます。ピペッティングで混合し、250 mM NaIO4の2 μLを追加し、再び混ぜます。
- 氷の上で45分間インキュベートし、光を避けるためにアルミホイルでプレートを覆います。
- 酸化ミックスに16μLのトリス-HCl(pH 8.5)を加えてpHを中和します。
- 酸化cDNA:SPRI磁気ビーズを用いてRNAハイブリッドを精製する。酸化ミックスの60 μLにSPRIビーズの108 μLを加えます(サンプル比に1.8:1ビーズ)。手順 2.6.1 ~ 2.6.6 に記載されているように、精製を繰り返します。42μLの水を用いて37°Cに予熱した溶出量。
注:NaIO4の1mg当たり18.7μLの水を加えて250mM NaIO4を作り出します。NaIO4は光に敏感です。したがって、溶液はアルミ箔で覆われたチューブまたは耐光管に保管してください。
4. バイオチニル化
- 精製酸化試料を含むチューブに1M NaOAc(pH 6.0)の4 μLを加え、ピペッティングで混合します。
- 10mMビオチン溶液の4 μLを加え、ピペッティングで混合し、光を避けるために熱サイクラーで23°Cで2時間インキュベートします。
注:50mgのビオチンを13.5mLのDMSOと混合してビオチン溶液を調作する。単一使用のアリコートを作り、-80 °Cで凍結します。 - SPRI磁気ビーズを用いてバイオチン化cDNA:RNAハイブリッドを精製する。2プロパノールの12 μLを追加し、ピペッティングで混合します。SPRIビーズの108 μL(サンプル比に1.8:1ビーズ)を追加し、手順2.6.1-2.6.6に記載されているように精製を繰り返します。42μLの水を37°Cで予熱した水を用いて溶出する。
注:プロトコルはここで一時停止することができ、サンプルは-80 °Cで凍結します。
5. RNase I ディディステリオン
- 10x RNase Iバッファーの4.5 μLを各サンプルあたり0.5 μLのRNase I(10 U/μL)と混合してRNase I混合を調調します。ピペッティングで混ぜます。
- 各精製サンプルにミックスの5 μLを加えます(合計で45μL)。ピペッティングで混合し、37°Cで30分間インキュベートします。
6. ストレプトアビジンビーズの調製
- 各サンプルについて、ストレプトアビジンビーズスラリーの30μLを20mg/mL tRNAの0.38 μLと混合します。氷の上で30分間インキュベートし、チューブをフリックして5分ごとに混ぜます。
注:ボトルをフリックしてピペッティングする前に、ストレプトアビジンビーズスラリーを再中断します。tRNA溶液は、村田ら11に従って調製する必要がある。 - 磁気スタンドのビーズを2~3分間分離し、上清を取り除きます。
- 15 μLのバッファーAでビーズを洗い直し、磁気スタンドのビーズを2~3分間分離し、上清を取り除きます。洗浄を繰り返し、上清を取り除きます。
- ビーズをバッファーAの105μLで再中断し、tRNAの20mg/mLの0.19 μLを加える。ピペッティングでよく混ぜます。
注:ビーズは、使用する前に新鮮に準備する必要があります。RNase I消化中にビーズの調製を開始します。複数のサンプルのために単一の管で一緒にビーズを準備する。
7. キャップトラップ
-
サンプルバインディング
- RNase I処理試料の45μLに、調製されたストレプトアビジンビーズの105μLを添加する。ピペッティングでよく混ぜ、37°Cで30分間インキュベートし、10分ごとにピペッティングしてミックスします。
- 磁気スタンドのビーズを2~3分間分離し、上清を取り除きます。
-
ビーズの洗浄
- 150 μLの洗浄バッファーAを追加し、ピペッティングでビーズを再サスペンドします。磁気スタンドのビーズを2~3分間分離し、上清を取り除きます。
- 洗浄バッファーBの150 μLを追加し、ピペッティングによってビーズを再中断します。磁気スタンドのビーズを2~3分間分離し、上清を取り除きます。
- 洗浄バッファーCの150 μLを追加し、ピペッティングによってビーズを再中断します。磁気スタンドのビーズを2~3分間分離し、上清を取り除きます。
注:バッファBとCは37°Cに予熱する必要があります。洗浄バッファーA、B、Cのレシピは、村田ら11に記載されている。
-
cDNAリリース
- 58.5 μL の水と 10x RNase I バッファーの 6.5 μL を混合して、1x RNase I バッファーを準備します。
- 1x RNase Iバッファの35 μLでビーズを再中断します。95°Cで5分間インキュベートし、cDNAの再結結を防ぐために2分間氷の上に直接移します。圧力が高まるため飛び出す可能性があるため、氷への移動中に蓋を保持します。
- 磁気スタンドで2~3分間ビーズを分離し、上清を新しいプレートに移します。
- 1x RNase Iバッファの30 μLでビーズを再中断します。磁気スタンドのビーズを2~3分間分離し、上清を以前に採取した上清に移します(eluted cDNAの総体積は約65μLでなければなりません)。
8. RNase HおよびRNase I消化によるRNA除去
- サンプルごとに、水の2.4 μL、10x RNase Iバッファーの0.5 μL、RNase Hの0.1 μL、およびRNase Iの2 μLを組み合わせます。
- 放出されたcDNAサンプルの65 μLにミックスの5 μLを加え、ピペッティングで混合します。37°Cで15分間インキュベートし、4°Cで保持します。
- SPRI磁気ビーズを用いてRNase消化ミックスからcDNAを精製します。70μLの劣化反応に126μLのSPRIビーズを加え、ピペッティングで混合します。2.6.1-2.6.6のSPRIビーズ精製について説明されているように精製手順に従ってください。記載されているように37°Cで予熱した水の42μLを用いて溶出する。
- 10x RNase Iバッファの4.5 μLとRNase Iの0.5 μLを組み合わせてRNase Iミックスを準備します。
- 精製されたcDNAサンプルの40 μLにRNaseミックスの5 μLを加えます。ピペッティングで混合し、37 °Cでインキュベートし、30分間4°Cで保持します。
- SPRI磁気ビーズを使用してサンプルを浄化します。45μLの分解反応に81μLのSPRIビーズを加え、ピペッティングにより混合します。2.6.1-2.6.6のSPRIビーズ精製について説明されているように精製手順に従ってください。記載されているように42μLの水を用いて溶出する。
9. 5' リンカーのライゲーション
- 真空濃縮器を使用して精製されたcDNAサンプルを4μLに濃縮します。温度は30~35°Cに保ちます。ピペットを使用してボリュームをテストします。試料が乾燥して完全性を発した場合は、4μLの水を加えて溶解する。
注:サンプルの損失を防ぐために完全性に乾燥を避ける方が良いです。 - 濃縮サンプルを95°Cで5分間インキュベートし、直ちに氷の上に2分間置き、発光を防ぎます。圧力の蓄積により蓋が飛び出す可能性があるため、チューブを移しながら蓋を保持します。
- 2.5 μM 5'リンカーの4 μLを5分間5分間5分間インキュベートし、直ちに2分間氷の上に置き、発色を防ぎます。
- 2.5 μM 5' リンカーの 4 μL をサンプルの 4 μL と混合します。
注:5' リンカーは、補足表2、補足表3、補足表4、および補足表 5に従って準備する必要があります。10 μM 5' リンカーを使用前に 100 mM NaCl を使用して 2.5 μM 濃度に希釈します。 - 混合5'リンカーとサンプルにライゲーションプリミックスの16 μL(材料の表を参照)を追加し、ピペッティングによってよく混合します。16 °Cで16時間インキュベートします。
- SPRI磁気ビーズを使用してライゲーションミックスを浄化します。SPRI ビーズの 43.2 μL を追加し、手順 2.6.1 ~ 2.6.6 に従います。42μLの水を用いて説明したように溶出する予熱は37°Cで予熱した。
- 転移した上清(1.8:1ビーズをサンプル比)に72μLのSPRIビーズを加えて、ステップ9.6で精製を繰り返します。
注:5'リンカーには、シーケンシング前に最大8つのサンプルのプールを可能にするバーコードが含まれています(ムラタら11および補足表1に記載されているように、8つのトリヌクレオチドバーコードが利用可能です)。
10. 3' リンカーのライゲーション
- ステップ9.1で説明するように真空濃縮器を使用して、精製されたサンプルを4μLに濃縮します。
- 濃縮サンプルを95°Cで5分間インキュベートし、直ちに氷の上に2分間置き、発光を防ぎます。圧力の蓄積のためにふたが飛び出す可能性があるため、チューブを移しながら蓋を保持します。
- 2.5 μM 3' リンカーの 4 μL を 65 °C で 5 分間インキュベートし、直ちに 2 分間氷の上に置き、発色を防ぎます。
- 濃縮サンプルの4μLに2.5 μM 3'リンカーの4 μLを追加します。
- ライゲーションプレミックスの16 μLを追加し、ピペッティングによってよく混ぜます。16 °Cで16時間インキュベートします。
- SPRI磁気ビーズを使用してライゲーションミックスを浄化します。SPRI ビーズの 43.2 μL を追加し、手順 2.6.1 ~ 2.6.6 に従います。42μLの水を用いて説明したように溶出する予熱水を37°Cに予熱した。
注:3' リンカーは、補足表 6および補足表 7に従って準備する必要があります。10μM 3'リンカーを100mM NaClを使用して2.5 μM濃度に希釈します。
11. 脱リン酸化
- 4 μL の水、10 倍の SAP バッファーの 5 μL、および SAP 酵素の 1 μL を組み合わせて SAP ミックスを準備します。
- 精製されたライゲーションサンプル(総体積50μL)にSAPミックスの10 μLを加え、サーモサイクラーでインキュベートします:30分間37°C、15分間65°C、4°Cで保持します。
12. ウラシル特異的切除酵素を用いた3'リンカー上鎖の劣化
- 脱リン酸化サンプルに2μLのウラシル特異的切除酵素(材料表参照)を加え、次のプログラムを使用してサーモサイクラーでピペッティングしてインキュベートして混合する:30分間37°C、5分間95°C、すぐに氷の上に2分間置く断片化した上鎖の再消し止めを防ぎます。
- 52 μl混合物にSPRI磁気ビーズの93.6 μLを加えて反応混合物を浄化し、ピペッティングによってよく混合します。精製手順を繰り返す 2.6.1 ~ 2.6.6.記載されているように37°Cで予熱した水の42 μLを有する溶出する。
13. 第2鎖合成
- 10x DNAポリメラーゼ反応バッファーの5 μL、水の2 μL、10mM dNTPの1 μL、50 μM nAnT-iCAGE第2ストランドプライマーの1μL(配列は補足表1)と1μLのDを組み合わせて、第2鎖合成ミックス(サンプル当たりの容積が発現)を調製するNAエキノールクレアーゼ欠損ポリメラーゼ(材料表の推奨ポリメラーゼを参照)。
- 精製サンプルにミックスの10 μLを追加し、ピペッティングによってよく混合します(総体積は50μLです)。次のプログラムを使用して熱サイクラーでインキュベート:5分間95°C、5分間55°C、30分間72°C、4°Cで保持します。
14. エキノクレアーゼIを用いた第2鎖合成プライマーの劣化
- 第2鎖合成混合物にエキノルクレアーゼIの1μLを添加する。ピペッティングでよく混ぜ、37°Cで30分間インキュベートし、その後4°Cで保持します。
- エキノクレアーゼI処理試料の51μLにSPRI磁気ビーズの91.8 μLを添加して二重鎖DNAを精製します。2.6.1~2.6.6に記載の精製手順を繰り返します。42 μLの水を予熱して37°Cに溶出する。
- ステップ9.1で説明するように、真空濃縮器を使用してサンプルを15 μLに濃縮します。
15. 品質と数量管理
- 濃縮されたサンプルの1 μLを使用し、DNA品質分析装置で高感度DNAチップを実行します。期待されるプロファイル/数量を図 3に示します。
16. キャリア劣化の第1ラウンド
- 水の2μL、10倍制限酵素バッファーの2μL、I-SceIの1μL、およびI-CeuIの1μLを組み合わせて分解ミックスを調製する。
- 濃縮試料の14μLに分解ミックスの6μLを加え、ピペッティングにより混合します。37°Cで3時間インキュベートし、その後65°Cで20分の不活性化を行い、4°Cで保持します。
- SPRI磁気ビーズを使用して分解ミックスを浄化します。5 μLの水を加えて分解ミックスの体積を増やし、SPRIビーズの45μLを加えます(サンプル比に1.8:1ビーズ)。手順 2.6.1 ~ 2.6.6 に記載されているように、精製を繰り返します。42 μLの水を37°Cに予熱して溶出します。
- 工程9.1で説明したように、総体積の42μLから20 μLまで、エラ出したサンプルを濃縮します。
17. 劣化レベルの制御とPCR増幅サイクル数の決定
- ライブラリ全体を増幅するための qPCR ミックス (アダプター ミックス) を準備します。水の3.8 μL、qPCRプレミックスの5 μL(2x)、10 μMアダプター_f1プライマーの0.1 μL(5'-AATGATACGCGACCACCGA-3')、および10 μMアダプター_r1プライマーの0.1 μL(推奨される各qRの推奨材料)を組み合わせます。
- ステップ16.4からサンプルの1 μLとqPCRアダプタミックスの9 μLを組み合わせ、ピペッティングによってよく混合します。
- キャリア(キャリアミックス)に由来するDNAを増幅するためのqPCRミックスを調調します。3.8 μL の水、5 μL の qPCR プレミックス (2x)、10 μM キャリア_f1 プライマーの 0.1 μl (5'-GCGGCAGTGTTCGCTATAACACACACACAC-3')、および各サンプルに対して 10 μM アダプター_r1 プライマーの 0.1 μL を組み合わせます。
- 工程16.4からサンプルの1 μLとqPCRキャリアミックスの9 μLを組み合わせ、ピペッティングによってよく混合します。
- qPCR プログラムを設定する: 95 °C 3 分間 (20 s の場合は 95 °C、20 s では 60 °C、2 分間は 72 °C) を 40 倍繰り返し、続いて計器特異的な脱退曲線 (65~ 95 °C) を繰り返し、4 °C で保持します。
注:サンプルを水に置き換えて、負のコントロールを準備します。
18. ターゲットライブラリのPCR増幅
- 6 μL の水、10 μM アダプター_f1 プライマーの 0.5 μL、10 μM アダプター_r1 プライマーの 0.5 μL、および 25 μL の PCR プレミックス (2 倍) を組み合わせて PCR 増幅ミックスを調作成します。ピペッティングでミックスします(推奨PCRプレミックスの材料表を参照)。
- ステップ16.4からサンプルの18 μLにPCRミックスの32 μLを追加します。ピペッティングで十分に混ぜます。
- PCR増幅を設定する:95 °Cを3分間、(20sで98°C、15sで60°C、2分間で72°C)12~18サイクル、72°Cを2分間、72°Cを2分間、4°Cで保持します。
注:PCRサイクルの正確な数はqPCR結果によって決定され、アダプタープライマーミックスで得られたCt値に対応します(PCRサイクルの数はCt値と等しい)。 - 増幅されたサンプルの50μLにSPRI磁気ビーズの90 μLを加えて増幅したサンプルを精製し、ピペッティングで十分に混合します。手順 2.6.1 ~ 2.6.6 で説明する精製手順を繰り返します。記載されているように42 μLの水を使用してサンプルを溶出します。
19. キャリア劣化の第2ラウンド
- 手順 16.1 ~ 16.3 を繰り返します。
- SPRI磁気ビーズを使用して分解ミックスを浄化します。サンプルに10 μLの水を加えて体積を増やし、SPRIビーズの30μL(サンプル比に対して1:1ビーズ)と混合します。手順 2.6.1 ~ 2.6.6 に記載されているように、精製を繰り返します。記載されているように37°Cで予熱した水の42 μLで溶出する。
- 総体積の42μLから30μLまで、エラ出したサンプルを濃縮します。
20. ライブラリサイズの選択
- ステップ19.3からサンプルの30 μLとSPRI磁気ビーズの24 μLを混合します。(0.8:1 ビーズとサンプル比)。手順 2.6.1 - 2.6.6 に記載されているように、精製手順を繰り返します。記載されているように42 μLの水でサンプルを溶出します。
- ステップ 9.1 で説明するように、サンプルを約 14 μL に濃縮します。
21. 品質管理
-
サイズ分布の評価
- 高感度DNAチップ上でサンプルの1 μLを実行します。期待される結果は図 4に示されています。
注:200 bp より短いフラグメントが表示されている場合 (図 4A、Cの例を参照)、短いフラグメントが削除されるまでサイズ選択 (手順 20.1 ~ 20.2) を繰り返す必要があります (図 4B、D)。通常、サイズの選択の 1 つの追加のラウンドで十分です。短いフラグメントの量が厳しい場合 (図 4Cのように)、ビーズとサンプルの比率を 0.6:1 に減らす必要があります。
- 高感度DNAチップ上でサンプルの1 μLを実行します。期待される結果は図 4に示されています。
-
キャリア劣化品質管理
- 手順 17.1 ~ 17.5 を繰り返します。
注:HS DNA チップラン(領域解析)で推定されるライブラリの濃度に応じて、サンプルはqPCRの前に希釈する必要があります。サンプルの損失を避け、水中で100~500倍希釈するためにサンプルの0.5 μLを使用します(希釈して1-20 pg/μL最終濃度)。アダプターとキャリアミックスで得られるCt値の予想差は5~10です。
- 手順 17.1 ~ 17.5 を繰り返します。
-
ライブラリの定量
- 1x TE の 980 μL と 100 mg/mL ラムダ DNA 標準の 20 μL を混合することにより、ラムダ DNA 標準の作業希釈を調用します (DNA 定量キットに提供される 20x TE を希釈して調記します)。ラムダDNAの希釈は-20°Cで保存することができる。
- 補足表8に従って希釈されたラムダ標準および1x TEを混合してラムダDNA標準シリアル希釈を調出す。
注:精度を高めるために、すべてのチューブに1x TEバッファーの100 μLを追加し、希釈されたラムダの体積ごとに必要に応じて1x TEボリュームを除去することをお勧めします。ライブラリの 1 μL を超える値を使用しないでください。この測定のための384の井戸版の使用は推薦される。
結果
本報告では、開始総RNA材料のナノグラムからシーケンシング対応ライブラリを取得するための完全なSLIC-CAGEプロトコルについて説明します(図1)。合成RNAキャリアミックスを得るためには、まず、PCR担体を調製し、PCR副産物を排除するためにゲル精製する必要がある(図2A)。各PCRテンプレート(合計10個)は、共通のフォワードを使用して生成されますが、異なるリバースプライマー(表2)を使用して、合成RNAキャリアのサイズ変動を可能にするためにPCRテンプレートの長さが異なります。精製されると、PCRテンプレートは、キャリア分子のインビトロ転写に使用される。テンプレートがゲル精製された場合、単一のRNAキャリア産物が期待されます(図2Bの代表的なゲル分析を参照)。キャリアの調製は、必要性に応じてアップスケールすることができ、準備すると、将来の使用のために-80 °Cで混合し、凍結します。
推奨されるサンプル総RNA(10ng)の推奨最小量と16-18サイクルのPCR増幅を組み合わせることで、高い複雑性SLIC-CAGEライブラリを実現できます。最終的なライブラリーを増幅するために必要なPCRサイクルの数は、使用される入力RNAの総数に大きく依存します(予想サイクル数は表4に示されています)。
劣化の最初のラウンドの後、qPCR結果(ステップ17)では、アダプター_f1またはcarrier_f1プライマーを使用して得られたCt値の予想差は1-2であり、アダプター_f1で得られたCt値はcarrier_f1よりも低い。
最終ライブラリにおけるフラグメント長の分布は200~2,000 bpで、平均フラグメントサイズは700~900bpです(Bioanalyzerソフトウェアを使用した領域解析に基づく図4B、D)。図4A、Cに示すように、短いフラグメントは、サイズ除外の追加ラウンドによって除去されなければならない(ステップ20〜21)。これらの短いフラグメントは、対象ライブラリではなく、PCR増幅アーティファクトです。フラグメントが短いほど、シーケンス フロー セルのクラスタが向上し、シーケンスの問題が発生する可能性があることに注意してください。
サンプル当たり得られるライブラリー材料の期待量は5〜50ngの間である。有意に低い量は、プロトコル中のサンプル損失を示します。取得した低い量がシーケンスに十分である場合 (プールされたライブラリの 2 ~ 3 ng が必要)、ライブラリの複雑さが低くなる可能性があります (下記参照)。
シーケンスマシンによっては、フローセルにロードされるライブラリの量を最適化する必要がある場合があります。Illumina HiSeq 2500 を使用して、8-12 pM SLIC-CAGE ライブラリを読み込むと、平均で 150 ~ 2 億の読み取りが得られますが、読み取りの 80% が品質スコア Q30 をしきい値として渡します。
得られた読み取りは、参照ゲノムにマッピングされます [50 bp 読み取り、Bowtie212は、シード シーケンスごとのミスマッチをゼロにするデフォルト パラメータ (22 bp)] で使用できます。予想されるマッピング効率は、RNA 入力の合計量に依存し、表 5に示します。一意にマップされた読み取りは、R グラフィカルおよび統計計算環境13にロードし、CAGEr (生体伝導体パッケージ14)を使用して処理できます。パッケージビネットは簡単に追従でき、マッピングされたデータのワークフローと処理を詳細に説明します。ライブラリの複雑さの容易な視覚的制御は、低複雑性ライブラリが人工的に狭いプロモーターを持つため、プロモーター幅の分布です(図5A、SLIC-CAGEライブラリは、総RNAの1 ngから派生した、詳細については、前を参照してください)パブリケーション 10)。ただし、複雑度の低い SLIC-CAGE ライブラリでも、低/中入力 TSS マッピングの代替方法よりも高い精度で真の CTSS を識別できます (図 5B、C)。
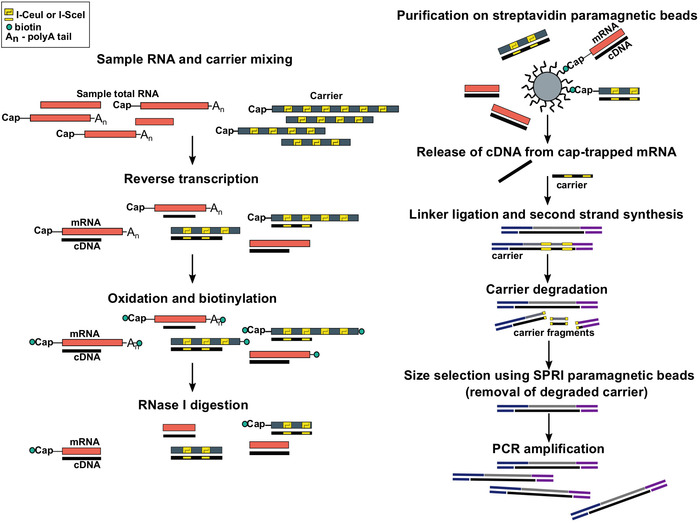
図 1:SLIC-CAGE プロトコルの手順。サンプルRNAは、全RNA材料の5μgを達成するためにRNAキャリアミックスと混合される。cDNAは、逆転写を介して合成され、キャップは、ナトリウム周期を使用して酸化されます。酸化は、ビオチンヒドラジドを使用してキャップにビオチンの添付を可能にします。ビオチンは、周期ナトリウムを使用して酸化されるので、mRNAの3′末端に付着します。mRNA:cDNAを不完全に合成したcDNAとmRNAの3′末端からビオチンを排除するために、サンプルはmRNAの5′末端に達したRNase I.cDNAで処理され、次にストレプトアビジン磁気ビーズの親和性精製によって選択されます(キャップトラップ)。cDNAの放出後、5'-および3'リンカーがライゲーションされる。キャリアに由来するライブラリー分子は、I-SceIおよびI-CeuIホーミングエンドヌクレアーゼを使用して分解され、断片はSPRI磁気ビーズを使用して除去されます。その後、ライブラリはPCR増幅されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
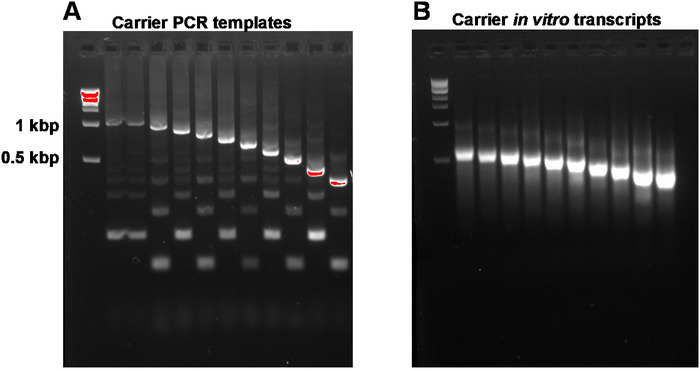
図2:インビトロ転写におけるキャリアPCRテンプレートおよびキャリアの代表的なゲル分析。(A)ゲル精製前のキャリアPCRテンプレート:第1ウェルは1kbpマーカーを含み、続いてキャリアPCRテンプレート1、1〜10が続く。(B)インビトロ転写物中のキャリア:第1のウェルは1kbpマーカーを含み、続いてキャリア転写物1〜10が続く。キャリア転写物は、積載前に95°Cで5分間加熱することにより変性した。この図のより大きなバージョンを表示するには、ここをクリックしてください。
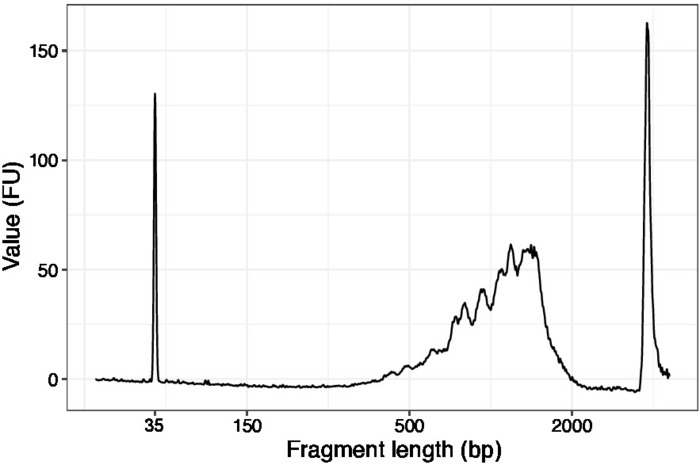
図3:キャリア劣化の第1ラウンド前のSLIC-CAGEの代表的なDNA品質(高感度DNAチップ)の痕跡。この図のより大きなバージョンを表示するには、ここをクリックしてください。
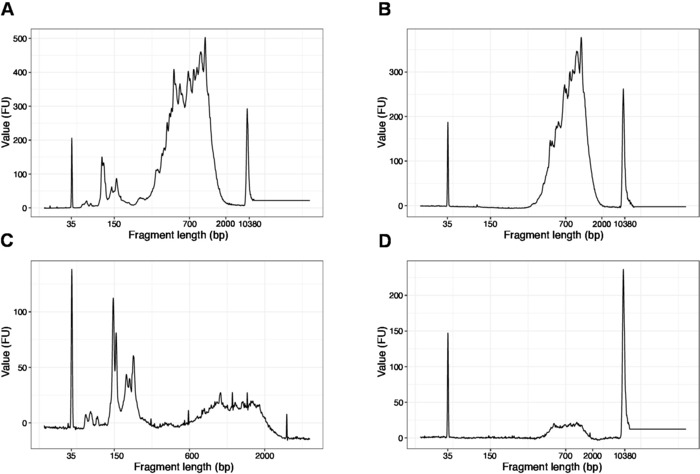
図4:PCR増幅後のSLIC-CAGEライブラリーの代表的なDNA品質(高感度DNAチップ)の痕跡。(A) SLIC-CAGE ライブラリで、短いフラグメントを削除するために追加のサイズ選択が必要です。(B) SLIC-CAGE ライブラリ 0.6x SPRI ビーズを使用してサンプル比を使用してサイズ選択後。(C) SLIC-CAGE ※短いフラグメントの除去にサイズ選択が必要な出力量が少ない。(D) SLIC-CAGEライブラリは、0.6:1 SPRIビーズをサンプル比に使用してサイズ選択後の出力量が少ない。この図のより大きなバージョンを表示するには、ここをクリックしてください。
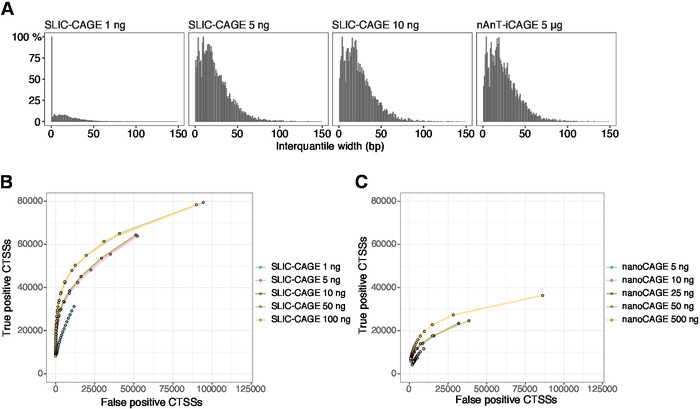
図 5:SLIC-CAGE ライブラリの検証。(A) SLIC-CAGEライブラリーにおけるタグクラスター間質幅の分布は、S.セレビシエ総RNAの1、5、または10ngから調製し、およびS.セレビシエ総RNAの5μgから調製されたnAnT-iCAGEライブラリー内で。1 ng SLIC-CAGE ライブラリ内の狭いタグ クラスタの量が多い場合は、その複雑さが低いことを示します。(B) S. セレビシエSLIC-CAGE ライブラリーにおける CTSS 識別のための ROC 曲線。すべてのS. セレビシエnAnT-iCAGE CTSSS が真のセットとして使用されました。(C) S.セレビシエナノケージライブラリーにおけるCTSS同定のためのROC曲線。すべてのS. セレビシエnAnT-iCAGE CTSSS が真のセットとして使用されました。ROC曲線の比較は、SLIC-CAGEがCTSS同定においてナノケージを強く上回っていることを示している。アレイエクスプレスE-MTAB-6519からのデータが使用されました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
表1:担体合成遺伝子の配列。I-SceI サイトは太字で紫色で斜体化され、I-CeuI 認識サイトは緑色です。このファイルをダウンロードするには、ここをクリックしてください。
| キャリア | リバースプライマー 5'-3' | PCR 製品の長さ / bp | |
| 1 | PCR_N6_r1: NnNNNNNCTACGTCGCAGAAATT | 1034年 | |
| 2 | PCR_N6_r2: NNNNNNTATCCAGATCGTTGAGCTGC | 966の | |
| 3 | PCR_N6_r3: NnNNNCACTGGGGCTCTCTTTACG | 889の | |
| 4 | PCR_N6_r4: NnNNNGCCGTCACTTTCGTTCGT | 821の | |
| 5 | PCR_N6_r5: NnNNNNNNナグットガットガガグトCTTC | 744の | |
| 6 | PCR_N6_r6: NNNNNNGTGAGAGAGAATTTTTCCCA | 676の | |
| 7 | PCR_N6_r7: NNNNNNCTCGCGGCTCCAGTAAC | 599の | |
| 8 | PCR_N6_r8: NNNNNNNNNNTCGCGGTGTCGTAC | 531の | |
| 9 | PCR_N6_r9: NNNNNNACCCGCGCCCCGCAGG | 454名 | |
| 10歳 | PCR_N6_r10: NNNNNNAGAGGGCGTTTTGCCCCA | 386の | |
| *フォワードプライマーは、すべてのキャリアテンプレートで同じです。下線が引かれているのはT7プロモーターシーケンスです。PCR_GN5_f1: タアダクアクトカタニャンNNCAGCGTTCGCTA | |||
表 2: キャリア テンプレート増幅用のプライマー。フォワード プライマーは、すべてのキャリア テンプレートで同じです。下線が引かれているのはT7プロモーターシーケンスです。PCR_GN5_f1:タアタクアクトカッタGNNNCAGCGTTCGCTA.異なるリバースプライマーを使用して、PCRテンプレート、したがって、異なる長さのキャリアRNAが生成されます。
| キャリア | 長さ | 上限なし/μg | 上限/μg |
| 1 | 1034年 | 3.96件 | 0.45分 |
| 2 | 966の | 8.36件 | 0.95件 |
| 3 | 889の | 4.4年 | 0.5年 |
| 4 | 821の | 6.6年 | 0.75年 |
| 5 | 744の | 4.4年 | 0.5年 |
| 6 | 676の | 3.08件 | 0.35 |
| 7 | 599の | 4.4年 | 0.5年 |
| 8 | 531の | 3.96件 | 0.45分 |
| 9 | 454名 | 2.64から | 0.3年 |
| 10歳 | 386の | 2.2年 | 0.25分 |
表3:RNAキャリアミックス。キャリアミックス0.3-1 kbpの合計49 μg:上限なし= 44 μg、キャップ= 5 μg。
| 合計 RNA 入力 /ng | PCR サイクル |
| 1 ng | 18歳 |
| 2 ng | 17歳 |
| 5 ng | 16歳 |
| 10 ng | 15-16日 |
| 25 ng | 14-15日 |
| 50 ng | 13-15日 |
| 100 ng | 12-14日 |
表 4:サンプル総RNA入力の依存性におけるPCRサイクルの予想数。概算サイクル数は、サッカロマイセスセレビシエ、ショウジョウバエメラノガスター、およびマスマス全RNAを用いて行われた実験に基づいている。
| 合計 RNA 入力/ng | 全体マップ率 | % 一意にマップ | % キャリア |
| 1 ng | 30歳 | 20-30日 | 30歳 |
| 2 ng | 60歳 | 20から50 | 10歳 |
| 5 ng | 60から70 | 40から60 | 5-10日 |
| 10 ng | 60から70 | 40から60 | 5-10日 |
| 25 ng | 65から80 | 40から70 | 0~5 |
| 50 ng | 65から80 | 40から70 | 0~3 |
| 100 ng | 70から85 | 40から70 | 0~2 |
表5:予想されるマッピング効率と総RNA入力量の依存性について。おおよその数が提示され、サッカロマイセスセレビシエとムスムスルムス総RNAを用いて行われた実験に基づいて提示される。
ディスカッション
SLIC-CAGEライブラリの準備を成功させるためには、サンプル吸着によるサンプル損失を防ぐために、低結合のヒントとチューブを使用することが重要です。上清の取り出しを伴うすべてのステップで、サンプル量全体を回収することをお勧めします。プロトコルには複数のステップがあるため、サンプルの連続的な損失はライブラリの失敗につながります。
CAGE(nAnT-iCAGE)が日常的に行われていない場合は、同じ総RNAサンプルの異なる入力量(10ng、20ng、50ng、100 ng、200 ng)でSLIC-CAGEをテストし、合計RNAの5μgを使用して調製されたnAnT-iCAGEライブラリと比較することをお知りください。nAnT-iCAGEライブラリーが失敗した場合(サンプルごとに得られたDNAライブラリーの0.5-1 ng未満)、SLIC-CAGEは機能しそうになく、サンプル損失を最小限に抑える必要があります。
上限のない劣化RNAまたはrRNAを欠いている高品質のライブラリーを確保するための重要なステップは、セクション7で説明するキャップトラップです。ストレプトアビジンビーズが洗浄バッファーで完全に再懸濁され、洗浄バッファーが次の洗浄工程またはcDNAの溶出に続く前に除去されることが非常に重要です。
キャリア劣化の最初のラウンド後のqPCRの結果が、アダプター_f1とcarrier_f1プライマーの使用との間に違いがない場合は、プロトコルを継続することをお勧めします。キャリア劣化の第2ラウンドの後、Ct値の差が5未満の場合、キャリア劣化の第3ラウンドが推奨される。我々は必要な劣化の第三ラウンドを見つけたことがなく、それが発生した場合は、ホーミングエンドヌクレアーゼ株を交換することをお勧めします。
取得したライブラリの最終的な量がシーケンスに十分でない場合は、PCR 増幅の追加ラウンドがプロトコルに追加される可能性があります。PCR増幅は、サイズ選択で避けることができないサンプル損失を考慮して、シーケンシングに十分な材料を得るために必要な増幅サイクルの最小数で設定できます。SPRI磁気ビーズを使用した精製またはサイズ選択は、すべての小さな(<200 bp)フラグメントが除去されるまで実行され(必要に応じて0.6:1ビーズをサンプル比に使用)、ライブラリはPicogreenを使用して定量化する必要があります。
ライブラリは、シングルエンドモードまたはペアエンドモードでシーケンスできます。ペアエンドシーケンスを使用すると、トランスクリプトアイソフォームに関する情報を取得できます。さらに、逆転写はランダムプライマー(TCT-N 6、N6はランダムヘキサマー)を用いて行われるので、配列された3'エンドからの情報は、PCRの重複を崩壊させる一意の分子識別子(UMI)として使用することができる。適度な数のPCR増幅サイクルが使用される(最大18)ため、UMIの使用は以前に不要であることが分かっています。
プロトコルのコアは nAnT-iCAGE11に依存しているため、SLIC-CAGE は 8 つのバーコードを使用します。したがって、現在、8 個を超えるサンプルの多重化はサポートされていません。さらに、SLIC-CAGEとnAnT-iCAGEの両方は、プロトコルがAMPure XPビーズを使用してサイズ除外を通じてリンカーとPCRアーティファクトを除去するように設計されているので、200 bpより短いRNAをキャプチャするのに適していません。
SLIC-CAGEは、全RNA材料のナノグラムを用いて転写開始開始部位をマッピングするための唯一の偏りのない低入力単一ヌクレオチド分解能法である。代替方法は、キャップトラップの代わりにバーコードキャップRNAに逆転写酵素のテンプレート切り替え活性に依存する(例えば、NanoCAGE15およびNanoPARE16)。テンプレートの切り替えにより、これらの方法はTSS検出において配列固有のバイアスを示し、偽陽性TSSの数が増加し、真のTSS9、10の数が減少します。
開示事項
分解性キャリアRNA/DNAの特許が満たされています。
謝辞
この研究は、ウェルカム・トラスト助成金(106954)がB.L.と医学研究評議会(MRC)コア・ファンディング(MC-A652-5QA10)に授与されました。N. C. は EMBO 長期フェローシップ (EMBO ALTF 1279-2016) によってサポートされました。E. P. は、英国医学研究評議会によって支援されました。B. L. は、英国医学研究評議会 (MC UP 1102/1) によってサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| 2-propanol, Bioultra, for molecular biology, ≥99.5% | Sigma-Aldrich | 59304-100ML-F | Used in RNAclean XP purification. |
| 3' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 3'linkers is described in the supplementary of this protocol. | ||
| 5' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 5'linkers is described in the supplementary of this protocol. | ||
| Agencourt AMPure XP, 60 mL | Beckman Coulter | A63881 | Purification of DNA |
| Agencourt RNAClean XP Kit | Beckman Coulter | A63987 | Purification of RNA and RNA:cDNA hybrids in CAGE steps. |
| Axygen 0.2 mL Polypropylene PCR Tube Strips and Domed Cap Strips | Axygen (available through Corning) | PCR-0208-CP-C | Or any 8-tube PCR strips (used only for water and mixes). |
| Axygen 1 x 8 strip domed PCR caps | Axygen (available through Corning) | PCR-02CP-C | Caps for PCR plates. |
| Axygen 1.5 mL Maxymum Recovery Snaplock Microcentrifuge Tube | Axygen (available through Corning) | MCT-150-L-C | Low-binding 1.5 mL tubes, used for enzyme mixes or sample concentration. |
| Axygen 96 well no skirt PCR microplate | Axygen (available through Corning) | PCR-96-C | Low-binding PCR plates - have to be used for all steps in the protocol. Note that plates should be cut to contain 2 x 8 wells for easier visibility of the samples |
| Bioanalyzer (or Tapestation): RNA nano and HS DNA kits | Agilent | To determine quality of RNA, efficient size selection and final quality of the library (Tapestation can also be used) | |
| Biotin (Long Arm) Hydrazide | Vector laboratories | SP-1100 | Biotinylation/tagging |
| Cutsmart buffer | NEB | Restriction enzyme buffer | |
| Deep Vent (exo-) DNA Polymerase | NEB | M0259S | Second strand synthesis |
| DNA Ligation Kit, Mighty Mix | Takara | 6023 | Used for 5' and 3'-linker ligation |
| dNTP mix (10 mM each) | ThermoFisher Scientific | 18427013 | dNTP mix for production of carrier templates (or any dNTPs suitable for PCR) |
| Dynabeads M-270 Streptavidin | Invitrogen | 65305 | Cap-trapping. Do not use other beads as these are optimised with the buffers used. |
| DynaMag-2 Magnet | ThermoFisher Scientific | 12321D | Magnetic stand for 1.5 mL tubes - used to prepare Streptavidin beads. |
| DynaMag-96 Side Skirted Magnet | ThermoFisher Scientific | 12027 | Magnetic stand for PCR plates (96 well-plates) - used with cut plates to contain 2 x 8 wells. |
| Ethanol, BioUltra, for molecular biology, ≥99.8% | Sigma-Aldrich | 51976-500ML-F | Used in AMPure washes. Any molecular biology suitable ethanol can be used. |
| Exonuclease I (E. coli) | NEB | M0293S | Leftover primer degradation |
| Gel Loading Dye, Purple (6x), no SDS | NEB | B7025S | agarose gel loading dye |
| HiScribe T7 High Yield RNA Synthesis Kit | New England Biolabs | E2040S | Kit for carrier in vitro transcription |
| Horizontal electrophoresis apparatus | purification of carrier DNA templates from agarose gels | ||
| I-Ceu | NEB | R0699S | Homing endonuclease used for carrier degradation. |
| I-SceI | NEB | R0694S | Homing endonuclease used for carrier degradation. |
| KAPA HiFi HS ReadyMix (2x) | Kapa Biosystems (Supplied by Roche) | KK2601 | PCR mix for target library amplification |
| KAPA SYBR FAST qPCR kit (Universal) 2x | Kapa Biosystems (Supplied by Roche) | KK4600 | qPCR mix to assess degradation efficiency and requiered number of PCR amplification cycles |
| Micropipettes and multichannel micropipettes (0.1-10 µL, 1-20 µL, 20-200 µL) | Gilson | Use of Gilson with the low-binding Sorenson tips is recommended. Other micropippetes might not be compatible. Different brand low-binding tips may not be of equal quality and may increase sample loss. | |
| Microplate reader | For Picogreen concentration measurement of the final library. Microplates are used to allow small volume measurement and reduce sample waste. | ||
| nuclease free water | ThermoFisher Scientific | AM9937 | Or any nuclease (DNase and RNase) free water |
| PCR thermal cycler | incubation steps and PCR amplficication | ||
| Phusion High-Fidelity DNA Polymerase | ThermoFisher Scientific | F530S | DNA polymerase for amplification of carrier templates (or any high fidelity polymerase) |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Purification of carrier PCR templates from agarose gels. |
| qPCR machine | determining PCR amplification cyle number and degree of carrier degradation | ||
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher Scientific | P11495 | Used to measure final library concentration - recommended as, in our hands, it is more accurate and reproducible than Qubit. |
| Quick-Load Purple 100 bp DNA Ladder | NEB | N0551S | DNA ladder |
| Quick-Load Purple 1 kb Plus DNA Ladder | NEB | N0550S | DNA ladder |
| Ribonuclease H | Takara | 2150A | Digestion of RNA after cap-trapping. |
| RNase ONE Ribonuclease | Promega | M4261 | Degradation of single stranded RNA not protected by cDNA. |
| RNase-Free DNase Set | Qiagen | 79254 | Removal of carrier DNA templates after in vitro transcription. |
| RNeasy Mini Kit | Qiagen | 74104 | For cleanup of carrier RNA from in vitro transcription or capping |
| Sodium acetate, 1 M, aq.soln, pH 4.5 RNAse free | VWR | AAJ63669-AK | Or any nuclease (DNase and RNase) free solution |
| Sodium acetate, 1 M, aq.soln, pH 6.0 RNAse free | Or any nuclease (DNase and RNase) free solution | ||
| Sodium periodate | Sigma-Aldrich | 311448-100G | Oxidation of vicinal diols |
| Sorenson low binding aerosol barrier tips, MicroReach Guard, volume range 10 μL, Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719390-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 1,000 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719463-1000EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 20 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719412-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 200 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719447-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| SpeedVac Vacuum Concentrator | concentrating samples in various steps to lower volume | ||
| SuperScript III Reverse Transcriptase | ThermoFisher Scientific | 18080044 | Used for reverse transcription (1st CAGE step) |
| Trehalose/sorbitol solution | Preparation is described in Murata et al. 2014. | ||
| Tris-HCl, 1 M aq.soln, pH 8.5 | 1 M solution, DNase and RNase free | ||
| tRNA (20 mg/mL) | tRNA solution. Preparation is described in Murata et al. 2014. | ||
| UltraPure Low Melting Point Agarose | ThermoFisher Scientific | 16520050 | Or any suitable pure low-melt agarose. |
| USB Shrimp Alkaline Phosphatase (SAP) | Applied Biosystems (Provided by ThermoFisher Scientific) | 78390500UN | |
| USER Enzyme | NEB | M5505S | Degradation of 3'linker's upper strand, Uracil Specific Excision Reagent/Enzyme |
| Vaccinia Capping System | NEB | M2080S | Enzymatic kit for in vitro capping of carrier molecules |
| Wash buffer A | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer B | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer C | Cap trapping washes. Preparation is described in Murata et al. 2014. |
参考文献
- Shiraki, T., et al. Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proceedings of the National Academy of Sciences of the United States of America. 100 (26), 15776-15781 (2003).
- Haberle, V., Lenhard, B. Promoter architectures and developmental gene regulation. Seminars in Cell and Developmental Biology. 57, 11-23 (2016).
- Haberle, V., Stark, A. Eukaryotic core promoters and the functional basis of transcription initiation. Nature Reviews Molecular Cell Biology. 19 (10), 621-637 (2018).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Celniker, S. E., et al. Unlocking the secrets of the genome. Nature. 459 (7249), 927-930 (2009).
- Consortium, F., et al. A promoter-level mammalian expression atlas. Nature. 507 (7493), 462-470 (2014).
- Boyd, M., et al. Characterization of the enhancer and promoter landscape of inflammatory bowel disease from human colon biopsies. Nature Communications. 9 (1), 1661 (2018).
- Adiconis, X., et al. Comprehensive comparative analysis of 5'-end RNA-sequencing methods. Nature Methods. , (2018).
- Cvetesic, N., et al. SLIC-CAGE: high-resolution transcription start site mapping using nanogram-levels of total RNA. Genome Research. 28 (12), 1943-1956 (2018).
- Murata, M., et al. Detecting expressed genes using CAGE. Methods in Molecular Biology. 1164, 67-85 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9, 357 (2012).
- A language and environment for statistical computing. Available from: https://www.R-project.org/ (2017)
- Haberle, V., Forrest, A. R., Hayashizaki, Y., Carninci, P., Lenhard, B. CAGEr: precise TSS data retrieval and high-resolution promoterome mining for integrative analyses. Nucleic Acids Research. 43 (8), e51 (2015).
- Poulain, S., et al. NanoCAGE: A Method for the Analysis of Coding and Noncoding 5'-Capped Transcriptomes. Methods in Molecular Biology. 1543, 57-109 (2017).
- Schon, M. A., Kellner, M. J., Plotnikova, A., Hofmann, F., Nodine, M. D. NanoPARE: parallel analysis of RNA 5' ends from low-input RNA. Genome Research. 28 (12), 1931-1942 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved