Method Article
Transkripsiyon başlangıç site haritalama süper düşük giriş taşıyıcı-kafes kullanarak
Bu Makalede
Özet
Gen ifadesinin Cap Analizi (Cage), RNA polimeraz II transkripsiyon başlangıç sitelerini tek nükle çözünürlüğe yakalamak için mRNA 5 ' ucu genom çapında nicel haritalama için bir yöntemdir. Bu çalışma, nanogram-toplam RNA miktarını kullanarak yüksek kaliteli kütüphaneler oluşturmak için düşük giriş (dılım-kafes) protokolünü açıklar.
Özet
Gen ifadesinin Cap Analizi (Cage), RNA polimeraz II transkripsiyon başlangıç sitelerinin (tsss) tek nükle çözünürlüğü tespiti için kullanılan bir yöntemdir. TSSs 'nin doğru tespiti, çekirdek promotörleri tanıma ve keşfi geliştirir. Ayrıca, aktif arttırıcılar çift yönlü transkripsiyon başlatma imzaları ile tespit edilebilir. Burada açıklanan süper düşük giriş taşıyıcı-kafes (dılım-kafes) gerçekleştirmek için bir protokoldür. CAGE protokolünün bu dılım adaptasyonu, ilgi örneğine eklenen bir in vitro transkripsiyonu RNA taşıyıcı karışımı kullanarak RNA miktarını yapay olarak artırarak RNA kaybını en aza indirir ve böylece nanogram-toplam miktardan Kütüphane hazırlığı sağlar RNA (yani, binlerce hücre). Taşıyıcı beklenen DNA Kütüphanesi parça uzunluğu dağılımı taklit, böylece homojen bir taşıyıcı bolluğu neden olabilir önyargıları ortadan kaldırmak. Protokolün son aşamalarında, taşıyıcı endonükleases ile bozulma yoluyla kaldırılır ve hedef Kütüphane amplifikatörlü. Hedef örnek kütüphane bozulmaya karşı korunmaktadır, homing endonükleaz tanıma siteleri uzun (arasında 18 ve 27 BP), ökaryotik genomlar içinde varoluşunun olasılığını çok düşük hale. Sonuç, yeni nesil sıralama için hazır bir DNA kütüphanesidir. Protokoldeki tüm adımlar, sıralama kadar, 6 gün içinde tamamlanabilir. Taşıyıcı hazırlığı tam bir çalışma günü gerektirir; Ancak, büyük miktarlarda hazırlanabilir ve dondurulmuş tutulması-80 °C. Sıralı olarak, genom çapında tek nükle çözünürlüğü elde etmek için okuma işlenebilir TSSs. TSSs, gen Yönetmeliğine ilişkin anlayış sağlayan çekirdek promotör veya arttırıcı keşif için kullanılabilir. Bir kez Promoters için toplanan veri 5 ' merkezli ifade profil oluşturma için de kullanılabilir.
Giriş
Gen ifadesinin Cap Analizi (Cage), RNA polimeraz II transkripsiyon başlangıç sitelerindeki (tsss)1tek nüklenotid çözünürlüğünde genom çapında haritalama için kullanılan bir yöntemdir. Nicel niteliği de 5 ' End merkezli ifade profilleme sağlar. Tsss (yaklaşık 40 BP upstream ve downstream) çevreleyen bölgeler çekirdek promotörler ve RNA polimeraz II ve genel transkripsiyon faktörleri bind (daha önce2,3gözden) fiziksel konumunu temsil eder. Tsss tam konumları hakkında bilgi temel Organizatör Discovery ve organizatör dinamikleri izlemek için kullanılabilir. Ayrıca, aktif arttırıcılar çift yönlü transkripsiyon imzaları sergiler olarak, CAGE verileri de arttırıcı keşif ve geliştirici dinamiklerinin izlenmesi için kullanılabilir4. CAGE metodolojisi son zamanlarda geniş uygulama ve ENCODE5, modencode6ve Fantom projeleri7gibi yüksek profilli araştırma projelerinde kullanımı nedeniyle popülaritesi artmıştır. Buna ek olarak, TSS bilgileri de hastalık özgü TSSs tanılama amacıyla8kullanılabilir olarak, sağlıklı ve hastalıklı doku ayırt etmek için önemli olduğunu kanıtlıyor.
TSS haritalama için çeşitli yöntemler kullanılabilir olsa da (CAGE, RAMPAGE, STRT, nanoCAGE, nanoCAGE-XL, oligo-capping), biz ve diğerleri son zamanlarda kafes en tarafsız yöntemi yanlış pozitif9 en az sayıda gerçek tsss yakalamak için gösterilmiştir , 10. son Cage protokolü, NAnT-ıcage11, TSS profil için en tarafsız protokoldür, bu kısıtlama enzimleri kullanarak kısa etiketlere parçaları kesme önler ve PCR amplifikasyon kullanmaz gibi. NAnT-ıcage protokolünün bir sınırlaması, büyük miktarda başlangıç malzemesi (örn. her örnek için 5 μg toplam RNA) gereksinimdir. Belirli, biyolojik olarak alakalı soruları yanıtlamak için, genellikle bu kadar yüksek miktarlarda başlangıç malzemesi elde etmek imkansız (örn., FACS sıralı hücreler veya erken embriyonik aşamaları için). Son olarak, nAnT-ıcage başarılı olursa, her örnekten sadece 1-2 ng DNA Kütüphane malzemesi kullanılabilir, böylece ulaşılabilir sıralama derinliği sınırlandırılabilir.
Sadece Nanogramlar toplam RNA kullanarak TSS profil etkinleştirmek için, son zamanlarda süper düşük giriş taşıyıcı-kafes10 geliştirdik (dılım-kafes, Şekil 1). DILIM-kafes yüksek karmaşıklık kütüphaneler elde etmek için toplam RNA sadece 10 ng gerektirir. Protokollerimiz, toplam 5 μg RNA malzemesi elde etmek için ilgi RNA 'ya eklenen özenle tasarlanan sentetik RNA taşıyıcısına dayanır. Sentetik taşıyıcı, aşırı homojen moleküllerin neden olabilecek potansiyel önyargıları önlemek için uzunluk dağılımı hedef DNA kütüphanesini taklit eder. Taşıyıcının sırası, iki nedenden ötürü Escherichia coli leucyl-tRNA sentetaz geni (Tablo 1) dizisine dayanır. İlk olarak, son kütüphanede taşıyıcı herhangi bir kalan, sıralı olsa bile, bir ökaryotik genom harita olmayacaktır. İkinci olarak, E. coli mezofilik türler olarak, temızlık genler dılım-kafes için uygun sıcaklık aralığı için optimize edilmiştir. Taşıyıcı dizisi aynı zamanda taşıyıcı RNA moleküllerinden türetilen DNA spesifik bozulması sağlamak için homing endonükleaz tanıma siteleri ile gömülür. Hedef, örnek-türetilen kitaplık sağlam kalır, homing endonükleaz tanıma siteleri uzun olduğu gibi (ı-ceui = 27 BP; I-SceI = 18 BP) ve istatistiksel olarak ökaryotik genomlar içinde bulunmak imkansız. Taşıyıcı ve boyutu dışlama tarafından parçaların çıkarılması belirli bozulması sonra, hedef Kütüphane PCR amplifikatörlüğünü ve yeni nesil sıralı için hazır. Başlangıç RNA miktarına (1-100 ng) bağlı olarak, 13-18 PCR amplifikasyon döngüleri arasında gerekli olması bekleniyor. 5-50 ng arasında her örnek aralıkları başına DNA son miktarı, çok derin sıralı için yeterli malzeme verimli. Toplam RNA sadece 1-2 ng kullanırken, gerçek TSSs tespit edilebilir; Ancak, kitaplıkları daha düşük karmaşıklık olması bekleniyor. Son olarak, SLıC-CAGE, nAnT-ıcage protokol11' e dayanır olarak, sıralamanın öncesinde sekiz örneğe kadar çoğullama sağlar.
Protokol
1. taşıyıcı hazırlanması
-
İn vitro transkripsiyon için DNA şablonlarının hazırlanması
- 41 μL su, 20 μL 5x HF tampon, 8 μL 2,5 mM dNTPs, 10 μL 10 μM benzersiz ileri astar (PCR_GN5_f1, Tablo 2; astarlar çözünür ve suda seyreltilmiş) ile her BIR PCR şablonu için PCR karışımını hazırlayın , sentetik taşıyıcı geni ve 1 μL Phusion polimeraz içeren 10 μL 2 ng/μL şablon Plasmid. PCR karışımı pipetleme ile karıştırın. Tüm 10 şablonlar için bir ana karışım aynı anda hazırlanabilir (11 reaksiyona hazırlanın).
- PCR karışımı 90 μL, 10 μM ters astar (PCR_N6_r1-R10, Tablo 2) Ile 10 μL 'ye ekleyin. Pipetleme ile karıştırın.
- PCR aşağıdaki programı kullanarak şablonları yükseltir: 98 °C için 60 s, (98 °C için 10 s, 50 °C 30 s, 72 °C için 30 s) 35 döngüleri, 72 °C 10 dakika, 4 °C ' de tutun.
-
PCR-amplifikatörleşmiş DNA jel arıtma şablonları
- % 1 agaroz jel hazırlayın (düşük erime agaroz önerilir).
- Hacmi azaltmak için, 100 μL 'den 20 μL 'ye kadar olan PCR reaksiyon karışımlarının düşük orta sıcaklıkta (30 – 40 °C) vakum yoğunlaştırıcıyı kullanarak konsantre olun.
- 6x yükleme boyasını 6 μL ekleyin, iyi karıştırın ve jel üzerine yükleyin. Electroforesis 'i, kullanılan Elektroforez tankı (5 – 10 V/cm) için uygun voltajda 1x TAE tampon olarak 30 dakika boyunca çalıştırın. Paralel bir 100 BP veya 1.000 BP DNA merdiven çalıştırın.
- Temiz bir neşter kullanarak, hedef PCR ürününü içeren jel dilimlerini çıkarın. Aşırı agaroz jel kaçının. Bir jel ekstraksiyon kiti kullanarak PCR ürünlerini arındırın (üreticinin talimatlarına göre).
Not: Agaroz jelinden izole edilen DNA A260/A230 oranları genellikle düşüktür (0.1 – 0.3). Beklenen hedef ürün ve yan ürünler Şekil 2a'da gösterilir. 100 μL PCR reaksiyonundan beklenen verimler 1,2 – 3 μg 'dir. reaksiyonlar daha yüksek bir verim elde etmek için ölçeklendirilebilir.
-
Taşıyıcı moleküllerin In vitro transkripsiyonu
- Üretici talimatlarına göre T7 RNA polimeraz kullanarak taşıyıcı RNA in vitro dönüştürme. 10 – 20 μL reaksiyonu ayarlayın (önerilen Kit malzeme tablosudur).
- RNA arıtma kiti kullanarak in vitro transkripsiyonu RNA 'yi arındırın. DNase ben üreticinin standart talimatları takip kullanarak çözüm DNA sindirim ayarlayın ve 50 μL su RNA elute. Elüsyon verimi artırmak için, santrifüjleme öncesinde 5 dakika boyunca suyu sütunda bırakın.
Not: Sütunların maksimum bağlayıcı kapasitesini aşmamaya dikkat edin ( malzeme tablosundabelirtilen Kit içinde, kapasite 100 μg 'ye kadar). PCR şablonlarından beklenen verim 1 – 10 (1 KBP ila 200 BP uzunluğunda), 10 μL in vitro transkripsiyon reaksiyonlarından 25 – 50 μg 'dir. Reaksiyonlar taşıyıcı moleküllerin daha büyük bir stok almak için ölçeklendirilebilir.
-
İn vitro transkripsiyonu yapan RNA moleküllerinin capping
- 2 μL 10X kapatma tamponu, 1 μL 10 mm GTP, 1 μL 2 mm Sam (taze seyreltilmiş) ve taşıyıcı RNA başına 1 μL Vaccinia kapatma enzimini birleştirerek kapak karışımını hazırlayın.
- 65 °C ' de 10 dak için 15 μL toplam hacimde 10 μg 'ye kadar her taşıyıcı molekülü karıştırın. İkincil yapı oluşumunu önlemek için hemen buz üzerine yerleştirin.
- Denatli taşıyıcı RNA 'nın 5 μL 'sini kapatma karışımı ile karıştırın ve 37 °c ' de 1 saat boyunca inküye yapın.
- RNA arıtma kiti kullanarak capped RNA moleküllerini arındırın — üreticinin temizleme protokolünü izleyin. 30 μL su içinde elute RNA. Elüsyon verimi artırmak için, santrifüjleme öncesinde 5 dakika boyunca suyu sütunda bırakın.
Not: Mikrohacim spektrofotometresini kullanarak konsantrasyonu ölçün. Beklenen A260/A280 oranı > 2 ve A260/A230 > 2 ' dir. Bazı RNA örnekleri için A260/A230 1.3 – 2 arasında olabilir. 10 μg 'sız RNA kullanıldığında beklenen verim 9 – 10 μg ' lık capped RNA 'dir.
- Tablo 3' te açıklanan tutarları birleştirerek, kaplı ve kullanılmayan taşıyıcının karışımını hazırlayın. Tüp Flicking ve mikrohacim spektrofotometre kullanarak konsantrasyon ölçmek iyi karıştırın.
Not: Taşıyıcı daha yüksek bir konsantrasyon Ters transkripsiyon reaksiyonu sığdırmak için gerekli ise (aşağıya bakın), taşıyıcı karışımı istenilen son konsantrasyona ulaşıncaya kadar düşük orta sıcaklıkta (30-35 °C) vakum konsantratörü kullanılarak konsantre olabilir. 2-14 adımları Murata ve ark.11 tarafından bildirilen standart NAnT-ıcage protokolünden değiştirilir.
2. Ters transkripsiyon
- RT astar 1 μL (2,5 mM TCT-N6 su içinde çözünür, sıra için bkz: Tamamlayıcı Tablo 1), 10 ng Toplam faiz RNA ve 4.990 ng taşıyıcı karışımı (Tablo 3) 10 μL toplam hacim içinde düşük bağlayıcı PCR plaka. Tüp Flicking ile karıştırın.
Not: Örnek RNA Ters transkripsiyon için çok seyreltilip (aşağıya bakın), taşıyıcının uygun miktarıyla birleştirin, vakum yoğunlaştırıcıyı 9 μL toplam hacmine odaklanın ve 1 μL RT astar ekleyin. Taşıyıcı Earl ekleme, toplam 5 μg RNA ulaşmak için örnek kaybı önler. - 5 dakika için 65 °C ' de adım 2,1 ' dan karışımı ısıtın ve renaturasyonu önlemek için hemen buz üzerine yerleştirin.
-
Ters transkripsiyon (RT) karışımı hazırlayın.
- Her örnek için 6,1 μL su birleştirin (RNase-ve DNase-Free), 7,6 μL 5x ilk iplikli tampon, 1,9 μL, 0,1 M DTT, 1 μL, 10 mm dntps, 7,6 μL trehalose/Sorbitol karışımı (bkz. Murata et al.11) ve 3,8 μL önerilen ters transkriptaz (bkz. < c 6. malzeme tablosu). Tüp Flicking iyi karıştırın.
- 10 μL RNA, taşıyıcı ve RT astar (Toplam hacim 38 μL) ile PCR tüpüne RT karışımı 28 μL ekleyin. Pipetleme ile iyi karıştırın.
Not: Mix trehalose/Sorbitol nedeniyle son derece viskoz. Gözle görülür homojen kadar karıştırın. - Aşağıdaki programı kullanarak bir termal bisikletçi içinde inkükleyici: 30 s için 25 °C, 60 dk için 50 °C, ve 4 °C ' de tutun.
-
CDNA arıtma: RNA melez SPRı manyetik boncuklar kullanarak
- Ekle 68,4 μL önerilen RNAse ve DNase-Free SPRı boncuk (bkz: malzeme tablosu) 38 ΜL RT karışımı (boncuk örnek oranı 1.8:1). Pipetleme ile iyi karıştırın ve oda sıcaklığında (RT) 5 dakika boyunca kuluçk yapın.
- Bir manyetik standı üzerinde boncuklar ayırın 5 dakika. süpernatant atın ve 200 μL ile iki kez boncuk yıkamak 70% etanol (taze hazırlanmış).
Not: Etanol karıştırıcı olmadan boncuklar eklenir ve tüp manyetik standı üzerinde iken. Eklenen etanol hemen kaldırılır. Bakım örnek kaybına neden olabilir gibi yıkanıyor sırasında herhangi bir boncuk kaybetmek değil alınmalıdır. - Tüp hala manyetik stand üzerinde iken, etanol tüm izlerini çıkarın. Etanol damlacıkları çıkarılabilir ve bir P10 pipet kullanarak tüp dışarı itti olabilir. Boncuk kuru izin vermeyin.
- 37 °C ' de boncuklar için önceden ısıtılan su 42 μL ekleyin ve 60x yukarı ve aşağı pipetleme ile örnek elute.
Not: Köpükte boncuk kaybına neden olabilir gibi pipetleme ile köpük neden değil (yani, bağlı örnek) dikkatli olun. - 37 °C ' de kapak olmadan 5 dakika boyunca inkübasyon yapın ve izleme miktarlarında etanol buharlaşma sağlar.
- 5 dakika boyunca bir manyetik stand üzerinde boncuklar ayırın ve yeni bir plaka süpernatant aktarmak.
Not: Boncuk çalışırken kaçınarak örnek kaybını önlemek için tüm süpernatant almak için deneyin. Son numune damlacıkları almak için P10 pipet kullanın.
3. oksidasyon
- Arıtılmış RT reaksiyonuna 2 μL 1 M NaOAc (pH 4,5) ekleyin. Pipetleme ile karıştırın, 2 μL 250 mM NaIO4 ekleyin ve tekrar karıştırın.
- 45 dakika boyunca buzun üzerinde kulvarın. ışık önlemek için alüminyum folyo ile plaka kapak.
- PH 'ı nötralize etmek için oksidasyon karışımından 16 μL Tris-HCl (pH 8,5) ekleyin.
- Oksitlenmiş cDNA: RNA melez SPRI manyetik boncuklar kullanarak arındırın. 60 μL oksidasyon karışımı (1.8:1 boncuk örnek oranı) için 108 μL SPRı boncuk ekleyin. (2.6.6) adımda açıklandığı gibi arıtma tekrarlayın. 42 μL su kullanan elute, 37 °C ' ye kadar ısıtılır.
Not: Taze hazırlamak 250 mM NaIO4 ekleyerek 18,7 μl su başına 1 mg naio4. NaIO4 ışığa duyarlıdır; Bu nedenle, solüsyonu alüminyum folyo veya ışığa dayanıklı bir tüple kaplı bir tüpte tutun.
4. biotinilasyon
- Saf oksitlenmiş numuneyi içeren tüpün içine 4 μL 1 M NaOAc (pH 6,0) ekleyin ve pipetleme ile karıştırın.
- 4 μL 10 mM biotin çözeltisi ekleyin, pipetleme ile karıştırın ve ışık önlemek için bir termal cycler içinde 2 saat 23 °C ' de inkük.
Not: 13,5 mL DMSO ile 50 mg biotin karıştırarak biotin çözümünü hazırlayın. Tek kullanımlık plakaya yapın ve-80 °c ' de dondurur. - Biyoinylated cDNA arındırmak: RNA melez SPRı manyetik boncuklar kullanarak. 12 μL 2-propanol ekleyin ve pipetleme ile karıştırın. 108 μL SPRı boncuk (1.8:1 boncuk örnek oranı) ekleyin ve adımlar 2.6.1 – 2.6.6 açıklandığı gibi arıtma yineleyin. 37 °C ' de önceden ısıtılan 42 μL su kullanarak elute.
Not: Protokol burada duraklatılır ve-80 °C ' de dondurulmuş numuneler alınabilir.
5. RNase ben Ddigestion
- Her örnek için 0,5 μL RNase ı (10 U/μL) ile% 10X RNase ı tampon 4,5 μL karıştırarak RNase ı karışımı hazırlayın. Pipetleme ile karıştırın.
- Her bir arıtılmış örneğe 5 μL Mix ekleyin (Toplam 45 μL). 37 °C ' de 30 dakika boyunca pipetleme ve inküye ile karıştırın.
6. streptavidin boncuk hazırlanması
- Her örnek için, streptavidin boncuk 30 μL Mix 0,38 μL 20 mg/mL tRNA ile bulamaç. 30 dakika boyunca buzun üzerinde kuluçat ve tüp Flicking her 5 dakika karıştırın.
Not: Streptavidin boncuklar şişeyi Flicking tarafından pipetleme önce iyi Bulamaç resuspend. TRNA çözeltisi Murata ve ark.11 ' e göre hazırlanmalıdır. - Manyetik standında boncukları 2 – 3 dakika ayırın. süper natant çıkarın.
- Boncukları, 15 μL tampon A ' da resuspending ile yıkayın. manyetik Standdaki boncukları 2 – 3 dakika ayırın ve supernatant çıkarın. Yıkama tekrarlayın ve supernatant çıkarın.
- 105 μL tampon A içinde boncuklar resuspend ve 0,19 μL 20 mg/mL tRNA ekleyin. Pipetleme ile iyi karıştırın.
Not: Boncukların kullanımından önce taze hazırlanması gerekir. RNase I sindirimi sırasında boncuk hazırlanması başlayın. Birden fazla numune için boncukları tek bir tüpte birlikte hazırlayın.
7. Cap-bindirme
-
Örnek bağlama
- 105 μL hazırlanmış streptavidin boncuk 45 μL RNase ı-tedavi örnek ekleyin. Pipetleme ile iyi karıştırın ve 37 °C ' de 30 dakika boyunca inküye yapın. her 10 dakikada bir pipetleme ile karıştırın.
- Manyetik standında boncukları 2 – 3 dakika ayırın. süper natant çıkarın.
-
Yıkama boncukları
- 150 μL yıkama tamponunu ekleyin ve boncukları pipetleme ile yeniden pelletini. Manyetik standında boncukları 2 – 3 dakika ayırın ve süpernatant çıkarın.
- 150 μL yıkama tamponunun B ekleyin ve boncuklar pipetleme ile yeniden pelletini. Manyetik standında boncukları 2 – 3 dakika ayırın ve süpernatant çıkarın.
- 150 μL yıkama tamponu C ekleyin ve boncuklar pipetleme ile yeniden pelletini. Manyetik standında boncukları 2 – 3 dakika ayırın ve süpernatant çıkarın.
Not: Tampon B ve C 37 °C ' ye önceden ısıtılmalıdır. A, B ve C yıkama tamponları için Tarifler Murata ve ark.11 ' de açıklanmıştır.
-
cDNA sürümü
- 1x RNase ı tamponunu 58,5 μL su ile 6,5 μL 10X RNase I tampon ile karıştırarak hazırlayın.
- 35 μL 1x RNase ı tampon içinde boncuk resuspend. 95 °C ' de 5 dakika boyunca kulyatın ve cDNA 'nın yeniden ilişkilendirmesini önlemek için doğrudan buz üzerinde 2 dakika transfer yapın. Basınç birikme nedeniyle pop-off olabilir gibi buz transferi sırasında kapakları tutun.
- Boncuk 2-3 dakika için bir manyetik standı ayırın ve yeni bir plaka süpernatant aktarmak.
- 30 μL 1x RNase ı tampon olarak boncuk resuspend. 2-3 dakika için manyetik stand üzerinde boncuk ayrı ve daha önce toplanan süpernatant (toplam hacmi cDNA yaklaşık 65 μL olmalıdır) için süpernatant aktarmak.
8. RNA kaldırma RNase H ve RNase ı sindirim
- Örnek başına, 2,4 μL su, 0,5 μL 10X RNase ı tampon, 0,1 μL RNase H ve 2 μL RNase ı birleştirir.
- Serbest bırakılan cDNA örneğinin 65 μL 'ye karışımı 5 μL ekleyin ve pipetleme ile karıştırın. 37 °C ' de 15 dakika boyunca kulyatın ve 4 °C ' de tutun.
- SPRı manyetik boncuklar kullanarak RNase sindirimi Mix cDNA arındırmak. 70 μL düşüş reaksiyonu ve pipetleme ile Mix için 126 μL SPRı boncuk ekleyin. , 2.6.1-2.6.6 SPRı boncuk arıtma için açıklandığı gibi arıtma adımlarını izleyin. 42 μL su kullanan elute, 37 °C ' de önceden ısıtılmıştır.
- Hazırlamak RNase ı karışımı 4,5 μL 10X RNase ı tampon ve 0,5 μL RNase ı birleştirerek.
- 5 μL RNase karışımı 40 μL temizleşmiş cDNA örnek ekleyin. Pipetleme ile karıştırın ve 37 °C ' de 30 dak. 4 °C ' de tutun.
- SPRı manyetik boncuk kullanarak örnek arındırın. 45 μL düşüş reaksiyonu ve pipetleme ile Mix için 81 μL SPRı boncuk ekleyin. , 2.6.1-2.6.6 SPRı boncuk arıtma için açıklandığı gibi arıtma adımlarını izleyin. 42 μL su kullanarak elute açıklandığı gibi.
9.5 ' bağlayıcı ligation
- Arıtılmış cDNA örneğini vakum yoğunlaştırıcıyı kullanarak 4 μL 'ye konsantre olun. Sıcaklığı 30 – 35 °C ' de tutun. Bir pipet kullanarak ses seviyesini test edin. Örnek bütünlüğü için kurutulduğunda, 4 μL su ekleyerek çözülür.
Not: Bu numune kaybını önlemek için bütünlüğü için kuruma önlemek için daha iyidir. - Konsantre numuneyi 95 °C ' de 5 dakika boyunca inkübasyon yapın ve renaturasyonu önlemek için hemen 2 dakika buz üzerine yerleştirin. Kapaklar basınç birikme nedeniyle pop-off olabilir gibi tüpler aktarırken kapakları tutun.
- 2,5 μM 5 ' ' in 4 μL 'sini 55 °C ' de 5 dakika boyunca inkübasyon yapın ve renaturasyonu önlemek için hemen 2 dakika buz üzerine yerleştirin.
- 4 μL 2,5 μM 5 ' bağlayıcı ile 4 μL örnekteki karıştırın.
Not: 5 ' bağlayıcı Tamamlayıcı tablo 2, tamamlayıcı tablo 3, tamamlayıcı tablo 4ve Tamamlayıcı Tablo 5göre hazırlanmalıdır. Kullanmadan önce 100 mM NaCl kullanarak 2,5 μm konsantrasyonuna 10 μM 5 ' bağlayıcı seyreltin. - Karma 5 ' bağlayıcı ve örnek için 16 μL ligasyon Premiks (bkz. malzeme tablosu) ekleyin ve pipetleme ile iyi karıştırın. 16 saat 16 °C ' de inkulat.
- SPRı manyetik boncuk kullanarak ligasyon karışımı arındırın. 43,2 μL SPRı boncuk ekleyin ve adımları, 2.6.1 – 2.6.6 izleyin. 42 kullanarak açıklandığı gibi elute 37 °C ' de önceden ısıtılan su μL.
- Transfer edilen süpernatant (1.8:1 boncuk örnek oranı) için 72 μL SPRı boncuklar ekleyerek 9,6 adımda yapılan arıtma tekrarlayın.
Not: 5 ' linkers sıralamadan önce sekiz numune havuzu sağlayan barkodlar içerir (sekiz trinükleotitler barkodlar mevcuttur, Murata ve ark.11 ve Tamamlayıcı Tablo 1' de açıklandığı gibi).
10.3 ' bağlayıcı ligation
- 9,1 adımda açıklandığı gibi vakum yoğunlaştırıcıyı kullanarak arıtılmış numuneyi 4 μL 'ye konsantre olun.
- Konsantre numuneyi 95 °C ' de 5 dakika boyunca inkübasyon yapın ve renaturasyonu önlemek için hemen 2 dakika buz üzerine yerleştirin. Kapaklar basınç birikme nedeniyle kapalı açılır gibi tüpler aktarırken kapakları tutun.
- 2,5 μM 3 ' ' lik 4 μL 'i 65 °C ' de 5 dakika boyunca inkübasyon yapın ve renaturasyonu önlemek için hemen 2 dakika buz üzerine yerleştirin.
- 2,5 μM 3 ' den 4 μL 'ye konsantre numunenin 4 μL 'sini ekleyin.
- Ligasyon Premiks 16 μL ekleyin ve pipetleme ile iyi karıştırın. 16 saat 16 °C ' de inkulat.
- SPRı manyetik boncuk kullanarak ligasyon karışımı arındırın. 43,2 μL SPRı boncuk ekleyin ve adımları, 2.6.1 – 2.6.6 izleyin. 42 kullanarak açıklandığı gibi elute 37 °C için önceden ısıtılmış su μL.
Not: 3 ' bağlayıcı Tamamlayıcı tablolar 6 ve ek tablo 7göre hazırlanmalıdır. 100 mM NaCl kullanarak 2,5 μM konsantrasyonuna 10 μM 3 ' bağlayıcı seyreltir.
11. dephosphorilasyon
- SAP karışımını 4 μL su, 5 μL 10X SAP tamponu ve 1 μL SAP enzimini birleştirerek hazırlayın.
- Temizleşmiş birleştirilmiş örneğe 10 μL SAP Mix ekleyin (Toplam hacim 50 μL) ve aşağıdaki programı kullanarak termocycler içinde inküye: 37 °C 30 dakika, 65 °C için 15 dakika ve 4 °C ' de tutun.
12. urasil özel eksizyon enzim kullanarak 3 ' bağlayıcı üst Strand bozulması
- 2 μL urasil spesifik eksizyon enzim ekleyin ( malzeme tablosunabakın) dephosphorylated örneğe, pipetleme ile karıştırın ve aşağıdaki programı kullanarak termocycler içinde kuluçk: 37 °c için 30 dakika, 95 °c için 5 dakika, ve hemen buz üzerinde yer 2 dakika , parçalanmış üst iplikteki retavlama önlemektir.
- 52 μL karışımı için 93,6 μL SPRı manyetik boncuk ekleyerek reaksiyon karışımı arındırın ve pipetleme ile iyi karıştırın. Tekrar arıtma adımları 2.6.1 – 2.6.6. 42 μL su ile elute, 37 °C ' de önceden ısıtılmıştır.
13. ikinci Strand sentezi
- 5 μL 10X DNA polimeraz reaksiyon tamponu, 2 μL su, 1 μL 10 mM dNTPs, 1 μL 50 μM nAnT-ıcage ikinci iplikli astar (dizi ek tablo 1' de) ve 1 μL D 'yi birleştirerek ikinci iplik sentezi karışımını hazırlayın (numune başına hacim ifade edilir) NA exonuclease-eksikliği polimeraz ( malzeme tablosundaönerilen polimeraz bakın).
- Arıtılmış örneğe 10 μL karışımı ekleyin ve pipetleme ile iyi karıştırın (Toplam hacim 50 μL 'dir). Aşağıdaki programı kullanarak termal bisikletçi içinde inküye: 95 °C için 5 dk, 55 °C için 5 dk, 72 °C 30 dakika ve 4 °C ' de tutun.
14. Exonuclease ı kullanarak Ikinci Strand sentezi primer bozulması
- 1 μL Exonuclease ı ikinci iplik sentezi karışımı ekleyin. Pipetleme ile iyi karıştırın ve 37 °C ' de 30 dakika boyunca 4 °C ' de tutarak kuluçk yapın.
- Exonuclease ı-tedavi numunesi 51 μL 'ye 91,8 μL SPRı manyetik boncuk ekleyerek çift telli DNA 'Yı arındırın. 2.6.1 – 2.6.6 'de açıklanan arıtma adımlarını tekrarlayın. ve 42 μL su ile elute, açıklandığı gibi 37 °C ' ye kadar ısıtılır.
- 9,1 adımda açıklandığı gibi, vakum yoğunlaştırıcıyı 15 μL 'ye kullanarak numuneyi konsantre olun.
15. kalite ve miktar kontrolü
- Konsantre örneklerden 1 μL kullanın ve DNA kalite analizörü üzerinde yüksek hassasiyet DNA yongası çalıştırın. Beklenen profil/miktar Şekil 3' te sunulmuştur.
16. taşıyıcı düşüşün ilk turu
- 2 μL su, 2 μL 10X kısıtlama enzim tamponu, 1 μL ı-SceI ve 1 μL ı-CeuI ' i birleştirerek bozulma karışımını hazırlayın.
- Konsantre numunenin 14 μL 'ye 6 μL azalma karışımı ekleyin ve pipetleme ile karıştırın. 37 °c ' de 3 saat, 65 °C ' de 20 dakika devre dışı bırakma ve 4 °C ' de tutun.
- SPRı manyetik boncuklar kullanarak bozulma karışımı arındırın. Bozulma karışımı hacmini artırmak için 5 μL su ekleyin ve 45 μL SPRı boncuk (1.8:1 boncuk numune oranı) ekleyin. Bu adımda açıklandığı gibi arıtma yineleyin 2.6.1 – 2.6.6. ve 42 μL su ile elute, 37 °C ' ye ısıtılır.
- 9,1 adımda açıklandığı gibi toplam birimin 42 μL 'den 20 μL 'ye kadar olan eltli numuneyi konsantre olun.
17. bozulma seviyesinin kontrolü ve PCR amplifikasyon döngülerinin sayısını belirleme
- Tüm kitaplıkları (adaptör karışımı) yükseltmek için qPCR karışımını hazırlayın. 3,8 μL su, 5 μL qPCR Premiks (2x) birleştirmek, 0,1 μL 10 μm adaptor_f1 Primer (5 '-aatgatacggcgaccaccga-3 ') ve 0,1 μL 10 μm adaptor_r1 Primer (5 '-caagcagaagacggcatacga-3 ') her numune için (bkz. önerilen qPCR Premiks için malzeme tablosu ).
- 9 μL qPCR adaptör karışımını, adım 16,4 ' dan 1 μL örnek ile birleştirin ve pipetleme ile iyi karıştırın.
- Taşıyıcı (taşıyıcı Mix) türetilen DNA amplifikasyon için qPCR Mix hazırlayın. Her numune için 3,8 μL su, 5 μL qPCR Premiks (2x), 0,1 μL 10 μm carrier_f1 Primer (5 '-gcggcagcgttcgctataac-3 ') ve 0,1 μL 10 μm adaptor_r1 astar birleştirin
- 9 μL qPCR taşıyıcı karışımı ile 1 μL örnek adım 16,4 ile birleştirin ve pipetleme ile iyi karıştırın.
- QPCR programını ayarlayın: 95 ° c için 3 dakika (95 °C için 20 s, 60 °C için 20 s, 72 °C 2 dk) tekrarlanan 40X, ardından enstrümana özgü denaturasyon eğrisi (65 – 95 °C) ve 4 °C ' de tutun.
Not: Numuneyi suyla değiştirerek negatif bir kontrol hazırlayın.
18. hedef kütüphanenin PCR amplifikasyonu
- 6 μL su, 0,5 μL 10 μm adaptor_f1 astar, 0,5 μL 10 μm adaptor_r1 astar ve 25 μL PCR Premiks (2x) birleştirerek PCR amplifikasyon karışımını hazırlayın. Pipetleme ile karıştırın (önerilen PCR premix için malzeme tablosuna bakın).
- Ekle 32 μL PCR Mix 18 μL için adım 16,4 örnek. Pipetleme ile iyice karıştırın.
- PCR amplifikasyonu ayarlayın: 95 °C için 3 dakika, (98 °C için 20 s, 60 °C için 15 s, 72 °C 2 dk) 12-18 döngüleri, 72 °C 2 dak ve 4 °C ' de tutun.
Not: PCR döngülerinin tam sayısı qPCR sonuçları tarafından belirlenir ve adaptör astar karışımı ile elde edilen CT değerine karşılık gelir (PCR döngülerinin sayısı CT değerine eşittir). - Güçlendirilmiş numunenin 50 μL 'ye 90 μL SPRı manyetik boncuk ekleyerek amplifikatördeki numuneyi arındırın ve pipetleme ile iyice karıştırın. Bu adımda açıklanan arıtma adımlarını yineleyin 2.6.1 – 2.6.6. ve örnek 42 μL su kullanılarak açıklandığı gibi elute.
19. ikinci tur taşıyıcı bozulması
- 16.1 – 16.3 arasındaki adımları yineleyin.
- SPRı manyetik boncuklar kullanarak bozulma karışımı arındırın. Hacmi artırmak ve 30 μL SPRı boncuk (1:1 boncuk numune oranı) ile karıştırın için örneğe 10 μL su ekleyin. Bu adımda açıklandığı gibi arıtma yineleyin 2.6.1 – 2.6.6. ve 42 μL su ile elute, 37 °C ' de önceden ısıtılmıştır.
- 42 μL 'den% 30 μL 'ye kadar olan eltilmiş numuneyi toplam hacmine konsantre olun.
20. kitaplık boyutu seçimi
- 24 μL SPRı manyetik boncuk 30 μL örnek adım 19,3 ile karıştırın. (0.8:1 boncuk örnek oranı). Bu adımlar 2.6.1 – 2.6.6 içinde açıklandığı gibi arıtma adımlarını tekrarlayın. ve 42 μL su örneğinden açıklandığı gibi elute.
- Örnek yaklaşık 14 μL 9,1 adımda açıklandığı gibi konsantre.
21. kalite kontrol
-
Boyut dağılımı değerlendirmesi
- Yüksek hassasiyet DNA yongası üzerinde örnek 1 μL çalıştırın. Beklenen sonuçlar Şekil 4' te sunulmuştur.
Not: 200 BP 'den daha kısa parçalar görünüyorsa ( Şekil 4A, C'de örneğe bakın), boyut seçimi (: 20.1 – 20.2 adımları) kısa parçalar kaldırılıncaya kadar tekrarlanmalıdır (Şekil 4b, D). Genellikle ek bir boyut seçimi turu yeterlidir. Kısa parça miktarı şiddetli ise ( Şekil 4c'de olduğu gibi), boncuk-numune oranı 0.6:1 ' e düşürülmelidir.
- Yüksek hassasiyet DNA yongası üzerinde örnek 1 μL çalıştırın. Beklenen sonuçlar Şekil 4' te sunulmuştur.
-
Taşıyıcı bozulması kalite kontrolü
- 17.1 – 17.5 arasındaki adımları yineleyin.
Not: HS DNA çip çalıştırma (bölge Analizi) tahmini kütüphaneler konsantrasyonuna bağlı olarak, numunelerin qPCR önce seyreltilmiş olması gerekir. Numune kaybını önlemek ve su içinde 100 – 500x seyreltmeli (1 – 20 pg/μL son konsantrasyona seyreltmeli) için örnek 0,5 μL kullanın. Adaptör ve taşıyıcı karışımı ile elde edilen CT değerleri arasında beklenen fark 5 – 10 ' dir.
- 17.1 – 17.5 arasındaki adımları yineleyin.
-
Kitaplık ölçümü
- 100 mg/mL lambda DNA standardını 980 μL 1x TE ile karıştırarak lambda DNA standardının çalışma seyreltmesini hazırlayın (DNA ölçme kiti içinde sağlanan 20 x TE seyreltme ile hazırlayın). Lambda DNA 'Sı seyreltme-20 °C ' de depolanabilir.
- Seyreltilmiş lambda standardını ve 1x TE 'yi Tamamlayıcı tablo 8' e göre KARıŞTıRARAK lambda DNA standardının seri dilüslerine hazırlanın.
Not: Daha yüksek doğruluk için, tüm tüplere 100 μL 1x TE tampon eklenmeli ve eklenecek seyreltilmiş lambda hacmi başına gerekli olarak 1x TE hacminin kaldırılması önerilir. Kütüphanenin 1 μL 'den fazlasını kullanmayın; Bu ölçüm için 384 iyi plaka kullanımı önerilir.
Sonuçlar
Bu raporda, toplam RNA malzemesi başlangıç nanogramlardan sıralı hazır kütüphaneler elde etmek için tam dılım kafes Protokolü açıklanmaktadır (Şekil 1). Sentetik RNA taşıyıcı karışımı elde etmek için, önce PCR taşıyıcı şablonlarının hazırlanması ve PCR yan ürünlerini ortadan kaldırmak için jel arıtması gerekir (Şekil 2a). Her PCR şablonu (Toplam on) ortak bir ileri kullanılarak üretilir, ancak farklı bir ters astar (Tablo 2), sentetik RNA taşıyıcıları boyutu değişkenliği sağlamak için PCR şablonunun farklı uzunluklarına yol. Bir kez arıtılmış, PCR şablonları taşıyıcı moleküllerin in vitro transkripsiyon için kullanılır. Şablonlar jel arıtılırsa tek RNA taşıyıcı ürün bekleniyor (bkz. Şekil 2B'de temsili jel Analizi). Taşıyıcının hazırlanması, ihtiyaca bağlı olarak ve gelecekteki kullanım için-80 °C ' de hazırlanmış, karışık ve dondurulmuş olduğunda yükseltilebilir.
16-18 PCR amplifikasyonu döngüleri ile birlikte önerilen en az miktarda numune toplam RNA (10 ng) kullanarak, yüksek karmaşıklık dılım-kafes kütüphaneler elde edilebilir. Son kütüphaneyi yükseltmek için gereken PCR döngülerinin sayısı çok kullanılan toplam giriş RNA sayısına bağlıdır (beklenen döngü sayısı Tablo 4' te sunulmuştur).
İlk düşüş turunun ardından, qPCR sonuçlarında (adım 17), adaptor_f1 veya carrier_f1 primer kullanılarak elde edilen CT değerleri arasındaki beklenen fark 1-2, adaptor_f1 ile carrier_f1 'den daha düşük olan CT değerleri ile elde edilir.
Son kütüphanede parça uzunlukları dağılımı 700-900 BP ortalama parça boyutu ile 200-2000 BP arasındadır (Bioanalyzer yazılımı kullanarak bölge analizine dayalı, Şekil 4b, D). Şekil 4A, C'de sunulan daha kısa parçalar, boyut-dışlama ek Tur tarafından kaldırılması gerekir (Steps 20-21). Bu kısa parçalar PCR amplifikasyon eserleri ve hedef kütüphanesidir. Daha kısa parçaları sıralama akışı hücrelerinde daha iyi küme ve sıralama sorunlarına neden olabilir unutmayın.
Örnek başına elde edilen Kütüphane malzemesi beklenen miktarda 5-50 ng arasındadır. Önemli ölçüde daha düşük tutarlar protokol sırasında örnek kaybın göstergesidir. Elde edilen düşük miktar sıralama için yeterli ise (2-3 ng havuzlanmış kitaplıkları gerekli), kitaplıkları daha düşük karmaşıklık olabilir (aşağıya bakın).
Sıralı makineye bağlı olarak, akış hücresine yüklenen kütüphanenin miktarı optimize edilmesi gerekebilir. Bir ıllıina HiSeq 2500 kullanarak, 8-12 pM SLıC-CAGE kütüphaneler yükleme ortalama 150-200 milyon okuma verir, >% 80 ile kalite puanı Q30 eşik olarak geçen okur.
Elde edilen okur daha sonra referans genomuna eşleştirilir [için 50 BP okur, Bowtie212 çekirdek sırası başına sıfır uyuşmazlık (22 BP)] izin varsayılan parametrelerle kullanılabilir. Beklenen eşleme verimliliği toplam RNA giriş miktarına bağlıdır ve Tablo 5' te sunulur. Benzersiz olarak eşlenen okuma daha sonra R grafik ve istatistiksel bilgi işlem ortamına yüklenebilir13 ve Cager (bioconductor paketi14) kullanılarak işlenir. Paket sahne takip etmek kolaydır ve iş akışı ve ayrıntılı olarak eşlenen verilerin işlenmesi açıklar. Kütüphane karmaşıklığının kolay bir görsel kontrolü, düşük karmaşıklık kitaplıkları yapay olarak dar promotörler (Şekil 5A, toplam RNA 1 ng dan türetilen dilim-kafes Kütüphanesi olacak gibi, organizatör genişliğinin dağılımı, Ayrıntılar için önceki görmek yayını10). Ancak, hatta düşük karmaşıklığı SLıC-CAGE kütüphaneler gerçek CTSSs, düşük/orta giriş TSS haritalama (Şekil 5B, C) için alternatif yöntemlerden daha fazla hassasiyet ile tanımlaması sağlar.
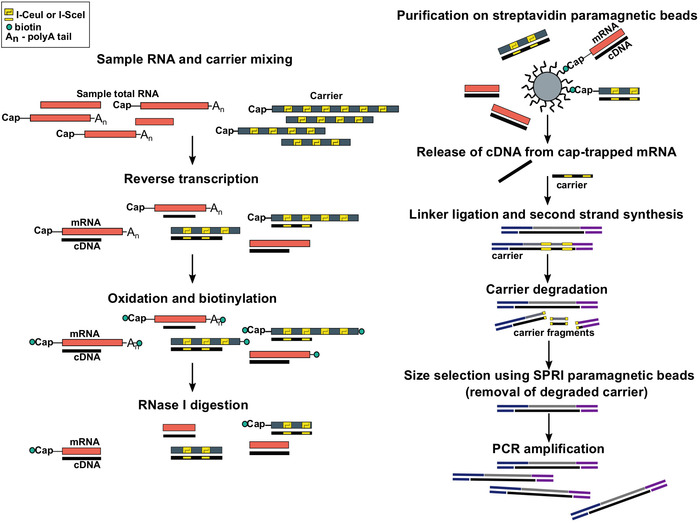
Şekil 1: SLıC-Cage protokolünde adımlar. Örnek RNA, 5 μg toplam RNA malzemesi elde etmek için RNA taşıyıcı karışımı ile karıştırılır. cDNA Ters transkripsiyon ile sentezlenir ve kap sodyum periodat kullanılarak oksitlenir. Oksidasyon biotin hidazid kullanarak kapağın biotin eki sağlar. Biotin mRNA 's bağlı alır 3 ′ End, aynı zamanda sodyum periodate kullanılarak oksitlenmiş olduğu gibi. MRNA gelen biotin ortadan kaldırmak için: cDNA tamamen sentezlenmiş CDA ile melez ve 3 ′ den mRNA biter, örnekler RNase ı. cDNA ile tedavi edilir 5 ulaştı ′ mRNA sonuna kadar daha sonra streptavidin manyetik boncuk üzerinde yakınlık arıtma tarafından seçilir ( Cap-bindirme). CDNA serbest bıraktıktan sonra, 5 ′-ve 3 ′-linkers bağlandı. Taşıyıcı kaynaklanan Kütüphane molekülleri ı-scei ve ı-ceui homing restriksiyon endonükleazlarına benzer ve parçaları SPRI manyetik boncuklar kullanılarak kaldırılır kullanılarak bozulur. Kütüphane daha sonra PCR amplifikatörlenmiştir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
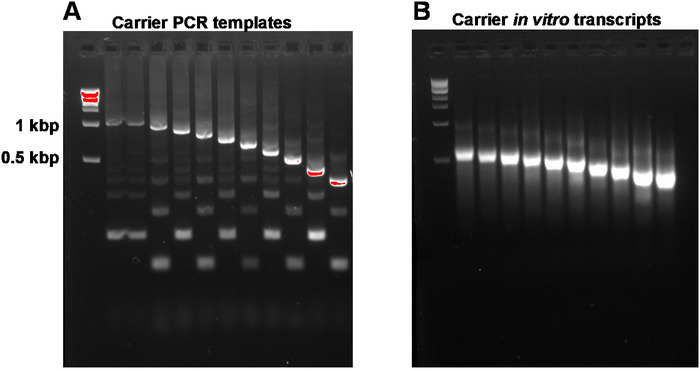
Şekil 2: taşıyıcı PCR şablonları ve taşıyıcı in vitro transkriptler temsilcisi jel-analizi. (A) TAŞıYıCı PCR şablonları jel arıtma önce: ilk iyi içerir 1 KBP Marker, takip taşıyıcı PCR şablonları 1, 1-10. (B) taşıyıcı in vitro transkriptler: ilk iyi 1 KBP Marker içerir, taşıyıcı transkriptler 1-10 takip. Taşıyıcı transkriptler, yüklemeden önce 95 °C ' de 5 dakika ısıtarak denattandı. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
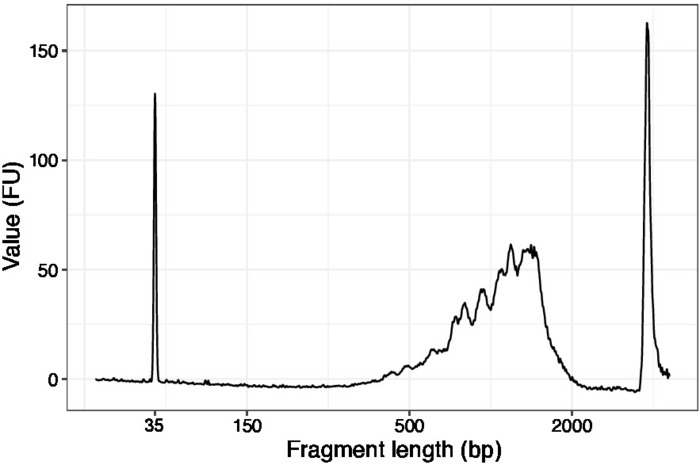
Şekil 3: temsılcı DNA kalitesi (yüksek HASSASIYET DNA çip) taşıyıcı bozulması ilk turda önce dilim-kafes izi. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
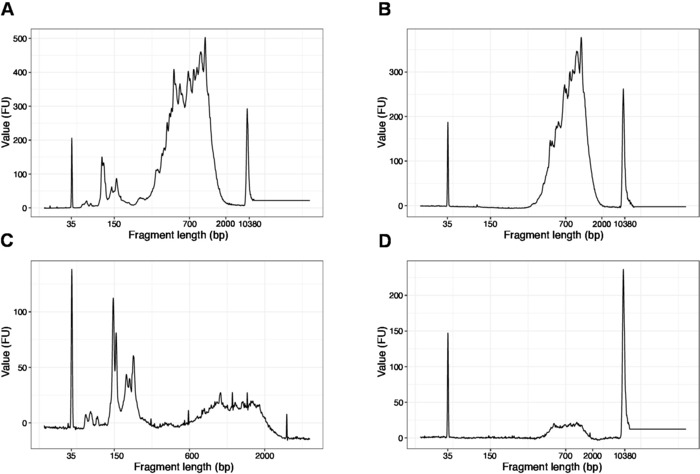
Şekil 4: PCR amplifikasyonu sonrasında temsılcı DNA kalitesi (yüksek HASSASIYET DNA çipi) dilim-kafes kütüphanelerinin izleri. (A) kısa parçaların çıkarılması için ek boyut seçimi GEREKTIREN dilim kafes Kütüphanesi. (B) boyut seçiminden sonra 0.6 x SPRI boncukları kullanarak dılım-kafes Kütüphanesi örnek oranı. (C) kısa parça çıkarılması için boyut seçimi gerektiren alt çıkış miktarının dılım-kafes Kütüphanesi. (D) boyut-seçim sonra 0.6:1 SPRI boncuk örnek oranı kullanarak daha düşük çıkış MIKTARı dilim kafes Kütüphanesi. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
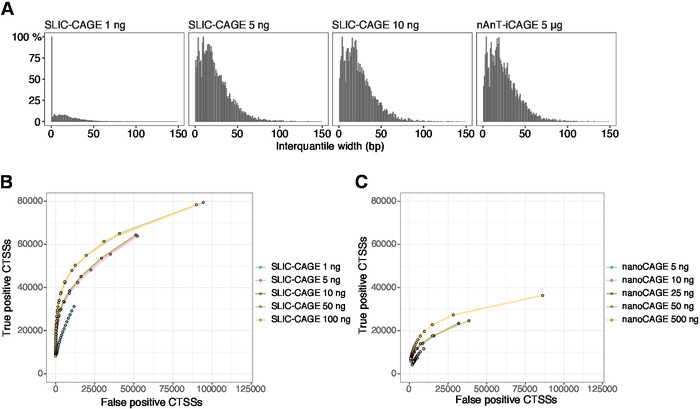
Şekil 5: SLıC-Cage kütüphanelerinin doğrulanması. (A) 1, 5 veya 10 ng s. cerevisiae toplam RNA 'dan hazırlanan ve 5 μg s. cerevisiae toplam RNA 'Dan hazırlanan nAnT-ıCAGE KÜTÜPHANESINDE bulunan dilim-kafes kütüphanelerinde etiket kümesi interquantile genişlikleri dağılımı. 1 ng SLıC-CAGE kitaplığında yüksek miktarda dar etiket kümeleri, düşük karmaşıklığı gösterir. (B) S. cerevisiae dilim-kafes kütüphanelerinde CTSS tanımlaması için Roc eğrileri. Tüm S. cerevisiae NAnT-ıcage CTSSS gerçek bir set olarak kullanıldı. (C) S. cerevisiae nanocage kütüphanelerinde CTSS tanımlaması için Roc eğrileri. Tüm S. cerevisiae NAnT-ıcage CTSSS gerçek bir set olarak kullanıldı. ROC eğrilerinin karşılaştırılması, SLıC-CAGE ' in CTSS kimliğinde nanoCAGE 'yi güçlü bir şekilde gerçekleştirdiğini gösterir. ArrayExpress E-MTAB-6519 verileri kullanıldı. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
Tablo 1: taşıyıcı sentetik geni dizisi. I-SceI siteleri kalın ve mor olarak italik ve ı-CeuI tanınırları siteleri yeşil. Bu dosyayı indirmek Için lütfen buraya tıklayın.
| Taşıyıcı | ters astar 5 '-3 ' | PCR ürün uzunluğu/BP | |
| 1 | PCR_N6_r1: NNNNNNCTACGTGTCGCAGACGAATT | 1034 | |
| 2 | PCR_N6_r2: NNNNNNTATCCAGATCGTTGAGCTGC | 966 | |
| 3 | PCR_N6_r3: NNNNNNCACTGCGGGATCTCTTTACG | 889 | |
| 4 | PCR_N6_r4: NNNNNNGCCGTCGATAACTTGTTCGT | 821 | |
| 5 | PCR_N6_r5: NNNNNNAGTTGACCGCAGAAGTCTTC | 744 | |
| 6 | PCR_N6_r6: NNNNNNGTGAAGAATTTCTGTTCCCA | 676 | |
| 7 | PCR_N6_r7: NNNNNNCTCGCGGCTCCAGTCATAAC | 599 | |
| 8 | PCR_N6_r8: NNNNNNTATACGCGATGTTGTCGTAC | 531 | |
| 9 | PCR_N6_r9: NNNNNNACCGCCGCGCCTTCCGCAGG | 454 | |
| 10 | PCR_N6_r10: NNNNNNCAGGACGTTTTTGCCCAGCA | 386 | |
| * Forward astar tüm taşıyıcı şablonları için aynıdır. Altı çizili T7 Organizatör dizisidir. PCR_GN5_f1: TAATACGACTCACTATAGNNNNNCAGCGTTCGCTA | |||
Tablo 2: taşıyıcı şablon amplifikasyon Için astar. İleri astar tüm taşıyıcı şablonları için aynıdır. Altı çizili T7 Organizatör dizisidir. PCR_GN5_f1: Taatacgactcactatagnnnnncagcgttcgcta. Farklı ters astar kullanarak, PCR şablonları ve dolayısıyla farklı uzunlukta taşıyıcı RNAs üretilmektedir.
| Taşıyıcı | Uzun -luğu | unapped/μg | capped/μg |
| 1 | 1034 | 3,96 | 0,45 |
| 2 | 966 | 8,36 | 0,95 |
| 3 | 889 | 4,4 | 0,5 |
| 4 | 821 | 6,6 | 0,75 |
| 5 | 744 | 4,4 | 0,5 |
| 6 | 676 | 3,08 | 0,35 |
| 7 | 599 | 4,4 | 0,5 |
| 8 | 531 | 3,96 | 0,45 |
| 9 | 454 | 2,64 | 0,3 |
| 10 | 386 | 2,2 | 0,25 |
Tablo 3: RNA taşıyıcı karışımı. Toplam 49 μg taşıyıcı karışımı 0.3-1 KBP: unapped = 44 μg, capped = 5 μg.
| Toplam RNA girişi/ng | PCR döngüleri |
| 1 Bisiklet | 18 |
| 2 ng üzerinde | 17 |
| 5 adede kadar | 16 |
| 10 ng | 15-16 |
| 25 ng 'ye kadar | 14-15 |
| 50 | 13-15 |
| 100 | 12-14 |
Tablo 4: Örnek Toplam RNA girişine bağımlılığında beklenen PCR döngüsü sayısı. Döngülerin yaklaşık sayısı, Saccharomyces cerevisiae, Drosophila melanogaster ve Mus musculus Total RNA kullanılarak gerçekleştirilen deneylere dayanır.
| Toplam RNA girişi/ng | % genel eşlenen | % benzersiz olarak eşlenen | % taşıyıcı |
| 1 Bisiklet | 30 | 20-30 | 30 |
| 2 ng üzerinde | 60 | 20-50 | 10 |
| 5 adede kadar | 60-70 | 40-60 | 5-10 |
| 10 ng | 60-70 | 40-60 | 5-10 |
| 25 ng 'ye kadar | 65-80 | 40-70 | 0-5 |
| 50 | 65-80 | 40-70 | 0-3 |
| 100 | 70-85 | 40-70 | 0-2 |
Tablo 5: beklenen eşleme verimliliği ve toplam RNA giriş tutarının bağımlılığı. Yaklaşık sayılar sunulur ve Saccharomyces cerevisiae ve Mus musculus Total RNA kullanılarak gerçekleştirilen deneylere dayanır.
Tartışmalar
Başarılı dılım kafes Kütüphane preparatları için, örnek adsorpsiyon nedeniyle numune kaybını önlemek için düşük ciltli ipuçları ve tüpler kullanmak önemlidir. Supernatant alma içeren tüm adımlarda, tüm hacmi kurtarmak için tavsiye edilir. Protokolün birden çok adımı olduğu için, sürekli örnek kaybı başarısız kitaplıklara yol açacaktır.
CAGE (nAnT-ıcage) rutin olarak yapılmadı ise, aynı toplam RNA örneğinin farklı giriş tutarları (10 ng, 20 ng, 50 ng, 100 ng, 200 ng) ile SLıC-CAGE test etmek ve toplam RNA 5 μg kullanılarak hazırlanan nAnT-ıcage kütüphaneleri karşılaştırmak en iyisidir. NAnT-ıcage Kütüphanesi başarısız olursa (numune başına elde edilen DNA kütüphanesinin 0,5-1 ng 'den az), dılım-kafes çalışma olasılığı düşüktür ve örnek kaybın minimize edilmesi gerekir.
Bozulmuş RNA veya rRNA yoksun yüksek kaliteli kütüphaneler sağlamak için kritik bir adım Bölüm 7 ' de açıklanan kap bindirme olduğunu. Streptavidin boncuk iyice yıkama tamponları resuspended ve yıkama tamponları önce bir sonraki yıkama adım veya cDNA elüsyon devam etmek kaldırılır önemlidir.
Taşıyıcı bozulması ilk turda qPCR sonuçları adaptor_f1 ve carrier_f1 astar kullanımı arasında hiçbir fark gösteriyorsanız, protokol devam hala önerilir. Taşıyıcı bozulması ikinci turda sonra, CT değerleri farkı beşten az, taşıyıcı bozulması üçüncü turda önerilir. Biz bozulması gerekli üçüncü bir turda bulamadık ve oluşursa, bu homing endonükleaz stokları yerine tavsiye edilir.
Alınan kütüphanenin son tutarı sıralama için yeterli değilse, ek PCR amplifikasyon turlarında protokole eklenebilir. PCR amplifikasyonu daha sonra boyut seçimi kaçınılamaz dikkate örnek kaybına alarak, sıralama için yeterli malzeme verim için gereken en az sayıda amplifikasyon döngüleri ile ayarlanabilir. SPRı manyetik boncuk kullanarak arıtma veya boyut seçimi daha sonra tüm küçük (< 200 BP) parçaları kaldırılıncaya kadar yapılmalıdır (gerekirse, 0.6:1 boncuk örnek oranı kullanın), ve Kütüphane Picogreen kullanılarak nicelik olmalıdır.
Kitaplıkları tek uç veya eşleştirilmiş uç modunda sıralanabilir. Eşleştirilmiş uç sıralamasını kullanarak, transkript izoformlarının hakkında bilgi elde edilebilir. Buna ek olarak, Ters transkripsiyon rasgele bir astar kullanılarak gerçekleştirilir gibi (TCT-N6, n6 rasgele bir hexamer olmak), sıralı 3 '-End bilgileri benzersiz moleküler TANıMLAYıCıLARı olarak kullanılabilir (UMı) PCR çoğaltır daraltmak için. Orta sayıda PCR amplifikasyon döngüsü kullanıldığında (18 ' e kadar), Umıs 'in kullanımı daha önce gereksiz olarak bulunmuştur.
Protokolün çekirdeği nAnT-ıcage11' e dayanır, dılım-Cage sekiz barkodları kullanır. Bu nedenle, sekiz örneklerden fazla çoğullama Şu anda desteklenmiyor. Buna ek olarak, hem dilim-kafes ve nAnT-ıcage Rnas 200 BP 'den daha kısa yakalamak için uygun değildir, protokolleri ampure XP boncuklar ile boyut-dışlama yoluyla Linkler ve PCR eserler kaldırmak için tasarlanmıştır gibi.
DILIM-kafes toplam RNA malzemesi Nanogramlar kullanarak transkripsiyon başlatılması başlangıç siteleri haritalama için tek tarafsız düşük giriş tek nüklemotid çözünürlük yöntemidir. Alternatif Yöntemler, Cap-bindirme (örn., nanocage15 ve nanopare16) yerine barkodlu RNA 'ya ters transkriptaz etkinliğinin geçiş şablonuna dayanır. Şablon geçişi nedeniyle, bu yöntemler tsss algılama serisine özgü önyargıları sergiler, yanlış pozitif tsss sayısı arttı ve gerçek tsss9,10azalan numaraları yol.
Açıklamalar
Çözünebilir taşıyıcı RNA/DNA için bir patent doldurulur.
Teşekkürler
Bu çalışma, B. L. ve tıbbi araştırma Konseyi (MRC) çekirdek finansmanı (MC-A652-5QA10) tarafından verilen Wellcome Trust Grant (106954) tarafından destekleniyordu. N. C. EMBO uzun dönem Bursu (EMBO ALTF 1279-2016) tarafından destekleniyordu; E. P. Tıbbi Araştırma Konseyi INGILTERE tarafından destekleniyordu; B. L. Tıbbi Araştırma Konseyi INGILTERE tarafından desteklenmektedir (MC UP 1102/1).
Malzemeler
| Name | Company | Catalog Number | Comments |
| 2-propanol, Bioultra, for molecular biology, ≥99.5% | Sigma-Aldrich | 59304-100ML-F | Used in RNAclean XP purification. |
| 3' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 3'linkers is described in the supplementary of this protocol. | ||
| 5' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 5'linkers is described in the supplementary of this protocol. | ||
| Agencourt AMPure XP, 60 mL | Beckman Coulter | A63881 | Purification of DNA |
| Agencourt RNAClean XP Kit | Beckman Coulter | A63987 | Purification of RNA and RNA:cDNA hybrids in CAGE steps. |
| Axygen 0.2 mL Polypropylene PCR Tube Strips and Domed Cap Strips | Axygen (available through Corning) | PCR-0208-CP-C | Or any 8-tube PCR strips (used only for water and mixes). |
| Axygen 1 x 8 strip domed PCR caps | Axygen (available through Corning) | PCR-02CP-C | Caps for PCR plates. |
| Axygen 1.5 mL Maxymum Recovery Snaplock Microcentrifuge Tube | Axygen (available through Corning) | MCT-150-L-C | Low-binding 1.5 mL tubes, used for enzyme mixes or sample concentration. |
| Axygen 96 well no skirt PCR microplate | Axygen (available through Corning) | PCR-96-C | Low-binding PCR plates - have to be used for all steps in the protocol. Note that plates should be cut to contain 2 x 8 wells for easier visibility of the samples |
| Bioanalyzer (or Tapestation): RNA nano and HS DNA kits | Agilent | To determine quality of RNA, efficient size selection and final quality of the library (Tapestation can also be used) | |
| Biotin (Long Arm) Hydrazide | Vector laboratories | SP-1100 | Biotinylation/tagging |
| Cutsmart buffer | NEB | Restriction enzyme buffer | |
| Deep Vent (exo-) DNA Polymerase | NEB | M0259S | Second strand synthesis |
| DNA Ligation Kit, Mighty Mix | Takara | 6023 | Used for 5' and 3'-linker ligation |
| dNTP mix (10 mM each) | ThermoFisher Scientific | 18427013 | dNTP mix for production of carrier templates (or any dNTPs suitable for PCR) |
| Dynabeads M-270 Streptavidin | Invitrogen | 65305 | Cap-trapping. Do not use other beads as these are optimised with the buffers used. |
| DynaMag-2 Magnet | ThermoFisher Scientific | 12321D | Magnetic stand for 1.5 mL tubes - used to prepare Streptavidin beads. |
| DynaMag-96 Side Skirted Magnet | ThermoFisher Scientific | 12027 | Magnetic stand for PCR plates (96 well-plates) - used with cut plates to contain 2 x 8 wells. |
| Ethanol, BioUltra, for molecular biology, ≥99.8% | Sigma-Aldrich | 51976-500ML-F | Used in AMPure washes. Any molecular biology suitable ethanol can be used. |
| Exonuclease I (E. coli) | NEB | M0293S | Leftover primer degradation |
| Gel Loading Dye, Purple (6x), no SDS | NEB | B7025S | agarose gel loading dye |
| HiScribe T7 High Yield RNA Synthesis Kit | New England Biolabs | E2040S | Kit for carrier in vitro transcription |
| Horizontal electrophoresis apparatus | purification of carrier DNA templates from agarose gels | ||
| I-Ceu | NEB | R0699S | Homing endonuclease used for carrier degradation. |
| I-SceI | NEB | R0694S | Homing endonuclease used for carrier degradation. |
| KAPA HiFi HS ReadyMix (2x) | Kapa Biosystems (Supplied by Roche) | KK2601 | PCR mix for target library amplification |
| KAPA SYBR FAST qPCR kit (Universal) 2x | Kapa Biosystems (Supplied by Roche) | KK4600 | qPCR mix to assess degradation efficiency and requiered number of PCR amplification cycles |
| Micropipettes and multichannel micropipettes (0.1-10 µL, 1-20 µL, 20-200 µL) | Gilson | Use of Gilson with the low-binding Sorenson tips is recommended. Other micropippetes might not be compatible. Different brand low-binding tips may not be of equal quality and may increase sample loss. | |
| Microplate reader | For Picogreen concentration measurement of the final library. Microplates are used to allow small volume measurement and reduce sample waste. | ||
| nuclease free water | ThermoFisher Scientific | AM9937 | Or any nuclease (DNase and RNase) free water |
| PCR thermal cycler | incubation steps and PCR amplficication | ||
| Phusion High-Fidelity DNA Polymerase | ThermoFisher Scientific | F530S | DNA polymerase for amplification of carrier templates (or any high fidelity polymerase) |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Purification of carrier PCR templates from agarose gels. |
| qPCR machine | determining PCR amplification cyle number and degree of carrier degradation | ||
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher Scientific | P11495 | Used to measure final library concentration - recommended as, in our hands, it is more accurate and reproducible than Qubit. |
| Quick-Load Purple 100 bp DNA Ladder | NEB | N0551S | DNA ladder |
| Quick-Load Purple 1 kb Plus DNA Ladder | NEB | N0550S | DNA ladder |
| Ribonuclease H | Takara | 2150A | Digestion of RNA after cap-trapping. |
| RNase ONE Ribonuclease | Promega | M4261 | Degradation of single stranded RNA not protected by cDNA. |
| RNase-Free DNase Set | Qiagen | 79254 | Removal of carrier DNA templates after in vitro transcription. |
| RNeasy Mini Kit | Qiagen | 74104 | For cleanup of carrier RNA from in vitro transcription or capping |
| Sodium acetate, 1 M, aq.soln, pH 4.5 RNAse free | VWR | AAJ63669-AK | Or any nuclease (DNase and RNase) free solution |
| Sodium acetate, 1 M, aq.soln, pH 6.0 RNAse free | Or any nuclease (DNase and RNase) free solution | ||
| Sodium periodate | Sigma-Aldrich | 311448-100G | Oxidation of vicinal diols |
| Sorenson low binding aerosol barrier tips, MicroReach Guard, volume range 10 μL, Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719390-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 1,000 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719463-1000EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 20 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719412-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 200 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719447-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| SpeedVac Vacuum Concentrator | concentrating samples in various steps to lower volume | ||
| SuperScript III Reverse Transcriptase | ThermoFisher Scientific | 18080044 | Used for reverse transcription (1st CAGE step) |
| Trehalose/sorbitol solution | Preparation is described in Murata et al. 2014. | ||
| Tris-HCl, 1 M aq.soln, pH 8.5 | 1 M solution, DNase and RNase free | ||
| tRNA (20 mg/mL) | tRNA solution. Preparation is described in Murata et al. 2014. | ||
| UltraPure Low Melting Point Agarose | ThermoFisher Scientific | 16520050 | Or any suitable pure low-melt agarose. |
| USB Shrimp Alkaline Phosphatase (SAP) | Applied Biosystems (Provided by ThermoFisher Scientific) | 78390500UN | |
| USER Enzyme | NEB | M5505S | Degradation of 3'linker's upper strand, Uracil Specific Excision Reagent/Enzyme |
| Vaccinia Capping System | NEB | M2080S | Enzymatic kit for in vitro capping of carrier molecules |
| Wash buffer A | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer B | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer C | Cap trapping washes. Preparation is described in Murata et al. 2014. |
Referanslar
- Shiraki, T., et al. Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proceedings of the National Academy of Sciences of the United States of America. 100 (26), 15776-15781 (2003).
- Haberle, V., Lenhard, B. Promoter architectures and developmental gene regulation. Seminars in Cell and Developmental Biology. 57, 11-23 (2016).
- Haberle, V., Stark, A. Eukaryotic core promoters and the functional basis of transcription initiation. Nature Reviews Molecular Cell Biology. 19 (10), 621-637 (2018).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Celniker, S. E., et al. Unlocking the secrets of the genome. Nature. 459 (7249), 927-930 (2009).
- Consortium, F., et al. A promoter-level mammalian expression atlas. Nature. 507 (7493), 462-470 (2014).
- Boyd, M., et al. Characterization of the enhancer and promoter landscape of inflammatory bowel disease from human colon biopsies. Nature Communications. 9 (1), 1661(2018).
- Adiconis, X., et al. Comprehensive comparative analysis of 5'-end RNA-sequencing methods. Nature Methods. , (2018).
- Cvetesic, N., et al. SLIC-CAGE: high-resolution transcription start site mapping using nanogram-levels of total RNA. Genome Research. 28 (12), 1943-1956 (2018).
- Murata, M., et al. Detecting expressed genes using CAGE. Methods in Molecular Biology. 1164, 67-85 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9, 357(2012).
- R Core Team. A language and environment for statistical computing. , Available from: https://www.R-project.org/ (2017).
- Haberle, V., Forrest, A. R., Hayashizaki, Y., Carninci, P., Lenhard, B. CAGEr: precise TSS data retrieval and high-resolution promoterome mining for integrative analyses. Nucleic Acids Research. 43 (8), e51(2015).
- Poulain, S., et al. NanoCAGE: A Method for the Analysis of Coding and Noncoding 5'-Capped Transcriptomes. Methods in Molecular Biology. 1543, 57-109 (2017).
- Schon, M. A., Kellner, M. J., Plotnikova, A., Hofmann, F., Nodine, M. D. NanoPARE: parallel analysis of RNA 5' ends from low-input RNA. Genome Research. 28 (12), 1931-1942 (2018).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır