Method Article
长距离反向PCR对热线1的逆变活性的检测
摘要
本文概述了一种基于PCR的简单测定,用于监测活性LINE-1逆转录的活性,并绘制给定基因组中反逆转位图。使用 MCF7 细胞系,我们在此演示如何应用此方法来检测位于 22q12.1 的 LINE-1 的活动。
摘要
长穿插的核元素1(LINE-1)是人类基因组中唯一可以自主移动的移动遗传元素家族。他们通过一种称为逆转的过程来这样做,其中他们转录形成mRNA中间体,然后通过逆转录插入基因组。尽管在正常细胞中保持静音,LINE-1在不同的上皮肿瘤中非常活跃。德诺沃LINE-1插入可能驱动肿瘤发生,因此系统地研究LINE-1在癌症中的逆转是很重要的。在人类基因组中存在的150个具有逆转能力的LINE-1中,只有少数LINE-1位点,也称为"热"LINE-1,占不同癌症类型的de novo LINE-1插入的大多数。我们开发了一种基于聚合酶链反应(PCR)的简单方法,用于监测这些热LINE-1的逆转活性。该方法基于长距离反(LDI)-PCR,利用3=转导,一种利用LINE-1调动其侧翼非重复区域的机制,随后可用于识别de novo LINE-1 3= 转导事件源自特定的热线-1。
引言
长穿插的核元件(LINE-1s)是一个称为逆转子的移动遗传元素家族,可以通过称为逆转的复制和粘贴机制从一个地方独立移动到另一个地方。在进化时期,人类基因组已经积累了超过50万份LINE-1重复1。然而,基因组中存在的大多数LINE-1拷贝是突变的,因此不能通过逆转移动;只有+150份有完整的DNA序列副本,他们移动2。在正常体细胞中,这些LINE-1的流动性受到不同宿主因子3的限制。这些限制在不同的上皮肿瘤被解除,导致LINE-1被压抑,并导致许多在肿瘤基因组4的denovo插入。 这些肿瘤相关的一些新插入已被证明导致插入诱变在基因中,从而驱动肿瘤进展5,6。因此,能够绘制肿瘤基因组中新插入图非常重要。
现有检测de novo LINE-1 插入的方法使用 (1) 全基因组测序方法4,7,8, 使用不同的计算算法来查找 de novo LINE-1 插入从WGS数据,或(2)下一代测序,针对3= 结束年轻的,潜在的活动LINE-1s9,10,11,12,13。然而,用这些方法在几千个几乎相同的拷贝中寻找新的插入体远非微不足道,而且与LINE-1插入4相关的肿瘤异质性和基因组变化进一步加剧了这一挑战。
使用这些现有方法的研究表明,只有几个LINE-1占大多数在肿瘤7,8中观察到的de novo LINE-1插入。因此,要回答特定的肿瘤样本是否显示LINE-1活性,只需绘制由这少数高度活跃的LINE-1位点引起的逆转事件就足够了。在本文中,我们描述了一种基于聚合酶链反应(PCR)的简单方法14,该方法可用于监测TTC28基因第一个内源在22q12.1中高度活跃的特定LINE-1位点的活性,该方法在结肠直肠癌中高度活跃7,8.本 LINE-1 位点将在整个文章中称为TTC28-LINE-1。此测定特别识别de novo LINE-1 的回变事件,通过称为 3 = 转导15的机制在源 LINE-1 的 3 + 侧翼区域调动非重复序列。3 = 转导发生是由于弱LINE-1多分化信号(PAS),导致转录机械跳过它,而是终止转录在更强的PAS下游,从而捕获侧翼非重复序列(从此称为"唯一标记"),然后与 LINE-1 序列一起插入目标位置。Philippe等人16日最近发现,不同的细胞类型可以表达不同的LINE-1位点。根据这一发现,这种方法可用于监测表达最高度的LINE-1的活动,在感兴趣的癌症类型中调动其独特的标签。
LDI-PCR 的第一步是消化基因组DNA与限制性酶,产生一个限制片段,其中包含LINE-1被测定(这里,TTC28-LINE-1)及其唯一标记(图1)。 然后,通过自结扎和PCR使用位于独特标签内的反向引体进行循环。这样,其"原生"位置的全长源LINE-1始终被放大,并与其一起,在包含唯一标记的不同目标位点的子代LINE-1插入也将被放大(图1),从而报告有问题的LINE-1的逆变活动。
研究方案
这项研究得到了赫尔辛基大学医院机构审查委员会和道德委员会的批准。签署知情同意从受试者的血液样本用于证明该协议。
1. 设计逆引体和选择限制性酶(生物信息学)
- 确定 LINE-1 关联的唯一标记
- 从LINE-1数据库下载FASTA格式的TTC28-LINE-1序列,如L1Base17。TTC28-LINE-1 的 L1base ID 为 135。
- 包括 5 kb 序列,在 LINE-1 序列的 5° 和 3 ° 两端进行齿面,并在字处理器中批字。
注:此处的LINE-1齿面序列以棕色字体进行注释,LINE-1 序列为灰色字体(补充文件)。 - 将所选LINE-1的共生PAS下游的1 kb序列输入到PAS预测工具中,如聚adq18或龙聚A点子19,并在此 1 kb 窗口中对所有聚化信号进行批别。
注: 如果 1 kb 窗口中没有 PAS,则在下一个 1 kb 窗口下游搜索 PAS。TTC28-LINE-1自身的弱 PAS 以粉红色突出显示,而下游 1 kb 窗口中的所有其他 PAS 以红色突出显示(补充文件)。 - 将LINE-1的共生PAS的末尾与下游最强的PAS之间的顺序作为"唯一标签"进行批别。
注: TTC28-LINE-1 的"唯一标签"以黄色突出显示(补充文件)。
- 设计逆引体
- 通过在基于 Web 的引体设计工具(如引注 3(http://bioinfo.ut.ee/primer3-0.4.0/)或 NCBI 的引基-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)中输入"唯一标记"序列来设计反向 PCR 引基。由于这些工具设计的引色对相互面,促进传统的PCR,因此使用底漆对的反向补码功能来执行反向PCR。
注:由于LINE-1转导在5端被严重截断,且换能区域的大小变化很大,因此通过在NCBI引物-BLAST中设置"PCR产品长度"参数,将两个反向引物之间的距离降至最低。最低。在唯一标记中的多个 PAS 的情况下,设计多个对应于不同 PAS 的引体对。设计引体接近PAS,因为LINE-1插入从RNA中间体的3端启动,5端被可变截断。这里设计了三个引色对,以青绿色突出显示,对应于TTC28 LINE-1(补充文件)的唯一标签中的三个强聚化信号。
- 通过在基于 Web 的引体设计工具(如引注 3(http://bioinfo.ut.ee/primer3-0.4.0/)或 NCBI 的引基-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)中输入"唯一标记"序列来设计反向 PCR 引基。由于这些工具设计的引色对相互面,促进传统的PCR,因此使用底漆对的反向补码功能来执行反向PCR。
- 选择限制性酶
- 使用基于 Web 的工具(如 RestrictionMapper)在silico 中消化 LINE-1 序列及其 5 kb 的上游和下游侧翼。这将给出一个综合的限制性酶列表,这些酶可以消化这个区域,产生不同的限制片段。
- 选择切割LINE-1原生位点的限制性酶,如下所示:在5=端,在LINE-1的上游5=5=结束或远5=结束LINE-1本身,并在3=末端,在LINE-1的唯一标签的下游。
注:所选限制性酶应对DNA甲基化不敏感,应热不可活化,并应产生交错的"粘性"端,相互补充。为了演示LDI-PCR,使用Sac I限制酶,在GAGCTC位点切割DNA,这里以浅绿色突出显示,使用(补充文件)。 - 注意所选限制性酶的限制片段大小。这不应超过 12 kb,因为它可能无法被 PCR 有效放大。
2. 为长距离反向PCR制作圆形DNA模板
- DNA提取和质量评估
- 使用市售的DNA提取试剂盒从样品(肿瘤或血液)中提取基因组DNA,可根据制造商的说明提取LDI-PCR所需的高质量、高分子量DNA。另外,高分子量DNA也可以用苯酚:氯仿20提取。
- 根据制造商的说明,使用荧光计测量DNA浓度,并在含有溴化乙二苯(0.5微克/升)的1xTris-醋酸-EDTA(TAE)缓冲液中运行100 ng的DNA,同时使用+-HindIIIDNA分子量标记,用于检查DNA的质量和数量。
- 消化基因组DNA
- 通过在冰上0.2 mL PCR管中加入20单位Sac I限制酶,5μL的10x反应缓冲液(材料表),在0.2 mL PCR管中加入100 ng(高达44μL),使消化反应混合(最终体积为50μL)(大多数制造商的1μL限制酶i足以完全消化100纳克的基因组DNA)。通过轻拂管子将溶液混合,并短暂离心。
- 使用热循环器在 37°C 孵育反应混合物 1 小时,随后在 65°C 下进行热灭活 5 分钟。
- 自处理消化的基因组DNA
- 在50μL消化混合物中(步骤2.2.2后),加入8μL的10x T4 DNA结合酶缓冲液、1μL(5单位)的T4DNA结合酶和21μL的超纯水,使最终反应体积为80μL。通过轻拂管混合溶液,并短暂离心。
- 在22°C的热循环器中孵育10分钟,在65°C下用热失活步骤终止10分钟。
3. 长距离反向PCR
- 通过梯度 PCR 确定引火退火温度
- 设置退火温度梯度 (A-4) °C, (A-2)°C, A,(A+2)°C, (A+4) °C,其中 A 是使用商业供应商的基于 Web 的工具计算的引结对的理论退火温度。
注:某些制造商的热循环器可能不允许手动设置温度梯度。在这种情况下,自动梯度设置可用于温度范围 (A-4) °C 到 (A+4) °C。 - 在 1.5 mL 微离心管中组合和混合以下组件,为 PCR 制备主混合物:4 μL 5x 反应缓冲液,0.4 μL 10 mM dNTP,5 μL 2 μM PCR 底漆(前向和反向,在步骤 1.3.1 中设计),每反应的DNA聚合酶0.2μL(0.1 U)。为梯度中的每个退火温度设置一个反应。
- 对于每种反应,将主混合物的等分19μL混入0.2 mL PCR管中,并加入第2节中制作的1μL(1.25 ng)循环DNA模板。
注:使用从正常血液DNA生成的循环自结DNA作为模板,以避免消耗潜在的珍贵肿瘤DNA进行优化步骤。 - 在热循环器上运行梯度 PCR 程序,如下所述:(i) 在 98°C(变性)下运行 3 分钟的循环;(三) 在 98°C 下运行梯度 PCR 程序;(三) 在 98°C 下运行梯度 PCR 程序(在 98°C 时运行梯度 PCR 程序)。(二) 35个周期(98°C[变性]时10秒),[A-4]至[A-4][A]°C[退火]温度梯度20s,在72°C[延长]时1⁄6分钟[每千分之30s预期PCR产物];(三) 在72°C(最终延伸)时一个周期10分钟。
- 在 1x TAE 缓冲液中以 4.5 V/cm 制备的 1% agarose 凝胶21中运行 6 μL PCR 产品,并分析在不同退火温度下产生的 PCR 产物。
- 选择产生与预期尺寸对应的 PCR 产品的退火温度。
- 设置退火温度梯度 (A-4) °C, (A-2)°C, A,(A+2)°C, (A+4) °C,其中 A 是使用商业供应商的基于 Web 的工具计算的引结对的理论退火温度。
- 检测肿瘤基因组中的de novo LINE-1逆转活性
- 在从肿瘤样本生成的圆形DNA模板上使用反向PCR引基对执行LDI-PCR(第2节)。遵循与梯度 PCR 相同的说明(第 3.1 节),但这次用最佳退火温度替换温度梯度。
- 如步骤 3.1.5 中所做的那样,通过胶质电泳分析 PCR 产品。已知尺寸的PCR产物或对应于LINE-1在其原生位点的"原生"PCR产物,对于每种反应都应该可见。德诺沃LINE-1 3 = 在肿瘤样本测定中的转导可检测为不同尺寸的PCR产物,以及阿加辛凝胶中的原生PCR产物。
4. 对LDI-PCR产品进行测序,以揭示LINE-1 3+转导的目标位点的身份
- 对每个LDI-PCR反应中产生的所有PCR放大阻子执行单分子长读测序,以识别这些LINE-1 3+转导事件的目标积分位点。
注:LDI-PCR产品的克隆和桑格测序也是一种可能的方法,尽管很麻烦。 - 使用标准序列对齐管道将单分子长读测序平台产生的读取与参考基因组对齐。使用 LDI-PCR 软件14分析对齐的读取,以识别de novo LINE-1 插入及其目标站点。
结果
对于 TTC28-LINE-1,在其共生 PAS 下游的 1 kb 窗口内有多个 PAS,因此TTC28-LINE-1PAS 和下游 811 bp 的最强 PAS 之间的区域被视为TTC28-LINE-1的唯一标记。 三个反向PCR引结对设计在这个独特的标签,对应于不同的PAS目前14。我们选择了三种限制酶:(i) NiI,该酶在TTC28-LINE-1 的上游切割 5 = 上部,3 = 唯一标记外,和 (ii) SacI 和 (iii) PstI 在其远 5 端的 LINE-1 内切割 5 = 和 3 = 唯一标记外。这些分别生成 10,288 bp、5,699 bp 和 6,305 bp 的限制片段。
为了证明这种方法,我们对从MCF7细胞系中提取的DNA进行了LDI-PCR。这种乳腺癌细胞系以前曾报道显示TTC28-LINE-1活性16。 为简单起见,我们使用一种限制性酶SacI 制作了一个圆形 DNA 模板,使用三种引体对中的一个引体对(表1) 来检测TTC28产生的de novo LINE-1 插入 -行 1。
从MCF7细胞系中提取的DNA质量良好,通过胶质电泳确保了良好质量(图2)。高分子量DNA表明,基因组DNA是这种测定的最佳质量。如果涂片是可见的,这表明提取的DNA质量较差,这反过来会阻碍下游程序。
图 3显示了梯度 PCR 实验的代表性结果,旨在确定TTC28-LINE-1反向引基器对的最佳退火温度。血液基因组DNA与Sac I一起消化,然后自我结合形成圆形DNA模板,用于这种反应。在 62、64 和 66°C 下,预期尺寸(5,649 bp)的高度特异性 PCR 产物表明,该引基对的最佳退火温度在 62-66 °C 范围内。
我们通过消化MCF7基因组DNA,然后结合自结酶,生成了圆形DNA模板。图4显示,TTC28-LINE-13 = 转导发生在MCF7细胞系中:de novo插入可检测为不同尺寸的LDI-PCR产物以及已知尺寸的本机PCR产物(5,649 bp)。为了确定de novo目标位点的基因组坐标,PCR 放大组可以排序(参见协议第 4 节)。
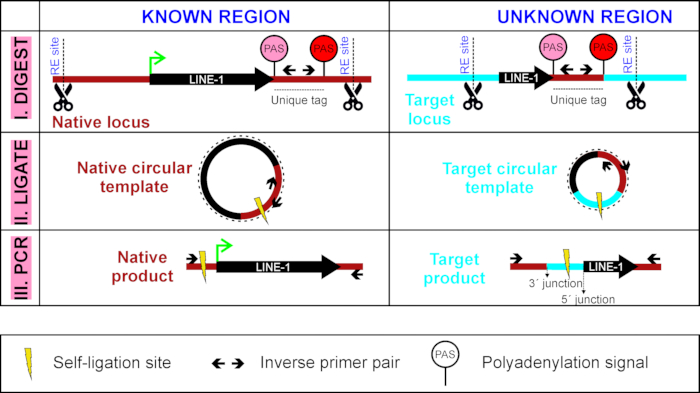
图 1:LDI-PCR 的概述,用于检测 LINE-1 3 = 转导。循环DNA模板是首先消化(I)它与限制酶和自控(II)它。此步骤后面是反向 PCR (III),其反向 PCR 引录针对感兴趣的 LINE-1 的唯一标记(LINE-1 自身较弱的 PAS(粉红色)和较强的 PAS 下行(红色)之间的顺序)。请点击此处查看此图的较大版本。
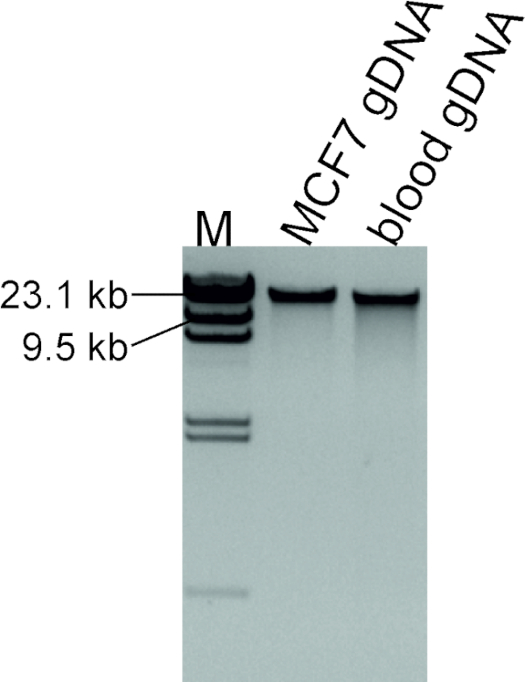
图 2:提取DNA的质量评估。从MCF7细胞系中提取的100 ng DNA和来自正常个体的血液,将用作对照样本,与1 μL和2μL的βDNA/HindIII标记一起运行,标记为M。请点击这里查看这个数字的较大版本。
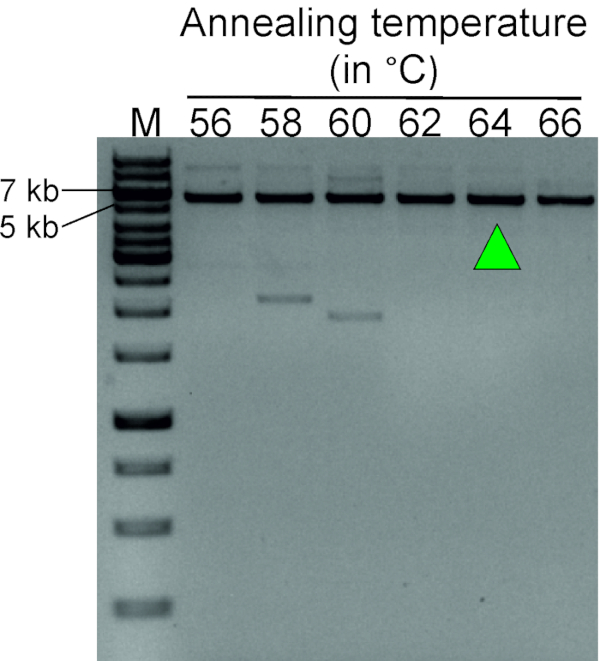
图 3:梯度PCR,用于确定反向PCR引漆的最佳退火温度(表1)。LDI-PCR在退火温度为56至66°C时使用逆底漆对,在62~66°C下显示独特的PCR产物。绿色箭头表示为将来实验选择的退火温度。通过消化来自具有SacI的正常个体的血液基因组DNA,然后进行自结扎,产生循环DNA模板用于此优化步骤。M,标记(1 kb加上DNA阶梯)。请点击此处查看此图的较大版本。
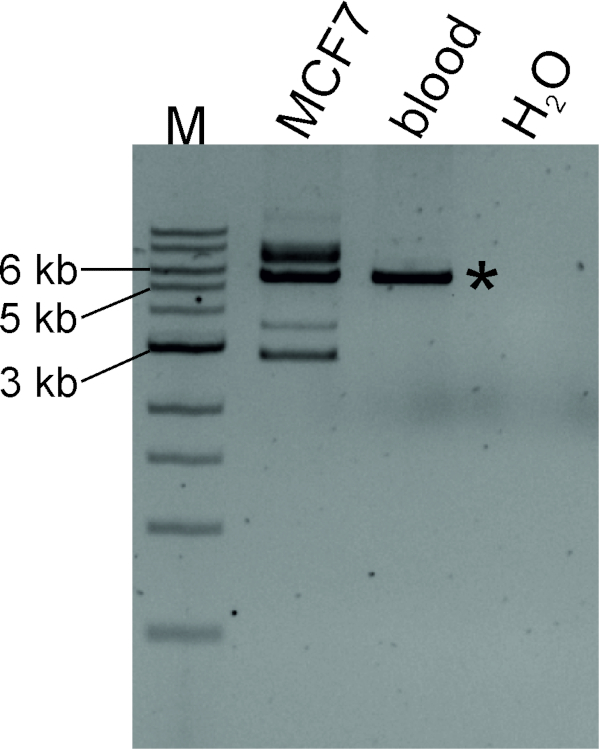
图 4: LDI-PCR 可识别 LINE-1 3 = 转导TTC28-线-1。通过消化MCF7和血液(来自正常个体)基因组DNA与Sac I然后进行自结扎产生的循环DNA模板在最佳退火温度下被反向引注放大。在MCF7 DNA和正常血液DNA中检测出"原生"PCR产物,标有星号,预期尺寸为5,649bp),而MCF7还生产了不同尺寸的附加PCR产物,表明de novo LINE-1逆转。M,标记(1 kb DNA阶梯)。请点击此处查看此图的较大版本。
| 引种名称 | 序列 (5 × 3 °) |
| L1_001 (修订) | TTCACTATATATATATATAGAAAC |
| L1_002 (fwd) | CCCAAATATACCCATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATATAT |
表1:为TTC28-线-114.
补充文件。请点击此处下载此文件。
讨论
在这里,我们描述了一个可用于识别来自任何感兴趣的活动LINE-1的插入方法。我们已经优化了这种方法的高度活跃的LINE-1,位于22q12.1,并先前证明它在检测结肠直肠癌14的亚克隆插入高度敏感。
LDI-PCR的成功取决于基因组DNA的质量。因此,我们增加了一个质量控制步骤,以确保在协议开始时存在高分子量DNA(步骤2.1.2)。我们建议将基因组DNA储存在-20°C进行长期储存,并准备等分物以避免冷冻和解冻的循环。强烈建议使用血液或患者匹配的正常组织的基因组DNA来区分检测到的LINE-1逆转是生殖系还是躯体事件。由于限制性酶的切割位点在基因组中是随机的,因此特定的de novo LINE-1 插入位点可能并不包含任何限制酶在其附近使用的切口位点。因此,为了增加检测肿瘤DNA中大多数de novo LINE-1插入的可能性,在单独的反应中应使用多个限制性酶来生成不同的循环DNA模板库。此外,如果感兴趣的 LINE-1 的唯一标记具有多个 PAS,则使用每个 PAS 相邻的引物对可提高检测严重截断的转导率的机会。
尽管存在用于全基因组检测de novo LINE-1 插入的优雅方法,但如果目的是在特定细胞环境中探索特定 LINE-1 的逆转能力,它们可能会令人难以承受。为此,LDI-PCR 可以是一种廉价、简单但可靠的方法来可视化 LINE-1 逆转事件。该方法使用的目标方法与TS-ATLAS22类似;然而,LDI-PCR避免了使用链接器寡核苷酸,并可以同时放大de novo LINE-1插入的5 Ω和3个接点。有关LINE-1插入的5+和3个连接点的信息,集成的目标位点,聚A尾部和目标位点修改,所有这些都是LINE-1逆转的标志,可以通过将LDI-PCR与单分子耦合获得长读测序技术。因此生成的长读取包含插入的 LINE-1 序列、其唯一标记和单个读取的目标序列,从而规避了在重复区域中映射短读的困难。
使用 LDI-PCR 检测 LINE-1 活动有两个主要限制。第一个是PCR固有的:它只能可靠地放大大小达10 kb的片段。在选择限制性酶时应考虑这一点,因为原生片段不应超过此限制。其次,该方法只能检测通过3+转导来调动LINE-1的3+侧翼区域的逆变事件。因此,不会使用此方法检测未表现出 3 = 转导的 LINE-1 的活动。此外,尽管被LDI-PCR放大,但一些LINE-1逆转事件(a)产生与"原生"位置或其他逆转位置相似的PCR目标,或(b)罕见或亚克隆,可能无法通过角胶检测电泳。这种LINE-1逆转事件可以通过使用单分子长读测序技术14对LDI-PCR产物进行测序来捕获。
此处描述的工作流程可以很容易地修改,通过使用适当的限制性酶和设计针对这些LINE-1的反向引体来检测其他"热"LINE-1的活性。除了检测LINE-1介导3=转导外,该方法还可以适应检测较不频繁的LINE-1介导5=转导23。类似的方法也用于识别在基于细胞的测定24和癌症25的病毒整合位点的LINE-1检测器的整合位点。除了LINE-1插入,这种方法还可用于检测其他基因组畸变,如DNA重排,其中有关重排易发生区域的信息预先存在26。
披露声明
作者没有什么可透露的。
致谢
我们想感谢所有共同作者在文章中,这种方法首先被描述14,特别是塔蒂亚娜·卡朱索,金莫·佩林,奥蒂·基尔皮瓦拉和埃萨·皮特卡宁在开发该方法时进行了有价值的讨论。L.K.由芬兰学院(赠款号25996、292789、306026和314394)、西格丽德·朱塞柳斯基金会和芬兰癌症协会资助。B.P. 是赫尔辛基大学研究基金会博士生奖学金、芬兰癌症协会论文资助和伊达·蒙蒂宁·塞蒂的博士研究资助。我们还感谢来自赫尔辛基大学的Teemu Masalin(赫尔辛基大学)和库尔·什雷斯塔的研究小组协助我们制作视频。
材料
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
参考文献
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16(2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343(2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. Jr High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043(2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521(2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Jr Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484(2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。