Method Article
Détection de l'activité de rétrotransposition des lignes-1 chaudes par PCR inverse à longue distance
Dans cet article
Résumé
Cet article décrit un simple test basé sur PCR pour surveiller l'activité d'un rétrotransposon line-1 actif et pour cartographier les rétrotranspositions de novo dans un génome donné. À l'aide de la lignée cellulaire MCF7, nous démontrons ici comment cette méthode peut être appliquée pour détecter l'activité d'une ligne-1 située au 22q12.1.
Résumé
Les éléments nucléaires entrecoupés de longue durée 1 (LINE-1) sont la seule famille d'éléments génétiques mobiles dans le génome humain qui peuvent se déplacer de façon autonome. Ils le font par un processus appelé rétrotransposition dans lequel ils transcritnt pour former un intermédiaire arnasquie qui est ensuite inséré dans le génome par transcription inversée. En dépit d'être silencieux dans les cellules normales, les LINE-1 sont fortement actifs dans différentes tumeurs épithéliales. De novo (En) Les insertions de LINE-1 peuvent potentiellement conduire la tumorigénèse, et il est donc important d'étudier systématiquement la rétrotransposition LINE-1 dans le cancer. Sur les 150 LINE-1 compétents en rétrotransposition présents dans le génome humain, seule une poignée de loci LINE-1, également appelés LINE-1 « chauds », représentent la majorité de l'insertion de novo LINE-1 dans différents types de cancer. Nous avons développé une méthode simple basée sur la réaction en chaîne de polymérase (PCR) pour surveiller l'activité de rétrotransposition de ces LIGNES-1 chaudes. Cette méthode, basée sur l'inverse de longue distance (LDI)-PCR, tire parti de la transduction de 3 euros, un mécanisme par lequel une LIGNE-1 mobilise sa région non répétitive, qui peut ensuite être utilisée pour identifier les événements de transduction novo LINE-1 3 provenant d'une ligne-1 chaude particulière.
Introduction
Les éléments nucléaires entrecoupés de longue durée (LINE-1) sont une famille d'éléments génétiques mobiles appelés rétrotransposons qui peuvent se déplacer indépendamment d'un endroit à l'autre par l'intermédiaire d'un mécanisme de copier-coller appelé rétrotransposition. Au fil du temps, le génome humain a accumulé plus de 500 000 copies de LINE-1 répète1. Cependant, la plupart des copies LINE-1 présentes dans le génome sont mutées et ne peuvent donc pas se déplacer par rétrotransposition; seulement 150 exemplaires ont une copie intacte de la séquence d'ADN nécessaire pour qu'ils se déplacent2. Dans les cellules somatiques normales, la mobilité de ces LINE-1 est limitée par différents facteurs d'hôte3. Ces restrictions sont soulagées dans différentes tumeurs épithéliales, causant des LINE-1 pour être déprimées et ayant pour résultat de nombreuses insertions de novo dans le génome de tumeur4. Certaines de ces insertions de novo associées à la tumeur ont été montrées pour causer la mutagénèse insertionnelle dans les gènes, conduisant ainsi la progression de tumeur5,6. Par conséquent, il est important d'être en mesure de cartographier de nouvelles insertions dans le génome de la tumeur.
Méthodes existantes pour détecter de novo LINE-1 insertions utilisation (1) approche de séquençage du génome entier4,7,8, où différents algorithmes de calcul sont utilisés pour trouver de novo LINE-1 insertions à partir des données WGS, ou (2) séquençage de nouvelle génération qui cible la fin de 3 'de jeunes, potentiellement actifs LINE-1s9,10,11,12,13. Cependant, trouver de nouvelles insertions parmi plusieurs milliers de copies quasi identiques avec ces méthodes est loin d'être trivial, et le défi est encore aggravé par l'hétérogénéité tumorale et les altérations génomiques associées à l'insertion LINE-14.
Des études utilisant ces méthodes existantes ont montré que seulement quelques LINE-1 représentent la majorité des insertions de novo LINE-1 observées dans les tumeurs7,8. Par conséquent, pour répondre si oui ou non un échantillon de tumeur particulier affiche l'activité LINE-1, il suffit de cartographier les événements de rétrotransposition causés par cette poignée de loci LINE-1 très actif. Dans cet article, nous décrivons une simple méthode14 basée sur la réaction en chaîne de polymérase (PCR) qui peut être utilisée pour surveiller l'activité d'un locus particulier de LINE-1 dans le premier intron du gène TTC28 à 22q12.1 qui est fortement actif dans le cancer colorectal 7 Annonces , 8. Ce locus LINE-1 sera appelé TTC28-LINE-1 tout au long de l'article. Cet exemple identifie spécifiquement les événements de rétrotransposition de novo LINE-1 qui mobilisent des séquences non répétitives sur la région de 3 flancs de la source LINE-1 par un mécanisme appelé 3 'transduction15. 3 - la transduction se produit en raison du faible signal de polyadenylation LINE-1 (PAS) qui fait sauter la machinerie transcriptionnelle et de mettre fin à la transcription à la PLUS forte PAS en aval, capturant ainsi la séquence non répétitive de flanc ( désormais appelée « balise unique ») qui est ensuite insérée dans l'emplacement cible le long de la séquence LINE-1. Philippe et coll.16 ont récemment montré que différents types de cellules peuvent exprimer différents loci LINE-1. À la lumière de cette constatation, cette méthode peut être appliquée pour surveiller l'activité de la ligne-1 la plus fortement exprimée qui mobilisent leur étiquette unique dans le type d'intérêt de cancer.
La première étape de LDI-PCR est la digestion de l'ADN génomique avec une enzyme de restriction qui génère un fragment de restriction contenant le LINE-1 étant évalué (ici, TTC28-LINE-1) et son étiquette unique (Figure 1). L'ADN digéré est ensuite circularisé par auto-ligation et PCR amplifié à l'aide d'amorces inverses situées dans l'étiquette unique. Ce faisant, la source pleine longueur LINE-1 à son emplacement "indigène" est toujours amplifiée et à côté d'elle, les insertions LINE-1 de progéniture à différents loci cibles contenant l'étiquette unique seront également amplifiées (figure 1), rapportant ainsi l'activité de rétrotransposition du LINE-1 en question.
Protocole
Cette recherche a été approuvée par le Comité d'examen institutionnel et le comité d'éthique de l'hôpital universitaire d'Helsinki. Le consentement éclairé signé a été obtenu du sujet pour l'échantillon de sang utilisé pour démontrer ce protocole.
1. Concevoir des amorces inverses et sélectionner des enzymes de restriction (bioinformatique)
- Détermination d'une balise unique associée à LINE-1
- Téléchargez la séquence TTC28-LINE-1en format FASTA à partir d'une base de données LINE-1 telle que L1Base17. L'ID L1base pour TTC28-LINE-1 est 135.
- Inclure une séquence de 5 kb flanquant à la fois 5 et 3 extrémités de la séquence LINE-1 et l'annoter dans un traitement de texte.
REMARQUE: Ici, la séquence de flanc LINE-1 est annotée en police brune, et la séquence LINE-1 est en police grise (Fichier supplémentaire). - Entrez une séquence de 1 kb en aval du PAS cognate de LINE-1 sélectionné dans un outil de prédiction PAS tel que polyadq18 ou Dragon PolyA spotter19 et annoter tout le signal de polyadenylation dans cette fenêtre de 1 kb.
REMARQUE : S'il n'y a pas de PAS dans la fenêtre de 1 kb, recherchez PAS dans la fenêtre suivante de 1 kb en aval. TTC28-LINE-1 propre PAS faible est mis en évidence en rose et tous les autres PAS en 1 kb fenêtre en aval est mis en évidence en rouge (Fichier supplémentaire). - Annoter la séquence entre la fin du PAS cognate de LINE-1 et le PAS le plus fort en aval comme « étiquette unique ».
REMARQUE: Le "tag unique" de TTC28-LINE-1 est mis en évidence en jaune (Fichier supplémentaire).
- Conception d'amorces inverses
- Concevez des amorces PCR inverses en entrant la séquence « étiquette unique » dans un outil de conception d'amorce basé sur le Web comme Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) ou l'amorce-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) de NCBI. Étant donné que les paires d'amorces conçues par ces outils se font face pour faciliter le PCR conventionnel, utilisez la fonction de complément inverse pour la paire d'amorce pour effectuer PCR inverse.
REMARQUE : Comme les transductions LINE-1 sont fortement tronquées à l'extrémité de 5 et que la taille de la région transductée est très variable, visez à maintenir la distance entre les deux amorces inverses minimale, en définissant le paramètre « longueur du produit PCR » dans l'amorce-BLAST NCBI minimum. Dans le cas de plusieurs PAS dans l'étiquette unique, concevez plusieurs paires d'apprêt qui correspondent à différents PAS. Amorces de conception près du PAS, comme les insertions LINE-1 initier à partir de la 3 'fin de l'ARN intermédiaire et l'extrémité de 5 'est variablement tronqué. Ici, trois paires d'amorces ont été conçues, mis en évidence en sarcelle et vert, correspondant à trois signaux de polyadenylation forte dans l'étiquette unique de TTC28 LINE-1 (Fichier supplémentaire).
- Concevez des amorces PCR inverses en entrant la séquence « étiquette unique » dans un outil de conception d'amorce basé sur le Web comme Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) ou l'amorce-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) de NCBI. Étant donné que les paires d'amorces conçues par ces outils se font face pour faciliter le PCR conventionnel, utilisez la fonction de complément inverse pour la paire d'amorce pour effectuer PCR inverse.
- Sélection d'enzymes de restriction
- Digdiérez la séquence LINE-1 avec ses flancs en amont et en aval de 5 kb en silico à l'aide d'outils Web tels que RestrictionMapper. Ceci donnera une liste complète des enzymes de restriction qui digère cette région, générant différents fragments de restriction.
- Sélectionnez les enzymes de restriction qui coupent le locus natif de LINE-1 comme suit : à l'extrémité 5' de l'extrémité, soit en amont de la ligne-1 5's'end ou loin 5 'end de la LIGNE-1 elle-même, et à l'extrémité 3', en aval de l'étiquette unique de la LIGNE-1.
REMARQUE : L'enzyme de restriction sélectionnée doit être insensible à la méthylation de l'ADN, doit être inactivatable par la chaleur et devrait générer des extrémités « collantes » décalées qui sont complémentaires les unes aux autres. Afin de démontrer LDI-PCR, SacI enzyme restriction qui coupe l'ADN sur les sites GAGCTC, mis en évidence ici en vert clair, est utilisé (Fichier supplémentaire). - Prenez note de la taille du fragment de restriction faite par des enzymes de restriction sélectionnées. Cela ne devrait pas être plus de 12 kb car il pourrait ne pas être efficacement amplifié par PCR.
2. Fabrication de modèles d'ADN circulaires pour PCR inverse à longue distance
- Extraction d'ADN et évaluation de la qualité
- Extraire l'ADN génomique d'échantillons (tumeur ou sang) à l'aide de kits d'extraction d'ADN disponibles dans le commerce qui peuvent extraire l'ADN de bonne qualité et de poids moléculaire élevé nécessaire pour LDI-PCR selon les instructions du fabricant. Alternativement, l'ADN de poids moléculaire élevé peut également être extrait par phénol:chloroforme20.
- Mesurer la concentration d'ADN à l'aide d'un fluoromètre selon les instructions du fabricant, et exécuter 100 ng d'ADN sur 1% (w/v) gel d'agarose contenant du bromure d'éhidium (0,5 g/mL) dans 1x Tris-acetate-EDTA (TAE) tampon à 4,5 V/cm21 à côté de -HindIII Marqueurs de poids moléculaires de l'ADN pour vérifier la qualité et la quantité de l'ADN.
- Digester l'ADN génomique
- Faire un mélange de réaction de digestion (volume final de 50 L) en ajoutant 20 unités SacI enzyme de restriction, 5 L de tampon de réaction 10x (Tableau des matériaux), 100 ng d'ADN (jusqu'à 44 L) dans un tube PCR de 0,2 mL sur la glace (1 l d'enzyme de restriction de la plupart des fabricants i s suffisant pour digérer complètement 100 ng d'ADN génomique). Mélanger la solution en faisant glisser le tube, et la centrifugeuse brièvement.
- À l'emploi d'un cycleur thermique, incuber le mélange de réaction à 37 oC pendant 1 h, puis d'inactivation de la chaleur à 65 oC pendant 5 min.
- Auto-ligating de l'ADN génomique digéré
- Pour le mélange de digestion de 50 l (après l'étape 2.2.2), ajouter 8 l de 10x T4 tampon de ligase d'ADN, 1 l (5 unités) de ligase d'ADN T4 et 21 l d'eau ultrapure pour faire un volume de réaction final de 80 'L. Mélanger la solution en faisant glisser les tubes , et la centrifugeuse brièvement.
- Incuber dans un cycleur thermique à 22 oC pendant 10 min, en terminant par une étape d'inactivation de la chaleur à 65 oC pendant 10 min.
3. PCR inverse de longue distance
- Détermination de la température d'anneling d'apprêt par gradient PCR
- Définir un gradient de température annealing de (A-4) 'C, (A-2) 'C, A, (A'2)'C, (A'4) 'C où A est la température d'annealing théorique de la paire d'amorce calculée à l'aide de l'outil Web du vendeur commercial.
REMARQUE : Les cyclistes thermiques de certains fabricants peuvent ne pas permettre de régler manuellement le gradient de température. Dans ce cas, le réglage automatique du gradient peut être utilisé avec une plage de température de (A-4) à 4 oC. - Préparer un mix maître pour le PCR en combinant et en mélangeant les composants suivants dans un tube microcentrifuge de 1,5 ml : 4 L de tampon de réaction 5x, 0,4 l de 10 ml de dNTP, 5 l de 2 apprêt PCR M (avant et inverse, conçu à l'étape 1.3.1) , 0,2 L (0,1 U) de polymérase d'ADN par réaction. Configurez une réaction pour chaque température d'annealing dans le gradient.
- Pour chaque réaction, aliquot 19 'L du mélange principal en tubes PCR de 0,2 mL et ajouter 1 l (1,25 ng) de modèle d'ADN circulaire fait à la section 2.
REMARQUE : Utilisez l'ADN auto-ligated circulaire généré de l'ADN normal de sang comme modèle de manière à éviter de consommer l'ADN potentiellement précieux de tumeur pour cette étape d'optimisation. - Exécuter le programme PCR de gradient sur un cycleur thermique tel que décrit ci-dessous : (i) un cycle de 3 min à 98 oC (dénaturation); (ii) 35 cycles de (10 s à 98 oC [dénaturation], 20 s au gradient de température de [A-4] à [A-4] 'C [Annealing] et de 1 à 6 min [30 s par kilobase du produit PCR prévu] à 72 oC [extension]); (iii) un cycle de 10 min à 72 oC (extension finale).
- Exécuter 6 ll du produit PCR en gel d'agarose21 de 1 % préparé dans un tampon TAE de 1 x 4,5 V/cm et analyser les produits PCR produits produits à différentes températures d'annealing.
- Sélectionnez la température d'annealing qui donne un produit PCR qui correspond à la taille prévue.
- Définir un gradient de température annealing de (A-4) 'C, (A-2) 'C, A, (A'2)'C, (A'4) 'C où A est la température d'annealing théorique de la paire d'amorce calculée à l'aide de l'outil Web du vendeur commercial.
- Détection de l'activité de rétrotransposition de novo LINE-1 dans le génome tumoral
- Effectuer LDI-PCR en utilisant la paire inverse d'amorce PCR sur les modèles circulaires d'ADN générés à partir d'échantillons de tumeur (section 2). Suivez les mêmes instructions que pour le gradient PCR (section 3.1), mais cette fois en remplaçant le gradient de température par la température d'annealing optimale.
- Analyser les produits PCR par électrophoresis gel agarose comme fait à l'étape 3.1.5. Le produit PCR de taille connue ou le produit PCR « indigène » correspondant au LINE-1 à son locus natif doit être visible pour chaque réaction. De novo (En) LA transduction LINE-1 3 dans l'échantillon de tumeur a été détectée est détectable comme produits PCR de différentes tailles, avec le produit PCR indigène dans le gel d'agarose.
4. Séquençage des produits LDI-PCR pour révéler l'identité des sites cibles pour la transduction LINE-1 3
- Effectuer un séquençage à longue lecture à longue molécule de tous les amplicons PCR générés dans chaque réaction LDI-PCR afin d'identifier les sites d'intégration cibles de ces événements de transduction LINE-1 3.
REMARQUE : Le séquençage du clonage et du sanger des produits LDI-PCR est également une approche possible, quoique lourde. - Alignez les lectures produites par les plates-formes de séquençage à longue lecture à une seule molécule au génome de référence à l'aide de pipelines d'alignement de séquence standard. Analyser les lectures alignées à l'aide du logiciel LDI-PCR14 pour identifier les insertions de novo LINE-1 et ses sites cibles.
Résultats
Dans le cas de TTC28-LINE-1, il y a plus d'un PAS dans une fenêtre de 1 kb en aval de son PAS cognate, d'où la région entre le PAS TTC28-LINE-1et le PAS le plus fort à 811 pb en aval a été considérée comme l'étiquette unique pour TTC28-LINE-1. Trois paires d'amorces PCR inverses ont été conçues à cette étiquette unique qui correspondent à différents PAS présents14. Nous avons sélectionné trois enzymes de restriction: (i) NsiI qui coupe 5 en amont de TTC28-LINE-1 et 3 - en dehors de l'étiquette unique, et (ii) SacI et (iii) PstI qui a coupé 5 'dans le LINE-1 dans son extrémité de 5 ', et 3 'en dehors de l'étiquette unique. Ceux-ci génèrent des fragments de restriction de 10 288 pb, 5 699 pb et 6 305 pb respectivement.
Afin de démontrer cette méthode, nous avons exécuté LDI-PCR sur l'ADN extrait de la lignée cellulaire MCF7. Cette lignée de cellules de cancer du sein a été précédemment rapportée pour afficher l'activité de TTC28-LINE-116. Par souci de simplicité, nous avons fait un modèle d'ADN circulaire à l'aide d'une enzyme de restriction, SacI, sur trois et effectué un LDI-PCR en utilisant une paire d'amorce sur trois (tableau 1) pour détecter de novo LINE-1 insertions provenant de TTC28- LIGNE 1.
La bonne qualité de l'ADN extrait de la lignée cellulaire MCF7 a été assurée par l'électrophoresis de gel d'agarose (figure 2). Intact ADN de poids moléculaire élevé montre que l'ADN génomique est de qualité optimale pour cet analyse. Si un frottis est visible à la place, cela indique une mauvaise qualité de l'ADN extrait, ce qui à son tour entravera les procédures en aval.
La figure 3 montre un résultat représentatif d'une expérience PCR de gradient, visant à déterminer la température d'annealing optimale de la paire d'amorce inverse TTC28-LINE-1. L'ADN génomique de sang digéré avec le sacI, suivi de l'auto-ligation pour former un modèle circulaire d'ADN, a été employé pour cette réaction. Un produit PCR très spécifique de taille prévue (5 649 pb) à 62, 64 et 66 oC montre que la température d'annealing optimale pour cette paire d'amorce se situe dans la fourchette de 62 à 66 oC.
Nous avons produit un modèle circulaire d'ADN en digérant l'ADN génomique de MCF7 avec l'enzyme de restriction de SacI suivie de l'auto-ligation. La figure 4 montre que la transduction TTC28-LINE-13 se produit dans les lignées cellulaires MCF7 : les insertions de novo peuvent être détectées sous forme de produits LDI-PCR de tailles variables ainsi que d'un produit PCR indigène de taille connue (5 649 pb). Pour identifier les coordonnées génomiques des sites cibles de novo, les amplicons PCR peuvent être séquencés (voir la section 4 du protocole).
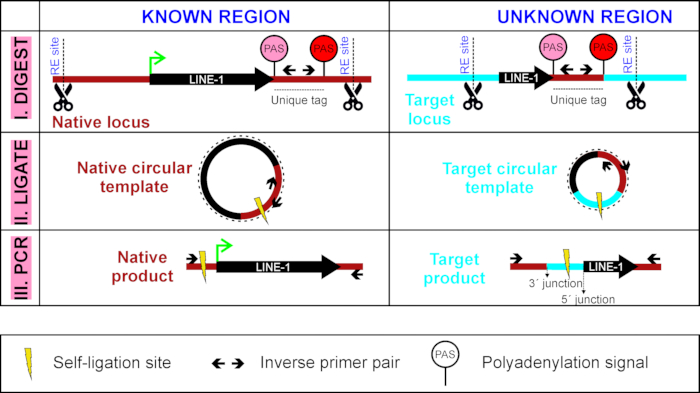
Figure 1 : Aperçu de LDI-PCR pour détecter la transduction LINE-1 3. Un modèle circulaire d'ADN est généré par la première digestion (I) il avec une enzyme de restriction et l'auto-ligating (II) il. Cette étape est suivie par PCR inverse (III) avec des amorces PCR inverses ciblées sur l'étiquette unique de la LIGNE-1 d'intérêt (séquence entre LE PAS plus faible de LINE-1, en rose, et PAS plus fort en aval, en rouge). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
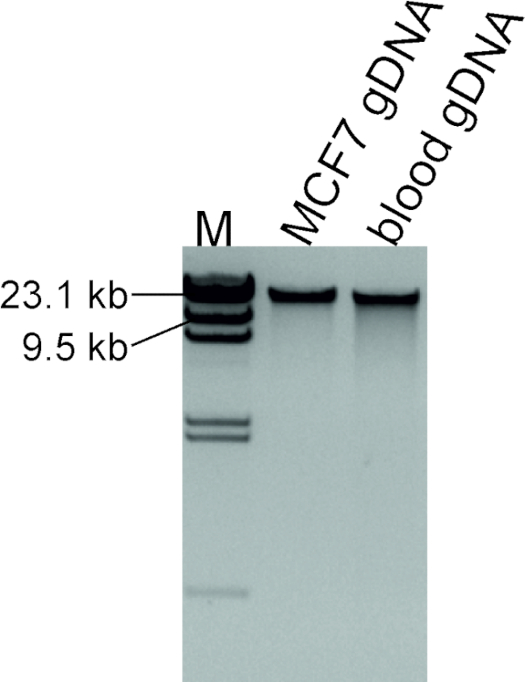
Figure 2 : Évaluation de la qualité de l'ADN extrait. 100 ng d'ADN extrait de la lignée cellulaire MCF7 et le sang d'un individu normal, qui sera utilisé comme un échantillon de contrôle, ont été exécutés à côté de 1 'L et 2 'L de marqueur d'ADN/HindIII étiqueté comme M. S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
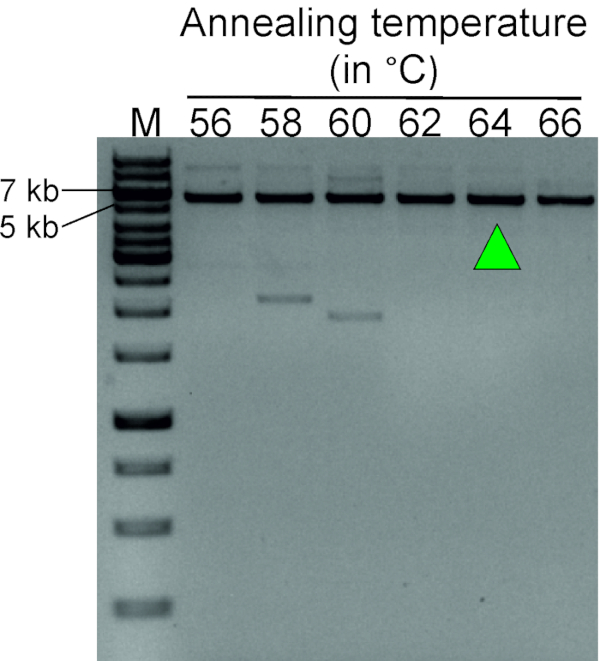
Figure 3 : Gradient PCR pour déterminer la température d'annealing optimale pour les amorces PCR inverses (tableau 1). LDI-PCR utilisant des paires d'apprêt inverse à la température d'annealing s'étendant de 56 à 66 oC montre un produit distinct de PCR à 62-66 oC. La flèche verte indique la température d'annealing choisie pour de futures expériences. Le modèle circulaire d'ADN généré en digérant l'ADN génomique de sang d'un individu normal avec SacI suivi par l'auto-ligation a été employé pour cette étape d'optimisation. M, marqueur (1 kb plus échelle d'ADN). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
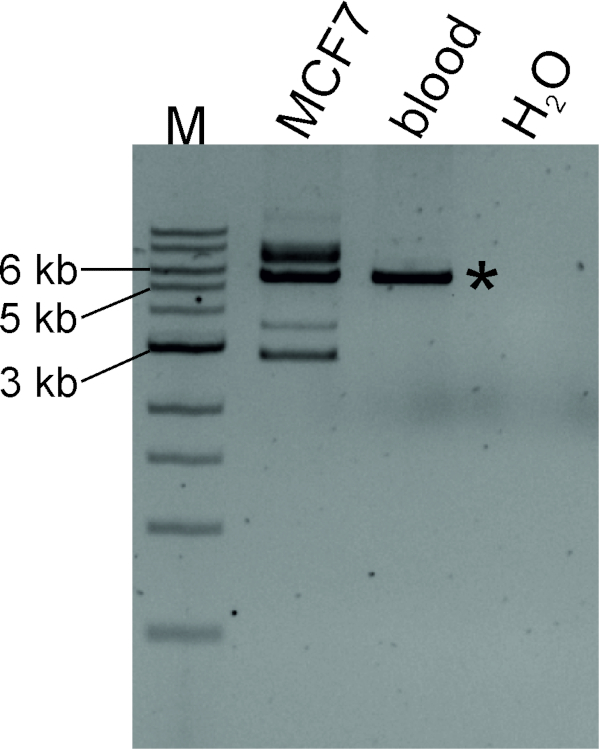
Figure 4 : LDI-PCR pour identifier la transduction LINE-1 3 TTC28 (en) -ligne 1. Les modèles circulaires d'ADN générés par la digestion mcF7 et le sang (de l'individu normal) L'ADN génomique avec SacI suivi par l'auto-ligation ont été amplifiés par des amorces inverses dans la température annealing optimale. Le produit PCR « indigène », marqué de l'astérisque, de taille prévue (5 649 pb) a été détecté dans l'ADN MCF7 et l'ADN sanguin normal, tandis que MCF7 a également produit d'autres produits PCR de tailles variables, indiquant la rétrotransposition de novo LINE-1. M, marqueur (1 kb échelle d'ADN). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Nom d'amorce | Séquence (5 '3 ') |
| L1-001 (rev) | TTCACTAAGCATGTTGTGGAAAAAC |
| L1-002 (fwd) | CCCAAAtATACCCAATTACTGGCA |
Tableau 1 : Paire d'amorce PCR inverse conçue pour l'étiquette unique de TTC28 (en) -LIGNE-1 14 (en) .
Fichier supplémentaire. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ici, nous décrivons une méthode qui peut être utilisée pour identifier les insertions de novo LINE-1 provenant de toute ligne active-1 d'intérêt. Nous avons optimisé cette méthode pour un LINE-1 très actif, situé à 22q12.1, et précédemment démontré qu'il est très sensible dans la détection des insertions sous-clonales dans le cancer colorectal14.
Le succès du LDI-PCR dépend de la qualité de l'ADN génomique. Par conséquent, nous avons inclus une étape supplémentaire de contrôle de la qualité pour nous assurer qu'au début du protocole, l'ADN de poids moléculaire élevé est présent (étape 2.1.2). Nous vous recommandons de stocker l'ADN génomique à -20 oC pour le stockage à long terme, et de préparer les aliquots afin d'éviter les cycles de congélation et de décongélation. L'utilisation de l'ADN génomique provenant du sang ou du tissu normal apparié par le patient est fortement recommandée pour distinguer si la rétrotransposition LINE-1 détectée est une lignée germinale ou un événement somatique. Étant donné que les sites de coupe pour les enzymes de restriction sont stochastiques dans le génome, il est possible qu'un site particulier d'insertion de novo LINE-1 ne puisse pas abriter de sites de coupe pour l'enzyme de restriction utilisée à proximité. Par conséquent, pour augmenter la probabilité de détecter la majorité des insertions de novo LINE-1 dans l'ADN tumoral, plus d'une enzyme de restriction devrait être utilisée dans des réactions distinctes pour générer différentes bibliothèques de modèle d'ADN circulaire. En outre, si l'étiquette unique de la LIGNE-1 d'intérêt a plus d'un PAS, puis l'utilisation de paires d'apprêt adjacentes à chaque PAS améliore les chances de détecter les transductions fortement tronquées.
Bien qu'il existe des méthodes élégantes pour la détection à l'échelle du génome des insertions de novo LINE-1, elles peuvent être écrasantes si l'objectif est de sonder la compétence de rétrotransposition d'une ligne-1 particulière dans un contexte cellulaire spécifique. À cette fin, LDI-PCR peut être une approche peu coûteuse et simple mais robuste pour visualiser les événements de rétrotransposition LINE-1. L'approche de ciblage utilisée dans cette méthode est similaire à TS-ATLAS22; cependant, LDI-PCR évite d'utiliser des oligonucléotides de liaison et peut amplifier simultanément les jonctions de 5 et 3 jonctions de l'insertion de novo LINE-1. Les informations concernant les deux jonctions de 5 et 3 de l'insertion LINE-1, le site cible de l'intégration, la queue polyA et les modifications du site cible, qui sont toutes des caractéristiques de la rétrotransposition LINE-1, peuvent être obtenues en couplant LDI-PCR avec une seule molécule technologies de séquençage à lecture longue. Les longues lectures ainsi générées contiennent la séquence LINE-1 insérée, son balise unique et les séquences cibles en une seule lecture, contournant les difficultés de cartographie des lectures courtes dans la région répétitive.
Il y a deux limitations majeures à l'utilisation de LDI-PCR pour la détection de l'activité LINE-1. La première est inhérente au PCR : elle ne peut amplifier de manière fiable les fragments jusqu'à 10 kb. Ceci devrait être pris en considération lors de la sélection de l'enzyme de restriction(s), comme le fragment indigène ne doit pas dépasser cette limite. Deuxièmement, cette méthode ne peut détecter que les événements de rétrotransposition qui mobilisent la région de 3 côtés de la LIGNE-1 par 3 transductions. Par conséquent, l'activité des LIGNES-1 qui ne présentent pas la transduction de 3 euros ne sera pas détectée à l'aide de cette méthode. De plus, bien qu'ils soient amplifiés par le LDI-PCR, certains événements de rétrotransposition LINE-1 qui (a) génèrent une cible DE PCR de taille similaire à l'emplacement « natif » ou à d'autres rétrotranspositions ou (b) sont rares ou sous-clonaux, peuvent ne pas être détectés par le gel d'agarose Électrophorèse. De tels événements de rétrotransposition LINE-1 peuvent être capturés en séquençant le produit LDI-PCR à l'aide de technologies de séquençage à lecture longue molécule unique14.
Le flux de travail décrit ici peut être facilement modifié pour détecter l'activité d'autres LINE-1 « chauds » en utilisant une enzyme de restriction appropriée et en concevant des amorces inverses ciblant ces LINE-1. En plus de la détection de la transduction de 3 -de la ligne-1, cette méthode peutêtre adaptée pour détecter les transductions de 5 '5 'par i de la ligne-1 moins fréquentes. Une méthode similaire a été utilisée pour identifier le site d'intégration des journalistes de LINE-1 dans les essais cellulaires24 et les sites d'intégration provirale dans le cancer25. Outre les insertions LINE-1, cette méthode peut également être utilisée pour détecter d'autres aberrations génomiques, telles que les réarrangements d'ADN, où l'information concernant la région sujette au réarrangement préexiste26.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous tenons à remercier tous nos co-auteurs dans l'article où cette méthode a été décrite pour la première fois14, en particulier Tatiana Cajuso, Kimmo Palin, Outi Kilpivaara et Esa Pitkunen pour des discussions précieuses tout en développant la méthode. L.K. est financé par l'Académie de Finlande (numéros de subvention 25996, 292789, 306026 et 314394), la Fondation Sigrid Juselius et la Société finlandaise du cancer. B.P. est titulaire d'un doctorat de la Fondation de recherche de l'Université d'Helsinki, d'une bourse de thèse de la Société finlandaise du cancer et d'une bourse de recherche doctorale d'Ida Montinin Sôtiô. Nous remercions également Teemu Masalin (Université d'Helsinki) et Kul Shrestha du groupe de recherche de L.K. de nous avoir aidés dans la production vidéo.
matériels
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
Références
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16(2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343(2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. Jr High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043(2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521(2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Jr Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484(2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon