Method Article
Rilevamento dell'attività di retrotraposizione di HOT LINE-1s da PCR inverso a lunga distanza
In questo articolo
Riepilogo
Questo articolo descrive un semplice saggio basato su PCR per monitorare l'attività di un retrotrasposon LINE-1 attivo e per mappare retrotrasposizioni de novo in un determinato genoma. Utilizzando la linea cellulare MCF7, dimostri qui come questo metodo può essere applicato per rilevare l'attività di un LINE-1 situato a 22q12.1.
Abstract
Gli elementi nucleari a lunga intersperatura 1 (LINE-1) sono l'unica famiglia di elementi genetici mobili nel genoma umano in grado di muoversi autonomamente. Lo fanno attraverso un processo chiamato retrotrasposizione in cui trascrivono per formare un mRNA intermedio che viene poi inserito nel genoma dalla trascrizione inversa. Nonostante siano silenziosi nelle cellule normali, i LINE-1 sono altamente attivi in diversi tumori epiteliali. De novo Gli inserimenti line-1 possono potenzialmente guidare la tumorigenesi, e quindi è importante studiare sistematicamente la retrotrasposizione LINE-1 nel cancro. Dei 150 LINE-1 retrotrassivi presenti nel genoma umano, solo una manciata di loci LINE-1, chiamati anche LINE-1 "caldi", rappresentano la maggior parte dell'inserimento de novo LINE-1 in diversi tipi di cancro. Abbiamo sviluppato un semplice metodo basato sulla reazione a catena di polimerasi (PCR) per monitorare l'attività di retrotrasposizione di questi line-1 caldi. Questo metodo, basato sulla PCR inverso a lunga distanza (LDI)-PCR, sfrutta la trasduzione 3, un meccanismo mediante il quale un LINE-1 mobilita la sua regione non ripetitiva di fianco, che può essere successivamente utilizzato per identificare gli eventi di trasduzione di de novo LINE-1 3 derivanti da un particolare line-1 caldo.
Introduzione
I lunghi elementi nucleari intervallati (LINE-1) sono una famiglia di elementi genetici mobili chiamati retrotrasposoni che possono spostarsi in modo indipendente da un luogo all'altro tramite un meccanismo di copia e incolla chiamato retrotrasposizione. Nel corso del tempo evolutivo, il genoma umano ha accumulato più di 500.000 copie di ripetizioni LINE-11. Tuttavia, la maggior parte delle copie LINE-1 presenti nel genoma sono mutate e quindi non possono muoversi attraverso la retrotrasposizione; solo 150 copie hanno una copia intatta della sequenza di DNA necessaria per spostarsi2. Nelle normali cellule somatiche, la mobilità di questi LINE-1 è limitata da diversi fattori ospiti3. Queste restrizioni sono alleviate in diversi tumori epiteliali, causando LINE-1 essere derepressi e con conseguente molti inserimenti de novo nel genoma del tumore4. Alcuni di questi inserimenti de novo associati al tumore hanno dimostrato di causare mutagenesi inserimento nei geni, quindi guidando la progressione tumorale5,6. Pertanto, è importante essere in grado di mappare nuovi inserimenti nel genoma del tumore.
I metodi esistenti per rilevare gli inserimenti de novo LINE-1 utilizzano (1) l'approccio di sequenziamento dell'intero genoma4,7,8, in cui vengono utilizzati diversi algoritmi computazionali per trovare inserimenti de novo LINE-1 dai dati WGS, o (2) sequenziamento di nuova generazione che si rivolge alla fine di 3, potenzialmente attivi LINE-1s9,10,11,12,13. Tuttavia, trovare nuovi inserimenti tra diverse migliaia di copie quasi identiche con questi metodi è tutt'altro che banale, e la sfida è ulteriormente aggravata dall'eterogeneità tumorale e dalle alterazioni genomiche associate all'inserimento LINE-14.
Studi che utilizzano questi metodi esistenti hanno mostrato che solo pochi LINE-1 rappresentano la maggior parte degli inserimenti de novo LINE-1 osservati nei tumori7,8. Pertanto, per verificare se un particolare campione di tumore mostra o meno l'attività line-1, è sufficiente mappare gli eventi retrotrasposizione causati da questa manciata di loci LINE-1 altamente attivi. In questo articolo, descriviamo un semplice metodo a catena di reazione alla polimerasi (PCR)14 che può essere utilizzato per monitorare l'attività di un particolare locus LINE-1 nel primo intron del gene TTC28 a 22q12.1 che è altamente attivo nel cancro colorettale 7 (in questo stato , 8. Questo locus LINE-1 sarà indicato come TTC28-LINE-1 in tutto l'articolo. Questo saggio identifica in modo specifico gli eventi retrotrasposizione de novo LINE-1 che mobilitano la sequenza non ripetitiva sulla regione di fiancheggiamento 3 della sorgente LINE-1 da un meccanismo chiamato 3 - trasduzione15. 3 - la trasduzione si verifica a causa del debole segnale di poliadenilazione LINE-1 (PAS) che fa sì che il meccanismo trascrizionale la salti e termini invece la trascrizione al PAS più forte a valle, catturando così la sequenza non ripetitiva di fianco ( d'ora in poi indicato come il "tag unico") che viene poi inserito nella posizione di destinazione insieme alla sequenza LINE-1. Philippe et al.16 recentemente ha dimostrato che diversi tipi di cellule possono esprimere diversi loci LINE-1. Alla luce di questa scoperta, questo metodo può essere applicato per monitorare l'attività del LINE-1 più altamente espresso che mobilitano il loro tag unico nel tipo di cancro di interesse.
Il primo passo in LDI-PCR è la digestione del DNA genomico con un enzima di restrizione che genera un frammento di restrizione contenente il LINE-1 che viene analisi (qui, TTC28-LINE-1) e il suo tag unico (Figura 1). Il DNA digerito viene quindi circolare con l'auto-legamento e la PCR amplificata utilizzando primer inversi situati all'interno del tag unico. In questo modo, la sorgente a tutta lunghezza LINE-1 nella sua posizione "nativa" è sempre amplificata e accanto ad essa, gli inserimenti LINE-1 della prostesa in diversi loci di destinazione contenenti il tag unico saranno amplificati (Figura1), segnalando così così la segnalazione l'attività di retrotransposition del LINE-1 in questione.
Protocollo
Questa ricerca è stata approvata dal Comitato Istituzionale di Revisione e dal Comitato Etico dell'Ospedale Universitario di Helsinki. Il consenso informato firmato è stato ottenuto dal soggetto per il campione di sangue utilizzato per dimostrare questo protocollo.
1. Progettazione di primer inversi e selezione degli enzimi di restrizione (bioinformatica)
- Determinazione di un tag univoco associato line-1
- Scaricare la sequenza TTC28-LINE-1 in formato FASTA da un database LINE-1, ad esempio L1Base17. L'ID L1base per TTC28-LINE-1 è 135.
- Includere una sequenza di 5 kb che fiancheggia sia le estremità 5 e 3 della sequenza LINE-1 e annotarla in un elaboratore di testi.
NOTA: Qui la sequenza di fianco line-1 è annotata in carattere marrone e la sequenza LINE-1 è in grigio (Filesupplementare ). - Inserisci 1 kb di sequenza a valle del cognato PAS di LINE-1 selezionato in uno strumento di previsione PAS come polyadq18 o Dragon PolyA spotter19 e annota tutto il segnale di poliadenilazione in questa finestra da 1 kb.
NOTA: Se non c'è PAS nella finestra da 1 kb, cercare PAS nella finestra successiva da 1 kb a valle. Il PAS debole di TTC28-LINE-1 è evidenziato in rosa e tutti gli altri PAS nella finestra di 1 kb a valle sono evidenziati in rosso (Filesupplementare ). - Annota la sequenza tra la fine del PAS cognato di LINE-1 e il più forte PAS a valle come "tag unico".
NOTA: il "tag univoco" di TTC28-LINE-1 è evidenziato in giallo (File supplementare).
- Progettazione di primer inversi
- Progetta primer PCR inversa inserendo la sequenza "tag unico" in uno strumento di progettazione di primer basato sul web come Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) o primer-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) di NCBI. Poiché le coppie di primer progettate da questi strumenti si affrontano facilitando la PCR convenzionale, utilizzare la funzione di complemento inverso per la coppia di primer per eseguire pcR inverso.
NOTA: poiché le trasduzioni LINE-1 sono fortemente troncate alla loro estremità 5 e la dimensione della regione trasdutta è altamente variabile, mira a mantenere la distanza tra i due primer inversi minima, impostando il parametro "lunghezza prodotto PCR" in NCBI primer-BLAST minimo. Nel caso di più PAS all'interno del tag univoco, progettare diverse coppie di primer che corrispondono a diversi PAL. Progettare i primer vicino al PAS, poiché gli inserimenti line-1 iniziano dalla fine 3 dell'RNA intermedia e l'estremità 5 viene variabilmente troncata. Qui sono state progettate tre coppie di primer, evidenziate in verde acqua e verde, corrispondenti a tre forti segnali di poliadenilazione nell'etichetta unica di TTC28 LINE-1 (Supplemental File).
- Progetta primer PCR inversa inserendo la sequenza "tag unico" in uno strumento di progettazione di primer basato sul web come Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) o primer-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) di NCBI. Poiché le coppie di primer progettate da questi strumenti si affrontano facilitando la PCR convenzionale, utilizzare la funzione di complemento inverso per la coppia di primer per eseguire pcR inverso.
- Selezione degli enzimi di restrizione
- Digerire la sequenza LINE-1 insieme ai suoi fianchi a monte e a valle di 5 kb in silico utilizzando uno strumento web-based come RestrictionMapper. Questo darà un elenco completo di enzimi di restrizione che digerisce questa regione, generando diversi frammenti di restrizione.
- Selezionare gli enzimi di restrizione che tagliano il locus nativo di LINE-1 come segue: alla fine 5 e, sia a monte del LINE-1 e 5 'fine del LINE-1 stesso, e alla fine 3, a valle del tag unico del LINE-1.
NOTA: l'enzima di restrizione selezionato deve essere insensibile alla metilazione del DNA, deve essere inattivabile dal calore e deve generare estremità "appiccicose" sfalsate complementari tra loro. Per dimostrare LDI-PCR, viene utilizzato l'enzima di restrizione SacI che taglia il DNA nei siti GAGCTC, evidenziato qui in verde chiaro (File supplementare). - Prendere nota della dimensione del frammento di restrizione effettuata da enzimi di restrizione selezionati. Questo non dovrebbe essere più lungo di 12 kb in quanto potrebbe non essere amplificato in modo efficiente da PCR.
2. Creazione circolare di modelli di DNA per PCR inverso a lunga distanza
- Estrazione del DNA e valutazione della qualità
- Estrarre il DNA genomico da campioni (tumore o sangue) utilizzando kit di estrazione del DNA disponibili in commercio in grado di estrarre DNA di buona qualità e di peso molecolare necessario per LDI-PCR secondo le istruzioni del produttore. In alternativa, il DNA ad alto peso molecolare può anche essere estratto da fenolo:cloroformio20.
- Misurare la concentrazione di DNA utilizzando un fluorometro secondo le istruzioni del produttore ed eseguire 100 ng di DNA su 1% (w/v) gel di agarose contenente bromuro di etidio (0,5 g/mL) nel buffer 1x Tris-acetate-EDTA (TAE) a 4,5 V/cm21 a fianco di Marcatori di peso molecolare del DNA per controllare la qualità e la quantità del DNA.
- Digerire il DNA genomico
- Fare un mix di reazione alla digestione (volume finale di 50 , L) aggiungendo 20 unità SacI enzima di restrizione, 5 l di 10x buffer di reazione (Tabella dei materiali), 100 ng di DNA (fino a 44 ) in un tubo PCR da 0,2 mL sul ghiaccio (1 - L di enzima di restrizione dalla maggior parte dei produttori i s sufficiente a digerire completamente 100 ng di DNA genomico). Mescolare la soluzione facendo scorrere il tubo e centrifugare brevemente.
- Utilizzare un ciclore termico per incubare la miscela di reazione a 37 gradi centigradi per 1 h, seguito da inattivazione di calore a 65 gradi centigradi per 5 minuti.
- Auto-litigazione del DNA genomico digerito
- Per la miscela di digestione 50 l (dopo il passo 2.2.2), aggiungere 8 L di 10x T4 DNA ligase buffer, 1 L (5 unità) di T4 DNA ligase e 21 L di acqua ultrapura per fare un volume di reazione finale di 80 . e centrifugare brevemente.
- Incubare in un ciclore termico a 22 gradi centigradi per 10 minuti, terminando con una fase di inattivazione del calore a 65 gradi centigradi per 10 minuti.
3. PCR inverso a lunga distanza
- Determinazione della temperatura di annessione del primer per gradiente PCR
- Impostare un gradiente di temperatura di annealing di (A-4) C, (A-2) , A, (A-2) , C, (A) C, dove A è la temperatura di annealing teorica della coppia di primer calcolata utilizzando lo strumento web del fornitore commerciale.
NOTA: i ciclisti termici di alcuni produttori potrebbero non consentire l'impostazione manuale del gradiente di temperatura. In tal caso, l'impostazione automatica del gradiente può essere utilizzata con un intervallo di temperatura compreso tra (A-4) e C. - Preparare un master mix per la PCR combinando e mescolando i seguenti componenti in un tubo di microcentrifuga da 1,5 ml: 4 -L di buffer di reazione 5x, 0,4 l di 10 mM dNTP, 5 ll di 2 M PCR primer (avanti e indietro, progettato nel passaggio 1.3.1) , 0,2 l (0,1 U) di polimerasi del DNA per reazione. Impostare una reazione per ogni temperatura di analing nel gradiente.
- Per ogni reazione, aliquote 19 L della miscela master in tubi PCR da 0,2 mL e aggiungere 1 L (1,25 ng) di modello di DNA circolare fatto nella sezione 2.
NOTA: Utilizzare il DNA auto-ligato circolare generato dal DNA sanguigno normale come modello in modo da evitare di consumare DNA tumorale potenzialmente prezioso per questa fase di ottimizzazione. - Eseguire il gradiente del programma PCR su un ciclore termico come descritto di seguito: (i) un ciclo di 3 min a 98 gradi (denaturazione); (ii) 35 cicli di (10 s a 98 s [denaturazione], 20 s al gradiente di temperatura di [A-4] a [A-4] C [Annealing] e 1/6 min [30 s per kilobase del prodotto PCR previsto] a 72 c [estensione]); (iii) un ciclo di 10 min a 72 gradi (estensione finale).
- Eseguire 6 Ll del prodotto PCR in 1% gel di agarose21 preparato in 1x tampone TAE a 4,5 V/cm e analizzare i prodotti PCR prodotti a diverse temperature di annealing.
- Selezionare la temperatura di annealing che produce un prodotto PCR che corrisponde alla dimensione prevista.
- Impostare un gradiente di temperatura di annealing di (A-4) C, (A-2) , A, (A-2) , C, (A) C, dove A è la temperatura di annealing teorica della coppia di primer calcolata utilizzando lo strumento web del fornitore commerciale.
- Rilevamento dell'attività retrotransposizione di de novo LINE-1 nel genoma del tumore
- Eseguire LDI-PCR utilizzando la coppia di primer PCR inversa su modelli di DNA circolari generati da campioni di tumore (sezione 2). Seguire le stesse istruzioni per la PCR con gradiente (sezione 3.1), ma questa volta sostituendo il gradiente di temperatura con la temperatura di annealing ottimale.
- Analizzare i prodotti PCR mediante elettroforesi gel agarose come fatto al passo 3.1.5. Il prodotto PCR di dimensioni note o il prodotto PCR "nativo" corrispondente al LINE-1 nel suo locus nativo dovrebbe essere visibile per ogni reazione. De novo La trasduzione line-1 3 nel campione tumorale ha evitato che è rilevabile come prodotti PCR di diverse dimensioni, insieme al prodotto PCR nativo nel gel di agarose.
4. Sequenziamento dei prodotti LDI-PCR per rivelare l'identità dei siti di destinazione per la trasduzione LINE-1 3
- Eseguire il sequenziamento a singola molecola di lettura a lungo di tutti gli amplificatori PCR generati in ogni reazione LDI-PCR per identificare i siti di integrazione di destinazione di questi eventi di trasduzione LINE-1 3.
NOTA: Anche la clonazione e il sequenziamento di Sanger dei prodotti LDI-PCR sono possibili, anche se ingombranti. - Allineare le letture prodotte dalle piattaforme di sequenziamento a lungo termine a singola molecola al genoma di riferimento utilizzando pipeline di allineamento della sequenza standard. Analizzare le letture allineate utilizzando il software LDI-PCR14 per identificare gli inserimenti di de novo LINE-1 e i siti di destinazione.
Risultati
Nel caso di TTC28-LINE-1, c'è più di un PAS all'interno di una finestra di 1 kb a valle del suo cognato PAS, da qui la regione tra il TTC28-LINE-1 PAS e il più forte PAS a 811 bp a valle è stata considerata come il tag unico per TTC28-LINE-1. Tre coppie di primer PCR inversa sono state progettate in questo tag unico che corrispondono a diversi PAS presenti14. Abbiamo selezionato tre enzimi di restrizione: (i) NsiI che taglia 5 a monte di TTC28-LINE-1 e 3 al di fuori del tag unico, e (ii) SacI e (iii) PstI che tagliano 5 all'interno del LINE-1 nella sua estremità lontano 5 e 3 al di fuori del tag unico. Questi generano frammenti di restrizione rispettivamente di 10.288 bp, 5.699 bp e 6.305 bp.
Per dimostrare questo metodo, abbiamo eseguito LDI-PCR sul DNA estratto dalla linea cellulare MCF7. Questa linea cellulare del cancro al seno è stato precedentemente segnalato per visualizzare tTC28-LINE-1 attività16. Per semplicità, abbiamo realizzato un modello di DNA circolare utilizzando un enzima di restrizione, SacI, su tre ed eseguito un LDI-PCR utilizzando una coppia di primer su tre (Tabella 1) per rilevare gli inserimenti de novo LINE-1 derivanti da TTC28- LINEA-1.
Una buona qualità del DNA estratto dalla linea cellulare MCF7 è stata garantita dall'elettroforesi gel di agarose (Figura 2). Il DNA intatto con peso molecolare dimostra che il DNA genomico è di qualità ottimale per questo test. Se invece uno striscio è visibile, ciò indica una scarsa qualità del DNA estratto, che a sua volta ostacolerà le procedure a valle.
La figura 3 mostra un risultato rappresentativo di un esperimento pcR con gradiente, volto a determinare la temperatura di annealing ottimale della coppia di primer inversi TTC28-LINE-1. Il DNA genomico del sangue digerito con SacI, seguito da auto-legazione per formare un modello di DNA circolare, è stato utilizzato per questa reazione. Un prodotto PCR altamente specifico di dimensioni attese (5.649 bp) a 62, 64 e 66 gradi centigradi mostra che la temperatura di fissazione ottimale per questa coppia di primer si trova all'interno dell'intervallo di 62,66 gradi centigradi.
Abbiamo generato un modello di DNA circolare digerindo il DNA genomico MCF7 con l'enzima di restrizione SacI seguito dall'auto-legazione. La figura 4 mostra che la trasduzione TTC28-LINE-1 3 è avvenuta nelle linee cellulari MCF7: gli inserimenti de novo possono essere rilevati come prodotti LDI-PCR di varie dimensioni insieme a un prodotto PCR nativo di dimensioni note (5.649 bp). Per identificare le coordinate genomiche dei siti di destinazione de novo, è possibile sequenziare gli amplificatori PCR (vedere la sezione protocollo 4).
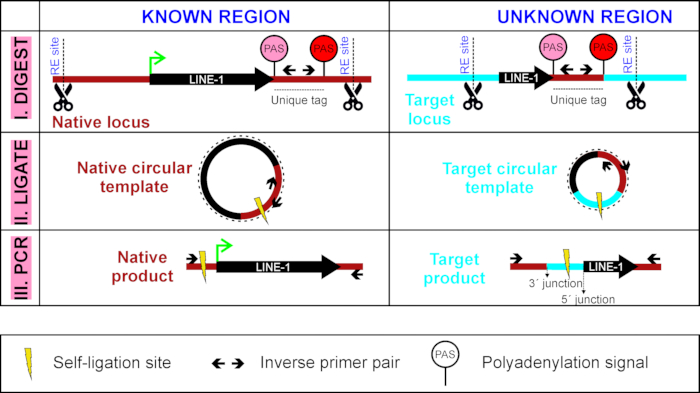
Figura 1 : Panoramica di LDI-PCR per rilevare la trasduzione LINE-1 3. Un modello di DNA circolare viene generato digerindo prima (I) con un enzima di restrizione e auto-ligating (II) esso. Questo passaggio è seguito da PCR inverso (III) con primer PCR inversa mirati al tag unico del LINE-1 di interesse (sequenza tra il PAS più debole di LINE-1, in rosa, e PIÙ forte PAS a valle, in rosso). Fare clic qui per visualizzare una versione più grande di questa figura.
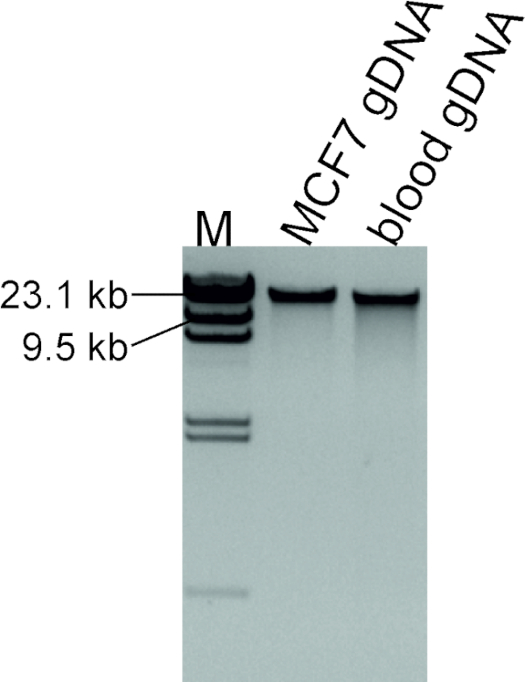
Figura 2 : valutazione della qualità del DNA estratto. 100 ng di DNA estratto dalla linea cellulare MCF7 e sangue da un individuo normale, che verrà utilizzato come campione di controllo, sono stati eseguiti insieme a 1 :L e 2 -L di marcatore DI DNA/HindIII etichettato come M. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
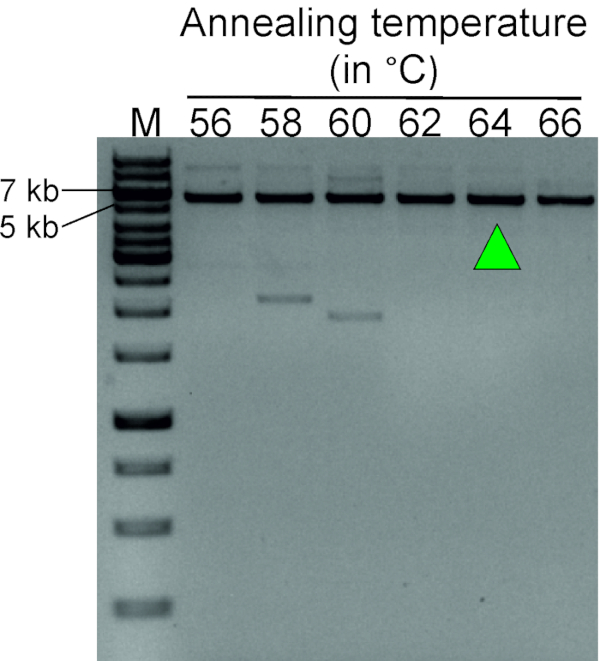
Figura 3 : Gradiente PCR per determinare la temperatura di analing ottimale per i primer PCR inversa (Tabella 1). LDI-PCR che utilizza coppie di primer inverse a temperature di annealing che vanno da 56 a 66 gradi centigradi mostra un prodotto PCR distinto a 62,66 gradi centigradi. La freccia verde indica la temperatura di analamento selezionata per esperimenti futuri. Modello di DNA circolare generato digerendo il DNA genomico del sangue da un individuo normale con SacI seguito da auto-legamento è stato utilizzato per questa fase di ottimizzazione. M, marcatore (1 kb più la scala del DNA). Fare clic qui per visualizzare una versione più grande di questa figura.
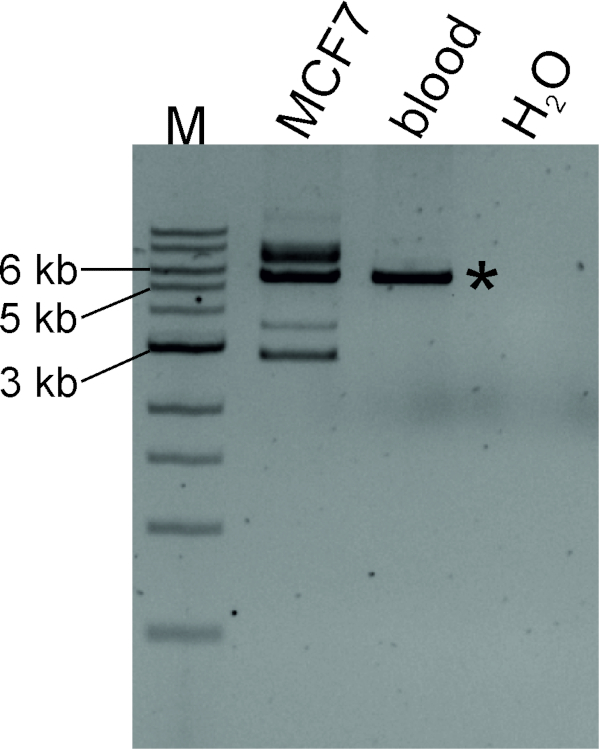
Figura 4 : LDI-PCR per identificare la trasduzione LINE-1 3 TTC28 -LINEA-1. I modelli circolari di DNA generati digerino il DNA genomico MCF7 e sangue (da individuo normale) con SacI seguito da auto-legamento sono stati amplificati da primer inversi in temperatura di accaralazione ottimale. Il prodotto PCR "nativo", contrassegnato con asterisco, delle dimensioni previste (5.649 bp) è stato rilevato sia nel DNA MCF7 che nel DNA sanguigno normale, mentre MCF7 produceva anche prodotti PCR aggiuntivi di varie dimensioni, indicando la retrotrasposizione de novo LINE-1. M, marcatore (1 kb di scala del DNA). Fare clic qui per visualizzare una versione più grande di questa figura.
| Nome primer | Sequenza (5 x 3) |
| L1_001 (rev) | TTCACTAAGCATGTGTGGGGAAAC |
| L1_002 (fwd) | CCCAAAATATACCCAATTATGGCA |
Tabella 1: coppia di primer PCR inversa progettata per l'etichetta TTC28 -LINEA-1 14 Del sistema .
File supplementare. Fare clic qui per scaricare questo file.
Discussione
Qui descriviamo un metodo che può essere utilizzato per identificare gli inserimenti de novo LINE-1 derivanti da qualsiasi LINE-1 attivo di interesse. Abbiamo ottimizzato questo metodo per un LINE-1 altamente attivo, situato a 22q12.1, e in precedenza dimostrato di essere altamente sensibile nel rilevamento di inserimenti sotto-clonali nel cancro colorettale14.
Il successo di LDI-PCR dipende dalla qualità del DNA genomico. Pertanto, abbiamo incluso un ulteriore passaggio di controllo della qualità per garantire che all'inizio del protocollo sia presente dna ad alto peso molecolare (passaggio 2.1.2). Si consiglia di conservare il DNA genomico a -20 gradi centigradi per lo stoccaggio a lungo termine e di preparare l'aliquota al fine di evitare cicli di congelamento e scongelamento. L'uso del DNA genomico proveniente dal sangue o dal tessuto normale abbinato al paziente è altamente raccomandato per distinguere se la retrotrasposizione LINE-1 rilevata è una linea germinale o un evento somatico. Poiché i siti tagliati per gli enzimi di restrizione sono stocastici nel genoma, è possibile che un particolare sito di inserimento de novo LINE-1 non possa ospitare siti tagliati per l'enzima di restrizione utilizzato nelle sue vicinanze. Quindi, per aumentare la probabilità di rilevare la maggior parte degli inserimenti de novo LINE-1 nel DNA tumorale, più di un enzima di restrizione dovrebbe essere utilizzato in reazioni separate per generare diverse librerie di modello di DNA circolare. Inoltre, se il tag unico del LINE-1 di interesse ha più di un PAS, l'utilizzo di coppie di primer adiacenti a ogni PAS migliora le possibilità di rilevare trasduzioni fortemente troncate.
Sebbene esistano metodi eleganti per il rilevamento a livello di genoma degli inserimenti di de novo LINE-1, possono essere schiaccianti se l'obiettivo è quello di sondare la competenza retrotrasposizione di un particolare LINE-1 in uno specifico contesto cellulare. A questo scopo, LDI-PCR può essere un approccio economico e semplice ma robusto per visualizzare gli eventi retrotrasposizione LINE-1. L'approccio di targeting utilizzato in questo metodo è simile a TS-ATLAS22; tuttavia, LDI-PCR evita di utilizzare oligonucleotidi del linker e può amplificare contemporaneamente sia le giunzioni a 5 e 3 giunzioni di inserimento de novo LINE-1. Le informazioni relative sia alle giunzioni a 5 e 3 giunzioni dell'inserimento LINE-1, al sito di destinazione dell'integrazione, alla coda poliA e alle modifiche del sito bersaglio, tutte caratteristiche della retrotrasposizione LINE-1, possono essere ottenute accolando LDI-PCR a una singola molecola tecnologie di sequenziamento a lunga lettura. Le letture lunghe così generate contengono la sequenza LINE-1 inserita, il suo tag unico e le sequenze di destinazione in una sola lettura, eludendo le difficoltà di mappatura delle letture brevi nella regione ripetitiva.
L'utilizzo di LDI-PCR per il rilevamento dell'attività line-1 è costituito da due limitazioni principali. Il primo è inerente alla PCR: può amplificare solo in modo affidabile frammenti fino a 10 kb di dimensioni. Questo dovrebbe essere considerato durante la selezione di enzimi di restrizione, come il frammento nativo non deve superare questo limite. In secondo luogo, questo metodo è in grado di rilevare solo gli eventi retrotrasposizione che mobilitano la regione di fiancheggiamento di LINE-1 di 3 di 3 di 3 traslazioni. Quindi, l'attività di quei LINE-1 che non presentano 3 - trasduzione non sarà rilevata utilizzando questo metodo. Inoltre, nonostante siano stati amplificati da LDI-PCR, alcuni eventi retrotrasposizione LINE-1 che (a) generano un bersaglio PCR di dimensioni simili a quelle della posizione "nativa" o di altre retrotrasposizioni o (b) sono rare o subclonali, potrebbero non essere rilevati da gel agarose Elettroforesi. Tali eventi di retrotrasposizione LINE-1 possono essere catturati sequenziando il prodotto LDI-PCR utilizzando tecnologie di sequenziamento a lunga lettura a singola molecola14.
Il flusso di lavoro qui descritto può essere facilmente modificato per rilevare l'attività di altri LINE-1 "caldi" utilizzando un enzima di restrizione adatto e progettando primer inversi mirati a questi LINE-1. Oltre al rilevamento della trasduzione MEDIAta da LINE-1 3', questo metodo può essere adattatoper rilevare le trasduzioni line-1 mediate meno frequenti 5. Metodo simile sono stati utilizzati per identificare il sito di integrazione dei reporter LINE-1 nei saggi basati sulle cellule24 e nei siti di integrazione provirale nel cancro25. Oltre agli inserimenti di LINE-1, questo metodo può essere utilizzato anche per rilevare altre aberrazioni genomiche, come il riarrangiamento del DNA, dove le informazioni riguardanti la regione soggetta a riarrangiamento pre-esistono26.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Vorremmo ringraziare tutti i nostri co-autori nell'articolo in cui questo metodo è stato descritto per la prima volta14, in particolare Tatiana Cajuso, Kimmo Palin, Outi Kilpivaara e Esa Pitkànen per le discussioni preziose durante lo sviluppo del metodo. L.K. è finanziato dall'Accademia di Finlandia (numeri di sovvenzione 25996, 292789, 306026 e 314394), dalla Fondazione Sigrid Juselius e dalla Società finlandese per il cancro. B.P. è stato insignito della borsa di studio PhD dell'Università di Helsinki Research Foundation, di una borsa di studio della Società finlandese per il cancro e di una borsa di ricerca di dottorato presso Ida Montinin S. Ringraziamo anche Teemu Masalin (Università di Helsinki) e Kul Shrestha del gruppo di ricerca di L.K. per averci aiutato nella produzione video.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
Riferimenti
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16(2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343(2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. Jr High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043(2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521(2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Jr Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484(2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon