Method Article
Uzun mesafe ters PCR tarafından sıcak hat-1s Retrotransposition aktivitesinin tespiti
Bu Makalede
Özet
Bu makalede, aktif bir hat-1 retrotransposon aktivitesini izlemek ve belirli bir genom de Novo retrotranspositions eşlemek için basit bir PCR tabanlı tahlil özetlenmektedir. MCF7 hücre hattını kullanarak, burada bu yöntemin 22q 12.1 'de bulunan bir satır 1 ' in aktivitesini algılamak için nasıl uygulanacağını gösteriyoruz.
Özet
Uzun serpiştirilmiş nükleer elemanları 1 (hat-1s) otonom hareket edebilir insan genomu mobil genetik öğelerin tek ailesidir. Onlar bu yüzden bir süreç olarak adlandırılan retrotransposition onlar sonra sonuç olarak ters transkripsiyon tarafından genom içine yerleştirilen bir mRNA ara oluşturmak için nasıl. Normal hücrelerde sessiz olmasına rağmen, LINE-1s farklı epitelyal tümörlerde son derece aktif. De Novo LINE-1 eklemeler potansiyel olarak tumorigenesis sürücü ve bu nedenle sistemik hat-1 retrotranspozis kanserde çalışma önemlidir. Out of ~ 150 retrotransposition-yetkili hat-1s insan genomu mevcut, LINE sadece bir avuç-1 loci, aynı zamanda "sıcak" LINE-1s olarak da adlandırılır, farklı kanser türlerinde de Novo Line-1 ekleme çoğunluğu için hesap. Biz basit bir polimeraz zincir reaksiyonu geliştirdik (PCR)-bu sıcak hat-1s retrotransposition etkinliğini izlemek için tabanlı yöntem. Uzun mesafe ters (LDı)-PCR dayalı bu yöntem, 3 ' dönüştürücü, bir hat-1 tarafından daha sonra da Novo LINE-1 3 ́ dönüştürücü olayları tanımlamak için kullanılabilir kendi çevreleme olmayan bölge, harekete geçen bir mekanizma yararlanır belirli bir sıcak hat-1 kaynaklanan.
Giriş
Uzun serpiştirilmiş nükleer elemanları (LINE-1s), geriye dönük olarak adlandırılan bir kopya ve yapıştırma mekanizması ile bağımsız olarak bir yerden diğerine hareket edebilir retrotranspozonları denilen mobil genetik elemanlar bir ailelerdir. Evrimsel zaman içinde, insan genomu satır-1 tekrarlar1' den fazla 500.000 kopyalarını birikmiş. Ancak, genomda bulunan satır-1 kopyalarının çoğu mutasyona uğramış ve bu nedenle retrotranspozite yoluyla hareket edemez; sadece ~ 150 kopya onlar2taşımak IÇIN gerekli DNA dizisi bozulmamış kopyası var. Normal somatik hücrelerde, bu LINE-1s mobilitesini farklı konak faktörleri3ile kısıtlanır. Bu kısıtlamalar farklı epitelyal tümörlerde rahatlıyor, LINE-1s ' l e neden derepressed ve tümörün genomu4' te birçok de Novo insersiyonları ortaya çıkacak. Bu tümör ilişkili de Novo eklemeler bazıları genlerde insersiyon mutagenez neden gösterildi, dolayısıyla tümör ilerlemesi sürüş5,6. Bu nedenle, tümör genomunda yeni eklemeler eşlemesini muktedir önemlidir.
De Novo Line-1 eklemeler algılamak için mevcut Yöntemler (1) tüm genom sıralama yaklaşımı4,7,8, burada farklı Hesaplamalı algoritmalar de Novo Line-1 eklemeler bulmak için kullanılır WGS verilerinden veya (2) yeni nesil sıralama, Genç, potansiyel olarak aktif hat-1s9,10,11,12,13' ün 3 ' ü sonuna kadar hedefler. Ancak, bu yöntemlerle birkaç bin yakın özdeş kopya arasında roman eklemeler bulmak önemsiz uzak, ve zorluk daha fazla tümör heterojenite ve hat-1 ekleme4ile ilişkili genomik değişiklikler tarafından ağırlanmış olduğunu.
Bu mevcut yöntemleri kullanarak çalışmalar gösterdi ki sadece birkaç satır-1s büyük çoğunluğu için hesap de Novo LINE-1 eklemleri tümörlerde gözlenen7,8. Bu nedenle, belirli bir tümör örneği ÇIZGI-1 aktivite görüntüler olup olmadığını yanıtlamak için, bu son derece aktif LINE-1 loci bu avuç neden retrotransposition olayları eşlemek için yeterlidir. Bu makalede, biz basit bir polimeraz zincir reaksiyonu açıklamak (PCR)-tabanlı Yöntem14 belırlı bir hat aktivitesini izlemek için kullanılabilir-1 Locus TTC28 geni ilk intron içinde 22q 12.1 son derece Kolorektal kanserde aktif 7 , 8. bu hat-1 Locus olarak adlandırılır TTC28-LINE-1 Makale boyunca. Bu tahlil özellikle de Novo Line-1 retrotransposition olayları, 3 ' dönüştürücü15denilen bir mekanizma tarafından kaynak satır-1 ' in 3 ' yan bölgesinde tekrarsız sırasını seferber tanımlar. 3 ' dönüştürücü, zayıf hat-1 polyadenilasyon sinyali (pas) nedeniyle gerçekleşir ve bunun yerine, transkripsiyon makinelerinin onu atlamasına ve daha güçlü pas alt kısmında transkripsiyon sonlandırılmasına neden olmaktadır ( Bundan sonra satır-1 dizisinin yanında hedef konuma eklenen "benzersiz etiket" olarak adlandırılır). Philippe ve ark.16 son zamanlarda farklı hücre TÜRLERI farklı LINE-1 loci ifade edebilir gösterdi. Bu bulgu ışığında, bu yöntem en çok ifade edilen satır-1 etkinliği izlemek için uygulanabilir, ilgi kanser türü kendi benzersiz etiket seferber.
LDı-PCR ' d a ilk adım, satır-1 ' i (burada, TTC28-Line-1) ve benzersiz etiketini (Şekil 1) içeren bir kısıtlama parçası üreten bir kısıtlama enzimiyle genomik DNA 'nın sindirimi. Sindirilmiş DNA daha sonra Self-ligasyon ve PCR benzersiz etiketi içinde bulunan ters astar kullanılarak amplifikatörlü tarafından circularized. Bunu yaparak, tam uzunluklu kaynak hattı-1 kendi "yerel" konumda her zaman amplifikatörleşmiş ve yanında, yavru ÇIZGI-1 eklemeler farklı hedef loci benzersiz etiketi içeren de amplifikatörleşmiş olacak (Şekil 1), böylece raporlama söz konusu satır-1 retrotransposition aktivitesi.
Protokol
Bu araştırma Helsinki Üniversitesi Hastanesi kurumsal Inceleme kurulu ve Etik Komitesi tarafından onaylanmıştır. Bu protokolü göstermek için kullanılan kan numunesi için konu tarafından imzalanan bilgilendirilmiş onay alındı.
1. ters astar tasarımı ve kısıtlama enzimleri seçme (Biyoinformatik)
- SATıR-1 ilişkili benzersiz etiketi belirleme
- TTC28-Line-1 DIZISINI FASTA formatında, L1Base17gibi bir satır-1 veritabanından indirin. TTC28-LINE-1 IÇIN L1base KIMLIĞI 135 ' dir.
- SATıR-1 dizisinin hem 5 ' ve 3 ́ uçlarını flanşla 5 KB serisi dahil et ve bir kelime işlemcisine ek açıklama ekle.
Not: burada satır-1 yan sıra kahverengi yazı tipi ve satır-1 sıra gri yazı tipi (ek dosya) olarak açıklamalı. - Seçilen LINE-1 ' i n bilat pas aşağı aşağı 1kb sıra polyadq18 veya Dragon Polya gözcü19 gibi bir pas tahmin aracına girin ve bu 1 KB penceresinde tüm Poliadenilasyon sinyali açıklama.
Not: 1 KB penceresinde PAS yok ise, sonraki 1 KB penceresinde aşağı aşağı PAS arayın. TTC28-LINE-1 ' i n kendı zayıf pas pembe olarak vurgulanır ve DIĞER tüm pas (ilave dosya) kırmızı vurgulanır - LINE-1 ' i n bilat PAS ve "benzersiz etiket" olarak en güçlü PAS akış sonu arasındaki sıraya açıklama ekleyin.
Not: "benzersiz etiket" TTC28-LINE-1 sarı (ek dosya) vurgulanır.
- Ters astar tasarımı
- Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) veya NCBı 'nın primer-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) gibi Web tabanlı bir astar tasarımı aracına "benzersiz etiket" dizisini girerek ters PCR astarları tasarlayın. Bu aletler tarafından tasarlanan astar çiftleri, geleneksel PCR kolaylaştırılması birbirlerine karşı karşıya beri, ters PCR gerçekleştirmek için astar çifti için ters tamamlayıcı işlevini kullanın.
Not: LINE-1 dönüştürücüleri ağır kendi 5 ́ sonunda kesilmiş ve transrik bölgenin boyutu çok değişkendir, iki ters astar minimum arasındaki mesafeyi tutmak amacı, NCBı primer-BLAST "PCR ürün uzunluğu" parametresini ayarlayarak Minimum. Benzersiz etiket içinde birden fazla PAS durumunda, farklı PASs karşılık çeşitli astar çiftleri tasarım. Hat-1 eklemeleri RNA orta ve 5 ' End den başlatmak olarak pas, yakın tasarım astarlar farklı kesilir. Burada üç astar çiftleri, TTC28 LINE-1 (ek dosya) benzersiz etiketinde üç güçlü Poliadenilasyon sinyalleri karşılık gelen, deniz göbeği ve yeşil vurgulanır, tasarlanmıştır.
- Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) veya NCBı 'nın primer-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) gibi Web tabanlı bir astar tasarımı aracına "benzersiz etiket" dizisini girerek ters PCR astarları tasarlayın. Bu aletler tarafından tasarlanan astar çiftleri, geleneksel PCR kolaylaştırılması birbirlerine karşı karşıya beri, ters PCR gerçekleştirmek için astar çifti için ters tamamlayıcı işlevini kullanın.
- Kısıtlama enzimleri seçme
- SATıR-1 dizisini, RestrictionMapper gibi Web tabanlı aracı kullanarak silico 'da 5 KB upstream ve downstream flanşları ile birlikte özetler. Bu, farklı kısıtlama parçaları üreten bu bölgeyi kıracak olan kısıtlama enzimlerinin kapsamlı bir listesini verecektir.
- LINE-1 ' i n Yerel yerlerini aşağıdaki gibi kestiği kısıtlama enzimleri seçin: 5 ́ sonunda, hat-1 ' i k i 5 ' End veya Far 5 ' End LINE-1 ' in kendisi ve 3 ́ sonunda, LINE-1in benzersiz etiketinin aşağı yukarı.
Not: seçilen kısıtlama enzimi DNA metilasyonu için duyarlı olmalıdır, ısı-inactivatable olmalıdır, ve birbirine tamamlayıcı olan "yapışkan" biten sendeleyerek oluşturmalıdır. LDı-PCR göstermek için, GAGCTC sitelerinde DNA keser sacı kısıtlama enzim, açık yeşil burada vurgulanır, kullanılır (ek dosya). - Seçilen kısıtlama enzimleri tarafından yapılan kısıtlama parçası boyutunu not alın. Bu, PCR tarafından verimli bir şekilde amplifikatörde olmayabilir gibi bu 12 KB 'den uzun olmamalıdır.
2. uzun mesafeli ters PCR için dairesel DNA şablonları yapma
- DNA ekstraksiyon ve kalite değerlendirmesi
- Üreticinin talimatlarına göre LDı-PCR için gerekli kaliteli, yüksek moleküler ağırlık DNA ayıklamak ticari olarak kullanılabilir DNA ekstraksiyon kitleri kullanarak örneklerden (tümör veya kan) genomik DNA ayıklamak. Alternatif olarak, yüksek moleküler ağırlık DNA da Phenol tarafından ayıklanabilir: kloroform20.
- Üreticinin talimatlarına göre bir Fluorometre kullanarak DNA konsantrasyonunu ölçün ve 100 ng DNA 'yı 1% (w/v) agaroz jel içeren etidium bromür (0,5 μg/ml) içinde 1x Tris-asetat-EDTA (Tae) tampon olarak 4,5 v/cm21 ' de λ-hindiii ile birlikte çalıştırın DNA Moleküler Ağırlık belirteçleri DNA kalitesini ve miktarını kontrol etmek.
- Genomik DNA 'Yı sindirme
- 20 adet sacı kısıtlama enzimi, 5 μL 10X reaksiyon tamponu (malzeme tablosu), 100 ng DNA (44 μL 'ye kadar), buz üzerindeki bir 0,2 ml PCR tüpte (en çok üreticilerden 1 μL kısıtlama enzimi) ekleyerek bir sindirim reaksiyonu karışımı (50 μL son hacmi) olun 100 ng genomik DNA 'yı tamamen sindirmek için yeterli s). Tüp Flicking ve kısaca santrifüjle çözüm karıştırın.
- 37 °C ' de 1 h için reaksiyon karışımından, 65 °C ' de 5 dakika ısı inaktivasyonu takiben bir termal cycler kullanın.
- Sindirilmiş genomik DNA 'Yı kendinden bağlama
- 50 μL sindirim karışımı (adım 2.2.2 sonra) için 8 μL 10X T4 DNA ligaz tampon, 1 μL (5 adet) T4 DNA ligaz ve 21 μL ultra saf su, 80 μL son reaksiyon hacminin yapmak için ekleyin. tüpler Flicking tarafından çözüm karıştırın ve kısa bir süre Santrifüjü.
- 22 °C ' de 10 dakika boyunca ısı inaktivasyon adımıyla 65 °C ' de 10 dk. sona erdirmek için bir termal bisikletçiyle inkükleyici.
3. uzun mesafeli ters PCR
- Degrade PCR ile primer tavlama sıcaklığını belirleme
- (A-4) °C, (a-2) °C, A, (A + 2) °C, (A + 4) °C ' nin bir tavlama sıcaklığı degradesini, ticari satıcının Web tabanlı aracı kullanılarak hesaplanan primer çifti teorik tavlama sıcaklığına ayarlayın.
Not: bazı üreticilerin termal cycler sıcaklık gradyan el ile ayarlanması izin vermeyebilir. Bu durumda, otomatik degrade ayarı (a-4) °C (a + 4) °C sıcaklık aralığı ile kullanılabilir. - 1,5 mL mikrosantrifüj tüpünde aşağıdaki bileşenleri birleştirerek ve karıştırarak PCR için bir ana karışım hazırlayın: 4 μL 5x reaksiyon tampon, 0,4 μL 10 mM dNTP, 5 μL 2 μM PCR astar (ileri ve geri, adım 1.3.1 ' de tasarlanmıştır) , 0,2 μL (0,1 U) reaksiyon başına DNA polimeraz. Degradenin her tavlama sıcaklığı için bir reaksiyon ayarlayın.
- Her reaksiyon için, 0,2 ml PCR tüplerine ana karışımını 19 μL kısım ve Bölüm 2 ' de yapılan dairesel DNA şablonu 1 μL (1,25 ng) ekleyin.
Not: Bu optimizasyon adım için potansiyel olarak değerli tümör DNA tüketen önlemek için şablon olarak normal kan DNA oluşturulan dairesel Self-ligated DNA kullanın. - Gradyan PCR programını aşağıda açıklandığı gibi termal bisikletçiler üzerinde çalıştırın: (i) 98 °C ' de 3 dk (denaturasyon) bir döngü; (ii) 35 (98 °C ' de 10 sn [denaturation], [A-4] ' a [a + 4] °C [tavlama] ve 1 − 6 dk [30 sn/beklenen PCR ürününün kiloüssü başına 20 sn), 72 °C ' de [uzatma]); (iii) 72 °C ' de 10 dakikalık bir döngü (Final uzatma).
- 4,5 V/cm 'de 1x Tae tampon olarak hazırlanan% 1 agaroz jel21 ' de PCR ürününün 6 μL 'ini çalıştırın ve farklı tavlama sıcaklıklarında elde edilen PCR ürünlerini analiz edin.
- Beklenen boyuta karşılık gelen bir PCR ürünü veren tavlama sıcaklığını seçin.
- (A-4) °C, (a-2) °C, A, (A + 2) °C, (A + 4) °C ' nin bir tavlama sıcaklığı degradesini, ticari satıcının Web tabanlı aracı kullanılarak hesaplanan primer çifti teorik tavlama sıcaklığına ayarlayın.
- Tümör genomunda de Novo LINE-1 retrotranspozit aktivitesinin tespiti
- Tümör örneklerinden oluşturulan dairesel DNA şablonlarında ters PCR astar çiftini kullanarak LDı-PCR gerçekleştirin (Bölüm 2). Degrade PCR (Bölüm 3,1) için aynı talimatları izleyin, ancak bu kez sıcaklık degradesini optimum tavlama sıcaklığına göre değiştirin.
- 3.1.5 adımda yapılan agaroz jel elektroforezi ile PCR ürünlerini analiz edin. PCR ürün bilinen boyutu veya "yerel" PCR ürün LINE-1 karşılık gelen yerel Locus her reaksiyon için görünür olmalıdır. De Novo Satır-1 3 ́ 'deki tümör örneğinde dönüştürücü, farklı boyutlarda PCR ürünleri olarak algılanabilir, agaroz jeldeki yerli PCR ürünü ile birlikte.
4. LINE-1 3 ́ dönüştürücü için hedef sitelerin kimliğini ortaya çıkarmak için LDı-PCR ürünlerini sıralama
- Bu hat-1 3 ' dönüştürücü olayların hedef entegrasyon sitelerini belirlemek için her LDI-PCR reaksiyonunda oluşturulan tüm PCR amplikonlar tek molekül uzun okuma sıralaması gerçekleştirin.
Not: LDı-PCR ürünlerinin klonlanması ve Sanger dizilimi, aynı zamanda hantal, yaklaşım da olabilir. - Standart sıralı hizalama ardışık düzenlerini kullanarak tek molekül uzun okunan sıralama platformları tarafından üretilen okuma başvuru genomuna hizalayın. De Novo LINE-1 eklemeler ve hedef sitelerini tanımlamak için LDı-PCR yazılımı14 kullanarak hizalanmış okuma analiz edin.
Sonuçlar
TTC28-Line-1 durumunda, onun BILAT pas 1 KB 'lik bir pencere içinde bırden fazla pas var, bu nedenle TTC28-Line-1 PAS ve en güçlü pas 811 BP aşağı arasındaki bölge TTC28-Line-1 için benzersiz etiket olarak kabul edildi. Üç ters PCR astar çifti, farklı PASs Present14' e karşılık gelen bu benzersiz etikette tasarlanmıştır. Biz üç kısıtlama enzimleri seçti: (i) nsiı keser 5 ' upstream TTC28-LINE-1 ve 3 ́ benzersiz etiket dışında, ve (ii) sacı ve (iii) PSTı bu kesme 5 ' LINE-1 içinde onun uzak 5 ́ sonu, ve 3 ' benzersiz etiket dışında. Bunlar sırasıyla BP, 5.699 BP ve 6.305 BP 10.288 kısıtlama parçaları üretir.
Bu yöntemi göstermek için, MCF7 hücre hattından çıkarılan DNA 'da LDı-PCR ' y i gerçekleştirdik. Bu meme kanseri hücre hattı daha önce TTC28-Line-1 aktivite16görüntülemek için bildirilmiştir. Basitlik için, bir kısıtlama enzimi kullanarak dairesel bir DNA şablonu yaptık, sacı, üç dışarı ve bır LDı-PCR bir astar çifti kullanarak gerçekleştirilen üç (Tablo 1) TTC28den kaynaklanan de Novo LINE-1 eklemeler algılamak için- SATıR-1.
MCF7 hücre hattından çıkarılan DNA 'nın iyi kalitesi agaroz jel elektroforezi ile sağlandı (Şekil 2). Bozulmamış yüksek moleküler ağırlığı DNA genomik DNA bu tahlil için optimum kalitede olduğunu gösterir. Bir smear yerine görünüyorsa, bu ayıklanan DNA kalitesiz gösterir, hangi sırayla aşağı aşağı prosedürleri engel olacaktır.
Şekil 3 , TTC28-LINE-1 ters astar çifti için optimum tavlama sıcaklığını belirlemeyi amaçlayan bir degrade PCR deneyinin temsili bir sonucunu gösterir. Sacı ile sindirilmiş kan genomik DNA, bu reaksiyon için DAIRESEL bir DNA şablonu oluşturmak için kendinden ligasyon izledi. 62, 64 ve 66 °C ' de beklenen boyutta (5.649 BP) son derece spesifik bir PCR ürünü, bu astar çifti için optimum tavlama sıcaklığının 62 − 66 °C aralığında olduğunu gösterir.
Sacı kısıtlama ENZIMIYLE MCF7 genomik DNA 'yı sindirerek DAIRESEL bir DNA şablonu ürettik. Şekil 4 TTC28-LINE-1 3 ' dönüştürücü MCF7 hücre çizgilerinin oluştuğunu gösterir: de novo eklemeler bilinen boyutu (5.649 BP) yerel bir PCR ürün ile birlikte farklı boyutlarda LDI-PCR ürünleri olarak tespit edilebilir. De Novo hedef sitelerinin genomik koordinatlarını belirlemek için PCR amplikonlar sıralı olabilir (bkz. protokol bölümü 4).
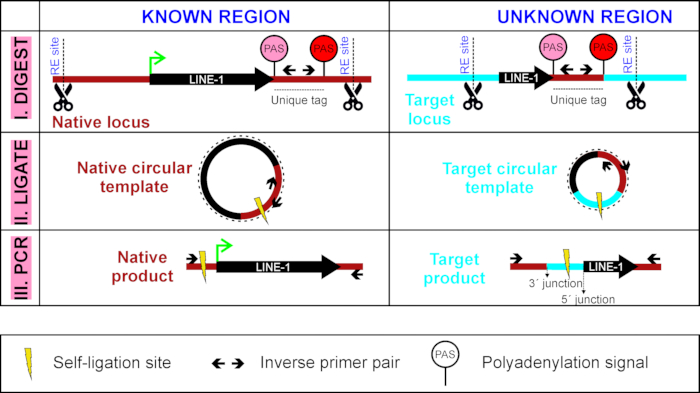
Şekil 1 : LINE-1 3 ' dönüştürücü algılamak için LDI-PCR ' e genel bakış. Dairesel bir DNA şablonu ilk sindirme (ı) ile bir kısıtlama enzim ve Self-ligating (II) ile oluşturulur. Bu adım, ters PCR (III) ile, hat-1 ' i n benzersiz etiketini hedefleyen (LINE-1' in kendi zayıf PAS, pembe ve daha güçlü PAS aşağı, kırmızı) bir ÇIZGI ile karşı çevrir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
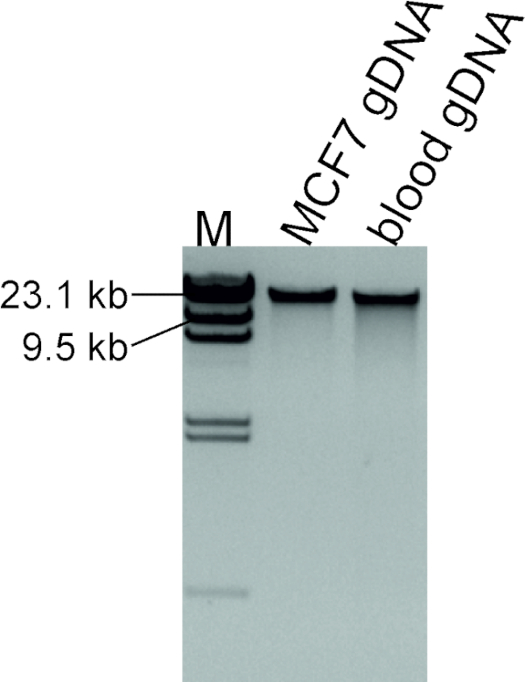
Şekil 2 : ÇıKARıLAN DNA 'Nın kalite değerlendirmesi. 100 bir kontrol örneği olarak kullanılacak olan normal bir bireyin MCF7 hücre hattından ve kandan çıkarılan DNA 'Sı, M. olarak etiketli 1 μL ve 2 μL, a. bu figürün daha büyük bir versiyonunu görüntülemek Için lütfen buraya tıklayın .
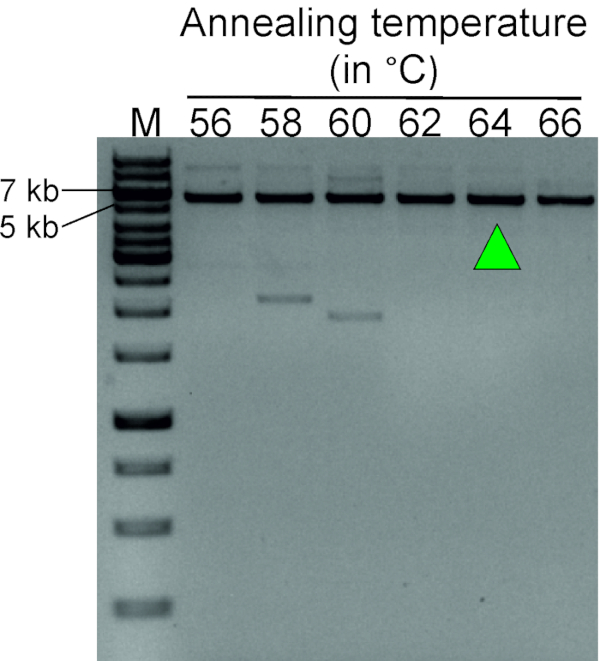
Şekil 3 : Ters PCR astar için optimum tavlama sıcaklığını belirlemek üzere Gradyan PCR (Tablo 1). 56 ila 66 °C arasındaki tavlama sıcaklığında ters astar çiftleri kullanan LDı-PCR, 62 − 66 °C ' de farklı bir PCR ürününü gösterir. Yeşil ok, gelecekteki deneyler için seçilen tavlama sıcaklığını gösterir. Dairesel DNA şablonunda kan genomik DNA 'Yı sindirerek oluşturulan normal bir bireyin sacile takip ettiğim bu optimizasyon adımı için Self-ligasyon kullanıldı. M, Marker (1 KB artı DNA merdiven). Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
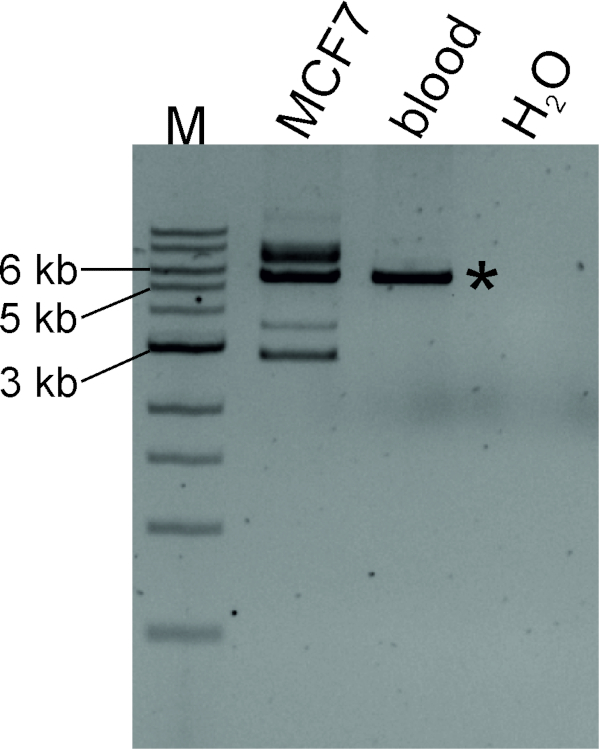
Şekil 4 : LDI-PCR, LINE-1 3 ' ı tespit etmek için TTC28 -LINE-1. MCF7 ve kan sindirerek oluşturulan dairesel DNA şablonları (normal bireysel) genomik DNA ile sacile birlikte kendi kendine ligasyon, optimum tavlama sıcaklığında ters astar tarafından güçlendirilmiş. "Yerli" PCR ürün, yıldız işareti ile işaretlenmiş, beklenen boyutu (5.649 BP) hem MCF7 DNA ve normal kan DNA algılandığında, MCF7 de Novo LINE-1 retrotranspozit gösteren farklı boyutlarda ek PCR ürünleri üretti. M, Marker (1 KB DNA merdiven). Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
| Primer adı | Sıra (5 ́ → 3 ́) |
| L1_001 (devir) | Mustafa aktaş |
| L1_002 (FWD) | Bu BIR |
Tablo 1: benzersiz etiket için tasarlanan ters PCR astar çifti TTC28 -LINE-1 14 .
Tartışmalar
Burada de Novo Line-1 eklemeleri herhangi BIR etkin hat-1 ilgi kaynaklanan tanımlamak için kullanılabilecek bir yöntem açıklanmaktadır. Bu yöntemi, 22q 12.1 'de bulunan son derece aktif bir hat-1 için optimize ettik ve daha önce kolorektal kanser14' te alt klonal eklemeler tespit etmek için son derece hassas olduğunu göstermiştir.
LDı-PCR başarısı genomik DNA 'nın kalitesine bağlıdır. Bu nedenle, protokolün başlangıcında, yüksek moleküler ağırlık DNA 'Sı mevcut (adım 2.1.2) olduğundan emin olmak için ek bir kalite kontrol adımı dahil ettik. Uzun süreli depolama için genomik DNA 'yı-20 °c ' de saklamanızı ve donma ve çözme döngülerini önlemek için plakaya hazırlamak öneririz. Kan veya hasta uyumlu normal dokudan genomik DNA 'Yı kullanarak, hat-1 retrotranspozin algılanıp bir germline veya somatik bir olay olup olmadığını ayırt etmek çok tavsiye edilir. Sınırlama enzimleri için kesme siteleri genomda Stokastik olduğundan, belirli bir de Novo LINE-1 ekleme sitesi çevresinde kullanılan kısıtlama enzimi için herhangi bir kesim siteleri liman olmayabilir mümkündür. Bu nedenle, tümör DNA 'sında de Novo LINE-1 eklemeler çoğunluğu tespit olasılığını artırmak için, birden fazla kısıtlama ENZIM dairesel DNA şablonu farklı kütüphaneler oluşturmak için ayrı reaksiyonlar kullanılmalıdır. Ayrıca, Eğer LINE-1 ilgi benzersiz etiket birden fazla PAS varsa, daha sonra her PAS bitişik primer çiftleri kullanarak ağır kesilmiş transssiyonları tespit şansını geliştirir.
De Novo Line-1 eklemeleri genom çapında tespiti için zarif yöntemler var olsa da, amaç belirli bir hücre bağlamında belırlı bir hat-1 retrotransposition yetkinlik prob ise, onlar ezici olabilir. Bu amaçla, LDı-PCR LINE-1 retrotransposition olaylarını görselleştirmek için ucuz ve basit ama sağlam bir yaklaşım olabilir. Bu yöntemde kullanılan hedefleme yaklaşımı TS-ATLAS22' ye benzer; Ancak LDı-PCR, bağlayıcı oligonükleotidler kullanmaktan kaçınır ve aynı zamanda Novo LINE-1 eklemeleri için 5 ' ve 3 ́ kavşlarını aynı anda güçlendirebilir. Hat-1 ekleme, hedef site entegrasyonu, polyA kuyruğu ve hedef-site modifikasyonları, her biri LINE-1 retrotranspozis, her ikisi de 5 ' ve 3 ' kavşağıyla ilgili bilgiler, LDı-PCR tek molekül ile bağlama ile elde edilebilir uzun okunan sıralama teknolojileri. Uzun okur böylece oluşturulan eklenen LINE-1 sıra, benzersiz etiketi ve hedef sıraları tek bir okuma, Çift eşleme sorunları, tekrarlayan bölgede kısa okur.
SATıR 1 etkinliğini algılamak için LDı-PCR kullanmanın iki büyük kısıtlaması vardır. İlk PCR doğaldır: sadece güvenli bir şekilde 10 KB boyutunda parçaları yükseltmek olabilir. Yerel parça bu sınırı aşmamalıdır gibi kısıtlama enzim (ler), seçerken bu dikkate alınmalıdır. İkincisi, bu yöntem yalnızca satır-1 ' i n 3 ' yan bölgesini 3 ' transmetitle seferber eden retrotranspozite olaylarını algılayabilir. Bu nedenle, bu hat-1s aktivite 3 ' dönüştürücü sergileyen bu yöntem kullanılarak algılanacaktır değil. Ayrıca, LDI-PCR tarafından güçlendirilmesine rağmen, (a) "yerli" konum veya diğer retrotranspozitlerle veya (b) nadir veya subclonal olarak benzer boyutta bir PCR hedefi üreten, bazı LINE-1 retrotransposition olaylar agaroz jel tarafından algılanamayabilir Elektroforez. Bu tür satır-1 retrotransposition olayları tek molekül uzun okunan sıralama teknolojileri14kullanarak LDI-PCR ürün sıralamayı tarafından yakalanabilir.
Burada açıklanan iş akışı, uygun bir kısıtlama enzimi kullanarak diğer "sıcak" satır-1s etkinliğini algılamak ve bu LINE-1s ' i hedefleyen ters astar tasarımı yaparak kolayca değiştirilebilir. LINE-1 aracılı 3 ' dönüştürücü tespitine ek olarak, bu yöntem daha az sıklıkta hat-1 aracılı 5 ' dönüştürücü23' ü tespit etmek için uyarlanabilir. Benzer yöntem, hücre tabanlı deneyleri24 ve kanser25proviral entegrasyon siteleri LINE-1 gazetecilerin entegrasyon site tanımlamak için kullanılmıştır. SATıR-1 eklemeler yanı sıra, bu yöntem de diğer genomik sapmaları algılamak için kullanılabilir, DNA yeniden düzenlemeleri gibi, burada yeniden düzenleme eğilimli bölge ile ilgili bilgiler önceden var26.
Açıklamalar
Yazarların ifşa etmesi gereken hiçbir şey yok.
Teşekkürler
Biz bu yöntem ilk14, özellikle Tatiana Cajuso, Kimmo Palin, Outi Kilpivaara ve ESA Pitkänen değerli tartışmalar için yöntem geliştirirken açıklanan makalede tüm ortak yazarlar teşekkür etmek istiyorum. L.K. Finlandiya Akademisi (Grant numaraları 25996, 292789, 306026 ve 314394), Sigrid Juselius Vakfı ve Finlandiyalı Kanser Derneği tarafından finanse edilmektedir. Tansiyon, Helsinki Üniversitesi Araştırma Vakfı doktora öğrencisi, Finlandiyalı Kanser Derneği tez hibe ve Ida Montinin Säätiö 'den doktora araştırması hibe sahibidir. Ayrıca L.K. 'in araştırma grubundan Teemu masalin 'e (Helsinki Üniversitesi) ve kul Shrestha 'ya video üretimiyle bize yardımcı olmak için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
Referanslar
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16(2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343(2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. Jr High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043(2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521(2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Jr Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484(2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır