Method Article
Detecção de atividade de retrotransposição de Hot LINE-1s por PCR inverso de longa distância
Neste Artigo
Resumo
Este artigo esboça um ensaio PCR-baseado simples para monitorar a atividade de um Retrotransposão linha-1 ativo e para mapear retrotransposições de de novo em um genoma dado. Usando a linha celular MCF7, demonstramos aqui como esse método pode ser aplicado para detectar a atividade de uma linha-1 localizada em 22q 12.1.
Resumo
Os elementos nucleares intercaladas longos 1 (linha-1s) são a única família de elementos genéticos móveis no genoma humano que pode se mover autonomamente. Eles fazem isso por um processo chamado retrotransposição em que transcripta para formar um intermediário mRNA que é então, consequentemente, inserido no genoma por transcrição reversa. Apesar de ser silencioso em células normais, a linha-1s é altamente ativa em diferentes tumores epiteliais. De novo As inserções LINE-1 podem potencialmente conduzir a tumorigênese e, portanto, é importante estudar sistematicamente a retrotransposição da linha 1 no câncer. Fora de ~ 150 retrotransposição-competente linha-1s presente no genoma humano, apenas um punhado de linha-1 loci, também referido como "quente" linha-1s, conta para a maioria de de novo line-1 inserção em diferentes tipos de câncer. Nós desenvolvemos um método simples da reacção em cadeia do polymerase (PCR)-based para monitorar a atividade do retrotransposição destes linha-1s quente. Esse método, baseado no inverso de longa distância (LDI)-PCR, aproveita a transdução de 3 ́, mecanismo pelo qual uma linha-1 mobiliza sua região não repetitiva flanqueada, que pode ser usada posteriormente para identificar eventos de transdução de linha-1 3 ́ de novo proveniente de uma linha quente particular-1.
Introdução
Os elementos nucleares intercaladas longos (linha-1s) são uma família de elementos genéticos móveis chamados retrotransposons que podem independentemente mover-se de um lugar a outro através de um mecanismo da copiar-e-pasta chamado retrotransposição. Durante o tempo evolutivo, o genoma humano acumulou mais de 500.000 cópias de repetições delinha 1. No entanto, a maioria das cópias da linha 1 presentes no genoma são mutadas e, portanto, não podem se mover via retrotransposição; somente ~ 150 cópias têm a cópia intata da seqüência do ADN necessária para que movam2. Em células somáticas normais, a mobilidade destes linha-1s é restrita por diferentes fatores hospedeiros3. Estas limitações são aliviadas em tumores epithelial diferentes, fazendo com que a linha-1s seja com e tendo por resultado muitas inserções de de novo no genoma4do tumor. Algumas destas inserções tumor-associadas de de novo foram mostradas para causar a mutagenese do insertional nos genes, daqui conduzindo a progressão do tumor5,6. Portanto, é importante ser capaz de mapear novas inserções no genoma tumoral.
Métodos existentes para detectar o uso da linha-1 das inserções de de novo (1) aproximação de sequenciamento do genoma inteiro4,7,8, onde os algoritmos computacionais diferentes são usados para encontrar a linha-1 inserções de de novo de dados WGS, ou (2) seqüenciamento de próxima geração que tem como alvo o final 3 ́ de jovens, potencialmente ativos linha-1s9,10,11,12,13. No entanto, encontrar novas inserções entre vários milhares de cópias Near-idênticos com esses métodos está longe de ser trivial, e o desafio é ainda mais agravado pela heterogeneidade tumoral e alterações genómicas associadas à inserção da linha 14.
Estudos que utilizam esses métodos existentes mostraram que apenas alguns linha-1s conta para a maioria das inserções de linha-1 de de novo observadas nos tumores7,8. Conseqüentemente, para responder se ou não uma amostra particular do tumor indica a atividade da linha-1, é suficiente mapear eventos do retrotransposição causados por este punhado de loci linha-1 altamente ativo. Neste artigo, nós descrevemos um método simples da reacção em cadeia do polymerase (PCR)-baseado14 que possa ser usado para monitorar a atividade de um locus específico da linha-1 no primeiro intron do gene TTC28 em 22q 12.1 que é altamente-ativo no cancro colorretal 7 anos de , 8. este Locus line-1 será referido como TTC28-line-1 em todo o artigo. Este ensaio identifica especificamente os eventos de retrotransposição de linha-1 de novo que mobilizam a sequência não repetitiva na região de flanquear 3 ́ da linha-1 de origem por um mecanismo denominado transdução de 3 ́15. a transdução 3 ́ ocorre devido ao fraco sinal de poliadenilação LINE-1 (PAS) que faz com que a maquinaria transcricional o ignore e, em vez disso, termine a transcrição no PAS mais forte a jusante, capturando assim a sequência não repetitiva ( doravante referida como a "marca exclusiva") que é então inserida no local de destino ao lado da seqüência de linha 1. Philippe et al.16 mostraram recentemente que diferentes tipos de células podem expressar diferentes Locos line-1. À luz deste achado, este método pode ser aplicado para monitorar a atividade do mais altamente expressado linha-1 que mobilizam sua Tag original no tipo de câncer de interesse.
O primeiro passo na LDI-PCR é a digestão do DNA genómico com uma enzima de restrição que gera um fragmento de restrição contendo a linha-1 sendo ensaiada (aqui, TTC28-line-1) e sua tag exclusiva (Figura 1). O DNA digerido é então circularizado por autoligadura e PCR amplificado usando primers inversos localizados dentro da tag exclusiva. Ao fazer isso, a fonte de comprimento total LINE-1 em seu local "nativo" é sempre amplificada e ao lado dela, as inserções LINE-1 de descendentes em diferentes loci-alvo contendo a marca exclusiva também serão amplificadas (Figura 1), relatando assim a atividade de retrotransposição da linha-1 em questão.
Protocolo
Esta pesquisa foi aprovada pelo Comitê de ética e revisão institucional do hospital universitário de Helsinque. O consentimento informado assinado foi obtido a partir do assunto para a amostra de sangue utilizada para demonstrar este protocolo.
1. projetando primers inversos e selecionando enzimas de restrição (bioinformática)
- Determinando uma marca exclusiva associada à linha-1
- Transfira a seqüência TTC28-line-1 no formato de FASTA de uma base de dados line-1 tal como L1Base17. O ID L1base para TTC28-line-1 é 135.
- Inclua a sequência de 5 KB flanqueando as extremidades 5 ́ e 3 ́ da sequência LINE-1 e anote-a em um processador de texto.
Observação: aqui a seqüência de flanquear linha 1 é anotada na fonte marrom, e linha-1 seqüência está na fonte cinza (arquivo suplementar). - Insira a sequência de 1 KB a jusante do PAS cognato da LINE-1selecionada em uma ferramenta de predição do PAS, como o polyadq18 ou o spotter19 do Dragon Polya, e anote todo o sinal de poliadenilação nesta janela de 1 KB.
Observação: se não houver nenhum PAS na janela de 1 KB, procure PAS na próxima janela de 1 KB downstream. TTC28-line-1 do próprio Pas fraco é destacado em rosa e todos os outros pas em 1 KB janela downstream é realçado em vermelho (arquivo suplementar). - Anote a sequência entre o fim da linha-1 ' s cognato Pas e o mais forte Pas downstream como "Unique tag".
Observação: a "marca exclusiva" de TTC28-line-1 é destacada em amarelo (arquivo suplementar).
- Projetando primers inversos
- Projete primers inversos do PCR introduzindo a seqüência do "tag original" em uma ferramenta Web-based da primeira demão-projetando como a primeira demão 3 (http://bioinfo.ut.ee/primer3-0.4.0/) ou o primer-BLAST de NCBI (https://www.ncbi.nlm.nih.gov/tools/primer-blast/). Desde que os pares da primeira demão projetados por estas ferramentas enfrentam-se que facilitam o PCR convencional, usam a função do reverso-complemento para que o par da primeira demão realize o PCR inverso.
Nota: uma vez que as transsecções LINE-1 são fortemente truncadas ao final de 5 ́ e o tamanho da região transdeada é altamente variável, visam manter a distância entre os dois primers inversos mínimos, definindo o parâmetro "comprimento do produto PCR" no NCBI primer-BLAST para o Mínimo. No caso de vários PAS dentro da tag exclusiva, projetar vários pares de primer que correspondem a diferentes PASs. Projete os primers próximos ao PAS, pois as inserções de linha 1 iniciam a partir do 3 ́fim do intermediário de RNA e a extremidade 5 ́ é truncada variavelmente. Aqui foram projetados três pares de primers, destacados em verde-azulado e verdes, correspondendo a três fortes sinais de poliadenilação na tag exclusiva de TTC28 line-1 (arquivo suplementar).
- Projete primers inversos do PCR introduzindo a seqüência do "tag original" em uma ferramenta Web-based da primeira demão-projetando como a primeira demão 3 (http://bioinfo.ut.ee/primer3-0.4.0/) ou o primer-BLAST de NCBI (https://www.ncbi.nlm.nih.gov/tools/primer-blast/). Desde que os pares da primeira demão projetados por estas ferramentas enfrentam-se que facilitam o PCR convencional, usam a função do reverso-complemento para que o par da primeira demão realize o PCR inverso.
- Seleção de enzimas de restrição
- Digerir a sequência LINE-1 juntamente com seus flancos upstream e downstream de 5 KB em silico usando a ferramenta baseada na Web, como RestrictionMapper. Isso dará uma lista abrangente de enzimas de restrição que digere esta região, gerando diferentes fragmentos de restrição.
- Selecione as enzimas de restrição que cortam o locus nativo da linha-1 da seguinte forma: na extremidade 5 ́, seja a montante da linha-1 ́s 5 ́end ou Far 5 ́end da linha-1 em si, e no final 3 ́, a jusante da marca exclusiva da LINE-1.
Nota: a enzima de restrição selecionada deve ser insensível à metilação do DNA, deve ser calor-inativável, e deve gerar escalonada "Sticky" extremidades que são complementares entre si. A fim demonstrar LDI-PCR, a enzima da limitação do SACmim que corta o ADN em locais de gagctc, destacado aqui no verde claro, é usado (arquivo suplementar). - Anote o tamanho do fragmento de restrição feito por enzimas de restrição selecionadas. Isto não deve ser mais longo do que 12 KB porque não pôde ser amplificado eficientemente pelo PCR.
2. fazendo modelos circulares do ADN para o PCR inverso interurbano
- Extração de DNA e avaliação da qualidade
- Extraia o ADN genomic das amostras (tumor ou sangue) usando os jogos comercialmente disponíveis da extração do ADN que podem extrair a boa qualidade, ADN do peso molecular elevado necessário para LDI-PCR de acordo com as instruções do fabricante. Alternativamente, o DNA de alto peso molecular também pode ser extraído por fenol: clorofórmio20.
- Meça a concentração do ADN usando um fluorómetro de acordo com as instruções do fabricante, e funcione 100 ng do ADN no gel do agarose de 1% (w/v) que contem o brometo do etídio (0,5 μg/ml) no tampão do Tris-Acetato-EDTA 1x (Tae) em 4,5 v/cm21 ao lado do λ-HindIII Marcadores do peso molecular do ADN para verific a qualidade e a quantidade do ADN.
- Digesting DNA genómico
- Faça uma mistura de reação de digestão (volume final de 50 μL) adicionando 20 unidades de enzima de restrição SACI, 5 μL de tampão de reação 10x (tabela de materiais), 100 ng de DNA (até 44 μL) em um tubo de PCR de 0,2 ml no gelo (1 μL de enzima de restrição da maioria dos fabricantes I s suficiente para digerir completamente 100 ng de DNA genómico). Misture a solução sacudendo o tubo e centrifugue brevemente.
- Utilize um termociclador para incubar a mistura de reacção a 37 ° c durante 1 h, seguido de inactivação do calor a 65 ° c durante 5 min.
- Auto-Ligating o DNA genomic digerido
- Para a mistura de digestão de 50 μL (após o passo 2.2.2), adicione 8 μL de tampão de ligase de ADN de 10x T4, 1 μL (5 unidades) de ligase de ADN T4 e 21 μL de água ultrapura para fazer um volume de reacção final de 80 μL. Misture a solução sacudendo os tubos e centrifugar brevemente.
- Incubar em um Thermal cycler em 22 ° c por 10 minutos, terminando com uma etapa da inactivação do calor em 65 ° c por 10 minutos.
3. PCR inverso de longa distância
- Determinando a temperatura do recozimento do primer pelo gradiente PCR
- Defina um gradiente de temperatura de recozimento de (A-4) ° c, (A-2) ° c, A, (A + 2) ° c, (A + 4) ° c onde a é a temperatura de recozimento teórica do par de primer calculado usando a ferramenta baseada na Web do fornecedor comercial.
Nota: os cicladores térmicos de alguns fabricantes podem não permitir a definição manual do gradiente de temperatura. Nesse caso, a configuração automática de gradiente pode ser usada com uma faixa de temperatura de (a-4) ° c a (A + 4) ° c. - Prepare uma mistura mestra para o PCR combinando e misturando os seguintes componentes em um tubo do microcentrifugador de 1,5 mL: 4 μL do amortecedor da reação 5x, 0,4 μL do dNTP de 10 milímetros, 5 μL do primer do PCR de 2 μM (para diante e reverso, projetados na etapa 1.3.1) , 0,2 μL (0,1 U) de DNA polimerase por reação. Configure uma reação para cada temperatura de recozimento no gradiente.
- Para cada reação, alíquota 19 μl da mistura mestra em 0,2 ml de tubos de PCR e adicione 1 μl (1,25 ng) de modelo de DNA circular feito na seção 2.
Nota: Use o DNA autoligated circular gerado do ADN normal do sangue como o molde para evitar consumir o ADN potencial precioso do tumor para esta etapa da optimização. - Execute o programa do PCR do Gradient em um Thermal cycler como descrito abaixo: (i) um ciclo de 3 minutos em 98 ° c (denaturation); (II) 35 ciclos de (10 s a 98 ° c [desnaturação], 20 s no gradiente de temperatura de [a-4] a [a + 4] ° c [recozimento] e 1 − 6 min [30 s por kilobase do produto de PCR esperado] a 72 ° c [extensão]); III) um ciclo de 10 min a 72 ° c (extensão final).
- Execute 6 μL do produto do PCR no gel21 do agarose de 1% preparado no amortecedor de 1x TAE em 4,5 V/cm e analise os produtos do PCR rendido em temperaturas diferentes do recozimento.
- Selecione a temperatura de recozimento que produz um produto de PCR que corresponda ao tamanho esperado.
- Defina um gradiente de temperatura de recozimento de (A-4) ° c, (A-2) ° c, A, (A + 2) ° c, (A + 4) ° c onde a é a temperatura de recozimento teórica do par de primer calculado usando a ferramenta baseada na Web do fornecedor comercial.
- Detectando a atividade de retrotransposição line-1 de novo no genoma tumoral
- Realize LDI-PCR usando o par de primers de PCR inverso em modelos de DNA circulares gerados a partir de amostras tumorais (seção 2). Siga as mesmas instruções que para o gradiente de PCR (seção 3,1), mas desta vez substituindo o gradiente de temperatura com a temperatura de recozimento ideal.
- Analise os produtos do PCR pela electroforese do gel do agarose como feito na etapa 3.1.5. O produto do PCR do tamanho conhecido ou do produto "nativo" do PCR que corresponde à linha-1 em seu locus nativo deve ser visível para cada reação. De novo A transdução LINE-1 3 ́ na amostra de tumor ensaiada é detectável como produtos do PCR de tamanhos diferentes, junto com o produto nativo do PCR no gel do agarose.
4. sequenciamento de produtos LDI-PCR para revelar a identidade dos sítios-alvo para a transdução LINE-1 3 ́
- Execute o sequenciamento de leitura longa de molécula única de todos os amplicões de PCR gerados em cada reação de LDI-PCR para identificar os locais de integração alvo desses eventos de transdução line-1 3 ́.
Nota: a clonagem e o sequenciamento de Sanger dos produtos LDI-PCR também são uma abordagem possível, embora complicada. - Alinhe as leituras produzidas por plataformas de sequenciamento de leitura longa de molécula única ao genoma de referência usando pipelines de alinhamento de sequência padrão. Analise as leituras alinhadas usando o software14 de LDI-PCR para identificar inserções de de novo line-1 e seus locais do alvo.
Resultados
Em caso de TTC28-line-1, há mais de um pas dentro de uma janela de 1 KB a jusante de seu pas cognato, daqui a região entre o Pas TTC28-line-1 e o Pas o mais forte em 811 BP a jusante foi considerado como a etiqueta original para TTC28-line-1. Três pares inversos da primeira demão do PCR foram projetados neste Tag original que correspondem à passagem diferente atual14. Selecionamos três enzimas de restrição: (i) NSIi que corta 5 ́ a montante de TTC28-line-1 e 3 ́ fora da tag única, e (II) SACi e (III) PSTi que Cut 5 ́ dentro da linha-1 em seu final de 5 ́, e 3 ́ fora da marca única. Estes geram fragmentos de restrição de 10.288 BP, 5.699 BP e 6.305 BP, respectivamente.
A fim demonstrar este método, nós executamos LDI-PCR no ADN extraído da linha celular MCF7. Esta linha celular do cancro da mama foi relatada previamente para indicar a atividade TTC28-line-116. Para simplificar, fizemos um modelo de DNA circular usando uma enzima de restrição, SACI, de três e realizei um LDI-PCR usando um par de primer de três (tabela 1) para detectar inserções de de novo line-1, decorrentes de TTC28- LINHA 1.
A boa qualidade do DNA extraído da linha celular MCF7 foi assegurada pela eletroforese em gel de agarose (Figura 2). O DNA de alto peso molecular intacto mostra que o DNA genómico é de ótima qualidade para este ensaio. Se um esfregaço é visível em vez disso, isso indica má qualidade do DNA extraído, que por sua vez vai dificultar os procedimentos a jusante.
A Figura 3 mostra um resultado representativo de um experimento de PCR com gradiente, visando determinar a temperatura ideal de recozimento do par de primer inverso TTC28-line-1. O ADN genomic do sangue digerido com o sacomim, seguido pelo Self-Ligation para dar forma a um molde circular do ADN, foi usado para esta reação. Um produto altamente específico do PCR do tamanho esperado (5.649 BP) em 62, em 64 e em 66 ° c mostra que a temperatura a melhor do recozimento para este par da primeira demão encontra-se dentro da escala de 62 − 66 ° c.
Nós geramos um molde circular do ADN digerindo o ADN genomic MCF7 com a enzima da limitação do SACmim seguida pelo Self-ligation. A Figura 4 mostra que a transdução TTC28-line-1 3 ́ ocorre em linhas celulares MCF7: inserções de novo podem ser detectadas como produtos LDI-PCR de tamanhos variados, juntamente com um produto de pcr nativo de tamanho conhecido (5.649 BP). Para identificar as coordenadas genómicas dos sítios de destino de novo , os amplicões de PCR podem ser sequenciados (ver protocolo secção 4).
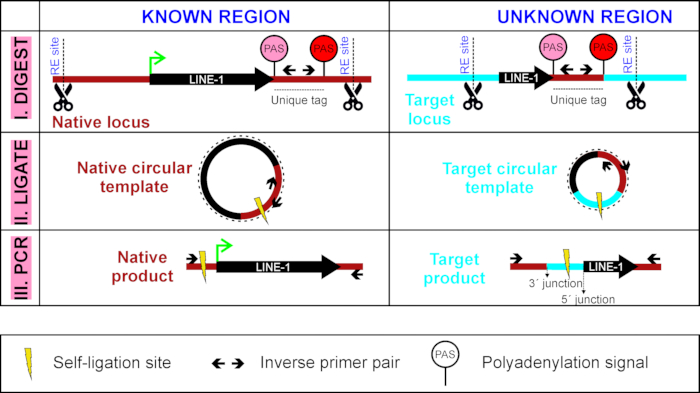
Figura 1 : Visão geral da LDI-PCR para detecção da transdução line-1 3 ́. Um molde circular do ADN é gerado primeiramente digerindo (I) ele com uma enzima da limitação e self-Ligating (II) ele. Esta etapa é seguida pelo inverso PCR (III) com os primers inversos do PCR alvejados ao Tag original da linha-1 do interesse (seqüência entre o PAS mais fraco da linha-1a, em cor-de-rosa, e mais forte PAS downstream, no vermelho). Por favor clique aqui para ver uma versão maior desta figura.
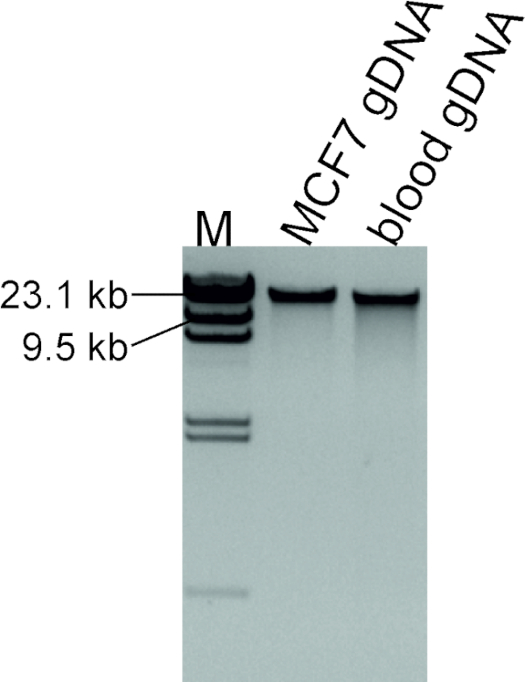
Figura 2 : Avaliação da qualidade do DNA extraído. 100 ng de DNA extraído da linha celular MCF7 e sangue de um indivíduo normal, que será usado como uma amostra de controle, foram executados ao lado de 1 μL e 2 μL de λ DNA/HindIII marcador rotulado como M. por favor clique aqui para ver uma versão maior desta figura.
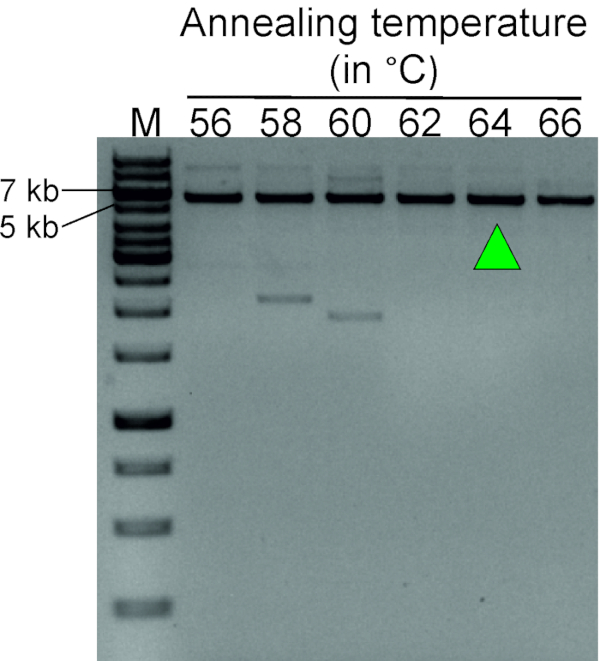
Figura 3 : PCR do Gradient para determinar a temperatura óptima do recozimento para primers inversos do PCR (tabela 1). LDI-PCR usando pares inversos da primeira demão na temperatura de recozimento que varia de 56 a 66 ° c mostra um produto distinto do PCR em 62 − 66 ° c. A seta verde indica a temperatura de recozimento selecionada para experimentos futuros. O molde circular do ADN gerado digerindo o ADN genomic do sangue de um indivíduo normal com o SACeu segui pelo Self-Ligation foi usado para esta etapa da optimização. M, marcador (1 KB mais a escada do ADN). Por favor clique aqui para ver uma versão maior desta figura.
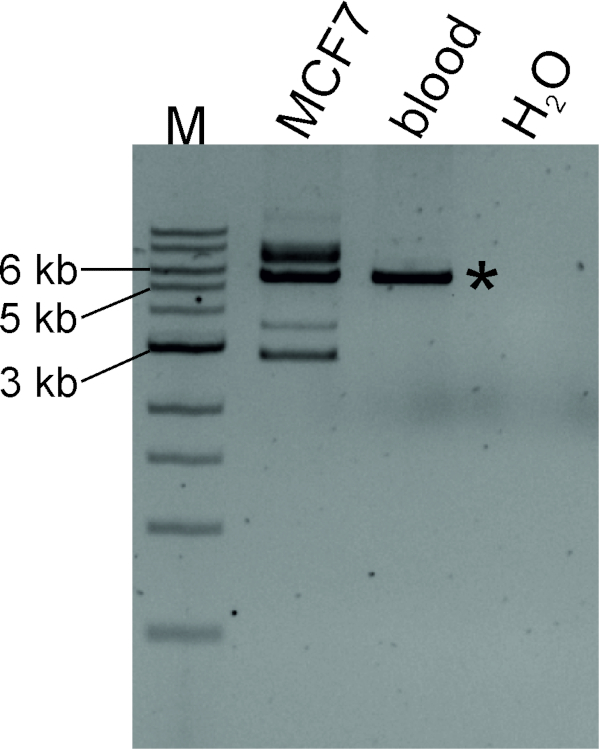
Figura 4 : LDI-PCR para identificar a transdução line-1 3 ́ decorrente TTC28 -Linha 1. Os moldes circulares do ADN gerados digerindo MCF7 e o ADN genomic do sangue (do indivíduo normal) com o SACque eu segui pela auto-ligadura foram amplificados por iniciadores inversos na temperatura óptima do recozimento. O produto do PCR do "nativo", marcado com o asterisco, do tamanho esperado (5.649 BP) foi detectado no ADN MCF7 e no ADN normal do sangue, quando MCF7 igualmente produziu produtos adicionais do PCR de tamanhos de variação, indicando o retrotransposição da linha-1 de de novo . M, marcador (1 KB de DNA Ladder). Por favor clique aqui para ver uma versão maior desta figura.
| Nome da cartilha | Sequência (5 ́ → 3 ́) |
| L1_001 (Rev) | TTCACTAAGCATGTATGTGGAAAAC |
| L1_002 (FWD) | CCCAAAATATACCCAATTACTGGCA |
Tabela 1: par de primer de PCR inverso projetado para a marca exclusiva de TTC28 -Line-1 14 anos de .
Arquivo suplementar. Por favor, clique aqui para baixar este arquivo.
Discussão
Aqui nós descrevemos um método que possa ser usado para identificar as inserções da linha-1 de de novo que resultam de toda a linha-1 ativa de interesse. Otimizamos este método para uma linha-1 altamente ativa, localizada em 22q 12.1, e previamente demonstrou ser altamente sensível na detecção de inserções subclonais no câncer colorretal14.
O sucesso da LDI-PCR depende da qualidade do DNA genómico. Conseqüentemente, nós incluímos uma etapa adicional do controle da qualidade para assegurar-se de que no começo do protocolo, o ADN high-molecular do peso esteja atual (etapa 2.1.2). Nós recomendamos armazenar o ADN genomic em-20 ° c para o armazenamento a longo prazo, e para preparar alíquotas a fim evitar ciclos de congelação e de thawing. Usar o ADN genomic do sangue ou do tecido normal paciente-combinado é altamente-recomendado distinguir se a linha-1 retrotransposição detectada é um germline ou um evento somático. Desde que os locais cortados para enzimas da limitação são estocásticos no genoma, é possível que um local particular da inserção line-1 de de novo não possa abrigar nenhuns locais cortados para a enzima da limitação que está sendo usada em sua vizinhança. Daqui para aumentar a probabilidade de detectar a maioria de inserções da linha-1 de de novo no ADN do tumor, mais de uma enzima da limitação deve ser usada em reações separadas para gerar bibliotecas diferentes do molde circular do ADN. Além disso, se a tag exclusiva da linha-1 de interesse tem mais de um PAS, em seguida, usando primer pares adjacentes a cada PAS melhora as chances de detectar transacionações fortemente truncados.
Embora os métodos elegantes para a deteção genoma-larga de inserções da linha-1 de de novo existam, podem ser esmagadores se o alvo é sondar a competência do retrotransposição de uma linha-1 particular em um contexto celular específico. Para esta finalidade, LDI-PCR pode ser uma aproximação barata e simples contudo robusta para visualizar a linha-1 eventos do retrotransposição. A abordagem de segmentação utilizada neste método é semelhante ao TS-ATLAS22; no entanto, a LDI-PCR evita o uso de oligonucleotídeos do vinculador e pode amplificar as junções 5 ́ e 3 ́ da inserção de de novo line-1 simultaneamente. As informações relativas às junções 5 ́ e 3 ́ da inserção LINE-1, ao local de integração, à cauda de polyA e às modificações no local de destino, todas as quais são marcas de linha-1 retrotransposição, podem ser obtidas através do acoplamento LDI-PCR com uma molécula única tecnologias de sequenciamento de leitura longa. Leituras longas assim geradas contêm a seqüência de linha-1 inserida, sua marca exclusiva e as sequências de destino em uma única leitura, contornando dificuldades de mapeamento de leituras curtas na região repetitiva.
Há duas limitações principais a usar o LDI-PCR para a deteção da atividade LINE-1. O primeiro é inerente ao PCR: pode somente confiantemente amplificar fragmentos até 10 KB no tamanho. Isto deve ser considerado ao selecionar enzima (s) da restrição, porque o fragmento nativo não deve exceder este limite. Em segundo lugar, este método só pode detectar eventos de retrotransposição que mobilizam a região de flanqueamento de 3 ́ da linha 1 por três ́ transsecções. Portanto, a atividade desses linha-1s que não exibem a transdução 3 ́ não será detectada usando esse método. Adicionalmente, apesar de ser amplificado por LDI-PCR, alguns eventos de retrotransposição LINE-1 que (a) geram um alvo do PCR do tamanho similar como a posição "nativa" ou outras retrotransposições ou (b) são raros ou subclonal, não podem ser detectados pelo gel do agarose Electroforese. Esses eventos de retrotransposição de linha 1 podem ser capturados por sequenciamento do produto LDI-PCR usando tecnologias de sequenciamento de leitura longa de molécula única14.
O fluxo de trabalho descrito aqui pode ser facilmente modificado para detectar a atividade de outros "Hot" LINE-1s usando uma enzima de restrição adequada e projetando iniciadores inversos direcionados a esses linha-1s. Além da detecção da transdução de 3 ́ mediada pela linha-1, esse método pode ser adaptado para detectar Duções de 5 ́ mediadas por linha 1 menos freqüentes23. O método similar foi usado para identificar o local da integração de repórteres da linha-1 em ensaios Cell-Based24 e em locais da integração de proviral no cancro25. Além das inserções da linha 1, esse método também pode ser utilizado para detectar outras aberrações genómicas, como rearranjos de DNA, onde a informação sobre a região propensa a rearranjo pré-existente26.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Gostaríamos de agradecer a todos os nossos coautores no artigo onde este método foi descrito pela primeira vez14, especialmente Tatiana Cajuso, Kimmo Palin, Outi Kilpivaara e ESA Pitkänen para discussões valiosas ao desenvolver o método. L.K. é financiado pela Academia da Finlândia (números de subvenção 25996, 292789, 306026 e 314394), a Fundação Sigrid Juselius, e da sociedade finlandesa de câncer. B.P. é beneficiária da Universidade de Helsinki Research Foundation PhD Studentship, uma bolsa de estudos da sociedade finlandesa do câncer, e uma bolsa de doutorado de ida Montinin Säätiö. Agradecemos também Teemu Masalin (Universidade de Helsínquia) e Kul Shrestha do grupo de pesquisa da L.K. para nos ajudar com a produção de vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
Referências
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16 (2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343 (2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043 (2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521 (2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484 (2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados