Method Article
Обнаружение ретротранспозиционной активности горячих линий-1 с помощью дальнего обратного ПЦР
В этой статье
Резюме
В этой статье излагается простой анализ на основе ПЦР для мониторинга активности активного ретротранспоза LINE-1 и для картирования ретротранспозиций de novo в данном геноме. Используя линию клеток MCF7, мы демонстрируем, как этот метод может быть применен для обнаружения активности LINE-1, расположенной на 22q12.1.
Аннотация
Длинные перемежаются ядерные элементы 1 (LINE-1s) являются единственным семейством мобильных генетических элементов в геноме человека, которые могут двигаться автономно. Они делают это путем процесса, называемого ретротранспозицией, в которой они транскрибируют для формирования промежуточных мРНК, который затем, следовательно, вставляется в геном путем обратной транскрипции. Несмотря на молчание в нормальных клетках, LINE-1s очень активны в различных эпителиальных опухолях. Де Ново Line-1 вставки потенциально может привести к опухолевому и, следовательно, важно систематически изучать LINE-1 ретротранспозиции при раке. Из 150 ретротранспозиционно-компетентных LINE-1, присутствующих в геноме человека, только горстка локусов LINE-1, также именуемых «горячими» ЛИНИЯ-1, приходится большая часть вставки de novo LINE-1 в различных типах рака. Мы разработали простой метод полимеразной цепной реакции (ПЦР) для мониторинга ретротранспозиционной активности этих горячих ЛИНИ-1. Этот метод, основанный на междугородной обратной (LDI)-PCR, использует 3 трансдукции, механизм, с помощью которого LINE-1 мобилизует его фланговый неповторяющийся регион, который впоследствии может быть использован для выявления de novo LINE-1 3 - трансдукционных событий вытекающих из конкретной горячей LINE-1.
Введение
Длинные перемежаются ядерных элементов (LINE-1s) являются семействомобильных генетических элементов, называемых ретротранспозоны, которые могут самостоятельно перемещаться из одного места в другое через копировать и вставить механизм называется ретротранспозиции. За эволюционное время геном человека накопил более 500 000 копийLINE-1 повторов 1. Тем не менее, большинство копий LINE-1, присутствующих в геноме, мутируют и, следовательно, не могут двигаться через ретротранспозицию; только 150 копий имеют нетронутую копию последовательности ДНК, необходимой для их перемещения2. В нормальных соматических клетках подвижность этих ЛИНИ-1ограничена различными принимающими факторами 3. Эти ограничения снимаются при различных эпителиальных опухолях, в результате чего LINE-1s будет подавлен и в результате многих de novo вставки в геноме опухоли4. Некоторые из этих опухолевых де Ново вставки были показаны, чтобы вызвать вставку мутагенеза в генах, следовательно, движущей прогрессии опухоли5,6. Поэтому важно иметь возможность составлять карты новых вставок в геном опухоли.
Существующие методы обнаружения вставок de novo LINE-1 используют (1) подход секвенирования всего генома4,7,8,где для поиска вставок de novo LINE-1 используются различные вычислительные алгоритмы из данных WGS, или (2) последовательности следующего поколения, которая нацелена на 3- конец молодых, потенциально активных LINE-1s 9,10,11,12,13. Тем не менее, найти новые вставки среди нескольких тысяч почти идентичных копий с этими методами далеко не тривиально, и проблема еще больше усугубляется неоднородности опухоли и геномных изменений, связанных с LINE-1 вставки4.
Исследования с использованием этих существующих методов показали, что лишь несколько LINE-1s составляют большинство de novo LINE-1 вставки наблюдается в опухолях7,8. Поэтому, чтобы ответить, отображает ли конкретный образец опухоли активность LINE-1, достаточно сопоставить события ретротранспозиции, вызванные этой горсткой высокоактивных локусов LINE-1. В этой статье мы описываем простой полимеразы цепной реакции (PCR) на основе метода14, которые могут быть использованы для мониторинга активности конкретного локуса LINE-1 в первом интрон гена TTC28 на 22q12.1, который очень активен в колоректального рака 7 (г. , 8. Этот локус LINE-1 будет называться TTC28-LINE-1на протяжении всей статьи. Этот ассс конкретно определяет de novo LINE-1 ретротранспозиции событий, которые мобилизуют неповторяющихся последовательности на 3 »фланговой области источника LINE-1 механизмом под названием 3 "трансдукции15. 3 трансдукция происходит из-за слабого сигнала полиаденилаации LINE-1 (PAS), который вызывает транскрипционного механизма, чтобы пропустить его и вместо прекращения транскрипции на сильнее PAS вниз по течению, таким образом захватывая фланговые неповторяющиеся последовательности ( отныне упоминается как "уникальный тег"), который затем вставляется в целевое место наряду с последовательностью LINE-1. Филипп и др.16 недавно показали, что различные типы клеток могут выражать различные локусы LINE-1. В свете этого вывода, этот метод может быть применен для мониторинга деятельности наиболее высоко выраженных LINE-1, которые мобилизуют их уникальный тег в рак типа интереса.
Первым шагом в LDI-PCR является переваривание геномной ДНК с ферментом ограничения, который генерирует фрагмент ограничения, содержащий анализ LINE-1 (здесь, TTC28-LINE-1) и его уникальный тег(рисунок 1). Переваренный ДНК затем циркуляризированы самолигии и ПЦР усиливается с помощью обратных грунтовки, расположенные в рамках уникального тега. Поступая таким образом, полнометражный источник LINE-1 в своем "родном" месте всегда усиливается и рядом с ним, потомство LINE-1 вставки на различных целевых локусов, содержащих уникальный тег также будет усилена (Рисунок 1), таким образом отчетности ретротранспозиционная деятельность LINE-1, о котором идет речь.
протокол
Это исследование было одобрено Советом по институциональному обзору и комитетом по этике больницы Хельсинкского университета. Подписанное информированное согласие было получено от субъекта для анализа крови, используемого для демонстрации этого протокола.
1. Проектирование обратных грунтовок и выбор ферментов ограничения (биоинформатика)
- Определение уникального тега LINE-1, связанного с ним
- Скачать последовательность TTC28-LINE-1в формате FASTA из базы данных LINE-1, такой как L1Base17. L1base ID для TTC28-LINE-1 составляет 135.
- Включите последовательность 5 кб, обрамленная как 5, так и 3 концами последовательности LINE-1, и аннотировать ее в текстовом процессоре.
ПРИМЕЧАНИЕ: Здесь line-1 фланговая последовательность аннотируется коричневым шрифтом, и LINE-1 последовательность в серый шрифт (Дополнительный файл). - Введите 1 кб последовательность вниз по течению выбранного LINE-1 в cognate PAS в PAS инструмент прогнозирования, таких как polyadq18 или Дракон PolyA корректировщик19 и аннотировать все полиамунилирования сигнала в этом 1 кб окно.
ПРИМЕЧАНИЕ: Если нет PAS в окне 1 кб, ищите PAS в следующем окне 1 кб вниз по течению. TTC28-LINE-1 собственные слабые PAS выделенв розовый и все другие PAS в 1 кб окно вниз по течению выделенкрасным красным цветом (Дополнительный файл). - Аннотировать последовательность между концом коньяка LINE-1 PAS и сильнейшим PAS вниз по течению как "уникальный тег".
ПРИМЕЧАНИЕ: "Уникальный тег" TTC28-LINE-1 выделен желтым цветом(дополнительный файл).
- Проектирование обратных грунтовок
- Дизайн обратных ПЦР праймеры, введя "уникальный тег" последовательность в веб-основе грунтового проектирования инструмент, как Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) или праймер NCBI-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/). Так как грунтовые пары, разработанные этими инструментами, сталкиваются друг с другом, облегчая обычные ПЦР, используйте функцию обратного дополнения для пары праймеров для выполнения обратного ПЦР.
ПРИМЕЧАНИЕ: Как LINE-1 трансуктокции сильно усечены на их 5 й конец и размер транскорпорированной области является весьма переменной, цель сохранить расстояние между двумя обратными грунтовки минимальным, установив "PCR длина продукта" параметр в NCBI праймер-BLAST к Минимальный. В случае нескольких PAS в уникальном теге, дизайн нескольких грунтовых пар, которые соответствуют различным PASs. Дизайн праймеров близко к PAS, как LINE-1 вставки инициировать из 3 "конец РНК промежуточных и 5 " конец вариативно truncated. Здесь были разработаны три грунтовки пары, выделенные в чирок и зеленый, соответствующий трем сильным сигналам полиаденилаации в уникальном теге TTC28 LINE-1 (Дополнительный файл).
- Дизайн обратных ПЦР праймеры, введя "уникальный тег" последовательность в веб-основе грунтового проектирования инструмент, как Primer 3 (http://bioinfo.ut.ee/primer3-0.4.0/) или праймер NCBI-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/). Так как грунтовые пары, разработанные этими инструментами, сталкиваются друг с другом, облегчая обычные ПЦР, используйте функцию обратного дополнения для пары праймеров для выполнения обратного ПЦР.
- Выбор ферментов ограничения
- Дайджест LINE-1 последовательности вместе с его 5 кб вверх по течению и вниз по течению фланги в силико с помощью веб-инструмента, такого как RestrictionMapper. Это даст полный список ферментов ограничения, который переваривает эту область, генерируя различные фрагменты ограничения.
- Выберите ферменты ограничения, которые разрезают родной локус LINE-1 следующим образом: в конце 5 и, либо вверх по течению LINE-1's 5 'end, либо далеко 5 «end» самой LINE-1, и в конце 3 ', вниз по течению уникального тега LINE-1.
ПРИМЕЧАНИЕ: Выбранный фермент ограничения должен быть нечувствительным к метилированию ДНК, должен быть тепло-неактивируемым, и должен генерировать шахматные "липкие" концы, которые дополняют друг друга. Для того, чтобы продемонстрировать LDI-PCR, SacI ограничение фермента, который режет ДНК на сайтах GAGCTC, выделенные здесь в светло-зеленый, используется(Дополнительный файл). - Обратите внимание на размер фрагмента ограничения, сделанный выбранными ферментами ограничения. Это не должно быть больше, чем 12 кб, как это не может быть эффективно усиливается ПЦР.
2. Создание циркулярных шаблонов ДНК для дальних обратных ПЦР
- Экстракция ДНК и оценка качества
- Извлекайте геномную ДНК из образцов (опухоли или крови) с использованием коммерчески доступных комплектов для извлечения ДНК, которые могут извлечь хорошее качество, дна высокого молекулярного веса, необходимое для LDI-PCR в соответствии с инструкциями производителя. Кроме того, дна высокого молекулярного веса также может быть извлечена с помощью фенола: хлороформ20.
- Измерьте концентрацию ДНК с помощью флюорометра в соответствии с инструкциями производителя, и запустите 100 нг ДНК на 1% (w/v) геля агароза, содержащего бромид этидия (0,5 мкг/мл) в буфере 1x Tris-acetate-EDTA (TAE) на уровне 4,5 В/см21 наряду с Хиндсий Молекулярные маркеры веса ДНК для проверки качества и количества ДНК.
- Переваривание геномной ДНК
- Сделать смесь реакции пищеварения (окончательный объем 50 qL) путем добавления 20 единиц SacI ограничение фермента, 5 Л 10x буфер реакции (Таблица материалов), 100 нг ДНК (до 44 л) в 0,2 мл ПЦР трубки на льду (1 л ограничения фермента от большинства производителей я s достаточно, чтобы полностью переварить 100 нг геномной ДНК). Смешайте раствор, щелкнув трубку, и центрифуга кратко.
- Используйте тепловой цикл, чтобы инкубировать реакционную смесь при температуре 37 градусов по Цельсию в течение 1 ч, а затем тепловой инактивации при температуре 65 градусов по Цельсию в течение 5 мин.
- Самолилигация переваренной геномной ДНК
- К смеси пищеварения 50 qL (после шага 2.2.2), добавьте 8 зл 10x буфера лигазы ДНК T4, 1 Зл (5 единиц) T4 ДНК лигазы и 21 л ультрачистой воды, чтобы сделать окончательный объем реакции 80 л. Смешайте раствор, щелкая трубками , и центрифуги кратко.
- Инкубировать в тепловом цикле при температуре 22 градусов по Цельсию в течение 10 мин, прекращая с тепловой инактивацией шаг на 65 градусов по Цельсию в течение 10 мин.
3. Дальние обратные ПЦР
- Определение температуры грунтовки по градиенту ПЦР
- Установите градиент температуры annealing (A-4) КС, (A-2)' C, A, (A 2)' C, (A'4) QC, где А является теоретической температурой аннулирования грунтовой пары, рассчитанной с помощью веб-инструмента коммерческого поставщика.
ПРИМЕЧАНИЕ: Тепловые циклизаторы от некоторых производителей могут не позволять устанавливать градиент температуры вручную. В этом случае автоматический параметр градиента можно использовать с температурным диапазоном (A-4) к C до (A-4) . - Подготовьте мастер-микс для ПЦР путем объединения и смешивания следующих компонентов в микроцентрифуговой трубке 1,5 мл: 4 л 5-x буфера реакции, 0,4 л 10 мМ dNTP, 5 л 2 ЦМ ПЦР грунтовка (вперед и обратном, разработанный в шаге 1.3.1) 0,2 л (0,1 У) полимеразы ДНК на одну реакцию. Навязаните одну реакцию для каждой annealing температуры в градиенте.
- Для каждой реакции, aliquot 19 qL мастер смесь в 0,2 мл ПЦР труб и добавить 1 КЛ (1,25 нг) кругового шаблона ДНК, сделанные в разделе 2.
ПРИМЕЧАНИЕ: Используйте круговую самолигированную ДНК, генерируемую из нормальной ДНК крови в качестве шаблона, чтобы избежать потребления потенциально ценной ДНК опухоли для этого шага оптимизации. - Запустите градиентную пЧР-программу на тепловом цикле, как описано ниже: (i) один цикл 3 мин при 98 градусах По кв/ (денатурация); ii) 35 циклов (10 с при 98 градусах по Цельсию, 20 с при температурном градиенте от «А-4» до «A»4» (Аннеалинг) и 1-6 мин на килобазу ожидаемого продукта ПЦР при 72 «C» (расширение); iii) один цикл по 10 мин при 72 градусах по Цельсию (окончательное расширение).
- Запустите 6 зЛ продукта ПЦР в 1% агарозный гель21, приготовленный в буфере 1x TAE при 4,5 В/см, и проанализируйте продукты ПЦР, произвданные при различных температурах annealing.
- Выберите температуру annealing, которая дает продукт ПЦР, соответствующий ожидаемому размеру.
- Установите градиент температуры annealing (A-4) КС, (A-2)' C, A, (A 2)' C, (A'4) QC, где А является теоретической температурой аннулирования грунтовой пары, рассчитанной с помощью веб-инструмента коммерческого поставщика.
- Обнаружение ретротранспозиционной активности de novo LINE-1 в геноме опухоли
- Выполните LDI-PCR с помощью обратной пары праймер ПЦР на круглых шаблонах ДНК, генерируемых из образцов опухоли (раздел 2). Следуйте тем же инструкциям, что и для градиента ПЦР (раздел 3.1), но на этот раз заменяя градиент температуры оптимальной температурой annealing.
- Проанализируйте продукты ПЦР с помощью электрофореза агарозного геля, как это делается в шаге 3.1.5. ПЦР-продукт известного размера или «родной» ПЦР продукт, соответствующий LINE-1 на его родном локусе, должен быть виден для каждой реакции. Де Ново LINE-1 3 - трансдукция в анализе образца опухоли обнаруживается как пЦР-продукты различных размеров, наряду с родным продуктом ПЦР в агарозном геле.
4. Секвенирование продуктов LDI-PCR для выявления идентичности целевых объектов для LINE-1 3 и трансдукции
- Выполняйте одномолекулярное длительное секвенирование всех амприконов ПЦР, генерируемых в каждой реакции LDI-PCR, для определения целевых интеграционных участков этих событий трансдукции LINE-1 3.
ПРИМЕЧАНИЕ: Клонирование и секвенирование сэнгера продуктов LDI-PCR также является возможным, хотя и громоздким, подходом. - Выровнять считывания, произведенные одномолекулярными долго считываемыми платформами секвенирования, с эталонным геномом с помощью стандартных трубопроводов выравнивания последовательности. Проанализируйте выровненные считывания с помощью программного обеспечения LDI-PCR14 для определения вставок de novo LINE-1 и целевых объектов.
Результаты
В случае TTC28-LINE-1, есть более чем один PAS в течение 1 кб окно вниз по течению его cognate PAS, следовательно, область между TTC28-LINE-1PAS и сильнейших PAS на 811 bp вниз по течению считался уникальным тегом для TTC28-LINE-1. Три обратные пары пЦР грунтовки были разработаны в этом уникальном теге, которые соответствуют различным PASs настоящее14. Мы выбрали три фермента ограничения: (i) NsiI, который режет 5 » вверх по течению TTC28-LINE-1 и 3 » за пределами уникального тега, и (ii) SacI и (iii) PstI, что сократить 5 » в пределах LINE-1 в его далеко 5 "конец, и 3" за пределами уникального тега. Они генерируют фрагменты ограничения 10,288 bp, 5,699 bp, и 6.305 bp соответственно.
Для того, чтобы продемонстрировать этот метод, мы выполнили LDI-PCR на ДНК, извлеченной из линии клеток MCF7. Эта линия клеток рака молочной железы ранее сообщалось, чтобы отобразить TTC28-LINE-1 деятельности16. Для простоты, мы сделали круговой шаблон ДНК с помощью одного фермента ограничения, SacI, из трех и выполняется LDI-PCR с помощью одной грунтовой пары из трех (Таблица 1) для обнаружения de novo LINE-1 вставки, вытекающие из TTC28- ЛИНИЯ-1.
Хорошее качество ДНК, извлеченной из линии клеток MCF7, было обеспечено электрофорезом агарозного геля(рисунок2). Intact высокий молекулярный вес ДНК показывает, что геномная ДНК имеет оптимальное качество для этого анализа. Если мазок виден вместо этого, это указывает на низкое качество извлеченной ДНК, что, в свою очередь, будет препятствовать вниз по течению процедур.
На рисунке 3 показан репрезентативный результат градиентного пЦР-эксперимента, направленного на определение оптимальной температуры аннулирования пары tTC28-LINE-1обратной грунтовки. Для этой реакции использовалась геномная ДНК крови, переваренная с помощью SacI, а затем самолигация для формирования кругового шаблона ДНК. Весьма специфический продукт ПЦР ожидаемого размера (5649 б.п.) при температуре 62, 64 и 66 градусов по Цельсию показывает, что оптимальная температура для этой грунтовочной пары находится в пределах 62-66 градусов по Цельсию.
Мы создали круговой шаблон ДНК путем переваривания геномной ДНК MCF7 с ферментом ограничения SacI с последующим самовылиганием. На рисунке 4 показано, что TTC28-LINE-1 3 трансдукция происходит в линиях ячейки MCF7: de novo вставки могут быть обнаружены как продукты LDI-PCR различных размеров наряду с родным продуктом ПЦР известного размера (5649 bp). Для определения геномных координат целевых сайтов de novo можно секвенировать амплаконы ПЦР (см. раздел протокола 4).
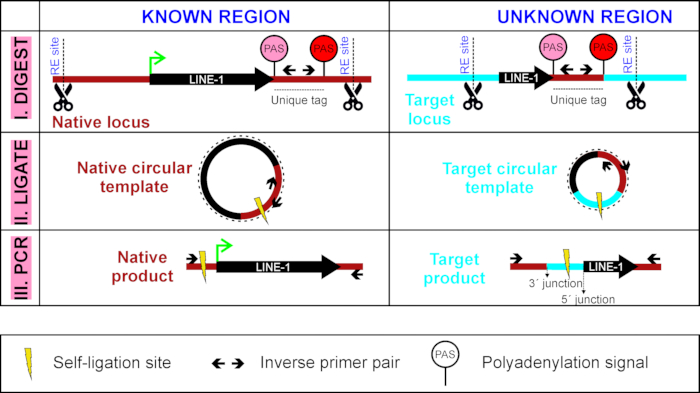
Рисунок 1 : Обзор LDI-PCR для обнаружения LINE-1 3 трансдукции. Круговой шаблон дна произведен first усваивая (I) оно с энзимом ограничения и собственн-ligating (II) оно. Этот шаг сопровождается обратным PCR (III) с обратными пЦР праймеры, ориентированные на уникальный тег LINE-1 интереса (последовательность между line-1 собственной слабее PAS, в розовом, и сильнее PAS вниз по течению, в красном цвете). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
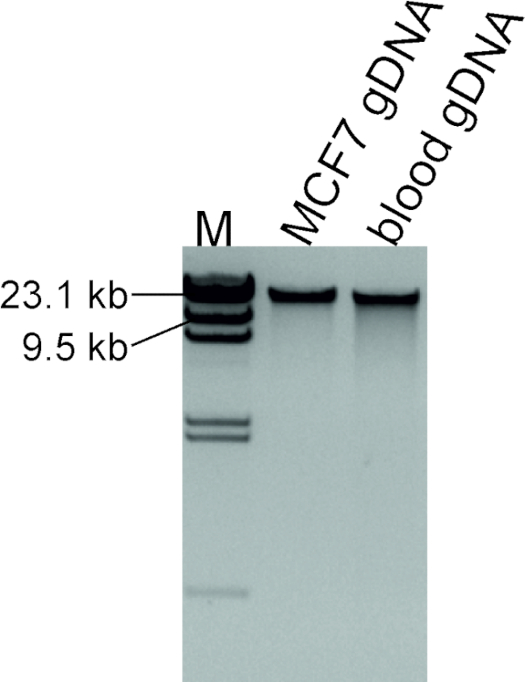
Рисунок 2 : Оценка качества извлеченной ДНК. 100 нг ДНК, извлеченных из линии клеток MCF7 и крови от нормального человека, который будет использоваться в качестве контрольного образца, были запущены вместе с 1 Зл и 2 Зл днк / Хиндии маркер помечены как M. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
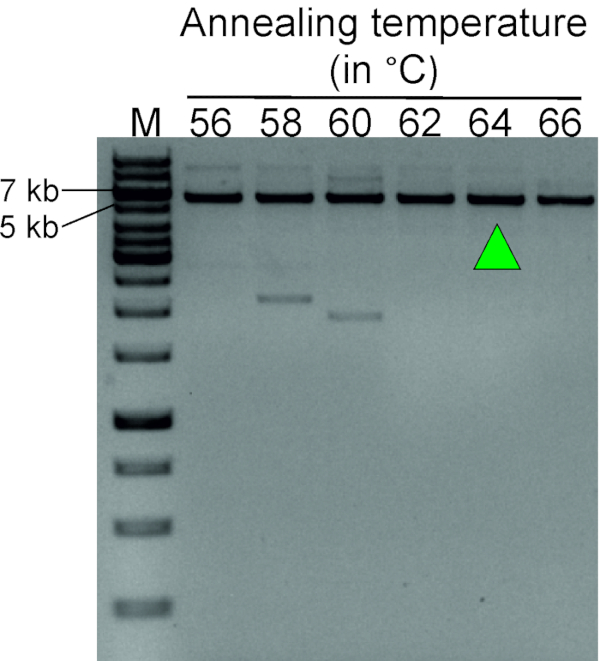
Рисунок 3 : Градиент ПЦР для определения оптимальной температуры annealing для обратных праймеров ПЦР (таблица 1). LDI-PCR с использованием обратных грунтовых пар при температуре annealing в диапазоне от 56 до 66 градусов по Цельсию показывает различный продукт ПЦР при температуре 62-66 градусов по Цельсию. Зеленая стрелка указывает на выбранную температуру аннулирования для будущих экспериментов. Для этого шага оптимизации использовался круговой шаблон ДНК, генерируемый путем переваривания геномной ДНК от нормального человека с помощью SacI, за которым следовала самолигация. M, маркер (1 кБ плюс ДНК лестница). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
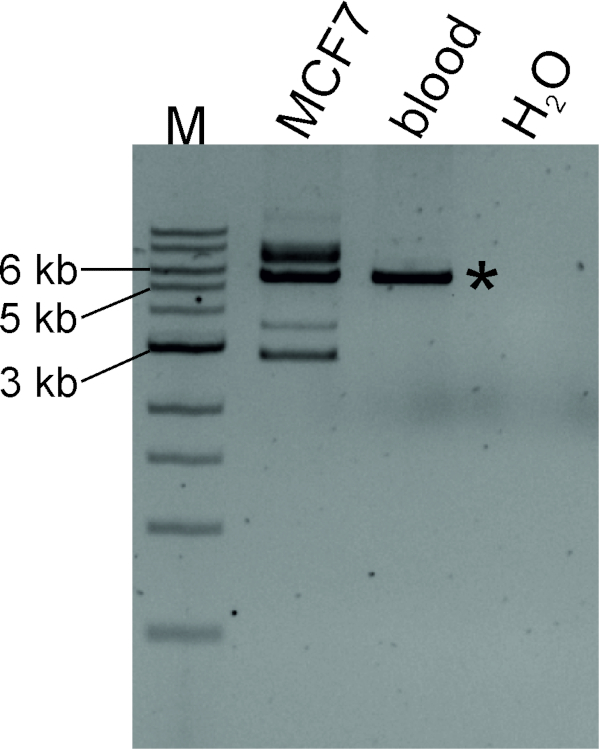
Рисунок 4 : LDI-PCR для идентификации LINE-1 3 и трансдукции, вытекающих из TTC28 -Линия-1. Круговые шаблоны ДНК, генерируемые путем переваривания MCF7 и крови (от нормального человека) геномной ДНК с SacI, за которыми следовала самолигация, были усилены обратными грунтовками в оптимальной температуре аннулирования. "Родной" продукт ПЦР, отмеченный звездочкой, ожидаемого размера (5649 б.п.) был обнаружен как в ДНК MCF7, так и в нормальной ДНК крови, в то время как MCF7 также производил дополнительные продукты ПЦР различных размеров, что указывает на ретротранспозицию de novo LINE-1. M, маркер (1 кБ ДНК лестница). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Имя праймер | Последовательность (5 х 3) |
| L1'001 (rev) | TTCACTAAGCATGTATGTGTGGAAAAC |
| L1'002 (fwd) | CCCAAAATACCCAATACTACTGGCA |
Таблица 1: Обратная пара праймер ПЦР, предназначенная для уникального тега TTC28 -Линия-1 14 Год .
Дополнительный файл. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Здесь мы описываем метод, который может быть использован для идентификации вставок de novo LINE-1, вытекающих из любого активного LINE-1, представляющих интерес. Мы оптимизировали этот метод для высокоактивной LINE-1, расположенной на 22q12.1, и ранее продемонстрировали, что он очень чувствителен при обнаружении субклональных вставок при колоректальном раке14.
Успех LDI-PCR зависит от качества геномной ДНК. Поэтому мы включили дополнительный шаг контроля качества, чтобы убедиться, что в начале протокола присутствует ДНК с высоким молекулярным весом (шаг 2.1.2). Мы рекомендуем хранить геномную ДНК при температуре -20 градусов для длительного хранения, а также готовить аликвоты, чтобы избежать циклов замораживания и оттаивания. Использование геномной ДНК из крови или пациента соответствия нормальной ткани настоятельно рекомендуется различать ли LINE-1 ретротранспозиции обнаружены зародышевой или соматические события. Поскольку места разреза для ферментов ограничения являются стохастичными в геноме, вполне возможно, что конкретный участок вставки de novo LINE-1 может не содержать в себе никаких участков для фермента ограничения, используемого в его окрестностях. Таким образом, чтобы увеличить вероятность обнаружения большинства de novo LINE-1 вставки в ДНК опухоли, более одного ограничения фермента должны быть использованы в отдельных реакциях для создания различных библиотек кругового шаблона ДНК. Кроме того, если уникальный тег LINE-1 интереса имеет более одного PAS, то использование грунтовки пар, прилегающих к каждому PAS повышает шансы обнаружения сильно усеченных трансдукторов.
Хотя элегантные методы для обнаружения генома в масштабах всего de novo LINE-1 существуют, они могут быть подавляющими, если цель состоит в том, чтобы исследовать компетенцию ретротранспозиции конкретной LINE-1 в определенном клеточном контексте. Для этой цели LDI-PCR может быть недорогим и простым, но надежным подходом к визуализации событий ретротранспозиции LINE-1. Подход к таргетингу, используемый в этом методе, аналогичен TS-ATLAS22; однако, LDI-PCR избегает использования олигонуклеотидов связующих и может одновременно усиливать как 5, так и 3 соединения вставки de novo LINE-1. Информация как о 5, так и о 3 развязках вставки LINE-1, целевом объекте интеграции, полиа-хвосте и модификациях целевого участка, которые являются отличительными чертами ретротранспозиции LINE-1, может быть получена путем соединения LDI-PCR с одномолекулярными технологии секвенирования долгочтения. Длинные чтения, таким образом, содержат вставленную последовательность LINE-1, ее уникальный тег и целевые последовательности в одном чтении, обходя трудности отображения коротких считываемых в повторяющейся области.
Существует два основных ограничения для использования LDI-PCR для обнаружения деятельности LINE-1. Первый присущ ПЦР: он может только надежно усиливать фрагменты до 10 кб в размерах. Это следует учитывать при выборе фермента ограничения (ы), так как родной фрагмент не должен превышать этот предел. Во-вторых, этот метод может обнаружить только события ретротранспозиции, которые мобилизуют 3-й фланговый регион LINE-1 на 3 трансдукции. Таким образом, активность тех LINE-1, которые не демонстрируют 3 трансдукции, не будет обнаружена с помощью этого метода. Кроме того, несмотря на усиление LDI-PCR, некоторые события ретротранспозиции LINE-1, которые (а) генерируют цель ПЦР такого же размера, как "родное" местоположение или другие ретротранспозиции или (b) редкие или подклональные, не могут быть обнаружены агарозным гелем Электрофорез. Такие события ретротранспозиции LINE-1 могут быть зафиксированы путем секвенирования продукта LDI-PCR с использованием одной молекулы длинночитаемых технологий секвенирования14.
Описанный здесь рабочий процесс может быть легко изменен для обнаружения активности других «горячих» ЛИНИ-1 с помощью подходящего фермента ограничения и проектирования обратных грунтовок, ориентированных на эти LINE-1. В дополнение к обнаружению LINE-1 опосредованный 3 трансдукции, этот метод может быть адаптирован для обнаружения менее частых LINE-1 опосредощенных 5 и трансдукции23. Аналогичный метод был использован для выявления интеграции сайта LINE-1 репортеров в клеточных анализов24 и провирусной интеграции сайтов в рак25. Помимо line-1 вставки, этот метод также может быть использован для обнаружения других геномных аберраций, таких как перестановки ДНК, где информация о перестановочных регионах, предшествуеет26.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Мы хотели бы поблагодарить всех наших соавторов в статье, где этот метод был впервые описан14, особенно Татьяна Cajuso, Киммо Пэйлин, Outi Kilpivaara и Эса Питкянен для ценных дискуссий при разработке метода. Л.К. финансируется Академией Финляндии (гранты 25996, 292789, 306026 и 314394), Фондом Сигрида Джузелиуса и Финским онкологическим обществом. Б.П. является лауреатом стипендий Исследовательского фонда Хельсинкского университета, диссертационного гранта Финского онкологического общества и докторского гранта от Иды Монтинин Сюти. Мы также благодарим Теему Масалина (Хельсинкский университет) и Кул Шреста из исследовательской группы Л.К. за помощь в видеопроизводстве.
Материалы
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
Ссылки
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16(2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343(2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. Jr High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043(2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521(2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Jr Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484(2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены