需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
从植物提取物和分数中识别新型抗菌和抗生物膜分子的系统方法,以防止牙科蛀牙
* 这些作者具有相同的贡献
摘要
天然产品是开发新药和治疗剂的有希望的起点。然而,由于化学多样性高,从植物中寻找新的治疗化合物是一项具有挑战性和耗时的任务。我们描述了从植物提取物和分数中识别抗菌和抗生物膜分子的简化方法。
摘要
天然产品提供结构不同的物质,具有无数的生物活动。然而,由于复杂的植物基质和耗时的隔离和鉴定程序,从植物中识别和分离活性化合物是具有挑战性的。因此,提出了一种从植物中筛选天然化合物的渐进方法,包括分离和识别潜在的活性分子。它包括植物材料的收集:原油提取物的准备和分馏;色谱和光谱学(UHPLC-DAD-HRMS和NMR)分析和化合物鉴定方法:生物分析(抗菌和抗生物膜活动;细菌"粘附力"到唾液颗粒和初始葡萄糖基质用选定的治疗治疗):和数据分析。该模型简单、可重复,可持续控制多种化合物的高通量筛选、浓度和处理步骤。获得的数据为今后的研究提供了基础,包括含有最活跃提取物和/或分数的配方、分子的分离、微生物细胞和生物膜中特定目标的分子建模。例如,控制致癌生物膜的一个目标是抑制链 球菌突变 体葡萄糖转移酶的活性,这种转移酶合成细胞外基质的葡萄糖。这些酶的抑制可以防止生物膜的积累,降低其毒性。
引言
社会中最早使用的药物模型以天然产品为基础。从那时起,人类一直在寻找新的化学物质在自然界中,可以转化为药物1。这一搜索使民族植物筛查的技术和方法不断改进。NPs 提供了结构多样化物质的丰富来源,具有广泛的生物活动,可用于开发替代疗法或辅助疗法。然而,固有的复杂植物矩阵使得活性化合物的分离和鉴定成为一项具有挑战性和耗时的任务。
基于NPs的药物或配方可用于预防和/或治疗影响口腔的几种疾病,包括牙科蛀牙4。牙科蛀牙是全球最普遍的慢性疾病之一,它源于牙齿表面形成的富含糖分的饮食和微生物生物膜(牙菌斑)的相互作用,导致微生物代谢产生的有机酸导致脱氧,如果不治疗,会导致牙齿脱落5、6。虽然其他微生物可能与7有关,但链球菌是一种关键的致癌细菌,因为它是酸性、酸性且细胞外基质构建者。该物种编码多个外酶(如糖素转移酶或Gtfs),使用蔗糖作为基材8,以建立富含外聚糖精的细胞外基质,这是一个毒性决定因素9。此外,真菌念珠菌可以推动细胞外矩阵7的生产。尽管氟化物以各种方式施用,仍然是预防蛀牙10的基础,但需要新的方法作为辅助剂,以提高其有效性。此外,现有的抗斑块模式是基于使用广谱微生物制剂(如氯河西丁)11。作为替代方案,NPs是控制生物膜和预防蛀牙12,13的潜在疗法。
从植物中发现新的生物活性化合物的进一步进展包括必要的步骤或方法,例如:(一) 使用可靠和可重复的协议进行采样,考虑到植物往往表现出特异性:(二) 编制小规模的综合提取物及其各自的分数:(三) 其化学特征的定性和/或复制认为获取多维数据,例如GC-MS、LC-DAD-MS或NMR:(四) 使用可行的高产模型来评估生物活性:(五) 根据多变量数据分析或其他统计工具选择潜在的新热门歌曲:(六) 对目标化合物或有前途的候选人进行隔离和净化:和(七)使用分离化合物2,14对应生物活动的验证。
复制是快速识别原油提取物中已知化合物的过程,允许将新化合物与已经研究过的化合物区分开来。此外,当某些化合物已经描述生物活性时,这个过程可以防止隔离,并且特别有助于检测"频繁的击球手"。它已用于不同的非目标工作流程,从主要化合物识别或活动引导分馏的加速到提取物集合的化学剖析。它可以与代谢学研究完全集成,用于CE的非目标化学分析或代谢物的有针对性识别。所有这些最终导致在隔离程序1,15,16,17之前优先考虑提取物。
因此,在本手稿中,我们描述了从植物提取物和分数中识别抗菌和抗生物膜分子的系统方法。它包括四个多学科步骤:(1) 收集植物材料:((二)制备粗制提取物和馏分,然后进行化学剖面分析:(3) 生物分析:(4)生物和化学数据分析(图1)。因此,我们提出了为分析 Cacasaria西尔维斯特里斯 提取物和针对 链球菌突变 体和 念珠菌13的分馏的抗菌和抗生物膜活动而制定的协议,以及植物化学特征和数据分析的程序。为了简单起眼,这里的重点是演示使用细菌筛选天然化合物的方法。
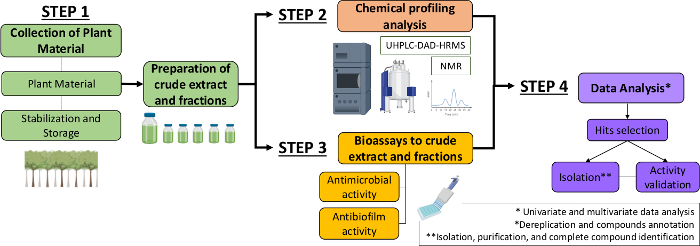
图1:系统方法的流程图,以识别植物提取物和分数中的活性分子。请点击这里查看此数字的较大版本。
研究方案
1. 植物材料收集
- 植物材料
- 在电子平台上记录获得植物材料的情况,该平台将规范在进行采集的国家获得遗传遗产的情况。例如,在巴西,向国家遗传遗产和相关传统知识管理系统注册-SisGen(网站 https://sisgen.gov.br/paginas/login.aspx)。
- 收集感兴趣的植物材料的样本(例如叶子、茎、根、花、水果)。如果材料是在生殖阶段或植物阶段收集的,请进行登记。
- 记录收集参数(日期、地理参考、年平均温度和平均湿度百分比)。
- 精确识别样本,分类学家必须确认真实性。
- 植物样品稳定和储存
- 收集后立即将植物器官分离在单独的塑料袋或烧瓶中。
- (i) 立即冻结液氮中,(ii) 在循环空气烤箱中脱水(40 °C),或 (iii) 通过冻干冷冻干燥样品,从而激活潜在的酶反应。
- 将稳定材料存放在室温或冰柜中密封的袋子中,直至使用(-20 或 -80 °C,具体取决于储存期或预期用途)。
- 在分析磨机中研磨样品(刀或球,取决于组织类型或可用性),并使用标准化筛子标准化颗粒大小。
- 为后续提取步骤单独称重样品。
2. 将原油提取物 (CE) 和分数 (CEF) 准备到化学剖面分析和生物分析
- 原油提取物的制备
注:图 2A中的流程图中显示了这些步骤。- 用水酒精混合物(例如乙醇 (EtOH) 70% 或水、EtOH 和其他修饰剂的三元混合物(根据先前报告的实验设计定义)制备提取溶剂。
- 使用比例样本重量(干重,毫克)/提取溶剂(mL)从50到100毫克不等,每个mL溶剂。
- 对于快速和可重复的萃取,使用微管使用批量萃取。
注:此时应至少使用三个副本,以便进行统计分析。 - 执行超声波辅助提取(阿联酋),使其快速、简单和便宜。
- 为了获得最佳效率,重复三次程序(每次 15 分钟)。
- 每次提取步骤后,通过离心清除固体残留物,并去除超纳坦。
- 将超级纳坦单独组合,过滤并保存 aliquot,以同时进行化学分析和生物分析。如有必要,在真空、氮流或冻素化下去除萃取溶剂,并登记称重和产量。
- 存放在-20°C保护免受光线照射。
注:协议可在此处暂停以筛选 CE 并选择显示所需活动的方案。
- 原油提取物的分馏
注:图 2B中的流程图中显示了这些步骤。- 使用至少 1 克配制的墨盒。如果生物碱可能存在于 CE 中,请使用含有 0.1% 的福米酸 (FA) 的溶剂。
- 在最合适的溶剂(或溶剂混合物)中稀释样品,以获得100毫克/mL的样品溶液。然后,将样品溶液的 1 mL 传输到预制的固相提取墨盒 - SPE(1 克抽调剂,6 mL 容量)。
- 使用每个萃取的大约三个死卷进行分馏(到 1 g 的墨盒,它将对应于每个溶剂的 2 mL )。如果生物碱可能存在于 CE 中,请使用含有 0.1% FA 的溶剂。
- 通过精英成分收集一小部分,并保存阿里引用以同时进行化学分析和生物分析。
- 在真空、氮流或冻毒下去除溶剂,并登记称重和产量。
注:如果 CE 难以溶解在初始的 elution 混合物中,则在将材料装载到墨盒顶部之前,以 1:1(w/w) 的比例将 CE 分散在实心相(例如 C18 或 celite)中。
注意事项: 提前称量微管,计算提取物和分数的质量产量。
- 化学分析
注:考虑到每个植物物种的化学分析都需要优化和具体的方法,在以下部分,我们描述了用于分析植物材料的最常见分析方法。作为一个实际的例子,开发并验证了反相液相色谱,用于同时分析酚类化合物和由两个品种 的Casaria西尔维斯· 斯沃茨(Salicacea)分化的苯酚化合物和克莱罗丹型二元体。UPLC-DAD 设备配备了除气器、第四纪泵、自动取样器、UV-Vis 光电二极管阵列探测器和烤箱(详见 Bueno 等人 2015年 18 日)。下列示例中描述的类似方法可根据其他植物物种和/或植物材料进行优化。- 色谱分析和连字符可能性
- 对于使用超高性能液体色谱 (UPLC) 的分离,请使用 C18 色谱柱(例如,150 × 2.1 毫米、2.6 微米、100°)受兼容的预柱保护。
注:其他柱相或色谱模式可根据植物种类/材料使用。传统 HPLC 也可用于:在这种情况下,必须选择合适的色谱列。要实现出色的分离,请根据流速 (μL/min)、柱温 (°C) 和注入体积 (μL) 调整色谱条件。移动相通常由水 (A) 和丙酮三醇或甲醇 (B) 组成,使用线性或多步排泄梯度或异质精英。也可以使用缓冲区、酸、碱或其他修饰剂。 - 执行植物材料分析 (CE 和/或 CEF),并根据可用的连字符记录所有相关数据,如光谱数据(使用紫外线和/或优先使用 MS 探测器)、保留时间(分钟)和其他数据。
注:液相色谱(LC)通常与高分辨率质谱仪(HRMS)连字符(耦合),作为LC+HRMS,通常用于CE OU CEF15中代谢物的快速注释。 - 如果需要定性数据且需要定量数据,则按照相同的协议仔细准备和注入校准曲线。
注:最佳色谱条件的开发可以在实验设计的帮助下进行,如Bueno等人描述的2015年18日,或类似的文献。在方法开发过程中考虑纳入内部标准非常重要。非常感谢他们,因为他们允许在样品准备和注射过程中正确的技术偏差,并进一步正常化的数据分析。
- 对于使用超高性能液体色谱 (UPLC) 的分离,请使用 C18 色谱柱(例如,150 × 2.1 毫米、2.6 微米、100°)受兼容的预柱保护。
- 色谱分析和连字符可能性
- 一次多变体数据分析
- 以适当的格式(例如ASCII、.txt或.csv格式)导出注册的色谱。如果分析多个样本,并且需要比较,则可以通过连接和对齐色度图来设置单个数据矩阵。由此产生的矩阵必须根据使用的内部标准使色谱规范化。
- 使用多变体和多变体方法分析植物代谢学数据。通过使用多变量统计方法(包括无人监督的主要组件分析 (PCA) 和分层聚类分析 (HCA) 或监督的部分最小方块分析(如 PLS、OPLS、PLS-DA)同时分析多个变量,探索和可视化代谢学数据集。单一的方法,如ANOVA,学生,图基和韦尔奇的t测试,是特别有趣的精确分析样品19之间的定量差异。
- 复制和化合物注释
注:此步骤的目标是快速在线识别已知的 NP,以避免可同时执行的单一或多变量数据分析的乏味隔离。- 执行检测到的化合物或目标化合物的识别水平:
- 已识别的化合物,包括完整的3D结构和立体化学(0级):
- 通过两个正交参数(如保留时间和 MS/MS 频谱(1 级)实现的识别:
- 假定注释化合物和化合物类(2级和3级):
- 不明或未分类的代谢物,可以根据分析数据(4级)19,20进行区分。
- 使用商业或公共数据库对已知化合物进行特征描述。在最重要的数据库中,可以突出显示:NIST(https://www.nist.gov)、威利(https://www.sisweb.com/software/ms/wiley.htm)、大众银行(https://massbank.eu/MassBank/)、GMD(http://gmd.mpimp-golm.mpg.de/)、METLIN(https://metlin.scripps.edu)和全球天然产品社会分子网络——GNPS数据库(https://gnps.ucsd.edu)19。
注:有不同级别的注释,它们依赖于研究期间使用的连字符技术,可能包括:基于MS(或NMR)的光谱数据库的辅助,以及硅胶光谱预测算法。 - 隔离、净化和完整的复合识别
注:如果给定化合物(其身份被统计方法怀疑)需要完整的结构识别,完成此任务的第一步是更大规模地分离和净化所需的化合物。可以通过扩展已开发的协议来实现。- 通过已建立和优化的预制色谱技术,对目标化合物进行快速和直接隔离。半准备HPLC与干负荷注入可用于避免妥协,通常需要在高负荷和样品溶解1之间。
- 完成分离化合物的完整结构特征和鉴定。这可以通过不同的技术组合来完成:
- 核磁共振(核磁共振):
- 质谱学( MS):
- 紫外线(UV)和红外(IR)区域的光谱技术对于功能组的表征也非常有用:
- 使用脊医光谱学,如电子和振动圆形二极论(ECD和VCD,分别),拉曼光学活性(ROA)和X射线结晶术是绝对配置特征的重要技术。
- 执行检测到的化合物或目标化合物的识别水平:
3. 生物分析
注:生物筛选:为了快速评估CE和CEF潜在的生物活性,应组织和直接对天然物质进行初步筛查。
- 为生物分析准备 CE 和 CEF
- 用最好的溶剂(可以实验确定)重组干物质。实验设计21、22定义了溶剂的库存溶液和浓度。
- 计算库存溶液的溶剂浓度。为此,请使用公式:C1 x V1 = C2 x V2,其中 C1 表示 CE 和/或 CEF 的股票解决方案 (mg):V1 表示溶剂的体积:C2 是 CE 和/或 CEF 的权重:V2 是库存解决方案的最终卷 (mL)。
注:例如,我们选择了84.15%EtOH和15%二甲基硫化物(DMSO)作为库存溶液的溶剂浓度。我们准备了CE的库存浓度为6毫克/mL,CEF的库存浓度为1毫克/mL13。稀释CE和CEF的溶剂将取决于生物活性的评价方法。用作车辆的溶剂不得干扰生物和毒理学活动。通常,水,DMSO,EtOH,或基于EtOH的水溶剂用于溶解植物提取物或植物衍生物4,13。
- 测试生物的制备
- 重新激活微生物菌株,例如血琼脂上的S.突变UA159(48小时, 37 °C, 5% CO2),并培养它在液体培养介质 (例如, tryptone-酵母提取物汤 [TYE: 2.5% (w/v) 尝试酮与 1.5% (w/v) 酵母提取物] 含有 1% 葡萄糖 (w/v) (TYEg) 16 小时, 37 ° C, 5% CO2.
- 在同一培养介质中对微生物的初始培养进行1:20稀释(初始培养的稀释比可能根据实验设计而改变)。
- 孵化,直到达到中日志生长阶段。
- 在TYEg中准备具有定义人群的生物测定(例如,每毫升2x106 个菌落形成单位-CFU/mL),用于抗微生物检测;TYE,配以1%蔗糖(w/v)(TYE)用于生物膜检测。
注:生长条件将取决于测试的微生物。
- 抗菌活性
注:这些步骤以图 3示例。- 在 96 井板中,添加 CE 和/或 CEF 库存解决方案(处理)的别名 (μL)。名言的体积由测试浓度定义,测试浓度必须根据以前的研究进行选择。例如,要在 0.5 毫克/mL 测试浓度下测试 CE,请在 6 mg/mL 下使用 16.67 μL 的库存溶液。为此计算,使用公式:C1 x V1 = C2 x V2,其中 C1 是库存浓度,V1 是库存溶液的体积,C2 是测试浓度,V2 是 96 井板的体积(对应 200μ L)。在此实验条件下,溶剂(车辆)的测试浓度为 7% EtOH 和 1.25% DMSO。
- 包括一组对照每个板:一列与治疗,没有不孕症(空白控制每个治疗,帮助区分浊度通过治疗本身与微生物生长):带车辆和气管的柱子(CE 或 CEF 或 0 毫克/mL 控制的凹痕);仅具有培养介质(培养介质控制)的列和仅具有不言自检(微生物生长控制)的列。
- 使用 TYEg,将音量调整为 100 μL。接下来,孵化,例如,24小时,37°C,5%二氧化碳 (取决于测试的微生物)。
- 将 100μL 的微生物接种到 96 井板中(1x10 6 CFU/mL)。
- 通过对油井的目视检查(晴朗或多云),根据浊度分析细菌生长。清晰:意味着微生物没有视觉增长。多云:意味着微生物有视觉增长。
- 测量每口井细菌培养物的吸收(光学密度或 O.D.)(使用 540 nm 的 ELISA 读卡器)。接下来,将100μL的培养物转移到含有900μL盐溶液(0.89%NaCl)的微管中,通过旋涡混合良好。接下来,继续执行十倍串行稀释,直到达到预期值。
- 在特定的琼脂板(重复)中接种所需稀释的别名。例如,血液琼脂板上特定稀释的 10 微升。
- 孵化。微生物之间的条件可以改变,例如,S.突变体:48小时,37°C,5%二氧化碳。
- 执行殖民地计数在板上以后转换为CFU/mL作为(一些殖民地x 10 n)/q。在此公式中,n等于稀释的绝对值(0、1、2 或 3),q等于在 mL 中,用于在 agar 板上镀上的每一个稀释板的移液量。此外,CFU/mL 可以转换为日志值。
注:当植物提取物被添加到培养介质中时,可能会产生萃取物颗粒的沉淀。这一事实可能使解释结果变得困难。当微板读取器测量浊度时,也会发生同样的情况,在某些情况下,细胞聚集在微板的底部。此外,根据所使用的提取物,植物叶提取物的颜色可能难以量化浊度23,24。另一种方法使用染料,揭示微生物细胞是否代谢活跃。
- 抗生物胶片活动
注:评估治疗对生物膜形成的影响的步骤见图4。- 生物膜的形成和加工
- 如抗微生物活性协议步骤所述,在 96 井板中稀释培养介质 (TYE) 中的稀释处理。
- 孵化板。在 S. mutans 的 示例中,孵化在 24 小时、37 °C 和 5% CO2期间进行。
- 孵化后,将板放在轨道摇床上(5分钟,37°C,75转/分钟),以松开不粘附于生物膜的细胞。然后,丢弃含有不粘附细胞的培养介质,用0.89%的NaCl清洗剩余的生物膜三次,以去除不粘附细胞。
- 处理过的生物膜生物质的量化
注:这些步骤在 图 4A中的流程图中进行了说明。- 将生物膜清洗在盘子上,为每口井添加 50 μL 的 1% 水晶紫水溶液。
- 在室温下孵化盘子35分钟。
- 用米利Q水清洗染色水井(三次),然后空气干燥60-90分钟。
- 通过在轨道摇床(5 分钟、37 °C、75 rpm)中孵化板,将染色油井中的水晶紫罗兰与 200 μL 的 99% EtOH 分离。
- 将每个油井的 150μL 点名与分离染料转移到另一个板,并量化样品生物量(ELISA 读取器 570 nm)。
- 对经过处理的生物膜的可行微生物群(CFU/mL)进行量化
注:图 4B中的流程图中显示了这些步骤。- 用移液器和 200 μL 的 NaCl 0.89% 从盘子中取出已洗过的生物膜,并将由此产生的悬架单独转移到无菌微管中。
- 每口井额外使用 200 μL 的 NaCl 0.89%,并将其转移到相应的管子中,该管子已经包含 200μL 的初始生物膜悬架。执行此过程,直到达到每个原始井 1 mL 生物膜的总悬浮。
- 使用每根管子的别名来执行十倍的串行稀释。
- 在特定的琼脂板(重复)中接种所需稀释的别名。例如,血液琼脂板上特定稀释的 10 微升。
- 孵化琼脂板(如48小时,37°C,5%二氧化碳),然后计算菌落,以确定CFU/mL如上所述。
- 生物膜的形成和加工
- 生物活性验证阶段
- 唾液颗粒形成
- 使用羟基阿帕特石 (HA) 珠(宏准备陶瓷羟基石类型 I 80 μm) 作为表面,形成唾液膜25.这些珠子表面模仿牙釉质。
- 在微管中称重 HA 珠(例如 10 毫克),并消毒。然后,使用吸附缓冲区(AB缓冲区:50 mM KCl, 1 mM KPO4,1 mm CaCl2,1 mm MgCl2,在 dd-H2O, pH 6.5]25 含有 0.1 m 苯甲基硫化物 (PMSF) 和 0.02% 钠阿齐德 (NaN3)洗珠。
- 收集和准备人类唾液26。有必要得到机构道德委员会的批准。
- 将500μL的唾液加入微管并孵育(40分钟,37°C,24转)。
- 接下来,取出唾液超纳坦并洗珠子(三次用AB缓冲液含有PMSF和NaN3)。sHA珠(带唾液颗粒的HA珠)现已准备好进行下游检测。
注:唾液是从健康志愿者那里收集的。收集后,用AB缓冲液和离心机稀释唾液(1:1 v/v)(1699 x g,20分钟,4°C)。通过过滤消毒(聚乙醚硫氟烃膜过滤器,低结合到0.22微米蛋白质)26.机构道德委员会必须批准这项研究。就我们而言,该机构的道德委员会批准了这项研究(CAAE: 68161417.0.000.5416)。
- 附着在薄膜唾液和葡萄糖上后分离 S. mutans, 用选定的提取物处理
- 如上所述,将微生物培养到中日志生长阶段。
- 当培养物达到所需的 O.D.时,离心机(4000 ×克为 20 分钟),用 0.89% NaCl 溶液清洗,使用与 0.89% NaCl 相同的培养基初始体积重新悬浮颗粒。
- 如果使用链球菌,如 S.突变体,用探针使培养物去链(30s,7W,三次)。如果使用单个细胞有机体,则可以跳过此步骤。
- 检查 O.D. (540 nm) 以将浓度调整为 2 x 106 CFU/mL。
- S. 突变体与唾液颗粒(sHA)的粘附和粘附细胞的分离
注:图 5中的流程图中显示了这些步骤。- 获取上述 sHA 样本。
- 添加一个aliquot(例如,我们添加500μL)的选定治疗(在测试浓度:例如,0.5毫克/mL)或控制在含有 sHA样本的微管中。
- 用治疗或控制(30分钟,37°C,24转/分钟)孵化 sHA 样品;然后,用AB缓冲器(包含PMSF和NaN3)清洗珠子三次。
- 添加微生物培养。在示例中,我们将 500 μL 的 S. mutans 培养(2 x 106 CFU/mL)添加到每个微管中。
- 孵化(1小时,37°C,24 rpm),然后用AB缓冲区清洗三次,去除未绑定的细胞。
- 重新悬念每个样品与阿利克索特(在示例中,我们添加1000μL)的AB缓冲区,并与探头声波(30s,7 W)。
- 使用每个悬架的点名进行十倍的串行稀释,通过在特定的琼脂板上电镀(48 小时、37 °C、5% CO2)来确定可行的菌落数量。接下来,数一数殖民地,以确定上述CFU/mL。
注:声波酸的步骤是执行分离细胞坚持sHA。
- S. 突变体与初始葡萄糖基质(gsHA)的粘附和粘附细胞的分离
注:图6中的流程图中显示了这些步骤。GtfB 酶是从培养超纳坦链球菌米勒里KSB8 中纯化的,该培养物旨在生产 GtfB。净化是使用含有两种蛋白酶抑制剂(0.1 mM PMSF和0.02%NaN 3)27、28的缓冲液,用含有羟基阿帕特石珠的色谱柱进行的。然后,在丙烯酰胺凝胶(SDS-PAGE)上检查酶,并染上硝酸银。酶的Aliquot被储存在-80°C,直到使用。- 获取上述sHA样本。接下来,添加一个别名(在示例中,我们添加 500 μL) 的 GtfB 酶到每个管子中,并在同质化剂中孵育(40 分钟,37 °C,24 rpm)。然后,用 AB 缓冲器清洗三次(包含 PMSF 和 NaN3)。
- 在每个微管中加入含有治疗(或测试浓度对照,例如0.5毫克/mL)的蔗糖基板(100毫醇)的别名(例如,500微升)。
- 在同质化剂中孵育样品(4小时37°C,24 rpm)。然后,用AB缓冲液(PMSF和NaN3)进行三次清洗,以去除未纳入合成葡萄糖 (gsHA样本)的治疗和过量的蔗糖。
- 在每个微管中添加 500μL 的 S. mutans (2 x 106 CFU/mL)的别名(在示例中,我们添加 500 μL)。
- 在同质化剂(1小时37°C,24 rpm)中孵化,用AB缓冲液(带PMSF和NaN3)清洗三次,以去除未绑定的细胞。
- 用 AB 缓冲液(带 PMSF 和 NaN3)的方位引用(例如 1000 μL)重新悬生每个样本,并使用探针进行声波处理,以分离粘附于 gsHA(30 s, 7 W)的细胞。
- 使用每个悬架的点名进行十倍的串行稀释,通过在特定的琼脂板上电镀(48 小时、37 °C、5% CO2)来确定可行的菌落数量。接下来,数一数殖民地,以确定上述CFU/mL。
- 唾液颗粒形成
4. 生物数据分析
- 生物分析数据
- 在电子表格中输入生物分析的原始数据。计算每个治疗的微生物生长抑制日志为(CFU/mL治疗 +1) x 日志10。然后,计算微生物生长抑制的日志百分比,与使用车辆控制(原木10 CFU/mL治疗/平均10 CFU/mL的车辆控制)x 100%相比。
- 经治疗(CE和CEF治疗组)和车辆控制(负控制)治疗的浮法培养物和生物量的正确 O.D.如要更正,请将仅含有培养介质(Ablank)的井中处理过的油井的吸收减去为(A处理组介质 /A负控制介质)x 100%。
- 此校正后,计算生物质抑制的百分比,与车辆控制(A处理生物质/平均车辆控制)x 100% 相比。
- 提交生成的原始数据,用于使用特定软件对数据进行统计分析。
注:对给定治疗有效性的解释使用断点(如 IC50/IC90)确定。这些值被定义为能够抑制细菌生长或生物膜形成24的治疗的最低浓度。这些参数可以帮助解释数据,并为选择活性更好的化合物提供基础。
结果
我们提供了一个例子,使用系统的方法来筛选植物提取物和分数的生物活性,以确定潜在的活性分子为可能的新抗胆碱疗法:抗微生物和抗生物膜活性Cacaaria sylvestris提取物从不同的巴西生物群系对链球菌突变体和念珠菌13。
背景
特定口服微生物-宿主因子-富含蔗糖和淀粉的饮食之间的复杂相互作用可以调节致病生物膜的形?...
讨论
与天然原油提取物工作相关的主要挑战包括其复杂的组成和经典生物制导隔离研究的不足。虽然这个过程是缓慢的,但它是有效的,并导致在NP研究的主要发现。为了合理化,需要优先级驱动的研究来合理化。因此,使用现代化学分析方法分析CE和分离前的复制是重要的特征研究材料,特别是有用的,以避免重新隔离已知化合物与已经描述的生物活性2,15。<...
披露声明
没有宣布任何利益冲突。
致谢
我们感谢联合国ESP化学研究所(NuBBE)的比奥莱奥·德·比奥恩萨奥斯、比奥森特塞·埃·普罗杜托斯·纳图拉伊斯(NuBBE)为植物材料的编写提供实验室。我们还感谢牙科材料和普罗斯特霍顿学部、UNESP、阿拉夸拉/SP的应用微生物实验室。这项研究得到了圣保罗研究基金会(FAPESP #2013/07600–3 至 AJC)的研究资助和奖学金以及间接费用基金 (FAPESP #2017/07408-6 和 FAPESP #2019/23175-7 到 SMR) 的支持: #2011/21440–3 和#2012/21921–4 至 PCPB)。国家科学和技术发展委员会与FAPESP合作提供了额外支持(国家科学技术中心#465637/2014-0和FAPESP#2014/50926-0至AJC)。
材料
| Name | Company | Catalog Number | Comments |
| 96-well microplates | Kasvi | Flat bottom | |
| Activated carbon | LABSYNTH | Clean up and/or fractionation step | |
| Analytical mill | Ika LabortechniK | Model A11 Basic | |
| Blood agar plates | Laborclin | ||
| Chromatographic column C18 | Phenomenex Kinetex | 150 × 2.1 mm, 2.6 µm, 100Â | |
| Dimethyl sulfoxide | Sigma-Aldrich | Vehicle solution | |
| ELISA plate reader | Biochrom Ez | ||
| Ethanol | J. T. Baker | For extraction and fractionation steps, and mobile phase composition | |
| Ethanol | Sigma-Aldrich | Vehicle solution | |
| Ethyl acetate | J. T. Baker | Fractionation step | |
| GraphPad Software | La Jolla | GraphPad Prism7 | |
| Hexane | J. T. Baker | Fractionation step | |
| Incubator | Thermo Scientific | ||
| Isopropanol | J. T. Baker | For extraction step | |
| Lyophilizer (a freeze dryer) | Savant | Modulyo | |
| Nylon Millipore | LAC | 0.22 µm x 13 mm | |
| Orbital shaker | Quimis | Model G816 M20 | |
| Polyamide solid phase extraction cartridge | Macherey-Nagel | Clean up and/or fractionation step | |
| Silica gel | Merck | 40–63 μm, 60 Â | |
| Sodium Chloride (NaCl) | Synth | 0,89% in water | |
| Solid phase extraction cartridges (SPE) | Macherey-Nagel | Clean up and/or fractionation step | |
| Tryptone | Difco | ||
| UHPLC-DAD | Dionex | Ultimate 3000 RS | |
| Ultrasonic bath | UNIQUE | Model USC 2800 | |
| Yeast extract | Difco |
参考文献
- Newman, D. J., Cragg, G. M. Natural Products as Sources of New Drugs over the Nearly Four Decades from 01/1981 to 09/2019. Journal of Natural Products. 83 (3), 770-803 (2020).
- Wolfender, J. L., Litaudon, M., Touboul, D., Queiroz, E. F. Innovative omics-based approaches for prioritisation and targeted isolation of natural products – new strategies for drug discovery. Natural Product Report. 36 (6), 855-868 (2019).
- Michel, T., Halabalaki, M., Skaltsounis, A. New Concepts, Experimental Approaches, and Dereplication Strategies for the Discovery of Novel Phytoestrogens from Natural Sources. Planta Medica. 79 (7), 514-532 (2013).
- Jeon, J. G., Rosalen, P. L., Falsetta, M. L., Koo, H. Natural products in caries research: current (limited) knowledge, challenges and future perspective. Caries Research. 45 (3), 243-263 (2011).
- Tonetti, M. S., Jepsen, S., Jin, L., Otomo-Corgel, J. Impact of the global burden of periodontal diseases on health, nutrition and wellbeing of mankind: A call for global action. Journal of Clinical Periodontology. 44 (5), 456-462 (2017).
- Peres, M. A., et al. Oral diseases: a global public health challenge. Lancet. 394 (10194), 249-260 (2019).
- Bowen, W. H., Burne, R. A., Wu, H., Koo, H. Oral biofilms: pathogens, matrix, and polymicrobial interactions in microenvironments. Trends Microbiology. 26 (3), 229-242 (2018).
- Paes Leme, A. F., Koo, H., Bellato, C. M., Bedi, G., Cury, J. A. The role of sucrose in cariogenic dental biofilm formation--new insight. Journal of Dental Research. 85 (10), 878-887 (2006).
- Koo, H., Falsetta, M. L., Klein, M. I. The exopolysaccharide matrix: a virulence determinant of cariogenic biofilm. Journal of Dental Research. 92 (12), 1065-1073 (2013).
- Cury, J. A., de Oliveira, B. H., dos Santos, A. P., Tenuta, L. M. Are dental fluoride releasing materials clinically effective on caries control. Dental Materials. 32 (3), 323-333 (2016).
- Mattos-Graner, R. O., Klein, M. I., Smith, D. J. Lessons Learned from Clinical Studies: Roles of Mutans Streptococci in the Pathogenesis of Dental Caries. Current Oral Health Reports. 1, 70-78 (2014).
- Rocha, G. R., Florez Salamanca, E. J., de Barros, A. L., Lobo, C. I. V., Klein, M. I. Effect of tt-farnesol and myricetin on in vitro biofilm formed by Streptococcus mutans and Candida albicans. BMC Complementary and Alternative Medicine. 18 (1), 61 (2018).
- Ribeiro, S. M., et al. Antimicrobial and antibiofilm activities of Casearia sylvestris extracts from distinct Brazilian Biomes against Streptococcus mutans and Candida albicans. BMC Complementary and Alternative Medicine. 19 (1), 308 (2019).
- Pilon, A. C., et al. Metabolômica de plantas: métodos e desafios. Quimica Nova. 43 (3), 329-354 (2020).
- Wolfender, J. L., Nuzillard, J. M., Hooft, J. J. J., Renault, J. H., Bertrand, S. Accelerating Metabolite Identification in Natural Product Research: Toward an Ideal Combination of Liquid Chromatography-High-Resolution Tandem Mass Spectrometry and NMR Profiling, in Silico Databases, and Chemometrics. Analytical Chemistry. 91 (1), 704-742 (2019).
- Allard, P. M., et al. Pharmacognosy in the digital era: shifting to contextualized metabolomics. Current opinion in biotechnology. 54, 57-64 (2018).
- Hubert, J., Nuzillard, J., Renault, J. Dereplication strategies in natural product research: How many tools and methodologies behind the same concept. Phytochemistry Reviews. 16, 55-95 (2017).
- Bueno, P. C. P., Pereira, F. M. V., Torres, R. B., Cavalheiro, A. J. Development of a comprehensive method for analysing clerodane-type diterpenes and phenolic compounds from Casearia sylvestris Swartz (Salicaceae) based on ultra-high performance liquid chromatography combined with chemometric tools. Journal of separation science. 38 (10), 1649-1656 (2015).
- Bueno, P. C. P., Lopes, N. P. Metabolomics to Characterize Adaptive and Signaling Responses in Legume Crops under Abiotic Stresses. American Chemical Society omega. 5 (4), 1752-1763 (2020).
- Blaženović, I., Kind, T., Ji, J., Fiehn, O. Software tools and approaches for compound identification of LC-MS/MS data in metabolomics. Metabolites. 8 (2), 31 (2018).
- Eloff, J. N. Quantifying the bioactivity of plant extracts during screening and bioassay-guided fractionation. Phytomedicine: International Journal Of Phytotherapy And Phytopharmacology. 11 (4), 370-371 (2004).
- Rios, J. L., Recio, M. C. Medicinal plants and antimicrobial activity. Journal of Ethnopharmacology. 100 (1-2), 80-84 (2005).
- Eloff, J. N. A sensitive and quick microplate method to determine the minimal inhibitory concentration of plant extracts for bacteria. Planta Medica. 64, 711-714 (1998).
- Eloff, J. N. Avoiding pitfalls in determining antimicrobial activity of plant extracts and publishing the results. BMC Complementary and Alternative Medicine. 19 (1), 106 (2019).
- Klein, M. I., Xiao, J., Heydorn, A., Koo, H. An analytical tool-box for comprehensive biochemical, structural and transcriptome evaluation of oral biofilms mediated by mutans streptococci. Journal of Visualized Experiments. (47), e2512 (2011).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to study the physiology of oral biofilms. Methods in molecular biology. 666, 87-102 (2010).
- Venkitaraman, A. R., Vacca-Smith, A. M., Kopec, L. K., Bowen, W. H. Characterization of glucosyltransferase B, GtfC, and GtfD in solution and on the surface of hydroxyapatite. Journal of Dental Research. 74, 1695-1701 (1995).
- Vacca-Smith, A. M., Venkitaraman, A. R., Quivey, R. G., Bowen, W. H. Interactions of streptococcal glucosyltransferases with alpha-amylase and starch on the surface of saliva-coated hydroxyapatite. Archives of Oral Biology. 41, 291-298 (1996).
- Van Dijck, P., et al. Methodologies for in vitro and in vivo evaluation of efficacy of antifungal and antibiofilm agents and surface coatings against fungal biofilms. Microbial Cell. 5 (7), 300-326 (2018).
- Marsh, P. D. Are dental diseases examples of ecological catastrophes. Microbiology. 149 (2), 279-294 (2003).
- Bowen, W. H., Koo, H. Biology of Streptococcus mutans-derived glucosyltransferases: role in extracellular matrix formation of cariogenic biofilms. Caries Research. 45 (1), 69-86 (2011).
- Lobo, C. I. V., et al. Dual-species biofilms of Streptococcus mutans and Candida albicans exhibit more biomass and are mutually beneficial compared with single-species biofilms. Journal of Oral Microbioly. 11 (1), 1581520 (2019).
- Kim, D., et al. Candida albicans stimulates Streptococcus mutans microcolony development via crosskingdom biofilm-derived metabolites. Scientific reports. 7, 41332 (2017).
- Ferreira, P. M. Folk uses and pharmacological properties of Casearia sylvestris: a medicinal review. Anais da Academia Brasileira de Ciencias. 83 (4), 1373-1384 (2011).
- Xia, L., Guo, Q., Tu, P., Chai, X. The genus Casearia: a phytochemical and pharmacological overview. Phytochemistry Reviews. 14, 99-135 (2015).
- Ferreira, P. M. P., et al. Toxicological findings about an anticancer fraction with casearins described by traditional and alternative techniques as support to the Brazilian Unified Health System (SUS). Journal of Ethnopharmacol. 15, 241 (2019).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. Journal of Bacteriology. 192 (12), 3024-3032 (2010).
- Maske, T. T., van de Sande, F. H., Arthur, R. A., Huysmans, M. -. C. D. N. J. M., Cenci, M. S. In vitro biofilm models to study dental caries: a systematic review. Biofouling. 33 (8), 661-675 (2017).
- Fu, Y., Luo, J., Qin, J., Yang, M. Screening techniques for the identification of bioactive compounds in natural products. Journal of Pharmaceutical and Biomedical Analysis. 168, 189-200 (2019).
- Sarker, S. D., Nahar, L. An introduction to natural products isolation. Methods in molecular biology. 864, 1-25 (2012).
- Clinical Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptibility testing; twenty-fifth informational supplement. Clinical Laboratory Standards Institute (CLSI). , (2015).
- Saputo, S., Faustoferri, R. C., Quivey, R. G. A drug repositioning approach reveals that Streptococcus mutans is susceptible to a diverse range of established antimicrobials and nonantibiotics. Antimicrobial Agents and Chemotherapy. 62 (1), 01674 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。