È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Approccio sistematico per identificare nuove molecole antimicrobiche e antibiofilm dagli estratti e dalle frazioni delle piante per prevenire la carie dentale
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I prodotti naturali rappresentano punti di partenza promettenti per lo sviluppo di nuovi farmaci e agenti terapeutici. Tuttavia, a causa dell'elevata diversità chimica, trovare nuovi composti terapeutici dalle piante è un compito impegnativo e dispendioso in termini di tempo. Descriviamo un approccio semplificato per identificare molecole antimicrobiche e antibiofilm da estratti e frazioni vegetali.
Abstract
I prodotti naturali forniscono sostanze strutturalmente diverse, con una miriade di attività biologiche. Tuttavia, l'identificazione e l'isolamento dei composti attivi dalle piante sono impegnativi a causa della complessa matrice vegetale e delle lunghe procedure di isolamento e identificazione. Pertanto, viene presentato un approccio graduale per lo screening dei composti naturali dalle piante, incluso l'isolamento e l'identificazione di molecole potenzialmente attive. Comprende la raccolta del materiale vegetale; preparazione e frazionamento di estratti grezzi; approcci cromatografici e spettrometrici (UHPLC-DAD-HRMS e NMR) per l'analisi e l'identificazione dei composti; test biometrici (attività antimicrobiche e antibiofilm; "forza di adesione" batterica al pellicolo salivare e matrice di glucano iniziale trattata con trattamenti selezionati); analisi dei dati. Il modello è semplice, riproducibile e consente lo screening ad alta produttività di più composti, concentrazioni e fasi di trattamento che possono essere controllati in modo coerente. I dati ottenuti forniscono le basi per studi futuri, tra cui formulazioni con gli estratti e/o frazioni più attivi, isolamento delle molecole, modellazione di molecole a target specifici in cellule microbiche e biofilm. Ad esempio, un obiettivo per controllare il biofilm cariogenico è quello di inibire l'attività dello Streptococcus mutans glucosyltransferasi che sintetizzano i glucani della matrice extracellulare. L'inibizione di questi enzimi impedisce l'accumulo di biofilm, diminuendone la virulenza.
Introduzione
I primi modelli di medicina utilizzati nelle società erano basati su prodotti naturali (NP). Da allora, gli esseri umani sono alla ricerca di nuove sostanze chimiche in natura che possono essere trasformate infarmaci 1. Questa ricerca ha causato un miglioramento continuo delle tecnologie e dei metodi per lo screening etnobotanico1,2,3. I PNP offrono una ricca fonte di sostanze strutturalmente diverse, con una vasta gamma di attività biologiche utili per lo sviluppo di terapie alternative o adiuvanti. Tuttavia, la matrice vegetale complessa intrinseca rende l'isolamento e l'identificazione dei composti attivi un compito impegnativo e dispendioso in termini di tempo4.
I farmaci o le formulazioni a base di PNP possono essere utilizzati per prevenire e/o trattare diverse condizioni che influenzano l'orale, compresa la carie dentale4. La carie dentale, una delle malattie croniche più diffuse a livello globale, deriva dall'interazione della dieta ricca di zucchero e dei biofilm microbici (placca dentale) formatisi sulla superficie dentale che porta alla demineralizzazione causata da acidi organici derivati dal metabolismo microbico e, se non trattati, porta alla perditadei denti 5,6. Sebbene altri microrganismi possano essere associati a7, Lo Streptococcus mutans è un batterio cariogenico critico perché acidogeno, acidurico e un generatore di matrici extracellulari. Questa specie codifica per esoenzimi multipli (ad esempio glicosiltransferasi o Gtfs) che usano il saccarosio come substrato8 per costruire la matrice extracellulare ricca di esopolisaccaridi, che sono un determinante della virulenza9. Inoltre, il fungo Candida albicans può aumentare la produzione di quella matrice extracellulare7. Sebbene il fluoruro, somministrato in varie modalità, rimanga la base per prevenire la carie dentale10, sono necessari nuovi approcci come coadiuvanti per aumentarne l'efficacia. Inoltre, le modalità anti-placca disponibili si basano sull'uso di agenti microbicidi ad ampio spettro (ad esempio, clorexidina)11. In alternativa, i PNP sono potenziali terapie per controllare i biofilm e prevenire la cariedentale 12,13.
L'ulteriore progresso nella scoperta di nuovi composti bioattivi dalle piante comprende misure o approcci necessari quali: i) l'uso di protocolli affidabili e riproducibili per il campionamento, considerando che le piante mostrano spesso variabilità intraspecifica; — la preparazione di estratti completi e delle rispettive frazioni su piccola scala; — la caratterizzazione e/o la dereplicazione dei loro profili chimici ha ritenuto che l'acquisizione di dati multidimensionali come GC-MS, LC-DAD-MS o NMR, per esempio; — l'uso di modelli validi e ad alto rendimento per valutare la bioattività; — la selezione di potenziali nuovi riscontri basati sull'analisi multivariata dei dati o su altri strumenti statistici; — effettuare l'isolamento e la purificazione dei composti mirati o dei candidati promettenti; e vii) la convalida delle attività biologiche corrispondenti utilizzando i composti isolati2,14.
La dereplicazione è il processo di identificazione rapida di composti noti nell'estratto grezzo e consente di differenziare nuovi composti da quelli che sono già stati studiati. Inoltre, questo processo previene l'isolamento quando la bioattività è già stata descritta per alcuni composti ed è particolarmente utile rilevare "battitori frequenti". È stato utilizzato in diversi flussi di lavoro non mirati che vanno dall'identificazione dei composti principali o all'accelerazione del frazionamento guidato dall'attività fino alla profilazione chimica delle raccolte di estratti. Può essere completamente integrato con studi metabolomici per la profilazione chimica non mirata di CE o l'identificazione mirata dei metaboliti. Tutto ciò alla fine porta a dare priorità alle estrazioni prima delle proceduredi isolamento 1,15,16,17.
Pertanto, nel presente manoscritto, descriviamo un approccio sistematico per identificare molecole antimicrobiche e antibiofilm da estratti e frazioni vegetali. Esso comprende quattro fasi multidisciplinari: (1) raccolta di materiale vegetale; 2) preparazione di estratti grezzi (CE) e frazioni (MCE), seguita dalla loro analisi del profilo chimico; 3) biosasay; e (4) analisi dei dati biologici e chimici(figura 1). Presentiamo così il protocollo sviluppato per analizzare le attività antimicrobiche e antibiofilmali degli estratti e frazioni di Casearia sylvestris contro Streptococcus mutans e Candida albicans13,nonché le procedure per la caratterizzazione fitochimica e l'analisi dei dati. Per semplicità, l'obiettivo qui è dimostrare l'approccio per lo screening dei composti naturali usando il batterio.
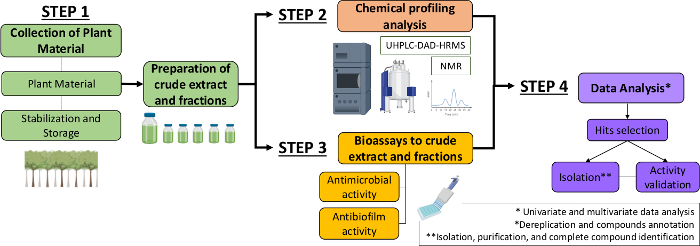
Figura 1: Diagramma di flusso dell'approccio sistematico per identificare molecole attive da estratti e frazioni vegetali. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Raccolta di materiale vegetale
- Materiale vegetale
- Registrare l'accesso al materiale vegetale su piattaforme elettroniche che regolano l'accesso al patrimonio genetico nel paese in cui si svolgerà la raccolta. Ad esempio, in Brasile, registrati al Sistema Nazionale per la Gestione del Patrimonio Genetico e delle Conoscenze Tradizionali Associate - SisGen (sito web https://sisgen.gov.br/paginas/login.aspx).
- Raccogliere campioni del materiale vegetale di interesse (ad esempio, foglie, steli, radici, fiori, frutti). Registrare se il materiale è stato raccolto durante la fase riproduttiva o vegetativa.
- Registrare i parametri di raccolta (data, georeferenziazione, temperatura media annuale e percentuale di umidità media).
- Identificare i campioni con precisione e un tassonomista deve confermare l'autenticità.
- Stabilizzazione e stoccaggio dei campioni vegetali
- Separare gli organi vegetali in singoli sacchetti di plastica o fiasche immediatamente dopo le raccolte.
- Inattivare le potenziali reazioni enzimatiche mediante i) congelamento immediato in azoto liquido, ii) disidratazione in un forno ad aria circolante (40 °C) o (iii) liofilizzazione dei campioni per liofilizzazione.
- Conservare il materiale stabilizzato in sacchetti ermeticamente sigillati a temperatura ambiente o in un congelatore fino all'uso (-20 o -80 °C, a seconda del periodo di conservazione o dell'uso previsto).
- Macinare i campioni in un mulino analitico (coltello o sfera, a seconda del tipo di tessuto o della disponibilità) e standardizzare la dimensione delle particelle utilizzando setacci standardizzati.
- Pesare i campioni singolarmente per le successive fasi di estrazione.
2. Preparazione di estratti grezzi (CE) e frazioni (CEF) all'analisi del profilo chimico e ai bioacidi
- Preparazione di estratti grezzi (CE)
NOTA: i passaggi sono illustrati nel diagramma di flusso nella figura 2A.- Preparare un solvente da estrazione con una miscela idroalcolica (ad esempio, etanolo (EtOH) 70% o miscele ternarie di acqua, EtOH e altri modificatori, definiti dalla progettazione sperimentale secondo i rapporti precedenti).
- Utilizzare il rapporto peso campione (peso secco, mg)/ solvente da estrazione (mL) variabile da 50 a 100 mg per ogni mL di solvente.
- Per estrazioni veloci e riproducibili, utilizzare estrazioni batch utilizzando microtubi.
NOTA: A questo punto devono essere utilizzate almeno tre repliche per consentire l'analisi statistica. - Esegui estrazioni assistite da ultrasuoni (UAE) per renderlo rapido, facile ed economico.
- Ripetere la procedura tre volte (15 minuti ciascuna) per la migliore efficienza.
- Dopo ogni fase di estrazione, decantare il residuo solido mediante centrifugazione e rimuovere il supernatante.
- Combinare i supernatanti singolarmente, filtrare e salvare le aliquote per analisi chimiche simultanee e biosatti. Se necessario, rimuovere il solvente estratto sotto vuoto, il flusso di azoto o la lofilizzazione e registrare il peso e la resa.
- Conservare a -20 °C protetto dalla luce.
NOTA: Il protocollo può essere messo in pausa qui per schermare il CE e selezionare quelli che presentano l'attività desiderata.
- Frazionamento degli estratti grezzi (MCE)
NOTA: i passaggi sono illustrati nel diagramma di flusso nella figura 2B.- Utilizzare cartucce con almeno 1 g di adsorbente. Se gli alcaloidi sono potenzialmente presenti nella CE, utilizzare solventi contenenti lo 0,1% di acido formico (FA).
- Diluire il campione nel solvente più adatto (o miscela di solventi) per ottenere una soluzione per campioni da 100 mg/mL. Quindi, trasferire 1 mL della soluzione campione in una cartuccia di estrazione in fase solida precondizione - SPE (1 g di adsorbente, 6 mL di capacità).
- Eseguire il frazionamento utilizzando circa tre volumi morti di ogni eluente di estrazione (ad una cartuccia di 1 g, corrisponderà a 2 mL di ogni solvente). Se gli alcaloidi sono potenzialmente presenti nella CE, utilizzare solventi contenenti lo 0,1% di FA.
- Raccogliere una frazione per composizione eluente e salvare le aliquote per l'analisi chimica simultanea e i biosatti.
- Rimuovere il solvente sotto vuoto, flusso di azoto o lyophilization e registrare il peso e la resa.
NOTA: Se il CE è difficile da sciogliere nella miscela di eluizione iniziale, disperdere il CE in una fase solida (ad esempio, C18 o celite) in proporzione 1:1 (w/w) prima di caricare il materiale sulla parte superiore della cartuccia.
ATTENZIONE: Pesare i microtubi in anticipo per calcolare la resa di massa di estratti e frazioni.
- Analisi della profilazione chimica
NOTA: Considerando che ogni specie vegetale richiede metodi ottimizzati e specifici per la sua analisi chimica, nelle sezioni seguenti descriviamo gli approcci analitici più comuni utilizzati per analizzare i materiali vegetali. A titolo pratico, è stata sviluppata e convalidata una cromatografia liquida in fase inversa per l'analisi simultanea di composti fenolici e diterpeni di tipo cleronica biosintesi differenzialmente biosintesi da due varietà di Casearia sylvestris Swartz (Salicaceae). L'apparato UPLC-DAD era dotato di un degassatore, una pompa quaternaria, un campionatore automatico, un rivelatore di array di fotodiodi UV-Vis e un forno (vedi dettagli in Bueno et al. 201518). Approcci simili descritti nei seguenti esempi possono essere ottimizzati in base ad altre specie vegetali e/o materiali vegetali.- Analisi cromatografica e possibilità di sillabazione
- Per le separazioni con cromatografia liquida ad altissime prestazioni (UPLC), utilizzare una colonna cromatografica C18 (ad esempio, 150 × 2,1 mm, 2,6 μm, 100 Å) protetta da una pre-colonna compatibile.
NOTA: Altre fasi di colonna o modalità cromatografiche possono essere utilizzate a seconda della specie/materiale vegetale. L'HPLC convenzionale può anche essere utilizzato; in tal caso, deve essere scelta un'adeguata colonna cromatografica. Per ottenere separazioni eccellenti, regolare le condizioni cromatografiche considerando la portata (μL/min), la temperatura della colonna (°C) e il volume di iniezione (μL). La fase mobile di solito consiste di acqua (A) e acetonitrile o metanolo (B) usando gradienti di eluizione lineari o multistep o eluizione isocratica. È inoltre possibile utilizzare modificatori come buffer, acidi, basi o altri. - Eseguire l'analisi del materiale vegetale (CE e/o CEF) e registrare tutti i dati correlati, come i dati spettrali (utilizzando un UV-Vis e/o, preferenzialmente, rivelatori MS), il tempo di ritenzione (min) e altri, a seconda della sillabazione disponibile.
NOTA: La cromatografia liquida (LC) è solitamente sillabata (accoppiata) con spettrometria di massa ad alta risoluzione (HRMS), come LC−HRMS, ed è comunemente usata per la rapida annotazione del metabolita in CE ou CEF15. - Se sono necessari dati qualitativi e sono necessari dati quantitativi, preparare e iniettare attentamente curve di calibrazione, seguendo lo stesso protocollo.
NOTA: Lo sviluppo delle migliori condizioni cromatografiche può essere eseguito con l'assistenza della progettazione di esperimenti, come descritto da Bueno et al. È importante considerare l'inclusione di norme interne durante lo sviluppo del metodo. Sono molto apprezzati poiché consentono deviazioni tecniche corrette durante la preparazione e le iniezioni del campione e un'ulteriore normalizzazione per l'analisi dei dati.
- Per le separazioni con cromatografia liquida ad altissime prestazioni (UPLC), utilizzare una colonna cromatografica C18 (ad esempio, 150 × 2,1 mm, 2,6 μm, 100 Å) protetta da una pre-colonna compatibile.
- Analisi cromatografica e possibilità di sillabazione
- Analisi dei dati univariata e multivariata
- Esportare i cromatogrammi registrati in un formato adatto (ad esempio, ASCII, .txt. o .csv formato). È possibile impostare una singola matrice di dati unendo e allineando i cromatogrammi se vengono analizzati più campioni e sono necessari confronti. Le matrici risultanti devono essere normalizzate i cromatogrammi secondo lo standard interno utilizzato.
- Analizzare i dati della metabolomica dell'impianto utilizzando metodi multivariati e univariati. Esplorare e visualizzare i set di dati di metabolomica attraverso l'analisi simultanea di più variabili utilizzando metodi statistici multivariati, tra cui l'analisi dei componenti principali non supervisionati (PCA) e l'analisi del clustering gerarchico (HCA), o l'analisi dei meno quadrati parziali supervisionati (come PLS, OPLS, PLS-DA). I metodi univariati, come ANOVA, Student's, Tukey e Welch's t-test, sono particolarmente interessanti per l'analisi precisa delle differenze quantitative tra icampioni 19.
- Annotazione di dereplicazione e composti
NOTA: L'obiettivo di questo passaggio è dedicato alla rapida identificazione on-line di NP noti per evitare noiosi isolamento che possono essere eseguiti contemporaneamente all'analisi dei dati uni- o multivariate.- Eseguire i livelli di identificazione dei composti rilevati o bersaglio:
- Composto identificato, compresa la struttura 3D completa e la stereochimica (livello 0);
- Identificazione ottenuta da due parametri ortogonali, come il tempo di ritenzione e lo spettro MS/MS (livello 1);
- Composti e classi composte annotati putativamente (livelli 2 e 3);
- Metaboliti non identificati o non classificati che possono essere differenziati in base a dati analitici (livello 4)19,20.
- Caratterizzare i composti noti utilizzando i database commerciali o pubblici. Tra le banche dati più importanti, si possono evidenziare: NIST (https://www.nist.gov), Wiley (https://www.sisweb.com/software/ms/wiley.htm), MassBank (https://massbank.eu/MassBank/), GMD (http://gmd.mpimp-golm.mpg.de/), METLIN (https://metlin.scripps.edu) e il Global Natural Products Social Molecular Networking – database GNPS (https://gnps.ucsd.edu)19.
NOTA: Ci sono diversi livelli di annotazioni e dipendono dalla tecnica sillabata utilizzata durante lo studio e possono includere: l'assistenza di database spettrali basati su MS- (o NMR) e negli algoritmi di previsione spettrale silico. - Isolamento, purificazione e identificazione completa del composto
NOTA: Se un dato composto (la cui identità è stata sospettata dai metodi statistici) necessita di un'identificazione strutturale completa, il primo passo per svolgere questo compito è isolare e purificare i composti desiderati su una scala maggiore. Può essere realizzato scalando i protocolli già sviluppati.- Eseguire l'isolamento rapido e diretto del composto o dei composti bersaglio mediante tecniche cromatografiche preparatorie ben consolidate e ottimizzate. L'HPLC semi-preparativo con iniezione di carico a secco può essere utilizzato per evitare i compromessi che generalmente devono essere fatti tra carico elevato e solubilizzazione del campione1.
- Realizzare la caratterizzazione strutturale completa e l'identificazione di composti isolati. Questo può essere fatto attraverso la combinazione di diverse tecniche:
- Risonanza magnetica nucleare (NMR);
- Spettrometria di massa (MS);
- Le tecniche spettrometriche nelle regioni ultravioletta (UV) e infrarossa (IR) sono anche molto utili per la caratterizzazione dei gruppi funzionali;
- L'uso della spettroscopia chiropticale come il dicroismo circolare elettronico e vibrazionale (ECD e VCD, rispettivamente), l'attività ottica Raman (ROA) e la cristallografia a raggi X sono tecniche importanti per la caratterizzazione assoluta della configurazione.
- Eseguire i livelli di identificazione dei composti rilevati o bersaglio:
3. Biosasay
NOTA: Screening biologico: Per valutare rapidamente la potenziale bioattività di CE e CEF, lo screening iniziale delle sostanze naturali dovrebbe essere organizzato e diretto.
- Preparazione di CE e CEF per i biosasay
- Ricostituire la sostanza secca con i migliori solventi possibili (che possono essere determinati sperimentalmente). Il progetto sperimentale21,22 definisce la soluzione stock e la concentrazione di solventi.
- Calcolare la concentrazione di solvente della soluzione stock. Per fare ciò, utilizzare la formula: C1 x V1 = C2 x V2, dove C1 rappresenta la soluzione stock (mg) di CE e/o CEF; V1 rappresenta il volume del solvente; C2 è il peso del CE e/o del CEF; V2 è il volume finale (mL) della soluzione stock.
NOTA: Ad esempio, abbiamo selezionato l'84,15% di EtOH e il 15% di solfossido di dimetile (DMSO) come concentrazione di solvente della soluzione stock. Abbiamo preparato la concentrazione di ce a 6 mg/mL e il CEF a 1 mg/mL13. Il solvente per diluire il CE e il CEF dipenderà dal metodo di valutazione dell'attività biologica. Il solvente utilizzato come veicolo non deve interferire con l'attività biologica e tossicologica. Tipicamente, acqua, DMSO, EtOH o un solvente acquoso a base di EtOH viene utilizzato per solubilizzare estratti vegetali o derivati vegetali4,13.
- Preparazione di organismi di prova
- Riattivare un ceppo microbico, ad esempio S. mutans UA159 sull'agar del sangue (48 h, 37 °C, 5% CO2), e coltura in mezzo di coltura liquida (ad esempio, brodo estratto di lievito di tripptone [TYE: 2,5% (w/v) triptone con estratto di lievito 1,5% (w/v) contenente 1% di glucosio (w/v) (TYEg) per 16 h, 37 ° C, 5% CO2.
- Eseguire una diluizione 1:20 della coltura iniziale del microrganismo nello stesso mezzo di coltura (il rapporto di diluizione della coltura iniziale può cambiare in base al progetto sperimentale).
- Incubare fino a raggiungere la fase di crescita a metà log.
- Preparare l'inoculo per i bioacidi con una popolazione definita (ad esempio, 2x106 unità di formazione di colonie per millilitro - CFU/mL) in TYEg per saggi antimicrobici e TYE con 1% di saccarosio (w/v) (TYE) per saggi di biofilm.
NOTA: Le condizioni di crescita dipenderanno dal microrganismo testato.
- Attività antimicrobica
NOTA: I passaggi sono illustrati nella figura 3.- In una piastra da 96 po', aggiungere un'aliquota (μL) della soluzione stock CE e/o CEF (trattamenti). Il volume dell'aliquota è definito dalla concentrazione di prova, che deve essere selezionata sulla base di studi precedenti. Ad esempio, per testare CE alla concentrazione di prova di 0,5 mg/mL, utilizzare un'aliquota di 16,67 μL della soluzione stock a 6 mg/mL. Per questo calcolo, utilizzare la formula: C1 x V1 = C2 x V2, dove C1 è la concentrazione del materiale, V 1 è il volume dell'aliquota dellasoluzione stock, C2 è la concentrazione di prova e V2 è il volume della piastra da 96 pozzi (che corrisponde a 200 μL). In questa condizione sperimentale, la concentrazione di prova dei solventi (veicolo) sarà del 7% EtOH e dell'1,25% DMSO.
- Includere una serie di controlli per ogni piastra: una colonna con trattamenti, senza l'inoculo (controllo vuoto per trattamento, aiuta a differenziare la torbidità dal trattamento utilizzato dalla crescita microbica); una colonna con veicolo e inoculo (diluente del controllo CE o CEF o 0 mg/mL); una colonna con solo il mezzo di coltura (controllo del mezzo di coltura) e una colonna con solo inoculo (controllo della crescita microbica).
- Utilizzando TYEg, regolare il volume a 100 μL. Successivamente, incubare, ad esempio, 24 ore, 37 °C, 5% CO2 (a seconda del microrganismo testato).
- Inoculare 100 μL di microrganismo inoculo (1x 106 CFU/mL) nella piastra a 96 porri.
- Analizzare la crescita batterica in base alla torbidità mediante ispezione visiva dei pozzi (chiari o torbidi). Chiaro: significa che non c'è crescita visiva del microrganismo. Nuvoloso: significa che c'è crescita visiva del microrganismo.
- Misurare l'assorbanza (densità ottica o O.D.) della coltura batterica in ogni pozzo (lettore ELISA utilizzando 540 nm). Successivamente, trasferire 100 μL delle colture su microtubi contenenti 900 μL di soluzione salina (0,89% NaCl), mescolare bene mediante vortice. Continuare quindi a eseguire una diluizione seriale dieci volte fino al valore desiderato.
- Inoculare un'aliquota della diluizione desiderata in specifiche piastre di agar (in duplicato). Ad esempio, 10 μL di una diluizione specifica sulle placche di agar del sangue.
- Incubare. Le condizioni possono cambiare tra i microrganismi, ad esempio S. mutans: 48 h, 37 °C, 5% CO2.
- Eseguire il conteggio delle colonie sulle piastre per la successiva trasformazione in CFU/mL come (Uncerto numero di colonie x 10n)/q. In questa formula,n è uguale al valore assoluto della diluizione (0, 1, 2 o 3), e q è uguale alla quantità, in mL, pipettata per ogni diluizione placcata sulla piastra di agar. Inoltre, il CFU/mL può essere convertito in valori di log.
NOTA: Quando gli estratti vegetali vengono aggiunti al mezzo di coltura, può verificarsi la precipitazione di particelle dagli estratti. Questo fatto può rendere difficile l'interpretazione dei risultati. Lo stesso accade quando un lettore di micropiatte misura la torbidità come, in alcuni casi, le cellule si aggrappano sul fondo della micropiatta. Inoltre, a seconda dell'estratto utilizzato, il colore degli estratti di foglie vegetali può rendere difficile quantificare latorbidità 23,24. Un metodo alternativo utilizza coloranti che rivelano se le cellule microbiche sono metabolicamente attive o meno24.
- Attività antibiofilm
NOTA: I passaggi per valutare l'effetto dei trattamenti sulla formazione di biofilm sono illustrati nella figura 4.- Formazione ed elaborazione di biofilm
- Diluire i trattamenti in mezzo di coltura (TYE) in una piastra da 96 pozzetti come descritto nelle fasi del protocollo Di attività antimicrobica.
- Incubare il piatto. Nell'esempio con S. mutans, l'incubazione viene eseguita durante 24 ore, a 37 °C e 5% CO2.
- Dopo l'incubazione, posizionare le piastre su uno shaker orbitale (5 min, 37 °C, 75 giri/min) per allentare le cellule non aderenti al biofilm. Quindi, scartare il mezzo di coltura contenente le cellule non aderenti e lavare i biofilm rimanenti tre volte con lo 0,89% di NaCl per rimuovere le cellule non aderenti.
- Quantificazione della biomassa da biofilm trattati
NOTA: i passaggi sono illustrati nel diagramma di flusso nella figura 4A.- Mantenere i biofilm lavati sul piatto e aggiungere 50 μL di soluzione acquosa viola cristallo all'1% ad ogni pozzo.
- Incubare la piastra a temperatura ambiente per 35 minuti.
- Lavare i pozzi macchiati con acqua MilliQ (tre volte) e poi asciugarli all'aria per 60-90 minuti.
- Elute il viola cristallino dai pozzi macchiati con 200 μL di 99% EtOH incubando la piastra in uno shaker orbitale (5 min, 37 °C, 75 giri/min).
- Trasferire un'aliquota di 150 μL da ogni pozzo con il colorante eluitato su un'altra piastra e quantificare la biomassa del campione (lettore ELISA 570 nm).
- Quantificazione della popolazione microbica vitale (CFU/mL) dei biofilm trattati
NOTA: i passaggi sono illustrati nel diagramma di flusso nella figura 4B.- Rimuovere i biofilm lavati dalla piastra con una pipetta e 200 μL di NaCl 0,89% e trasferire la sospensione risultante singolarmente su microtubi sterili.
- Utilizzare altri 200 μL di NaCl 0,89% per pozzo e trasferirlo nel tubo corrispondente, contenente già 200 μL della sospensione iniziale del biofilm. Eseguire questo processo fino a raggiungere una sospensione totale di 1 mL di biofilm per pozzo originale.
- Utilizzare un'aliquota da ogni tubo per eseguire una diluizione seriale di dieci volte.
- Inoculare un'aliquota della diluizione desiderata in specifiche piastre di agar (in duplicato). Ad esempio, 10 μL di una diluizione specifica sulle placche di agar del sangue.
- Incubare le piastre di agar (ad esempio, 48 h, 37 °C, 5% CO2), quindi contare le colonie per determinare il CFU/mL come descritto sopra.
- Formazione ed elaborazione di biofilm
- Fase di convalida dell'attività biologica
- Formazione pellicle salivare
- Utilizzare perline idrossiapatite (HA) (Macro-Prep Ceramic Hydroxyapatite Type I 80 μm) come superficie per formare il film salivare25. Queste perline di superficie imitano lo smalto dentale.
- Pesare le perline ha (ad esempio, 10 mg) in microtubi e sterilizzare. Quindi, utilizzare il buffer di adsorbimento (buffer AB: 50 mM KCl, 1 mM KPO4, 1 mM CaCl2, 1 mM MgCl2, in dd-H2O, pH 6.5]25 contenente 0,1 mM di fluoruro di fenilmetilsulfonil (PMSF) e 0,02% di azide di sodio (NaN3)per lavare le perline.
- Raccogliere e preparare la salivaumana 26. È necessario ottenere l'approvazione del comitato etico istituzionale.
- Aggiungere 500 μL di saliva nei microtubi e incubare (40 min, 37 °C, 24 giri/min).
- Quindi, rimuovere il supernatante saliva e lavare le perline (tre volte con tampone AB contenente PMSF e NaN3). Le perle sHA (perline HA con pellicle salivare) sono ora pronte per i test a valle.
NOTA: La saliva viene raccolta da volontari sani. Dopo la raccolta, diluire la saliva (1: 1 v/v) con tampone AB e centrifuga (1699 x g, 20 min, 4 °C). Sterilizzare mediante filtrazione (filtro a membrana in polieteresolfone a basso legame a 0,22 μm di proteine)26. Il Comitato Etico Istituzionale deve approvare lo studio. Nel nostro caso, il Comitato Etico dell'Istituzione ha approvato lo studio (CAAE: 68161417.0.0000.5416).
- Distacco di S. mutans dopo adesione al film salivare e glucani trattati con estratti selezionati
- Coltivare il microrganismo fino alla fase di crescita a metà log, come descritto sopra.
- Quando le colture hanno raggiunto l'O.D. desiderato, centrifuga (4000 × g per 20 min), lavare con soluzione nacl allo 0,89% e rimescolare il pellet con 0,89% NaCl utilizzando lo stesso volume iniziale del mezzo di coltura.
- Se si utilizza uno streptococchi, come S. mutans, sonicare le colture con una sonda da dechain (30 s, 7 W, tre volte). Se si utilizza un singolo organismo cellulare, questo passaggio può essere saltato.
- Controllare l'O.D. (540 nm) per regolare la concentrazione a 2 x 106 CFU/mL.
- Adesione di S. mutans al pellicolo salivare (sHA) e distacco delle cellule aderenti
NOTA: i passaggi sono illustrati nel diagramma di flusso nella figura 5.- Ottenere i campioni di sHA come descritto in precedenza.
- Aggiungere un'aliquota (nell'esempio, aggiungiamo 500 μL) di trattamenti selezionati (alla concentrazione di prova; ad esempio, 0,5 mg/mL) o controlli in microtubi contenenti campioni di sHA.
- Incubare i campioni di sHA con trattamenti o controlli (30 min, 37 °C, 24 giri/min); quindi, lavare le perline tre volte con tampone AB (contenente PMSF e NaN3).
- Aggiungere la cultura del microrganismo. Nell'esempio, aggiungiamo 500 μL di coltura di S. mutans (2 x 106 CFU/mL) a ogni microtubo.
- Incubare (1 h, 37 °C, 24 giri/min) e quindi rimuovere le celle non vincolate lavandosi tre volte con tampone AB.
- Rimospendare ogni campione con un'aliquota (nell'esempio, aggiungiamo 1000 μL) di buffer AB e sonicare con una sonda (30 s, 7 W).
- Utilizzare un'aliquota di ciascuna sospensione per una diluizione seriale di dieci volte per determinare il numero di colonie vitali placcando su piastre di agar specifiche (48 h, 37 °C, 5% CO2). Quindi, conta le colonie per determinare il CFU/mL come descritto sopra.
NOTA: Il passaggio del sonicato viene eseguito per staccare le cellule aderenti a sHA.
- Adesione di S. mutans alla matrice glucano iniziale (gsHA) e distacco di cellule aderenti
NOTA: i passaggi sono illustrati nel diagramma di flusso nella figura 6. L'enzima GtfB è stato purificato dal supernatante coltura Streptococcus milleri KSB8 progettato per produrre GtfB. La purificazione è stata eseguita con una colonna cromatografica contenente perline di idrossiapatite utilizzando tamponi contenenti due inibitori della proteasi (0,1 mM PMSF e 0,02% NaN3)27,28. Quindi, l'enzima è stato controllato su gel di acrilammide (SDS-PAGE) e macchiato con nitrato d'argento. Le aliquote dell'enzima sono state conservate a -80 °C fino all'uso.- Ottenere i campioni di sHA come descritto in precedenza. Quindi, aggiungere un'aliquota (nell'esempio, aggiungiamo 500 μL) di enzima GtfB a ciascun tubo e incubare in un omogeneizzatore (40 min, 37 °C, 24 giri/min). Quindi, lavare tre volte con buffer AB (contenente PMSF e NaN3).
- Aggiungere ad ogni microtubo un'aliquota (ad esempio, 500 μL) di substrato di saccarosio (100 mmol di saccarosio) contenente i trattamenti (o i controlli alla concentrazione di prova, ad esempio 0,5 mg/mL).
- Incubare i campioni in un omogeneizzatore (4 h, 37 °C, 24 giri/min). Quindi, eseguire tre lavaggi con tampone AB (con PMSF e NaN3) per rimuovere i trattamenti e l'eccesso di saccarosio non incorporato nei glucani sintetizzati (campioni di gsHA).
- Aggiungere un'aliquota (nell'esempio, aggiungiamo 500 μL) di S. mutans inoculum (2 x 106 CFU/mL) a ciascun microtubo.
- Incubare in un omogeneizzatore (1 h, 37 °C, 24 giri/min) e lavare tre volte con tampone AB (con PMSF e NaN3)per rimuovere le cellule non vincolate.
- Resuspend ogni campione con un'aliquota (ad esempio, 1000 μL) di tampone AB (con PMSF e NaN3) e sonicare con una sonda per staccare le cellule aderenti al gsHA (30 s, 7 W).
- Utilizzare un'aliquota di ciascuna sospensione per una diluizione seriale di dieci volte per determinare il numero di colonie vitali placcando su piastre di agar specifiche (48 h, 37 °C, 5% CO2). Quindi, conta le colonie per determinare il CFU/mL come descritto sopra.
- Formazione pellicle salivare
4. Analisi biologica dei dati
- Dati sui biosasay
- Inserire dati grezzi per i biosasay in un foglio di calcolo. Calcolare il registro dell'inibizione della crescita microbica per ogni trattamento come (ACFU/mL di trattamenti + 1) x log10. Quindi, calcolare la percentuale di log dell'inibizione della crescita microbica, rispetto al controllo del veicolo utilizzando (Alog10 CFU/mLdi trattamento /media Alog10 CFU/mL del controllo del veicolo) x 100%.
- O.D. corretto delle colture planctoniche e della biomassa trattate da trattamenti (gruppi trattati ce e CEF) e dal controllo del veicolo (controllo negativo). Per la correzione, sottrarre l'assorbanza dei pozzi trattati da quella ottenuta in pozzi contenenti solo mezzi di coltura (Ablank) come (Agruppi trattati medio /Un mezzo dicontrollonegativo ) x 100%.
- Dopo questa correzione, calcolare la percentuale di inibizione della biomassa, rispetto al controllo del veicolo come(biomassa trattata/controllo medio delveicoloA ) x 100%.
- Inviare i dati grezzi generati per l'analisi statistica dei dati utilizzando software specifico.
NOTA: L'interpretazione dell'efficacia di un determinato trattamento è determinata utilizzando punti di interruzione, come l'IC50/IC90. Questi valori sono definiti come la concentrazione minima di un trattamento in grado di inibire rispettivamente il 50% e il 90%, della crescita batterica o della formazione di biofilm24. Questi parametri possono aiutare a interpretare i dati e fornire una base per la selezione di composti con unamigliore attività 13,29.
Risultati
Forniamo un esempio di utilizzo di un approccio sistematico per migliorare l'attività biologica degli estratti vegetali e delle frazioni per identificare molecole potenzialmente attive per possibili nuove terapie anti-carie: attività antimicrobiche e antibiofilm degli estratti di Casearia sylvestris da distinti biomi brasiliani contro Streptococcus mutans e Candida albicans13.
Priorità bassa
Interazioni complesse tra spe...
Discussione
Le principali sfide legate al lavoro con estratti grezzi naturali comprendono la loro composizione complessa e le inadeguatezze dei classici studi di isolamento bioguidato. Sebbene questo processo sia lento, è efficace e ha portato a importanti risultati nella ricerca NP. Per razionalizzare, sono necessari studi basati sulla definizione delle priorità per razionalizzare. Pertanto, l'uso di moderni approcci di profilazione chimica per l'analisi del CE e della dereplicazione prima dell'isolamento è importante per caratt...
Divulgazioni
Nessun conflitto di interessi dichiarato.
Riconoscimenti
Esprimiamo la nostra gratitudine a Núcleo de Bioensaios, Biossíntese ed Ecofisiologia de Produtos Naturais (NuBBE) dell'Istituto di Chimica dell'UNESP, Araraquara/SP per aver fornito i laboratori per la preparazione del materiale vegetale. Ringraziamo anche il Laboratorio di Microbiologia Applicata del Dipartimento di Materiali Dentali e Protesi, UNESP, Araraquara/SP. Questa ricerca è stata sostenuta da una sovvenzione di ricerca della São Paulo Research Foundation (FAPESP #2013/07600-3 all'AJC) e da borse di studio più fondi generali (FAPESP #2017/07408-6 e FAPESP #2019/23175-7 alla SMR; #2011/21440-3 e #2012/21921-4 al PCPB). Il Consiglio nazionale per lo sviluppo scientifico e tecnologico in collaborazione con fapesp ha fornito ulteriore sostegno (INCT CNPq #465637/2014-0 e FAPESP #2014/50926–0 all'AJC).
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well microplates | Kasvi | Flat bottom | |
| Activated carbon | LABSYNTH | Clean up and/or fractionation step | |
| Analytical mill | Ika LabortechniK | Model A11 Basic | |
| Blood agar plates | Laborclin | ||
| Chromatographic column C18 | Phenomenex Kinetex | 150 × 2.1 mm, 2.6 µm, 100Â | |
| Dimethyl sulfoxide | Sigma-Aldrich | Vehicle solution | |
| ELISA plate reader | Biochrom Ez | ||
| Ethanol | J. T. Baker | For extraction and fractionation steps, and mobile phase composition | |
| Ethanol | Sigma-Aldrich | Vehicle solution | |
| Ethyl acetate | J. T. Baker | Fractionation step | |
| GraphPad Software | La Jolla | GraphPad Prism7 | |
| Hexane | J. T. Baker | Fractionation step | |
| Incubator | Thermo Scientific | ||
| Isopropanol | J. T. Baker | For extraction step | |
| Lyophilizer (a freeze dryer) | Savant | Modulyo | |
| Nylon Millipore | LAC | 0.22 µm x 13 mm | |
| Orbital shaker | Quimis | Model G816 M20 | |
| Polyamide solid phase extraction cartridge | Macherey-Nagel | Clean up and/or fractionation step | |
| Silica gel | Merck | 40–63 μm, 60 Â | |
| Sodium Chloride (NaCl) | Synth | 0,89% in water | |
| Solid phase extraction cartridges (SPE) | Macherey-Nagel | Clean up and/or fractionation step | |
| Tryptone | Difco | ||
| UHPLC-DAD | Dionex | Ultimate 3000 RS | |
| Ultrasonic bath | UNIQUE | Model USC 2800 | |
| Yeast extract | Difco |
Riferimenti
- Newman, D. J., Cragg, G. M. Natural Products as Sources of New Drugs over the Nearly Four Decades from 01/1981 to 09/2019. Journal of Natural Products. 83 (3), 770-803 (2020).
- Wolfender, J. L., Litaudon, M., Touboul, D., Queiroz, E. F. Innovative omics-based approaches for prioritisation and targeted isolation of natural products – new strategies for drug discovery. Natural Product Report. 36 (6), 855-868 (2019).
- Michel, T., Halabalaki, M., Skaltsounis, A. New Concepts, Experimental Approaches, and Dereplication Strategies for the Discovery of Novel Phytoestrogens from Natural Sources. Planta Medica. 79 (7), 514-532 (2013).
- Jeon, J. G., Rosalen, P. L., Falsetta, M. L., Koo, H. Natural products in caries research: current (limited) knowledge, challenges and future perspective. Caries Research. 45 (3), 243-263 (2011).
- Tonetti, M. S., Jepsen, S., Jin, L., Otomo-Corgel, J. Impact of the global burden of periodontal diseases on health, nutrition and wellbeing of mankind: A call for global action. Journal of Clinical Periodontology. 44 (5), 456-462 (2017).
- Peres, M. A., et al. Oral diseases: a global public health challenge. Lancet. 394 (10194), 249-260 (2019).
- Bowen, W. H., Burne, R. A., Wu, H., Koo, H. Oral biofilms: pathogens, matrix, and polymicrobial interactions in microenvironments. Trends Microbiology. 26 (3), 229-242 (2018).
- Paes Leme, A. F., Koo, H., Bellato, C. M., Bedi, G., Cury, J. A. The role of sucrose in cariogenic dental biofilm formation--new insight. Journal of Dental Research. 85 (10), 878-887 (2006).
- Koo, H., Falsetta, M. L., Klein, M. I. The exopolysaccharide matrix: a virulence determinant of cariogenic biofilm. Journal of Dental Research. 92 (12), 1065-1073 (2013).
- Cury, J. A., de Oliveira, B. H., dos Santos, A. P., Tenuta, L. M. Are dental fluoride releasing materials clinically effective on caries control. Dental Materials. 32 (3), 323-333 (2016).
- Mattos-Graner, R. O., Klein, M. I., Smith, D. J. Lessons Learned from Clinical Studies: Roles of Mutans Streptococci in the Pathogenesis of Dental Caries. Current Oral Health Reports. 1, 70-78 (2014).
- Rocha, G. R., Florez Salamanca, E. J., de Barros, A. L., Lobo, C. I. V., Klein, M. I. Effect of tt-farnesol and myricetin on in vitro biofilm formed by Streptococcus mutans and Candida albicans. BMC Complementary and Alternative Medicine. 18 (1), 61 (2018).
- Ribeiro, S. M., et al. Antimicrobial and antibiofilm activities of Casearia sylvestris extracts from distinct Brazilian Biomes against Streptococcus mutans and Candida albicans. BMC Complementary and Alternative Medicine. 19 (1), 308 (2019).
- Pilon, A. C., et al. Metabolômica de plantas: métodos e desafios. Quimica Nova. 43 (3), 329-354 (2020).
- Wolfender, J. L., Nuzillard, J. M., Hooft, J. J. J., Renault, J. H., Bertrand, S. Accelerating Metabolite Identification in Natural Product Research: Toward an Ideal Combination of Liquid Chromatography-High-Resolution Tandem Mass Spectrometry and NMR Profiling, in Silico Databases, and Chemometrics. Analytical Chemistry. 91 (1), 704-742 (2019).
- Allard, P. M., et al. Pharmacognosy in the digital era: shifting to contextualized metabolomics. Current opinion in biotechnology. 54, 57-64 (2018).
- Hubert, J., Nuzillard, J., Renault, J. Dereplication strategies in natural product research: How many tools and methodologies behind the same concept. Phytochemistry Reviews. 16, 55-95 (2017).
- Bueno, P. C. P., Pereira, F. M. V., Torres, R. B., Cavalheiro, A. J. Development of a comprehensive method for analysing clerodane-type diterpenes and phenolic compounds from Casearia sylvestris Swartz (Salicaceae) based on ultra-high performance liquid chromatography combined with chemometric tools. Journal of separation science. 38 (10), 1649-1656 (2015).
- Bueno, P. C. P., Lopes, N. P. Metabolomics to Characterize Adaptive and Signaling Responses in Legume Crops under Abiotic Stresses. American Chemical Society omega. 5 (4), 1752-1763 (2020).
- Blaženović, I., Kind, T., Ji, J., Fiehn, O. Software tools and approaches for compound identification of LC-MS/MS data in metabolomics. Metabolites. 8 (2), 31 (2018).
- Eloff, J. N. Quantifying the bioactivity of plant extracts during screening and bioassay-guided fractionation. Phytomedicine: International Journal Of Phytotherapy And Phytopharmacology. 11 (4), 370-371 (2004).
- Rios, J. L., Recio, M. C. Medicinal plants and antimicrobial activity. Journal of Ethnopharmacology. 100 (1-2), 80-84 (2005).
- Eloff, J. N. A sensitive and quick microplate method to determine the minimal inhibitory concentration of plant extracts for bacteria. Planta Medica. 64, 711-714 (1998).
- Eloff, J. N. Avoiding pitfalls in determining antimicrobial activity of plant extracts and publishing the results. BMC Complementary and Alternative Medicine. 19 (1), 106 (2019).
- Klein, M. I., Xiao, J., Heydorn, A., Koo, H. An analytical tool-box for comprehensive biochemical, structural and transcriptome evaluation of oral biofilms mediated by mutans streptococci. Journal of Visualized Experiments. (47), e2512 (2011).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to study the physiology of oral biofilms. Methods in molecular biology. 666, 87-102 (2010).
- Venkitaraman, A. R., Vacca-Smith, A. M., Kopec, L. K., Bowen, W. H. Characterization of glucosyltransferase B, GtfC, and GtfD in solution and on the surface of hydroxyapatite. Journal of Dental Research. 74, 1695-1701 (1995).
- Vacca-Smith, A. M., Venkitaraman, A. R., Quivey, R. G., Bowen, W. H. Interactions of streptococcal glucosyltransferases with alpha-amylase and starch on the surface of saliva-coated hydroxyapatite. Archives of Oral Biology. 41, 291-298 (1996).
- Van Dijck, P., et al. Methodologies for in vitro and in vivo evaluation of efficacy of antifungal and antibiofilm agents and surface coatings against fungal biofilms. Microbial Cell. 5 (7), 300-326 (2018).
- Marsh, P. D. Are dental diseases examples of ecological catastrophes. Microbiology. 149 (2), 279-294 (2003).
- Bowen, W. H., Koo, H. Biology of Streptococcus mutans-derived glucosyltransferases: role in extracellular matrix formation of cariogenic biofilms. Caries Research. 45 (1), 69-86 (2011).
- Lobo, C. I. V., et al. Dual-species biofilms of Streptococcus mutans and Candida albicans exhibit more biomass and are mutually beneficial compared with single-species biofilms. Journal of Oral Microbioly. 11 (1), 1581520 (2019).
- Kim, D., et al. Candida albicans stimulates Streptococcus mutans microcolony development via crosskingdom biofilm-derived metabolites. Scientific reports. 7, 41332 (2017).
- Ferreira, P. M. Folk uses and pharmacological properties of Casearia sylvestris: a medicinal review. Anais da Academia Brasileira de Ciencias. 83 (4), 1373-1384 (2011).
- Xia, L., Guo, Q., Tu, P., Chai, X. The genus Casearia: a phytochemical and pharmacological overview. Phytochemistry Reviews. 14, 99-135 (2015).
- Ferreira, P. M. P., et al. Toxicological findings about an anticancer fraction with casearins described by traditional and alternative techniques as support to the Brazilian Unified Health System (SUS). Journal of Ethnopharmacol. 15, 241 (2019).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. Journal of Bacteriology. 192 (12), 3024-3032 (2010).
- Maske, T. T., van de Sande, F. H., Arthur, R. A., Huysmans, M. -. C. D. N. J. M., Cenci, M. S. In vitro biofilm models to study dental caries: a systematic review. Biofouling. 33 (8), 661-675 (2017).
- Fu, Y., Luo, J., Qin, J., Yang, M. Screening techniques for the identification of bioactive compounds in natural products. Journal of Pharmaceutical and Biomedical Analysis. 168, 189-200 (2019).
- Sarker, S. D., Nahar, L. An introduction to natural products isolation. Methods in molecular biology. 864, 1-25 (2012).
- Clinical Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptibility testing; twenty-fifth informational supplement. Clinical Laboratory Standards Institute (CLSI). , (2015).
- Saputo, S., Faustoferri, R. C., Quivey, R. G. A drug repositioning approach reveals that Streptococcus mutans is susceptible to a diverse range of established antimicrobials and nonantibiotics. Antimicrobial Agents and Chemotherapy. 62 (1), 01674 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon