É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Abordagem Sistemática para Identificar Novas Moléculas antimicrobianas e antibiofilmes de extratos e frações de plantas para prevenir cárie dentária
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Os produtos naturais representam pontos de partida promissores para o desenvolvimento de novas drogas e agentes terapêuticos. No entanto, devido à alta diversidade química, encontrar novos compostos terapêuticos das plantas é uma tarefa desafiadora e demorada. Descrevemos uma abordagem simplificada para identificar moléculas antimicrobianas e antibiofilmes a partir de extratos e frações vegetais.
Resumo
Os produtos naturais fornecem substâncias estruturalmente diferentes, com uma miríade de atividades biológicas. No entanto, a identificação e o isolamento de compostos ativos das plantas são desafiadores devido à complexa matriz vegetal e aos procedimentos demorados de isolamento e identificação. Por isso, é apresentada uma abordagem stepwise para a triagem de compostos naturais das plantas, incluindo o isolamento e identificação de moléculas potencialmente ativas. Inclui a coleta do material vegetal; preparação e fracionamento de extratos brutos; abordagens de cromatografia e espectrometria (UHPLC-DAD-HRMS e NMR) para análise e identificação de compostos; bioensações (atividades antimicrobianas e antibiofilmes; "força de adesão" bacteriana à pellícula salivar e matriz glucana inicial tratada com tratamentos selecionados); e análise de dados. O modelo é simples, reprodutível e permite a triagem de alto rendimento de múltiplos compostos, concentrações e passos de tratamento podem ser consistentemente controlados. Os dados obtidos fornecem a base para estudos futuros, incluindo formulações com extratos e/ou frações mais ativos, isolamento de moléculas, modelagem de moléculas para alvos específicos em células microbianas e biofilmes. Por exemplo, um alvo para controlar o biofilme cariogênico é inibir a atividade de glucosyltransferases da matriz extracelular que sintetizam os glúcanos da matriz extracelular. A inibição dessas enzimas impede o acúmulo de biofilme, diminuindo sua virulência.
Introdução
Os primeiros modelos de medicina utilizados nas sociedades foram baseados em produtos naturais (NPs). Desde então, os humanos têm procurado por novos produtos químicos na natureza que possam ser transformados em drogas1. Essa pesquisa causou uma melhoria contínua das tecnologias e métodos para a triagem etnobotânica1,2,3. Os NPs oferecem uma rica fonte de substâncias estruturalmente diversas, com uma ampla gama de atividades biológicas úteis para o desenvolvimento de terapias alternativas ou adjuvantes. No entanto, a matriz vegetal complexa inerente torna o isolamento e identificação dos compostos ativos uma tarefa desafiadora e demorada4.
Medicamentos ou formulações baseadas em NPs podem ser usados para prevenir e/ou tratar várias condições que afetam a oral, incluindo cárie dentária4. A cárie dentária, uma das doenças crônicas mais prevalentes globalmente, deriva da interação da dieta rica em açúcar e dos biofilmes microbianos (placa dentária) formados na superfície dentária que leva à desmineralização causada por ácidos orgânicos derivados do metabolismo microbiano e, se não tratados, leva à perda de dentes5,6. Embora outros microrganismos possam estar associados7, Streptococcus mutans é uma bactéria cariogênica crítica porque é acidogênica, aciduric, e um construtor de matriz extracelular. Esta espécie codifica múltiplos exoenzymes (por exemplo, glicosyltransferases ou Gtfs) que usam sacarose como substrato8 para construir a matriz extracelular rica em exopolysacarídeos, que são um determinante virulência9. Além disso, os fungos Candida albicans podem impulsionar a produção dessa matriz extracelular7. Embora o flúor, administrado em diversas modalidades, continue sendo a base para a prevenção da cárie dentária10, novas abordagens são necessárias como adjuvantes para aumentar sua eficácia. Além disso, as modalidades anti-placa disponíveis baseiam-se no uso de agentes microbicidais de amplo espectro (por exemplo, clorexidina)11. Como alternativa, as NPs são terapias potenciais para o controle de biofilmes e prevenção de cárie dentária12,13.
O avanço adicional na descoberta de novos compostos bioativos das plantas inclui etapas ou abordagens necessárias, tais como: (i) o uso de protocolos confiáveis e reprodutíveis para amostragem, considerando que as plantas frequentemente apresentam variabilidade intraespecífica; (ii) a elaboração de extratos abrangentes e suas respectivas frações em pequena escala; (iii) a caracterização e/ou dereplicação de seus perfis químicos pensou na aquisição de dados multidimensionais como GC-MS, LC-DAD-MS ou NMR, por exemplo; (iv) a utilização de modelos viáveis e de alto rendimento para avaliar a bioatividade; v A seleção de novos hits potenciais com base na análise de dados multivariados ou outras ferramentas estatísticas; vi Realizar o isolamento e purificação dos compostos direcionados ou candidatos promissores; e (vii) a validação das atividades biológicas correspondentes utilizando os compostos isolados2,14.
A dereplicação é o processo de identificação rápida de compostos conhecidos em extrato bruto e permite diferenciar novos compostos daqueles que já foram estudados. Além disso, esse processo previne o isolamento quando a bioatividade já foi descrita para determinados compostos, e é particularmente útil detectar "rebatedores frequentes". Tem sido usado em diferentes fluxos de trabalho não-alvo, desde a identificação de compostos principais ou a aceleração do fracionamento guiado pela atividade até o perfil químico das coleções de extratos. Pode ser totalmente integrado com estudos metabolômicos para o perfil químico não direcionado de CE ou a identificação direcionada de metabólitos. Tudo isso, em última análise, leva à priorização de extratos antes dos procedimentos de isolamento1,15,16,17.
Portanto, no presente manuscrito, descrevemos uma abordagem sistemática para identificar moléculas antimicrobianas e antibiofilmes a partir de extratos e frações vegetais. Inclui quatro etapas multidisciplinares: (1) coleta de material vegetal; (2) elaboração de extratos brutos (CE) e frações (CEF), seguidos de sua análise de perfil químico; (3) bioensaios; e (4) análises de dados biológicos e químicos(Figura 1). Assim, apresentamos o protocolo desenvolvido para analisar as atividades antimicrobianas e antibiofilmes de extratos e frações de Casearia sylvestris contra Streptococcus mutans e Candida albicans13,bem como os procedimentos para a caracterização fitoquímica e análise de dados. Para simplificar, o foco aqui é demonstrar a abordagem para a triagem de compostos naturais usando a bactéria.
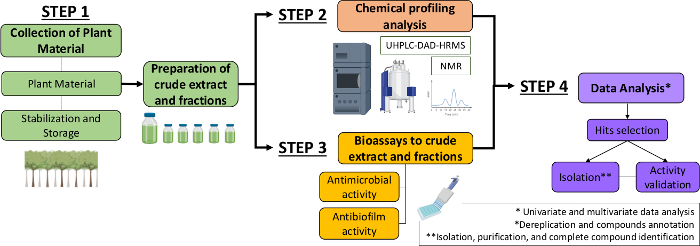
Figura 1: Fluxograma da Abordagem Sistemática para identificar moléculas ativas de extratos e frações de plantas. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Coleta de Material Vegetal
- Material vegetal
- Regissei o acesso ao material vegetal em plataformas eletrônicas que regulam o acesso ao patrimônio genético no país onde ocorrerá a coleta. Por exemplo, no Brasil, registre-se no Sistema Nacional de Gestão do Patrimônio Genético e do Conhecimento Tradicional Associado – SisGen (site https://sisgen.gov.br/paginas/login.aspx).
- Coletar amostras do material vegetal de interesse (por exemplo, folhas, caules, raízes, flores, frutas). Registre se o material foi coletado durante a fase reprodutiva ou vegetativa.
- Regissos parâmetros de coleta (data, georreferenciamento, temperatura média anual e percentual médio de umidade).
- Identifique as amostras com precisão, e um taxonomista deve confirmar a autenticidade.
- Estabilização e armazenamento de amostras de plantas
- Separe os órgãos vegetais em sacos plásticos individuais ou frascos imediatamente após as coletas.
- Inativar reações enzimáticas potenciais por (i) congelamento imediato em nitrogênio líquido, (ii) desidratação em um forno de ar circulante (40 °C), ou (iii) congelamento das amostras por liofilização.
- Armazene o material estabilizado em sacos hermeticamente lacrados à temperatura ambiente ou em um congelador até o uso (-20 ou -80 °C, dependendo do período de armazenamento ou do uso pretendido).
- Triture as amostras em um moinho analítico (faca ou bola, dependendo do tipo de tecido ou disponibilidade) e padronize o tamanho das partículas usando peneiras padronizadas.
- Pesar as amostras individualmente para as etapas de extração subsequentes.
2. Preparação de Extratos Brutos (CE) e Frações (CEF) para Análise de Perfil Químico e Bioensaões
- Elaboração de Extratos Brutos (CE)
NOTA: As etapas são ilustradas no fluxograma na Figura 2A.- Prepare um solvente de extração com uma mistura hidroalcoólica (por exemplo, etanol (EtOH) 70% ou misturas ternárias de água, EtOH e outros modificadores, definidos pelo desenho experimental de segundo relatórios anteriores).
- Use a razão de peso amostral (peso seco, mg)/ solvente de extração (mL) variando de 50 a 100 mgs para cada mL de solvente.
- Para extrações rápidas e reprodutíveis, use extrações em lote usando microtubos.
NOTA: Pelo menos três réplicas devem ser utilizadas neste momento para permitir a análise estatística. - Realize extrações assistidas por ultrassom (EAU) para torná-lo rápido, fácil e barato.
- Repita o procedimento três vezes (15 min cada) para obter a melhor eficiência.
- Após cada etapa de extração, decante o resíduo sólido por centrifugação e remova o sobrenante.
- Combine os supernantes individualmente, filtre e salve alíquotas para análise química simultânea e bioensações. Se necessário, remova o solvente extrato sob vácuo, fluxo de nitrogênio ou liofilização, e registre a pesagem e o rendimento.
- Armazene a -20 °C protegido contra a luz.
NOTA: O protocolo pode ser pausado aqui para tela do CE e selecionar os que apresentam a atividade desejada.
- Fracionamento de Extratos Brutos (CEF)
NOTA: As etapas são ilustradas no fluxograma na Figura 2B.- Use cartuchos com pelo menos 1 g de adsorbent. Se os alcaloides estiverem potencialmente presentes no CE, utilize solventes contendo 0,1% de ácido fórmico (FA).
- Diluir a amostra na mistura de solvente (ou solvente) mais adequada para obter uma solução de amostra de 100 mg/mL. Em seguida, transfira 1 mL da solução amostral para um cartucho de extração em fase sólida - SPE (1 g de adsorbent, 6 mL de capacidade).
- Realize o fracionamento utilizando cerca de três volumes mortos de cada eluente de extração (a um cartucho de 1 g, ele corresponderá a 2 mL de cada solvente). Se os alcaloides estiverem potencialmente presentes no CE, utilize solventes contendo 0,1% de FA.
- Colete uma fração por composição eluente e salve alíquotas para análise química simultânea e bioensações.
- Remova o solvente sob vácuo, fluxo de nitrogênio ou liofilização e registre a pesagem e o rendimento.
NOTA: Se o CE for difícil de dissolver na mistura inicial de elução, disperse o CE em uma fase sólida (por exemplo, C18 ou celite) em proporção de 1:1 (w/w) antes de carregar o material na parte superior do cartucho.
ATENÇÃO: Pesar os microtubos com antecedência para calcular o rendimento em massa de extratos e frações.
- Análise de perfil químico
NOTA: Considerando que cada espécie de planta requer métodos otimizados e específicos para sua análise química, nas seguintes seções, descrevemos as abordagens analíticas mais comuns utilizadas para analisar materiais vegetais. Como exemplo prático, foi desenvolvida e validada uma cromatografia líquida em fase inversa para a análise simultânea de compostos fenólicos e diterpenos do tipo clerodano, autenticadas diferenciadamente por duas variedades de Casearia sylvestris Swartz (Salicaceae). O aparelho UPLC-DAD foi equipado com um degasser, uma bomba quaternária, um sampler automático, um detector de matriz de fotodiodo UV-Vis e um forno (veja detalhes em Bueno et al. 201518). Abordagens semelhantes descritas nos exemplos a seguir podem ser otimizadas de acordo com outras espécies vegetais e/ou materiais vegetais.- Análise cromatográfica e possibilidades de hifenização
- Para separações utilizando cromatografia líquida de alto desempenho (UPLC), use uma coluna cromatográfica C18 (por exemplo, 150 × 2,1 mm, 2,6 μm, 100 Å) protegida por uma pré-coluna compatível.
NOTA: Outras fases de coluna ou modos cromatográficos podem ser usados dependendo da espécie/material da planta. O HPLC convencional também pode ser usado; Nesse caso, deve-se escolher uma coluna cromatográfica adequada. Para obter excelentes separações, ajuste as condições cromatográficas considerando a taxa de fluxo (μL/min), temperatura da coluna (°C) e volume de injeção (μL). A fase móvel geralmente consiste em água (A) e acetonitrilo ou metanol (B) usando gradientes de elução linear ou multicamada ou eluição isocrática. Modificadores como buffers, ácidos, bases ou outros também podem ser usados. - Realize a análise do material vegetal (CE e/ou CEF) e registre todos os dados relacionados, como dados espectrais (utilizando um UV-Vis e/ou, preferencialmente, detectores de MS), tempo de retenção (min) e outros, dependendo da hifenização disponível.
NOTA: A cromatografia líquida (LC) é geralmente hifenizada (acoplada) com espectrometria de massa de alta resolução (HRMS), como LC-HRMS, e é comumente usada para a rápida anotação de metabólito em CE ou CEF15. - Se forem necessários dados qualitativos e dados quantitativos, prepare-se cuidadosamente e injete curvas de calibração, seguindo o mesmo protocolo.
NOTA: O desenvolvimento das melhores condições cromatográficas pode ser realizado com o auxílio do desenho de experimentos, conforme descrito por Bueno et al. 201518, ou literatura semelhante. É importante considerar a inclusão de normas internas durante o desenvolvimento do método. Eles são muito apreciados, pois permitem desvios técnicos corretos durante a preparação e injeções da amostra, e uma maior normalização para análise de dados.
- Para separações utilizando cromatografia líquida de alto desempenho (UPLC), use uma coluna cromatográfica C18 (por exemplo, 150 × 2,1 mm, 2,6 μm, 100 Å) protegida por uma pré-coluna compatível.
- Análise cromatográfica e possibilidades de hifenização
- Análise de dados univariados e multivariados
- Exporte os cromatógramas registrados em formato adequado (por exemplo, ASCII, .txt. ou .csv formato). Uma única matriz de dados pode ser definida unindo e alinhando os cromatógrafos se várias amostras estiverem sendo analisadas, e comparações são necessárias. As matrizes resultantes devem ser normalizadas os cromatogramas de acordo com o padrão interno utilizado.
- Analise os dados de metabolômica da planta usando métodos multivariados e univariados. Explorar e visualizar conjuntos de dados metabolômicos através da análise simultânea de múltiplas variáveis utilizando métodos estatísticos multivariados, incluindo análise de componentes principais não supervisionadas (PCA) e análise hierárquica de clustering (HCA), ou análise de menos quadrados parciais supervisionados (como PLS, OPLS, PLS-DA). Métodos univariados, como ANOVA, Student's, Tukey e Welch's t-test, são especialmente interessantes para a análise precisa das diferenças quantitativas entre as amostras19.
- Anotação de dereplicação e compostos
NOTA: O objetivo desta etapa é dedicar-se à rápida identificação on-line de NPs conhecidos para evitar o isolamento tedioso que possa ser realizado simultaneamente à análise de dados uni ou multivariada.- Realize os níveis de identificação dos compostos detectados ou alvos:
- Composto identificado, incluindo estrutura 3D completa e estereoquímica (nível 0);
- Identificação alcançada por dois parâmetros ortogonais, como tempo de retenção e espectro MS/MS (nível 1);
- Compostos anotados e classes compostas (níveis 2 e 3);
- Metabólitos não identificados ou não classificados que podem ser diferenciados com base em dados analíticos (nível 4)19,20.
- Caracterizar os compostos conhecidos utilizando os bancos de dados comerciais ou públicos. Entre as bases de dados mais importantes, destacam-se: NIST (https://www.nist.gov), Wiley (https://www.sisweb.com/software/ms/wiley.htm), MassBank (https://massbank.eu/MassBank/), GMD (http://gmd.mpimp-golm.mpg.de/), METLIN (https://metlin.scripps.edu) e a Rede Molecular Social global de Produtos Naturais – Banco de Dados GNPS (https://gnps.ucsd.edu)19.
NOTA: Existem diferentes níveis de anotações e dependem da técnica hifenizada empregada durante o estudo e podem incluir: a assistência de bancos de dados espectrais baseados em MS- (ou NMR) e em algoritmos de previsão espectral silico. - Isolamento, purificação e identificação completa de compostos
NOTA: Se um determinado composto (cuja identidade foi suspeita pelos métodos estatísticos) precisa de identificação estrutural completa, o primeiro passo para realizar essa tarefa é isolar e purificar os compostos desejados em maior escala. Pode ser realizado ampliando os protocolos já desenvolvidos.- Realizar o isolamento rápido e direto do composto alvo (s) por técnicas cromatográficas preparatórias bem estabelecidas e otimizadas. O HPLC semi-preparatório com injeção de carga seca pode ser usado para evitar os compromissos que geralmente precisam ser feitos entre o carregamento elevado e a solubilização da amostra1.
- Realizar a caracterização estrutural completa e identificação de compostos isolados. Isso pode ser feito através da combinação de diferentes técnicas:
- Ressonância Magnética Nuclear (RMN);
- Espectrometria de massa (MS);
- Técnicas espectrométricas nas regiões ultravioleta (UV) e infravermelha (IR) também são muito úteis para a caracterização dos grupos funcionais;
- O uso de espectroscopia quiroptical como o dicroísmo circular eletrônico e vibracional (ECD e VCD, respectivamente), a atividade óptica de Raman (ROA) e a cristalografia de raios-X são técnicas importantes para caracterização de configuração absoluta.
- Realize os níveis de identificação dos compostos detectados ou alvos:
3. Bioensações
NOTA: Triagem biológica: Para avaliar rapidamente a bioatividade potencial da CE e da CEF, a triagem inicial de substâncias naturais deve ser organizada e direta.
- Preparação de CE e CEF para bioensa de ensaios
- Reconstitua a matéria seca com os melhores solventes possíveis (que podem ser determinados experimentalmente). O projeto experimental21,22 definem a solução de estoque e a concentração de solventes.
- Calcule a concentração de solventes da solução de estoque. Para isso, utilize a fórmula: C1 x V1 = C2 x V2, onde C1 representa a solução de estoque (mg) de CE e/ou CEF; V1 representa o volume do solvente; C2 é o peso do CE e/ou CEF; V2 é o volume final (mL) da solução de estoque.
NOTA: Por exemplo, selecionamos 84,15% EtOH e 15% de sulfóxido de dimetil (DMSO) como a concentração de solventes da solução de estoque. Preparamos a concentração de estoque do CE a 6 mg/mL e da CEF a 1 mg/mL13. O solvente para diluir o CE e a CEF dependerá do método de avaliação da atividade biológica. O solvente utilizado como veículo não deve interferir na atividade biológica e toxicológica. Normalmente, a água, DMSO, EtOH ou um solvente aquoso à base de EtOH é usado para solubilizar extratos de plantas ou derivados de plantas4,13.
- Preparação de organismos de teste
- Reativar uma cepa microbiana, por exemplo S. mutans UA159 em ágar de sangue (48 h, 37 °C, 5% CO2), e cultvá-lo em meio de cultura líquida (por exemplo, broto extrato de fônton-levedura [TYE: 2,5% (w/v) triptona com 1,5% (w/v) extrato de levedura] contendo 1% de glicose (w/v) (TYEg) para 16 h, 37° C, 5% CO2.
- Realizar uma diluição de 1:20 da cultura inicial do microrganismo no mesmo meio de cultura (a razão de diluição da cultura inicial pode mudar de acordo com o desenho experimental).
- Incubar até chegar à fase de crescimento do registro médio.
- Prepare o inóculo para bioensaudidos com uma população definida (por exemplo, 2x106 unidades formadoras de colônias por mililitro - CFU/mL) em TYEg para ensaios antimicrobianos e TYE com 1% de sacarose (w/v) (TYEs) para ensaios de biofilmes.
NOTA: As condições de crescimento dependerão do microrganismo testado.
- Atividade antimicrobiana
NOTA: As etapas são ilustradas na Figura 3.- Em uma placa de 96 poços, adicione uma alíquota (μL) da solução de estoque CE e/ou CEF (tratamentos). O volume da alíquota é definido pela concentração do teste, que deve ser selecionada com base em estudos anteriores. Por exemplo, para testar ce na concentração de teste de 0,5 mg/mL, use uma alíquota de 16,67 μL da solução de estoque a 6 mg/mL. Para este cálculo, use a fórmula: C1 x V1 = C2 x V2, onde C1 é a concentração de estoque, V1 é o volume da alíquota da solução de estoque, C2 é a concentração de teste, e V2 é o volume da placa de 96-well (que corresponde a 200 μL). Nesta condição experimental, a concentração de teste dos solventes (veículo) será de 7% EtOH e 1,25% DMSO.
- Incluir um conjunto de controles para cada placa: uma coluna com tratamentos, sem o inóculo (controle em branco por tratamento, ajuda a diferenciar a turbidez pelo tratamento utilizado a partir do crescimento microbiano); uma coluna com veículo e inóculo (diluído de controle CE ou CEF ou 0 mg/mL); uma coluna com apenas o meio de cultura (controle médio da cultura) e uma coluna com apenas inóculo (controle de crescimento microbiano).
- Usando TYEg, ajuste o volume para 100 μL. Em seguida, incubar, por exemplo, 24h, 37 °C, 5% de CO2 (dependendo do microrganismo testado).
- Inocular 100 μL de microrganismo inóculo (1x 106 CFU/mL) na placa de 96 poços.
- Analisar o crescimento bacteriano de acordo com a turbidez por inspeção visual dos poços (claro ou nublado). Claro: significa que não há crescimento visual do microrganismo. Nublado: significa que há crescimento visual do microrganismo.
- Medindo a absorvância (densidade óptica ou O.D.) da cultura bacteriana em cada poço (leitor ELISA usando 540 nm). Em seguida, transfira 100 μL das culturas para microtubos contendo 900 μL de solução salina (0,89% NaCl), misture bem por vórtice. Em seguida, continue realizando uma diluição serial dez vezes maior até o valor desejado.
- Inocular uma alíquota da diluição desejada em placas específicas de ágar (em duplicata). Por exemplo, 10 μL de uma diluição específica em placas de ágar de sangue.
- Incubar. As condições podem mudar entre microrganismos, por exemplo, S. mutans: 48 h, 37 °C, 5% CO2.
- Realizar colônia conta com as placas para posterior transformação em UFC/mL como (Umnúmero de colônias x 10n)/q. Nesta fórmula,n é igual ao valor absoluto da diluição (0, 1, 2 ou 3), e q é igual à quantidade, em mL, pipetada para cada diluição banhada na placa de ágar. Além disso, a CFU/mL pode ser convertida em valores de log.
NOTA: Quando os extratos vegetais são adicionados ao meio da cultura, pode ocorrer precipitação de partículas dos extratos. Esse fato pode dificultar a interpretação dos resultados. O mesmo ocorre quando um leitor de microplacândia mede a turbidez como, em alguns casos, as células se aglomeram na parte inferior da microplacão. Além disso, dependendo do extrato utilizado, a cor dos extratos de folhas vegetais pode dificultar a quantificação da turbidez23,24. Um método alternativo usa corantes que revelam se as células microbianas são metabolicamente ativas ou não24.
- Atividade antibiofilm
NOTA: As etapas para avaliar o efeito dos tratamentos na formação de biofilme são ilustradas na Figura 4.- Formação e processamento de biofilmes
- Tratamentos diluídos em meio de cultura (TYEs) em uma placa de 96 poços como descrito nas etapas do protocolo de Atividade Antimicrobiana.
- Incubar a placa. No exemplo com S. mutans, a incubação é realizada durante 24 h, a 37 °C, e 5% DE CO2.
- Após a incubação, coloque as placas em um agitador orbital (5 min, 37 °C, 75 rpm) para soltar as células não aderidas ao biofilme. Em seguida, descarte o meio de cultura contendo as células não aderidas e lave os biofilmes restantes três vezes com 0,89% de NaCl para remover células não aderentes.
- Quantificação da biomassa a partir de biofilmes tratados
NOTA: As etapas são ilustradas no fluxograma na Figura 4A.- Mantenha os biofilmes lavados na placa e adicione 50 μL de solução aquosa cristalina de 1% para cada poço.
- Incubar a placa em temperatura ambiente por 35 minutos.
- Lave os poços manchados com água MilliQ (três vezes) e depois seque-os por 60-90 min.
- Elute o violeta cristalina dos poços manchados com 200 μL de 99% EtOH incubando a placa em um agitador orbital (5 min, 37 °C, 75 rpm).
- Transfira uma alíquota de 150 μL de cada poço com o corante elucido para outra placa e quantifique a biomassa amostral (leitor ELISA 570 nm).
- Quantificação da população microbiana viável (UFC/mL) dos biofilmes tratados
NOTA: As etapas são ilustradas no fluxograma na Figura 4B.- Remova os biofilmes lavados da placa com uma pipeta e 200 μL de NaCl 0,89% e transfira a suspensão resultante individualmente para microtubos estéreis.
- Utilize um adicional de 200 μL de NaCl 0,89% por poço e transfira-o para o tubo correspondente, já contendo 200 μL da suspensão inicial do biofilme. Realize este processo até atingir uma suspensão total de 1 mL biofilm por poço original.
- Use uma alíquota de cada tubo para realizar uma diluição serial de dez vezes.
- Inocular uma alíquota da diluição desejada em placas específicas de ágar (em duplicata). Por exemplo, 10 μL de uma diluição específica em placas de ágar de sangue.
- Incubar placas de ágar (por exemplo, 48 h, 37 °C, 5% CO2), em seguida, contar as colônias para determinar a UFC/mL como descrito acima.
- Formação e processamento de biofilmes
- Fase de validação da atividade biológica
- Formação de pellicle salivar
- Use contas de hidroxiapatita (HA) (Macro-Prep Cerâmica Hydroxyapatite Tipo I 80 μm) como superfície para formar o filme salivar25. Essas contas superfamem esmalte dentário.
- Pesar as contas HA (por exemplo, 10 mg) em microtubos e esterilizar. Em seguida, use o buffer de adsorção (buffer AB: 50 mM KCl, 1 mM KPO4, 1 mM CaCl2, 1 mM MgCl2, em dd-H2O, pH 6.5]25 contendo 0,1 mM de fenilmtilasulfonil fluoreto (PMSF) e 0,02% de azida de sódio (NaN3) para lavar as contas.
- Coletar e preparar saliva humana26. É necessário ter aprovação do Comitê de Ética Institucional.
- Adicione 500 μL de saliva em microtubos e incubar (40 min, 37 °C, 24 rpm).
- Em seguida, remova a supernasa de saliva e lave as contas (três vezes com tampão AB contendo PMSF e NaN3). As contas sHA (contas HA com pellicle salivar) estão agora prontas para ensaios a jusante.
NOTA: A saliva é coletada de voluntários saudáveis. Após a coleta, dilui a saliva (1: 1 v/v) com tampão AB e centrífuga (1699 x g, 20 min, 4 °C). Esterilizar por filtração (filtro de membrana polietroésulfone com baixa ligação a 0,22 μm de proteínas)26. O Comitê de Ética Institucional deve aprovar o estudo. No nosso caso, o Comitê de Ética da Instituição aprovou o estudo (CAAE: 68161417.0.0000.5416).
- Desprendimento de S. mutans após adesão ao filme salivar e glucas tratados com extratos selecionados
- Cultive o microrganismo até a fase de crescimento do registro médio, conforme descrito acima.
- Quando as culturas atingiram o desejado O.D., centrífuga (4000 × g por 20 min), lave com solução de 0,89% NaCl e resuspenja a pelota com 0,89% NaCl utilizando o mesmo volume inicial do meio de cultura.
- Se usar um estreptococos, como S. mutans,sonicar as culturas com uma sonda para dechain (30 s, 7 W, três vezes). Se usar um único organismo celular, este passo pode ser ignorado.
- Verifique a O.D. (540 nm) para ajustar a concentração para 2 x 106 UFC/mL.
- Adesão de S. mutans à pellicle salivar (sHA) e desprendimento de células aderidas
NOTA: As etapas são ilustradas no fluxograma na Figura 5.- Obtenha as amostras de sHA conforme descrito acima.
- Adicione uma alíquota (por exemplo, adicionamos 500 μL) de tratamentos selecionados (na concentração de teste; por exemplo, 0,5 mg/mL) ou controles em microtubos contendo amostras de sHA.
- Incubar as amostras de sHA com tratamentos ou controles (30 min, 37 °C, 24 rpm); em seguida, lave as contas três vezes com tampão AB (contendo PMSF e NaN3).
- Adicione a cultura do microrganismo. No exemplo, adicionamos 500 μL de cultura S. mutans (2 x 106 UFC/mL) a cada microtubo.
- Incubar (1h, 37 °C, 24 rpm) e, em seguida, remover células sem saída lavando três vezes com tampão AB.
- Resuspend cada amostra com uma alíquota (no exemplo, adicionamos 1000 μL) de tampão AB e sonicato com uma sonda (30 s, 7 W).
- Use uma alíquota de cada suspensão para uma diluição serial de dez vezes para determinar o número de colônias viáveis, emplacando em placas específicas de ágar (48 h, 37 °C, 5% CO2). Em seguida, conte as colônias para determinar a UFC/mL como descrito acima.
NOTA: A etapa do sonicato é realizada para desacopr as células aderidas ao sHA.
- Adesão de S. mutans à matriz glucano inicial(gsHA) e desprendimento de células aderidas
NOTA: As etapas são ilustradas no fluxograma na Figura 6. A enzima GtfB foi purificada da cultura supernacante Streptococcus milleri KSB8 projetada para produzir GtfB. A purificação foi realizada com uma coluna de cromatografia contendo contas de hidroxiapatita utilizando tampões contendo dois inibidores de protease (0,1 mM PMSF e 0,02% NaN3)27,28. Em seguida, a enzima foi verificada em gel de acrilamida (SDS-PAGE) e manchada com nitrato de prata. As alíquotas da enzima foram armazenadas a -80 °C até o uso.- Obtenha as amostras de sHA conforme descrito acima. Em seguida, adicione uma alíquota (no exemplo, adicionamos 500 μL) de enzima GtfB a cada tubo e incubamos em um homogeneizador (40 min, 37 °C, 24 rpm). Em seguida, lave três vezes com tampão AB (contendo PMSF e NaN3).
- Adicione uma alíquota (por exemplo, 500 μL) de substrato de sacarose (100 mmol de sacarose) contendo os tratamentos (ou controles na concentração de teste, por exemplo, 0,5 mg/mL) a cada microtubo.
- Incubar as amostras em um homogeneizador (4h, 37 °C, 24 rpm). Em seguida, realize três lavagens com tampão AB (com PMSF e NaN3) para remover os tratamentos e o excesso de sacarose não incorporados nos glucas sintetizados (amostras de gsHA).
- Adicione uma alíquota (no exemplo, adicionamos 500 μL) de S. mutans inóculo (2 x 106 CFU/mL) a cada microtubo.
- Incubar em um homogeneizador (1 h, 37 °C, 24 rpm) e lavar três vezes com tampão AB (com PMSF e NaN3) para remover células sem saída.
- Resuspend cada amostra com uma alíquota (por exemplo, 1000 μL) de tampão AB (com PMSF e NaN3) e sonicato com uma sonda para desacoplar células aderidas a gsHA (30 s, 7 W).
- Use uma alíquota de cada suspensão para uma diluição serial de dez vezes para determinar o número de colônias viáveis, emplacando em placas específicas de ágar (48 h, 37 °C, 5% CO2). Em seguida, conte as colônias para determinar a UFC/mL como descrito acima.
- Formação de pellicle salivar
4. Análise de Dados Biológicos
- Bioensaensiagens Dados
- Insira dados brutos para os bioensadores em uma planilha. Calcule o registro da inibição do crescimento microbiano por cada tratamento como (UMAUFC/mL de tratamentos + 1) x log10. Em seguida, calcule a porcentagem de registro da inibição do crescimento microbiano, em comparação com o uso do controle do veículo (Alog10 CFU/mL de tratamento/média Alog10 CFU/mL de controle do veículo) x 100%.
- Correto O.D. de culturas planctônicas e biomassa tratadas por tratamentos (grupos tratados ce e CEF) e por controle veicular (controle negativo). Para correção, subtraia a absorção de poços tratados daquele obtido em poços contendo apenas meio de cultura (Ablank) como(Agrupos tratados médio /A meio de controle negativo ) x 100%.
- Após essa correção, calcule o percentual da inibição da biomassa, em comparação com o controle do veículo como(Biomassa tratada/controle médiode veículo A ) x 100%.
- Envie os dados brutos gerados para análise estatística dos dados utilizando software específico.
NOTA: A interpretação da eficácia de um determinado tratamento é determinada por meio de pontos de ruptura, como o IC50/IC90. Esses valores são definidos como a concentração mínima de um tratamento capaz de inibir 50% e 90%, respectivamente, de crescimento bacteriano ou formação de biofilme24. Esses parâmetros podem ajudar a interpretar os dados e fornecer uma base para a seleção de compostos com melhor atividade13,29.
Resultados
Fornecemos um exemplo de utilização de uma abordagem sistemática para triagem da atividade biológica de extratos e frações vegetais para identificar moléculas potencialmente ativas para possíveis novas terapias anticaspas: atividades antimicrobianas e antibiofilmas de extratos de Casearia sylvestris de biomas brasileiros distintos contra estreptococos mutadores e Candida albicans13.
Fundo
Interações complexas entr...
Discussão
Os principais desafios relacionados ao trabalho com extratos naturais brutos compreendem sua composição complexa e as inadequações de estudos clássicos de isolamento biogui guiado. Embora esse processo seja lento, é eficaz e levou a grandes descobertas na pesquisa de PN. Para racionalizar, são necessários estudos orientados à priorização para racionalizar. Assim, o uso de abordagens químicas modernas para análise de CE e desreplicação antes do isolamento são importantes para caracterizar o material estuda...
Divulgações
Nenhum conflito de interesses declarado.
Agradecimentos
Expressamos nossa gratidão ao Núcleo de Bioensaios, Biossíntese e Ecofisiologia de Produtos Naturais (NuBBE) do Instituto de Química da UNESP, Araraquara/SP, por fornecer os laboratórios para a preparação do material vegetal. Agradecemos também ao Laboratório de Microbiologia Aplicada do Departamento de Materiais Odontológicos e Prostodontia, UNESP, Araraquara/SP. Esta pesquisa foi apoiada por uma bolsa de pesquisa da Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP #2013/07600-3 à AJC) e bolsas de estudo (FAPESP #2017/07408-6 e FAPESP #2019/23175-7 à RMS; #2011/21440-3 e #2012/21921-4 para PCPB). O Conselho Nacional de Desenvolvimento Científico e Tecnológico em parceria com a FAPESP prestou apoio adicional (INCT CNPq #465637/2014-0 e FAPESP #2014/50926-0 à AJC).
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well microplates | Kasvi | Flat bottom | |
| Activated carbon | LABSYNTH | Clean up and/or fractionation step | |
| Analytical mill | Ika LabortechniK | Model A11 Basic | |
| Blood agar plates | Laborclin | ||
| Chromatographic column C18 | Phenomenex Kinetex | 150 × 2.1 mm, 2.6 µm, 100Â | |
| Dimethyl sulfoxide | Sigma-Aldrich | Vehicle solution | |
| ELISA plate reader | Biochrom Ez | ||
| Ethanol | J. T. Baker | For extraction and fractionation steps, and mobile phase composition | |
| Ethanol | Sigma-Aldrich | Vehicle solution | |
| Ethyl acetate | J. T. Baker | Fractionation step | |
| GraphPad Software | La Jolla | GraphPad Prism7 | |
| Hexane | J. T. Baker | Fractionation step | |
| Incubator | Thermo Scientific | ||
| Isopropanol | J. T. Baker | For extraction step | |
| Lyophilizer (a freeze dryer) | Savant | Modulyo | |
| Nylon Millipore | LAC | 0.22 µm x 13 mm | |
| Orbital shaker | Quimis | Model G816 M20 | |
| Polyamide solid phase extraction cartridge | Macherey-Nagel | Clean up and/or fractionation step | |
| Silica gel | Merck | 40–63 μm, 60 Â | |
| Sodium Chloride (NaCl) | Synth | 0,89% in water | |
| Solid phase extraction cartridges (SPE) | Macherey-Nagel | Clean up and/or fractionation step | |
| Tryptone | Difco | ||
| UHPLC-DAD | Dionex | Ultimate 3000 RS | |
| Ultrasonic bath | UNIQUE | Model USC 2800 | |
| Yeast extract | Difco |
Referências
- Newman, D. J., Cragg, G. M. Natural Products as Sources of New Drugs over the Nearly Four Decades from 01/1981 to 09/2019. Journal of Natural Products. 83 (3), 770-803 (2020).
- Wolfender, J. L., Litaudon, M., Touboul, D., Queiroz, E. F. Innovative omics-based approaches for prioritisation and targeted isolation of natural products – new strategies for drug discovery. Natural Product Report. 36 (6), 855-868 (2019).
- Michel, T., Halabalaki, M., Skaltsounis, A. New Concepts, Experimental Approaches, and Dereplication Strategies for the Discovery of Novel Phytoestrogens from Natural Sources. Planta Medica. 79 (7), 514-532 (2013).
- Jeon, J. G., Rosalen, P. L., Falsetta, M. L., Koo, H. Natural products in caries research: current (limited) knowledge, challenges and future perspective. Caries Research. 45 (3), 243-263 (2011).
- Tonetti, M. S., Jepsen, S., Jin, L., Otomo-Corgel, J. Impact of the global burden of periodontal diseases on health, nutrition and wellbeing of mankind: A call for global action. Journal of Clinical Periodontology. 44 (5), 456-462 (2017).
- Peres, M. A., et al. Oral diseases: a global public health challenge. Lancet. 394 (10194), 249-260 (2019).
- Bowen, W. H., Burne, R. A., Wu, H., Koo, H. Oral biofilms: pathogens, matrix, and polymicrobial interactions in microenvironments. Trends Microbiology. 26 (3), 229-242 (2018).
- Paes Leme, A. F., Koo, H., Bellato, C. M., Bedi, G., Cury, J. A. The role of sucrose in cariogenic dental biofilm formation--new insight. Journal of Dental Research. 85 (10), 878-887 (2006).
- Koo, H., Falsetta, M. L., Klein, M. I. The exopolysaccharide matrix: a virulence determinant of cariogenic biofilm. Journal of Dental Research. 92 (12), 1065-1073 (2013).
- Cury, J. A., de Oliveira, B. H., dos Santos, A. P., Tenuta, L. M. Are dental fluoride releasing materials clinically effective on caries control. Dental Materials. 32 (3), 323-333 (2016).
- Mattos-Graner, R. O., Klein, M. I., Smith, D. J. Lessons Learned from Clinical Studies: Roles of Mutans Streptococci in the Pathogenesis of Dental Caries. Current Oral Health Reports. 1, 70-78 (2014).
- Rocha, G. R., Florez Salamanca, E. J., de Barros, A. L., Lobo, C. I. V., Klein, M. I. Effect of tt-farnesol and myricetin on in vitro biofilm formed by Streptococcus mutans and Candida albicans. BMC Complementary and Alternative Medicine. 18 (1), 61 (2018).
- Ribeiro, S. M., et al. Antimicrobial and antibiofilm activities of Casearia sylvestris extracts from distinct Brazilian Biomes against Streptococcus mutans and Candida albicans. BMC Complementary and Alternative Medicine. 19 (1), 308 (2019).
- Pilon, A. C., et al. Metabolômica de plantas: métodos e desafios. Quimica Nova. 43 (3), 329-354 (2020).
- Wolfender, J. L., Nuzillard, J. M., Hooft, J. J. J., Renault, J. H., Bertrand, S. Accelerating Metabolite Identification in Natural Product Research: Toward an Ideal Combination of Liquid Chromatography-High-Resolution Tandem Mass Spectrometry and NMR Profiling, in Silico Databases, and Chemometrics. Analytical Chemistry. 91 (1), 704-742 (2019).
- Allard, P. M., et al. Pharmacognosy in the digital era: shifting to contextualized metabolomics. Current opinion in biotechnology. 54, 57-64 (2018).
- Hubert, J., Nuzillard, J., Renault, J. Dereplication strategies in natural product research: How many tools and methodologies behind the same concept. Phytochemistry Reviews. 16, 55-95 (2017).
- Bueno, P. C. P., Pereira, F. M. V., Torres, R. B., Cavalheiro, A. J. Development of a comprehensive method for analysing clerodane-type diterpenes and phenolic compounds from Casearia sylvestris Swartz (Salicaceae) based on ultra-high performance liquid chromatography combined with chemometric tools. Journal of separation science. 38 (10), 1649-1656 (2015).
- Bueno, P. C. P., Lopes, N. P. Metabolomics to Characterize Adaptive and Signaling Responses in Legume Crops under Abiotic Stresses. American Chemical Society omega. 5 (4), 1752-1763 (2020).
- Blaženović, I., Kind, T., Ji, J., Fiehn, O. Software tools and approaches for compound identification of LC-MS/MS data in metabolomics. Metabolites. 8 (2), 31 (2018).
- Eloff, J. N. Quantifying the bioactivity of plant extracts during screening and bioassay-guided fractionation. Phytomedicine: International Journal Of Phytotherapy And Phytopharmacology. 11 (4), 370-371 (2004).
- Rios, J. L., Recio, M. C. Medicinal plants and antimicrobial activity. Journal of Ethnopharmacology. 100 (1-2), 80-84 (2005).
- Eloff, J. N. A sensitive and quick microplate method to determine the minimal inhibitory concentration of plant extracts for bacteria. Planta Medica. 64, 711-714 (1998).
- Eloff, J. N. Avoiding pitfalls in determining antimicrobial activity of plant extracts and publishing the results. BMC Complementary and Alternative Medicine. 19 (1), 106 (2019).
- Klein, M. I., Xiao, J., Heydorn, A., Koo, H. An analytical tool-box for comprehensive biochemical, structural and transcriptome evaluation of oral biofilms mediated by mutans streptococci. Journal of Visualized Experiments. (47), e2512 (2011).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to study the physiology of oral biofilms. Methods in molecular biology. 666, 87-102 (2010).
- Venkitaraman, A. R., Vacca-Smith, A. M., Kopec, L. K., Bowen, W. H. Characterization of glucosyltransferase B, GtfC, and GtfD in solution and on the surface of hydroxyapatite. Journal of Dental Research. 74, 1695-1701 (1995).
- Vacca-Smith, A. M., Venkitaraman, A. R., Quivey, R. G., Bowen, W. H. Interactions of streptococcal glucosyltransferases with alpha-amylase and starch on the surface of saliva-coated hydroxyapatite. Archives of Oral Biology. 41, 291-298 (1996).
- Van Dijck, P., et al. Methodologies for in vitro and in vivo evaluation of efficacy of antifungal and antibiofilm agents and surface coatings against fungal biofilms. Microbial Cell. 5 (7), 300-326 (2018).
- Marsh, P. D. Are dental diseases examples of ecological catastrophes. Microbiology. 149 (2), 279-294 (2003).
- Bowen, W. H., Koo, H. Biology of Streptococcus mutans-derived glucosyltransferases: role in extracellular matrix formation of cariogenic biofilms. Caries Research. 45 (1), 69-86 (2011).
- Lobo, C. I. V., et al. Dual-species biofilms of Streptococcus mutans and Candida albicans exhibit more biomass and are mutually beneficial compared with single-species biofilms. Journal of Oral Microbioly. 11 (1), 1581520 (2019).
- Kim, D., et al. Candida albicans stimulates Streptococcus mutans microcolony development via crosskingdom biofilm-derived metabolites. Scientific reports. 7, 41332 (2017).
- Ferreira, P. M. Folk uses and pharmacological properties of Casearia sylvestris: a medicinal review. Anais da Academia Brasileira de Ciencias. 83 (4), 1373-1384 (2011).
- Xia, L., Guo, Q., Tu, P., Chai, X. The genus Casearia: a phytochemical and pharmacological overview. Phytochemistry Reviews. 14, 99-135 (2015).
- Ferreira, P. M. P., et al. Toxicological findings about an anticancer fraction with casearins described by traditional and alternative techniques as support to the Brazilian Unified Health System (SUS). Journal of Ethnopharmacol. 15, 241 (2019).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. Journal of Bacteriology. 192 (12), 3024-3032 (2010).
- Maske, T. T., van de Sande, F. H., Arthur, R. A., Huysmans, M. -. C. D. N. J. M., Cenci, M. S. In vitro biofilm models to study dental caries: a systematic review. Biofouling. 33 (8), 661-675 (2017).
- Fu, Y., Luo, J., Qin, J., Yang, M. Screening techniques for the identification of bioactive compounds in natural products. Journal of Pharmaceutical and Biomedical Analysis. 168, 189-200 (2019).
- Sarker, S. D., Nahar, L. An introduction to natural products isolation. Methods in molecular biology. 864, 1-25 (2012).
- Clinical Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptibility testing; twenty-fifth informational supplement. Clinical Laboratory Standards Institute (CLSI). , (2015).
- Saputo, S., Faustoferri, R. C., Quivey, R. G. A drug repositioning approach reveals that Streptococcus mutans is susceptible to a diverse range of established antimicrobials and nonantibiotics. Antimicrobial Agents and Chemotherapy. 62 (1), 01674 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados