需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
单细胞阵列的活细胞成像 (LISCA) - 量化细胞动力学的多功能技术
摘要
我们提出了一种利用微模式阵列从单个细胞获取荧光记者时间课程的方法。该协议描述了单细胞阵列的准备、活细胞扫描延时显微镜的设置和操作,以及用于自动预选、可视控制和跟踪每个粘附站点的细胞集成荧光时间课程的开源图像分析工具。
摘要
单细胞阵列的活细胞成像 (LISCA) 是一种多功能方法,可收集来自高吞吐量单个细胞的荧光信号的时间过程。一般来说,从培养细胞获得单细胞时间课程受到细胞移动性和细胞形状多样性的阻碍。粘合微阵列标准化单细胞条件,便于图像分析。LISCA 将单细胞微阵列与扫描延时显微镜和自动图像处理相结合。在这里,我们描述了以 LISCA 格式参加单细胞荧光时间课程的实验步骤。我们通过 mRNA 编码对增强型绿色荧光蛋白 (eGFP) 进行微模式阵列的传输,并通过扫描延时显微镜同步监测数百个细胞的 eGFP 表达动力学。图像数据堆栈由新开发的软件自动处理,该软件集成了选定细胞轮廓上的荧光强度,以生成单细胞荧光时间过程。我们演示了 mRNA 转染后的 eGFP 表达时间过程,通过一个简单的动能转换模型很好地描述了 mRNA 的表达和退化率。讨论了 LISCA 在信号凋亡背景下多个标记事件时间相关性进一步应用。
引言
近年来,单细胞实验的重要性日益显现。来自单个细胞的数据允许研究细胞对细胞的变异性,细胞内参数相关性的分辨率和细胞动力学的检测,这些都隐藏在合奏测量1、2、3中。为了同时研究数千个单个细胞的细胞动力学,需要采用新的方法,在长达数小时、持续数天的标准化条件下对细胞进行监测,然后进行定量数据分析4。在这里,我们介绍单细胞阵列的活细胞成像 (LISCA),它结合了微结构阵列的使用与延时显微镜和自动图像分析。
在文献5、6中,已经建立并出版了几种产生微结构单细胞阵列的方法。在这里,我们简要地描述了微尺度等离子启动蛋白模式 (μPIPP)。参考文献7中还发现了使用μPIPP制造的单细胞阵列制造的详细协议。单细胞阵列的使用使数千个细胞对齐标准化粘附点,为每个细胞呈现定义的微环境,从而减少实验变异性来源(图1A)。单细胞阵列用于监控荧光标记的时间过程,这些标记旨在指示各种蜂窝过程。扫描延时模式的长期显微镜允许监测大面积的单细胞阵列,从而在几个小时甚至几天的观测时间内以高通量采样单细胞数据。这会生成来自阵列每个位置的时线图像堆栈(图1B)。为了减少大量的图像数据,并有效地提取所需的单细胞荧光时间课程,需要利用细胞定位(图1C)进行自动图像处理。
LISCA 面临的挑战是调整实验协议和计算工具,形成高通量检测,生成蜂窝动力学的定量和可重复数据。在本文中,我们逐步描述了各个方法以及它们如何在 LISCA 分析中组合。例如,我们讨论了人工mRNA传递后增强绿色荧光蛋白(eGFP)表达的时间过程。mRNA交付后的eGFP表达通过mRNA的转化和降解反应速率方程来描述。将 eGFP 浓度的时间过程的模型功能与 LISCA 读出每个细胞的荧光强度(随着时间的推移)相适应,可对 mRNA 退化率等模型参数进行最佳估计。作为一个具有代表性的结果,我们讨论了两种不同脂基转染剂的 mRNA 输送效率及其参数分布的差异。
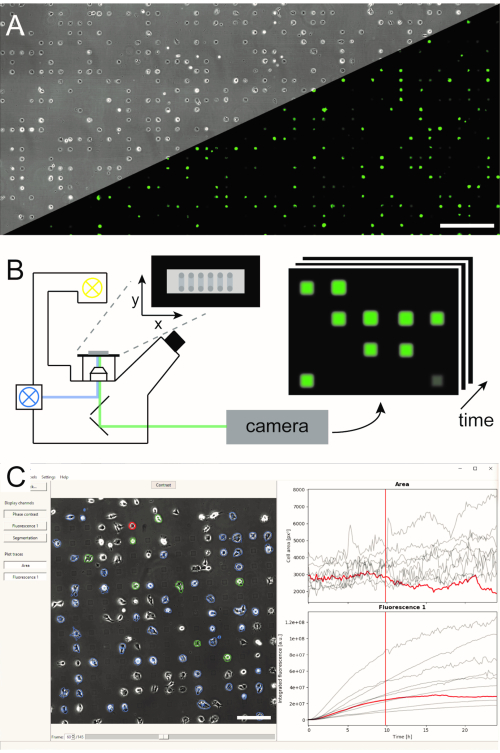
图1:LISCA 工作流程的表示,结合 (A) 微模式单细胞阵列 (B) 扫描延时显微镜和 (C) 记录图像系列的自动图像分析。单细胞阵列由细胞粘合方块的二维模式组成,具有细胞排斥的间空,可在微观图案上排列细胞,如相对比图像以及 eGFP 表达细胞(A)的荧光图像。整个微结构区域以扫描延时模式进行成像,在一系列位置(B) 中反复拍摄图像。记录的图像系列经过处理,以读出每个细胞的荧光强度(C)。比例杆:500微米(A),200微米(C)。请单击此处查看此图的较大版本。
研究方案
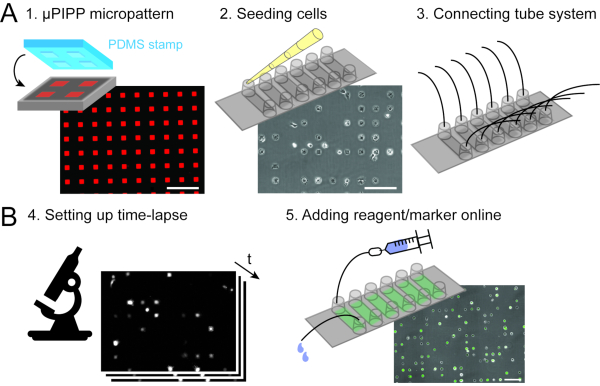
图2:将单细胞微阵拉 (A) 与扫描延时显微镜 (B) 相结合的数据采集。作为延时实验的准备,准备了带有 2D 微模式粘附方块的单细胞阵列 (1),然后是细胞播种和微模式 (2) 上细胞的对齐,以及灌注系统与六通道滑梯的连接,从而在延时测量 (3) 期间实现液体处理。在延时实验 (5) 期间,通过灌注系统注入 mRNA 脂质溶液,在显微镜上对细胞进行微镜传输扫描延时实验 (4)。比例栏: 200μm.请单击此处查看此图的更大版本。
1. 微结构单细胞阵列制造 (图2A)
- 准备 μPIPP 阵列制造所需的材料。
- 在 pH 7.4 中准备无菌磷酸盐缓冲盐水 (PBS)。
- 在 25 °C 下准备电阻度至少为 18 MΩcm 的无菌超纯水。
- 准备PLL(20 kDa)-g[3.5]PEG(2 kDa)(PLL-PEG)工作解决方案,在含有150毫克纳克和10m 4-(2-羟基)-1-管道氨基硫酸(HEPES)的超纯水中,PLL-PEG浓度为2毫克/mL。
- 准备细胞外基质蛋白溶液用于表面涂层:PBS 中的 1 毫克/mL 纤维素 (FN)。
- 用光刻8 制作的微型制图准备硅晶片,该晶片可作为可重复使用的主。微型图由边缘长度为 30μm 的正方形组成,深度为 12μm,方形间距离为 60 μm,按六条条纹排列,每个条纹宽度为 6 mm,高度为 18 mm。
- 将聚二甲基硅氧烷 (PDMS) 单体与 9% 的交联器(质量%) 混合使用硅胶弹性体套件,并将其除气约30分钟,直到它是无气泡使用干燥器。将硅晶片与大约 3-5 毫米厚的 PDMS 层一起铸造,再除气约 30 分钟,直到无气泡。
- 将带有 PDMS 的硅晶片放入 50 °C 的烘烤炉中,以至少 4 小时治愈 PDMS。
- 剪下 PDMS 邮票。
- 使用手术刀,并切出 PDMS 第一层 PDMS 杰作,其中包含六个微模式条纹。
- 将 PDMS 杰作放在长凳上,微型模式朝上。
- 将 PDMS 杰作的六个微模式条纹中的每一个都切成 PDMS 邮票。请注意,PDMS 邮票的边缘通过切断某些图案区域而打开。
- 将 PDMS 邮票放在六通道幻灯片的盖片上(图 3-1)。
- 使用未涂层的盖片,仔细抓起盖片的保护箔,标记六通道滑梯的通道位置。然后将盖片放在长凳上,保护箔朝下。
- 使用钳子将 PDMS 邮票与微模式向下朝下放置在标记通道位置的盖片上。
- 检查显微镜下 PDMS 邮票的附件。如果 PDMS 印章完全连接到盖唇上,则接触的正方形看起来比间空暗。PDMS 邮票与盖子的附件对于微型模式质量至关重要。
- 将盖片上印有六枚 PDMS 邮票放入等离子清洁剂中,用氧等离子体(压力 0.2 mbar,约 40 W 3 分钟)处理,使 PDMS 邮票和盖片亲水(图 3-2)之间的表面。
- 在生物安全柜中执行微模式制造的所有进一步步骤。使用 15 μL 的 PLL-PEG 解决方案,并在每个 PDMS 邮票旁边一滴移液,以便将 PLL-PEG 解决方案吸收到 PDMS 邮票的亲水模式中(图 3-3)。让PLL-PEG在室温下孵育20分钟。
- 在盖片上冲洗1mL的超纯水,上面印有PDMS邮票,并使用钳子取出PDMS邮票(图3-4)。然后用 1 mL 的超纯水第二次冲洗盖滑,使其干燥。
- 当盖片完全干燥时,将六通道粘滑动粘在盖片上(图3-4)。注意微模式区域与通道底部对齐。
- 使粘附方块与 FN 功能化。
- 将 40μL 的 PBS 填充到每个通道中。
- 在 PBS 中准备 100μg/mL FN 解决方案。
- 向每个通道添加 40 μL 的 FN 解决方案(图 3-5)。通过从一个储液库中取出 40 μL 并将其添加到同一通道的对面储液库 3 次,将 FN 解决方案与通道中的 PBS 彻底混合,从而生成同质解决方案。在室温下孵化 FN 解决方案 45 分钟。
- 用 120μL 的 PBS(图 3 -6)清洗每个通道三次。
- 为了检查图案质量,请使用步骤 9.2 中标有荧光标记的 FN。(图4A) 。
注意:我们建议在细胞播种前不超过一天准备 μPIPP 阵列,因为 PLL-PEG 和 FN 与基板没有共效性,并且模式的质量可能会随着时间的推移而降低。将准备好的μPIPP阵列存放在冰箱中。
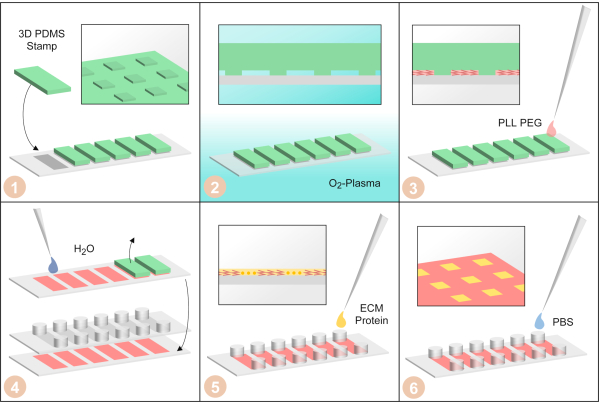
图3:由μPIPP制作的单细胞微阵列(1)表面有三维微模式结构的PDMS邮票被安排在六通道幻灯片的盖片上。(2) 盖上 PDMS 印章的盖片用氧浆处理,使表面亲水。(3) 添加 PLL-PEG。它被毛细管力吸收到微观结构中,使表面不被PDMS标记细胞排斥剂所覆盖。(4) 盖片用水冲洗,以去除剩余的 PLL-PEG。然后,PDMS 邮票被移除,六通道粘滑梯卡在盖子上。(5) 添加细胞外基质的蛋白质纤维素,使这些区域无PLL-PEG细胞粘合剂。(6) 六通道滑梯用磷酸盐缓冲盐水清洗。请单击此处查看此图的较大版本。
2. 细胞播种 (图2A)
注意:对于以下洗涤步骤,将相应的液体添加到一个储液库中,然后从通道的对面储液库中去除等量的液体。
- 用 37 °C 的 120 μL 完全补充细胞生长介质清洗每个通道。在添加单元格悬架之前,确保只有通道中填充,而不是储液层。
- 按照细胞传递的标准协议,将 HuH7 细胞从细胞培养瓶中分离,并将细胞悬浮浓度调整为 4 x 105 细胞/mL。
- 加入 40 μL 的细胞悬浮液,将细胞生长介质与细胞悬浮混合,从一个储液库中取出 40 μL,并将其添加到同一通道的对面储液层中 3 次,以达到同质细胞分布(图 4B)。
- 从通道中删除 40 μL 的悬架,以便只有通道充满细胞悬架。
- 使用相对比显微镜播种后,将幻灯片放入孵化器中,并检查细胞粘附 1 小时。
- 添加 120 μL 的 37 °C 暖细胞生长介质。
- 但滑回孵化器中3小时,使细胞在微观模式(图4C)上实现自我组织。

图4:μPIPP 阵列的细胞自我组织和质量控制。 (A) 微结构表面由方形 FN 涂层粘附点组成,红色被细胞排斥聚合物包围。(B) 细胞播种后,HuH7细胞随机分布,(C) 在4小时的时间内主要粘附在粘附点上。转载许可7.比例栏: 200μm.请单击此处查看此图的更大版本。
3. 灌注系统 (图2A)
注意:只有在延时测量过程中需要添加试剂或荧光标记时,才需要使用灌注系统。根据您的需求,您可以将每个通道连接到一个单独的灌注系统,或将多个系列通道连接到同一个灌注系统。灌注系统的数量与独立实验条件的数量相对应。将无菌条件下的管子连接到生物安全柜中,避免在灌注系统中加入气泡。如果不使用灌注系统,请在延时测量前将试剂/标记添加到生物安全柜中。灌注系统是内部制造的,用过的材料列在材料表中。灌注系统的组装已经描述过以前9。
- 使用 1 mL 注射器(更换孢子),用 1 mL 的 37 °C 细胞生长介质填充注射器。
- 使用阀门将注射器连接到入口管,并用介质填充管。
- 将入口管连接到通道的储液池,确保没有气泡被夹住。
- 要将另一个系列通道连接到此灌注系统,将串行连接器连接到当前通道入口管对面的储液库。转到下一个通道,并将串行连接器的免费端连接到其储液库之一。
- 重复以前的步骤,直到所需的频道数以系列方式连接。
- 将出水管直接连接到当前通道的免费储液池。用介质填充连接的管子,以检查灌注系统是否漏水。
- 重复以前的步骤,直到幻灯片的所有六个通道连接到灌注系统。
- 将带连接的灌注系统的滑梯放回孵化器中,直到进一步使用或直接将其放置在显微镜预热至 37 °C 的加热室中,以便进行延时测量。
4. 延时显微镜 (图2B)
注意:对于长期测量,保持 37 °C 的稳定温度和稳定的 CO2 水平。作为 CO2依赖细胞生长介质的替代品,使用 L15 介质,无需气体孵化系统。
注意:对于定量成像,在延时测量期间使用无酚红色的细胞生长介质来减少背景荧光,并使用延时协议的相同设置以及相同的显微镜进行技术复制。
- 设置延时协议,使用 10 倍目标和适当的荧光滤光过滤器记录相对比图像和荧光图像,曝光时间为 750 ms(取决于相机),连续循环通过位置列表之间的 10 分钟时间间隔,以及 30 h 的观测时间。
- 将带单元的六通道滑梯放在 37 °C 暖加热室的样品架的单细胞阵列上。如果灌注系统连接到六通道滑梯,请使用一些胶带将管子固定到舞台上,以确保在液体交换过程中不会移动六通道滑梯。通过 15 mL 反应管的孔插入出口管的免费端以收集液体废物。
- 设置扫描延时测量的位置列表。确保在通过位置列表的连续循环之间的定义时间间隔内扫描职位数量。每个通道的定位为 10 倍,可根据相机芯片大小设置为扫描整个微模式区域。
- 开始延时测量。为了提高长期测量的图像质量,请使用自动对焦校正系统。
5. 荧光标记 - mRNA 转染 (图 2B)
注:对于通过管道系统连接的两个通道的传输,需要总容量为 600 μL 的传输组合(一个通道为 300 μL)。指示的卷是指两个连接通道中的转染。
- 通过在 200 μL 的血清减少介质中稀释 1μL 的转染剂来准备转染剂解决方案,让溶液在室温下孵育 5 分钟。
- 通过在 150 μL 的血清减少介质中稀释 300 ng 的 mRNA 编码来准备 mRNA 解决方案。
- 通过在 mRNA 解决方案中添加 150 μL 的转染剂溶液并将其很好地混合,来准备转染混合物。让转染混合物在室温下孵育20分钟。
- 在输血混合物的孵化过程中,使用注射器使用 1 mL 的 37 °C 暖 PBS 冲洗管系统。冲洗管子时,确保显微镜阶段不会移动。如有必要,暂停延时测量。
- 通过添加 300 μL 血清减少介质,将转染混合物稀释到 0.5 ng/μL 的最终 mRNA 浓度。
- 使用注射器使用转染混合冲洗管系统,让 mRNA 脂质孵育 1 小时(如有必要,暂停延时测量)。
- 停止转染孵化,用 1 mL 的 37 °C 暖全补充细胞生长介质清洗,冲洗未绑定的 mRNA 脂质(如有必要,暂停延时测量)。
6. 图像分析和荧光读出
- 首次运行图像分析时,根据所提供的说明,从引用的位置 10 处安装开源软件"Python 中的自动微结构分析"(PyAMA) 的版本0.1.6。
- 确保图像通道(相对比度和荧光)可作为多图像 16 位 TIFF 文件提供。如有必要,相应地转换它们。
- 启动PyAMA并单击打开堆栈。。。
- 要打开每个多图像 TIFF 文件,请单击"打开"并选择文件,以便将其显示在对话框左侧加载的文件列表中(图 5-1)。
- 标记分析中要包含的通道。对于每个通道,执行以下步骤。
- 在包含通道的 TIFF 文件加载文件列表中进行选择。
- 在添加 新通道的部分中,选择 TIFF 文件中的通道索引。索引是零基的:第一个通道有指数 0,第二个通道有索引 1 等。
- 选择通道类型。选择 相位对比 度或 荧光 为相应的图像通道和 细分 的二进制通道指示细胞轮廓。
- 可选地输入区分不同荧光通道的通道标签:eGFP 和 DAPI。
- 配置频道后,单击 "添加"。
- 当所有添加的通道都显示在对话框右侧的通道列表中时,单击 "确定 "以加载堆栈。
- 要使用 PyAMA 的内置分割算法执行基于相对比图像(图 5 -2)的单元识别,请转到工具|二元化...并输入带有双化通道的NumPy文件的文件名称。
注意:在当前版本中,加载二进制通道需要重新加载所有通道。 - 要在荧光通道(图 5-3)上执行背景校正11,请确保加载荧光通道和细分通道。如果没有加载分割通道,请确保加载相对比通道进行自动分割。转到"工具>背景校正。。。"并选择一个文件名称,为由此产生的TIFF文件与更正的荧光通道。
注意:在当前版本中,加载后台校正通道需要重新加载所有通道。 - 通过滚动浏览时间框架、查看左侧通道菜单中列出的通道并单击单元以突出显示其荧光时间课程(图 1C),检查预先选定的单元(图 5-4)及其集成荧光信号(图5 -5)。通过进一步分析,使用细胞选择排除不可行的细胞,不局限于粘附点或连接到其他细胞的细胞。通过按下"移位"和单击单元格,或通过突出显示单元格并按"输入"来切换单元格的读取选择。
- 通过单击文件|,为细胞区域和集成荧光(图 5-6)保存单细胞时间课程 保存 并选择要保存到的目录。

图5:使用PyAMA自动处理延时图像系列。(1)每个成像位置的相对比度和荧光图像系列导入。(2) 细胞轮廓由相对比图像堆栈上的细分决定。(3) 荧光图像应用背景校正。(4) 细胞轮廓随时间而跟踪,并预选为导出。(5) 荧光强度根据跟踪的细胞轮廓进行集成。(6) 评估单细胞细胞区域和综合荧光强化度,并输出每个细胞的时间课程。比例栏: 100μm.请单击此处查看此图的更大版本。
- 要分析 mRNA 转染后的翻译动力学,请将基于生化速率方程的翻译模型与 Reiser 等人之前描述的每个单细胞时间过程进行转换。该研究中使用的数据和代码是公开的13。
- 对于每个单单元格时间过程,检索代表 mRNA 退化率和翻译发病时间点的翻译模型的估计拟合参数。示例数据集在代表性结果部分进行讨论。
- 进一步分析不同实验条件参数的最佳估计值的分布,以调查细胞群中的细胞对细胞的变异性。
结果
LISCA 方法能够有效地从单个细胞中收集荧光时间课程。作为一个具有代表性的例子,我们概述了如何应用 LISCA 方法测量转染后的单细胞 eGFP 表达。LISCA实验的数据用于评估mRNA输送动力学,这对高效mRNA药物的开发非常重要。
我们特别展示了两个基于脂质的 mRNA 递送系统对单细胞水平的翻译发病时间点和表达速率的不同影响。我们培养细胞,并将该批次分成两个群体。一个亚人...
讨论
在这里,我们描述 LISCA 是一种多功能技术,在单细胞水平上遵循细胞内荧光标签的细胞动力学。为了执行成功的 LISCA 实验,协议部分的每个描述步骤必须单独确定,然后必须合并所有步骤。LISCA 的三个主要方面都具有关键步骤。
单细胞微阵拉制造
微传讯的质量至关重要,因为微传管上的细胞对齐不仅对所有进一步的实验步骤都很重要,而且对数据质量也有影响。?...
披露声明
作者宣称他们没有相互竞争的经济利益。
致谢
这项工作得到了德国科学基金会(DFG)向合作研究中心(SFB)1032的赠款的支持。德国联邦教育、研究和技术部 (BMBF) 根据合作项目 05K2018-2017-06716 美帝索夫的支持以及巴耶里舍·福松施蒂夫通的赠款,都表示感谢。安妮塔·赖瑟通过慕尼黑定量生物科学研究生院(QBM)得到了DFG奖学金的支持。
材料
| Name | Company | Catalog Number | Comments |
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
参考文献
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: "Correlation of mRNA delivery timing and protein expression in lipid-based transfection". Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。