É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem de células vivas de matrizes unicelulares (LISCA) - uma técnica versátil para quantificar cinética celular
Neste Artigo
Resumo
Apresentamos um método para a aquisição de cursos de tempo de repórter fluorescência de células únicas usando matrizes micropatteradas. O protocolo descreve a preparação de matrizes unicelulares, a configuração e o funcionamento de microscopia de lapso de tempo de varredura de células vivas e uma ferramenta de análise de imagem de código aberto para pré-seleção automatizada, controle visual e rastreamento de cursos de tempo de fluorescência integrados por células por local de adesão.
Resumo
A imagem de células vivas de Matrizes Unicelulares (LISCA) é um método versátil para coletar cursos de tempo de sinais de fluorescência de células individuais em alta produtividade. Em geral, a aquisição de cursos de tempo unicelulares de células cultivadas é dificultada pela motilidade celular e diversidade de formas celulares. Micro arrays adesivos padronizam as condições unicelulares e facilitam a análise de imagens. O LISCA combina microarrays unicelulares com microscopia de lapso de tempo de varredura e processamento automatizado de imagens. Aqui, descrevemos os passos experimentais de fazer cursos de tempo de fluorescência unicelulares em um formato LISCA. Transfectamos células aderentes a uma matriz micropatterada usando codificação mRNA para proteína fluorescente verde aprimorada (eGFP) e monitoramos a cinética de expressão eGFP de centenas de células em paralelo através da microscopia de lapso de tempo de varredura. As pilhas de dados de imagem são automaticamente processadas por um software recém-desenvolvido que integra a intensidade da fluorescência sobre contornos celulares selecionados para gerar cursos de tempo de fluorescência unicelulares. Demonstramos que os cursos de tempo de expressão do eGFP após a transfecção do mRNA são bem descritos por um simples modelo de tradução cinética que revela taxas de expressão e degradação do mRNA. Outras aplicações do LISCA para correlações de tempo de evento de múltiplos marcadores no contexto de sinalização da apoptose são discutidas.
Introdução
Nos últimos anos, a importância de experimentos unicelulares tornou-se aparente. Os dados de células únicas permitem a investigação da variabilidade celular-célula, a resolução de correlações de parâmetros intracelulares e a detecção de cinéticas celulares que permanecem escondidas nas medições do conjunto1,2,3. Para investigar cinética celular de milhares de células únicas em paralelo, são necessárias novas abordagens que permitam monitorar as células em condições padronizadas durante um período de várias horas até vários dias seguido de uma análise quantitativa de dados 4. Aqui, apresentamos imagens de células vivas de matrizes unicelulares (LISCA), que combina o uso de matrizes microestruturadas com microscopia de lapso de tempo e análise automatizada de imagens.
Vários métodos de geração de matrizes unicelulares microestruturadas foram estabelecidos e publicados na literatura5,6. Aqui, descrevemos brevemente a padronização de proteína iniciada por plasma de microescala (μPIPP). Um protocolo detalhado da fabricação de matriz unicelular usando μPIPP também é encontrado na referência7. O uso de matrizes unicelulares permite o alinhamento de milhares de células em pontos de adesão padronizados apresentando microambientes definidos para cada célula e, assim, reduz uma fonte de variabilidade experimental(Figura 1A). Matrizes unicelulares são usadas para monitorar os cursos de tempo de marcadores fluorescentes com propósito para indicar uma variedade de processos celulares. A microscopia de longo prazo no modo de verificação de lapso de tempo permite monitorar uma grande área das matrizes unicelulares e, portanto, amostrar dados unicelulares em alta taxa de distribuição durante um tempo de observação de várias horas ou até dias. Isso gera pilhas de imagens de linha de tempo de cada posição da matriz(Figura 1B). Para reduzir a grande quantidade de dados de imagem e extrair os cursos de tempo de fluorescência unicelular desejados de forma eficiente, é necessário um processamento automatizado de imagens que aproveita o posicionamento das células(Figura 1C).
O desafio do LISCA é adaptar os protocolos experimentais e ferramentas computacionais para formar um ensaio de alto rendimento que gere dados quantitativos e reprodutíveis de cinética celular. Neste artigo, fornecemos uma descrição passo a passo dos métodos individuais e como eles são combinados em um ensaio LISCA. Como exemplo, discutimos o curso temporal da expressão aprimorada de proteína fluorescente verde (eGFP) após a entrega artificial de mRNA. A expressão eGFP após a entrega do mRNA é descrita por equações de taxa de reação modelando tradução e degradação do mRNA. A montagem da função do modelo para o curso de tempo da concentração eGFP à leitura LISCA da intensidade de fluorescência para cada célula individual ao longo do tempo produz melhores estimativas de parâmetros de modelo, como a taxa de degradação do mRNA. Como resultado representativo, discutimos a eficiência de entrega de mRNA de dois diferentes agentes de transfecção à base de lipídios e como suas distribuições de parâmetros diferem.
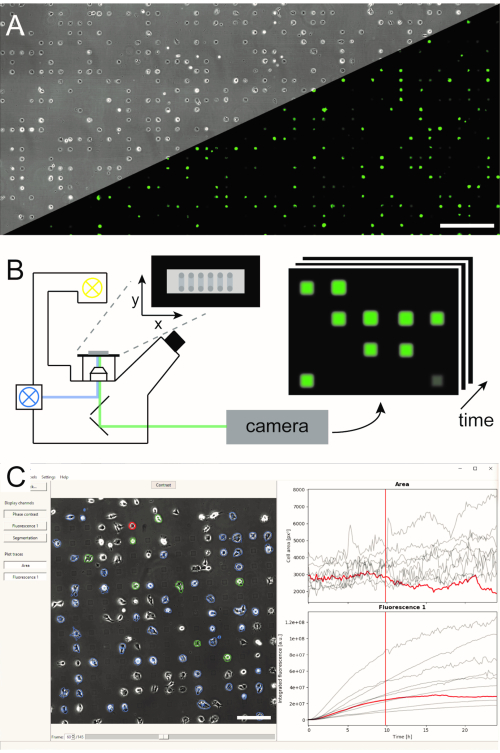
Figura 1: Representação do fluxo de trabalho LISCA que combina (A) matrizes unicelulares (B) micro-padronizadas de micro-padrão e análise automatizada de imagens (C) da série de imagens gravadas. As matrizes unicelulares consistem em um padrão bidimensional de quadrados adesivos celulares com um interespaço repelente celular levando a um arranjo das células no micropattern, como pode ser visto na imagem de contraste de fase, bem como na imagem de fluorescência das células expressas eGFP(A). Toda a área microestruturada é imagem em um modo de fluxo de tempo de varredura repetidamente tirando imagens em uma sequência de posições(B). Séries de imagens gravadas são processadas para ler a intensidade da fluorescência por célula ao longo do tempo(C). Barras de escala: 500 μm(A),200 μm(C). Clique aqui para ver uma versão maior desta figura.
Protocolo
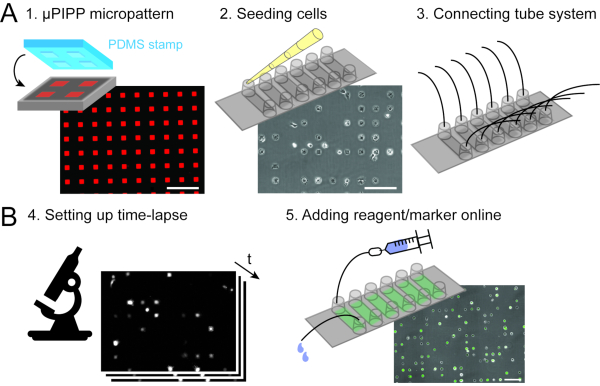
Figura 2: Aquisição de dados combinando microarrays unicelulares (A) com microscopia de lapso de tempo de varredura (B). Como preparação do experimento de lapso de tempo, uma matriz unicelular com um micropattern 2D de quadrados de adesão é preparada (1), seguida pela semeadura celular e o alinhamento das células no micropêrnio (2), bem como a conexão de um sistema de perfusão ao slide de seis canais, que permite o manuseio líquido durante a medição do lapso de tempo (3). Um experimento de lapso de tempo de varredura é configurado (4) e as células são transfeinadas no microscópio injetando uma solução lipoplex de mRNA através do sistema de perfusão durante o experimento de lapso de tempo (5). Barras de escala: 200 μm. Clique aqui para ver uma versão maior desta figura.
1. Fabricação de matriz unicelular microestruturada(Figura 2A)
- Prepare os materiais necessários para a fabricação da matriz μPIPP.
- Prepare a solução salina tamponada de fosfato estéril (PBS) em pH 7.4.
- Prepare água ultrauso estéril com uma resistividade de pelo menos 18 MΩcm a 25 °C.
- Prepare a solução de trabalho PLL(20 kDa)-g[3.5] PEG(2 kDa) (PLL-PEG) com uma concentração de 2 mg/mL de PLL-PEG em água ultrapura contendo 150 mM NaCl e 10 mM 4-(2-hidroxitil)-1-piperazineethanesulfonic acid (HEPES).
- Prepare uma solução de proteína de matriz extracelular para revestimento superficial: 1 mg/mL fibronectina (FN) em PBS.
- Prepare um wafer de silício com um micropattern fabricado pela fotolitografia8 que funciona como um mestre reutilizável. O micropattern consiste em quadrados com um comprimento de borda de 30 μm, uma profundidade de 12 μm e uma distância interquadrada de 60 μm, disposta em seis listras cada uma com uma largura de 6 mm e uma altura de 18 mm.
- Misture um monômero de polidimimetilaxitano (PDMS) com 9% de crosslinker (massa %) usando um kit de elastômero de silicone e degas-lo por cerca de 30 minutos até que ele esteja livre de bolhas usando um desiccator. Lance o wafer de silício com uma camada PDMS de aproximadamente 3-5 mm de espessura e desgase-o novamente por cerca de 30 minutos até que esteja livre de bolhas.
- Coloque o wafer de silício com o PDMS em forno a 50 °C para curar o PDMS por pelo menos 4h.
- Corte os selos PDMS.
- Use um bisturi e recorte da camada PDMS uma obra-prima PDMS que contém as seis listras de micropattern.
- Coloque a obra-prima do PDMS em um banco com o micropattern voltado para cima.
- Corte cada uma das seis listras de micropattern da obra-prima PDMS com um razorblade em um selo PDMS. Tome cuidado para que as bordas dos selos PDMS estejam abertas cortando parte da área padronizada.
- Coloque os selos PDMS em um deslizamento de tampa de um slide de seis canais(Figura 3-1).
- Use um deslizamento de cobertura não revestido e marque as posições do canal do slide de seis canais, arranhando cuidadosamente a folha de proteção do deslizamento de tampas. Em seguida, coloque a mancha de cobertura no banco com a folha de proteção voltada para baixo.
- Coloque os selos PDMS com o micropattern voltado para baixo na tampa nas posições do canal marcadas usando pinças.
- Verifique a fixação dos selos PDMS sob um microscópio. Se um selo PDMS estiver totalmente conectado ao deslizamento de tampa, os quadrados em contato parecem mais escuros do que o interespaço. A fixação do selo PDMS ao deslizamento de cobertura é crucial para a qualidade do micropattern.
- Coloque a tampa com os seis selos PDMS sobre ele em um limpador de plasma e trate-o com plasma de oxigênio (pressão 0,2 mbar, ~40 W por 3 min) para fazer as superfícies entre os selos PDMS e as coberturas hidrofílicas(Figura 3-2).
- Realize todas as etapas adicionais da fabricação de micropattern em um armário de biossegurança. Use 15 μL da solução PLL-PEG e pipeta uma gota dela ao lado de cada selo PDMS para que a solução PLL-PEG seja absorvida no padrão hidrofílico do selo PDMS(Figura 3-3). Deixe o PLL-PEG incubar por 20 minutos em temperatura ambiente.
- Enxágüe 1 mL de água ultrauso sobre o deslizamento de tampas com os selos PDMS e remova os selos PDMS usando pinças(Figura 3-4). Em seguida, enxágue a tampa uma segunda vez com 1 mL de água ultrauso e deixe secar.
- Quando a tampa secar completamente, coloque um slide pegajoso de seis canais na tampa(Figura 3-4). Tome cuidado para que as áreas micropatadas se alinhem com a parte inferior dos canais.
- Funcionalize os quadrados de adesão com FN.
- Encha 40 μL de PBS em cada canal.
- Prepare uma solução de 100 μg/mL FN em PBS.
- Adicione 40 μL da solução FN a cada canal(Figura 3-5). Misture a solução FN com a PBS no canal completamente, removendo 40 μL de um reservatório e adicionando-a ao reservatório oposto do mesmo canal por 3 vezes para gerar uma solução homogênea. Incubar a solução FN por 45 min a temperatura ambiente.
- Lave cada canal três vezes com 120 μL de PBS (Figura 3-6).
- Para verificar se há a qualidade do padrão, use uma FN fluorescentemente rotulada na etapa 9.2. (Figura 4A).
NOTA: Recomendamos preparar a matriz μPIPP não mais do que um dia antes da semeadura celular como PLL-PEG e FN não estão covalentemente ligadas ao substrato e a qualidade do padrão pode diminuir com o tempo. Armazene a matriz μPIPP preparada na geladeira.
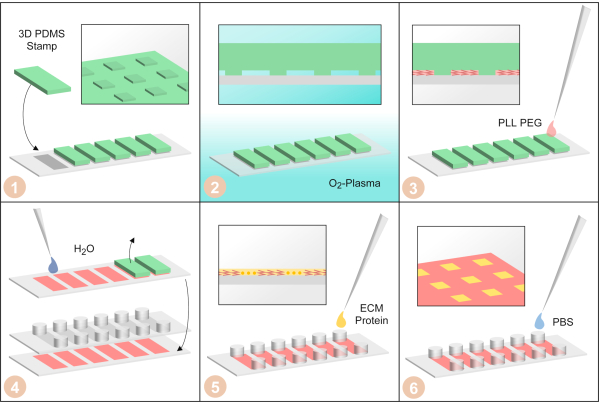
Figura 3: A fabricação de microarraia de célula única por μPIPP. (1) Selos PDMS com uma estrutura de micropattern tridimensional na superfície são dispostos em uma mancha de cobertura de um slide de seis canais. (2) A mancha de cobertura com os selos PDMS é tratada com plasma de oxigênio para tornar as superfícies hidrofílicas. (3) Adicionado PLL-PEG. É absorvida na microestrutura por forças capilares e faz com que as superfícies não cobertas pelo selo PDMS repelente celular. (4) A mancha de cobertura é lavada com água para remover o PLL-PEG restante. Em seguida, os selos PDMS são removidos e um slide pegajoso de seis canais é preso ao deslizamento de tampa. (5) A fibronectina, uma proteína da matriz extracelular, é adicionada para fazer as áreas sem adesivo celular PLL-PEG. (6) O slide de seis canais é lavado com soro fisiológico tamponado por fosfato. Clique aqui para ver uma versão maior desta figura.
2. Semeadura celular (Figura 2A)
NOTA: Para as seguintes etapas de lavagem, adicione o respectivo líquido a um reservatório e, em seguida, remova um volume igual de líquido do reservatório oposto de um canal.
- Lave cada canal com 120 μL de 37 °C totalmente suplementado meio de crescimento celular. Antes de adicionar a suspensão da célula, certifique-se de que apenas os canais estão cheios de médio, mas não os reservatórios.
- Desprender as células HuH7 de um frasco de cultura celular seguindo seu protocolo padrão de passagem celular e ajustar a concentração de suspensão celular para 4 x 105 células/mL.
- Adicione 40 μL de suspensão celular e misture o meio de crescimento celular com a suspensão celular removendo 40 μL de um reservatório e adicionando-o ao reservatório oposto do mesmo canal por 3 vezes para alcançar uma distribuição celular homogênea(Figura 4B).
- Remova 40 μL de suspensão do canal para que apenas o canal seja preenchido com suspensão celular.
- Coloque o slide em uma incubadora e verifique a adesão celular 1h após a semeadura usando um microscópio de contraste de fase.
- Adicione 120 μL de 37 °C de crescimento de células quentes.
- Mas o slide de volta na incubadora por mais 3h para permitir a auto-organização celular na micropattern(Figura 4C).

Figura 4: Auto-organização celular e controle de qualidade da matriz μPIPP. (A) A superfície microestruturada consiste em manchas de adesão revestidas de FN ao quadrado mostradas em vermelho cercadas por um polímero repelente celular. (B) Após a semeadura celular, as células HuH7 são distribuídas aleatoriamente e (C) aderem principalmente nos pontos de adesão durante um período de tempo de 4h. Reimpresso com permissão 7. Barras de escala: 200 μm. Clique aqui para ver uma versão maior desta figura.
3. Sistema de perfusão(Figura 2A)
NOTA: O uso de um sistema de perfusão só é necessário se os reagentes ou marcadores fluorescentes precisarem ser adicionados durante o curso da medição do lapso de tempo. Dependendo de suas necessidades, você pode conectar cada canal a um sistema de perfusão separado ou conectar vários canais em série ao mesmo sistema de perfusão. O número de sistemas de perfusão corresponde ao número de condições experimentais independentes. Conecte os tubos em condições estéreis em um armário de biossegurança e evite a inclusão de bolhas de ar no sistema de perfusão. Se não for utilizado nenhum sistema de perfusão, adicione os reagentes/marcadores em um armário de biossegurança antes da medição do lapso de tempo. O sistema de perfusão é fabricado internamente, o material usado está listado na Tabela de Materiais. A montagem do sistema de perfusão foi descrita anteriormente9.
- Use uma seringa de 1 mL (com esporão de substituição) e encha a seringa com 1 mL de meio de crescimento celular de 37 °C.
- Conecte a seringa ao tubo de entrada usando a válvula e encha o tubo com meio.
- Conecte o tubo de entrada a um reservatório de um canal e certifique-se de que nenhuma bolha de ar esteja presa.
- Para conectar outro canal em série a este sistema de perfusão, conecte um conector serial ao reservatório em frente ao tubo de entrada do canal atual. Vá para o próximo canal e conecte a extremidade livre do conector serial a um de seus reservatórios.
- Repita as etapas anteriores até que o número necessário de canais esteja conectado em séries.
- Conecte o tubo de saída diretamente ao reservatório livre do canal atual. Encha o tubo conectado com meio para verificar se o sistema de perfusão não vaza.
- Repita as etapas anteriores até que todos os seis canais do slide estejam conectados a um sistema de perfusão.
- Coloque o slide com o sistema de perfusão conectado de volta na incubadora até que use ou coloque-o diretamente na câmara de aquecimento do microscópio pré-aquecido a 37 °C para medição de lapso de tempo.
4. Microscopia de lapso de tempo(Figura 2B)
NOTA: Para medições de longo prazo, mantenha uma temperatura estável de 37 °C e um nível de CO2 estável. Como alternativa ao meio de crescimento celular dependente de CO2,use meio L15 para o qual não é necessário um sistema de incubação de gás.
NOTA: Para imagens quantitativas, use o meio de crescimento celular sem fenol vermelho durante a medição do lapso de tempo para reduzir a fluorescência de fundo e use as mesmas configurações do protocolo time-lapse, bem como o mesmo microscópio para réplicas técnicas.
- Configure um protocolo de lapso de tempo para gravar uma imagem de contraste de fase e uma imagem de fluorescência com tempos de exposição de 750 ms (dependendo da câmera), intervalo de tempo de 10 minutos entre loops consecutivos através da lista de posição e um tempo de observação de 30h, usando um filtro de fluorescência objetivo e apropriado de 10x.
- Coloque o slide de seis canais com as células nas matrizes unicelulares no suporte de amostra da câmara de aquecimento quente de 37 °C. Se os sistemas de perfusão estiverem conectados ao slide de seis canais, fixe os tubos no estágio usando alguma fita para garantir que o slide de seis canais não seja movido durante a troca líquida. Insira as extremidades livres dos tubos de saída através de um orifício de um tubo de reação de 15 mL para coletar os resíduos líquidos.
- Defina a lista de posições para a medição do lapso de tempo de varredura. Certifique-se de que o número de posições pode ser escaneado dentro do intervalo de tempo definido entre loops consecutivos através da lista de posições. Com um objetivo de 10x, 10-30 posições por canal podem ser definidas para digitalizar a área total de micropattern, dependendo do tamanho do chip da câmera.
- Inicie a medição do lapso de tempo. Para uma melhor qualidade de imagem de medições de longo prazo, use um sistema automatizado de correção de foco.
5. Marcador fluorescente - transfecção de mRNA(Figura 2B)
NOTA: Para uma transfecção em dois canais conectados por um sistema de tubulação, é necessário um volume total de 600 μL de mistura de transfecção (300 μL para um canal). Os volumes indicados referem-se a uma transfecção em dois canais conectados.
- Prepare uma solução de agente de transfecção diluindo 1 μL de agente de transfecção em 200 μL de meio reduzido por soro e deixe a solução incubar por 5 minutos à temperatura ambiente.
- Prepare uma solução mRNA diluindo 300 ng de codificação mRNA para eGFP em 150 μL de meio reduzido por soro.
- Prepare o mix de transfecção adicionando 150 μL da solução do agente de transfecção à solução mRNA e misture-a bem. Deixe a mistura de transfecção incubar por 20 minutos à temperatura ambiente.
- Lave o sistema de tubulação com 1 mL de PBS quente de 37 °C usando uma seringa durante a incubação da mistura de transfecção. Ao lavar os tubos, certifique-se de que o estágio do microscópio não se mova. Pausar a medição do lapso de tempo, se necessário.
- Diluir a mistura de transfecção para a concentração final de mRNA de 0,5 ng/μL adicionando 300 μL de meio reduzido por soro.
- Lave o sistema de tubulação com a mistura de transfecção usando uma seringa e deixe os lipoplexes mRNA incubarem por 1h (pausar a medição do lapso de tempo, se necessário).
- Pare a incubação de transfecção e limpe os lipoplexes mRNA não ligados lavando com 1 mL de 37 °C meio de crescimento celular totalmente suplementado usando uma seringa (pausar a medição do lapso de tempo, se necessário).
6. Análise de imagem e leitura de fluorescência
- Ao executar a análise de imagem pela primeira vez, instale a versão 0.1.6 do software de código aberto "Análise automatizada de microestrutura em Python" (PyAMA) a partir do localcitado 10 de acordo com as instruções ali fornecidas.
- Certifique-se de que os canais de imagem (contraste de fase e fluorescência) estejam disponíveis como arquivos TIFF de 16 bits de várias imagens. Se necessário, converta-os em conformidade.
- Inicie o PyAMA e clique em Abrir pilha... para abrir imagens para análise.
- Para que cada arquivo TIFF de várias imagens seja aberto, clique em Abrir e selecione o arquivo para que ele seja exibido na lista de arquivos carregados no lado esquerdo da caixa de diálogo (Figura 5-1).
- Marque os canais para incluir na análise. Para cada canal, execute as seguintes etapas.
- Selecione na lista de arquivos carregados o arquivo TIFF contendo o canal.
- Na seção Adicionar novo canal,selecione o índice do canal no arquivo TIFF. A indexação é baseada em zero; o primeiro canal tem índice 0, o segundo canal tem índice 1 e assim por diante.
- Selecione o tipo de canal. Selecione O contraste de fase ou fluorescência para os canais de imagem correspondentes e segmentação para um canal binário indicando os contornos celulares.
- Opcionalmente, digite um rótulo do canal para distinguir diferentes canais de fluorescência: eGFP e DAPI.
- Depois de configurar o canal, clique em Adicionar.
- Quando todos os canais adicionados forem exibidos na lista de canais no lado direito da caixa de diálogo, clique em OK para carregar a pilha.
- Para realizar a segmentação usando o algoritmo de segmentação incorporado do PyAMA para reconhecimento celular com base nas imagens de contraste de fase(Figura 5-2),vá para Ferramentas | Binarize... e digite um nome de arquivo para o arquivo NumPy com o canal binarizado.
NOTA: Na versão atual, carregar o canal binarizado requer recarregar todos os canais. - Para realizar uma correção de fundo11 em um canal de fluorescência (Figura 5-3 ), certifique-sede que o canal de fluorescência e um canal de segmentação estejam carregados. Se nenhum canal de segmentação for carregado, certifique-se de que um canal de contraste de fase seja carregado para segmentação automática. Vá para "Ferramentas > correção de fundo..." e selecione um nome de arquivo para o arquivo TIFF resultante com o canal de fluorescência corrigido.
NOTA: Na versão atual, carregar o canal corrigido em segundo plano requer recarregar todos os canais. - Inspecione as células pré-selecionadas(Figura 5-4) e seu sinal de fluorescência integrada(Figura 5-5) percorrendo os períodos de tempo, visualizando os canais listados no menu do canal no lado esquerdo e clicando nas células para destacar seus cursos de tempo de fluorescência(Figura 1C). Use a seleção celular para excluir células que não são viáveis, não se limitando a um ponto de adesão ou anexadas a outra célula de análises posteriores. Alterne a seleção de células para leitura pressionando Shift e clicando na célula, ou destacando a célula e pressionando Enter.
- Salve os cursos de tempo unicelular para a área celular e a fluorescência integrada(Figura 5-6) clicando em Arquivo | Salve e selecione um diretório para salvar.

Figura 5: O processamento automatizado de imagens da série de imagens de lapso de tempo usando pyama. (1) Séries de imagens de contraste de fase e fluorescência para cada posição de imagem são importadas. (2) Os contornos celulares são determinados pela segmentação na pilha de imagens de contraste de fase. (3) Uma correção de fundo é aplicada às imagens de fluorescência. (4) Os contornos celulares são rastreados ao longo do tempo e pré-selecionados para exportação. (5) A intensidade da fluorescência é integrada com base nos contornos celulares rastreados. (6) Áreas de células unicelulares e intensidades integradas de fluorescência são avaliadas e cursos de tempo para cada célula são exportados. Barras de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.
- Para analisar a cinética de tradução após a transfecção do mRNA, encaixe um modelo de tradução baseado em equações de taxa bioquímica para cada curso de tempo unicelular, conforme descrito anteriormente por Reiser et al.12. Os dados e códigos utilizados nesse estudo estão disponíveis publicamente13.
- Para cada curso de tempo de célula única, recupere os parâmetros estimados de montagem do modelo de tradução que representam a taxa de degradação do mRNA e o ponto de tempo do início da tradução. Um conjunto de dados de exemplo é discutido na seção de resultados representativos.
- Realizar uma análise mais aprofundada sobre as distribuições das melhores estimativas dos parâmetros para condições experimentais variadas para investigar a variabilidade célula-celular dentro das populações celulares.
Resultados
A abordagem LISCA permite coletar eficientemente cursos de tempo de fluorescência a partir de células únicas. Como exemplo representativo, descrevemos como o método LISCA é aplicado para medir a expressão eGFP unicelular após a transfecção. Os dados do experimento LISCA são utilizados para avaliar cinética de entrega de mRNA, o que é importante para o desenvolvimento de medicamentos mRNA eficientes.
Em particular, demonstramos o impacto diferente de dois sistemas de entrega de mRNA...
Discussão
Aqui descrevemos o LISCA como uma técnica versátil para seguir cinética celular de rótulos fluorescentes intracelulares no nível de célula única. Para realizar um experimento LISCA bem-sucedido, cada uma das etapas descritas da seção de protocolo deve ser estabelecida individualmente e, em seguida, todas as etapas devem ser combinadas. Cada um dos três principais aspectos do LISCA apresentam etapas cruciais.
Fabricação de microarraia unicelular
A qualidade da microarray...
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi apoiado por bolsas da Fundação Alemã de Ciência (DFG) ao Centro de Pesquisa Colaborativa (SFB) 1032. Apoio do Ministério Federal alemão da Educação, Pesquisa e Tecnologia (BMBF) no âmbito do projeto cooperativo 05K2018-2017-06716 Medisoft, bem como uma bolsa da Bayerische Forschungsstiftung são reconhecidas com gratidão. Anita Reiser foi apoiada por uma Bolsa DFG através da Escola de Pós-Graduação em Biociências Quantitativas de Munique (QBM).
Materiais
| Name | Company | Catalog Number | Comments |
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: "Correlation of mRNA delivery timing and protein expression in lipid-based transfection". Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados