需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
微殖器的高通量实时成像,测量生长和基因表达中的异质性
摘要
酵母生长表型通过高度平行的延时成像精确测量生长成微殖子的固定细胞。同时,可以监测应力耐受性、蛋白质表达和蛋白质定位,生成综合数据集,研究环境和遗传差异以及同源细胞之间的基因表达异质性如何调节生长。
摘要
精确测量微生物生长速率的菌株之间和菌株内异质性,对于理解遗传和环境投入对压力耐受性、致病性以及健身的其他关键组成部分至关重要。这份手稿描述了一个基于显微镜的检测,跟踪每个实验大约10 5个糖精微殖子。在多井板中固定酵母的自动延时成像后,通过自定义图像分析软件轻松分析微殖速增长率。对于每个微殖质,也可以监测荧光蛋白的表达和本地化以及急性应激的生存情况。这种测定允许精确估计菌株的平均增长率,以及全面测量克隆种群中生长、基因表达和应力耐受性的异质性。
引言
生长表型对酵母健康有至关重要的贡献。自然选择可以有效地区分与增长率不同的血统与相反的有效人口规模,可以超过108个人1。此外,人口内个体增长率的变化是一个进化相关的参数,因为它可以作为生存策略的基础,如押注对冲2,3,4,5,6。因此,能够对生长表型及其分布进行高精度测量的检测对于微生物的研究至关重要。此处描述的微殖器生长测定可以生成每个实验约 105微殖子的个体增长率测量值。因此,这种检测为研究酵母进化遗传学和基因组学提供了强有力的协议。它特别适合测试基因相同的单个细胞种群中的变异性是如何产生、维持和促进种群适应7、8、9、10的。
这里描述的方法(图1)使用定期捕获的低放大亮场图像,在96或384井玻璃底板的液体介质中生长的细胞,以跟踪生长成微殖子。细胞粘附在覆盖显微镜板底部的果胶康卡纳瓦林A上,形成二维菌落。由于微殖质生长在单层中,微殖区与7号细胞高度相关。因此,可以通过自定义图像分析软件生成微殖器增长率和滞后时间的准确估计,该软件可跟踪每个微殖器区域的变化速度。此外,实验设置可以监测这些微殖质中表达的荧光标记蛋白质的丰度,甚至亚细胞定位。这种微殖增长测定数据的下游处理可以通过自定义分析或现有的图像分析软件实现,如易处理图像 (PIE)11,这是一种用于强效聚落区域识别的算法,以及来自低放大、亮场图像的高吞吐量增长分析算法,可通过 GitHub12获得。
由于从微殖民增长测定得出的增长率估计来自大量单殖民地测量,因此它们非常准确,标准误差比合理大小的实验本身的估计值小几个数量级。因此,检测不同基因类型、治疗方法或环境条件之间的生长速率差异的功率很高。多井板格式允许在单个实验中比较许多不同的环境和基因型组合。如果菌株构成表达不同的荧光标记,它们可能混合在同一油井中,并通过随后的图像分析进行区分,从而通过允许逐井数据正常化进一步增加功率。
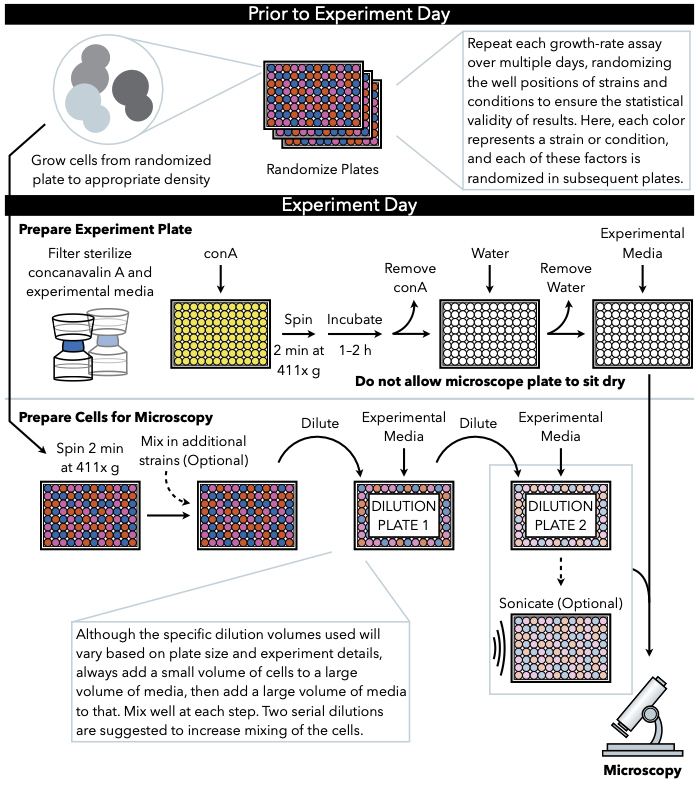
图1:协议的示意图表示。 此协议遵循两个主要步骤,即准备实验板和准备细胞成像。板块的随机化和细胞的生长应在实验日前进行。在稀释过程中,在每一步重复混合细胞在阶梯中必须进行,直到电镀,因此建议首先准备实验板,以便在细胞稀释完成后立即准备电镀。 请单击此处查看此图的更大版本。
研究方案
1. 随机板的准备(实验日前)
- 计划应变和条件测试与生长测定。此时,随机向任何井分配菌株和条件。
注意:在考虑板设置时,建议在单个板上包括多个每个菌株的复制和生长条件,以考虑测量中与之相关的噪音。有关详细信息,请参阅讨论。 - 计算随机化每个菌株的位置和环境条件的板复制将在不同的天运行。
- 在30°C(或任何其他适当温度)的摇床中生长所有用于实验的细胞,以饱和酵母提取物-肽-葡萄糖(YEPD;2%葡萄糖)介质。
- 手动或使用液体处理机器人创建随机库存板。将指定饱和细胞的 10μL 添加到无菌 U 底组织培养板的每口井中。如果在一口井中测试多个菌株,此时不要将其组合在一起:这种组合将在实验当天的细胞稀释之前完成,以确保所有菌株在镀为微殖细胞时都处于正确的浓度。
- 在每个盘子的每口井中加入10微升30%的甘油。上下吹笛,使细胞和甘油很好地混合。
- 用铝箔盖密封每个盘子,并立即冻结在 -70 °C,直到准备使用。
注:重要的是在同一天创建所有随机板,并冻结它们,以便每个板中的细胞的预生长条件是相同的,不会在生长率检测中产生技术变异。
2. 酵母的预生长
注意:通常,这从实验日之前开始,并且高度依赖于实验问题。有关详细信息,请参阅讨论。
- 从 -70 °C 冰柜中取出一个股票板(10 μL 酵母,每口井 10 μL 甘油),并添加 180 μL 的介质用于实验。如果实验将使用营养限制介质进行,则不要在营养限制介质中预生长酵母饱和,因为酵母的孢子会发生。相反,在非限制媒体中预先成长。
- 在30°C时摇晃时生长酵母。 考虑是否从日志阶段或静止阶段的细胞开始运行测定,以确定是否有必要在实验前多次稀释细胞。如果酵母菌株或检测条件下的增长率预计将有显著差异,那么需要两天的预生长期,以便所有不同的条件都达到静止阶段。
3. 显微镜设置
- 显微镜板制备
- 确保显微镜培养箱打开,并将显微镜室加热到实验条件所需的生长温度。对于使用 糖精细胞 的标准实验,孵化器应将显微镜室加热到 30 °C,以确保细胞的生长条件在生长速度检测期间正确无误。
- 用 70% 乙醇消毒工作台、移液器和其他工具。取回显微镜板,放在无绒毛和无静态擦拭的长凳上。
注意:切勿触摸显微镜板的底部,即使戴上手套,并且始终将显微镜板放在绒毛和无静电擦拭的顶部,无论它接触到任何表面。这样可以防止一旦对实验进行成像,污迹或划痕就会阻碍增长率测量。 - 解冻 5mL 的 5 倍康卡纳瓦林 A 溶液,用水稀释至 1 倍,并通过装有 0.2-μm 过滤器的注射器进行滤光消毒。
- 过滤器用 0.2μm 过滤器(包括实验介质)对检测中将使用的所有其他液体进行灭菌,以去除溶液中可能实现的任何晶体或碎屑。晶体的存在会降低显微镜图像的质量。
- 将200μL的康卡纳瓦林溶液放入显微镜板的每一口井中。
- 在 411 x 重力(g)下用绒毛和无静态擦拭将板切成 2 分钟,以确保协和蛋白 A 溶液均匀地覆盖每个油井的底部,并且没有气泡。
- 用盖子盖住盘子,让它坐1-2小时。板坐的确切时间是灵活的,但重要的是要在实验的不同运行之间保持一致。
- 通过吸气或强行将其排入水槽或插座,从盘子中取出所有协和素 A 溶液。小心不要碰盘子的玻璃部分。如果油井中还保留着几滴康卡纳瓦林A溶液,这是可以接受的。
- 通过添加 400 μL 的无菌水来清洗显微镜板井。像前一步的康卡纳瓦林 A 一样取出水。 不要让盘子坐干。
- 立即将 185 μL 实验生长介质添加到板中。15 μL 的 正确稀释 细胞将被添加到此板中。
- 酵母细胞稀释
注意:下面的步骤描述了饱和培养(约108 个细胞/毫升)400倍的酵母稀释,以达到250,000个细胞/毫升的浓度,其中15μL将被稀释为玻璃底板中的400μL,在96井板中每口井提供大约4000个细胞的最终数量。如果使用 384 井板,每口井的最终细胞数应约为 700 个,稀释应相应调整。此比率应根据在日志阶段收集的细胞进行调整,这些细胞生长在较丰富或较差的预生长介质中,或来自不同的菌株。运行超过 10 小时的时间段的增长率检测时,应降低每口井的细胞最终密度。- 设置两个 96 井培养板进行连续稀释:标记为板 1 和 2,并在每个串行稀释板中添加 90 μL 实验生长介质(即酵母将在显微镜上生长的介质)。
注:无论使用何种最终稀释剂,都建议至少进行两次连续的细胞稀释,其中每一个细胞中都会有少量酵母被输送到更大的实验介质中,然后大量实验介质与管道大力混合(如下面步骤 3.2.5 和 3.2.6)。 - 从预生长中取回细胞板,以411 x g将板离心机进行2分钟。
注意:不要在盘子中交叉污染不同的油井是非常重要的。在去除板材上的铝箔覆盖物之前,这一离心步骤的目的是确保一口井中充满酵母的液滴不会从铝箔上飞出,最终进入其他油井。小心切入或搅拌板,以避免酵母在离心后与铝箔覆盖物接触。 - 小心地剥回铝箔并重新输送细胞,用一根管子将细胞用一根管子固定在板内总体积的一半左右,同时将管道绕在井周围混合。检查所有细胞是否已从井底恢复。
- 如果在单独的井内使用多个菌株,应在此时以实验所需的比例混合菌株。如果参考菌株用于生成增长率测量,则参考菌株与测试菌株的参考比率应为 1:1。
- 从生长介质到稀释板 1 的 10 μL 酵母。将 100 μL 的实验生长介质添加到每口井中,最终体积为每口井 200 μL。大力上下吹笛。
- 从板 1 到板 2 的 10 μL 酵母。在每口井中加入 100 μL 的实验生长介质,并大力上下吹笛混合。
注意:这些稀释步骤对于帮助在生长前阶段结束时粘在一起的酵母集群,并确保每口井中最终的酵母细胞数量大致相等至关重要。每口井中酵母数量一致有助于消除实验噪音和增长率测量中的偏差(参见代表性结果)。
- 设置两个 96 井培养板进行连续稀释:标记为板 1 和 2,并在每个串行稀释板中添加 90 μL 实验生长介质(即酵母将在显微镜上生长的介质)。
- 声波
注: 声波是可选的,只需要执行酵母菌株,有很高的倾向,以坚持彼此(例如,一些野生菌株)。对于实验室菌株,声波通常没有必要,可以通过步骤 3.4 跳过。- 将 96 针声波器头与 70% 乙醇一起消毒,将其放入装满 70% 乙醇的 96 井板中,然后用无绒和无静电擦拭干燥。
- 设置一个足够强大的声波程序,以打破絮凝酵母细胞,但不会杀死细胞或导致压力反应升高。可能需要一些测试来确定给定实验的最佳声波程序。此实验中使用的声波程序是:振幅 = 10,过程时间 = 10s,脉冲打开 = 1 s,脉冲关闭 = 1 s。此精确程序可能不适用于所有声波器,因此建议在实验日前进行测试。
- 将酵母混合在串行稀释板 2 中,通过大力吹奏上下五次再次混合酵母。
- 将稀释板 2 放在平台上,用细胞悬架中的声波引脚固定它,但不要触摸板的底部。使用适当的耳部保护运行声波程序。
- 程序运行后,用70%的乙醇清洁声波器头,然后用水,然后 立即 进行显微镜板制备,使细胞不再絮凝。
- 为显微镜准备板:
- 将 15 μL 酵母从串行稀释板 2 放入显微镜板中,体积为 200 μL。 将 200 μL 的实验生长介质添加到每口井中,最终体积为每口井 400 μL,并大力上下输送以混合。
- 用透气膜盖住盘子。用这种膜密封好板很重要,例如使用橡胶辊。
- 要将酵母细胞粘附在玻璃表面的康卡纳瓦林 A 上,请在 411 x g下用绒毛和无静电擦拭离心板 2 分钟。
- 在显微镜下,用无绒毛和无静态擦拭板的顶部和底部,并将压缩空气吹到板上以清除碎屑。
- 将盘子放在显微镜上,确保其水平,并确保 A1 井位于左上角。
4. 延时显微镜增长率测量
注意:在延时显微镜中,以下功能由计算机控制:x、y 和 z 位置、百叶窗和荧光滤镜。基于硬件的自动对焦系统是防止延时成像期间焦点平面漂移的最佳选择。或者,还可以使用基于软件的自动对焦循环。为了保持显微镜室的湿度,建议在实验期间在腔室中保留一个有纯净水的烧嘴。
- 创建一个位置列表 (x,y) 以成像,以便每个显微镜板井完全成像。避免图像重叠,以免对任何单元格进行多次分析。
- 图像在明亮的领域与透镜照明(DIA)的放大倍数为15倍。设置曝光至~5毫秒。
- 以数字方式放大图像,使单元格清晰可见。使用聚焦旋钮在板角的四口井和板中央的一口井中确定实验的理想焦点。聚焦的方式,以获得细胞的最大对比度。
- 将 z 位置(或自动对焦位置)设置为每个油井所识别的 z/自动对焦位置的平均值。如果显微镜板制作良好,玻璃底部没有缺陷,则每口井的理想焦点位置应相似。
注意:当用易处理图像(PIE)图像分析管道11,12分析图像时,有助于细胞稍微脱离显微镜的焦点,使细胞外有一个黑暗的边缘和浅色的内部,这有助于准确的群落识别和大小估计。 - 如果使用荧光菌株,确定图像的通道和曝光,确保不会过度曝光像素。在为荧光通道设置曝光时间时,关闭显微镜上的"实时捕获"模式,以避免细胞长时间暴露在荧光激发中,因为这既能使细胞产生光泡,又会造成压力。
- 设置时间序列采集,以所需的时间间隔拍摄图像,以达到所需的时间长度。
- 运行实验。
结果
此协议的新颖之处在于,通过延时成像(图 2A),可以计算种群中单个细胞的生长速度,从而跟踪其生长成微殖体。由于由于存在康卡纳瓦林A,微殖民以平面方式生长了数小时,因此在整个实验过程中可以跟踪它们的区域,并且可以用来计算观察到的每个观察到的7、9、10、13个殖民地的线性适应该地区自然日志的变化。
讨论
这里描述的协议是一个多功能的检测,允许细胞生长和基因表达同时监测在个别微殖系的水平。结合这两种模式可以产生独特的生物学见解。例如,以前的工作已经使用这个测定显示TSL1基因的表达和同源野生型细胞的微殖体增长率之间的负相关性,同时测量7,10。也可以通过描述的检测来监测荧光标记蛋白质的生长速率和亚细胞定位动力学之间?...
披露声明
作者没有什么可透露的。
致谢
我们感谢娜奥米·齐夫、萨莎·利维和双李为制定本议定书、大卫·格雷舍姆共享设备和玛丽莎·诺尔在视频制作方面所做的贡献。这项工作得到了国家卫生研究院R35GM118170的支持。
材料
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
参考文献
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。