JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
성장과 유전자 발현의 이질성을 측정하는 마이크로콜로니의 높은 처리량 라이브 이미징
요약
효모 성장 표현형은 마이크로콜로 성장하는 고정 된 세포의 매우 병렬 시간 경과 이미징을 통해 정확하게 측정됩니다. 동시에, 스트레스 내성, 단백질 발현 및 단백질 국소화를 모니터링할 수 있고, 통합된 데이터 세트를 생성하여 환경 및 유전적 차이뿐만 아니라 동종 세포 간의 유전자 발현 이질성, 변조 성장을 연구할 수 있습니다.
초록
미생물 성장 속도에서 스트레인 내이질성의 정확한 측정은 유전적 및 환경 입력을 스트레스 내성, 병원성 및 기타 피트니스 구성 요소로 이해하는 데 필수적입니다. 이 원고는 실험 당 약 105 Saccharomyces cerevisiae 미세 식민지를 추적하는 현미경 기반 분석서를 설명합니다. 멀티웰 플레이트에 고정된 효모의 자동 타임랩스 이미징 후 맞춤형 이미지 분석 소프트웨어로 미세 콜로니 성장률을 쉽게 분석할 수 있습니다. 각 미세 식민지에 대 한, 발현 및 형광 단백질의 국소화 및 급성 스트레스의 생존또한 모니터링 될 수 있습니다. 이 분석법은 균주의 평균 성장률의 정확한 추정뿐만 아니라 클로나 집단 내에서 성장, 유전자 발현 및 스트레스 내성의 이질성의 포괄적 인 측정을 허용합니다.
서문
성장 표현형은 효모 피트니스에 매우 기여합니다. 자연 선택은 108명을 초과할 수 있는 유효 인구 규모의 역에 따라 성장률이 다른 혈통을 효율적으로 구별할 수있다. 더욱이, 인구 내개인의 성장률의 변동성은 2,3,4,5,6과같은 생존 전략의 기초역할을 할 수 있기 때문에 진화적으로 관련된 매개 변수이다. 따라서, 성장 표현형및 그 분포의 고도로 정확한 측정을 허용하는 아스약은 미생물의 연구에 중추적인 이다. 여기에 설명된 미세 식민지 성장 분석은 실험 당 ~105 마이크로 콜로니에 대한 개별 성장 속도 측정을 생성 할 수 있습니다. 이 분석법은 따라서 효모 진화 유전학 및 유전체학을 연구하기 위하여 강력한 프로토콜을 제공합니다. 그것은 유전적으로 동일한 단일 세포의 인구 내의 가변성이 생성되고, 유지되고, 인구 적합성7,8,9,10에기여하는 방법을 테스트하는 데 특히 잘 빌려준다.
여기서 설명된 방법은(도 1)을주기적으로 캡처한, 96-384웰 유리 바닥 플레이트에서 액체 미디어에서 성장하는 세포의 저배율 밝은 필드 이미지를 사용하여 마이크로콜로니로의 성장을 추적한다. 세포는 현미경 판의 바닥을 코팅하고 2 차원 식민지를 형성하는 렉틴 concanavalin A를 부착합니다. 마이크로콜로니가 단층에서 자라기 때문에 마이크로콜로니 영역은 세포 번호7과매우 상관관계가 있다. 따라서, 마이크로콜로니 의 성장 속도와 지연 시간의 정확한 추정은 각 마이크로 콜로니의 영역의 변화의 속도를 추적하는 사용자 정의 이미지 분석 소프트웨어로 생성 될 수있다. 더욱이, 실험용 설정은 이러한 마이크로콜로니에서 발현된 형광 표지 단백질의 풍부하고 세포전 소세포 국소화를 모니터링할 수 있다. 이러한 마이크로콜로니 성장 분석에서 데이터의 다운스트림 처리는 맞춤 분석 또는 기존 이미지 분석 소프트웨어(예: 쉽게(PIE)11,GitHub12를통해 사용할 수 있는 저배율, 브라이트필드 이미지로부터의 견고한 콜로니 영역 인식 및 고처리량 성장 분석을 위한 알고리즘으로 달성될 수 있다.
마이크로콜로니-성장 분석에서 파생된 성장률 추정치는 많은 수의 단일 콜로니 측정에서 생성되기 때문에, 표준 오류로 합리적으로 크기의 실험을 위한 추정치 자체보다 몇 배 더 작은 크기로 매우 정확합니다. 따라서 상이한 유전자형, 치료 또는 환경 조건 간의 성장률 차이를 검출하는 분석의 힘이 높다. 멀티웰 플레이트 포맷은 단일 실험에서 수많은 다른 환경과 유전자형 조합을 비교할 수 있게 해줍니다. 균주가 다른 형광 마커를 구성적으로 표현하는 경우, 동일한 우물로 혼합되고 후속 이미지 분석에 의해 구별될 수 있으며, 이는 잘 함으로써 잘 데이터 정규화를 허용함으로써 전력을 더욱 증가시킬 수 있습니다.
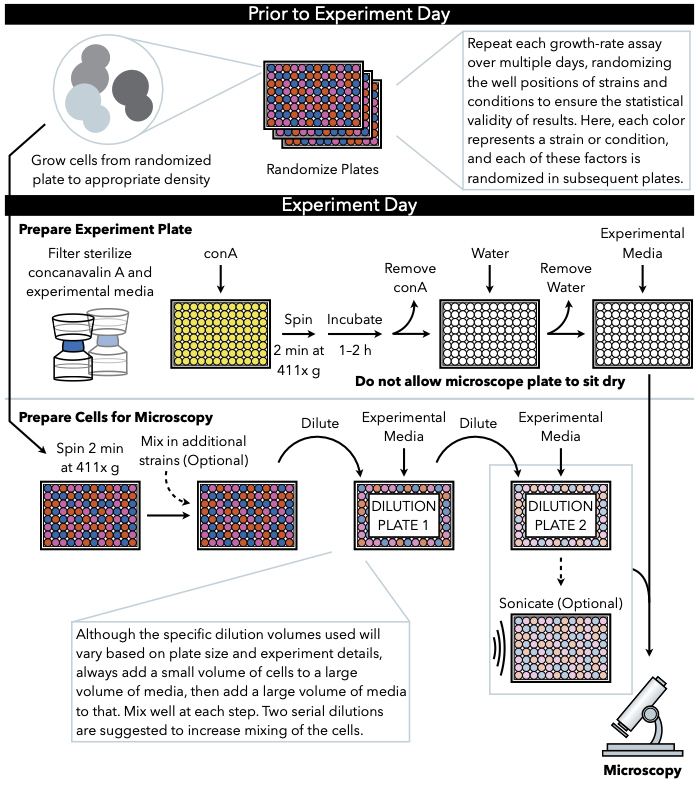
그림 1: 프로토콜의 회로도 표현. 이 프로토콜은 실험 판의 준비및 화상에 세포의 준비인 두 가지 주요 단계를 따릅니다. 플레이트의 무작위화와 세포의 성장은 실험의 날까지 전에 실시되어야한다. 희석 하는 동안 각 단계에서 세포의 반복 혼합 도금 까지 단계에서 필수적 이다, 따라서 먼저 실험 판을 준비 하는 것이 좋습니다 세포 희석의 완료 시 즉시 도금에 대 한 준비가. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. 무작위 판 준비 (실험일 이전)
- 성장 분석으로 테스트할 균주와 조건을 계획합니다. 이 시점에서, 임의로 어떤 우물에 균주와 조건을 할당합니다.
참고: 플레이트 설정을 고려할 때, 측정에서 잘 관련된 소음을 설명하기 위해 단일 플레이트에 변형 및 성장 조건당 둘 이상의 복제본을 포함하는 것이 좋습니다. 자세한 내용은 토론을 참조하십시오. - 다른 날에 실행되는 플레이트 복제에 대한 각 변형 및 환경 조건의 위치를 계산적으로 임의화합니다.
- 30°C(또는 기타 적절한 온도)에서 셰이커에서 효모 추출물-펩톤-덱스트로스(YEPD; 2% 포도당) 배지에서 포화를 위해 실험에 사용될 모든 세포를 성장시다.
- 수동으로 또는 액체 처리 로봇으로 무작위 스톡 플레이트를 만듭니다. 지정된 포화 세포의 10 μL을 멸균 U-바닥 조직 배양 플레이트의 각 웰에 추가합니다. 여러 균주가 하나의 우물에서 테스트될 경우 이 시점에서 결합하지 마십시오. 이 결합은 모든 균주가 마이크로 콜로니 창시자 세포로 도금 될 때 올바른 농도에 있는지 확인하기 위해 실험 당일 세포 희석 직전에 수행됩니다.
- 각 플레이트의 각 웰에 30% 글리세롤의 10 μL을 추가합니다. 세포와 글리세롤이 잘 섞이되도록 위아래로 파이프.
- 호일 커버로 각 접시를 밀봉하고 사용할 준비가 될 때까지 -70 °C에서 즉시 얼어 붙입니다.
참고: 같은 날 임의 플레이트를 모두 생성하고 동결하여 각 플레이트의 세포의 사전 성장 조건이 동일하고 성장속도 분석에서 기술적 변동을 일으키지 않도록 하는 것이 중요합니다.
2. 효모의 성장 전
참고: 일반적으로 실험 일 이전에 시작되며 실험 질문에 크게 의존합니다. 자세한 내용은 토론을 참조하십시오.
- -70°C 냉동고에서 스톡 플레이트(10μL 효모, 웰당 10 μL 글리세롤)를 제거하고 실험에 사용되는 180 μL의 매체를 추가한다. 실험이 영양 제한 매체를 사용하여 수행될 경우 효모의 포자가 발생할 수 있기 때문에 영양 제한 매체의 포화로 효모를 미리 성장시키지 마십시오. 대신, 비 제한 미디어에서 미리 성장.
- 30 °C에서 흔들면서 효모를 성장시다. 실험 전에 세포를 여러 번 희석할 지 여부를 결정하기 위해 로그 상 또는 고정 된 단계에서 세포로 시작하는 분석법을 실행할지 여부를 고려하십시오. 분석의 효모 균주 또는 조건이 크게 다른 성장 속도를 가질 것으로 예상되는 경우, 모든 다른 조건이 고정 된 단계에 도달하기 위해서는 이틀 전 성장 기간이 필요합니다.
3. 현미경 설정
- 현미경 플레이트 준비
- 현미경 인큐베이터가 현미경 챔버를 실험 조건에 대해 원하는 성장 온도로 가열하고 있는지 확인합니다. Saccharomyces cerevisiae 세포를 이용한 표준 실험의 경우, 인큐베이터는 현미경 챔버를 30°C로 가열하여 세포에 대한 성장 조건이 성장 속도 분석 중에 올바른지 확인해야 합니다.
- 작업대, 파이펫 및 기타 공구를 70%의 에탄올로 소독합니다. 현미경 플레이트를 검색하고 보풀과 정전기가없는 닦기 위에 벤치에 놓습니다.
참고: 장갑을 낀 경우에도 현미경 플레이트의 바닥을 만지지 말고, 표면에 닿을 때마다 항상 현미경 플레이트를 보풀과 정적 없는 닦기 위에 놓습니다. 이렇게 하면 실험이 이미지화되면 얼룩이나 긁힘이 증가율 측정을 방해하지 않습니다. - 5x concanavalin A 용액의 5mL를 해동하고, 물로 1x로 희석하고, 필터는 0.2-μm 필터가 장착된 주사기를 통해 살균합니다.
- 필터는 실험 용액에서 구체화될 수 있는 결정이나 이물질을 제거하기 위해 실험용 매체를 포함한 0.2 μm 필터로 분석에서 사용될 다른 모든 액체를 살균합니다. 결정의 존재는 현미경 심법의 질을 감소시킬 것입니다.
- 파이프 200 μL 의 concanavalin 현미경 플레이트의 각 우물에 용액.
- 플레이트 밑에 보풀및 정전기가 없는 닦아411 x중력(g)에서2분 동안 플레이트를 원심분리하여, 콩카나베일 A 용액이 각 우물의 바닥을 고르게 덮고 기포가 없음을 보장한다.
- 뚜껑으로 접시를 덮고 1-2 시간 동안 앉게하십시오. 플레이트가 앉아 정확한 시간은 유연하지만 실험의 다른 실행 간에 일관성을 두는 것이 중요합니다.
- 흡입또는 강제로 싱크대 또는 리셉터클에 배출하여 플레이트에서 모든 콩카나베일린 A 용액을 제거합니다. 접시의 유리 부분을 만지지 않도록주의하십시오. 일부 concanavalin A 용액이 우물에 남아 있으면 허용됩니다.
- 멸균 수의 400 μL을 추가하여 현미경 플레이트 우물을 세척하십시오. 이전 단계에서 concanavalin A와 마찬가지로 물을 제거합니다. 접시가 건조하게 앉지 마십시오.
- 즉시 플레이트에 185 μL 실험 성장 매체를 추가합니다. 15 μL의 올바르게 희석된 셀이 이 판에 첨가됩니다.
- 효모 세포 희석
참고: 아래 단계는 포화 배양(약 108세포/mL) 400배의 효모 희석을 설명하여 250,000개의 세포/mL의 농도를 달성하고, 15μL은 유리 바닥 판에서 400 μL로 희석되어 96개의 잘 된 플레이트당 약 4000개의 세포수를 제공합니다. 384-well 플레이트를 사용하는 경우 웰 당 최종 세포 수는 약 700이어야하며 희석제는 그에 따라 조정되어야합니다. 이 비율은 로그 단계에서 수집된 세포, 더 풍부하거나 가난한 사전 성장 매체또는 다른 균주에서 자라는 세포에 대해 조정되어야 합니다. 10 h 보다 더 긴 기간에 대 한 성장 속도 분석 을 실행할 때 잘 당 세포의 최종 밀도 감소 해야 합니다.- 직렬 희석을 위한 96웰 배양 플레이트 2개를 설정하고, 플레이트 1과 2라벨을 추가하고, 각 직렬 희석 플레이트에 90 μL 실험 성장 미디어(즉, 효모가 현미경으로 성장할 매체)를 추가합니다.
참고: 어떤 최종 희석이 사용되는지에 관계없이, 소량의 효모가 더 큰 실험 용으로 파이프로 파이프를 삽입한 다음 대량의 실험 용 미디어가 파이펫과 격렬하게 혼합되는 각각의 셀의 적어도 두 개의 직렬 희석이 권장됩니다(아래 단계 3.2.5 및 3.2.6 단계와 같이). - 411 x g에서 2 분 동안 플레이트를 사전 성장 및 원심 분리에서 세포의 플레이트를 검색하십시오.
참고 : 접시에 다른 우물을 교차 오염하지 않는 것이 매우 중요합니다. 플레이트에서 덮는 호일을 제거하기 전에이 원심 분리 단계의 목적은 효모가 채워진 방울이 호일을 날아 다른 우물에서 끝내지 않도록하는 것입니다. 원심분리 후 포일과 접촉하는 효모를 피하기 위해 접시를 기울이거나 교반하지 않도록 주의하십시오. - 조심스럽게 호일을 다시 껍질을 벗기고 혼합 하는 우물 주위 파이펫을 이동 하는 동안 접시에 있는 총 부피의 약 절반으로 설정 하는 파이펫으로 세포를 적극적으로 파이펫에 의해 세포를 다시 중단. 모든 세포가 우물 바닥에서 다시 중단되었는지 확인합니다.
- 개별 우물 내의 여러 균주가 사용되는 경우, 균주는 실험에 필요한 비율로이 시간에 혼합되어야한다. 기준 균주를 사용하여 성장률 측정을 생성하는 경우 테스트 스트레인에 대한 참조 비율은 1:1이어야 합니다.
- 성장 매체에서 희석 판 1로 효모의 파이프 10 μL. 100 μL의 실험 성장 매체를 각 웰에 추가하여 우물당 200 μL의 최종 부피에 추가합니다. 힘차게 위아래로 파이프.
- 플레이트 1에서 플레이트 2로 효모의 파이프 10 μL. 각 웰에 100 μL의 실험 성장 매체를 추가하고, 혼합을 위해 적극적으로 위아래로 파이프.
참고: 이러한 희석 단계는 성장 전 단계의 끝에 함께 붙어 있는 효모의 분리클러스터를 돕고 대략 동일한 수의 효모 세포가 각 우물에서 끝나는지 확인하는 데 중요합니다. 각 웰에 일관된 효모 수를 갖는 것은 성장 속도 측정에서 실험 소음과 편견을 제거하는 데 도움이됩니다 (대표 결과 참조).
- 직렬 희석을 위한 96웰 배양 플레이트 2개를 설정하고, 플레이트 1과 2라벨을 추가하고, 각 직렬 희석 플레이트에 90 μL 실험 성장 미디어(즉, 효모가 현미경으로 성장할 매체)를 추가합니다.
- 쥡니다
참고 : 초음파 는 선택 사항이며,서로를 준수하는 경향이 높은 효모 균주에 대해서만 수행해야합니다 (예 : 일부 야생 균주). 실험실 균주의 경우 초음파 처리는 일반적으로 필요하지 않으며 3.4 단계로 진행하여 건너 뛸 수 있습니다.- 96핀 소닉레이터 헤드를 70% 에탄올로 소독하여 70% 에탄올로 채워진 96웰 플레이트에 넣고 보풀과 정전기가 없는 닦아냅니다.
- 팽창 효모 세포를 분해하기에 충분히 강한 초음파 프로그램을 설정하지만 세포를 죽이거나 높은 응력 반응을 일으키지 않습니다. 일부 테스트는 주어진 실험에 가장 적합한 초음파 처리 프로그램을 식별하기 위해 필요할 수 있습니다. 이 실험에 사용되는 초음파 처리 프로그램은 진폭 = 10, 공정 시간 = 10 s, 펄스 온 = 1 s, 펄스 오프 = 1 s입니다. 이 정확한 프로그램은 모든 초음파 검사에 적용되지 않을 수 있으므로 실험 일 전에 테스트가 제안됩니다.
- 효모를 연속 희석 플레이트 2에 한 번 더 섞어 5번 위아래로 힘차게 피펫을 드시면 됩니다.
- 희석 플레이트 2를 플랫폼에 놓고 셀 서스펜션의 초음파 처리기 핀으로 고정하지만 플레이트의 바닥을 만지지 않습니다. 적절한 귀 보호 기능을 사용하여 초음파 처리 프로그램을 실행합니다.
- 프로그램이 실행된 후 초음파 처리기 헤드를 70% 에탄올로 청소한 다음 물로 즉시 현미경 플레이트 준비로 진행하여 세포가 다시 울지 않도록 합니다.
- 현미경을 위한 플레이트 준비:
- 시리얼 희석 플레이트 2에서 현미경 플레이트에 200 μL의 부피에 효모의 파이프 15 μL. 각 웰에 실험 성장 매체의 200 μL을 추가하여 우물당 400 μL의 최종 부피에 넣고, 양열적으로 혼합하도록 배관한다.
- 통기성 멤브레인으로 접시를 덮습니다. 예를 들어 고무 롤러를 사용하여이 멤브레인으로 플레이트를 잘 밀봉하는 것이 중요합니다.
- 유리 표면의 콩카보린 A에 효모 세포를 부착하려면 411 x g에서2 분 동안 보풀과 정적 무정성 닦아 플레이트를 원심 분리합니다.
- 현미경에서, 보풀과 정적 없는 닦아접시의 상단과 아래쪽을 닦고, 이물질을 제거하기 위해 접시에 압축 공기를 날려 버린다.
- 접시를 현미경에 올려 놓고, 그것이 수준인지 확인하고 A1이 왼쪽 상단 모서리에 있는지 확인합니다.
4. 시간 경과 현미경 검사기 성장 속도 측정
참고: 경과 현미경 검사 중에 x, y 및 z 위치, 셔터 및 형광 필터와 같은 컴퓨터 제어 기능이 있습니다. 하드웨어 기반 자동 초점 시스템은 시간 경과 이미징 중에 초점 평면 드리프트를 방지하는 데 최적입니다. 또는 소프트웨어 기반 자동 초점 루프를 사용할 수 있습니다. 현미경 챔버의 습도를 유지하기 위해 실험 기간 내내 챔버에 정제 된 물로 비커를 유지하는 것이 좋습니다.
- 각 현미경 플레이트가 완전히 이미지되도록 이미지에 위치(x,y) 목록을 만듭니다. 셀을 여러 번 분석하지 않도록 이미지가 겹치지 않도록 하십시오.
- 15배의 배율에서 횡반조명 (DIA)이있는 밝은 필드의 이미지. ~ 5 ms에 노출을 설정합니다.
- 셀이 명확하게 보이게 되도록 이미지를 디지털로 확대합니다. 초점 손잡이를 사용하여 플레이트 모서리에 있는 4개의 우물과 플레이트 중앙에 있는 4개의 우물에서 실험에 이상적인 초점을 식별합니다. 세포의 최대 대비를 얻기 위해 같은 방법으로 초점을 맞춥니다.
- 실험에 대한 z 위치(또는 자동 초점 위치)를 이러한 각 우물에 대해 식별된 z/자동 초점 위치의 평균으로 설정합니다. 현미경 플레이트가 잘 만들어지고 유리 바닥에 결함이없는 경우 이상적인 초점 위치는 각 우물마다 유사해야합니다.
참고: 프로세싱 이미지(PIE) 이미지 분석파이프라인(11,12)을사용하여 이미지를 분석할 때, 세포가 현미경에 약간 초점을 맞추지 않도록 세포가 현미경에 약간 초점을 맞추지 않도록 하고, 정확한 콜로니 인식 및 크기 추정치에 도움이 되는 밝은 색상의 내부가 있는 것이 도움이 된다. - 형광균을 사용하는 경우 이미지에 대한 채널과 노출을 식별하여 픽셀이 과도하게 노출되지 않도록 합니다. 형광 채널에 대한 노출 시간을 설정할 때, 현미경의 "라이브 캡처"모드를 끄고 장기간 형광 흥분에 세포를 노출하지 않도록, 이것은 모두 세포를 표백하고 스트레스를 일으킬 수 있기 때문에.
- 원하는 시간 간격으로 이미지를 캡처하도록 시간 시퀀스 수집을 설정합니다.
- 실험을 실행합니다.
결과
이 프로토콜의 참신은 시간 경과 이미징(그림 2A)을통해 마이크로 콜로니로의 성장을 추적하여 인구 내의 개별 세포에 대해 성장 속도를 계산할 수 있다는 것입니다. 마이크로콜로니가 파랑자 방식으로 수시간 동안 자라기 때문에, 그 영역은 실험 전반에 걸쳐 추적될 수 있으며, 시간이 지남에 따라 지역의 자연 로그의 변화에 선형적 맞춤을 관찰한 각 개별 식민지에 대한 성...
토론
여기서 설명된 프로토콜은 세포 성장 및 유전자 발현이 개별 마이크로콜로니의 수준에서 동시에 모니터링될 수 있도록 하는 다목적 분석이다. 이 두 가지 양식이 결합하면 고유한 생물학적 통찰력을 얻을 수 있습니다. 예를 들어, 이전 연구는 이 분석을 사용하여 TSL1 유전자의 발현과 동종 야생형 세포에서 마이크로콜로니 성장률 사이의 부정적인 상관관계를 동시에7,...
공개
저자는 공개 할 것이 없습니다.
감사의 말
나오미 지브, 사샤 레비, 슈앙 리(Shuang Li)가 이 프로토콜 개발에 기여한 것에 대해 감사를 표하고, 데이비드 그레샴(David Gresham)은 공유 장비를 제공했으며, 마리사 크놀이 비디오 제작에 도움을 준 것에 대해 감사드립니다. 이 작품은 건강 보조금 R35GM118170의 국가 학회에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
참고문헌
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유