このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
成長と遺伝子発現の不均一性を測定するマイクロコロニーのハイスループットライブイメージング
要約
酵母増殖型は、マイクロコロニーに成長する固定化細胞の高度に平行なタイムラプスイメージングを通じて正確に測定される。同時に、ストレス耐性、タンパク質発現、タンパク質の局在化を監視し、統合データセットを生成して、環境と遺伝的の違い、およびイソジェニック細胞間の遺伝子発現不均一性を研究し、成長を調節することができます。
要約
微生物の成長速度における菌株間および内の不均一性の正確な測定は、ストレス耐性、病原性、およびフィットネスの他の重要な構成要素への遺伝的および環境的な入力を理解するために不可欠です。この原稿は、実験ごとに約105 サッカロミセスセレビシエ の微小コロニーを追跡する顕微鏡ベースのアッセイを記述しています。マルチウェルプレートに固定化された酵母の自動タイムラプスイメージング後、マイクロコロニーの成長率はカスタム画像解析ソフトウェアで容易に解析できます。各マイクロコロニーについて、蛍光タンパク質の発現と局在化、急性ストレスの生存もモニタリングできます。このアッセイにより、株の平均成長率を正確に推定できるほか、クローン集団内の増殖、遺伝子発現、ストレス耐性の不均一性を総合的に測定できます。
概要
成長フェノタイプは酵母のフィットネスに重要に貢献します。自然選択は、有効人口規模の逆数によって異なる成長率を有する系統間を効率的に区別することができ、これは1個1の1を超える可能性がある。さらに、人口内の個体間の成長率の変動は、ベットヘッジ2、3、4、5、6などの生存戦略の基礎となる可能性があるため、進化的に関連するパラメータである。したがって、増殖表現型とその分布を極めて正確に測定できるアッセイは、微生物の研究にとって極めて重要です。ここで述べたマイクロコロニー成長アッセイは、実験ごとに〜105マイクロコロニーの個々の増殖速度測定を生成することができる。したがって、このアッセイは、酵母進化遺伝学とゲノミクスを研究するための強力なプロトコルを提供します。遺伝的に同一の単一細胞の集団内の変動が生成され、維持され、集団適合性7、8、9、10にどのように寄与するかをテストするのに特に適しています。
ここで説明する方法(図1)は、96または384ウェルガラス底板上の液体媒体に成長する細胞の周期的に捕捉された低倍率の明視野画像を使用して、マイクロコロニーへの成長を追跡する。細胞は、顕微鏡板の底部を覆うレクチン・コンカナジンAに付着し、二次元コロニーを形成する。マイクロコロニーは単層で成長するため、マイクロコロニー領域は細胞番号7と高い相関性を持つ。したがって、マイクロコロニーの成長率とラグタイムの正確な推定値は、各マイクロコロニーの面積の変化率を追跡するカスタム画像解析ソフトウェアで生成することができます。さらに、実験のセットアップは、これらのマイクロコロニーで発現する蛍光標識タンパク質の存在量および細胞内局在化を監視することができる。このマイクロコロニー成長アッセイからのデータのダウンストリーム処理は、カスタム分析または既存の画像解析ソフトウェア(画像処理容易(PIE)11)、低倍率、明視野画像からの堅牢なコロニー領域認識およびハイスループット成長解析のためのアルゴリズムによって達成され得る。
マイクロコロニー成長アッセイから得られた増殖率推定値は、多数の単一コロニー測定から生成されるため、非常に正確であり、合理的な規模の実験では、推定値自体よりも数桁小さい標準誤差が存在します。したがって、異なる遺伝子型、治療、または環境条件間の成長速度差を検出するアッセイのパワーは高い。マルチウェルプレート形式により、多くの異なる環境と遺伝子型の組み合わせを1回の実験で比較できます。株が構成的に異なる蛍光マーカーを発現する場合、それらは同じウェルで混合され、その後の画像分析によって区別され、ウェルバイウェルのデータの正規化を可能にすることによってさらに電力を増加させる可能性があります。
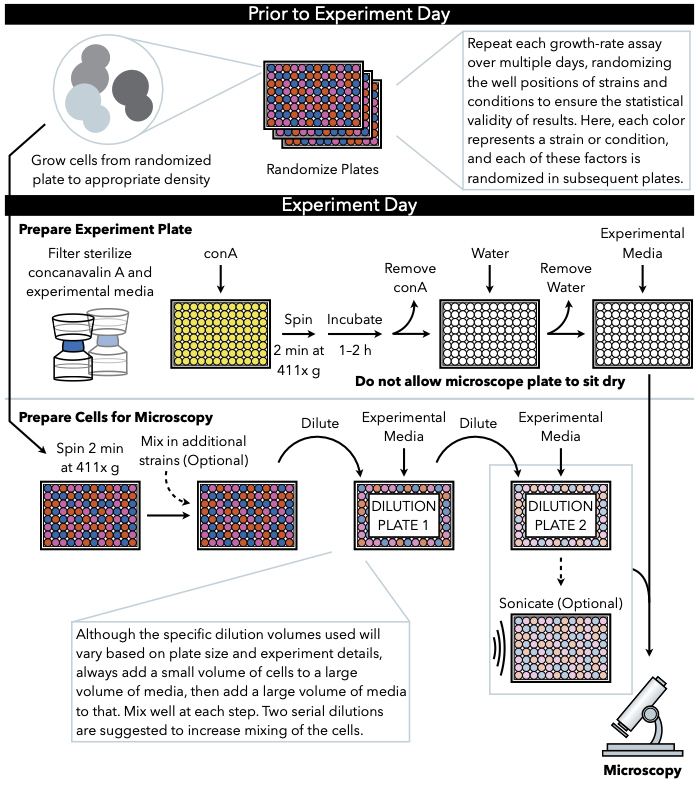
図1: プロトコルの概略図 このプロトコルは、実験プレートの調製と画像への細胞の調製である2つの主要なステップに従います。プレートのランダム化と細胞の増殖は、実験日の前に、そして実験日に至るまで行われるべきである。希釈時の各工程での細胞の繰り返し混合は、めっきまでの工程において必須であり、したがって、細胞希釈が完了した直後にめっきできるように、実験プレートを最初に準備することが推奨される。 この図の大きなバージョンを表示するには、ここをクリックしてください。
プロトコル
1. ランダムプレートの作成(実験日前)
- 成長アッセイで試験される株および条件を計画する。この時点で、任意のウェルにひずみと条件をランダムに割り当てます。
注:プレートの設定を検討する場合、測定で適切に関連するノイズを考慮するために、1つのプレートに1つの歪みと成長条件ごとに複数の反復を含めるのが賢明です。詳細については、「ディスカッション」を参照してください。 - 異なる日に実行されるプレート複製の各ひずみと環境条件の位置を計算的にランダム化します。
- 30°C(または他の適切な温度)でシェーカー中の酵母エキスペプトンデキストロース(YEPD;2%グルコース)培地で飽和する実験で使用されるすべての細胞を成長させます。
- ランダム化ストックプレートを手動で作成するか、液体処理ロボットで作成します。滅菌U-底組織培養プレートの各ウェルに、指定された飽和細胞の 10 μL を加えます。複数の株が単一のウェルでテストされる場合は、この時点でそれらを組み合わせないでください。この結合は、実験の日に細胞希釈の直前に行われ、マイクロコロニー形成細胞としてメッキされた場合にすべての株が正しい濃度であることを確認する。
- 各プレートの各ウェルに30%のグリセロールの10 μLを加えます。細胞とグリセロールがよく混ざるように上下にピペット。
- 各プレートをホイルカバーで密封し、すぐに-70°Cで凍結して使用できる状態にします。
注:ランダム化されたプレートをすべて同じ日に作成し、凍結して、各プレートの細胞の成長前の状態が同じになり、成長率アッセイに技術的な変動が生じないようにすることが重要です。
2. 酵母の前生育
注: 通常、これは実験日より前に始まり、実験の質問に大きく依存します。詳細については、「ディスカッション」を参照してください。
- ストックプレート(10 μL酵母、1ウェルあたり10μLグリセロール)を-70°C冷凍庫から取り出し、実験に使用する培地の180μLを加えます。栄養制限培地を用いて実験を行う場合は、酵母の胞子形成が起こり得る栄養制限培地中で酵母を飽和に前成長させないでください。代わりに、非制限メディアで事前に成長します。
- 30°Cで揺れながら酵母を育てる。 実験の前に細胞を複数回希釈する必要があるかどうかを判断するために、対数相または定常相の細胞から始まるアッセイを実行するかどうかを検討してください。酵母株またはアッセイの条件が有意に異なる増殖速度を有すると予想される場合、すべての異なる条件が定常段階に達するためには、2日間の前成長期間が必要となる。
3. 顕微鏡のセットアップ
- 顕微鏡板の準備
- 顕微鏡インキュベーターがオンであることを確認し、実験条件のために所望の成長温度に顕微鏡室を加熱します。 サッカロミセス・セレビシエ 細胞を用いた標準的な実験では、インキュベーターは、増殖速度アッセイ中に細胞の成長条件が正しいことを確認するために、顕微鏡室を30°Cに加熱する必要があります。
- 70%エタノールでワークベンチ、ピペット、その他のツールをサニタイズします。顕微鏡プレートを取り出し、lintと静的なワイプの上のベンチに置きます。
注:手袋をはめても顕微鏡プレートの底部に触れないでください、そして、それは表面に触れるときはいつでも、常にlintと静電気のワイプの上に顕微鏡プレートを下に置きます。これにより、実験がイメージ化されると、汚れや傷が成長速度の測定を妨げなくなります。 - 5xコンカナリンA溶液の5 mLを解凍し、水で1倍に希釈し、0.2μmフィルターを取り付けた注射器を通してフィルター滅菌します。
- フィルターは、実験媒体を含む0.2 μmフィルターでアッセイに使用される他のすべての液体を殺菌し、溶液中で具体化した可能性のある結晶または破片を除去します。結晶の存在は、顕微鏡画像の品質を低下させるであろう。
- コンカナリンのピペット200 μL顕微鏡板の各ウェルに溶液。
- プレートの下に、リントと静的なワイプを使用して411x重力(g)でプレートを2分間遠心分離し、コンカナバリンA溶液が各ウェルの底部を均等に覆い、気泡がないことを確認します。
- 蓋でプレートを覆い、1〜2時間座らせます。プレートが座っている正確な時間は柔軟ですが、実験の異なる実行間で一貫性を保うことが重要です。
- コンカナリン A 溶液をすべて、吸引するか、シンクまたはレセプタクルに強制的に排出してプレートから取り外します。プレートのガラス部分に触れないように注意してください。コンカナバリンA溶液のいくつかの滴が井戸に残っている場合、それは受け入れられます。
- 400μLの滅菌水を加えて顕微鏡プレートウェルを洗います。前の手順でコンカナバリンAで行ったように水を取り除きます。 プレートを乾かしてはなりません。
- すぐにプレートに185 μLの実験成長培地を加えます。15μLの 正しく希釈された 細胞がこのプレートに添加されます。
- 酵母細胞希釈液
注:以下のステップは、飽和培養物(約108 細胞/mL)から酵母を400倍に希釈して250,000細胞/mLの濃度を達成し、そのうち15μLはガラス底板で400μLに希釈し、96ウェルプレートでウェルあたり約4000細胞の最終数を与えることを説明しています。384ウェルプレートを使用する場合、ウェルあたりの細胞の最終的な数は約700であり、希釈はそれに応じて調整する必要があります。この比率は、ログ段階で収集された細胞、より豊かで貧しい前成長培地で成長、または異なる株から成長するために調整する必要があります。10時間より長い期間の増殖率アッセイを実行する場合、ウェル当たりの細胞の最終的な密度を減少させる必要があります。- シリアル希釈用の2つの96ウェル培養プレートをセット:プレート1と2としてラベルし、各シリアル希釈板に90μL実験成長培地(すなわち、酵母が顕微鏡上で成長する培地)を追加します。
注意:最終的な希釈が使用されるかどうかにかかわらず、少なくとも2つの連続希釈細胞が推奨され、それぞれに少量の酵母が実験媒体のより大きなボリュームにピペットに配管され、その後、大量の実験媒体がピペットと激しく混合されます(下記のステップ3.2.5および3.2.6)。 - 細胞のプレートを成長前から取り出し、411 x gで2分間プレートを遠心分離します。
注:プレート内の異なる井戸をクロス汚染しないことは非常に重要です。プレートからカバーするホイルを取り除く前のこの遠心分離ステップの目的は、1つの井戸から酵母で満たされた液滴がホイルから飛んで他の井戸に終わるのを確実にすることです。遠心分離後に覆うホイルに酵母が接触するのを避けるために、プレートを傾けたり攪拌したりしないように注意してください。 - ホイルを慎重に剥がし、ミキシングするウェルの周りにピペットを動かしながら、プレート内の総体積の約2分の1に設定されたピペットで細胞を激しくピペット化して細胞を再中断します。すべての細胞が井戸の底から再懸濁されていることを確認してください。
- 個々のウェル内の複数の株が使用される場合、株は実験に必要な比率でこの時点で混合する必要があります。参照歪みを使用して成長率の測定値を生成する場合、検定歪みに対する参照の比率は 1:1 にする必要があります。
- 成長培地から希釈板1に酵母の10μLをピペット。1ウェルあたり200μLの最終容積に、各ウェルに100μLの実験成長培地を加えます。激しく上下にピペット。
- プレート1からプレート2に酵母の10μLをピレットする。各ウェルに100μLの実験成長培地を加え、激しく上下にパイプして混ぜます。
注:これらの希釈ステップは、成長前段階の終わりに一緒にくっついている酵母のクラスターを分離し、酵母細胞のほぼ同じ数が各井戸で終わるようにするために重要です。各ウェルに一貫した数の酵母を持つことは、成長速度測定における実験的なノイズとバイアスを取り除くのに役立ちます(代表結果を参照)。
- シリアル希釈用の2つの96ウェル培養プレートをセット:プレート1と2としてラベルし、各シリアル希釈板に90μL実験成長培地(すなわち、酵母が顕微鏡上で成長する培地)を追加します。
- 超音波
注: 超音波処理は、オプションであり、お互いに付着する傾向が高い酵母株(例えば、いくつかの野生株)のために行われる必要があります。ラボ株の場合、超音波処理は一般的に必要ではなく、ステップ3.4に進んでスキップされる可能性があります。- 70%エタノールで満たされた96ウェルプレートに入れ、糸くずと静電気の拭き取りで乾燥させることによって、70%エタノールで96ピンのソニエーターヘッドを消毒します。
- 凝集酵母細胞を分解するのに十分強い超音波処理プログラムを設定しますが、細胞を殺したり、ストレス応答の上昇を引き起こしません。特定の実験に最適な超音波処理プログラムを特定するために、いくつかのテストが必要な場合があります。この実験で使用される超音波処理プログラムは、振幅= 10、処理時間 = 10 s、パルスオン = 1 s、パルスオフ= 1 sです。この正確なプログラムは、実験日の前にテストが推奨されているので、すべての超音波処理器には適用されない可能性が高いです。
- シリアル希釈板2に酵母を5回激しく上下にピペット化してもう一度混ぜる。
- 希釈板2をプラットフォーム上に置き、セルサスペンションのソニエーターピンで固定しますが、プレートの底部には触れないでください。適切な耳の保護を使用して超音波処理プログラムを実行します。
- プログラムが実行された後、70%エタノールでソニッチャルヘッドを洗浄し、次に水で 、すぐに細胞 が再び凝集しないように顕微鏡プレート調製に進みます。
- 顕微鏡用プレートを準備する:
- シリアル希釈板2から顕微鏡プレートから200μLの体積に酵母の15μLをパイプで入れ、200μLの実験成長培地を各ウェルに200 μLずつ加えて、井戸あたり400μLの最終体積に加え、激しく上下にピペットして混合します。
- 通気性のある膜でプレートを覆います。例えばゴム製ローラーを用いて、この膜でプレートを密封することが重要です。
- ガラス表面上のコンカナリンAに酵母細胞を接着するために、411 x gで2分間、その下に糸くずと静電気のないワイプでプレートを遠心分離する。
- 顕微鏡では、プレートの上部と下部を糸くずと静電気のワイプで拭き取り、圧縮空気をプレートに吹き込んで破片を取り除きます。
- プレートを顕微鏡に置き、レベルであり、A1ウェルが左上隅であることを確認します。
4. タイムラプス顕微鏡の成長速度測定
注意:タイムラプス顕微鏡では、X、y、z位置、シャッター、蛍光フィルタのコンピュータ制御機能があります。ハードウェアベースのオートフォーカスシステムは、タイムラプスイメージング中の焦点面ドリフトを防ぐのに最適です。あるいは、ソフトウェアベースの自動焦点ループを使用することもできます。顕微鏡室の湿気を維持するために、実験の間ずっとチャンバーの浄化された水とビーカーを保つことを勧める。
- 各顕微鏡プレートウェルが完全に画像化されるように、イメージに位置(x,y)のリストを作成します。セルが複数回解析されないように、画像の重なりを避けます。
- 15倍の倍率で、ディジカル照明(DIA)を持つ明視野の画像。露出を~5 ミリ秒に設定します。
- セルがはっきりと見えるように、画像をデジタルで拡大します。焦点を合わせるノブを使用して、プレートの隅にある4つのウェルとプレートの中央にある1つの井戸で実験に理想的なフォーカスを特定します。細胞の最大のコントラストを得るように焦点を合わせます。
- 実験の z 位置 (または自動焦点位置) を、これらのウェルごとに識別される z/オートフォーカス位置の平均値に設定します。顕微鏡板がうまく作られ、ガラス底に欠陥がない場合、理想的な焦点位置は各ウェルに類似しているはずです。
注:画像処理を容易に(PIE)画像解析パイプライン11,12で解析する場合、細胞の外側に暗い縁があり、明るい色の内部が存在するため、細胞が顕微鏡に少し焦点を合わせず、正確なコロニー認識とサイズの推定に役立ちます。 - 蛍光株を使用する場合は、チャンネルと画像に使用する露出を特定し、ピクセルが露出過多にならないようにします。蛍光チャネルの露光時間を設定する場合は、細胞を光漂白してストレスを引き起こす可能性がありますので、長期間蛍光励起に細胞を曝露しないように、顕微鏡上の「ライブキャプチャ」モードをオフにします。
- 時間シーケンス取得を設定して、目的の時間間隔で画像を取得します。
- 実験を実行します。
結果
このプロトコルの新しい点は、タイムラプスイメージングを通じてマイクロコロニーへの成長を追跡することによって、集団内の個々の細胞の成長率を計算できることである(図2A)。コンカナリアニンAの存在により、マイクロコロニーは平面的に何時間も成長するため、その領域は実験全体を通して追跡することができ、時間の経過とともに領域の自然対数の変化に直?...
ディスカッション
ここで説明するプロトコルは、細胞増殖と遺伝子発現を個々のマイクロコロニーのレベルで同時に監視することを可能にする汎用性の高いアッセイです。これら2つのモダリティを組み合わせることで、ユニークな生物学的洞察が得られます。例えば、以前の研究では、このアッセイを用いて、同時に7,10の両方を測定することにより、アイソジェニ...
開示事項
著者らは開示するものは何もない。
謝辞
ナオミ・ジヴ、サーシャ・レヴィ、双李は、このプロトコルの開発に貢献してくれたことに感謝し、共有機器のデビッド・グレシャム、ビデオ制作に協力してくれたマリッサ・ノールに感謝します。この研究は、国立衛生研究所の助成金R35GM118170によってサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
参考文献
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved