É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem ao vivo de alta taxa de microcolônias para medir heterogeneidade no crescimento e expressão genética
Neste Artigo
Resumo
Fenótipos de crescimento de leveduras são precisamente medidos através de imagens de lapso de tempo altamente paralelas de células imobilizadas crescendo em microcolônias. Simultaneamente, a tolerância ao estresse, a expressão proteica e a localização da proteína podem ser monitoradas, gerando conjuntos de dados integrados para estudar como as diferenças ambientais e genéticas, bem como a heterogeneidade de expressão genética entre as células isogênicas, modulam o crescimento.
Resumo
Medições precisas da heterogeneidade entre e dentro da tensão nas taxas de crescimento microbiano são essenciais para a compreensão de insumos genéticos e ambientais na tolerância ao estresse, patogenicidade e outros componentes-chave da aptidão. Este manuscrito descreve um ensaio baseado em microscópio que rastreia aproximadamente 105 microcolônias de Saccharomyces por experimento. Após a imagem automatizada de lapso de tempo de levedura imobilizada em uma placa multiwell, as taxas de crescimento de microcolonia são facilmente analisadas com software personalizado de análise de imagem. Para cada microcolonia, a expressão e a localização de proteínas fluorescentes e a sobrevivência do estresse agudo também podem ser monitoradas. Este ensaio permite uma estimativa precisa das taxas médias de crescimento das cepas, bem como uma medição abrangente da heterogeneidade no crescimento, expressão genética e tolerância ao estresse dentro das populações clonais.
Introdução
Fenótipos de crescimento contribuem criticamente para o condicionamento físico da levedura. A seleção natural pode distinguir eficientemente entre linhagens com taxas de crescimento diferidas pelo inverso do tamanho populacional efetivo, que pode exceder 108 indivíduos1. Além disso, a variabilidade das taxas de crescimento entre indivíduos dentro de uma população é um parâmetro evolutivamente relevante, pois pode servir de base para estratégias de sobrevivência como cobertura de apostas2,3,4,5,6. Portanto, ensaios que permitem medições altamente precisas de fenótipos de crescimento e suas distribuições são fundamentais para o estudo de microrganismos. O ensaio de crescimento da microcolônia descrito aqui pode gerar medições individuais de taxa de crescimento para ~105 microcolônias por experimento. Este ensaio fornece, portanto, um protocolo poderoso para estudar a genética evolutiva da levedura e a genômica. Ele se presta particularmente bem a testar como a variabilidade dentro de populações de células únicas geneticamente idênticas é gerada, mantida e contribui para a aptidão populacional7,8,9,10.
O método descrito aqui (Figura 1) usa imagens de campo brilhante de baixa ampliação capturadas periodicamente de células crescendo em mídia líquida em uma placa de fundo de vidro de 96 ou 384 poços para rastrear o crescimento em microcolônias. As células aderem à lectina concanavalina A, que reveste a parte inferior da placa de microscópio, e formam colônias bidimensionais. Como as microcolônias crescem em uma monocamada, a área de microcolonia é altamente correlacionada com o celular número7. Portanto, estimativas precisas da taxa de crescimento da microcolonia e do tempo de defasagem podem ser geradas com software personalizado de análise de imagem que rastreia a taxa de mudança da área de cada microcolonia. Além disso, a configuração experimental pode monitorar as abundâncias e até mesmo as localizações subcelulares de proteínas fluorescentes rotuladas expressas nessas microcolônias. O processamento a jusante de dados deste ensaio de crescimento de microcolonia pode ser alcançado por meio de análise personalizada ou por softwares de análise de imagem existentes, como Processing Images Easily (PIE)11, um algoritmo para reconhecimento robusto da área de colônia e análise de crescimento de alto rendimento a partir de imagens de baixa ampliação e brightfield, que está disponível via GitHub12.
Como as estimativas de taxa de crescimento derivadas do ensaio de crescimento da microcolonia são geradas a partir de um grande número de medições de uma única colônia, elas são extremamente precisas, com erros padrão várias ordens de magnitude menores do que as próprias estimativas para um experimento de tamanho razoável. Portanto, o poder do ensaio para detectar diferenças de taxa de crescimento entre diferentes genótipos, tratamentos ou condições ambientais é alto. O formato multiwell-plate permite que inúmeras combinações diferentes de ambiente e genótipo sejam comparadas em um único experimento. Se as cepas expressam com constitutivamente diferentes marcadores fluorescentes, elas podem ser misturadas no mesmo poço e distinguidas pela análise de imagem subsequente, o que poderia aumentar ainda mais a energia, permitindo a normalização dos dados bem-por-bem.
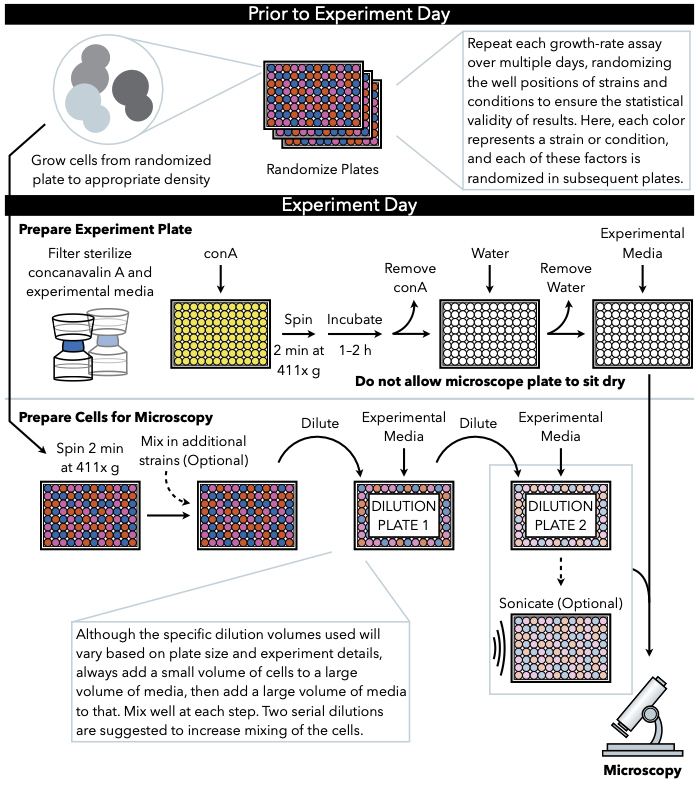
Figura 1: Representação esquemática do protocolo. Este protocolo segue dois passos principais, que são a preparação da placa experimental e a preparação das células para a imagem. A randomização das placas e o crescimento das células devem ser realizados antes e levando ao dia do experimento. A mistura repetida de células em cada etapa durante a diluição é imperativa nas etapas até o revestimento e, portanto, a preparação da placa experimental primeiro é recomendada para que ela esteja pronta para chapeamento imediatamente após a conclusão da diluição celular. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Preparação de placas aleatórias (antes do Dia do Experimento)
- Planeje as cepas e condições a serem testadas com o ensaio de crescimento. Neste ponto, atribuem aleatoriamente cepas e condições a qualquer poço.
NOTA: Ao considerar a configuração da placa, é aconselhável incluir mais de uma réplica por tensão e condição de crescimento em uma única placa para explicar o ruído bem relacionado nas medições. Consulte Discussão para obter mais detalhes. - Computacionalmente randomizar a localização de cada cepa e condição ambiental para réplicas de placas que serão executadas em dias diferentes.
- Cultivar todas as células que serão usadas no experimento para saturação em extrato de levedura-peptone-dextrose (YEPD; 2% glicose) médio em um agitador a 30 °C (ou qualquer outra temperatura apropriada).
- Crie as placas de estoque aleatórias manualmente ou com um robô de manuseio de líquidos. Adicione 10 μL das células saturadas designadas a cada poço de uma placa de cultura de tecido estéril do fundo U. Se várias cepas serão testadas em um único poço, não as combine neste momento; esta combinação será feita pouco antes das diluições celulares no dia do experimento para garantir que todas as cepas estejam nas concentrações corretas quando banhadas como células fundadoras da microcolonia.
- Adicione 10 μL de 30% de glicerol a cada poço de cada placa. Pipet para cima e para baixo para que as células e o glicerol se tornem bem misturados.
- Sele cada placa com uma tampa de papel alumínio e congele imediatamente a -70 °C até estar pronto para usar.
NOTA: É importante criar todas as placas aleatórias no mesmo dia, e congelá-las, para que as condições de pré-crescimento das células em cada placa tenham sido idênticas e não gerem variação técnica no ensaio de taxa de crescimento.
2. Pré-Crescimento da Levedura
NOTA: Normalmente, isso começa antes do dia do experimento e é altamente dependente da questão experimental. Consulte Discussão para obter detalhes.
- Retire uma placa de caldo (levedura de 10 μL, 10 μL glicerol por poço) do congelador -70 °C e adicione 180 μL da mídia a ser usada para o experimento. Se o experimento for realizado usando meios limitantes de nutrientes, não pré-cresça levedura para saturação na mídia limitante de nutrientes, pois a esporulação da levedura pode ocorrer. Em vez disso, pré-crescer em mídia não limitante.
- Cresça levedura enquanto treme a 30 °C. Considere se executar o ensaio começando com células em fase de registro ou em fase estacionária para determinar se a diluição das células várias vezes antes do experimento será necessária. Se as cepas ou condições de levedura no ensaio tiverem taxas de crescimento significativamente diferentes, então um período pré-crescimento de dois dias será necessário para que todas as condições diferentes cheguem à fase estacionária.
3. Configuração do microscópio
- Preparação da placa de microscópio
- Certifique-se de que a incubadora de microscópio está ligado e aquecendo a câmara de microscópio à temperatura de crescimento desejada para as condições experimentais. Para experimentos padrão usando células de saccharomyces cerevisiae, a incubadora deve aquecer a câmara de microscópio a 30 °C para garantir que as condições de crescimento das células estejam corretas durante o ensaio da taxa de crescimento.
- Higienize a bancada, pipetas e outras ferramentas com 70% de etanol. Recupere uma placa de microscópio e coloque-a no banco em cima de uma limpeza sem fiapos e estáticas.
NOTA: Nunca toque na parte inferior da placa do microscópio, mesmo com luvas, e sempre coloque a placa de microscópio em cima de uma limpeza sem fiapos e estáticas sempre que tocar em qualquer superfície. Isso evita que manchas ou arranhões impeçam as medições da taxa de crescimento uma vez que o experimento esteja sendo imagedo. - Descongele 5 mL de concanavalina 5x Uma solução, diluir para 1x com água e filtrar esterilizar através de uma seringa equipada com um filtro de 0,2-μm.
- O filtro esteriliza todos os outros líquidos que serão usados no ensaio com um filtro de 0,2 μm, incluindo mídia experimental, para remover quaisquer cristais ou detritos que possam ter se materializado nas soluções. A presença de cristais reduziria a qualidade das imagens de microscopia.
- Pipet 200 μL de concanavalina Uma solução em cada poço da placa de microscópio.
- Centrifugar a placa por 2 min a 411 x gravidade(g) com um lenço de fiapo e estático sob a placa, para garantir que a solução concanavalina Uma solução cobre uniformemente o fundo de cada poço e que não há bolhas de ar.
- Cubra a placa com a tampa e deixe-a sentada por 1-2 h. O tempo exato em que a placa é flexível, mas é importante ser consistente entre diferentes corridas do experimento.
- Remova toda a solução concanavalina Uma da placa por sucção ou descarregando-a à força na pia ou em um recipiente. Tenha cuidado para não tocar na parte de vidro da placa. É aceitável que algumas gotas de concanavalina uma solução permaneçam nos poços.
- Lave os poços da placa de microscópio adicionando 400 μL de água estéril. Remova a água como feito com a concanavalina A na etapa anterior. Não deixe a placa secar.
- Adicione imediatamente 185 μL de mídia de crescimento experimental na placa. 15 μL de células corretamente diluídas serão adicionadas a esta placa.
- Diluição celular de levedura
NOTA: As etapas abaixo descrevem uma diluição da levedura de uma cultura saturada (aproximadamente 108 células/mL) 400 vezes para alcançar uma concentração de 250.000 células/mL, 15 μL dos quais serão diluídos em 400 μL na placa de fundo de vidro, dando um número final de aproximadamente 4000 células por poço em uma placa de 96 poços. Se usar uma placa de 384 poços, o número final de células por poço deve ser de aproximadamente 700 e as diluições devem ser ajustadas em conformidade. Essa razão deve ser ajustada para células coletadas em fase de registro, crescendo em meios pré-crescimento mais ricos ou mais pobres, ou de diferentes cepas. A densidade final das células por poço deve ser reduzida ao executar o ensaio da taxa de crescimento por períodos de tempo superiores a 10 h.- Configure duas placas de cultura de 96 poços para diluições seriais: rotule como placa 1 e 2, e adicione 90 μL de mídia de crescimento experimental (ou seja, a mídia em que a levedura crescerá no microscópio) a cada placa de diluição serial.
NOTA: Independentemente da diluição final, pelo menos duas diluições seriais de células são recomendadas, em cada uma das quais um pequeno volume de levedura é pipetado em um volume maior de mídia experimental e, em seguida, um grande volume de mídia experimental é misturado vigorosamente com uma tubulação (como nas etapas 3.2.5 e 3.2.6 abaixo). - Recupere a placa de células do pré-crescimento e centrifugar a placa por 2 min a 411 x g.
NOTA: É muito importante não contaminar diferentes poços na placa. O objetivo desta centrifugação antes de remover a folha que cobre as placas é garantir que as gotículas cheias de leveduras de um poço não voem da folha e acabem em outros poços. Tenha cuidado para nunca inclinar ou agitar as placas para evitar que o fermento entre em contato com a folha cobrindo após a centrifugação. - Retire cuidadosamente a folha e resuspend as células, encanar vigorosamente as células com uma tubulação definida para aproximadamente metade do volume total da placa enquanto move a tubulação ao redor do poço para misturar. Verifique se todas as células foram resuspendidas do fundo dos poços.
- Se múltiplas cepas dentro de poços individuais forem usadas, as cepas devem ser misturadas neste momento na razão necessária para o experimento. Se uma cepa de referência for usada para gerar medições de taxa de crescimento, a razão de referência à cepa de teste deve ser de 1:1.
- Pipet 10 μL de levedura da mídia de crescimento para a placa de diluição 1. Adicione 100 μL de mídia de crescimento experimental a cada poço a um volume final de 200 μL por poço. Pipet para cima e para baixo vigorosamente.
- Pipet 10 μL de levedura da placa 1 para a placa 2. Adicione 100 μL de mídia de crescimento experimental a cada poço, e pipeta para cima e para baixo vigorosamente para misturar.
NOTA: Essas etapas de diluição são fundamentais para ajudar a separar os aglomerados de levedura que estão presos juntos no final do estágio pré-crescimento e garantir que aproximadamente o número igual de células de levedura acabe em cada poço. Ter um número consistente de levedura em cada poço ajuda a remover ruídos experimentais e vieses em medições de taxa de crescimento (ver Resultados Representativos).
- Configure duas placas de cultura de 96 poços para diluições seriais: rotule como placa 1 e 2, e adicione 90 μL de mídia de crescimento experimental (ou seja, a mídia em que a levedura crescerá no microscópio) a cada placa de diluição serial.
- Sonication
NOTA: A sônicação é opcional, e só precisa ser realizada para cepas de leveduras que tenham alta propensão a aderir umas às outras (por exemplo, algumas cepas selvagens). Para cepas de laboratório, a sônica geralmente não é necessária e pode ser ignorada procedendo à etapa 3.4.- Higienize uma cabeça sônica de 96 pinos com 70% de etanol colocando-a em uma placa de 96 poços cheia de 70% de etanol e seca com um lenço de fiapo e estático.
- Estabeleça um programa de sônica que seja suficientemente forte para quebrar células de levedura floculadas, mas não mate células ou cause respostas elevadas de estresse. Alguns testes podem ser necessários para identificar o melhor programa de sônica para um determinado experimento. O programa de sônica utilizado neste experimento é: amplitude = 10, tempo de processo = 10 s, pulse-on = 1 s, pulse-off = 1 s. Este programa exato provavelmente não é aplicável a todos os sonicadores, por isso os testes são sugeridos antes do dia do experimento.
- Misture o fermento na placa de diluição serial 2 mais uma vez, pipetando para cima e para baixo vigorosamente cinco vezes.
- Coloque a placa de diluição 2 na plataforma e fixe-a com os pinos do sonicator na suspensão da célula, mas não toque na parte inferior da placa. Execute o programa de sônica usando proteção auditiva apropriada.
- Após a ção do programa, limpe a cabeça do sonicator com 70% de etanol e depois com água, e depois prossiga imediatamente para a preparação da placa de microscópio para que as células não se desembrareça novamente.
- Prepare a placa para microscópio:
- Pipet 15 μL de levedura da placa de diluição serial 2 na placa de microscópio a um volume de 200 μL. Adicione 200 μL de mídia de crescimento experimental a cada poço a um volume final de 400 μL por poço, e pipet para cima e para baixo vigorosamente para misturar.
- Cubra a placa com uma membrana respirável. É importante selar bem a placa com esta membrana, por exemplo, usando um rolo de borracha.
- Para aderir as células de levedura à concanavalina A na superfície do vidro, centrifugar a placa com um lenço de fiapos e estáticos abaixo dele por 2 min a 411 x g.
- No microscópio, limpe a parte superior e inferior da placa com uma limpeza sem fiapos e estáticas, e sopre ar comprimido na placa para se livrar dos detritos.
- Coloque a placa sobre o microscópio, certificando-se de que está nivelado e que o poço A1 está no canto superior esquerdo.
4. Medições de taxa de crescimento da microscopia de lapso de tempo
NOTA: Durante a microscopia de lapso de tempo, as seguintes características são controladas por computador: x, y e z, persianas e filtros de fluorescência. Um sistema de foco automático baseado em hardware é ideal para evitar a deriva do avião focal durante a imagem de lapso de tempo. Alternativamente, um loop de foco automático baseado em software pode ser usado. Para manter a umidade na câmara de microscópio, é aconselhável manter um béquer com água purificada na câmara durante toda a duração do experimento.
- Crie uma lista de posições (x,y) para imagem, de modo que cada poço de placa de microscópio seja totalmente imagem. Evite imagens sobrepostas para que nenhuma célula seja analisada várias vezes.
- Imagem em brightfield com iluminação diascópica (DIA) em uma ampliação de 15x. Definir exposição a ~5 ms.
- Amplie a imagem digitalmente para que as células sejam claramente visíveis. Use os botões de foco para identificar o foco ideal para o experimento nos quatro poços no canto da placa e em um bem no centro da placa. Concentre-se de forma a obter o máximo contraste das células.
- Defina a posição z (ou posição de foco automático) para que o experimento seja uma média das posições de foco automático z identificadas para cada um desses poços. Se a placa do microscópio for bem feita e a parte inferior do vidro não tiver defeitos, as posições de foco ideais devem ser semelhantes para cada poço.
NOTA: Ao analisar imagens com o pipeline de análise de imagens Facilmente (PIE) do Pipeline de Análise de Imagens de Processamento11,12, é útil que as células fiquem ligeiramente fora de foco no microscópio para que haja uma borda escura fora da célula e um interior de cor clara, o que auxilia no reconhecimento preciso das colônias e estimativas de tamanho. - Se usar cepas fluorescentes, identifique os canais e as exposições com as quais a imagem, garantindo que nenhum pixel seja superexposto. Ao definir o tempo de exposição para canais fluorescentes, desligue o modo de "captura ao vivo" no microscópio para evitar expor as células à excitação da fluorescência por longos períodos de tempo, pois isso pode tanto fotoblear as células quanto causar estresse.
- Configure a aquisição de sequência de tempo para capturar imagens no intervalo de tempo desejado pelo tempo desejado.
- Executar o experimento.
Resultados
A novidade deste protocolo é que a taxa de crescimento pode ser calculada para células individuais dentro de uma população, rastreando seu crescimento em microcolônias através de imagens de lapso de tempo(Figura 2A). Como as microcolônias crescem por muitas horas de forma planar devido à presença de concanavalina A, suas áreas podem ser rastreadas ao longo do experimento, e um ajuste linear à mudança no tronco natural da área ao longo do tempo pode ser usado para calcular a taxa...
Discussão
O protocolo descrito aqui é um ensaio versátil que permite que o crescimento celular e a expressão genética sejam monitorados simultaneamente ao nível de microcolônias individuais. A combinação dessas duas modalidades produz insights biológicos únicos. Por exemplo, trabalhos anteriores usaram este ensaio para mostrar uma correlação negativa entre a expressão do gene TSL1 e a taxa de crescimento da microcolonia em células selvagens isogênicas medindo simultaneamente7,
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos a Naomi Ziv, Sasha Levy e Shuang Li por suas contribuições para o desenvolvimento deste protocolo, David Gresham por equipamentos compartilhados, e Marissa Knoll por ajuda na produção de vídeo. Este trabalho foi apoiado pelo Instituto Nacional de Saúde do Instituto R35GM118170.
Materiais
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Referências
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados