需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
利用时间分辨蛋白诱导的荧光增强来鉴定稳定的局部构象,一次一α-突触核蛋白单体
摘要
时间分辨单分子蛋白诱导的荧光增强是一种有用的荧光光谱接近传感器,对蛋白质的局部结构变化敏感。在这里,我们展示了它可用于揭示α-突触核蛋白中稳定的局部构象,当使用较长距离的FRET标尺测量时,这也称为球状非结构化和不稳定。
摘要
使用光谱标尺来跟踪单个生物分子及其动力学的多种构象,彻底改变了对结构动力学及其对生物学的贡献的理解。虽然基于FRET的标尺报告了3-10nm范围内的染料间距离,但其他光谱技术,如蛋白质诱导荧光增强(PIFE),报告了染料与蛋白质表面在较短的0-3nm范围内的接近度。无论选择哪种方法,它在测量自由扩散的生物分子时的使用一次一个检索实验参数的直方图,产生单独的中心分布的生物分子亚群,其中每个亚群代表在毫秒内保持不变的单个构象,或者相互转换速度远快于毫秒的多个构象,因此平均输出子群。在单分子FRET中,直方图中报告的参数是染料间FRET效率,一种内在无序的蛋白质,例如缓冲液中的α-突触核蛋白单体,先前被报道为表现出多个构象的单个平均亚群快速相互转化。虽然这些过去的发现取决于基于FRET的标尺的3-10nm范围,但我们试图使用单分子PIFE对这种蛋白质进行测试,在那里我们跟踪位点特异性sCy3标记的α突触核蛋白的荧光寿命一次一个。有趣的是,使用这种较短距离的光谱接近传感器,sCy3标记的α-突触核蛋白表现出几个具有明显不同平均寿命的寿命亚群,在10-100 ms内相互转换。这些结果表明,虽然α-突触核蛋白可能在全球范围内紊乱,但它仍然获得了稳定的局部结构。总之,在这项工作中,我们强调了使用不同光谱接近传感器的优势,这些传感器一次跟踪一个生物分子的局部或全局结构变化。
引言
在过去的二十年中,基于单分子荧光的方法已成为测量生物分子1,2的强大工具,探索不同的生物分子参数如何分布以及它们如何在亚毫秒分辨率3,4,5下动态地在这些参数的不同亚群之间相互转换。这些技术中的参数包括FRET测量的能量传递效率6,7,荧光各向异性8,9,荧光量子产率和寿命10,11,作为不同荧光猝灭12或增强13机制的函数。其中一种机制,更广为人知的是蛋白质诱导荧光增强(PIFE)14,引入了荧光量子产率和寿命的增强,作为激发态时荧光团自由异构化的空间位阻的函数,由染料附近的蛋白质表面引起14,15,16,17,18,19.FRET和PIFE都被认为是光谱尺或接近传感器,因为它们的测量参数与被测量的标记生物分子内的空间测量直接相关。虽然FRET效率与3-10 nm20范围内一对染料之间的距离有关,但PIFE跟踪荧光量子产量的增加或寿命与染料与附近蛋白质表面之间的距离在0-3 nm19范围内有关。
单分子FRET已被广泛用于为许多不同的蛋白质系统提供结构见解,包括内在无序蛋白质(IDPs)21,如α-突触核蛋白(α-Syn)22。α-Syn在与不同的生物分子结合后,在不同的条件下可以形成有序的结构23,24,25,26,27,28,29,30。然而,当未结合时,α-Syn单体的特征在于具有高构象异质性,具有快速相互转化的构象31,32。
α-Syn的构象以前已经使用各种不同的技术进行了研究,这些技术有助于识别这种高度异质和动态的蛋白质系统的构象动力学33,34,35,36,37,38,39。有趣的是,缓冲液中α-Syn的单分子FRET(smFRET)测量报告了单个FRET群体39,40,这是构象的时间平均动态相互转换的结果,其时间比α-Syn通过共聚焦点的典型扩散时间快得多(相对于典型的毫秒扩散时间,时间快到几微秒,甚至比这更快)40, 41.然而,使用具有3-10nm距离灵敏度的FRET光谱标尺有时只能报告小蛋白质(如α-Syn)的整体结构变化。利用具有较短距离灵敏度的光谱接近传感器进行单分子测量有可能报告局部结构的动力学。在这里,我们对α-Syn进行单分子PIFE测量,并确定荧光寿命的不同亚群映射到不同的局部结构,它们之间的过渡速度慢至100 ms。这项工作总结了自由扩散的α-Syn分子的时间分辨smPIFE测量值,一次一个,在缓冲液中,以及当与基于SDS的膜结合时作为短程单分子光谱接近传感器。
研究方案
1. 质粒转化
- 制备0.5 L SOC培养基
- 称取10克色蛋白酮,2.5克酵母提取物,0.25克氯化钠(NaCl),0.1克氯化钾(KCl)。
- 加入双蒸馏水(DDW),直至总体积为0.5升。
- 通过加入氢氧化钠(NaOH)调节至pH 7。
- 准备100 mL的配分试样和高压灭菌器。
- 使用前,将0.5毫升无菌氯化镁(MgCl2)和1.8毫升无菌葡萄糖加入100毫升SOC中。
- 在冰上解冻BL21(DE3)感受态 大肠杆菌 细胞。
- 轻轻混合感受态细胞。
- 准备两个 15 mL 试管。将一个标记为转化反应管,另一个标记为控制管。
- 等分100μL感受态细胞到管中。
- 将1μL质粒以0.1ng / μL的浓度加入转化反应管中。
- 在42°C水浴中加热SOC培养基,以便在步骤1.8中使用。
- 将两个管子在42°C水浴中加热脉冲 45秒。(关键步骤!
- 向每个管中加入900μL加热的SOC培养基。
- 将管在37°C下孵育45分钟,以225rpm的速度振荡。
- 使用无菌细胞扩增器将200μL具有转化质粒的细胞分散到含有100μg/ mL氨苄西林的LB(Luria-Bertani)琼脂平板上。
- 使用无菌扩散器 (阳性对照)将200μL具有转化质粒的细胞分散到一个没有氨苄西林的LB琼脂平板上。
- 使用无菌扩散器(阴性对照)将200μL对照细胞(不含质粒)分散到一个含有100μg/ mL氨苄西林的LB琼脂平板上。
- 将板在37°C下孵育过夜。
2. 蛋白质制备
- 重组α-突触核蛋白表达和纯化
- 选择单个菌落,并在100mL高压灭菌的LB(Luria-Bertani)液体培养基中生长,其中含有100μg/ mL氨苄西林,在37°C( 发酵剂)下。
- 准备四个2L锥形瓶,每个烧瓶含有1L高压灭菌的LB培养基和100μg/ mL氨苄西林 (大剂量接种)。
- 测量细胞溶液的光密度(λ = 600 nm处的外径)。当发酵剂的细胞密度达到0.6-0.7 的OD λ = 600nm时,每1 L LB培养基加入10 mL发酵剂溶液。
- 在37°C和200rpm速度的培养箱振荡器中培养细菌培养基。
- 当细胞密度达到ODλ=600nm 的0.6-0.8时,通过每1L生长培养基加入异丙基β-d-1-硫代乳糖吡喃苷(IPTG)至终浓度1mM来诱导蛋白质表达。
注意:将0.96gIPTG溶解在4mLDDW中,每1L细菌生长加入1mL所得IPTG溶液。 - 将细胞培养4小时,并通过以5,170×g的速度在50 mL管中离心它们来收集它们,持续时间为8分钟。
注意:在每一轮中,丢弃上清液,保留沉淀并再次填充具有细菌生长的相同管。 - 将细菌沉淀储存在深度冷冻储存(例如,-80°C)中,以备第二天使用。
- 第二天,制备200毫升裂解缓冲液:40%(w / v)蔗糖和缓冲液A,其中包括30mM Tris-HCl,2mM乙二胺四乙酸(EDTA),2mM二硫甲状腺素(DTT),pH 8.0。
- 向每个管中加入25mL裂解缓冲液,并加入细菌沉淀。
- 将沉淀(使用无菌塑料巴斯德移液管)重新悬浮在裂解缓冲液中(见2.1.8)。
- 在冰上用搅拌器磁铁准备无菌的250 mL锥形瓶。
- 加入5mL裂解缓冲液,然后将均质的再悬浮细胞转移到其中。
- 在室温下以200rpm搅拌细胞20分钟。
- 将溶液分成50mL管,并在4°C下以17,400×g的速度离心30分钟。
- 丢弃上清液。
- 准备无菌的250 mL锥形瓶,在冰上用搅拌器磁铁。
- 将15 mL溶解缓冲液(90 mL冷却缓冲液A和37μL溶解超过溶解度限值并过滤的MgCl2) 加入每个管中。使用无菌塑料巴斯德移液器并溶解沉淀。
- 当溶液的一部分变得均匀时,将其移至冰上搅拌的锥形瓶中。
- 确定危险品中的溶液卷。
- 每1mL溶液加入10mg硫酸链霉素(在加入之前,将硫酸链霉素溶解在2mL缓冲液A中)。
注意:执行此步骤是为了去除DNA和核糖体。 - 在室温下搅拌溶液10-20分钟。
- 将溶液分成50 mL管,以4°C和20,700×g的速度离心30分钟。
- 收集上清液并丢弃沉淀。
- 用搅拌器在冰上准备一个无菌的250 mL锥形瓶。
- 使用0.22μm注射器过滤器过滤上清液,放入干净的锥形瓶中。
- 确定溶液体积,每1mL溶液称量0.3g硫酸铵。
- 逐渐地,将硫酸铵加入到搅拌的溶液中。
注意:执行此步骤是为了沉淀蛋白质。当硫酸铵结合蛋白质时,溶液变得模糊不清,显示为白色。 - 在室温下搅拌30分钟。
- 将溶液分成50mL管,并在4°C和20,700g的速度下离心30分钟。
- 小心地丢弃上清液并保持沉淀。
- 用20 mL缓冲液A溶解沉淀。
- 将所有溶液统一到一个管中,然后测量溶液的紫外线吸收光谱。
注意:如果光谱显示聚集特征(吸收波长高于300nm),请继续下一步。如果没有,请跳过下一步并转到步骤 2.1.34。
注:根据紫外光谱计算的聚集指数(A.I.)为ODλ=350nm/ (ODλ=280nm - ODλ=350nm)×100。40 - 使用100 kDa截止离心管,并在4°C下以3,590×g的速度离心15分钟。收集通过过滤器的液体。
- 将溶液在4°C下透析过夜,对缓冲液A,使用具有3.5 kDa截止值的透析袋。
- 进行另一轮透析,持续时间至少为4小时。
- 将透析样品上样到1 mL MonoQ阴离子交换柱中。使用pH 7.5的30mM Tris盐酸盐(Tris-HCl)缓冲液作为洗涤缓冲液,使用30 mM Tris-HCl缓冲液pH 7.5和500 mM NaCl作为洗脱缓冲液。
- 将样品注入色谱柱。然后用20柱体积(CV)的0%洗脱缓冲液和100%洗涤缓冲液洗涤,以除去未结合的蛋白质。
- 用7 CV的30%洗脱缓冲液洗涤。
- 使用梯度从30%变为100%的洗脱缓冲液洗脱α-Syn蛋白样品,持续30 CV,流速为1.5 mL / min。
注:α-Syn以46-66 CV的电导率20-24 mS/cm洗脱,这指的是38-50%的洗脱缓冲液。 - 收集主洗脱峰的分数。在用考马斯蓝或快速透明带染色溶液染色后,通过在15%十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)中运行样品来验证α-Syn的存在。预计能段为15 kDa。
- 统一相关馏分,并在缓冲液A下使用透析袋在4°C下以3.5 kDa的截止值进行透析过夜。
- 准备蛋白质样品的等分试样并将其储存在-20°C。
- Cy3 标记的α-突触核蛋白
- 通过在室温下将DTT加入至2mM的终浓度持续1小时,减少α-Syn A56C突变体中半胱氨酸残基的硫醇。
- 通过使用3.5 kDa截止透析袋进行两轮透析来去除DTT。对于第一轮,对30 mM Tris-HCl pH 8.0和2 mM EDTA进行透析。对于第二轮,对50 mM HEPES pH 7.2和2 mM EDTA进行透析。
- 通过将Tris(2-羧乙基)膦盐酸盐(TCEP)加入蛋白质样品中,在室温下持续30分钟,将半胱氨酸残基降低至50μM的终浓度。
注意:TCEP是一种非巯基还原剂,因此它可以保持硫醇基团的还原而不与染料反应。我们从1 M TCEP的储备溶液中加入0.5μL。 - 计算最终反应体积1mL所需的蛋白质量。
注意:以下是我们使用的计算示例:
20 μM 最终蛋白浓度 × 1 mL 最终体积 = 56 μM 初始蛋白浓度 × V 蛋白加入 - 计算最终反应体积1 mL所需的染料量。单个半胱氨酸的染料标记应用过量染料进行,染料:蛋白质摩尔比至少为3:1。
注意:我们使用的计算示例:
60 μM 最终染料浓度 × 1 mL 最终体积 = 370 μM 初始染料浓度 × 要添加的 V 染料 - 计算将总反应体积调节至1 mL所需的透析液中缓冲液的量。
注:计算示例:1 mL - (V根据步骤 2.2.4计算 + V根据步骤 2.2.5 计算) - 在搅拌器顶部用磁铁制备反应瓶。
- 首先,加入计算出的蛋白质和缓冲液的量。之后,加入计算量的染料(磺酰基-Cy3马来酰亚胺)。
- 将反应保持在室温下,在黑暗中持续3-5小时。
- 通过加入2 mM DTT终止反应,并继续持续1小时。
- 对缓冲液A进行三轮透析,使用具有3.5 kDa截止值的透析袋从溶液中除去多余的游离染料。
- 将样品加载到尺寸排除柱上,以进一步将标记的α-Syn与游离染料分离。
- 通过测量染料的吸收来确定纯标记α-Syn的浓度(磺基-Cy3的吸收系数在λ= 548nm处为162,000M-1 cm -1)。
- 如果需要,使用真空浓缩器(例如 SpeedVac)浓缩纯标记的 α-Syn 溶液。然后,使用具有3.5 kDa截止值的透析袋对缓冲液A进行一轮透析,并再次确定标记蛋白质的浓度。
- 准备标记蛋白质样品的等分试样并将其储存在-20°C。
3. 测量
- smPIFE实验设置
注:使用以下基于共聚焦的设置或类似设置。- 使用脉冲激光源(在我们的例子中,λ= 532 nm皮秒脉冲激光器,脉冲宽度约为100 ps FWHM),以合适的重复速率(在我们的例子中为20 MHz)工作,并路由到时间相关单光子计数(TCSPC)卡的SYNC,作为磺基-Cy3(sCy3)激发的来源。
- 使用在532nm处具有高反射率的二向色镜,将激发和散射与荧光分开。
- 在准直的发射光束被透镜聚焦之后,在发射光束被另一个透镜重新准直之前,在发射光束的焦点处使用直径为100μm的针孔。
- 使用带通滤光片(在本例中为 585/40 nm)进一步过滤来自其他光源的 sCy3 荧光。
- 使用检测器(在我们的例子中是单光子雪崩二极管或混合光电倍增管)路由(在我们的例子中是通过4比1路由器)到TCSPC模块(在我们的例子中是Becker&Hickl SPC-150)来检测荧光。
注意:在我们的例子中,数据采集是通过VistaVision软件(ISSTM)以时间标记时间解析(TTTR)文件格式执行的。
- smPIFE样品制备
- 在测量缓冲液中制备25 pM sCy3标记的α-Syn:10 mM乙酸钠,10 mM磷酸二氢钠,10 mM甘氨酸pH 8.0,20 mM NaCl,10 mM半胱胺和1mM 6-羟基-2,5,7,8-四甲基色蛋白-2-羧酸(TROLOX),在低蛋白结合管中。
注意:如果在存在SDS囊泡的情况下测量α-Syn,则还要添加适当的SDS量。 - 用100μL 1mg / mL牛血清白蛋白(BSA)冲洗18腔显微镜盖玻片,持续1分钟,然后取出BSA。
- 将100μL的25 pM sCy3标记的α-Syn样品加入盖玻片中的腔室中。
- 使用所述设置对样品进行 smPIFE 测量,如下步骤所示。
- 在测量缓冲液中制备25 pM sCy3标记的α-Syn:10 mM乙酸钠,10 mM磷酸二氢钠,10 mM甘氨酸pH 8.0,20 mM NaCl,10 mM半胱胺和1mM 6-羟基-2,5,7,8-四甲基色蛋白-2-羧酸(TROLOX),在低蛋白结合管中。
- smPIFE数据采集
- 使用高数值孔径水浸式物镜(在本例中为奥林巴斯UPLSAPO 60x N.A. 1.2),并在物镜顶部加入一滴超纯水。
- 将盖玻片固定在载物台室中,并将其安装在显微镜载物台的顶部。
- 将物镜向上提起,直到物镜顶部的水滴涂抹在盖玻片的底部。
- 打开激光快门并将物镜向上,同时检查CMOS相机上的图案,收集从样品中散射的光。观察Airy环图案:第一个表示水玻璃界面处的焦点,然后第二个代表玻璃与样品溶液之间界面处的焦点。
- 将物镜的高度再增加75μm,将激光焦点深入溶液,以最大限度地减少来自盖玻片玻璃表面的自动荧光。
- 将物镜处的激光功率调谐至~100 μW。
- 在预定义的时间内开始采集检测到的光子(在我们的测量中为2小时)。
注意:大多数采集信号应具有与仅测量缓冲器时采集的速率相似的速率(以每秒计数,cps为单位测量);毫秒级数据应显示稀缺的光子突发事件,大多数箱的平均速率与典型的探测器背景速率相当(在我们的例子中为<1,000 cps)。
4. 脉冲群分析
- 原始数据转换
注意:数据通常存储在二进制文件中,其格式由制造TCSPC卡的公司预定义(在我们的例子中为.spc文件)。- 使用软件套件phconvert(https://github.com/Photon-HDF5/phconvert)将原始数据文件转换为photon-HDF5通用文件格式42。调用原始数据文件作为输入,并使用phconvert套件中的相应代码转换为原始数据 .hdf5 文件(在本例中,将 ns-ALEX Becker-Hickl 文件转换为 Photon-HDF5.ipynb Jupyter 笔记本)。
注:转换为.hdf5文件包括:(1)确定原始数据中的相关光子流(即相关探测器ID的注册光子的光子流);(2)相关光子的纳时(光子检测次数相对于激发同步时间);和 (3) 添加了元数据。
- 使用软件套件phconvert(https://github.com/Photon-HDF5/phconvert)将原始数据文件转换为photon-HDF5通用文件格式42。调用原始数据文件作为输入,并使用phconvert套件中的相应代码转换为原始数据 .hdf5 文件(在本例中,将 ns-ALEX Becker-Hickl 文件转换为 Photon-HDF5.ipynb Jupyter 笔记本)。
- 突发搜索和选择使用 FRETbursts43
注:smPIFE测量的所有光子-HDF5原始数据文件以及总结原始数据分析的代码都存储在Zenodo(https://doi.org/10.5281/zenodo.4587698)中。以下所有步骤都很详细,并显示在Jupyter笔记本中,也在Zenodo存储库链接中提供。- 打开Jupyter Notebooks(在我们的例子中是在Anaconda框架内)。
- 打开笔记本 smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (Final notebook).ipynb (可以在 Zenodo 链接中找到)。
- 加载 烦恼爆发 。
- 加载光子 HDF5 数据文件。
- BG速率评估:使用光子间时间的直方图,计算光子HDF5数据文件中每采集30秒的数据的背景(BG)速率。
注意:以下步骤描述了使用滑动窗口算法44,45,46的突发搜索。 - 移动m=20个连续光子的时间窗,一次一个光子。
- 仅当瞬时光子速率
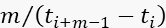 ()至少比数据采集期间的BG速率大11倍时,才收集光子数据。
()至少比数据采集期间的BG速率大11倍时,才收集光子数据。
注意:突发是由所有连续的光子构成的,这些光子是通过一次滑动一个光子(步骤4.2.6)并同意光子速率准则(步骤4.2.7)来收集的。 - 计算以下突发特性:
突发大小:突发中的光子量。
突发持续时间:突发中最后一个光子和第一个光子检测时间之间的时间差。
突发亮度:突发中瞬时光子速率的最大值。
突发分离:连续突发之间的时间间隔。
注:以下几点描述了进一步的突发选择过程。 - 绘制突发亮度值(突发中最高的瞬时光子速率)的直方图,事件轴以对数刻度表示。
- 将突发亮度阈值定义为直方图显示衰减模式的最小突发亮度值。
- 选择亮度值大于连拍亮度阈值的连拍。
注:接下来的步骤描述了突发平均荧光寿命。 - 绘制所有选定突发中所有光子的光子纳米时间直方图,光子计数轴以对数刻度表示。
- 将纳米时间阈值定义为光子纳米时间直方图表现出衰减模式的最小纳米时间值。
- 仅选择纳米时间大于纳米时间阈值的光子。
- 计算所有选定光子纳时的代数平均值。
- 从光子纳米时间代数平均值中减去纳米时间阈值。结果是突发的平均光子纳米时间,它与平均荧光寿命成正比。
- 绘制所有突发平均荧光寿命的直方图。可能出现荧光寿命的中心分布亚群。具有低值平均值的亚群代表具有sCy3且未被空间阻滞的分子物种,而具有较高值平均值的亚群代表具有sCy3的分子物种,其空间受阻性更强。
注:后续步骤将介绍基于突发重复分析的慢突发间动态47 - 绘制突发分离时间的直方图,分离时间轴以对数刻度表示。
注意:将出现两个突发分离时间的子群:
分离时间为秒的主要亚群,表示来自不同连续测量的分子的连续爆发。
分离时间约为<100 ms的次要亚群,表示来自同一分子的连续爆发,通过共聚焦体积重复出现。 - 选择此选项可保存所有连续脉冲群,这些脉冲串的间隔时间小于定义同分子子群的最大分离时间(在我们的例子中为 <100 ms)。
- 绘制低于特定分离时间阈值的所有突发对的第一和第二次突发的平均荧光寿命的直方图或散点图
结果
作为IDP,当它不与另一种生物分子结合时,α-Syn表现出多种构象之间的结构动力学,在几微秒40甚至数百纳秒41下过渡 。当α-Syn穿过共聚焦点时,它可能会在构象之间经历数千次转变。事实上,当smFRET使用39,40时就是这种情况。在这里,我们进行smPIFE测量,以探测α-Syn的局部构象动力学。
测量记...
讨论
进行了广泛的生化和生物物理研究,以研究α-Syn的结构特征及其无序性质33,34,35,36,37,38。一些工作已经利用自由扩散的smFRET来研究无结合的α-Syn单体的分子内动力学。这些工作报告了α-Syn的高动态异质性,这导致在通过共聚焦点的典型扩散时...
披露声明
所有作者均无利益冲突。
致谢
编码A56C α-Syn突变体的pT-t7质粒是作为Asaf Grupi博士,Dan Amir博士和Elisha Haas博士的礼物送给我们的。本文得到了美国国立卫生研究院(NIH,授予R01 GM130942给E.L.作为子奖),以色列科学基金会(KillCorona - 遏制冠状病毒研究计划中的3565/20赠款),米尔纳基金和耶路撒冷希伯来大学(启动基金)的支持。
材料
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
参考文献
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。