A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניצול שיפור פלואורסצנטיות הנגרמת על-ידי חלבון בזמן כדי לזהות קונפורמציות מקומיות יציבות אחת α-Synuclein Monomer בכל פעם
In This Article
Summary
שיפור פלואורסצנטיות המושרה בחלבון המושרה בחלבון בזמן הוא חיישן קרבה פלואורסצנטי שימושי רגיש לשינויים מבניים מקומיים בחלבונים. כאן אנו מראים שניתן להשתמש בו כדי לחשוף קונפורמציות מקומיות יציבות α-Synuclein, אשר ידוע גם בשם globulared ולא יציב כאשר נמדד באמצעות סרגל FRET לטווח ארוך יותר.
Abstract
שימוש בסרגלים ספקטרוסקופיים כדי לעקוב אחר קונפורמציות מרובות של ביומולקולות בודדות והדינמיקה שלהם חוללה מהפכה בהבנת הדינמיקה המבנית ותרומתה לביולוגיה. בעוד שהסרגל מבוסס FRET מדווח על מרחקים בין צבעים בטווח של 3-10 ננומטר, טכניקות ספקטרוסקופיות אחרות, כגון שיפור פלואורסצנטיות הנגרמת על-ידי חלבון (PIFE), מדווחות על הקרבה בין צבע למשטח חלבון בטווח הקצר יותר של 0-3 ננומטר. ללא קשר לשיטת הבחירה, השימוש בו במדידת ביו-מולוקולים המפזרים בחופשיות בזה אחר זה מאחזר היסטוגרמה של הפרמטר הניסיוני המניב אוכלוסיות תת-אוכלוסיות נפרדות בחלוקה מרכזית של ביו-מוקולות, כאשר כל תת-אוכלוסייה מייצגת התאמה אחת שנותרה ללא שינוי בתוך אלפיות השניה, או קונפורמציות מרובות המתחברות הרבה יותר מהר מאלפיות שניה, ומכאן תת-אוכלוסייה ממוצעת. במולקולה אחת FRET, שבו הפרמטר המדווח בהיסטוגרמה הוא יעילות FRET בין צבעים, חלבון הפרעה מהותית, כגון מונומר α-Synuclein במאגר, דווח בעבר כמפגין תת-אוכלוסייה ממוצעת אחת של קונפורמציות מרובות המתחברות במהירות. בעוד שממצאי העבר האלה תלויים בטווח של 3-10 ננומטר של השליט מבוסס FRET, ביקשנו לשים את החלבון הזה במבחן באמצעות PIFE של מולקולה אחת, שם אנו עוקבים אחר חיי הפלואורסצנטיות של חלבוני α-Synuclein ספציפיים לאתר. מעניין, באמצעות חיישן קרבה ספקטרוסקופי לטווח קצר זה, sCy3-תווית α-Synuclein מציג מספר אוכלוסיות משנה לכל החיים עם תקופות חיים ממוצעות שונות במובהק כי interconvert ב 10-100 ms. תוצאות אלה מראות כי בעוד α-Synuclein עלול להיות מופרע ברחבי העולם, הוא בכל זאת משיג מבנים מקומיים יציבים. לסיכום, בעבודה זו אנו מדגישים את היתרון של שימוש בחיישני קרבה ספקטרוסקופיים שונים העוקבים אחר שינויים מבניים מקומיים או גלובליים ביומולקול אחד בכל פעם.
Introduction
במהלך שני העשורים האחרונים, שיטות מבוססות פלואורסצנטיות של מולקולה אחת הפכו לכלי רב עוצמה למדידת ביומולקולות1,2, בודקים כיצד פרמטרים ביומולקולריים שונים מפיצים, כמו גם כיצד הם מתחברים באופן דינמי בין אוכלוסיות משנה שונות שלפרמטריםאלה ברזולוציה תת-אלפית השנייה 3,4,5. הפרמטרים בטכניקות אלה כוללים את יעילות העברת האנרגיה במדידות FRET 6,7, פלואורסצנטיות אניזוטרופיה8,9, פלואורסצנטיות תשואות קוונטיות ואורך חיים10,11, כפונקציה של פלואורסצנטיות שונה מרווה12 או שיפור13 מנגנונים. אחד ממנגנונים אלה, הידוע יותר בשם שיפור פלואורסצנטיות הנגרמת על ידי חלבון (PIFE)14 מציג את השיפור של תשואה קוונטית פלואורסצנטית ואורך חיים כפונקציה של חסימה סטרית לאיזומריזציה חופשית של הפלואורופור כאשר במצב נרגש, הנגרמת על ידי משטחי חלבון בקרבת הצבע14,15,16,17,18,19 . הן FRET והן PIFE נחשבים לשליטים ספקטרוסקופיים או חיישני קרבה מכיוון שהפרמטר הנמדד שלהם מקושר ישירות למדידה מרחבית בתוך הביומולקול המסומן תחת מדידה. בעוד יעילות FRET קשורה למרחק בין זוג צבעים בטווח של 3-10 ננומטר20, PIFE עוקב אחר עלייה בתפוקות קוונטיות פלואורסצנטיות או תקופות חיים הקשורות למרחק בין הצבע לבין משטח של חלבון סמוך בטווח של 0-3 ננומטר19.
FRET מולקולה אחת שימשה באופן נרחב למתן תובנות מבניות למערכות חלבון רבות ושונות, כולל חלבונים עם הפרעה מהותית (עקורים)21, כגון α-סינוקלאין (α-Syn)22. α-Syn יכול ליצור מבנים מסודרים בעקבות קשירה ביומולקולים שונים ובתנאים שונים23,24,25,26,27,28,29,30. עם זאת, כאשר לא מאוגד, מונומר α-Syn מאופיין בהטרוגניות קונפורמציה גבוהה עם קונפורמציות הדדיות במהירות31,32.
הקונפורמציות של α-Syn נחקרו בעבר באמצעות טכניקות שונות ושונות המסייעות בזיהוי דינמיקה קונפורמיאלית של מערכות חלבון הטרוגניות ודינמיות כאלה33,34,35,36,37,38,39. מעניין, מדידות FRET מולקולה אחת (smFRET) של α-Syn במאגר דיווחו על אוכלוסיית FRET יחיד39,40 כי הוא תוצאה של זמן ממוצע של קונפורמציות באופן דינמי interconverting לפעמים הרבה יותר מהר מאשר זמן דיפוזיה טיפוסי של α-Syn דרך נקודת הקונפוקציה (פעמים מהר כמו מיקרו שניות מעטות ואפילו מהר יותר מזה, יחסית זמני דיפוזיה אלפיות השנייה טיפוסית)40, 41. עם זאת, באמצעות סרגל ספקטרוסקופי FRET עם רגישות למרחק של 3-10 ננומטר מדווח לפעמים רק על שינויים מבניים כוללים בחלבון קטן כגון α-Syn. למדידות של מולקולה אחת המשתמשות בחיישני קרבה ספקטרוסקופיים עם רגישויות מרחק קצרות יותר יש פוטנציאל לדווח על דינמיקה של מבנים מקומיים. בזאת אנו מבצעים מדידות PIFE של α-Syn ומזהים תת-אוכלוסיות שונות של מיפוי אורך חיים פלואורסצנטי למבנים מקומיים שונים עם מעברים ביניהם איטיים כמו 100 אלפיות שני. עבודה זו מסכמת מדידות smPIFE שנפתרו בזמן של מולקולות α-Syn המתפזרות בחופשיות אחת בכל פעם, במאגר וכאשר היא מאוגדת לממברנות מבוססות SDS כחיישן קרבה ספקטרוסקופי לטווח קצר.
Protocol
1. טרנספורמציה פלסמיד
- הכנת 0.5 ליטר של SOC בינוני
- שקול 10 גרם של טריפטון, 2.5 גרם של תמצית שמרים, 0.25 גרם של נתרן כלורי (NaCl), 0.1 גרם של אשלגן כלורי (KCl).
- יש להוסיף מים מזוקקים כפולים (DDW) עד לנפח כולל של 0.5 ליטר.
- הסתגלו ל-pH 7 על ידי הוספת נתרן הידרוקסיד (NaOH).
- הכן aliquots מלאי של 100 מ"ל ו autoclave.
- לפני השימוש, להוסיף 0.5 מ"ל של מגנזיום כלוריד סטרילי (MgCl2)ו 1.8 מ"ל של גלוקוז סטרילי ל 100 מ"ל של SOC.
- הפשרת BL21 (DE3) תאי Escherichia coli מוסמכים על קרח.
- מערבבים בעדינות את התאים המוסמכים.
- הכן שני צינורות 15 מ"ל. תייג אחד כצינור תגובה טרנספורמציה והשני כצינור הבקרה.
- Aliquot 100 μL של תאים מוסמכים לתוך הצינורות.
- הוסף 1 μL של plasmid בריכוז של 0.1 ng / μL לתוך צינור תגובת הטרנספורמציה.
- חום SOC בינוני ב 42 °C אמבט מים לשימוש בשלב 1.8.
- חום דופק את שני הצינורות באמבט מים 42 °C (42 °F) למשך 45 שניות. (צעד קריטי!)
- הוסף 900 μL של בינוני SOC מחומם לכל צינור.
- לדגור על הצינורות ב 37 °C (55 °F) למשך 45 דקות עם רועד במהירות של 225 סל"ד.
- להפיץ 200 μL של התאים עם plasmid המרה על צלחת LB אחת (לוריא-ברטאני) -אגר המכיל 100 μg / mL אמפצילין, באמצעות מפיץ תאים סטרילי.
- להפיץ 200 μL של התאים עם plasmid המרה על צלחת LB אגר אחת ללא אמפיצלין, באמצעות מפזר סטרילי (שליטה חיובית).
- להפיץ 200 μL של תאי הבקרה (לא מכיל plasmid) על צלחת LB-אגר אחת המכילה 100 μg / mL אמפצילין, באמצעות מפזר סטרילי(שליטה שלילית).
- לדגור על הצלחות לילה ב 37 °C (50 °F).
2. הכנת חלבונים
- ביטוי וטיהור α-סינוקלאין רקומביננטיים
- בחר מושבה אחת ולגדול 100 מ"ל של LB אוטומטי (לוריא-ברטאני) מדיום נוזלי המכיל 100 מיקרוגרם / mL אמפצילין ב 37 °C (המתנע).
- הכן ארבעה בקבוקוני ארלנמייר 2 ליטר, כל אחד מכיל 1 ליטר של בינוני LB autoclaved ו 100 מיקרוגרם / mL אמפצילין (חיסון גדול).
- מדוד את הצפיפות האופטית (OD ב- λ = 600 ננומטר) של פתרון התא. כאשר צפיפות התא של המתנע מגיעה ODλ = 600nm של 0.6-0.7, להוסיף 10 מ"ל של פתרון המתנע לכל 1 ליטר של LB בינוני מוכן בשלב הקודם.
- לגדל את המדיומים החיידקיים בשייקר אינקובטור ב 37 °C (50 °F) ומהירות של 200 סל"ד.
- כאשר צפיפות התא מגיעה ODλ = 600nm של 0.6-0.8, לגרום ביטוי חלבון על ידי הוספת איזופרופיל β-d-1-thiogalactopyranoside (IPTG) לכל 1 ליטר של בינוני צמיחה לריכוז סופי של 1 mM.
הערה: להמיס 0.96 גרם של IPTG ב 4 מ"ל של DDW ולהוסיף 1 מ"ל של פתרון IPTG וכתוצאה מכך לכל 1 L של צמיחה חיידקית. - לגדל את התאים למשך 4 שעות ולאסוף אותם על ידי צנטריפוגה בצינורות 50 מ"ל במהירות של 5,170 x g למשך 8 דקות.
הערה: בכל סיבוב, להשליך את supernatant, לשמור על הכדור ולמלא שוב את אותם צינורות עם צמיחה חיידקית. - לאחסן את הכדור החיידקי באחסון הקפאה עמוקה (למשל, -80 °C (80 °F) ליום המחרת.
- למחרת, להכין 200 מ"ל של חוצץ תמוגה: 40% (w/v) סוכרוז חוצץ A, הכולל 30 mM Tris-HCl, 2 mM אתילנדימינשטראקטי חומצה (EDTA), 2 mM dithiothreitol (DTT) ב pH 8.0.
- הוסף 25 מ"ל של חוצץ תמוז לכל צינור עם גלולה חיידקית.
- להשעות מחדש את הכדור (השתמש בפיפטות פסטר פלסטיק סטרילי) במאגר ליסיס (ראה 2.1.8).
- הכן בקבוק ארלנמאייר סטרילי 250 מ"ל עם מגנט stirrer על קרח.
- הוסף 5 מ"ל של מאגר תמוגה ולאחר מכן העבר אליו את התאים ההומוגניים המושעים מחדש.
- מערבבים את התאים למשך 20 דקות ב 200 סל"ד בטמפרטורת החדר.
- חלק את הפתרון לצינורות 50 מ"ל וצנטריפוגה ב 4 °C (5 °F) למשך 30 דקות במהירות של 17,400 x g.
- זרוק את סופר-טבעי.
- הכן בקבוק ארלנמאייר סטרילי 250 מ"ל עם מגנט stirrer על קרח.
- הוסף 15 מ"ל של מאגר פירוק (90 מ"ל חיץ מקורר A ו 37 μL של MgCl2 מומס מעבר למגבלת המסיסות ומסונן) לתוך כל צינור. השתמש פיפטות פסטר פלסטיק סטרילי ולהמיס את הכדור.
- כאשר חלק מהפתרון הופך הומוגני, להעביר אותו בקבוק ארלנמאייר מעורבב על קרח.
- קבע את אמצעי האחסון של הפתרון בארנמאייר.
- הוסף 10 מ ג של סטרפטומיצין סולפט לכל 1 מ"ל של פתרון (לפני הוספת, להמיס סטרפטומיצין סולפט ב 2 מ"ל של חוצץ A).
הערה: שלב זה מבוצע על מנת להסיר את ה- DNA ואת הריבוזומים משם. - מערבבים את הפתרון למשך 10-20 דקות בטמפרטורת החדר.
- חלק את הפתרון לצינורות 50 מ"ל וצנטריפוגה ב 4 °C (7 °F) ובמהירות של 20,700 x g למשך 30 דקות.
- לאסוף את supernatant ולהשליך את הכדור.
- הכן בקבוק ארלנמייר סטרילי 250 מ"ל על קרח עם מערבל.
- סנן את supernatant באמצעות 0.22 מיקרומטר מזרק מסנן לתוך בקבוק Erlenmeyer נקי.
- לקבוע את נפח הפתרון ולשקול 0.3 גרם של אמוניום גופרתי לכל 1 מ"ל של פתרון.
- בהדרגה, מוסיפים את אמוניום גופרתי לתמיסה stirred.
הערה: שלב זה מבוצע כדי לזרז חלבונים. כאשר האמוניום הגופרתי קושר את החלבונים, התמיסה הופכת מעורפלת ומטושטשת, אשר מופיעה כצבע לבן. - מערבבים במשך 30 דקות בטמפרטורת החדר.
- חלק את הפתרון לצינורות 50 מ"ל וצנטריפוגה ב 4 °C (7 °F) ובמהירות של 20,700 גרם למשך 30 דקות.
- בזהירות להשליך את supernatant ולשמור את הכדור.
- להמיס את הכדור עם 20 מ"ל של חוצץ A.
- לאחד את כל הפתרונות לתוך צינור אחד, ולאחר מכן למדוד את ספקטרום ספיגת UV של הפתרון.
הערה: אם הספקטרום מציג חתימה של צבירה (ספיגה מעל אורך גל של 300 ננומטר), המשך לשלב הבא. אם לא, דלג על השלב הבא ועבור לשלב 2.1.34.
הערה: מדד הצבירה (A.I.) המחושב מפרטטרום UV כ- ODλ = 350nm/ (ODλ = 280nm - ODλ = 350nm) ×100. 41 - השתמש 100 kDa חיתוך צנטרינות צינורות צנטריפוגה במהירות של 3,590 x גרם ב 4 °C (5 °F) למשך 15 דקות. לאסוף את הנוזל שעבר דרך המסנן.
- חייג את הפתרון ב 4 °C (5 °F) לילה, נגד חוצץ A, באמצעות שקיות דיאליזה עם ניתוק 3.5 kDa.
- בצע סבב נוסף של דיאליזה למשך 4 שעות לפחות.
- טען את הדוגמה המחויגת לעמודת חילופי אניון MonoQ של 1 מ"ל. השתמש 30 mM טריס הידרוכלוריד (Tris-HCl) חוצץ ב pH 7.5 כחוצץ כביסה 30 mM חוצץ Tris-HCl pH 7.5 עם 500 mM NaCl כחוצץ elution.
- הזרק את הדגימה לעמודה. לאחר מכן לשטוף עם 20 נפחי עמודות (קורות מים) של 0% חוצץ elution ו 100% לשטוף חוצץ על מנת להסיר את החלבונים לא מאוגדים.
- לשטוף עם 7 קורות מים של 30% חוצץ elution.
- יש לבחון את דגימת החלבון α-Syn באמצעות שיפוע שמשתנה מ-30% ל-100% חוצץ אלוטיון עבור 30 קורות חיים, בקצב זרימה של 1.5 מ"ל/דקה.
הערה: α-Syn elutes ב 46-66 CVs במוליכות 20-24 mS/ס"מ, אשר מתייחס 38-50% חוצץ אלוטיון. - לאסוף את השברים של פסגת ההתחמקות הראשית. לאמת את נוכחותו של α-Syn על ידי הפעלת דגימות ב 15% נתרן דודסיל סולפט אלקטרופורזה ג'ל פולאקרילמיד (SDS-PAGE), בעקבות כתמים עם פתרון מכתים Coomassie כחול או מהיר SeeBand כתמים. להקה ב 15 kDa צפויה.
- לאחד את השברים הרלוונטיים ולחייג אותם בן לילה, נגד חוצץ A, באמצעות שקיות דיאליזה עם 3.5 kDa ניתוק ב 4 °C (5 °F).
- הכן aliquots של דגימת החלבון ולאחסן אותם ב -20 °C (50 °F).
- α-סינוקלאין עם תווית Cy3
- להפחית את thiol של שאריות ציסטאין במוטנט α-Syn A56C על ידי הוספת DTT לריכוז סופי של 2 מ"מ למשך שעה אחת בטמפרטורת החדר.
- הסר DTT על-ידי ביצוע שני סבבים של דיאליזה באמצעות שקיות דיאליזה מנותקות 3.5 kDa. בסיבוב הראשון, דיאליזה נגד 30 מ"מ טריס-HCl pH 8.0 ו 2 mM EDTA. לסיבוב השני, דיאליזה נגד 50 mM HEPES pH 7.2 ו 2 mM EDTA.
- להפחית את שאריות ציסטאין על ידי הוספת טריס (2-carboxyethyl)פוספין הידרוכלוריד (TCEP) לדגימת החלבון, לריכוז סופי של 50 מיקרומטר למשך 30 דקות בטמפרטורת החדר.
הערה: TCEP הוא סוכן הפחתת שאינו סולפיהידיל, ולכן הוא שומר על קבוצות תיול מופחתות מבלי להגיב עם הצבע. הוספנו 0.5 μL מפתרון מלאי של 1 M TCEP. - חשב את כמויות החלבון הנדרשות עבור נפח התגובה הסופי של 1 מ"ל.
הערה: הנה דוגמה לחישוב שבו השתמשנו:
ריכוז חלבון סופי של 20μM × נפח סופי של 1 מ"ל = ריכוז חלבון ראשוני של 56 מיקרומטר × חלבון V שיש להוסיף - חשב את כמות הצבע הנדרש עבור נפח התגובה הסופי של 1 מ"ל. תיוג צבע של ציסטאין יחיד צריך להתבצע עם צבע עודף, ביחס טוחנת צבע:חלבון של לפחות 3:1.
הערה: דוגמה לחישוב שבו השתמשנו:
ריכוז צבע סופי של 60μM × נפח סופי של 1 מ"ל = ריכוז צבע ראשוני של 370 מיקרומטר × צבע V שיש להוסיף - חשב את כמות המאגר מהדיאליזה הנדרשת להתאמת עוצמת התגובה הכוללת ל- 1 מ"ל.
הערה: דוגמה לחישוב: 1 מ"ל - (V מחושב בשלב 2.2.4 + V מחושב בשלב 2.2.5) - הכן את בישון התגובה עם מגנט על גבי מערבל.
- ראשית, מוסיפים את הכמות המחושבת של החלבון ואת המאגר. לאחר מכן, מוסיפים את הכמות המחושבת של הצבע (סולפו-Cy3 maleimide).
- שמור את התגובה בטמפרטורת החדר בחושך למשך 3-5 שעות.
- לסיים את התגובה על ידי הוספת 2 mM DTT ולהמשיך למשך שעה אחת.
- בצע שלושה סבבים של דיאליזה נגד Buffer A, באמצעות שקיות דיאליזה עם ניתוק 3.5 kDa כדי להסיר צבע חופשי עודף מהפתרון.
- טען את המדגם בעמודת אי-הכללת גודל כדי להפריד עוד יותר את התווית α-Syn מהצבע החינמי.
- לקבוע את הריכוז של טהור מסומן α-Syn על ידי מדידת ספיגת הצבע (מקדם ספיגה עבור sulfo-Cy3 הוא 162,000 M-1ס"מ-1ב λ = 548 ננומטר).
- במידת הצורך, לרכז את הפתרון α-Syn המסומן באמצעות רכז ואקום (למשל, SpeedVac). לאחר מכן, בצע סיבוב אחד של דיאליזה נגד חוצץ A, באמצעות שקיות דיאליזה עם ניתוק 3.5 kDa ושוב לקבוע את הריכוז של חלבון שכותרתו.
- הכן aliquots של דגימת חלבון שכותרתו ולאחסן אותם ב -20 °C (50 °F).
3. מדידות
- התקנה ניסיונית של smPIFE
הערה: השתמש בהתקנה הבאה המבוססת על קונפוקלי או בדומה.- השתמש במקור לייזר פעמו (במקרה שלנו, λ = 532 ננומטר picosecond פעמו לייזר עם רוחב דופק של ~ 100 ps FWHM), פועל בקצב חזרה מתאים (20 MHz במקרה שלנו) ומנותב לסנכרון של כרטיס ספירת פוטון יחיד מתואם בזמן (TCSPC), כמקור של עירור גופרתי-Cy3 (sCy3).
- השתמש במראה dichroic עם רפלקטיביות גבוהה ב 532 ננומטר, כדי להפריד עירור ופיזור פלואורסצנטיות.
- השתמש חור סיכה בקוטר 100 מיקרומטר במוקד האור הנפלט, לאחר קרן הפליטה הקולימט התמקדה בעדשה, ולפני קרן הפליטה כבר מחדש collimated על ידי עדשה אחרת.
- השתמש במסנן מעבר רצועה (585/40 ננומטר במקרה שלנו) כדי לסנן עוד יותר פלואורסצנטיות sCy3 ממקורות אור אחרים.
- לזהות את הפלואורסצנטיות באמצעות גלאי (דיודת מפולת פוטון יחיד או פוטומולטיפלייר היברידי, במקרה שלנו) מנותב (באמצעות נתב 4 ל -1, במקרה שלנו) למודול TCSPC (בקר והיקל SPC-150, במקרה שלנו).
הערה: במקרה שלנו, רכישת נתונים מתבצעת באמצעות תוכנת VistaVision (ISSTM)בתבנית הקובץ שתויגה בזמן (TTTR).
- הכנת מדגם smPIFE
- הכן 25 pM sCy3-Syn בצורת α-Syn במאגר מדידות: 10 mM נתרן אצטט, 10 mM נתרן דיהידרוגן פוספט, 10 mM גליצין pH 8.0, 20 mM NaCl, 10 mM ציסטאמין ו 1 mM 6-הידרוקסי-2,5,7,8-tetramethylchroman-2-קרבוקסיליד (TROLOX), בצינור כריכה חלבון נמוך.
הערה: אם α-Syn נמדד בנוכחות שלל SDS, הוסף גם את כמות ה- SDS המתאימה. - יש לשטוף שקופית מיקרוסקופיה בת 18 תאים עם 100 μL של סרום בקר אחד מ"ג/מ"ל (BSA) למשך דקה אחת, ולאחר מכן להסיר את ה- BSA.
- הוסף 100 μL של 25 pM sCy3 תווית α-Syn מדגם לתא בשקופית כיסוי.
- בצע מדידת smPIFE של המדגם באמצעות ההתקנה המתוארת, כדלקמן בשלבים הבאים.
- הכן 25 pM sCy3-Syn בצורת α-Syn במאגר מדידות: 10 mM נתרן אצטט, 10 mM נתרן דיהידרוגן פוספט, 10 mM גליצין pH 8.0, 20 mM NaCl, 10 mM ציסטאמין ו 1 mM 6-הידרוקסי-2,5,7,8-tetramethylchroman-2-קרבוקסיליד (TROLOX), בצינור כריכה חלבון נמוך.
- רכישת נתוני smPIFE
- השתמש בעדשה אובייקטיבית גבוהה של טבילת מי צמצם מספרית (במקרה שלנו, אולימפוס UPLSAPO 60x N.A. 1.2), ולהוסיף טיפה של מים טהורים במיוחד על גבי העדשה האובייקטיבית.
- תקן את שקופית כיסוי בתא הבמה והתקן אותה על גבי שלב המיקרוסקופ.
- מביאים את העדשה האובייקטיבית כלפי מעלה, עד שקצה המים מעל העדשה האובייקטיבית מרוח בתחתית מגלשת הכיסוי.
- פתחו את תריס הלייזר והבימו את העדשה האובייקטיבית כלפי מעלה, תוך כדי בדיקת התבנית במצלמת CMOS, איסוף אור המפוזר מהדגימה. שימו לב לתבנית הטבעות האוורייניות: הראשונה מייצגת את המוקד בממשק זכוכית המים, ואז השנייה מייצגת את המוקד בממשק שבין הזכוכית לפתרון לדוגמה.
- הגדל את גובה העדשה האובייקטיבית ב-75 מיקרומטר נוספים, והבא את מיקוד הלייזר עמוק לתוך הפתרון, כדי למזער פלואורסצנטיות אוטומטית מפני השטח של הזכוכית של כיסוי.
- כוונן את כוח הלייזר בעדשה האובייקטיבית להיות ~ 100 μW.
- התחל ברכישת פוטונים שזוהו לזמן מוגדר מראש (שעתיים, במדידות שלנו).
הערה: רוב אות הרכישה צריך להיות שיעור (נמדד בספירות לשניות, cps) הדומה לזה שנרכש בעת מדידת המאגר בלבד; הנתונים המנופחים באלפי השניה אמורים להראות אירועי התפרצות פוטון נדירים, כאשר לרוב הפחים יש שיעור ממוצע הדומה לשיעור הרקע האופייני לגלאי (<1,000 cps, במקרה שלנו).
4. ניתוח פרץ smPIFE
- המרת נתונים גולמיים
הערה: הנתונים מאוחסנים בדרך כלל בקובץ בינארי עם תבנית שהוגדרה מראש על ידי החברה המייצרת את כרטיס TCSPC (קבצי .spc במקרה שלנו).- המר את קובץ הנתונים הגולמיים לפורמט הקובץ האוניברסלי פוטון-HDF542, באמצעות phconvert חבילת התוכנה (https://github.com/Photon-HDF5/phconvert). התקשר לקובץ הנתונים הגולמיים כקלט והמר לקובץ .hdf5 נתונים גולמיים באמצעות הקוד המתאים בחבילת phconvert (קבצי המרת ns-ALEX בקר-היקל למחברת Photon-HDF5.ipynb Jupyter, במקרה שלנו).
הערה: ההמרה לקובץ .hdf5 כוללת: (1) קביעת זרמי הפוטונים הרלוונטיים בנתונים הגולמיים (כלומר, זרמי פוטון של פוטונים רשומים של מזהה הגלאי הרלוונטי); (2) ננו-זמן הפוטן הרלוונטי (זמני זיהוי הפוטונים ביחס לזמן ה- SYNC העירור); וספנו (3) מטא-נתונים.
- המר את קובץ הנתונים הגולמיים לפורמט הקובץ האוניברסלי פוטון-HDF542, באמצעות phconvert חבילת התוכנה (https://github.com/Photon-HDF5/phconvert). התקשר לקובץ הנתונים הגולמיים כקלט והמר לקובץ .hdf5 נתונים גולמיים באמצעות הקוד המתאים בחבילת phconvert (קבצי המרת ns-ALEX בקר-היקל למחברת Photon-HDF5.ipynb Jupyter, במקרה שלנו).
- חיפוש ובחירה מתפרצים באמצעות FRETbursts43
הערה: כל קבצי הנתונים הגולמיים פוטון-HDF5 של מדידות smPIFE, כמו גם את הקוד המסכם את הניתוח של הנתונים הגולמיים, מאוחסנים Zenodo (https://doi.org/10.5281/zenodo.4587698). כל השלבים שלהלן מפורטים ומוצגים בתוך מחברות Jupyter, המסופקות גם בקישור מאגר Zenodo.- פתח את מחברות יופיטר (במסגרת אנקונדה, במקרה שלנו).
- פתח את המחברת smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (מחברת סופית).ipynb (ניתן למצוא אותו בקישור Zenodo).
- טען התפרצויות.
- טען את קובץ הנתונים של פוטון HDF5.
- הערכת קצב BG: באמצעות היסטוגרמה של הזמנים הבין-פוטוניים, לחשב את שיעורי הרקע (BG) עבור כל 30 שניות של רכישת נתונים בקובץ הנתונים HDF5 פוטון.
הערה: השלבים הבאים מתארים את חיפוש הפרץ באמצעות אלגוריתם חלון הזזה44,45,46. - הזז חלון זמן של m = 20 פוטונים רצופים, פוטון אחד בכל פעם.
- אסוף את נתוני הפוטון רק אם קצב הפוטון המיידי,
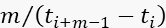 (), גדול לפחות פי 11 משיעור ה- BG לתקופה זו של רכישת הנתונים.
(), גדול לפחות פי 11 משיעור ה- BG לתקופה זו של רכישת הנתונים.
הערה: פרץ בנוי מכל הפוטונים הרצופים שנאספו על ידי הזזת החלון פוטון אחד בכל פעם (שלב 4.2.6.) והסכמה עם קריטריון שיעור הפוטון (שלב 4.2.7). - חשב את מאפייני הפרץ הבאים:
גודל פרץ: כמות הפוטונים בפרץ.
משך ההתפרצות: הפרש הזמן בין זמני זיהוי הפוטונים האחרונים והראשון בפרץ.
בהירות פרץ: הערך הגדול ביותר של קצב הפוטוס המיידי בפרץ.
הפרדת פרץ: מרווח הזמן בין התפרצויות רצופות.
הערה: הנקודות הבאות מתארות את הליך בחירת הפרץ הנוסף. - התווה את ההיסטוגרמה של ערכי בהירות פרץ (קצב הפוטוסים המיידי הגבוה ביותר בפרץ), כאשר ציר האירועים הוא בקנה מידה לוגריתמי.
- הגדר את סף בהירות הפרץ כערך בהירות פרץ מינימלי שממנו היסטוגרמה מציגה תבנית מתפוררת.
- בחר התפרצויות עם ערכי בהירות הגדולים מסף הבהירות של הפרץ.
הערה: השלבים הבאים מתארים פרץ פירושו אורך חיים פלואורסצנטי. - התווה את ההיסטוגרמה של ננו-זמן פוטון עבור כל הפוטונים בכל התפרצויות שנבחרו עם ציר ספירת הפוטונים בקנה מידה לוגריתמי.
- הגדר את סף הננו-זמן כערך ננו-זמן מינימלי שממנו היסטוגרמה של ננו-זמן פוטון מציגה תבנית מתפוררת.
- בחר רק פוטונים עם ננו-זמן גדול יותר מסף הננו-זמן.
- חשב את הממוצע האלגברי של כל ננו-זמן הפוטן שנבחר.
- הפחת את סף הננו-זמן מהממוצע האלגברי של הננו-זמן בפוטון. התוצאה היא הננו-זמן הפוטן הממוצע של הפרץ, שהוא פרופורציונלי ישירות לתוחלת החיים הממוצעת של הפלואורסצנטיות.
- שרטט את ההיסטוגרמה של כל תקופות החיים הממוצעות של פלואורסצנטיות. תת-אוכלוסיות בעלות תוחלת חיים פלואורסצנטית בחלוקה מרכזית עשויות להופיע. תת-אוכלוסיות עם ממוצעי ערך נמוכים מייצגות מינים של מולקולות עם sCy3 שלא נחסמו באופן סטרי, בעוד שתת-אוכלוסיות עם ממוצעי ערך גבוהים יותר מייצגות מינים של מולקולות עם sCy3 שנחסום בצורה סטרית יותר.
הערה: השלבים הבאים מתארים דינמיקה איטית בין התפרצויות המבוססת על ניתוח מופע חוזרשל פרץ 47 - התווה את ההיסטוגרמה של זמני הפרדת פרץ, עם ציר זמן ההפרדה בקנה מידה לוגריתמי.
הערה: יופיעו שתי תתי-אוכלוסיות של זמני הפרדת פרץ:
תת-אוכלוסייה גדולה עם זמני הפרדה של שניות, המייצגת התפרצויות רצופות שמקורן במולקולות שונות שנמדדו ברציפות.
תת-אוכלוסייה מינורית עם זמני הפרדה ~ <100 ms, המייצגת התפרצויות רצופות שמקורן באותה מולקולה, חוזרות על עצמן דרך נפח הקונפוקלי. - בחר כדי לשמור את כל זוגות התפרצויות רצופות המופרדים על ידי פחות מזמן הפרדה מקסימלי המגדיר את תת-אוכלוסיית אותה מולקולה (<100 אלפיות שני, במקרה שלנו).
- התוויית היסטוגרמה או חלקת פיזור של אורך החיים הממוצע של הפלואורסצנטיות של ההתפרצויות הראשונה והשנייה עבור כל זוגות התפרצויות שחוזרות על עצמן מתחת לסף זמן הפרדה מסוים
תוצאות
כ- IDP, כאשר הוא אינו מאוגד לביומולקול אחר, α-Syn מציג דינמיקה מבנית בין קונפורמציות מרובות, עם מעברים במיקרו שניות מעטות40 ואפילו במאות ננו-שניות41. כאשר α-סיסן חוצה את נקודת הקונפוציה, היא עשויה לעבור אלפי מעברים בין קונפורמציות. אכן, זה היה המקרה כאשר smFRET שימש
Discussion
מחקרים ביוכימיים וביופיזיים נרחבים בוצעו כדי לחקור את המאפיינים המבניים של α-Syn וטבעו המופרע33,34,35,36,37,38. מספר עבודות כבר השתמשו ב- smFRET המתפזר בחופשיות כדי לחקור את הדינמיקה התוך-מול?...
Disclosures
כל המחברים חולקים ניגוד אינטרסים.
Acknowledgements
ה-pT-t7 קידוד A56C α-Syn ניתנה לנו במתנה מד"ר אסף גרופי, ד"ר דן אמיר וד"ר אלישע האס. מאמר זה נתמך על ידי המכונים הלאומיים לבריאות (NIH, מענק R01 GM130942 לא"ל כסובה), הקרן הישראלית למדע (מענק 3565/20 במסגרת תוכנית קילקורונה - בלימת נגיף הקורונה), קרן מילנר והאוניברסיטה העברית בירושלים (קרנות הזנק).
Materials
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
References
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved