Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kararlı Yerel Konformasyonları Belirlemek için Zaman Çözümlenmiş Protein Kaynaklı Floresan Geliştirmeyi Kullanmak Bir Seferde Bir α-Synuclein Monomer
Bu Makalede
Özet
Zaman çözümlenmiş tek moleküllü protein kaynaklı floresan geliştirme, proteinlerdeki yerel yapısal değişikliklere duyarlı yararlı bir floresan spektroskopik yakınlık sensörüdür. Burada, daha uzun menzilli FRET cetveli kullanılarak ölçüldüğünde küresel olarak yapılandırılmamış ve kararsız olarak bilinen α-Synuclein'deki kararlı yerel konformasyonları ortaya çıkarmak için kullanılabileceğini gösteriyoruz.
Özet
Tek biyomoleküllerin ve dinamiklerinin birden fazla konformasyonunu izlemek için spektroskopik cetvellerin kullanılması, yapısal dinamiklerin anlaşılmasında ve biyolojiye katkılarında devrim yaratmasına neden oldu. FRET tabanlı cetvel 3-10 nm aralığındaki boyalar arası mesafeleri raporlarken, protein kaynaklı floresan geliştirme (PIFE) gibi diğer spektroskopik teknikler, daha kısa 0-3 nm aralığında bir boya ile protein yüzeyi arasındaki yakınlığı rapor eder. Tercih yöntemi ne olursa olsun, serbestçe dağılan biyomolekülleri teker teker ölçmedeki kullanımı, deneysel parametrenin histogramlarını alarak, her alt popülasyonun milisaniyeler içinde değişmeden kalan tek bir uyumu veya milisaniyeden çok daha hızlı bir şekilde kesişen birden fazla uyumu ve dolayısıyla ortalama bir alt popülasyonu temsil ettiği ayrı merkezi olarak dağıtılmış biyomolekül alt popülasyonlarını verir. Histogramlarda bildirilen parametrenin inter-boya FRET verimliliği olduğu tek moleküllü FRET'te, tampondaki α-Synuclein monomer gibi özünde düzensiz bir proteinin, daha önce hızla birbirine keneşen birden fazla konfektörden oluşan tek bir ortalama alt popülasyon sergilediği bildirilmiştir. Bu geçmiş bulgular FRET tabanlı cetvelin 3-10 nm aralığına bağlı olsa da, bu proteini, mekâna özgü sCy3 etiketli α-Synuclein proteinlerinin floresan ömrünü tek tek izlediğimiz tek moleküllü PIFE kullanarak test etmeye çalıştık. İlginçtir ki, bu daha kısa menzilli spektroskopik yakınlık sensörunu kullanarak, sCy3 etiketli α-Synuclein, 10-100 ms'de kesişen belirgin şekilde farklı ortalama ömürlere sahip birkaç ömür boyu alt popülasyon sergiler. Bu sonuçlar, α-Synuclein'in küresel olarak düzensiz olabileceğini, ancak yine de istikrarlı yerel yapılara sahip olduğunu göstermektedir. Özetle, bu çalışmada, yerel veya küresel yapısal değişiklikleri aynı anda bir biyomolekülden izleyen farklı spektroskopik yakınlık sensörleri kullanmanın avantajını vurguluyoruz.
Giriş
Son yirmi yılda, tek moleküllü floresan bazlı yöntemler biyomolekülleri ölçmek için güçlü bir araçhalinegelmiştir 1,2, farklı biyomoleküler parametrelerin nasıl dağılacağını ve bu parametrelerin farklı alt popülasyonları arasında milisaniyenin altında çözünürlük 3 , 4,5arasında dinamik olarak nasıl bir araya geldiklerini yoklar. Bu tekniklerdeki parametreler, FRET ölçümlerinde enerji transfer verimliliğini içerir 6,7, floresan anizotropi8,9, floresan kuantum verimleri ve ömürleri10,11, farklı floresan söndürme bir fonksiyon olarak12 veya geliştirme13 mekanizmaları. Protein kaynaklı floresan geliştirme (PIFE) 14 olarak bilinen bu mekanizmalardan biri,14, 15 , 16 ,17,18,19 boyasının çevresindeki protein yüzeylerinin neden olduğu, heyecan verici durumdayken floroforun serbest izomerizasyonunasteriktıkanıklığın bir işlevi olarak floresan kuantum veriminin ve ömrünün artırılmasınıtanıtır. . Hem FRET hem de PIFE spektroskopik cetveller veya yakınlık sensörleri olarak kabul edilir, çünkü ölçülen parametreleri ölçüm altında etiketli biyomolekül içindeki uzamsal bir ölçüye doğrudan bağlıdır. FRET verimliliği 3-10 nm20aralığındaki bir çift boya arasındaki mesafe ile ilgili olsa da, PIFE izleri floresan kuantum veriminde veya 0-3 nm19aralığındaki yakındaki bir proteinin yüzeyi arasındaki mesafe ile ilgili yaşam sürelerinde artar.
Tek moleküllü FRET, α-Synuclein (α-Syn) 22 gibi özünde düzensiz proteinler (IDP'ler)21de dahil olmak üzere birçokfarklı protein sistemine yapısal içgörüler sağlamak için yaygın olarak kullanılmıştır. α-Syn, farklı biyomoleküllere ve farklı koşullar altında 23 , 24 , 25 ,26,27,28,29,30'abağlanarak sıralı yapılar oluşturabilir. Bununla birlikte, ilişkisiz olduğunda, α-Syn monomer, hızla birbirine keneşen konformasyonlarla yüksek konformasyonlu heterojenlik ile karakterize edilir31,32.
α-Syn'nin konformasyonları daha önce, bu tür yüksek heterojen ve dinamik protein sistemlerinin konformasyonsal dinamiklerinin belirlenmesine yardımcı olan çeşitli farklı teknikler kullanılarak çalışılmıştır33,34,35,36,37,38,39. İlginçtir ki, tampondaki α-Syn'nin tek moleküllü FRET (smFRET)ölçümleri,konfokal nokta aracılığıyla α-Syn'nin tipik difüzyon zamanından çok daha hızlı zaman zaman dinamik olarak kesişen konfeksiyonların zaman ortalamasının bir sonucu olan tek bir FRET popülasyonu39,40 bildirdi (birkaç mikrosaniye kadar hızlı ve hatta bundan daha hızlı, tipik milisaniye difüzyon sürelerine göre)40, 41. Bununla birlikte, 3-10 nm mesafe hassasiyetine sahip bir FRET spektroskopik cetveli kullanmak bazen sadece α-Syn gibi küçük bir proteindeki genel yapısal değişiklikleri raporlar. Daha kısa mesafe hassasiyetlerine sahip spektroskopik yakınlık sensörlerini kullanan tek moleküllü ölçümler, yerel yapıların dinamiklerini raporlama potansiyeline sahiptir. Burada α-Syn'nin tek moleküllü PIFE ölçümlerini gerçekleştiriyor ve floresan ömürlerinin farklı alt popülasyonlarını, aralarında 100 ms kadar yavaş geçişlerle farklı yerel yapılara eşleşiyor. Bu çalışma, serbestçe dağılan α-Syn moleküllerinin zaman çözümlenmiş smPIFE ölçümlerini birer birer, tamponda ve SDS tabanlı membranlara kısa menzilli tek moleküllü spektroskopik yakınlık sensörü olarak bağlandığında özetlemektedir.
Protokol
1. Plazmid dönüşümü
- 0,5 L SOC ortamının hazırlanması
- 10 g tripton, 2,5 g Maya özü, 0,25 g sodyum klorür (NaCl), 0,1 g potasyum klorür (KCl) ağırlığındadır.
- Toplam 0,5 L hacme kadar çift damıtılmış su (DDW) ekleyin.
- Sodyum hidroksit (NaOH) ekleyerek pH 7'ye ayarlayın.
- 100 mL ve otoklav stok aliquots hazırlayın.
- Kullanmadan önce, 100 mL SOC'ye 0,5 mL steril magnezyum klorür (MgCl2)ve 1,8 mL steril glikoz ekleyin.
- Buz üzerinde BL21 (DE3) yetkin Escherichia coli hücrelerini çözün.
- Yetkin hücreleri hafifçe karıştırın.
- İki adet 15 mL tüp hazırlayın. Birini dönüşüm reaksiyon tüpü, diğerini kontrol tüpü olarak etiketle.
- Aliquot 100 μL yetkin hücreler tüplere.
- Dönüşüm reaksiyon tüpüne 0,1 ng/μL konsantrasyonda 1 μL plazmid ekleyin.
- 1.8. adımda kullanılmak üzere 42 °C su banyosunda SOC ortamını ısıtın.
- 42 °C su banyosunda iki tüpü 45 saniyeboyunca ısı darbeli. (Kritik adım!)
- Her tüpe 900 μL ısıtmalı SOC ortamı ekleyin.
- Tüpleri 37 °C'de 45 dakika boyunca 225 rpm hızında sallayarak kuluçkaya yatırın.
- Dönüştürülmüş plazmid ile hücrelerin 200 μL'sini steril bir hücre serpme kullanarak 100 μg/mL ampisilin içeren bir LB (Luria-Bertani)-agar plakasına yayın.
- Dönüştürülmüş plazmid ile hücrelerin 200 μL'sini steril bir serpme (pozitif kontrol)kullanarak ampisilin olmadan bir LB-agar plakasına yayın.
- Kontrol hücrelerinin 200 μL'lik kısmını (plazmid içermeyen) steril bir serpme(negatif kontrol)kullanarak 100 μg/mL ampisilin içeren bir LB-agar plakasına yayın.
- Plakaları gece boyunca 37 °C'de kuluçkaya yatırın.
2. Protein hazırlama
- Rekombinant α-Synuclein ekspresyyönetı ve saflaştırma
- Tek bir koloni seçin ve 37 °C'de (marş)100 μg/ mL ampisilin içeren 100 mL otoklavlı LB (Luria-Bertani) sıvı ortamda büyüyün.
- Her biri 1 L otomatik kapatılmış LB ortamı ve 100 μg/mL ampisilin (büyük aşılama)içeren dört adet 2 L Erlenmeyer şişesi hazırlayın.
- Hücre çözeltisinin optik yoğunluğunu (OD φ = 600 nm)ölçün. Marşın hücre yoğunluğu0,6-0,7 arasında bir OD φ=600nm'ye ulaştığında, önceki adımda hazırlanan her 1 L LB ortamı başına 10 mL başlangıç çözeltisi ekleyin.
- Bakteriyel ortamları 37 °C'de bir inkübatör çalkalayıcıda ve 200 rpm hızında yetiştirin.
- Hücre yoğunluğu0.6-0.8'in OD φ=600nm'sine ulaştığında, her 1 L büyüme ortamı başına izopropil β-d-1-tiyogalactopyranoside (IPTG) ekleyerek protein ekspresyonuna neden olarak 1 mM'lik son konsantrasyona ulaşır.
NOT: 0,96 g IPTG'yi 4 mL DDW'da çözün ve her 1 L bakteri üremesi başına elde edilen IPTG çözeltisinin 1 mL'si ekleyin. - Hücreleri 4 saat boyunca büyütün ve 8 dakika boyunca 5.170 x g hızında 50 mL tüplerde santrifüjleme ile toplayın.
NOT: Her turda, süpernatant atın, peletin tutun ve aynı tüpleri bakteri üremesi ile tekrar doldurun. - Bakteri peletini ertesi gün derin dondurucu depoda (örneğin, -80 °C) saklayın.
- Ertesi gün, 200 mL lizis tamponu hazırlayın: 30 mM Tris-HCl, 2 mM etileniamintetraasetik asit (EDTA), 2 mM dithiothreitol (DTT) içeren% 40 (w / v) sakkaroz ve tampon A, pH 8.0'de.
- Bakteriyel pelet ile her tüpe 25 mL lizis tamponu ekleyin.
- Peleti Lysis tamponunda yeniden askıya alın (steril plastik Pastör pipetler kullanın) (bkz. 2.1.8).
- Buz üzerinde bir karıştırıcı mıknatıs ile steril 250 mL Erlenmeyer şişesi hazırlayın.
- 5 mL lizis tamponu ekleyin ve ardından homojen yeniden askıya alınan hücreleri ona aktarın.
- Hücreleri oda sıcaklığında 200 rpm'de 20 dakika boyunca karıştırın.
- Çözeltiyi 17.400 x g hızında 30 dakika boyunca 4 °C'de 50 mL tüplere ve santrifüje bölün.
- Üstnatant atın.
- Buz üzerinde karıştırıcı mıknatıs ile steril 250 mL Erlenmeyer şişesi hazırlayın.
- Her tüpe 15 mL çözünme tamponu (90 mL soğutmalı tampon A ve 37 μL MgCl2 çözünürlük sınırının ötesinde çözünmüş ve filtrelenmiş) ekleyin. Steril plastik Pasteur pipetler kullanın ve peleti çözün.
- Çözeltinin bir kısmı homojen hale geldiğinde, buz üzerinde karıştırılmış bir Erlenmeyer şişesine taşıyın.
- Erlenmeyer'deki çözüm hacmini belirleyin.
- Her 1 mL çözelti başına 10 mg streptomisin sülfat ekleyin (eklemeden önce, streptomisin sülfatını 2 mL tampon A'da çözün).
NOT: Bu adım DNA ve ribozomları uzaklaştırmak için gerçekleştirilir. - Çözeltiyi oda sıcaklığında 10-20 dakika boyunca karıştırın.
- Çözeltiyi 4 °C'de ve 30 dakika boyunca 20.700 x g hızında 50 mL tüplere ve santrifüje bölün.
- Üstnatant toplayın ve peletin atın.
- Bir karıştırıcı ile buz üzerinde steril bir 250 mL Erlenmeyer şişesi hazırlayın.
- 0,22 μm şırınna filtresini kullanarak süpernatantı temiz Erlenmeyer şişesine filtreleyin.
- Çözelti hacmini belirleyin ve her 1 mL çözelti başına 0,3 g amonyum sülfat ağırlığındadır.
- Yavaş yavaş, amonyum sülfat karıştırılmış çözeltiye ekleyin.
NOT: Bu adım proteinleri çökeltmek için gerçekleştirilir. Amonyum sülfat proteinleri bağladığında, çözelti beyaz renk olarak ortaya çıkan belirsiz ve bulanık hale gelir. - Oda sıcaklığında 30 dakika karıştırın.
- Çözeltiyi 4 °C'de 50 mL tüplere ve santrifüje ve 30 dakika boyunca 20.700 g hıza bölün.
- Üstnatant dikkatlice atın ve pelet saklayın.
- Peletin 20 mL Tampon A ile çözün.
- Tüm çözümleri tek bir tüp halinde birleştirin ve ardından çözeltinin UV emilim spektrumunun ölçülmesi.
NOT: Spektrum toplama imzası gösteriyorsa (300 nm dalga boyunun üzerinde emilim), bir sonraki adıma geçin. Değilse, sonraki adımı atlayın ve 2.1.34 adımına geçin.
NOT: Toplama indeksi (A.I.), UV spektrumundan ODφ=350nm/ (ODφ=280nm - ODφ=350nm) ×100 olarak hesaplanır. 41 - 15 dakika boyunca 4 °C'de 3.590 x g hızında 100 kDa kesme centriprep tüpü ve santrifüj kullanın. Filtreden geçen sıvıyı toplayın.
- Çözeltiyi bir gecede 4 °C'de Tampon A'ya karşı 3,5 kDa kesmeli diyaliz torbaları kullanarak dialyze edin.
- En az 4 saatlik bir süre için başka bir diyaliz turu gerçekleştirin.
- Çaprazlanmış örneği 1 mL MonoQ anion değişim sütununa yükleyin. Yıkama tamponu olarak pH 7.5'te 30 mM Tris hidroklorür (Tris-HCl) tamponu ve bir elüasyon tamponu olarak 500 mM NaCl ile 30 mM Tris-HCl tampon pH 7.5 kullanın.
- Örneği sütuna enjekte edin. Daha sonra ilişkisiz proteinleri çıkarmak için% 0 elution tamponu ve% 100 yıkama tamponu 20 sütun hacmi (CV) ile yıkayın.
- %30'luk 7 CV elüasyon tamponu ile yıkayın.
- α-Syn protein örneğini, 30 CV için %30'dan %100 elution tampona değişen bir gradyan kullanarak 1,5 mL/dk akış hızında sıvılaşın.
NOT: α-Syn, iletkenlik 20-24 mS/cm'de 46-66 CV'de elutes, bu da% 38-50 elution tamponudur. - Ana elution zirvesinin fraksiyonlarını toplayın. Coomassie Blue veya Fast SeeBand boyama çözeltisi ile lekelenmeyi takiben% 15 sodyum dodecyl sülfat poliakrilamid jel elektroforezinde (SDS-PAGE) numuneler çalıştırarak α-Syn varlığını doğrulayın. 15 kDa'da bir grup bekleniyor.
- İlgili fraksiyonları birleştirin ve 4 °C'de 3,5 kDa kesmeli diyaliz torbaları kullanarak A tamponuna karşı bir gecede çevirin.
- Protein örneğinin aliquotlarını hazırlayın ve -20 °C'de saklayın.
- Cy3 etiketli α-Synuclein
- Oda sıcaklığında 1 saat süreyle 2 mM'lik son konsantrasyona DTT ekleyerek α-Syn A56C mutanttaki sistein kalıntısının tiyollarını azaltın.
- 3,5 kDa kesme diyaliz torbaları kullanarak iki tur diyaliz yaparak DTT'yi çıkarın. İlk tur için 30 mM Tris-HCl pH 8.0 ve 2 mM EDTA'ya karşı dialyze. İkinci tur için 50 mM HEPES pH 7.2 ve 2 mM EDTA'ya karşı dialyze.
- Protein örneğine Tris (2-karboksit)fosfin hidroklorür (TCEP) ekleyerek, oda sıcaklığında 30 dakika boyunca 50 μM'lik son konsantrasyona ekleyerek sistein kalıntılarını azaltın.
NOT: TCEP sülfülhidrit olmayan bir azaltıcı maddedir, bu nedenle boya ile reaksiyona vermeden tiyol gruplarını azaltır. 1 M TCEP'lik bir stok çözeltisinden 0,5 μL ekledik. - 1 mL'lik son reaksiyon hacmi için gerekli protein miktarlarını hesaplayın.
NOT: Kullandığımız hesaplama için bir örnek aşağıda verilmiştir:
20 μM nihai protein konsantrasyonu × 1 mL son hacim = 56 μM başlangıç protein konsantrasyonu × V proteini eklenecek - 1 mL'lik son reaksiyon hacmi için gereken boya miktarını hesaplayın. Tek bir sisteinin boya etiketlemesi, en az 3:1'lik bir boya:protein azı dişi oranında, fazla boya ile yapılmalıdır.
NOT: Kullandığımız hesaplama için bir örnek:
60 μM son boya konsantrasyonu × 1 mL son hacim = 370 μM başlangıç boya konsantrasyonu × V boyası eklenecek - Toplam reaksiyon ses seviyesini 1 mL'ye ayarlamak için gereken dialysate'den arabellek miktarını hesaplayın.
NOT: Hesaplama örneği: 1 mL - (Adım 2.2.4 +V'den hesaplanan V adım 2.2.5'ten hesaplanır) - Bir karıştırıcının üzerine bir mıknatısla reaksiyon şişesi hazırlayın.
- İlk olarak, proteinin ve tamponun hesaplanan miktarını ekleyin. Daha sonra, hesaplanan boya miktarını ekleyin (sülfo-Cy3 maleimid).
- Reaksiyonun oda sıcaklığında karanlıkta 3-5 saat boyunca tutulmasını sağlamak.
- Reaksiyona 2 mM DTT ekleyerek son haline alıp 1 saat boyunca devam edin.
- Çözeltiden fazla serbest boyayı çıkarmak için 3,5 kDa kesmeli diyaliz torbaları kullanarak Tampon A'ya karşı üç tur diyaliz gerçekleştirin.
- Etiketli α-Syn'yi serbest boyadan daha fazla ayırmak için örneği bir boyut dışlama sütununa yükleyin.
- Boyanın emilimini ölçerek α-Syn olarak etiketlenmiş saf konsantrasyonu belirleyin (sülfo-Cy3 için emilim katsayısı 162.000 M-1cm-1at φ=548 nm'dir).
- Gerekirse, saf etiketli α-Syn çözeltisini vakum konsantratör (örneğin, SpeedVac) kullanarak konsantre edin. Ardından, 3,5 kDa kesmeli diyaliz torbaları kullanarak A tamponuna karşı bir tur diyaliz gerçekleştirin ve etiketli proteinin konsantrasyonu tekrar belirleyin.
- Etiketli protein örneğinin aliquotlarını hazırlayın ve -20 °C'de saklayın.
3. Ölçümler
- smPIFE deneysel kurulumu
NOT: Aşağıdaki konfokal tabanlı kurulumu veya benzerini kullanın.- Sülfo-Cy3 (sCy3) ekscitasyon kaynağı olarak, uygun bir tekrarlama hızında (bizim durumumuzda 20 MHz) çalışan ve zaman ilişkili tek foton sayımı (TCSPC) kartının SYNC'ine yönlendirilmiş, darbeli bir lazer kaynağı kullanın (bizim durumumuzda, φ= 532 nm darbeli lazer ve ~100 ps FWHM darbe genişliğinde).
- Heyecan ve saçılmayı floresan'dan ayırmak için 532 nm'de yüksek yansıtıcılığa sahip dikroik bir ayna kullanın.
- Yayılan ışığın odağında, kolimlenmiş emisyon ışını bir mercekle odaklandıktan sonra ve emisyon ışını başka bir mercek tarafından yeniden harmanlanmadan önce 100 μm çapında bir iğne deliği kullanın.
- SCy3 floresanını diğer ışık kaynaklarından daha fazla filtrelemek için bir bant geçiş filtresi (bizim durumumuzda 585/40 nm) kullanın.
- Floresan'ı bir TCSPC modülüne (bizim durumumuzda Becker & Hickl SPC-150) yönlendirilmiş bir dedektör (tek foton çığ diyot veya hibrit fotomultiplier) kullanarak tespit edin.
NOT: Bizim durumumuzda, veri toplama VistaVision yazılımı (ISSTM)aracılığıyla zaman etiketli zaman çözümlenen (TTTR) dosya biçiminde gerçekleştirilir.
- smPIFE numune hazırlama
- Ölçüm tamponunda 25 pM sCy3 etiketli α-Syn hazırlayın: 10 mM sodyum asetat, Düşük protein bağlayıcı bir tüpte 10 mM sodyum dihidrojen fosfat, 10 mM glisin pH 8.0, 20 mM NaCl, 10 mM sisteamin ve 1 mM 6-hidroksi-2,5,7,8-tetrametrik krom-2 karboksilik asit (TROLOX).
NOT: α-Syn SDS veziküllerinin varlığında ölçülürse, uygun SDS miktarını da ekleyin. - 18 odalı bir mikroskopi kapak kaydıranı 100 μL 1 mg/mL sığır serum albümini (BSA) 1 dakika boyunca durulayın ve ardından BSA'yı çıkarın.
- Kapak kaydırağındaki bir odaya 25 pM sCy3 etiketli α-Syn örneğinin 100 μL'sini ekleyin.
- Açıklanan kurulumu kullanarak numunenin smPIFE ölçümünü, sonraki adımlarda aşağıdaki gibi gerçekleştirin.
- Ölçüm tamponunda 25 pM sCy3 etiketli α-Syn hazırlayın: 10 mM sodyum asetat, Düşük protein bağlayıcı bir tüpte 10 mM sodyum dihidrojen fosfat, 10 mM glisin pH 8.0, 20 mM NaCl, 10 mM sisteamin ve 1 mM 6-hidroksi-2,5,7,8-tetrametrik krom-2 karboksilik asit (TROLOX).
- smPIFE veri toplama
- Yüksek sayısal diyaframlı su daldırma objektif lensi kullanın (bizim durumumuzda, Olympus UPLSAPO 60x N.A. 1.2) ve objektif lensin üzerine bir damla ultra saf su ekleyin.
- Kapak kaydıranı bir sahne odasına sabitleyin ve mikroskop aşamasının üzerine takın.
- Objektif lensin üstündeki su damlası kapak kaymasının altındaki lekelere kadar objektif lensi yukarı getirin.
- Lazer deklanşörü açın ve bir CMOS kameradaki deseni incelerken objektif lensi yukarı doğru getirin ve numuneden saçılan ışığı toplar. Havadar halka desenini gözlemleyin: birincisi su camı ara uzaylarındaki odağı, ikincisi ise cam ve örnek çözüm arasındaki arayüzdeki odağı temsil eder.
- Kapak kapağının cam yüzeyinden otomatik floresanlığı en aza indirmek için objektif lensin yüksekliğini 75 μm daha artırarak lazer odağını çözeltinin derinliklerine getirin.
- Objektif lensteki lazer gücünü ~100 μW olacak şekilde ayarlayın.
- Önceden tanımlanmış bir süre için tespit edilen fotonların edinimine başlayın (ölçümlerimizde 2 saat).
NOT: Alım sinyalinin çoğunluğu, yalnızca arabelleği ölçerken elde edilene benzer bir orana (saniye başına sayımlarla ölçülür, cps) sahip olmalıdır; milisaniyelik veriler, kutuların çoğunluğunun tipik dedektör arka plan oranıyla karşılaştırılabilir ortalama bir orana sahip olmasıyla (bizim durumumuzda 1.000 cps <) kıt foton patlaması olayları göstermelidir.
4. smPIFE patlama analizi
- Ham veri dönüştürme
NOT: Veriler genellikle TCSPC kartını (bizim durumumuzdaki spc dosyaları) üreten şirket tarafından önceden tanımlanmış bir formata sahip ikili bir dosyada saklanır.- Ham veri dosyasını foton-HDF5 evrensel dosya formatına dönüştürün42, yazılım paketi phconvert (https://github.com/Photon-HDF5/phconvert) kullanarak. Ham veri dosyasını giriş olarak çağırın ve phconvert paketindeki uygun kodu kullanarak ham veri .hdf5 dosyasına dönüştürün (bizim durumumuzda ns-ALEX Becker-Hickl dosyalarını Photon-HDF5.ipynb Jupyter dizüstü bilgisayarına dönüştür).
NOT: .hdf5 dosyasına dönüştürme şunları içerir: (1) ham verilerdeki ilgili foton akışlarının belirlenmesi (yani, ilgili dedektör kimliğine sahip kayıtlı fotonların foton akışları); (2) ilgili foton nanotimes (heyecan SYNC süresine göre foton algılama süreleri); ve (3) meta veriler ekledi.
- Ham veri dosyasını foton-HDF5 evrensel dosya formatına dönüştürün42, yazılım paketi phconvert (https://github.com/Photon-HDF5/phconvert) kullanarak. Ham veri dosyasını giriş olarak çağırın ve phconvert paketindeki uygun kodu kullanarak ham veri .hdf5 dosyasına dönüştürün (bizim durumumuzda ns-ALEX Becker-Hickl dosyalarını Photon-HDF5.ipynb Jupyter dizüstü bilgisayarına dönüştür).
- Kullanarak Arama ve Seçim Patlaması FRETbursts43
NOT: smPIFE ölçümlerinin tüm foton-HDF5 ham veri dosyalarının yanı sıra ham verilerin analizini özetleyen kod Zenodo'da (https://doi.org/10.5281/zenodo.4587698) saklanır. Aşağıdaki tüm adımlar ayrıntılı olarak ayrıntılı olarak ve Zenodo depo bağlantısında da bulunan Jupyter dizüstü bilgisayarlarda gösterilmiştir.- Jupyter Notebook'ları açın (Anaconda çerçevesinde, bizim durumumuzda).
- Dizüstü bilgisayarı açın smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (Son not defteri).ipynb (Zenodo bağlantısında bulunabilir).
- FRET patlamalarını yükleyin.
- Foton HDF5 veri dosyasını yükleyin.
- BG oranı değerlendirmesi: fotonlararası zamanların histogramını kullanarak, foton HDF5 veri dosyasındaki her 30 saniyelik veri toplama için arka plan (BG) oranlarını hesaplayın.
NOT: Aşağıdaki adımda, kayan pencere algoritması44 , 45,46kullanılarak yapılan seri çekim açıklanmaktadır. - m=20 ardışık foton zaman penceresini, her seferinde bir foton taşıyın.
- Foton verilerini yalnızca anlık foton hızı (
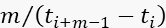 ), veri toplama döneminin BG oranından en az F=11 kat daha büyükse toplayın.
), veri toplama döneminin BG oranından en az F=11 kat daha büyükse toplayın.
NOT: Pencereyi bir seferde bir foton kaydırarak (adım 4.2.6.) ve foton oranı kriterini (adım 4.2.7) kabul ederek toplanan ardışık fotonların tümünden bir patlama oluşturulur. - Aşağıdaki seri çekim özelliklerini hesaplayın:
Patlama boyutu: bir patlamadaki foton miktarı.
Patlama süresi: bir patlamadaki son ve ilk foton algılama süreleri arasındaki zaman farkı.
Patlama parlaklığı: bir patlamada anlık foton hızının en büyük değeri.
Seri ayrım: ardışık patlamalar arasındaki zaman aralığı.
NOT: Aşağıdaki noktalarda daha fazla patlama seçim yordamı açıklanmaktadır. - Olayların ekseni logaritmik ölçekteyken, patlama parlaklığı değerlerinin histogramını (bir patlamada en yüksek anlık foton hızı) çizin.
- Patlama parlaklığı eşiğini, histogramın çürüyen bir desen sergilediği minimum patlama parlaklık değeri olarak tanımlayın.
- Patlama parlaklığı eşiğinden daha büyük parlaklık değerlerine sahip seri patlamaları seçin.
NOT: Sonraki adımlar patlama anlamına gelen floresan ömürlerini açıklar. - Logaritmik ölçekte foton sayım ekseni ile seçilen tüm patlamalardaki tüm fotonlar için foton nanotimes histogramını çizin.
- Nanotime eşiğini, foton nanotimes histogramının çürüyen bir desen sergilediği minimal nano zaman değeri olarak tanımlayın.
- Yalnızca nanotime eşiğinden daha büyük nanotime'lara sahip fotonları seçin.
- Seçilen tüm foton nanotimes cebirsel ortalamasını hesaplayın.
- Nanotime eşiğini foton nanotime cebirsel ortalamasından çıkarın. Sonuç, ortalama floresan ömrü ile doğru orantılı olan patlamanın ortalama foton nano zamanıdır.
- Tüm patlama ortalama floresan ömürlerinin histogramını çizin. Floresan ömrünün merkezi olarak dağıtılmış alt popülasyonları ortaya çıkabilir. Düşük değer ortalamalarına sahip alt popülasyonlar, sCy3 ile daha fazla sterik olarak engellenmemiş molekül türlerini temsil ederken, daha yüksek değer ortalamalarına sahip alt popülasyonlar, daha steretik olarak engellenen sCy3 ile molekül türlerini temsil eder.
NOT: Sonraki adımlarda, patlama yineleme analizine dayalı yavaş patlama arası dinamikler açıklanmaktadır47 - Logaritmik ölçekte ayırma süresi ekseni ile seri ayrım sürelerinin histogramını çizin.
NOT: Patlama ayırma sürelerinin iki alt popülasyonu görünecektir:
Ardışık olarak ölçülen farklı moleküllerden kaynaklanan ardışık patlamaları temsil eden, saniyelerin ayrılma sürelerine sahip büyük bir alt popülasyon.
Ayrılma süreleri ~<100 ms olan küçük bir alt popülasyon, her ikisi de aynı molekülden kaynaklanan ve konfokal hacimde tekrar eden ardışık patlamaları temsil eder. - Aynı moleküllü alt popülasyonu tanımlayan maksimum ayırma süresinden daha az bir süreyle ayrılan tüm ardışık patlama çiftlerini kaydetmek için seçin (bizim durumumuzda <100 ms).
- Belirli bir ayırma süresi eşiğinin altında yinelenen tüm patlama çiftleri için birinci ve ikinci patlamaların ortalama floresan ömürlerinin bir histogramını veya dağılım grafiğini çizin
Sonuçlar
Bir IDP olarak, başka bir biyomoleküle bağlı olmadığında, α-Syn, birkaç mikrosaniye40 ve hatta yüzlerce nanosaniye41'degeçişlerle birden fazla konformasyon arasında yapısal dinamikler sergiler. α-Syn konfokal noktayı geçtiğinde, konformasyonlar arasında binlerce geçişe maruz kalabilir. Gerçekten de, smFRET kullanıldığında durum böyleydi39,40. Burada α-Syn'nin yerel konformasyon dinamikler...
Tartışmalar
α-Syn'nin yapısal özelliklerini ve düzensiz doğasını incelemek için kapsamlı biyokimyasal ve biyofiziksel çalışmalar yapıldı33,34,35,36,37,38. Α-Syn monomerinin bağdan arındırılmış moleküler içi dinamiklerini araştırmak için birçok çalışma serbestçe dağıtıcı smFRET'i kullanmıştır. Bu çal...
Açıklamalar
Tüm yazarlar çıkar çatışması paylaşmaz.
Teşekkürler
PT-t7 plazmid kodlu A56C α-Syn mutant bize Dr. Asaf Grupi, Dr. Dan Amir ve Dr. Elisha Haas tarafından hediye edildi. Bu makale Ulusal Sağlık Enstitüleri (NIH, R01 GM130942'yi E.L.'ye subaward olarak vermek), İsrail Bilim Vakfı (KillCorona - Curbing Coronavirus Araştırma Programı dahilinde 3565/20 hibe), Milner Fonu ve Kudüs İbrani Üniversitesi (başlangıç fonları) tarafından desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
Referanslar
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır