Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Использование флуоресценции с временным разрешением, вызванного белком, для идентификации стабильных локальных конформаций по одному мономеру синуклеина α за раз
В этой статье
Резюме
Усиление флуоресценции с одномолекулярным белком, индуцированным временем, является полезным флуоресцентным спектроскопическим датчиком приближения, чувствительным к локальным структурным изменениям в белках. Здесь мы показываем, что он может быть использован для выявления стабильных локальных конформаций в α-синуклеине, который иначе известен как глобулярно неструктурированный и нестабильный при измерении с использованием линейки FRET с более длинным диапазоном.
Аннотация
Использование спектроскопических линеек для отслеживания множественных конформаций отдельных биомолекул и их динамики произвело революцию в понимании структурной динамики и ее вклада в биологию. В то время как линейка на основе FRET сообщает о межкрасивых расстояниях в диапазоне 3-10 нм, другие спектроскопические методы, такие как усиление флуоресценции, индуцированное белком (PIFE), сообщают о близости между красителем и поверхностью белка в более коротком диапазоне 0-3 нм. Независимо от выбранного метода, его использование при измерении свободно-диффузных биомолекул по одной за раз извлекает гистограммы экспериментального параметра, дающие отдельные централизованно распределенные субпуляции биомолекул, где каждая субоплюдион представляет собой либо единую конформацию, которая оставалась неизменной в течение миллисекунд, либо множественные конформации, которые взаимопреобразуются намного быстрее миллисекунд, и, следовательно, усредненную субпуляцию. В одномолекулярном FRET, где сообщаемым параметром в гистограммах является эффективность FRET между красителями, внутренне неупорядоцированный белок, такой как мономер α-синуклеина в буфере, ранее сообщал как демонстрирующий одну усредненную субпопуляцию множественных конформаций, быстро взаимопреобразующихся. Хотя эти прошлые результаты зависят от диапазона 3-10 нм линейки на основе FRET, мы стремились проверить этот белок с использованием одномолекулярного PIFE, где мы отслеживаем время жизни флуоресценции сайтов-специфических α-синуклеиновых белков sCy3 по одному. Интересно, что используя этот спектроскопический датчик приближения с более коротким диапазоном, меченый sCy3 α-Synuclein демонстрирует несколько подпопущений времени жизни с совершенно разными средними сроками жизни, которые взаимоконвертируются за 10-100 мс. Эти результаты показывают, что, хотя α-синуклеин может быть неупорядочешен в глобальном масштабе, он, тем не менее, достигает стабильных местных структур. Таким образом, в этой работе мы подчеркиваем преимущество использования различных спектроскопических датчиков приближения, которые отслеживают локальные или глобальные структурные изменения по одной биомолекуле за раз.
Введение
За последние два десятилетия одномолекулярные флуоресцентные методы стали мощным инструментом для измерения биомолекул1,2,исследования того, как распределяются различные биомолекулярные параметры, а также как они динамически взаимоконвертируются между различными субопциплонатами этих параметров при субмиллисекундном разрешении3,4,5. Параметры в этих методах включают эффективность передачи энергии в измерениях FRET 6,7,флуоресцентную анизотропию8,9,квант флуоресценции и время жизни10,11,как функцию различных флуоресцентных гасящих12 или13 улучшений механизмов. Один из этих механизмов, более известный как белково-индуцированное флуоресцентное усиление (PIFE)14, вводит увеличение квантового выхода флуоресценции и времени жизни в функции стерической обструкции к свободной изомеризации флуорофора в возбужденном состоянии, вызванном белковыми поверхностями в непосредственной близости от красителя14,15,16,17,18,19 . Как FRET, так и PIFE считаются спектроскопическими линейками или датчиками приближения, поскольку их измеряемый параметр напрямую связан с пространственной мерой в меченой биомолекуле при измерении. В то время как эффективность FRET связана с расстоянием между парой красителей в диапазоне 3-10 нм20,треки PIFE увеличивают квантовые выходы флуоресценции или время жизни, связанное с расстоянием между красителем и поверхностью соседнего белка в диапазоне 0-3 нм19.
Одномолекулярный FRET широко используется для обеспечения структурного понимания многих различных белковых систем, включая внутренне неупорядоженные белки (IMP)21,такие как α-synuclein (α-Syn)22. α-Syn может образовывать упорядоченные структуры после связывания с различными биомолекулами и в разных условиях23,24,25, 26,27,28,29,30. Однако при несвязывке мономер α-Syn характеризуется высокой конформационной гетерогенностью с быстро взаимоконвертируемыми конформациями31,32.
Конформации α-Syn были изучены ранее с использованием различных методов, которые помогают в выявлении конформационной динамики таких высоко гетерогенных и динамических белковых систем33,34,35,36,37,38,39. Интересно, что одномолекулярные измерения FRET (smFRET) α-Syn в буфере сообщили об одной популяции FRET39,40, что является результатом усреднения по времени конформаций, динамически взаимопреобразующихся в разы намного быстрее, чем типичное время диффузии α-Syn через конфокальное пятно (в разы быстрее, чем несколько микросекунд и даже быстрее, чем это, относительно типичного миллисекундного времени диффузии)40, 41г. Однако использование спектроскопической линейки FRET с чувствительностью расстояния 3-10 нм иногда сообщает только об общих структурных изменениях в небольшом белке, таком как α-Syn. Одномолекулярные измерения с использованием спектроскопических датчиков приближения с более короткой чувствительностью расстояния могут сообщить о динамике локальных структур. При этом мы выполняем одномолекулярные измерения PIFE α-Syn и идентифицируем различные субпопущенности флуоресцентных жизней, отображающих различные локальные структуры с переходами между ними медленно до 100 мс. В этой работе обобщены измерения smPIFE со временным разрешением свободно диффундируемых молекул α-Syn по одной, в буфере и при связываниях с мембранами на основе SDS в качестве одномолекулярного спектроскопического датчика приближения малого диапазона.
протокол
1. Плазмидная трансформация
- Приготовление 0,5 л SOC среды
- Взвесьте 10 г триптона, 2,5 г дрожжевого экстракта, 0,25 г хлорида натрия (NaCl), 0,1 г хлорида калия (KCl).
- Добавляют двойную дистиллированную воду (DDW) до общего объема 0,5 л.
- Отрегулируйте до pH 7, добавив гидроксид натрия (NaOH).
- Готовят запас аликвот по 100 мл и автоклав.
- Перед применением добавьте 0,5 мл стерильного хлорида магния (MgCl2)и 1,8 мл стерильной глюкозы к 100 мл SOC.
- Оттаивать BL21 (DE3) компетентные ячейки кишечной палочки на льду.
- Аккуратно перемешайте грамотные клетки.
- Подготовьте два туба по 15 мл. Обозначьте одну как реакционную трубку трансформации, а другую как контрольную трубку.
- Аликвота 100 мкл компетентных клеток в пробирки.
- Добавьте 1 мкл плазмиды в концентрации 0,1 нг/мкл в реакционную трубку трансформации.
- Нагрейте среду SOC на водяной бане 42 °C для использования на этапе 1.8.
- Нагревать-импульсные две трубки на водяной бане при температуре 42 °C в течение 45 секунд. (Критический шаг!)
- Добавьте 900 мкл нагретой среды SOC в каждую пробирку.
- Инкубируют трубки при 37 °C в течение 45 минут с встряхиванием со скоростью 225 об/мин.
- Распределите 200 мкл клеток с преобразованной плазмидой на одну пластину LB (Luria-Bertani)-агар, содержащую 100 мкг/мл ампициллина, используя стерильный клеточный распределитель.
- Распределите 200 мкл клеток с преобразованной плазмидой на одну пластину LB-агара без ампициллина, используя стерильный разбрасыватель (положительный контроль).
- Распределите 200 мкл контрольных клеток (не содержащих плазмиду) на одну пластину LB-агара, содержащую 100 мкг/мл ампициллина, используя стерильный разбрасыватель(отрицательный контроль).
- Инкубировать пластины в течение ночи при 37 °C.
2. Белковый препарат
- Экспрессия и очистка рекомбинантного α-синуклеина
- Соберите одну колонию и вырастите в 100 мл автоклавной жидкой среды LB (Luria-Bertani), содержащей 100 мкг/мл ампициллина при 37 °C (стартер).
- Приготовьте четыре колбы Эрленмейера по 2 л, каждая из которых содержит 1 л автоклавной среды LB и 100 мкг/мл ампициллина (большая прививка).
- Измерьте оптическую плотность (OD при λ = 600 нм) клеточного раствора. Когда плотность клеток стартера достигнет ODλ=600 нм 0,6-0,7, добавляют 10 мл стартового раствора на каждый 1 л LB-среды, приготовленной на предыдущей ступени.
- Выращивайте бактериальные среды в шейкере инкубатора при 37 °C и скорости 200 об/мин.
- Когда плотность клеток достигает ODλ=600 нм 0,6-0,8, индуцируют экспрессию белка путем добавления изопропил-β-d-1-тиогалакопиранозида (IPTG) на каждый 1 л питательной среды до конечной концентрации 1 мМ.
ПРИМЕЧАНИЕ: Растворить 0,96 г IPTG в 4 мл DDW и добавить 1 мл полученного раствора IPTG на каждый 1 л роста бактерий. - Выращивайте клетки в течение 4 часов и собирайте их центрифугированием в пробирках по 50 мл со скоростью 5 170 х г в течение 8 минут.
ПРИМЕЧАНИЕ: В каждом раунде отбросьте супернатант, сохраните гранулу и снова заполните те же пробирки с ростом бактерий. - Храните бактериальную гранулу в глубокой заморозке (например, -80 °C) в течение следующего дня.
- На следующий день готовят 200 мл лизисного буфера: 40% (мас./об.) сахарозы и буфера А, который включает 30 мМ Tris-HCl, 2 мМ этилендиаминтетрауксусной кислоты (ЭДТА), 2 мМ дитиотрейтола (ДТТ) при рН 8,0.
- Добавьте 25 мл лизисного буфера в каждый пробирку с бактериальной гранулой.
- Повторно суспендируют гранулы (используйте стерильные пластиковые пипетки Пастера) в буфере лизиса (см. 2.1.8).
- Приготовьте стерильную колбу Эрленмейера 250 мл с магнитом мешалки на льду.
- Добавьте 5 мл лизисного буфера, а затем переложите в него однородные повторно взвешенные клетки.
- Перемешивать ячейки в течение 20 минут при 200 об/мин при комнатной температуре.
- Разделить раствор на 50 мл пробирки и центрифугу при 4 °C в течение 30 минут со скоростью 17 400 х г.
- Выбросьте супернатант.
- Приготовьте стерильную колбу Эрленмейера 250 мл с магнитом мешалки на льду.
- Добавьте 15 мл буфера растворения (90 мл охлажденного буфера А и 37 мклMgCl2, растворенного сверх предела растворимости и отфильтрованного) в каждую пробирку. Используйте стерильные пластиковые пипетки Пастера и растворите гранулы.
- Когда часть раствора станет однородной, переместите ее в перемешанный на льду колбу Эрленмейера.
- Определите объем раствора в Erlenmeyer.
- Добавляют 10 мг стрептомицина сульфата на каждый 1 мл раствора (перед добавлением растворяют стрептомицина сульфат в 2 мл буфера А).
ПРИМЕЧАНИЕ: Этот шаг выполняется для того, чтобы удалить ДНК и рибосомы. - Перемешивать раствор длительностью 10-20 минут при комнатной температуре.
- Разделите раствор на 50 мл пробирки и центрифугу при 4 °C и со скоростью 20 700 х г в течение 30 минут.
- Соберите супернатант и выбросьте гранулу.
- Приготовьте стерильную колбу Эрленмейера 250 мл на льду с мешалкой.
- Отфильтруйте супернатант с помощью шприцевого фильтра 0,22 мкм в чистую колбу Эрленмейера.
- Определяют объем раствора и взвесят 0,3 г сульфата аммония на каждый 1 мл раствора.
- Постепенно добавляйте сульфат аммония в перемешанный раствор.
ПРИМЕЧАНИЕ: Этот этап выполняется для осаждения белков. Когда сульфат аммония связывает белки, раствор становится затемненным и размытым, что проявляется в виде белого цвета. - Перемешивать в течение 30 минут при комнатной температуре.
- Разделите раствор на 50 мл пробирки и центрифугу при 4 °C и со скоростью 20 700 г в течение 30 минут.
- Осторожно выбросьте супернатант и сохраните гранулу.
- Растворить гранулу с 20 мл буфера А.
- Объедините все растворы в одну трубку, а затем измерьте спектр поглощения УФ-излучения раствора.
ПРИМЕЧАНИЕ: Если спектр показывает сигнатуру агрегации (поглощение выше длины волны 300 нм), перейдите к следующему шагу. Если нет, пропустите следующий шаг и перейдите к шагу 2.1.34.
ПРИМЕЧАНИЕ: Индекс агрегации (A.I.), рассчитанный из УФ-спектров как ODλ=350nm/ (ODλ=280nm - ODλ=350nm) ×100. 41 41 г. - Используйте отсечковые центрипрепные трубки и центрифугу со скоростью 100 кДа со скоростью 3 590 х г при 4 °C в течение 15 минут. Соберите жидкость, прошедшую через фильтр.
- Диализуйте раствор при 4 °C в течение ночи против буфера А, используя диализные мешки с отсечкой 3,5 кДа.
- Выполните еще один раунд диализа продолжительностью не менее 4 часов.
- Загрузите диализованный образец на 1 мл ангионообменный столбец MonoQ. Используйте буфер триса гидрохлорида триса (Tris-HCl) 30 мМ при рН 7,5 в качестве промывного буфера и 30 мМ буфера Трис-HCl рН 7,5 с 500 мМ NaCl в качестве буфера элюдения.
- Ввести образец в столбец. Затем промыть 20 объемами колонки (РЕЗЮМ) 0% буфера элюения и 100% буфера промывки, чтобы удалить несвязанные белки.
- Промывайте 7 резюме 30% буфера элюения.
- Элюируют образец белка α-Syn с помощью градиента, который изменяется от 30% до 100% буфера элюирования для 30 РЕЗЮМ, при скорости потока 1,5 мл/мин.
ПРИМЕЧАНИЕ: α-Syn элюируется при 46-66 РЕЗЮМ при проводимости 20-24 мСм/см, что относится к 38-50% буферу элюирования. - Соберите фракции основного пика элюирования. Проверьте наличие α-Syn, запустив образцы в 15% полиакриламидном электрофорезе из полиакриламида натрия (SDS-PAGE) после окрашивания раствором для окрашивания Coomassie Blue или Fast SeeBand. Ожидается полоса на уровне 15 кДа.
- Унифицируйте соответствующие фракции и диализуйте их в течение ночи против буфера А, используя диализные мешки с отсечкой 3,5 кДа при 4 °C.
- Подготовьте аликвоты образца белка и храните их при -20 °C.
- Cy3-меченый α-синуклеин
- Уменьшают тиол остатка цистеина в мутанте α-Syn A56C путем добавления DTT к конечной концентрации 2 мМ в течение 1 часа при комнатной температуре.
- Удалите DTT, выполнив два раунда диализа с использованием отсечного диализного мешка 3,5 кДа. Для первого раунда диализуйте против 30 мМ Tris-HCl pH 8,0 и 2 мМ ЭДТА. Для второго раунда диализуйте против 50 мМ HEPES pH 7,2 и 2 мМ ЭДТА.
- Уменьшают остатки цистеина путем добавления трис(2-карбоксиэтил)фосфин гидрохлорида (TCEP) в образец белка до конечной концентрации 50 мкМ в течение 30 минут при комнатной температуре.
ПРИМЕЧАНИЕ: TCEP является несульфгидрилредуцированием, поэтому он сохраняет тиольные группы восстановленными, не вступая в реакцию с красителем. Мы добавили 0,5 мкл из запасного раствора 1 М TCEP. - Рассчитайте количество белка, необходимое для конечной реакции объемом 1 мл.
ПРИМЕЧАНИЕ: Вот пример для расчета, который мы использовали:
Конечная концентрация белка 20 мкМ × конечном объеме 1 мл = начальная концентрация белка 56 мкМ × V белка, подлежащего добавлению - Рассчитайте количество красителя, необходимое для конечной реакции объемом 1 мл. Маркировка красителем одного цистеина должна выполняться с избытком красителя, при молярном соотношении краситель:белок не менее 3:1.
ПРИМЕЧАНИЕ: Пример для расчета, который мы использовали:
Конечная концентрация красителя 60 мкМ × конечном объеме 1 мл = начальная концентрация красителя 370 мкМ × V красителя, подлежащего добавлению - Рассчитайте количество буфера из диализата, необходимое для регулировки общего реакционного объема до 1 мл.
ПРИМЕЧАНИЕ: Пример расчета: 1 мл - (V вычисляется из шага 2.2.4 + V вычисляется из шага 2.2.5) - Приготовьте реакционный флакон с магнитом поверх мешалки.
- Во-первых, добавьте расчетное количество белка и буфер. После этого добавляют расчетное количество красителя (сульфо-Cy3 малеймид).
- Держите реакцию при комнатной температуре в темноте в течение 3-5 часов.
- Прекратить реакцию, добавив 2 мМ DTT и продолжать в течение 1 часа.
- Выполните три раунда диализа против буфера А, используя диализные мешки с отсечкой 3,5 кДа для удаления избытка свободного красителя из раствора.
- Загрузите образец в столбец исключения размера, чтобы дополнительно отделить помеченный α-Syn от свободного красителя.
- Определяют концентрацию чистой меченой α-Syn путем измерения поглощения красителя (коэффициент поглощения для сульфо-Cy3 составляет 162 000М-1см-1при λ=548 нм).
- При необходимости сконцентрируйте чистый меченый раствор α-Syn с помощью вакуумного концентратора (например, SpeedVac). Затем выполните один раунд диализа против буфера А, используя диализные мешки с отсечкой 3,5 кДа и снова определите концентрацию меченого белка.
- Подготовьте аликвоты меченого образца белка и храните их при -20 °C.
3. Измерения
- Экспериментальная установка smPIFE
ПРИМЕЧАНИЕ: Используйте следующую конфокальную установку или аналогичную.- Используйте импульсный лазерный источник (в нашем случае λ= 532 нм пикосекундный импульсный лазер с шириной импульса ~100 пс FWHM), работающий с подходящей частотой повторения (20 МГц в нашем случае) и направленный на SYNC карты подсчета одиночных фотонов (TCSPC), в качестве источника возбуждения сульфо-Cy3 (sCy3).
- Используйте дихроичное зеркало с высокой отражательной способностью при 532 нм, чтобы отделить возбуждение и рассеяние от флуоресценции.
- Используйте точечное отверстие диаметром 100 мкм в фокусе излучаемого света, после того, как луч коллимированного излучения был сфокусирован линзой, и до того, как пучок излучения был повторно коллимирован другой линзой.
- Используйте полосовой фильтр (585/40 нм в нашем случае) для дальнейшего фильтра флуоресценции sCy3 от других источников света.
- Обнаружение флуоресценции с помощью детектора (однофотонного лавинного диода или гибридного фотоумножителя, в нашем случае), маршрутизируемого (через маршрутизатор 4-к-1, в нашем случае) в модуль TCSPC (Becker & Hickl SPC-150, в нашем случае).
ПРИМЕЧАНИЕ: В нашем случае сбор данных осуществляется с помощью программного обеспечения VistaVision (ISSTM)в формате файлов с временными метками и временным разрешением (TTTR).
- Пробоподготовка smPIFE
- Приготовьте 25 pM sCy3-меченого α-Syn в буфере измерений: 10 мМ ацетата натрия, 10 мМ дигидрофосфата натрия, 10 мМ глицина pH 8,0, 20 мМ NaCl, 10 мМ цистеамина и 1 мМ 6-гидрокси-2,5,7,8-тетраметилхроман-2-карбоновой кислоты (ТРОЛОКС) в трубке с низким связыванием с белками.
ПРИМЕЧАНИЕ: Если α-Syn измеряется в присутствии везикул SDS, добавьте также соответствующее количество SDS. - Промыть 18-камерный микроскопический покровный слайд 100 мкл 1 мг/мл бытового сывороточного альбумина (BSA) в течение 1 минуты, а затем удалить BSA.
- Добавьте 100 мкл образца α-Syn с маркировкой sCy3 25 pM в камеру на слайде крышки.
- Выполните измерение образца smPIFE с помощью описанной установки, как показано ниже в следующих шагах.
- Приготовьте 25 pM sCy3-меченого α-Syn в буфере измерений: 10 мМ ацетата натрия, 10 мМ дигидрофосфата натрия, 10 мМ глицина pH 8,0, 20 мМ NaCl, 10 мМ цистеамина и 1 мМ 6-гидрокси-2,5,7,8-тетраметилхроман-2-карбоновой кислоты (ТРОЛОКС) в трубке с низким связыванием с белками.
- Сбор данных smPIFE
- Используйте объектив с высокой числовой диафрагмой погружения в воду (в нашем случае Olympus UPLSAPO 60x N.A. 1.2) и добавьте каплю сверхчистой воды поверх объектива.
- Закрепите затвор крышки в сценической камере и установите его поверх сцены микроскопа.
- Поднесите объектив вверх, пока капля воды сверху объектива не смазывается в нижней части затвора.
- Откройте лазерный затвор и поднесите объектив вверх, одновременно осматривая рисунок на КМОП-камере, собирая свет, рассеянный от образца. Обратите внимание на рисунок колец Airy: первый представляет фокус на границе раздела вода-стекло, а затем второй представляет фокус на границе раздела между стеклом и раствором образца.
- Увеличьте высоту объектива еще на 75 мкм, уводя лазерный фокус глубоко в раствор, чтобы свести к минимуму автофлуоресценцию от стеклянной поверхности крышки.
- Настройте мощность лазера на объективе на ~100 мкВт.
- Начните сбор обнаруженных фотонов за заранее определенное время (2 часа, в наших измерениях).
ПРИМЕЧАНИЕ: Большая часть сигнала сбора должна иметь скорость (измеряемую в количестве в секунду, cps), аналогичную той, которая получена при измерении только буфера; миллисекундные данные должны показывать редкие события фотонового всплеска, причем большинство бункеров имеют среднюю скорость, сопоставимую с типичной фоновой скоростью детектора (<1000 cps, в нашем случае).
4. Анализ разрывов smPIFE
- Преобразование необработанных данных
ПРИМЕЧАНИЕ: Данные обычно хранятся в двоичном файле с форматом, который был предопределен компанией, производящей карту TCSPC (файлы .spc в нашем случае).- Преобразование файла необработанных данных в файл photon-HDF5 универсального формата42,используя программный пакет phconvert (https://github.com/Photon-HDF5/phconvert). Вызовите файл необработанных данных в качестве входных данных и преобразуйте в файл необработанных данных .hdf5 с помощью соответствующего кода в пакете phconvert (в нашем случае конвертируйте файлы ns-ALEX Becker-Hickl в записную книжку Jupyter Photon-HDF5.ipynb).
ПРИМЕЧАНИЕ: Преобразование в файл .hdf5 включает: (1) определение соответствующих потоков фотонов в необработанных данных (т.е. потоки фотонов зарегистрированных фотонов соответствующего идентификатора детектора); (2) соответствующие фотонные нано-времена (время обнаружения фотонов относительно времени sync возбуждения); и 3) добавлены метаданные.
- Преобразование файла необработанных данных в файл photon-HDF5 универсального формата42,используя программный пакет phconvert (https://github.com/Photon-HDF5/phconvert). Вызовите файл необработанных данных в качестве входных данных и преобразуйте в файл необработанных данных .hdf5 с помощью соответствующего кода в пакете phconvert (в нашем случае конвертируйте файлы ns-ALEX Becker-Hickl в записную книжку Jupyter Photon-HDF5.ipynb).
- Пакетный поиск и выбор с помощью FRETbursts43
ПРИМЕЧАНИЕ: Все файлы необработанных данных photon-HDF5 измерений smPIFE, а также код, обобщающий анализ необработанных данных, хранятся в Zenodo (https://doi.org/10.5281/zenodo.4587698). Все приведенные ниже шаги подробно описаны и показаны в записных книжках Jupyter, также поставляемых по ссылке на репозиторий Zenodo.- Откройте записные книжки Jupyter (в рамках Anaconda, в нашем случае).
- Откройте ноутбук smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (Final notebook).ipynb (его можно найти по ссылке Zenodo).
- Загрузка ФРЕТСПУГОВОВ.
- Загрузите файл данных фотона HDF5.
- Оценка скорости BG: используя гистограмму межфотонного времени, рассчитайте фоновые (BG) скорости для каждых 30 секунд сбора данных в файле данных фотона HDF5.
ПРИМЕЧАНИЕ: Следующие шаги описывают пакетный поиск с использованием алгоритма скользящего окна44,45,46. - Перемещение временного окна из m=20 последовательных фотонов, по одному фотону за раз.
- Собирать данные фотонов только в том случае, если мгновенная частота фотонов (
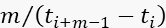 ), по крайней мере, F = 11 раз больше, чем скорость BG за этот период сбора данных.
), по крайней мере, F = 11 раз больше, чем скорость BG за этот период сбора данных.
ПРИМЕЧАНИЕ: Всплеск строится из всех последовательных фотонов, которые были собраны путем скольжения окна по одному фотону за раз (шаг 4.2.6.) и согласования с критерием скорости фотона (шаг 4.2.7). - Рассчитайте следующие характеристики пакетов:
Размер вспышки: количество фотонов во вспышке.
Длительность всплеска: разница во времени между временем обнаружения последнего и первого фотона во время всплеска.
Яркость всплеска: наибольшее значение мгновенной скорости фотона в всплеске.
Разделение пакетов: временной интервал между последовательными всплесками.
ПРИМЕЧАНИЕ: Следующие пункты описывают дальнейшую процедуру выбора пакетов. - Построение гистограммы значений яркости всплеска (самая высокая мгновенная частота фотонов в всплеске) с осью событий в логарифмической шкале.
- Определите порог яркости серийной очереди как минимальное значение яркости серийной вспышки, от которого гистограмма демонстрирует затухающий паттерн.
- Выберите серии со значениями яркости, превышаемыми пороговым значением яркости серийной очереди.
ПРИМЕЧАНИЕ: Следующие шаги описывают время жизни вспышки со средним временем жизни флуоресценции. - Построение гистограммы фотоновых нановрем для всех фотонов во всех выбранных всплесках с осью подсчета фотонов в логарифмическом масштабе.
- Определите порог нановрема как минимальное значение нановрема, из которого гистограмма фотоновых нано-времен демонстрирует паттерн распада.
- Выбирайте только фотоны с нано-временами, превышаемыми порогом нановрем.
- Вычислите алгебраическое среднее всех выбранных фотоновых нановрей.
- Вычтите порог нановрема из среднего алгебраического значения фотона нановремени. Результатом является среднее фотональное нановремя всплеска, которое прямо пропорционально среднему времени жизни флуоресценции.
- График гистограммы всех средних сроков жизни флуоресценции всплеска. Могут появиться централизованно распределенные субпудрения флуоресцентного времени жизни. Субпункции с низкими средними значениями представляют собой молекулярные виды с sCy3, которые не были стерически заблокированы, в то время как субпункции с более высокими средними значениями представляют собой молекулярные виды с sCy3, которые были более стерически заблокированы.
ПРИМЕЧАНИЕ: Следующие шаги описывают медленную динамику между всплесками на основе анализа повторения всплесков47 - Построение гистограммы времени разделения разрывов с осью времени разделения в логарифмической шкале.
ПРИМЕЧАНИЕ: Появятся две субпудрения времени разрыва разделения:
Основная субпунь с временем разделения в секундах, представляющая собой последовательные всплески, происходящие из разных последовательно измеренных молекул.
Незначительная субпопуляция со временем разделения ~ <100 мс, представляющая собой последовательные всплески, происходящие из одной и той же молекулы, повторяющиеся обратно через конфокальный объем. - Выберите, чтобы сохранить все пары последовательных всплесков, которые разделены менее чем максимальным временем разделения, которое определяет субпопулярную популяцию одной молекулы (в нашем случае <100 мс).
- Построение гистограммы или диаграммы рассеяния среднего времени жизни флуоресценции первого и второго всплесков для всех пар всплесков, которые повторялись ниже определенного порога времени разделения
Результаты
Как IDP, когда он не связан с другой биомолекулой, α-Syn демонстрирует структурную динамику между несколькими конформациями с переходами в несколько микросекунд40 и даже в сотни наносекунд41. Когда α-Син пересекает конфокальное пятно, оно может претерпевать тысячи...
Обсуждение
Проведены обширные биохимические и биофизические исследования для изучения структурных характеристик α-Син и его неупорядоченной природы33, 34,35,36,37,38. В нескольких работах уже ис...
Раскрытие информации
Всех авторов нет конфликта интересов.
Благодарности
Плазмида pT-t7, кодировавщая A56C α-Syn мутант, была подарена нам доктором Асафом Групи, доктором Дэном Амиром и доктором Элишей Хаасом. Эта статья была поддержана Национальными институтами здравоохранения (NIH, грант R01 GM130942 для E.L. в качестве субавадры), Израильским научным фондом (грант 3565/20 в рамках программы KillCorona - Сдерживание коронавируса), Фондом Милнера и Еврейским университетом Иерусалима (стартовые фонды).
Материалы
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
Ссылки
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены