このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
時間分解タンパク質誘導蛍光強化を利用して、一度に1つのαシヌクレインモノマーの安定した局所構造を同定する
要約
時間分解された単一分子タンパク質誘導蛍光増強は、タンパク質の局所的な構造変化に敏感な蛍光分光性近接センサとして有用である。ここでは、より長い範囲のFRET定規を使用して測定した場合、球状に非構造化および不安定として知られているα-Synucleinの安定した局所的な立体構造を発見するために使用できることを示す。
要約
分光定規を使用して単一の生体分子の複数の立体構造とそのダイナミクスを追跡することは、構造ダイナミクスの理解と生物学への貢献に革命をもたらしています。FRETベースの定規は3〜10nmの範囲の色間距離について報告するが、タンパク質誘発蛍光増強(PIFE)のような他の分光技術は、色素とタンパク質表面の間の近接性についてより短い0〜3nmの範囲で報告する。選択方法に関係なく、自由に拡散する生体分子を一度に1つずつ測定する際の使用は、実験パラメータのヒストグラムを取り出し、各サブ集団がミリ秒以内に変化しない単一の立体構造、またはミリ秒よりもはるかに速く変換する複数の立体構造を表す実験パラメータのヒストグラムを取得します。ヒストグラム中で報告されたパラメータが色素間FRET効率である単一分子FRETでは、バッファー内のα-Synucleinモノマーのような本質的に障害を持つタンパク質が、急速に相互変換する複数の立体構造の単一の平均アウトサブ集団を示していると以前に報告された。これらの過去の知見はFRETベースの定規の3〜10nmの範囲に依存するが、我々は一度に1つずつサイト特異的なsCy3標識αシヌクレインタンパク質の蛍光寿命を追跡する単一分子PIFEを使用してこのタンパク質を試験に入れようとした。興味深いことに、この短い範囲分光近接センサを使用して、sCy3ラベルα-Synucleinは、10〜100 msで相互変換する明らかに異なる平均寿命を有するいくつかの生涯サブ集団を示す。これらの結果は、α-シヌクレインが世界的に乱れるかもしれないが、それにもかかわらず安定した局所構造を達成することを示している。要約すると、この研究では、局所的またはグローバルな構造変化を一度に1つの生体分子で追跡する異なる分光近接センサーを使用することの利点を強調する。
概要
過去20年間にわたり、単一分子蛍光ベースの方法は、生体分子1、2を測定するための強力なツールとなっており、異なる生体分子パラメータがどのように分布し、これらのパラメータの異なるサブ集団間でこれらのパラメータの異なるサブ集団間でどのように動的に相互変換するかを探究する3、4、5。これらの技術のパラメータは、FRET測定におけるエネルギー伝達効率6、7、蛍光異方性8、9、蛍光量子収率および寿命10、11、異なる蛍光クエンチング12または増強13機構の関数として含まれる。タンパク質誘導蛍光増強(PIFE)14としてよく知られているこれらのメカニズムの1つは、色素14、15、16、17、18、18の近傍のタンパク質表面によって引き起こされる励起状態の蛍光性の自由異性化に対する立体閉塞の機能として、蛍光量子収量および寿命の増強を導入する.FRETとPIFEは、測定されたパラメータが測定中の標識された生体分子内の空間的尺度に直接リンクされているため、分光定規または近接センサーと見なされます。FRET効率は、3〜10 nm20の範囲内の一対の色素間の距離に関連しているが、PIFEは0〜3nm19の範囲で色素と近くのタンパク質の表面との間の距離に関連する蛍光量子収量または寿命の増加を追跡する。
単一分子FRETは、α-シヌクレイン(α-Syn)22など、本質的に障害を持つタンパク質(IDP)21を含む多くの異なるタンパク質系に構造的な洞察を提供するために広く使用されている。α-Synは、異なる生体分子に結合し、異なる条件下で23、24、25、26、27、28、29、30の順序構造を形成することができます。しかし、非結合の場合、α-Synモノマーは、急速に結合する立体31,32との高い立体異質性を特徴とする。
α-Synの立体構造は、このような高度に不均一かつ動的なタンパク質系の立体ダイナミクスを同定するのに役立つ様々な異なる技術を用いて以前に研究されてきた33,34,35,36, 37,38,39.興味深いことに、バッファー内のα-Synの単一分子FRET(smFRET)測定は、単一のFRET集団39,40を報告し、コンフォーカルスポットを介したα-Synの典型的な拡散時間よりもはるかに速く動的に変換されるコンフォメーションの時間平均化の結果である(典型的な拡散時間よりも速い時間、ミリ秒時間に比べて)。 41.しかし、3-10 nm距離感度を有するFRET分光定規を用いて、α-Synのような小さなタンパク質の全体的な構造変化についてしか報告しないこともある。距離感度の低い分光近接センサを利用した単一分子計測は、局所構造のダイナミクスを報告する可能性を秘めています。本明細書では、α-Synの単一分子PIFE測定を行い、蛍光寿命の異なるサブ集団を特定し、それらの間の遷移を100ミリ秒と遅くして異なる局所構造にマッピングする。この研究は、緩衝剤中および短距離単一分子分光近接センサとしてSDSベースの膜に結合した場合に、一度に1つずつ、自由に拡散するα-Syn分子の時間分解smPIFE測定を要約する。
プロトコル
1. プラスミド変換
- SOC培地の0.5Lの調製
- トリプトン10g、酵母エキス2.5g、0.25gの塩化ナトリウム(NaCl)、0.1gの塩化カリウム(KCl)を秤量する。
- 総容量が0.5 Lになるまで、二重蒸留水(DDW)を加えます。
- 水酸化ナトリウム(NaOH)を加えてpH7に調整します。
- 100 mL とオートクレーブのストックアリコートを用意します。
- 使用前に、無菌塩化マグネシウム(MgCl2)と1.8mLの無菌グルコースを100mLのSOCに加えます。
- 氷上BL21(DE3)有能 な大腸菌 細胞を解凍します。
- 有能な細胞を優しく混ぜます。
- 2つの15 mLチューブを準備します。1つを変換反応チューブとしてラベルし、もう一方をコントロールチューブとしてラベル付けします。
- 管に有能な細胞のアリコート100 μL。
- 0.1 ng/μLの濃度で1μLのプラスミドを変換反応チューブに加えます。
- SOC培地をステップ1.8で使用するために42°Cの水浴に加熱する。
- 42°Cの水浴で2本のチューブを 45秒間熱パルスします。(重要なステップ!
- 各チューブに900μLの加熱されたSOC培地を加えます。
- 225 rpmの速度で振って45分間、37°Cでチューブをインキュベートします。
- 200 μLの細胞を、形質転換したプラスミドを含む細胞を1つのLB(ルリア・ベルタニ)-アンピシリンを含む寒天プレートに、無菌細胞スプレッダーを使用して広げます。
- 200 μLの細胞を、形質転換したプラスミドをアンピシリンを使わずに1つのLB-寒天プレートに広げ、滅菌拡散機 (陽性対照)を使用する。
- 滅菌スプレッダー(陰性対照)を使用して、100μg/mLアンピシリンを含む1つのLB寒天プレート上にコントロールセル(プラスミドを含まない)の200μLを広げる。
- プレートを一晩37°Cでインキュベートする。
2. タンパク質調製
- 組換えα-シヌクレインの発現と精製
- 1つのコロニーを選び、37°C( スターター)で100 μg/mLアンピシリンを含むオートクレーブLB(ルリア・ベルタニ)液体培地の100 mLで成長する。
- 4つの2 Lエルレンマイヤーフラスコを準備し、それぞれ1 LのオートクレーブLB培地と100 μg/mLアンピシリン (大接種)を含む。
- セル溶液の光学密度(Λ=600nmのOD)を測定します。スターターの細胞密度が0.6-0.7のODλ=600nm に達したら、前のステップで調製したLB培地の1Lごとに10mLのスターター溶液を加えます。
- 37°Cで培養器シェーカーで細菌培地を成長させ、200rpmの速度を持ちます。
- 細胞密度が0.6-0.8のODλ=600nm に達すると、1Lの成長培地毎にイソプロピルβ-d-1-チオガラクトピラノシド(IPTG)を1mMの最終濃度に添加してタンパク質発現を誘導します。
注:DDWの4 mLにIPTGの0.96 gを溶解し、細菌の増殖の各1 Lごとに得られるIPTG溶液の1 mLを加えます。 - 4時間の持続時間のために細胞を成長させ、8分間の間5,170 x gの速度で50 mLの管で遠心分離によってそれらを集める。
注:各ラウンドでは、上清を捨て、ペレットを保持し、細菌の成長で再び同じチューブを充填します。 - 翌日の深い凍結保存(例えば、-80°C)に細菌ペレットを保管してください。
- 翌日、200 mLのリシスバッファーを調製します:40%(w/v)ショ糖およびバッファA、これには30 mM Tris-HCl、2 mMエチレンアミンテア酢酸(EDTA)、2 mMジチオスライトール(DTT)が含まれています。
- 細菌ペレットを含む各チューブに25 mLのライシスバッファーを加えます。
- リシスバッファー内のペレットを再中断(滅菌プラスチックパスツールピペットを使用)(2.1.8参照)。
- 氷の上にスターラーマグネットで滅菌250 mLエルレンマイヤーフラスコを準備します。
- 5 mLのライシスバッファーを加え、均質な再懸濁細胞をそれに移します。
- 室温で200rpmで20分間細胞をかき混ぜます。
- 溶液を50mLチューブに分け、4°Cで遠心分離機を17,400 x gの速度で30分間分けます。
- 上清を捨てます。
- 氷の上にスターラーマグネットと滅菌250 mLエルレンマイヤーフラスコを準備します。
- 溶解バッファー(90 mL冷却バッファーAとMgCl2 の37 μL)を溶解度を超えて溶解し、各チューブに加えます。滅菌プラスチックパスツールピペットを使用し、ペレットを溶解します。
- 溶液の一部が均質になったら、氷の上で攪拌されたエルレンマイヤーフラスコに移動します。
- エルレンマイヤーの溶液容積を決定します。
- 1 mLの溶液毎に10mgのストレプトマイシン硫酸塩を加える(添加前に、ストレプトマイシン硫酸塩を2mLの緩衝液Aの中に溶解する)。
注:このステップは、DNAとリボソームを取り除くために行われます。 - 室温で10〜20分間溶液をかき混ぜます。
- 溶液を4°Cで50mLチューブと遠心分離機に分け、30分間20,700 x gの速度で分けます。
- 上清を集め、ペレットを捨てます。
- スターラーで氷の上に滅菌250 mLエルレンマイヤーフラスコを準備します。
- 0.22 μmのシリンジフィルターを使用して上清をきれいなエルレンマイヤーフラスコにフィルターします。
- 溶液量を決定し、溶液1mL当たり0.3gの硫酸アンモニウムを測定する。
- 徐々に、攪拌液に硫酸アンモニウムを加える。
注:このステップは、タンパク質を沈殿させるために行われます。硫酸アンモニウムがタンパク質に結合すると、溶液が不明瞭になり、ぼやけて、白色で現れる。 - 室温で30分間かき混ぜます。
- 溶液を4°Cで50mLチューブと遠心分離機に分け、30分間20,700gの速度で割ります。
- 上清を慎重に捨て、ペレットを保管してください。
- ペレットを20mLの緩衝Aで溶解する。
- すべての溶液を1つのチューブに統一し、溶液のUV吸収スペクトルを測定します。
注: スペクトルが凝集のシグネチャ(波長300 nm以上の吸収)を示している場合は、次のステップに進みます。ない場合は、次のステップをスキップし、ステップ 2.1.34 に進みます。
注: UV スペクトルから ODλ=350nm/ (ODλ=280nm - ODλ=350nm)×100として計算される凝集指数(A.I.)41 - 15分間、4°Cで3,590 x gの速度で100kDaカットオフ遠心管と遠心分離機を使用してください。フィルターを通過した液体を収集します。
- 3.5 kDaカットオフの透析袋を使用して、バッファーAに対して、一晩4°Cで溶液を透析する。
- 少なくとも4時間の間、透析の別のラウンドを行います。
- 透析サンプルを1 mL MonoQアニオン交換カラムに積み込みます。pH 7.5で30 mM塩酸塩(Tris-HCl)バッファーを洗浄バッファーとして使用し、溶出バッファーとして500 mM NaClを備えた30 mM Tris-HClバッファーpH 7.5を使用します。
- 列にサンプルを挿入します。次に、20カラムボリューム(CV)を0%溶出バッファと100%洗浄バッファで洗浄し、アンバウンドタンパク質を除去します。
- 30%溶出バッファーの7 CVで洗浄します。
- α-Synタンパク質サンプルを、30 CVの30%から100%溶出バッファーに、流量1.5mL/minで変化する勾配を使用して溶出します。
注:α-Synは、38-50%溶出バッファを指す導電率20-24 mS/cmで46-66 CVで溶出します。 - 主溶出ピークの分数を収集します。15%のドデシル硫酸ナトリウムポリアクリルアミドゲル電気泳動(SDS-PAGE)でサンプルを実行し、クーマシーブルーまたはファストシーバンド染色液のいずれかで染色することにより、α-Synの存在を確認します。15 kDa のバンドが期待されます。
- 関連する分数を一元化し、4°Cで3.5 kDaカットオフした透析袋を使用して、バッファAに対して一晩透析します。
- タンパク質サンプルのアリコートを準備し、-20°Cで保存します。
- Cy3ラベルのα-シヌクレイン
- α-Syn A56C変異体中のシステイン残基のチオールを、室温で1時間の間、2 mMの最終濃度に加えることによって、システイン残基のチオールを減少させる。
- 3.5 kDa カットオフ透析バッグを使用して 2 ラウンドの透析を行うことで DTT を取り外します。第1ラウンドでは、30 mMトリス-HCl pH 8.0および2 mM EDTAに対して透析を行います。第2ラウンドでは、50 mM HEPES pH 7.2および2 mM EDTAに対して透析を行います。
- トリス(2-カルボキイェチル)ホスフィン塩酸塩(TCEP)をタンパク質サンプルに加え、室温で30分間の最終濃度50μMにすることで、システイン残基を減らします。
注:TCEPは非スルフヒドリル還元剤であるため、色素と反応することなくチオール基を減少させ続けます。1 M TCEPのストック溶液から0.5 μLを追加しました。 - 1 mLの最終反応量に必要なタンパク質の量を計算します。
注: ここでは、使用した計算の例を示します。
20 μM 最終タンパク質濃度 × 1 mL 最終体積 = 56 μM 初期タンパク質濃度 × V タンパク質を追加する - 1mLの最終反応量に必要な染料量を計算します。単一システインの色素標識は、過剰な色素で、染料:タンパク質モル比で少なくとも3:1で行われるべきである。
注: 使用した計算の例:
60 μM 最終染料濃度 ×1 mL 最終体積 = 370 μM 初期染料濃度 ×V 染料を追加する - 合計反応量を1mLに調整するために必要な透析液からバッファー量を計算します。
注:計算例:1 mL - (ステップ2.2.4 + Vから計算されたV) - 攪拌機の上に磁石で反応バイアルを準備します。
- まず、計算量のタンパク質とバッファーを加算します。その後、計算した量の色素(スルホ-Cy3マレイミド)を加える。
- 3〜5時間の間、暗闇の中で室温で反応を保ちます。
- 2 mM DTTを加えて反応を終了し、1時間継続する。
- バッファAに対して3ラウンドの透析を行い、3.5kDaカットオフの透析バッグを使用して溶液から余分な自由染料を除去します。
- サイズ除外列にサンプルをロードして、ラベルの付いたα-Synをフリー仕分けからさらに分離します。
- 色素の吸収を測定することにより、純正標識α-Synの濃度を決定します(sulfo-Cy3の吸収係数はλ=548 nmで162,000 M-1cm-1です)。
- 必要に応じて、真空濃縮器(SpeedVacなど)を使用して、純粋なラベル付きα-Syn溶液を濃縮します。次に、3.5kDaカットオフを有する透析袋を用いて、バッファーAに対して1ラウンドの透析を行い、標識タンパク質の濃度を再び決定する。
- 標識したタンパク質サンプルのアリコートを調製し、-20°Cで保存します。
3. 測定
- smPIFE 実験セットアップ
注: 次の共焦点ベースのセットアップまたは同様の設定を使用します。- パルスレーザーソース(当社の場合、パルス幅が〜100 ps FWHMのパルス幅を持つλ = 532 nmピコ秒パルスレーザー)を使用し、適切な繰り返しレート(当社の場合は20MHz)で動作し、時間相関単一光子計数(TCSPC)カードのSYNCに対して、スルフォ-Cy3(sCy3)励起のソースとして使用します。
- 532 nmで高反射率の二色性ミラーを使用して、励起および散乱を蛍光から分離します。
- 照射された光の焦点で直径100μmのピンホールを使用し、コリメートされた発光ビームをレンズで集めた後、放出ビームが別のレンズによって再コリメートされる前に使用します。
- バンドパスフィルタ(当社の場合は585/40 nm)を使用して、他の光源からsCy3蛍光をさらにフィルタリングします。
- 検出器(当社の場合はシングルフォトテンアバランシェダイオードまたはハイブリッドフォトマルチプライヤ)を使用して蛍光を検出し、(当社の場合は4対1のルータを介して)TCSPCモジュール(当社の場合はベッカー&ヒックルSPC-150)にルーティングします。
注: この場合、データ取得は、時間タグ付き時間解決(TTTR)ファイル形式で VistaVision ソフトウェア (ISSTM)を介して実行されます。
- smPIFE サンプル調製
- 測定バッファーに25 pM sCy3標識α-Synを調製:10 mM酢酸ナトリウム、10 mM二水素リン酸ナトリウム、10 mMグリシンpH 8.0、20 mM NaCl、10 mMシステアミンおよび1mM 6-ヒドロキシ-2、5,7,8-テトラメチルクロマン-2-カルボリック酸(TROX結合)、低タンパク質
注:α-SynがSDS小胞の存在下で測定される場合は、同様に適切なSDS量を追加します。 - 18室の顕微鏡カバースリップスライドを1mg/mLのウシ血清アルブミン(BSA)の100 μLで1分間リンスし、BSAを取り除きます。
- カバースリップスライドのチャンバーに、25 pM sCy3 ラベルのα Syn サンプルの 100 μL を追加します。
- 次の手順で、次の手順で説明した設定を使用してサンプルのsmPIFE測定を行います。
- 測定バッファーに25 pM sCy3標識α-Synを調製:10 mM酢酸ナトリウム、10 mM二水素リン酸ナトリウム、10 mMグリシンpH 8.0、20 mM NaCl、10 mMシステアミンおよび1mM 6-ヒドロキシ-2、5,7,8-テトラメチルクロマン-2-カルボリック酸(TROX結合)、低タンパク質
- smPIFE データ取得
- 高い開口水浸し対物レンズ(我々の場合はオリンパスUPLSAPO 60x N.A.1.2)を使用し、対物レンズの上に超純水の滴を加えます。
- ステージチャンバーのカバースリップスライドを固定し、顕微鏡ステージの上に取り付けます。
- カバースリップスライドの下部にある対物レンズの上の水滴が塗りつぶされるまで、対物レンズを上向きに持ち上げます。
- レーザーシャッターを開き、対物レンズを上方に持ち上げ、CMOSカメラでパターンを検査しながら、サンプルから散乱した光を集めます。Airyリングパターンを観察:最初の1つは水ガラスインターフェースでの焦点を表し、2つ目はガラスとサンプル溶液の間の界面での焦点を表します。
- 対物レンズの高さをさらに75 μm高くし、レーザーフォーカスを溶液の奥深くに持ち込み、カバースリップのガラス表面からの自動蛍光を最小限に抑えます。
- 対物レンズでレーザーパワーを100μW程度に調整します。
- 検出されたフォトンの取得を事前に定義された時間(測定では2時間)開始します。
注: 取得信号の大部分は、バッファのみを測定する場合に取得したレート(秒あたりのカウント数、cps)を持っている必要があります。ミリ秒単位のデータは、ビトンバーストイベントが不足しており、ビンの大部分は典型的な検出器のバックグラウンドレート(<1,000 cps)に匹敵する平均レートを持つ必要があります。
4. smPIFE バースト解析
- 生データ変換
注: データは通常、TCSPC カード (私たちの場合は .spc ファイル) を製造する会社によって事前定義された形式のバイナリ ファイルに格納されます。- ソフトウェアスイートphconvert(https://github.com/Photon-HDF5/phconvert)を使用して、生のデータファイルをphoton-HDF5ユニバーサルファイル形式42に変換します。生データファイルを入力として呼び出し、phconvertスイート(私たちの場合は、photon-HDF5.ipynb Jupyterノートブックにns-ALEXベッカー-ヒッケルファイルを変換する)で適切なコードを使用して、生データ.hdf5ファイルに変換します。
注:hdf5ファイルへの変換には、(1)生データ内の関連するフォトンストリーム(すなわち、関連する検出器IDの登録されたフォトンのフォトンストリーム)を決定するが含まれます。(2)関連する光子ナノタイム(励起SYNC時間に対する光子検出時間)。(3) メタデータを追加しました。
- ソフトウェアスイートphconvert(https://github.com/Photon-HDF5/phconvert)を使用して、生のデータファイルをphoton-HDF5ユニバーサルファイル形式42に変換します。生データファイルを入力として呼び出し、phconvertスイート(私たちの場合は、photon-HDF5.ipynb Jupyterノートブックにns-ALEXベッカー-ヒッケルファイルを変換する)で適切なコードを使用して、生データ.hdf5ファイルに変換します。
- バースト検索と選択 FRETbursts43
注: smPIFE 測定値のフォトン HDF5 の生データ ファイルの全てと、生データの分析をまとめたコードは、ゼノド(https://doi.org/10.5281/zenodo.4587698)に保存されています。以下のすべての手順は、Zenodoリポジトリリンクにも記載されているJupyterノートブック内で詳細かつ表示されています。- ジュピター ノートブックを開きます (この場合、アナコンダ のフレームワーク内)。
- ノートブック smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (最終ノートブック) を開きます(ゼノドリンクにあります)。
- フレツバーストをロードする。
- フォトン HDF5 データ ファイルを読み込みます。
- BGレート評価:光間時間のヒストグラムを用いて、フォトンHDF5データファイル内のデータ取得の30秒ごとにバックグラウンド(BG)レートを計算する。
注: 次の手順では、スライディング ウィンドウ アルゴリズム44、45、46を使用したバースト検索について説明します。 - 連続するフォトン m=20 のタイム ウィンドウを移動します(一度に 1 つのフォトン)。
- 瞬間光子レート(
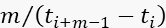 )が、データ取得のその期間のBGレートよりも少なくともF=11倍大きい場合にのみ、フォトンデータを収集します。
)が、データ取得のその期間のBGレートよりも少なくともF=11倍大きい場合にのみ、フォトンデータを収集します。
注:バーストは、一度に1光子を窓をスライドさせて集めた連続するすべてのフォトン(ステップ4.2.6.)から構築され、フォトンレート基準(ステップ4.2.7)に同意します。 - 次のバースト特性を計算します。
バーストサイズ:バースト内のフォトンの量。
バースト時間: バーストの最後のフォトン検出時間と最初のフォトン検出時間の時間差。
バースト輝度: バーストの瞬間光子レートの最大値。
バースト分離: 連続するバースト間の時間間隔。
注: 次の点は、さらにバースト選択手順を説明します。 - バースト輝度値のヒストグラム (バーストで最も高い瞬間光度) を、イベントの軸を対数スケールでプロットします。
- バースト輝度しきい値を、ヒストグラムが減衰パターンを示す最小バースト輝度値として定義します。
- 明るさの値がバースト輝度しきい値より大きいバーストを選択します。
注: 次のステップでは、バースト平均蛍光寿命について説明します。 - 選択したすべてのバーストのすべてのフォトンのフォトンナノタイムのヒストグラムを、フォトンカウント軸を対数スケールでプロットします。
- ナノ時間閾値を、光ナノ時間のヒストグラムが減衰パターンを示す最小ナノタイム値として定義する。
- ナノタイムしきい値よりナノ倍大きいフォトンのみを選択します。
- 選択したすべてのフォトンナノタイムの代数平均を計算します。
- 光子ナノ時間代数平均からナノタイム閾値を差し引く。結果はバーストの平均光子ナノタイムであり、平均蛍光寿命に直接比例する。
- 全バースト平均蛍光寿命のヒストグラムをプロットする。蛍光寿命の中央分布サブ集団が出現し得る。低い値の平均を持つサブ集団は、立体的に妨害されなかったsCy3を有する分子種を表し、より高い値平均を有するサブ集団は、より立体的に妨害されたsCy3を有する分子種を表す。
注:次のステップは、バースト再発分析に基づいて低速のバースト間ダイナミクスを説明します 47 - バースト分離時間のヒストグラムを、分離時間軸を対数スケールでプロットします。
注: バースト分離時間の 2 つのサブ集団が表示されます。
分離時間が秒の主要なサブ集団で、連続して測定された異なる分子から発生する連続バーストを表す。
分離時間が~<100ミリ秒のマイナーサブ集団で、同じ分子から発生した連続バーストを表し、共焦点体積を通して繰り返される。 - 同一分子サブ集団(当社の場合は <100 ms)を定義する最大分離時間未満で分離された連続するバーストのペアをすべて保存します。
- 特定の分離時間しきい値を下回るバーストのすべてのペアに対して、最初と2番目のバーストの平均蛍光寿命のヒストグラムまたは散布図をプロットする
結果
IDPとして、別の生体分子に結合していない場合、α-Synは、数マイクロ秒40、さらには数百ナノ秒41での遷移を伴う複数の立体間の構造ダイナミクスを示す。α-Synが共焦点を越えるとき、それは立体構造間の何千もの遷移を受けるかもしれません。確かに、これはsmFRETが39、40を使用した場合でした。ここでは、α-Syn...
ディスカッション
広範な生化学的および生物物理学的研究は、α-Synとその無秩序な性質33、34、35、36、37、38の構造的特徴を研究するために行われた。すでに自由に拡散するsmFRETを利用して、結合のないα-Synモノマーの分子内ダイナミクスを調べるいくつかの作品...
開示事項
すべての著者は利益相反を共有していません。
謝辞
A56C α-Syn変異体をコードするpT-t7プラスミドは、アサフ・フルピ博士、ダン・アミール博士、エリシャ・ハース博士からのプレゼントとして私たちに与えられました。この論文は、国立衛生研究所(NIH、サブアワードとしてE.L.にR01 GM01 GM130942を付与)、イスラエル科学財団(キルコロナ - コロナウイルス研究プログラムの抑制内で3565/20を付与)、ミルナー基金、エルサレムヘブライ大学(スタートアップ資金)によって支持されました。
資料
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
参考文献
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved