Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verwendung einer zeitaufgelösten proteininduzierten Fluoreszenzverstärkung zur Identifizierung stabiler lokaler Konformationen jeweils α-Synuclein-Monomer
In diesem Artikel
Zusammenfassung
Die zeitaufgelöste Einzelmolekül-Protein-induzierte Fluoreszenzverstärkung ist ein nützlicher fluoreszenzspektroskopischer Näherungssensor, der empfindlich auf lokale Strukturveränderungen in Proteinen reagiert. Hier zeigen wir, dass es verwendet werden kann, um stabile lokale Konformationen in α-Synuclein aufzudecken, das auch als globulär unstrukturiert und instabil bezeichnet wird, wenn es mit dem FRET-Lineal mit größerer Reichweite gemessen wird.
Zusammenfassung
Die Verwendung spektroskopischer Lineale zur Verfolgung mehrerer Konformationen einzelner Biomoleküle und ihrer Dynamik hat das Verständnis der Strukturdynamik und ihrer Beiträge zur Biologie revolutioniert. Während das FRET-basierte Lineal über Interfarbstoffabstände im Bereich von 3-10 nm berichtet, berichten andere spektroskopische Techniken, wie die proteininduzierte Fluoreszenzverstärkung (PIFE), über die Nähe zwischen einem Farbstoff und einer Proteinoberfläche im kürzeren Bereich von 0-3 nm. Unabhängig von der gewählten Methode ruft seine Verwendung bei der Messung frei diffuser Biomoleküle einzeln Histogramme des experimentellen Parameters ab, die separate zentral verteilte Teilpopulationen von Biomolekülen ergeben, wobei jede Teilpopulation entweder eine einzelne Konformation darstellt, die innerhalb von Millisekunden unverändert blieb, oder mehrere Konformationen, die viel schneller als Millisekunden ineinander übergehen, und damit eine gemittelte Teilpopulation. Bei Einzelmolekül-FRET, bei dem der in Histogrammen berichtete Parameter die Interfarbstoff-FRET-Effizienz ist, wurde zuvor berichtet, dass ein intrinsisch ungeordnetes Protein, wie das α-Synuclein-Monomer im Puffer, eine einzige gemittelte Teilpopulation mehrerer Konformationen aufweist, die sich schnell ineinander überlagern. Während diese früheren Ergebnisse vom 3-10 nm-Bereich des FRET-basierten Lineals abhängen, haben wir versucht, dieses Protein mit Einzelmolekül-PIFE zu testen, wo wir die Fluoreszenzlebensdauer von ortsspezifischen sCy3-markierten α-Synuclein-Proteinen nacheinander verfolgen. Interessanterweise zeigt der sCy3-markierte α-Synuclein mit diesem spektroskopischen Näherungssensor mit kürzerer Reichweite mehrere lebenslange Subpopulationen mit deutlich unterschiedlichen mittleren Lebenszeiten, die sich in 10-100 ms interkonvertieren. Diese Ergebnisse zeigen, dass α-Synuclein zwar global ungeordnet ist, aber dennoch stabile lokale Strukturen erreicht. Zusammenfassend heben wir in dieser Arbeit den Vorteil der Verwendung verschiedener spektroskopischer Näherungssensoren hervor, die lokale oder globale Strukturveränderungen jeweils ein Biomolekül nach dem anderen verfolgen.
Einleitung
In den letzten zwei Jahrzehnten haben sich Einzelmolekül-Fluoreszenz-basierte Methoden zueinemleistungsfähigen Werkzeug zur Messung von Biomolekülen 1 ,2entwickelt,um zu untersuchen, wie sich verschiedene biomolekulare Parameter verteilen und wie sie dynamisch zwischen verschiedenen Subpopulationen dieser Parameter mit einer Auflösung von submillisekunden3,4,5interkonvertieren. Die Parameter in diesen Techniken umfassen die Energieeffizienz in den FRET-Messungen 6,7, Fluoreszenzanisotropie8,9, Fluoreszenzquantenausbeuten undLebensdauern 10,11, als Funktion verschiedener Fluoreszenzlöschungsmechanismen12 oderVerstärkung 13. Einer dieser Mechanismen, besser bekannt als proteininduzierte Fluoreszenzverstärkung (PIFE)14, führt die Verbesserung der Fluoreszenzquantenausbeute und -lebensdauer als Funktion der sterischen Obstruktion der freien Isomerisierung des Fluorophors im angeregten Zustand ein, verursacht durch Proteinoberflächen in der Nähe des Farbstoffs14,15,16,17,18,19 . Sowohl FRET als auch PIFE gelten als spektroskopische Lineale oder Näherungssensoren, da ihr gemessener Parameter direkt mit einer räumlichen Messung innerhalb des markierten zu messenden Biomoleküls verbunden ist. Während die FRET-Effizienz mit dem Abstand zwischen einem Farbstoffpaar in einem Bereich von 3-10 nm20zusammenhängt, verfolgt PIFE Erhöhungen der Fluoreszenzquantenausbeuten oder Lebensdauern in Bezug auf den Abstand zwischen dem Farbstoff und einer Oberfläche eines nahe gelegenen Proteins im Bereich von 0-3 nm19.
Einzelmolekül-FRET wurde häufig verwendet, um strukturelle Einblicke in viele verschiedene Proteinsysteme zu geben, einschließlich intrinsisch ungeordneter Proteine (IDPs)21,wie α-Synuclein (α-Syn)22. α-Syn kann nach Bindung an verschiedene Biomoleküle und unter verschiedenenBedingungen geordneteStrukturen bilden 23,24,25,26,27,28,29,30. Wenn es jedoch ungebunden ist, zeichnet sich das α-Syn-Monomer durch eine hohe Konformationsheterogenität mit schnell ineinander überlagernden Konformationenaus 31,32.
Die Konformationen von α-Syn wurden zuvor mit verschiedenen Techniken untersucht, die bei der Identifizierung der Konformationsdynamik solcher hochheterogenen und dynamischen Proteinsystemehelfen 33,34,35,36,37,38,39. Interessanterweise ergaben Einzelmolekül-FRET-Messungen (smFRET) von α-Syn im Puffer eine einzelne FRET-Population39,40, die ein Ergebnis der Zeitmittelung von Konformationen ist, die sich manchmal viel schneller als die typische Diffusionszeit von α-Syn durch den konfokalen Punkt dynamisch interkonvertieren (mal so schnell wie wenige Mikrosekunden und sogar schneller als das, relativ zu typischen Millisekunden-Diffusionszeiten)40, 41. Die Verwendung eines SPEKT-SPEKT-Lineals mit der Entfernungsempfindlichkeit von 3-10 nm berichtet jedoch manchmal nur über allgemeine strukturelle Veränderungen in einem kleinen Protein wie α-Syn. Einzelmolekülmessungen mit spektroskopischen Näherungssensoren mit kürzeren Entfernungsempfindlichkeiten haben das Potenzial, über die Dynamik lokaler Strukturen zu berichten. Hierin führen wir Einzelmolekül-PIFE-Messungen von α-Syn durch und identifizieren verschiedene Subpopulationen von Fluoreszenzlebenszeiten, die auf verschiedene lokale Strukturen mit Übergängen zwischen ihnen von bis zu 100 ms abgebildet sind. Diese Arbeit fasst zeitaufgelöste smPIFE-Messungen von frei diffundierenden α-Syn-Molekülen einzeln, in Puffer und gebunden an SDS-basierte Membranen als SDS-basierten Einzelmolekül-Näherungssensor mit kurzer Reichweite zusammen.
Protokoll
1. Plasmid-Transformation
- Herstellung von 0,5 l SOC-Medium
- Wiegen Sie 10 g Trypton, 2,5 g Hefeextrakt, 0,25 g Natriumchlorid (NaCl), 0,1 g Kaliumchlorid (KCl).
- Doppelt destilliertes Wasser (DDW) bis zum Gesamtvolumen von 0,5 l zugeben.
- Auf pH 7 einstellen, indem Sie Natriumhydroxid (NaOH) hinzufügen.
- Bereiten Sie Vorrat aliquots von 100 ml und Autoklav vor.
- Vor der Anwendung werden 0,5 ml steriles Magnesiumchlorid(MgCl2)und 1,8 ml sterile Glukose zu 100 ml SOC gegeben.
- Bl21 (DE3) kompetente Escherichia coli-Zellen auf Eis auftauen.
- Mischen Sie die kompetenten Zellen vorsichtig.
- Bereiten Sie zwei 15-ml-Röhrchen vor. Kennzeichnen Sie eines als Transformationsreaktionsrohr und das andere als Steuerrohr.
- Aliquot 100 μL kompetente Zellen in die Röhrchen.
- 1 μL Plasmid in einer Konzentration von 0,1 ng/μL in das Umwandlungsreaktionsröhrchen geben.
- SOC-Medium im 42 °C Wasserbad zur Verwendung in Schritt 1.8 erhitzen.
- Wärmeimpuls der beiden Röhren im 42 °C Wasserbad für eine Dauer von 45 Sekunden. (Kritischer Schritt!)
- Jedem Röhrchen werden 900 μL erhitztes SOC-Medium zugegeben.
- Inkubieren Sie die Röhren bei 37 °C für eine Dauer von 45 Minuten mit einer Geschwindigkeit von 225 U / min.
- Verteilen Sie 200 μL der Zellen mit dem transformierten Plasmid auf einer LB (Luria-Bertani)-Agarplatte mit 100 μg/ml Ampicillin unter Verwendung eines sterilen Zellspreaders.
- Verteilen Sie 200 μL der Zellen mit dem transformierten Plasmid auf eine LB-Agarplatte ohne Ampicillin mit einem sterilen Spreader (Positivkontrolle).
- Verteilen Sie 200 μL der Kontrollzellen (ohne Plasmid enthaltend) auf eine LB-Agarplatte mit 100 μg/ml Ampicillin unter Verwendung eines sterilen Spreaders (Negativkontrolle).
- Die Platten über Nacht bei 37 °C inkubieren.
2. Proteinzubereitung
- Rekombinante α-Synuclein-Expression und -Reinigung
- Wählen Sie eine einzelne Kolonie und züchten Sie in 100 ml autoklaviertem LB (Luria-Bertani) flüssigem Medium mit 100 μg / ml Ampicillin bei 37 ° C (Starter).
- Vier 2 L Erlenmeyerkolben mit jeweils 1 l autoklaviertem LB-Medium und 100 μg/ml Ampicillin (Großimpfung)herstellen.
- Messen Sie die optische Dichte (OD bei λ = 600 nm) der Zelllösung. Wenn die Zelldichte des Starters einen ODλ = 600 nm von 0,6-0,7 erreicht, fügen Sie 10 mL Starterlösung pro 1 l LB-Medium hinzu, das im vorherigen Schritt hergestellt wurde.
- Züchten Sie die Bakterienmedien in einem Inkubator-Shaker bei 37 °C und einer Geschwindigkeit von 200 U /min.
- Wenn die Zelldichte einen ODλ = 600 nm von 0,6-0,8 erreicht, induzieren Sie die Proteinexpression durch Zugabe von Isopropyl-β-d-1-thiogalactopyranosid (IPTG) pro 1 l Wachstumsmedium bis zu einer Endkonzentration von 1 mM.
ANMERKUNG: Lösen Sie 0,96 g IPTG in 4 ml DDW auf und fügen Sie 1 mL der resultierenden IPTG-Lösung pro 1 l Bakterienwachstum hinzu. - Züchten Sie die Zellen für eine Dauer von 4 Stunden und sammeln Sie sie durch Zentrifugation in 50 ml Röhrchen mit einer Geschwindigkeit von 5.170 x g für eine Dauer von 8 Minuten.
HINWEIS: Verwerfen Sie in jeder Runde den Überstand, bewahren Sie das Pellet auf und füllen Sie die gleichen Röhrchen erneut mit Bakterienwachstum. - Lagern Sie das Bakterienpellet für den nächsten Tag im Tiefkühllager (z.B. -80 °C).
- Bereiten Sie am nächsten Tag 200 ml Lysepuffer vor: 40% (w / v) Saccharose und Puffer A, der 30 mM Tris-HCl, 2 mM Ethylendiamintetraessigsäure (EDTA), 2 mM Dithiothreitol (DTT) bei pH 8,0 enthält.
- Fügen Sie 25 ml Lysepuffer zu jedem Röhrchen mit Bakterienpellet hinzu.
- Suspendieren Sie das Pellet (sterile Pasteur-Pipetten aus Kunststoff verwenden) im Lysepuffer (siehe 2.1.8).
- Sterilen 250 mL Erlenmeyerkolben mit einem Rührmagneten auf Eis vorbereiten.
- Fügen Sie 5 ml Lysepuffer hinzu und übertragen Sie dann die homogenen, wieder suspendierten Zellen darauf.
- Rühren Sie die Zellen für eine Dauer von 20 Minuten bei 200 U / min bei Raumtemperatur um.
- Die Lösung in 50-ml-Röhrchen teilen und bei 4 °C für eine Dauer von 30 Minuten mit einer Geschwindigkeit von 17.400 x g zentrifugieren.
- Verwerfen Sie den Überstand.
- Sterilen 250 mL Erlenmeyerkolben mit Rührermagnet auf Eis vorbereiten.
- In jedes Röhrchen werden 15 mL Auflösungspuffer (90 ml gekühlter Puffer A und 37 μLMgCl2 über die Löslichkeitsgrenze hinaus gelöst und filtriert) zugegeben. Verwenden Sie sterile Pasteur-Pipetten aus Kunststoff und lösen Sie das Pellet auf.
- Wenn ein Teil der Lösung homogen wird, wird er in einen gerührten Erlenmeyerkolben auf Eis bewegt.
- Bestimmen Sie das Lösungsvolumen im Erlenmeyer.
- Fügen Sie 10 mg Streptomycinsulfat pro 1 ml Lösung hinzu (vor der Zugabe lösen Sie Streptomycinsulfat in 2 ml Puffer A auf).
HINWEIS: Dieser Schritt wird durchgeführt, um die DNA und ribosomen zu entfernen. - Rühren Sie die Lösung für eine Dauer von 10-20 Minuten bei Raumtemperatur um.
- Die Lösung in 50-ml-Röhrchen teilen und bei 4 °C und einer Geschwindigkeit von 20.700 x g für eine Dauer von 30 Minuten zentrifugieren.
- Sammeln Sie den Überstand und entsorgen Sie das Pellet.
- Bereiten Sie einen sterilen 250 ml Erlenmeyerkolben auf Eis mit einem Rührer vor.
- Den Überstand wird mit einem 0,22 μm Spritzenfilter in den sauberen Erlenmeyerkolben filtriert.
- Bestimmen Sie das Lösungsvolumen und wiegen Sie 0,3 g Ammoniumsulfat pro 1 ml Lösung.
- Nach und nach das Ammoniumsulfat in die gerührte Lösung geben.
HINWEIS: Dieser Schritt wird durchgeführt, um Proteine auszufällen. Wenn das Ammoniumsulfat die Proteine bindet, wird die Lösung verdeckt und verschwommen, was sich als weiße Farbe zeigt. - 30 Minuten bei Raumtemperatur umrühren.
- Die Lösung wird auf 50-ml-Röhrchen aufgeteilt und bei 4 °C und einer Geschwindigkeit von 20.700 g für eine Dauer von 30 Minuten zentrifugiert.
- Entsorgen Sie den Überstand vorsichtig und bewahren Sie das Pellet auf.
- Lösen Sie das Pellet mit 20 ml Puffer A auf.
- Vereinheitlichen Sie alle Lösungen in einem Röhrchen und messen Sie dann das UV-Absorptionsspektrum der Lösung.
HINWEIS: Wenn das Spektrum eine Aggregationssignatur (Absorption oberhalb einer Wellenlänge von 300 nm) aufweist, fahren Sie mit dem nächsten Schritt fort. Wenn nicht, überspringen Sie den nächsten Schritt und fahren Sie mit Schritt 2.1.34 fort.
ANMERKUNG: Der Aggregationsindex (KI), berechnet aus den UV-Spektren als ODλ=350nm/ (ODλ=280nm - ODλ=350nm)×100. Nr. 41 - Verwenden Sie 100 kDa Cutoff-Zentrifropröhrchen und zentrifugieren Sie mit einer Geschwindigkeit von 3.590 x g bei 4 °C für eine Dauer von 15 Minuten. Sammeln Sie die Flüssigkeit, die durch den Filter gelangt ist.
- Dialysieren Sie die Lösung bei 4 °C über Nacht gegen Puffer A mit Dialysebeuteln mit einem 3,5 kDa-Cutoff.
- Führen Sie eine weitere Dialyserunde für eine Dauer von mindestens 4 Stunden durch.
- Laden Sie die dialysierte Probe auf eine 1 ml MonoQ-Anionenaustauschsäule. Verwenden Sie 30 mM Tris-Hydrochlorid (Tris-HCl) Puffer bei pH 7,5 als Waschpuffer und 30 mM Tris-HCl Puffer pH 7,5 mit 500 mM NaCl als Elutionspuffer.
- Injizieren Sie die Probe in die Spalte. Anschließend mit 20 Säulenvolumina (CVs) von 0% Elutionspuffer und 100% Waschpuffer waschen, um die ungebundenen Proteine zu entfernen.
- Waschen Sie mit 7 Lebensläufen mit 30% Elutionspuffer.
- Eluieren Sie die α-Syn-Proteinprobe mit einem Gradienten, der sich von 30% bis 100% Elutionspuffer für 30 CVs bei einer Durchflussrate von 1,5 ml/min ändert.
HINWEIS: α-Syn eluiert bei 46-66 Lebensläufen bei einer Leitfähigkeit von 20-24 mS/cm, was sich auf 38-50% Elutionspuffer bezieht. - Sammeln Sie die Bruchteile des Hauptelutionspeaks. Überprüfen Sie das Vorhandensein von α-Syn, indem Sie Nach der Färbung mit Coomassie Blue oder Fast SeeBand Färbelösung Proben in 15%iger Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) durchführen. Eine Band bei 15 kDa wird erwartet.
- Vereinheitlichen Sie die relevanten Fraktionen und dialysieren Sie sie über Nacht gegen Puffer A mit Dialysebeuteln mit einem 3,5 kDa-Cutoff bei 4 °C.
- Aliquots der Proteinprobe zubereiten und bei -20 °C lagern.
- Cy3-gekennzeichnete α-Synuclein
- Reduzieren Sie das Thiol des Cysteinrests in der α-Syn A56C-Mutante durch Zugabe von DTT auf eine Endkonzentration von 2 mM für eine Dauer von 1 Stunde bei Raumtemperatur.
- Entfernen Sie das DVB-T, indem Sie zwei Dialyserunden mit 3,5 kDa-Cutoff-Dialysebeuteln durchführen. Für die erste Runde dialysieren Sie gegen 30 mM Tris-HCl pH 8,0 und 2 mM EDTA. Für die zweite Runde dialysieren Sie gegen 50 mM HEPES pH 7,2 und 2 mM EDTA.
- Die Cysteinreste werden durch Zugabe von Tris(2-carboxyethyl)phosphinhydrochlorid (TCEP) zur Proteinprobe auf eine Endkonzentration von 50 μM für eine Dauer von 30 Minuten bei Raumtemperatur reduziert.
HINWEIS: TCEP ist ein Nicht-Sulfhydryl-Reduktionsmittel, daher hält es die Thiolgruppen reduziert, ohne mit dem Farbstoff zu reagieren. Wir fügten 0,5 μL aus einer Stammlösung von 1 M TCEP hinzu. - Berechnen Sie die Proteinmengen, die für das endgültige Reaktionsvolumen von 1 ml erforderlich sind.
HINWEIS: Hier ist ein Beispiel für die Berechnung, die wir verwendet haben:
20 μM Endproteinkonzentration × 1 ml Endvolumen = 56 μM Ausgangsproteinkonzentration × zuzusetzenden V-Protein - Berechnen Sie die Menge an Farbstoff, die für das endgültige Reaktionsvolumen von 1 ml erforderlich ist. Die Farbstoffmarkierung eines einzelnen Cysteins sollte mit überschüssigem Farbstoff in einem Farbstoff-Protein-Molar-Verhältnis von mindestens 3:1 durchgeführt werden.
HINWEIS: Ein Beispiel für die von uns verwendete Berechnung:
60 μM Endfarbstoffkonzentration × 1 ml Endvolumen = 370 μM Ausgangsfarbstoffkonzentration × zuzusetzenden V-Farbstoff - Berechnen Sie die Puffermenge aus dem Dialysat, die erforderlich ist, um das Gesamtreaktionsvolumen auf 1 ml einzustellen.
ANMERKUNG: Berechnungsbeispiel: 1 ml - (V berechnet aus Schritt 2.2.4 + V berechnet aus Schritt 2.2.5) - Bereiten Sie die Reaktionsfläschchen mit einem Magneten auf einem Rührer vor.
- Fügen Sie zunächst die berechnete Menge des Proteins und den Puffer hinzu. Anschließend die berechnete Menge des Farbstoffs (Sulfo-Cy3-Maleimid) hinzufügen.
- Halten Sie die Reaktion bei Raumtemperatur im Dunkeln für eine Dauer von 3-5 Stunden.
- Beenden Sie die Reaktion durch Zugabe von 2 mM DVB-T und fahren Sie für eine Dauer von 1 Stunde fort.
- Führen Sie drei Dialyserunden gegen Puffer A durch und verwenden Sie Dialysebeutel mit einem 3,5 kDa-Cutoff, um überschüssigen freien Farbstoff aus der Lösung zu entfernen.
- Laden Sie die Probe auf eine Größenausschlusssäule, um das markierte α-Syn weiter vom freien Farbstoff zu trennen.
- Bestimmen Sie die Konzentration von reinem markiertem α-Syn durch Messung der Absorption des Farbstoffs (Absorptionskoeffizient für Sulfo-Cy3 beträgt 162.000 M-1cm-1bei λ = 548 nm).
- Bei Bedarf konzentrieren Sie die rein markierte α-Syn-Lösung mit einem Vakuumkonzentrator (z. B. SpeedVac). Führen Sie dann eine Runde Dialyse gegen Puffer A durch, verwenden Sie Dialysebeutel mit einem 3,5 kDa-Cutoff und bestimmen Erneut die Konzentration des markierten Proteins.
- Aliquots der markierten Proteinprobe zubereiten und bei -20 °C lagern.
3. Messungen
- smPIFE Versuchsaufbau
HINWEIS: Verwenden Sie das folgende konfokale Setup oder ähnliches.- Verwenden Sie eine gepulste Laserquelle (in unserem Fall λ = 532 nm Pikosekunden-gepulster Laser mit einer Pulsbreite von ~100 ps FWHM), die mit einer geeigneten Wiederholrate (in unserem Fall 20 MHz) arbeitet und zum SYNC einer zeitkorrelierten Einzelphotonenzählkarte (TCSPC) als Quelle der Sulfo-Cy3 (sCy3) -Anregung geleitet wird.
- Verwenden Sie einen dichroitischen Spiegel mit hohem Reflexionsvermögen bei 532 nm, um Anregung und Streuung von der Fluoreszenz zu trennen.
- Verwenden Sie ein Loch mit einem Durchmesser von 100 μm im Fokus des emittierten Lichts, nachdem der kollimierte Emissionsstrahl durch eine Linse fokussiert wurde und bevor der Emissionsstrahl durch eine andere Linse wieder kollimiert wurde.
- Verwenden Sie einen Bandpassfilter (in unserem Fall 585/40 nm), um die sCy3-Fluoreszenz aus anderen Lichtquellen weiter zu filtern.
- Detektieren Sie die Fluoreszenz mit einem Detektor (in unserem Fall Einzelphotonen-Lawinendiode oder Hybrid-Photomultiplier), der (in unserem Fall über einen 4-zu-1-Router) an ein TCSPC-Modul (Becker & Hickl SPC-150, in unserem Fall) geleitet wird.
HINWEIS: In unserem Fall erfolgt die Datenerfassung über die VistaVision-Software (ISSTM)im TTTR-Dateiformat (Time-Tagged-Time-Resolved).
- smPIFE Probenvorbereitung
- 25 pM sCy3-markierte α-Syn in Messungen Puffer: 10 mM Natriumacetat, 10 mM Natriumdihydrogenphosphat, 10 mM Glycin pH 8,0, 20 mM NaCl, 10 mM Cysteamin und 1 mM 6-Hydroxy-2,5,7,8-tetramethylchroman-2-carbonsäure (TROLOX) in einem proteinarmen Bindungsröhrchen herstellen.
HINWEIS: Wenn α-Syn in Gegenwart von SDS-Vesikeln gemessen wird, fügen Sie auch die entsprechende SDS-Menge hinzu. - Spülen Sie einen 18-Kammer-Mikroskopie-Deckglas-Objektträger mit 100 μL 1 mg/ml Rinderserumalbumin (BSA) für eine Dauer von 1 Minute aus und entfernen Sie dann das BSA.
- 100 μL der 25 pM sCy3-markierten α-Syn-Probe in eine Kammer im Deckglasobjektträger geben.
- Führen Sie die smPIFE-Messung der Probe mit dem beschriebenen Setup durch, wie in den nächsten Schritten wie folgt.
- 25 pM sCy3-markierte α-Syn in Messungen Puffer: 10 mM Natriumacetat, 10 mM Natriumdihydrogenphosphat, 10 mM Glycin pH 8,0, 20 mM NaCl, 10 mM Cysteamin und 1 mM 6-Hydroxy-2,5,7,8-tetramethylchroman-2-carbonsäure (TROLOX) in einem proteinarmen Bindungsröhrchen herstellen.
- smPIFE Datenerfassung
- Verwenden Sie ein Wasser-Tauchobjektiv mit hoher numerischer Apertur (in unserem Fall Olympus UPLSAPO 60x N.A. 1.2) und fügen Sie einen Tropfen ultrareines Wasser auf das Objektiv hinzu.
- Befestigen Sie den Deckglas-Objektträger in einer Bühnenkammer und installieren Sie ihn oben auf dem Mikroskoptisch.
- Bringen Sie die Objektivlinse nach oben, bis der Wassertropfen oben auf der Objektivlinse an der Unterseite des Deckglasschiebers verschmiert.
- Öffnen Sie den Laserverschluss und bringen Sie das Objektiv nach oben, während Sie das Muster auf einer CMOS-Kamera untersuchen und das von der Probe gestreute Licht sammeln. Beobachten Sie das Muster der Luftigen Ringe: Der erste stellt den Fokus an der Wasser-Glas-Grenzfläche dar, und der zweite stellt den Fokus an der Grenzfläche zwischen dem Glas und der Probenlösung dar.
- Erhöhen Sie die Höhe der Objektivlinse um zusätzliche 75 μm und bringen Sie den Laserfokus tief in die Lösung, um die Autofluoreszenz von der Glasoberfläche des Deckglases zu minimieren.
- Stellen Sie die Laserleistung an der Objektivlinse auf ~100 μW ein.
- Starten Sie die Erfassung der detektierten Photonen für eine vordefinierte Zeit (2 Stunden, in unseren Messungen).
HINWEIS: Der Großteil des Erfassungssignals sollte eine Rate (gemessen in Zählungen pro Sekunde, cps) aufweisen, die derjenigen ähnelt, die bei der Messung nur des Puffers erfasst wird. Die Millisekunden-gebundenen Daten sollten knappe Photonen-Burst-Ereignisse zeigen, wobei die Mehrheit der Bins eine durchschnittliche Rate aufweist, die mit der typischen Hintergrundrate des Detektors vergleichbar ist (in unserem Fall <1.000 cps).
4. smPIFE-Burst-Analyse
- Rohdatenkonvertierung
HINWEIS: Die Daten werden normalerweise in einer Binärdatei mit einem Format gespeichert, das von der Firma, die die TCSPC-Karte herstellt, vordefiniert wurde (in unserem Fall .spc-Dateien).- Konvertieren Sie die Rohdatendatei mit der Software-Suite phconvert (https://github.com/Photon-HDF5/phconvert) in das universelle Photon-HDF5-Dateiformat42. Rufen Sie die Rohdatendatei als Eingabe auf und konvertieren Sie sie in eine .hdf5-Rohdatendatei mit dem entsprechenden Code in der phconvert-Suite (in unserem Fall das Notizbuch ns-ALEX Becker-Hickl in Photon-HDF5.ipynb Jupyter konvertieren).
HINWEIS: Die Konvertierung in eine .hdf5-Datei umfasst: (1) Bestimmung der relevanten Photonenströme in den Rohdaten (d.h. Photonenströme von registrierten Photonen der relevanten Detektor-ID); (2) die relevanten Photonen-Nanozeiten (die Photonendetektionszeiten relativ zur SYNC-Zeit der Anregung); und (3) Metadaten hinzugefügt.
- Konvertieren Sie die Rohdatendatei mit der Software-Suite phconvert (https://github.com/Photon-HDF5/phconvert) in das universelle Photon-HDF5-Dateiformat42. Rufen Sie die Rohdatendatei als Eingabe auf und konvertieren Sie sie in eine .hdf5-Rohdatendatei mit dem entsprechenden Code in der phconvert-Suite (in unserem Fall das Notizbuch ns-ALEX Becker-Hickl in Photon-HDF5.ipynb Jupyter konvertieren).
- Burst-Suche und -Auswahl mit FRETbursts43
HINWEIS: Alle Photon-HDF5-Rohdatendateien der smPIFE-Messungen sowie der Code, der die Analyse der Rohdaten zusammenfasst, sind in Zenodo (https://doi.org/10.5281/zenodo.4587698 gespeichert. Alle folgenden Schritte sind detailliert beschrieben und werden in den Jupyter-Notebooks angezeigt, die auch im Zenodo-Repository-Link enthalten sind.- Öffnen Sie die Jupyter Notebooks (in unserem Fall innerhalb des Anaconda-Frameworks).
- Öffnen Sie das Notebook smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (Final notebook).ipynb (es finden Sie im Zenodo-Link).
- LADEN SIE FRETbursts.
- Laden Sie die Photon HDF5-Datendatei.
- BG-Ratenbewertung: Berechnen Sie anhand des Histogramms der Interphotonenzeiten die Hintergrundraten (BG) für jeweils 30 Sekunden der Datenerfassung in der Photon HDF5-Datendatei.
HINWEIS: Die folgenden Schritte beschreiben die Burst-Suche mit dem Schiebefensteralgorithmus44,45,46. - Verschieben Sie ein Zeitfenster von m = 20 aufeinanderfolgenden Photonen, ein Photon nach dem anderen.
- Sammeln Sie die Photonendaten nur, wenn die momentane Photonenrate (
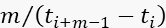 ) mindestens F= 11-mal größer ist als die BG-Rate für diesen Zeitraum der Datenerfassung.
) mindestens F= 11-mal größer ist als die BG-Rate für diesen Zeitraum der Datenerfassung.
HINWEIS: Ein Burst wird aus allen aufeinanderfolgenden Photonen konstruiert, die gesammelt wurden, indem das Fenster ein Photon nach dem anderen verschoben wurde (Schritt 4.2.6.) und mit dem Photonenratenkriterium (Schritt 4.2.7) übereinstimmt. - Berechnen Sie die folgenden Burst-Eigenschaften:
Burstgröße: Die Menge an Photonen in einem Burst.
Burst-Dauer: Die Zeitdifferenz zwischen der letzten und der ersten Photonen-Detektionszeit in einem Burst.
Burst-Helligkeit: der größte Wert der momentanen Photonenrate in einem Burst.
Burst-Trennung: das Zeitintervall zwischen aufeinanderfolgenden Bursts.
HINWEIS: Die folgenden Punkte beschreiben das weitere Burst-Auswahlverfahren. - Zeichnen Sie das Histogramm der Burst-Helligkeitswerte (die höchste momentane Photonenrate in einem Burst) mit der Ereignisachse in logarithmischer Skala.
- Definieren Sie den Bursthelligkeitsschwellenwert als den minimalen Bursthelligkeitswert, ab dem das Histogramm ein abklingendes Muster aufweist.
- Wählen Sie Bursts mit Helligkeitswerten aus, die größer als der Bursthelligkeitsschwellenwert sind.
HINWEIS: Die nächsten Schritte beschreiben die mittlere Fluoreszenzlebensdauer von Bursts. - Zeichnen Sie das Histogramm der Photonen-Nanozeiten für alle Photonen in allen ausgewählten Bursts mit der Photonenzählachse in logarithmischer Skala auf.
- Definieren Sie den Nanozeitschwellenwert als den minimalen Nanozeitwert, ab dem das Histogramm der Photonennanozeiten ein zerfallendes Muster aufweist.
- Wählen Sie nur Photonen aus, deren Nanozeit größer als der Nanozeitschwellenwert ist.
- Berechnen Sie den algebraischen Mittelwert aller ausgewählten Photonen-Nanozeiten.
- Subtrahieren Sie den Nanozeitschwellenwert vom algebraischen Mittelwert der Photonennanozeit. Das Ergebnis ist die mittlere Photonen-Nanozeit des Bursts, die direkt proportional zur mittleren Fluoreszenzlebensdauer ist.
- Zeichnen Sie das Histogramm aller geplatzten mittleren Fluoreszenzlebenszeiten auf. Zentral verteilte Subpopulationen der Fluoreszenzlebensdauer können auftreten. Subpopulationen mit niedrigen Wertdurchschnitten repräsentieren Molekülspezies mit sCy3, die nicht sterisch behindert wurden, während Subpopulationen mit höheren Wertdurchschnitten Molekülspezies mit sCy3 darstellen, die sterisch stärker behindert wurden.
HINWEIS: Die nächsten Schritte beschreiben die langsame Dynamik zwischen den Bursts basierend auf der Burst-Rekurrenzanalyse47 - Zeichnen Sie das Histogramm der Burst-Trennzeiten mit der Trennungszeitachse in logarithmischer Skala auf.
HINWEIS: Es werden zwei Teilpopulationen von Burst-Separationszeiten angezeigt:
Eine große Teilpopulation mit Sekundentreuzeiten, die aufeinanderfolgende Bursts darstellt, die von verschiedenen nacheinander gemessenen Molekülen stammen.
Eine kleine Teilpopulation mit Trennungszeiten ~ <100 ms, die aufeinanderfolgende Ausbrüche darstellt, die beide aus demselben Molekül stammen und durch das konfokale Volumen wiederkehren. - Wählen Sie diese Option aus, um alle Paare aufeinanderfolgender Bursts zu speichern, die durch weniger als eine maximale Trennzeit getrennt sind, die die Subpopulation mit demselben Molekül definiert (in unserem Fall <100 ms).
- Zeichnen Sie ein Histogramm oder ein Streudiagramm der mittleren Fluoreszenzlebensdauer des ersten und zweiten Bursts für alle Paare von Bursts, die unterhalb einer bestimmten Trennzeitschwelle aufgetreten sind
Ergebnisse
Als IDP, wenn es nicht an ein anderes Biomolekül gebunden ist, zeigt α-Syn strukturelle Dynamik zwischen mehreren Konformationen, mit Übergängen bei wenigen Mikrosekunden40 und sogar bei Hunderten von Nanosekunden41. Wenn α-Syn den konfokalen Punkt kreuzt, kann er Tausende von Übergängen zwischen konformationen durchlaufen. In der Tat war dies der Fall, als smFRET verwendet wurde39,40. Hier führen wir smPIFE...
Diskussion
Umfangreiche biochemische und biophysikalische Studien wurden durchgeführt, um die strukturellen Eigenschaften von α-Syn und seine ungeordnete Natur zu untersuchen33,34,35,36,37,38. Mehrere Arbeiten haben bereits frei diffundierendes smFRET verwendet, um die intramolekulare Dynamik des bindungsfreien α-Syn-Monomers zu unte...
Offenlegungen
Alle Autoren teilen keinen Interessenkonflikt.
Danksagungen
Das pT-t7-Plasmid, das für A56C α-Syn-Mutant kodiert, wurde uns von Dr. Asaf Grupi, Dr. Dan Amir und Dr. Elisha Haas geschenkt. Dieses Papier wurde von den National Institutes of Health (NIH, Grant R01 GM130942 an E.L. als Unterpreis), der Israel Science Foundation (Grant 3565/20 im Rahmen des KillCorona - Curbing Coronavirus Research Program), dem Milner Fund und der Hebrew University of Jerusalem (Startfonds) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
Referenzen
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten