È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Utilizzo del miglioramento della fluorescenza indotta da proteine risolte nel tempo per identificare le conformazioni locali stabili un monomero α-sinucleina alla volta
In questo articolo
Riepilogo
Il potenziamento della fluorescenza indotto da proteine a singola molecola risolto nel tempo è un utile sensore di prossimità spettroscopico a fluorescenza sensibile ai cambiamenti strutturali locali nelle proteine. Qui mostriamo che può essere usato per scoprire conformazioni locali stabili in α-sinucleina, che è altrimenti nota come globulare non strutturata e instabile quando misurata usando il righello FRET a lungo raggio.
Abstract
L'uso di righelli spettroscopici per tracciare più conformazioni di singole biomolecole e le loro dinamiche hanno rivoluzionato la comprensione delle dinamiche strutturali e dei suoi contributi alla biologia. Mentre il righello basato su FRET riporta le distanze inter-coloranti nell'intervallo 3-10 nm, altre tecniche spettroscopiche, come il potenziamento della fluorescenza indotto da proteine (PIFE), riportano la vicinanza tra un colorante e una superficie proteica nell'intervallo più breve di 0-3 nm. Indipendentemente dal metodo di scelta, il suo uso nella misurazione di biomolecole a diffusione libera una alla volta recupera gli istogrammi del parametro sperimentale producendo sotto-popolazioni separate distribuite centralmente di biomolecole, dove ogni sotto-popolazione rappresenta una singola conformazione che è rimasta invariata entro millisecondi, o più conformazioni che si interconvertono molto più velocemente dei millisecondi, e quindi una sotto-popolazione mediata. Nel FRET a singola molecola, dove il parametro riportato negli istogrammi è l'efficienza FRET inter-colorante, una proteina intrinsecamente disordinata, come il monomero α-sinucleina in tampone, è stata precedentemente segnalata come esibiva una singola sotto-popolazione mediata di conformazioni multiple che si interconvertono rapidamente. Mentre questi risultati passati dipendono dall'intervallo di 3-10 nm del righello basato su FRET, abbiamo cercato di mettere alla prova questa proteina usando PIFE a singola molecola, dove tracciamo la durata di fluorescenza delle proteine α-sinucleina labelte sCy3 sito-specifiche una alla volta. È interessante notare che, utilizzando questo sensore di prossimità spettroscopico a corto raggio, la α-sinucleina etichettata sCy3 presenta diverse sotto-popolazioni di vita con una vita media nettamente diversa che si interconverte in 10-100 ms. Questi risultati mostrano che mentre α-sinucleina potrebbe essere disordinata a livello globale, raggiunge comunque strutture locali stabili. In sintesi, in questo lavoro evidenziamo il vantaggio di utilizzare diversi sensori di prossimità spettroscopici che tracciano i cambiamenti strutturali locali o globali una biomolecola alla volta.
Introduzione
Negli ultimi due decenni, i metodi basati sulla fluorescenza a singola molecola sono diventati un potente strumento per misurare le biomolecole1,2,sondando come si distribuiscono i diversi parametri biomolecolari e come si interconvertono dinamicamente tra diverse sotto-popolazioni di questi parametri a risoluzione inferiore al millisecondo3,4,5. I parametri in queste tecniche includono l'efficienza di trasferimento di energia nelle misure FRET 6,7,l'anisotropia di fluorescenza8,9,i rendimenti quantistici di fluorescenza e le durate10,11,in funzione di diversi meccanismi di temprafluorescenza 12 o di potenziamento13. Uno di questi meccanismi, meglio noto come protein-induced fluorescence enhancement (PIFE)14 introduce il potenziamento della resa quantistica e della durata della fluorescenza in funzione dell'ostruzione sterica all'isomerizzazione libera del fluoroforo quando in stato eccitato, causata da superfici proteiche in prossimità del colorante14,15,16,17,18,19 . Sia FRET che PIFE sono considerati righelli spettroscopici o sensori di prossimità poiché il loro parametro misurato è direttamente collegato a una misura spaziale all'interno della biomolecola etichettata in fase di misurazione. Mentre l'efficienza FRET è correlata alla distanza tra una coppia di coloranti entro un intervallo di 3-10 nm20, PIFE traccia aumenti delle rese quantistiche di fluorescenza o della durata di vita legate alla distanza tra il colorante e una superficie di una proteina vicina nell'intervallo di 0-3 nm19.
La FRET a singola molecola è stata ampiamente utilizzata per fornire informazioni strutturali su molti sistemi proteici diversi, tra cui proteine intrinsecamente disordinate (IDP)21, come α-Sinucleina (α-Syn)22. α-Syn può formare strutture ordinate in seguito al legame a diverse biomolecole e in diverse condizioni23,24,25,26,27,28,29,30. Tuttavia, quando non legato, il monomero α-Syn è caratterizzato da un'elevata eterogeneità conformazionale con conformazioni rapidamente interconvertite31,32.
Le conformazioni di α-Syn sono state studiate in precedenza utilizzando varie tecniche diverse che aiutano a identificare la dinamica conformazionale di tali sistemi proteici altamente eterogenei e dinamici33,34,35,36,37,38,39. È interessante notare che le misurazioni FRET a singola molecola (smFRET) di α-Syn nel buffer hanno riportato una singola popolazione FRET39,40 che è il risultato della media temporale delle conformazioni che si interconvertono dinamicamente a volte molto più velocemente del tempo di diffusione tipico di α-Syn attraverso il punto confocale (tempi veloci come pochi microsecondi e anche più veloci di quello, rispetto ai tipici tempi di diffusione al millisecondo)40, 41. Tuttavia, l'uso di un righello spettroscopico FRET con la sensibilità alla distanza di 3-10 nm a volte riporta solo i cambiamenti strutturali complessivi in una piccola proteina come α-Syn. Le misurazioni a singola molecola che utilizzano sensori di prossimità spettroscopici con sensibilità a distanza più breve hanno il potenziale per riferire sulla dinamica delle strutture locali. Qui eseguiamo misurazioni PIFE a singola molecola di α-Syn e identifichiamo diverse sotto-popolazioni di vite di fluorescenza mappate a diverse strutture locali con transizioni tra loro lente fino a 100 ms. Questo lavoro riassume le misurazioni smPIFE risolte nel tempo di molecole α-Syn a diffusione libera una alla volta, in buffer e quando legate a membrane basate su SDS come sensore di prossimità spettroscopico a singola molecola a corto raggio.
Protocollo
1. Trasformazione del plasmide
- Preparazione di 0,5 L di mezzo SOC
- Pesare 10 g di triptone, 2,5 g di estratto di lievito, 0,25 g di cloruro di sodio (NaCl), 0,1 g di cloruro di potassio (KCl).
- Aggiungere acqua a doppia distillazione (DDW) fino a un volume totale di 0,5 L.
- Regolare a pH 7 aggiungendo idrossido di sodio (NaOH).
- Preparare aliquote di riserva di 100 ml e autoclave.
- Prima dell'uso, aggiungere 0,5 mL di cloruro di magnesio sterile (MgCl2) e 1,8 mL di glucosio sterile a 100 mL di SOC.
- Scongelare BL21 (DE3) cellule competenti di Escherichia coli su ghiaccio.
- Mescolare delicatamente le cellule competenti.
- Preparare due tubi da 15 ml. Etichettare uno come tubo di reazione di trasformazione e l'altro come tubo di controllo.
- Aliquota 100 μL delle cellule competenti nei tubi.
- Aggiungere 1 μL di plasmide ad una concentrazione di 0,1 ng/μL nel tubo di reazione di trasformazione.
- Riscaldare il mezzo SOC a bagnomaria a 42 °C per l'uso nel passaggio 1.8.
- Impulso termico dei due tubi a bagnomaria a 42 °C per una durata di 45 secondi. (Passaggio critico!)
- Aggiungere 900 μL di mezzo SOC riscaldato a ciascun tubo.
- Incubare i tubi a 37 °C per una durata di 45 minuti con agitazione ad una velocità di 225 giri/min.
- Distribuire 200 μL delle cellule con il plasmide trasformato su una piastra LB (Luria-Bertani)-agar contenente 100 μg/mL di ampicillina, utilizzando uno spandicellulare sterile.
- Distribuire 200 μL delle cellule con il plasmide trasformato su una piastra LB-agar senza ampicillina, utilizzando uno spargitore sterile (controllo positivo).
- Distribuire 200 μL delle cellule di controllo (non contenenti un plasmide) su una piastra LB-agar contenente 100 μg/mL di ampicillina, utilizzando uno spargitore sterile(controllo negativo).
- Incubare le piastre durante la notte a 37 °C.
2. Preparazione proteica
- Espressione e purificazione ricombinante α-sinucleina
- Scegliere una singola colonia e coltivare in 100 ml di terreno liquido autoclavato LB (Luria-Bertani) contenente 100 μg/mL di ampicillina a 37 °C (starter).
- Preparare quattro palloni Erlenmeyer da 2 L, ciascuno contenente 1 L di LB medio autoclavato e 100 μg/mL di ampicillina (inoculazione di grandi dimensioni).
- Misurare la densità ottica (OD a λ = 600 nm) della soluzione cellulare. Quando la densità cellulare dello starter raggiunge un ODλ=600nm di 0,6-0,7, aggiungere 10 mL di soluzione di avviamento per ogni 1 L di terreno LB preparato nella fase precedente.
- Coltivare i mezzi batterici in uno shaker incubatore a 37 °C e una velocità di 200 giri/min.
- Quando la densità cellulare raggiunge un ODλ =600nm di 0,6-0,8, indurre l'espressione proteica aggiungendo isopropil β-d-1-tiogalattopoliranoside (IPTG) per ogni 1 L di terreno di crescita ad una concentrazione finale di 1 mM.
NOTA: Sciogliere 0,96 g di IPTG in 4 mL di DDW e aggiungere 1 mL della soluzione IPTG risultante per ogni 1 L di crescita batterica. - Far crescere le cellule per una durata di 4 ore e raccoglierle per centrifugazione in tubi da 50 ml ad una velocità di 5.170 x g per una durata di 8 minuti.
NOTA: In ogni round, scartare il surnatante, tenere il pellet e riempire nuovamente gli stessi tubi con la crescita batterica. - Conservare il pellet batterico in un congelatore (ad esempio, -80 °C) per il giorno successivo.
- Il giorno successivo, preparare 200 mL di tampone di lisi: 40% (p / v) saccarosio e tampone A, che include 30 mM Tris-HCl, 2 mM di acido etilendiamminotetraacetico (EDTA), 2 mM ditiotreitolo (DTT) a pH 8,0.
- Aggiungere 25 ml di tampone di lisi a ciascun tubo con pellet batterico.
- Sospendere di sospendere il pellet (utilizzare pipette di plastica sterile Pasteur) nel tampone di Lisi (vedere 2.1.8).
- Preparare il matraccio Erlenmeyer sterile da 250 ml con un magnete agitatore su ghiaccio.
- Aggiungere 5 mL di tampone di lisi, quindi trasferire le celle omogenee ri-sospese ad esso.
- Mescolare le celle per una durata di 20 minuti a 200 giri / min a temperatura ambiente.
- Dividere la soluzione in tubi da 50 mL e centrifugare a 4 °C per una durata di 30 minuti ad una velocità di 17.400 x g.
- Scartare il surnatante.
- Preparare il matraccio Erlenmeyer sterile da 250 ml con magnete agitatore su ghiaccio.
- Aggiungere 15 mL di tampone di dissoluzione (90 mL di tampone raffreddato A e 37 μL di MgCl2 disciolto oltre il limite di solubilità e filtrato) in ciascun tubo. Utilizzare pipette Pasteur di plastica sterili e sciogliere il pellet.
- Quando una parte della soluzione diventa omogenea, spostarla in un matraccio Erlenmeyer mescolato su ghiaccio.
- Determinare il volume della soluzione in Erlenmeyer.
- Aggiungere 10 mg di streptomicina solfato per ogni 1 mL di soluzione (prima di aggiungere, sciogliere la streptomicina solfato in 2 mL di tampone A).
NOTA: Questo passaggio viene eseguito al fine di rimuovere il DNA e i ribosomi. - Mescolare la soluzione per una durata di 10-20 minuti a temperatura ambiente.
- Dividere la soluzione in tubi da 50 mL e centrifugare a 4 °C e ad una velocità di 20.700 x g per una durata di 30 minuti.
- Raccogliere il surnatante ed scartare il pellet.
- Preparare un matraccio Erlenmeyer sterile da 250 ml su ghiaccio con un agitatore.
- Filtrare il surnatante utilizzando un filtro a siringa da 0,22 μm nel matraccio Erlenmeyer pulito.
- Determinare il volume della soluzione e pesare 0,3 g di solfato di ammonio per ogni 1 mL di soluzione.
- A poco a poco, aggiungere il solfato di ammonio alla soluzione mescolata.
NOTA: Questo passaggio viene eseguito per precipitare le proteine. Quando il solfato di ammonio lega le proteine, la soluzione diventa oscurata e sfocata, che si presenta come colore bianco. - Mescolare per 30 minuti a temperatura ambiente.
- Dividere la soluzione in tubi da 50 mL e centrifugare a 4 °C e ad una velocità di 20.700 g per una durata di 30 minuti.
- Scartare con attenzione il surnatante e conservare il pellet.
- Sciogliere il pellet con 20 ml di Tampone A.
- Unificare tutte le soluzioni in un unico tubo, quindi misurare lo spettro di assorbimento UV della soluzione.
NOTA: se lo spettro mostra una firma di aggregazione (assorbimento superiore a una lunghezza d'onda di 300 nm), continuare con il passaggio successivo. In caso contrario, saltare il passaggio successivo e passare al passaggio 2.1.34.
NOTA: L'indice di aggregazione (A.I.) calcolato dagli spettri UV come ODλ=350nm/ (ODλ=280nm - ODλ=350nm) ×100. 41 anni - Utilizzare tubi centriprep cutoff da 100 kDa e centrifugare ad una velocità di 3.590 x g a 4 °C per una durata di 15 minuti. Raccogliere il liquido che è passato attraverso il filtro.
- Dializzare la soluzione a 4 °C durante la notte, contro il tampone A, utilizzando sacche per dialisi con un cutoff di 3,5 kDa.
- Eseguire un altro ciclo di dialisi per una durata di almeno 4 ore.
- Caricare il campione dializzato su una colonna di scambio anionico MonoQ da 1 mL. Utilizzare un tampone Tris cloridrato (Tris-HCl) da 30 mM a pH 7,5 come tampone di lavaggio e un tampone Tris-HCl da 30 mM pH 7,5 con NaCl da 500 mM come tampone di eluizione.
- Inserire l'esempio nella colonna. Quindi lavare con 20 volumi di colonna (CV) di tampone di eluizione allo 0% e tampone di lavaggio al 100% per rimuovere le proteine non legate.
- Lavare con 7 CV di tampone di eluizione al 30%.
- Eluire il campione di proteina α-Syn utilizzando un gradiente che passa dal 30% al 100% tampone di eluizione per 30 CV, ad una portata di 1,5 ml/min.
NOTA: α-Syn eluisce a 46-66 CV con conducibilità 20-24 mS/cm, che si riferisce al tampone di eluizione al 38-50%. - Raccogli le frazioni del picco di eluizione principale. Verificare la presenza di α-Syn eseguendo campioni in elettroforesi su gel di poliacrilammide dodecilsalfato di sodio al 15% (SDS-PAGE), dopo la colorazione con soluzione di colorazione Coomassie Blue o Fast SeeBand. È prevista una banda a 15 kDa.
- Unificare le frazioni rilevanti e dializzarle durante la notte, contro il tampone A, utilizzando sacche di dialisi con un cutoff di 3,5 kDa a 4 °C.
- Preparare aliquote del campione proteico e conservarle a -20 °C.
- Sinucleina α marcata Cy3
- Ridurre il tiolo del residuo di cisteina nel mutante α-Syn A56C aggiungendo DTT ad una concentrazione finale di 2 mM per una durata di 1 ora a temperatura ambiente.
- Rimuovere la DTT eseguendo due cicli di dialisi utilizzando sacche per dialisi da 3,5 kDa. Per il primo round, dializzare contro 30 mM Tris-HCl pH 8,0 e 2 mM EDTA. Per il secondo round, dializzare contro 50 mM HEPES pH 7,2 e 2 mM EDTA.
- Ridurre i residui di cisteina aggiungendo Tris(2-carbossietil)fosfina cloridrato (TCEP) al campione proteico, ad una concentrazione finale di 50 μM per una durata di 30 minuti a temperatura ambiente.
NOTA: TCEP è un agente riducente non sulfidrilico, quindi mantiene i gruppi tiolici ridotti senza reagire con il colorante. Abbiamo aggiunto 0,5 μL da una soluzione stock di 1 M TCEP. - Calcolare le quantità di proteine necessarie per il volume di reazione finale di 1 mL.
NOTA: Ecco un esempio per il calcolo che abbiamo usato:
20 μM di concentrazione proteica finale × 1 mL di volume finale = 56 μM di concentrazione proteica iniziale × proteina V da aggiungere - Calcolare la quantità di colorante necessaria per il volume di reazione finale di 1 mL. L'etichettatura del colorante di una singola cisteina deve essere eseguita con colorante in eccesso, con un rapporto molare colorante/proteina di almeno 3:1.
NOTA: un esempio per il calcolo che abbiamo usato:
Concentrazione finale del colorante di 60 μM × 1 mL di volume finale = 370 μM di concentrazione iniziale del colorante × V da aggiungere - Calcolare la quantità di tampone dal dializzato necessario per regolare il volume totale della reazione a 1 mL.
NOTA: Esempio di calcolo: 1 mL - (V calcolato dal passo 2.2.4 + V calcolato dal passaggio 2.2.5) - Preparare il flaconcino di reazione con un magnete sopra un agitatore.
- In primo luogo, aggiungere la quantità calcolata della proteina e il buffer. Successivamente, aggiungere la quantità calcolata del colorante (sulfo-Cy3 maleimmide).
- Mantenere la reazione a temperatura ambiente al buio per una durata di 3-5 ore.
- Terminare la reazione aggiungendo 2 mM DTT e continuare per una durata di 1 ora.
- Eseguire tre cicli di dialisi contro il tampone A, utilizzando sacche per dialisi con un cutoff di 3,5 kDa per rimuovere il colorante libero in eccesso dalla soluzione.
- Caricare il campione su una colonna di esclusione delle dimensioni per separare ulteriormente il α-Syn etichettato dal colorante libero.
- Determinare la concentrazione di α-Syn puro etichettato misurando l'assorbimento del colorante (il coefficiente di assorbimento per sulfo-Cy3 è 162.000 M-1cm-1a λ=548 nm).
- Se necessario, concentrare la soluzione pura etichettata α-Syn utilizzando un concentratore a vuoto (ad esempio, SpeedVac). Quindi, eseguire un ciclo di dialisi contro il tampone A, utilizzando sacche di dialisi con un cutoff di 3,5 kDa e determinare nuovamente la concentrazione della proteina etichettata.
- Preparare aliquote del campione proteico etichettato e conservarle a -20 °C.
3. Misurazioni
- Configurazione sperimentale smPIFE
NOTA: utilizzare la seguente configurazione basata su confocale o simile.- Utilizzare una sorgente laser pulsata (nel nostro caso, λ = laser pulsato a picosecondi da 532 nm con larghezza di impulso di ~ 100 ps FWHM), operante a una velocità di ripetizione adeguata (20 MHz nel nostro caso) e instradata al SYNC di una scheda di conteggio a singolo fotone correlato al tempo (TCSPC), come fonte di eccitazione sulfo-Cy3 (sCy3).
- Utilizzare uno specchio dicroico con elevata riflettività a 532 nm, per separare l'eccitazione e la dispersione dalla fluorescenza.
- Utilizzare un foro stenopeico di 100 μm di diametro al fuoco della luce emessa, dopo che il fascio di emissione collimato è stato focalizzato da una lente e prima che il raggio di emissione sia stato ri-collimato da un'altra lente.
- Utilizzare un filtro passa banda (585/40 nm nel nostro caso) per filtrare ulteriormente la fluorescenza sCy3 da altre sorgenti luminose.
- Rileva la fluorescenza utilizzando un rilevatore (diodo a valanga a singolo fotone o fotomoltiplicatore ibrido, nel nostro caso) instradato (attraverso un router 4 a 1, nel nostro caso) a un modulo TCSPC (Becker & Hickl SPC-150, nel nostro caso).
NOTA: Nel nostro caso, l'acquisizione dei dati viene eseguita tramite il software VistaVision (ISSTM)nel formato di file time-tagged-time-resolved (TTTR).
- Preparazione del campione smPIFE
- Preparare 25 pM di α-Syn con etichetta sCy3 nel tampone di misurazione: 10 mM di acetato di sodio, 10 mM di sodio diidrogeno fosfato, 10 mM di glicina pH 8,0, 20 mM NaCl, 10 mM di cisteamina e 1 mM di acido 6-idrossi-2,5,7,8-tetrametilcromo-2-carbossilico (TROLOX), in un tubo a basso legame proteico.
NOTA: se α-Syn viene misurato in presenza di vescicole SDS, aggiungere anche la quantità SDS appropriata. - Risciacquare un vetrino per microscopia a 18 camere con 100 μL di albumina sierica bovina da 1 mg/mL (BSA) per una durata di 1 minuto, quindi rimuovere la BSA.
- Aggiungere 100 μL del campione α-Syn da 25 pM con etichetta sCy3 a una camera nella diapositiva del coperchio.
- Eseguire la misurazione smPIFE del campione utilizzando l'impostazione descritta, come riportato di seguito nei passaggi successivi.
- Preparare 25 pM di α-Syn con etichetta sCy3 nel tampone di misurazione: 10 mM di acetato di sodio, 10 mM di sodio diidrogeno fosfato, 10 mM di glicina pH 8,0, 20 mM NaCl, 10 mM di cisteamina e 1 mM di acido 6-idrossi-2,5,7,8-tetrametilcromo-2-carbossilico (TROLOX), in un tubo a basso legame proteico.
- acquisizione dati smPIFE
- Utilizzare un obiettivo ad immersione in acqua ad alta apertura numerica (nel nostro caso, Olympus UPLSAPO 60x N.A. 1.2) e aggiungere una goccia di acqua ultrapura sopra l'obiettivo.
- Fissare il vetrino di copertura in una camera di palcoscenico e installarlo sopra lo stadio del microscopio.
- Portare la lente dell'obiettivo verso l'alto, fino a quando la goccia d'acqua sulla parte superiore della lente dell'obiettivo si spalma nella parte inferiore della diapositiva del coperchio.
- Aprire l'otturatore laser e portare l'obiettivo verso l'alto, mentre si ispeziona il modello su una fotocamera CMOS, raccogliendo la luce diffusa dal campione. Osserva il modello degli anelli Airy: il primo rappresenta il focus sull'interfaccia acqua-vetro, e poi il secondo rappresenta il focus sull'interfaccia tra il vetro e la soluzione campione.
- Aumentare l'altezza della lente dell'obiettivo di ulteriori 75 μm, portando la messa a fuoco laser in profondità nella soluzione, per ridurre al minimo l'autofluorescenza dalla superficie di vetro del coperchio.
- Sintonizzare la potenza del laser sulla lente dell'obiettivo in modo che sia ~ 100 μW.
- Iniziare l'acquisizione dei fotoni rilevati per un tempo predefinito (2 ore, nelle nostre misurazioni).
NOTA: la maggior parte del segnale di acquisizione dovrebbe avere una velocità (misurata in conteggi al secondo, cps) simile a quella acquisita quando si misura solo il buffer; i dati binned al millisecondo dovrebbero mostrare scarsi eventi di burst di fotoni, con la maggior parte dei contenitori che ha una velocità media paragonabile alla tipica frequenza di sfondo del rivelatore (< 1.000 cps, nel nostro caso).
4. analisi del burst smPIFE
- Conversione di dati grezzi
NOTA: I dati vengono solitamente memorizzati in un file binario con un formato predefinito dalla società che produce la scheda TCSPC (file .spc nel nostro caso).- Convertire il file di dati grezzi nel formato di file universale fotone-HDF542, utilizzando la suite di software phconvert (https://github.com/Photon-HDF5/phconvert). Chiamare il file di dati grezzi come input e convertirlo in un file .hdf5 di dati grezzi utilizzando il codice appropriato nella suite phconvert (il Converti i file ns-ALEX Becker-Hickl in Photon-HDF5.ipynb Jupyter notebook, nel nostro caso).
NOTA: la conversione in un file .hdf5 include: (1) la determinazione dei flussi di fotoni rilevanti nei dati grezzi (ad es. flussi di fotoni di fotoni registrati dell'ID del rivelatore pertinente); (2) i nanotempi di fotone pertinenti (i tempi di rilevamento dei fotone relativi al tempo SYNC di eccitazione); e (3) aggiunto di metadati.
- Convertire il file di dati grezzi nel formato di file universale fotone-HDF542, utilizzando la suite di software phconvert (https://github.com/Photon-HDF5/phconvert). Chiamare il file di dati grezzi come input e convertirlo in un file .hdf5 di dati grezzi utilizzando il codice appropriato nella suite phconvert (il Converti i file ns-ALEX Becker-Hickl in Photon-HDF5.ipynb Jupyter notebook, nel nostro caso).
- Burst Search and Selection utilizzando FRETbursts43
NOTA: Tutti i file di dati grezzi fotoni-HDF5 delle misurazioni smPIFE, così come il codice che riassume l'analisi dei dati grezzi, sono memorizzati in Zenodo (https://doi.org/10.5281/zenodo.4587698). Tutti i passaggi seguenti sono dettagliati e mostrati all'interno dei notebook Jupyter, forniti anche nel collegamento al repository Zenodo.- Apri i Quaderni Jupyter (all'interno del framework Anaconda, nel nostro caso).
- Aprire il notebook smPIFE-aSyn 56C(Cy3) 25 pM newBuffer (Final notebook).ipynb (si trova nel link Zenodo).
- Carica FRETbursts.
- Caricare il file di dati del fotone HDF5.
- Valutazione della velocità BG: utilizzando l'istogramma dei tempi inter-fotoni, calcolare le velocità di sfondo (BG) per ogni 30 secondi di acquisizione dati nel file di dati HDF5 del fotone.
NOTA: i seguenti passaggi descrivono la ricerca burst utilizzando l'algoritmo della finestra scorrevole44,45,46. - Spostare una finestra temporale di m=20 fotoni consecutivi, un fotone alla volta.
- Raccogliere i dati fotonici solo se la velocità istantanea del fotone, (
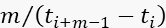 ), è almeno F=11 volte maggiore della velocità BG per quel periodo di acquisizione dei dati.
), è almeno F=11 volte maggiore della velocità BG per quel periodo di acquisizione dei dati.
NOTA: un burst è costruito da tutti i fotoni consecutivi che sono stati raccolti facendo scorrere la finestra un fotone alla volta (passo 4.2.6.) e concordando con il criterio della velocità del fotone (passo 4.2.7). - Calcola le seguenti caratteristiche di scoppio:
Dimensione del burst: la quantità di fotoni in una raffica.
Durata dello scoppio: la differenza di tempo tra l'ultimo e il primo tempo di rilevamento del fotone in una raffica.
Luminosità di raffica: il valore più grande della velocità istantanea di fotone in una raffica.
Burst separation: l'intervallo di tempo tra raffiche consecutive.
NOTA: i seguenti punti descrivono l'ulteriore procedura di selezione burst. - Traccia l'istogramma dei valori di luminosità di burst (la più alta velocità istantanea di fotone in una raffica), con l'asse degli eventi in scala logaritmica.
- Definite la soglia di luminosità di burst come il valore minimo di luminosità di burst da cui l'istogramma presenta un pattern di decadimento.
- Selezionare burst con valori di luminosità superiori alla soglia di luminosità burst.
NOTA: i passaggi successivi descrivono la durata media della fluorescenza di burst. - Traccia l'istogramma dei nanotempi di fotoni per tutti i fotoni in tutte le esplosioni selezionate con l'asse del conteggio dei fotoni in scala logaritmica.
- Definire la soglia nanotime come il valore nanotime minimo da cui l'istogramma dei nanotime fotoni mostra un pattern di decadimento.
- Selezionare solo fotoni con nanotime più grandi della soglia nanotime.
- Calcola la media algebrica di tutti i nanotempi di fotoni selezionati.
- Sottrarre la soglia nanotime dalla media algebrica nanotime del fotone. Il risultato è il nanotempo medio dei fotone del burst, che è direttamente proporzionale alla durata media della fluorescenza.
- Traccia l'istogramma di tutte le vite di fluorescenza media di burst. Possono comparire sotto popolazioni distribuite centralmente della vita di fluorescenza. Le sotto-popolazioni con medie di valore basse rappresentano specie di molecole con sCy3 che non è stato ostruito stericamente, mentre le sotto-popolazioni con medie di valore più elevate rappresentano specie di molecole con sCy3 che è stato ostruito più stericamente.
NOTA: i passaggi successivi descrivono la dinamica lenta tra burst basata sull'analisi della recidiva di burst47 - Traccia l'istogramma dei tempi di separazione burst, con l'asse del tempo di separazione in scala logaritmica.
NOTA: appariranno due sotto-popolazioni di tempi di separazione burst:
Una sotto-popolazione importante con tempi di separazione di secondi, che rappresenta raffiche consecutive originate da diverse molecole misurate consecutivamente.
Una sotto-popolazione minore con tempi di separazione ~ < 100 ms, che rappresenta esplosioni consecutive entrambe originate dalla stessa molecola, ricorrenti indietro attraverso il volume confocale. - Selezionare per salvare tutte le coppie di raffiche consecutive separate da meno di un tempo di separazione massimo che definisce la sotto-popolazione della stessa molecola (<100 ms, nel nostro caso).
- Tracciare un istogramma o un grafico a dispersione della durata media della fluorescenza della prima e della seconda raffica per tutte le coppie di esplosioni che si sono ripresentate al di sotto di una certa soglia temporale di separazione
Risultati
Come IDP, quando non è legato a un'altra biomolecola, α-Syn mostra dinamiche strutturali tra più conformazioni, con transizioni a pochi microsecondi40 e persino a centinaia di nanosecondi41. Quando α-Syn attraversa il punto confocale, può subire migliaia di transizioni tra le conformazioni. In effetti, questo è stato il caso quando smFRET è stato utilizzato39,40. Qui eseguiamo misure smPIFE al fine di sondare...
Discussione
Sono stati condotti ampi studi biochimici e biofisici per studiare le caratteristiche strutturali di α-Syn e la sua natura disordinata33,34,35,36,37,38. Diversi lavori hanno già utilizzato smFRET a diffusione libera per studiare la dinamica intra-molecolare del monomero α-Syn privo di legame. Questi lavori hanno riportato ...
Divulgazioni
Tutti gli autori non condividono alcun conflitto di interessi.
Riconoscimenti
Il plasmide pT-t7 che codifica per il mutante A56C α-Syn ci è stato regalato dal Dr. Asaf Grupi, dal Dr. Dan Amir e dal Dr. Elisha Haas. Questo documento è stato sostenuto dal National Institutes of Health (NIH, grant R01 GM130942 to E.L. come subaward), dalla Israel Science Foundation (grant 3565/20 nell'ambito del KillCorona - Curbing Coronavirus Research Program), dal Milner Fund e dall'Università Ebraica di Gerusalemme (fondi di avvio).
Materiali
| Name | Company | Catalog Number | Comments |
| Amicon Ultra-15 Centrifugal Filter Units | Merc | C7715 | cutoff: 100 kDa |
| ammonium sulfate | Sigma-Aldrich | A4418 | |
| BSA | Sigma-Aldrich | A9647 | |
| cysteamine | Sigma-Aldrich | 30070 | |
| dialysis bags - MEGA GeBaFlex-tube | Gene Bio-Application | MEGA320 | |
| dithiothreitol (DTT) | Sigma-Aldrich | 43815 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| Fast SeeBand staining solution | Gene Bio-Application | SB050 | |
| Glycine | Sigma-Aldrich | 50046 | |
| D-Glucose | Sigma-Aldrich | G7021 | |
| HEPES | Sigma-Aldrich | 54457 | |
| HiTrap Desalting 5 mL | Sigma-Aldrich | GE17-1408 | |
| 6-hydroxy-2,5,7,8-tetramethylchroman-2-carboxylic acid (TROLOX) | Sigma-Aldrich | 238813 | |
| isopropyl β-d-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| LB broth | Sigma-Aldrich | L3152 | |
| Magnesium chloride | Sigma-Aldrich | 63068 | |
| MonoQ column | Sigma-Aldrich | 54807 | |
| protein LoBind tube | Sigma-Aldrich | EP0030108094 | 0.5 mL |
| Rinse a µ-slide 18 | Ibidi | 81816 | |
| SDS | Sigma-Aldrich | 75746 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | S8045 | |
| Sodium phosphate monobasic monohydrate | Sigma-Aldrich | 71507 | |
| Sterile Cell spreaders, Drigalski spatulas | mini-plast | 815-004-05-001 | |
| streptomycin sulfate | Sigma-Aldrich | S9137 | |
| sulfo-Cy3 maleimide | abcam | ab146493 | |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | 75259 | |
| Tris-HCl | Sigma-Aldrich | 93363 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Yeast Extract | Sigma-Aldrich | Y1625 |
Riferimenti
- Gust, A., et al. A starting point for fluorescence-based single-molecule measurements in biomolecular research. Molecules. 19 (10), 15824-15865 (2014).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Haran, G. Single-molecule fluorescence spectroscopy of biomolecular folding. Journal of Physics Condensed Matter. 15 (32), R1291 (2003).
- Schuler, B., Lipman, E. A., Eaton, W. A. Probing the free-energy surface for protein folding with single-molecule fluorescence spectroscopy. Nature. 419 (6908), 743-747 (2002).
- Michalet, X., Weiss, S., Jäger, M. Single-molecule fluorescence studies of protein folding and conformational dynamics. Chemical Reviews. 106 (5), 1785-1813 (2006).
- Ha, T., Enderle, T., Ogletree, D. F., Chemla, D. S., Selvin, P. R., Weiss, S. Probing the interaction between two single molecules: Fluorescence resonance energy transfer between a single donor and a single acceptor. Proceedings of the National Academy of Sciences of the United States of America. 93 (13), 6264-6268 (1996).
- Deniz, A. A., et al. Single-molecule protein folding: Diffusion fluorescence resonance energy transfer studies of the denaturation of chymotrypsin inhibitor 2. Proceedings of the National Academy of Sciences of the United States of America. 97 (10), 5179-5184 (2000).
- Bialik, C. N., Wolf, B., Rachofsky, E. L., Ross, J. B. A., Laws, W. R. Dynamics of biomolecules: Assignment of local motions by fluorescence anisotropy decay. Biophysical Journal. 75 (5), 2564-2573 (1998).
- Jameson, D. M., Sawyer, W. H. Fluorescence anisotropy applied to biomolecular interactions. Methods in Enzymology. 246, 283-300 (1995).
- Kempe, D., Schöne, A., Fitter, J., Gabba, M. Accurate Fluorescence Quantum Yield Determination by Fluorescence Correlation Spectroscopy. Journal of Physical Chemistry B. 119 (13), 4668-4672 (2015).
- Callis, P. R., Liu, T. Quantitative Prediction of Fluorescence Quantum Yields for Tryptophan in Proteins. Journal of Physical Chemistry B. 108, 4248-4259 (2004).
- Lehrer, S. S. Solute Perturbation of Protein Fluorescence. The Quenching of the Tryptophyl Fluorescence of Model Compounds and of Lysozyme by Iodide Ion. Biochemistry. 10 (17), 3254-3263 (1971).
- Xie, K. X., Liu, Q., Jia, S. S., Xiao, X. X. Fluorescence enhancement by hollow plasmonic assembly and its biosensing application. Analytica Chimica Acta. 1144, 96-101 (2021).
- Stennett, E. M. S., Ciuba, M. A., Lin, S., Levitus, M. Demystifying PIFE: The Photophysics behind the Protein-Induced Fluorescence Enhancement Phenomenon in Cy3. Journal of Physical Chemistry Letters. 6 (10), 1819-1823 (2015).
- Nguyen, B., Ciuba, M. A., Kozlov, A. G., Levitus, M., Lohman, T. M. Protein Environment and DNA Orientation Affect Protein-Induced Cy3 Fluorescence Enhancement. Biophysical Journal. 117 (1), 66-73 (2019).
- Song, D., Graham, T. G. W., Loparo, J. J. A general approach to visualize protein binding and DNA conformation without protein labelling. Nature Communications. 7, 10976 (2016).
- Ploetz, E., et al. Förster resonance energy transfer and protein-induced fluorescence enhancement as synergetic multi-scale molecular rulers. Scientific Reports. 6, 33257 (2016).
- Hwang, H., Myong, S. Protein induced fluorescence enhancement (PIFE) for probing protein-nucleic acid interactions. Chemical Society Reviews. 43 (4), 1221-1229 (2014).
- Hwang, H., Kim, H., Myong, S. Protein induced fluorescence enhancement as a single molecule assay with short distance sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 108 (18), 7414-7418 (2011).
- Ray, P. C., Fan, Z., Crouch, R. A., Sinha, S. S., Pramanik, A. Nanoscopic optical rulers beyond the FRET distance limit: Fundamentals and applications. Chemical Society Reviews. 43, 6370-6404 (2014).
- Chen, H., Rhoades, E. Fluorescence characterization of denatured proteins. Current Opinion in Structural Biology. 18 (4), 516-524 (2008).
- Alderson, T. R., Markley, J. L. Biophysical characterization of α-synuclein and its controversial structure. Intrinsically Disordered Proteins. 1 (1), 18-39 (2013).
- Mane, J. Y., Stepanova, M. Understanding the dynamics of monomeric, dimeric, and tetrameric α-synuclein structures in water. FEBS Open Bio. 6 (7), 666-686 (2016).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Trexler, A. J., Rhoades, E. α-Synuclein binds large unilamellar vesicles as an extended helix. Biochemistry. 48 (11), 2304-2306 (2009).
- Stephens, A. D., Zacharopoulou, M., Kaminski Schierle, G. S. The Cellular Environment Affects Monomeric α-Synuclein Structure. Trends in Biochemical Sciences. 44 (5), 453-466 (2019).
- Illes-Toth, E., Dalton, C. F., Smith, D. P. Binding of dopamine to alpha-synuclein is mediated by specific conformational states. Journal of the American Society for Mass Spectrometry. 24 (9), 1346-1354 (2013).
- Frimpong, A. K., Abzalimov, R. R., Uversky, V. N., Kaltashov, I. A. Characterization of intrinsically disordered proteins with electrospray inization mass spectrometry: Conformationl heterogeneity of α-synuciein. Proteins: Structure, Function and Bioinformatics. 78 (3), 714-722 (2010).
- Sandal, M., et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level. PLoS Biology. 6 (1), e6 (2008).
- Brodie, N. I., Popov, K. I., Petrotchenko, E. V., Dokholyan, N. V., Borchers, C. H. Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations. PLoS Computational Biology. 15 (3), e1006859 (2019).
- Ullman, O., Fisher, C. K., Stultz, C. M. Explaining the structural plasticity of α-synuclein. Journal of the American Chemical Society. 133 (48), 19536-19546 (2011).
- Binolfi, A., Theillet, F. X., Selenko, P. Bacterial in-cell NMR of human α-synuclein: A disordered monomer by nature?. Biochemical Society Transactions. 40 (5), 950-954 (2012).
- Waudby, C. A., et al. In-Cell NMR Characterization of the Secondary Structure Populations of a Disordered Conformation of α-Synuclein within E. coli Cells. PLoS ONE. 8 (8), e72286 (2013).
- Yu, J., Malkova, S., Lyubchenko, Y. L. α-Synuclein Misfolding: Single Molecule AFM Force Spectroscopy Study. Journal of Molecular Biology. 384 (4), 992-1001 (2008).
- Guerrero-Ferreira, R., Kovacik, L., Ni, D., Stahlberg, H. New insights on the structure of alpha-synuclein fibrils using cryo-electron microscopy. Current Opinion in Neurobiology. 61, 89-95 (2020).
- Trexler, A. J., Rhoades, E. Single molecule characterization of α-synuclein in aggregation-prone states. Biophysical Journal. 99 (9), 3048-3055 (2010).
- Ferreon, A. C. M., Gambin, Y., Lemke, E. A., Deniz, A. A. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence. Proceedings of the National Academy of Sciences of the United States of America. 106 (14), 5645-5650 (2009).
- Rezaei-Ghaleh, N., et al. Local and Global Dynamics in Intrinsically Disordered Synuclein. Angewandte Chemie - International Edition. 57 (46), 15262-15266 (2018).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An Open File Format for Timestamp-Based Single-Molecule Fluorescence Experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Ingargiola, A., Lerner, E., Chung, S. Y., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing Single-molecule FRET. PLoS ONE. 11 (8), e0160716 (2016).
- Ingargiola, A., et al. Multispot single-molecule FRET: Highthroughput analysis of freely diffusing molecules. PLoS ONE. 12 (4), e0175766 (2017).
- Eggeling, C., Fries, J. R., Brand, L., Günther, R., Seidel, C. A. M. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. A. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102, 6601-6613 (1998).
- Hoffmann, A., et al. Quantifying heterogeneity and conformational dynamics from single molecule FRET of diffusing molecules: Recurrence analysis of single particles (RASP). Physical Chemistry Chemical Physics. 13 (5), 1857-1871 (2011).
- Eakin, C. M., Berman, A. J., Miranker, A. D. A native to amyloidogenic transition regulated by a backbone trigger. Nature Structural and Molecular Biology. 13 (3), 202-208 (2006).
- Jahn, T. R., Parker, M. J., Homans, S. W., Radford, S. E. Amyloid formation under physiological conditions proceeds via a native-like folding intermediate. Nature Structural and Molecular Biology. 13 (3), 195-201 (2006).
- Chen, D., et al. Tau local structure shields an amyloid-forming motif and controls aggregation propensity. Nature Communications. 10 (1), 2493 (2019).
- Lerner, E., Ploetz, E., Hohlbein, J., Cordes, T., Weiss, S. A Quantitative Theoretical Framework for Protein-Induced Fluorescence Enhancement-Förster-Type Resonance Energy Transfer (PIFE-FRET). Journal of Physical Chemistry. B. 120 (26), 6401-6410 (2016).
- Valuchova, S., Fulnecek, J., Petrov, A. P., Tripsianes, K., Riha, K. A rapid method for detecting protein-nucleic acid interactions by protein induced fluorescence enhancement. Scientific Reports. 6, 39653 (2016).
- Qiu, Y., Myong, S. Single-Molecule Imaging With One Color Fluorescence. Methods in Enzymology. 581, 33-51 (2016).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), eaan1133 (2018).
- Lerner, E., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Wang, W., et al. A soluble α-synuclein construct forms a dynamic tetramer. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17797-17802 (2011).
- Bartels, T., Choi, J. G., Selkoe, D. J. α-Synuclein occurs physiologically as a helically folded tetramer that resists aggregation. Nature. 477 (7362), 107-110 (2011).
- Ulmer, T. S., Bax, A., Cole, N. B., Nussbaum, R. L. Structure and dynamics of micelle-bound human α-synuclein. Journal of Biological Chemistry. 280 (10), 9595-9603 (2005).
- Georgieva, E. R., Ramlall, T. F., Borbat, P. P., Freed, J. H., Eliezer, D. Membrane-bound α-synuclein forms an extended helix: Long-distance pulsed ESR measurements using vesicles, bicelles, and rodlike micelles. Journal of the American Chemical Society. 130 (39), 12856-12857 (2008).
- Chen, J., et al. The structural heterogeneity of α-synuclein is governed by several distinct subpopulations with interconversion times slower than milliseconds. Structure. 29 (9), (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon