Method Article
量化草甘膦产品对微生物组的潜在影响
摘要
草甘膦产品(GBP)是全球最常见的广谱除草剂。在本文中,我们介绍了量化GBP对微生物组影响的一般指南,从现场实验到生物信息学分析。
摘要
草甘膦产品(GBP)是全球最常见的广谱除草剂。草甘膦的靶标是莽草酸途径中的5-烯醇丙酮基敷素-3-磷酸合酶(EPSPS),这在植物中几乎普遍存在。酶的抑制阻止了三种必需氨基酸的产生:苯丙氨酸,酪氨酸和色氨酸。EPSPS也存在于真菌和原核生物中,如古菌和细菌;因此,GBP的使用可能会对土壤,植物,食草动物和次级消费者的微生物组组成产生影响。本文旨在提供一般指南,以评估GBP对微生物组的影响,从现场实验到生物信息学分析,并提供一些可测试的假设。提出了两个现场实验来测试非目标生物体上的GBP。首先,对10个模拟免耕种植的复制对照和GBP处理样地的植物相关微生物进行采样和分析。在第二个实验中,从含有草甘膦残留物的家禽粪便或未经处理的对照粪便施肥的实验样地中获得样品。利用EPSPS蛋白序列的生物信息学分析来确定微生物对草甘膦的潜在敏感性。估计GBP对微生物组影响的第一步是确定它们对靶酶(EPSPS)的潜在敏感性。微生物序列可以从公共存储库或通过PCR扩增获得。然而,在大多数现场研究中,微生物组组成是基于通用DNA标记物(如16S rRNA和内部转录间隔体(ITS))确定的。在这些情况下,对草甘膦的敏感性只能通过使用密切相关物种对EPSPS序列进行概率分析来估计。基于EPSPS酶的生物体对草甘膦潜在敏感性的定量,为进一步研究靶标和非靶标抗性机制的实验提供了一种可靠的方法。
引言
现代农业中大量使用杀虫剂显然是导致生物多样性下降的主要原因1。本文重点介绍草甘膦,因为草甘膦基产品(GBPs)由于其效率和可承受的价格而成为全球使用最广泛的农药2,3。除了在农田中杀死杂草外,GBPs还通常用于造林,城市环境和家庭花园;此外,如果按照制造商的说明使用,它们已被宣布为对非目标生物体无毒。然而,越来越多的近期研究表明,草甘膦及其降解产物的残留物可能保留在土壤中并运输,从而对非目标生物体产生级联作用4,5,6,7,8 。草甘膦的作用不仅限于植物 - 莽草酸途径也存在于许多真菌和原核生物中。草甘膦靶向莽草酸途径中的5-烯醇丙酮基志糖酸-3-磷酸合酶(EPSPS),也称为aroA9。该酶是三种必需芳香氨基酸(苯丙氨酸,酪氨酸和色氨酸)合成中莽草酸途径的中心,并且存在于大多数原核生物,植物和真菌10,11中。一些微生物物种通过几种机制(包括EPSPS序列中的突变)对草甘膦产生了部分或绝对的抗性。因此,有人提出,使用GBPs可能对植物和动物微生物组产生直接影响,包括人类肠道微生物组12,13,14。然而,英镑的使用可能会对依赖微生物和微生物促进过程的几乎任何生态系统功能和服务产生不利影响。随之而来的威胁可能涉及生化土壤过程,授粉生物学以及动物和人类福祉。这需要更全面地了解草甘膦如何影响莽草酸盐途径和方法来评估微生物对草甘膦的敏感性。
在该协议中,我们提出了一个管道来测试草甘膦和GBP对微生物组的影响,从现场实验到生物信息学分析。我们详细描述了最近发表的一种生物信息学方法,可用于确定生物体对草甘膦的潜在敏感性12。据研究人员所知,这是第一个也是迄今为止唯一一个评估EPSPS酶对GBPs活性成分的内在敏感性的生物信息学工具。这种生物信息学方法基于对草甘膦靶酶(EPSPS)12中已知氨基酸标记物的检测。该管道分为五个主要工作阶段(图1):1)简要介绍两个现场实验以测试GBPs的效果,2)微生物组分析(16S rRNA,ITS和 EPSPS 基因)的简要摘要,3)从公共存储库收集EPSPS序列,4)确定生物体对草甘膦的潜在敏感性,以及5)从通用微生物标记物(16S rRNA和ITS)评估EPSPS类。
研究方案
1. 两次现场实验测试GBPs的效果
注意:该协议提供了两个现场实验设计示例,以测试GBP对植物相关微生物的影响。这两项实验都是在芬兰图尔库鲁伊萨洛大学植物园(60º26'N,22º10'E)没有除草剂或农业用途史的闲置田地进行的。土壤为沙质粘土,有机质比例高。
- 实验 1
注意:该实验旨在模拟免耕农业的一般农业实践,在生长季节之前和之后应用GBP来对抗杂草。- 将实验场划分为10个复制对照和GBP处理图(23 m x 1.5 m),并在地块之间具有植被缓冲条(在2014年春季的这项研究中为15)(图2)。
- 确保用旋耕机将地块耕作到5厘米的深度,并每年处理两次。在这里,这些地块在生长季节的开始(5月)和结束(10月)进行处理。
- 自来水处理对照样地(5 L/地块),商业GBP处理GBP样地(草甘膦浓度450 g·L-1,施用率6.4 L·ha-1 /5 L自来水)以模仿农业实践中草甘膦的最大允许用量(3 kg·ha-1)。
- 使用带有手动喷雾器的手动压力罐进行处理。在GBP应用两周后,根据农业实践在地块中种燕麦(Avena sativa),蚕豆(Vicia faba),萝卜油菜(Brassica rapa subsp. oleifera)和植物马铃薯(Solanum tuberosum)。
- 在生长季节,对地块进行手工除草,以保持植物竞争和土壤结构在对照和GBP处理的地块中尽可能相似。
- 从实验植物中取样微生物群。在这项研究中,从2017年到2020年,在GBP处理的和对照处理地块中连续对微生物群进行采样,在整个研究过程中,每个生长季节一次。
- 从田间收集十个重复的植物样品(根和叶),立即将它们放在冰上,并将它们带到实验室进行进一步处理,如第2.1节所述。
- 实验 2
注意:该实验旨在测试与循环食品经济相关的风险;更准确地说,它旨在检查粪便中GBP残留物作为肥料施用于作物植物2 的后果(图3)。- 在为期12个月的鸟舍实验中,从以GBP污染或对照饲料喂养的鹌鹑中收集垫料,包括木屑,粪便和一些溢出的饲料16,17。
注:受GBP污染的饲料包括用于蛋鸡的有机饲料,相当于160毫克草甘膦/公斤,相当于成年日本鹌鹑每公斤体重每天摄入12-20毫克草甘膦16,17。 - 为了进行验证,将样品送到认可的实验室,以六批次进料测量草甘膦浓度。
- 此外,在暴露12个月后测量鹌鹑排泄物样品中的草甘膦残留物。对照组喂食相同的有机饲料,没有增加16,17英镑。
- 在鸟舍实验期间,每两周更换一次床上用品。从GBP处理和控制中定期收集8-12个月的暴露中使用的床上用品,每次处理池,并在6°C的干燥,黑暗的储藏室中储存在密闭容器中,然后用作肥料。
- 在实验场的6 x 6棋盘网格中,在两个时间点,在18 GBP和18个控制图(尺寸1 m x 1 m)上手动铺设12 L。在这项研究中,床上用品在2018年8月和2019年5月进行了铺设。
- 将铺垫样品在铺展后立即送到认可的实验室进行草甘膦浓度测量(在2019年5月的这项研究中)。
- 在每个地块种植多年生草和草莓植物,以研究其根和叶微生物群。
注意:在这项研究中,将四种多年生草本植物(Festuca pratensis)和两种草莓植物(Fragaria x vescana)种植到每个地块,并研究其根和叶微生物群。
- 在为期12个月的鸟舍实验中,从以GBP污染或对照饲料喂养的鹌鹑中收集垫料,包括木屑,粪便和一些溢出的饲料16,17。
2. 微生物组分析(16S rRNA、 ITS 和 EPSPS 基因)
注意:大多数微生物组研究都是基于使用下一代测序技术对细菌和真菌群落内部转录间隔(ITS)区域的16S rRNA基因的分析。因此,该论文没有关于EPSPS类型的信息。来自数千个物种的EPSPS序列可在公共存储库中找到(协议第3节)(图4)。
- 16S rRNA基因
- 从上述实验中收集的分离的叶子和根部样品中,鉴定内生微生物(即生活在植物组织内的微生物)。
- 用自来水清洗植物样品,然后对其进行灭菌以除去附生微生物(即植物组织表面的微生物)。使用3%漂白剂灭菌3分钟,然后用70%乙醇溶液灭菌1分钟,并用高压灭菌的超纯水洗涤三次,每次1分钟。
- 在-80°C下冷冻样品,直到基因组DNA提取。
- 按照制造商的协议,使用市售植物DNA提取试剂盒进行基因组DNA提取。
- 使用嵌套方法靶向从提取的DNA样品中提取的16S rRNA基因的可变区域V6-V8,该方法具有与细菌DNA18特异性结合的判别引物,从而最大限度地减少宿主植物DNA的扩增。
- 经过三轮聚合酶链反应(PCR)后,用条形码和适配器序列标记靶基因,以将其制备为测序模板。按照步骤 2.1.7- 2.1.11 进行 PCR 扩增
- 为所需数量的样品制备PCR预混液,使每个反应的总体积为30 μLof,由30 ng DNA,1x PCR缓冲液,0.2mM dNTPs,每个引物的0.3μM和2000 U / mL DNA聚合酶组成。在第二轮和第三轮PCR中遵循相同的步骤。
- 对于第一轮PCR,使用引物799F19 和1492R(从20修改而来)(表1)。在热循环仪上设置扩增曲线(在95°C下初始变性3分钟,然后在95°C下变性35个循环45秒,在54°C下退火45秒,并在72°C下延伸1分钟)。在72°C下进行最终延伸5分钟。
- 作为第二轮PCR的模板,通过电泳(5μLPCR产物在1.5%琼脂糖凝胶上)验证扩增,然后以1:10的比例在高压灭菌的超纯水中稀释剩余的25μLPCR产物。
- 用稀释的PCR模板和引物Uni-1062F21 和uni-1390R22重复PCR(表1)。保持相同的PCR反应条件和扩增曲线(步骤2.1.8),除了将循环次数减少到25次。
- 将所得PCR产物以1:1的比例在高压灭菌的超纯水中稀释。进行第三轮PCR,用条形码和P1适配器序列标记产品,其8个周期与步骤2.1.8中提到的相同PCR曲线。
- 文库准备
- 在生物分析仪上验证PCR产物的浓度和质量,并将每个样品的30 ng DNA体积池化在1.5 mL管中以制备等摩尔文库。
- 使用琼脂糖凝胶盒上的自动DNA尺寸选择系统,通过尺寸分馏选择大小为350-550 bp的扩增子。请注意,这也消除了文库中的非特异性扩增子和PCR试剂。将由指定大小的扩增子组成的洗脱液收集到盒中的小瓶中,从而获得纯化的16S rRNA基因文库。
- 将洗脱液移出到1.5 mL管中,并验证生物分析仪上的纯度和浓度。使用高压灭菌的超纯水将DNA文库稀释至终浓度为26 pM;样品已准备好进行测序。
- 其
注意:使用 ITS 特异性引物扩增 ITS区域(表1),并且所得PCR产物用条形码和P1适配器序列标记以进行测序。- 根据与第2.1节中提到的相同方案与 ITS 引物制备PCR预混液。
- 将热循环仪上的扩增曲线设置为在95°C下5分钟的初始变性,然后在95°C下进行35次变性,退火和延伸30 s,55°C持续30 s,72°C持续1分钟,最终延伸72°C持续7分钟。
- 在1.5%琼脂糖凝胶上分析5μLPCR产物,并使用高压灭菌的超纯水将剩余的25μL稀释至1:10。使用稀释的PCR产物作为第二轮PCR的模板。
- 使用条形码标记的正向引物和P1适配器标记的反向引物制备所需数量的样品的PCR预混液(参见步骤2.1.6)。使用与步骤2.3.2中相同的扩增曲线进行扩增,但使用8个周期。
- 根据2.2节中提到的方案制备所得的PCR产物进行测序。
- 断续器基因
- 对微生物的 EPSPS 基因进行测序和分析。
注意:为了确定GBP暴露是否改变了群落中草甘膦敏感和抗性微生物的组成,需要对微生物的EPSPS基因进行测序和分析。因此,从可对齐紧密基因组簇(ATGC)数据库中的广泛微生物分类群集合中收集了353个EPSPS基因序列,并且所有蛋白质序列均对齐了22个。这些比对可在ATGC数据库23获得,并可用于从保守区域生成引物。一个易于使用的生物信息学工具旨在从多个序列比对中识别保守区域,这可以在Pere Puigbo研究第24页找到。但是,提供此 Web 服务器的详细说明超出了本出版物的范围。然而,图4中提供了利用这些引物扩增EPSPS基因以发现微生物组对草甘膦的敏感性的前瞻性方案。
- 对微生物的 EPSPS 基因进行测序和分析。
3. 从公共储存库收集EPSPS蛋白序列
- 用于宏观进化研究的EPSPS序列
- 从公共存储库收集EPSPS蛋白,例如PFAM23 (蛋白质家族25的数据库),GenBank24(基因,基因组和蛋白质26 的数据库),COG25(直系群27的簇;来自古细菌和细菌的直系蛋白质数据库);和PDB26(蛋白质数据库28;蛋白质结构数据库)。
注意:研究人员最近进行的一项研究表明,这些蛋白质可用于对草甘膦对具有莽草酸酯途径12的生物体的潜在影响进行微观进化和比较分析。作者开发了一个用户友好的网站,收集了数以万计的EPSPS蛋白质序列29的信息,包括来自人类肠道微生物组12的手动策划的蛋白质数据集。这些预先计算的数据集中的信息包括当前EPSPS分类为假定的敏感和抗草甘膦,物种分类信息,EPSPS活动站点的注释以及PDB和NCBI数据库的链接。此外,Web 服务器还包括 EPSPS 的 ID 代码和指向多个外部数据库的链接(表 2)。
- 从公共存储库收集EPSPS蛋白,例如PFAM23 (蛋白质家族25的数据库),GenBank24(基因,基因组和蛋白质26 的数据库),COG25(直系群27的簇;来自古细菌和细菌的直系蛋白质数据库);和PDB26(蛋白质数据库28;蛋白质结构数据库)。
- EPSPS序列将用于微进化研究ATGC
注:蛋白质序列的一般储存库可用于在相对较远的生物体中进行比较研究;然而,从进化的角度来看,草甘膦的潜在作用是相对较新的。因此,在一些研究中,有必要比较密切相关的物种(例如,来自同一细菌物种的不同菌株)以确定草甘膦14的作用。在这些情况下,可对齐紧密基因组簇(ATGC)30的数据库是一个更适合的资源,其中包含密切相关的古细菌和细菌基因组的完整列表。ATGC数据库包含来自数千个基因组的数百万个蛋白质的信息,这些基因组组织成数百个簇30。每个基因组簇都是可对齐的(基因组在其长度的≥85%以上共享同源性)和紧密(具有低于饱和度的同义词取代率)。研究人员在最近的一项研究中使用ATGC数据集来分析EPSPS蛋白14的微观进化变化。需要以下步骤来鉴定ATGC中的EPSPS蛋白序列:- 将ATGC的整个数据库从链接31 和COG0128的所有蛋白质(数据库中与EPSPS蛋白质相对应的代码)32 下载到本地项目中。
注意:如果研究人员/实验者位于芬兰,CSC-IT Center for Science33 提供存储和软件设施。以 FASTA 格式收集所有序列非常重要。 - 建立了COG0128的爆炸数据库,其中包含一组代表性原核生物物种中EPSPS蛋白的同源物。CSC预装了爆炸程序34 ,允许使用命令 makeblastdb -in COG0128.fa -dbtype prot 来创建EPSPS序列的参考数据库。
- 使用迭代爆炸搜索将ATGC数据库映射到COG0128.fa(EPSPS蛋白),命令 blastp -query [ATGC_X.fa] -db [COG0128.fa] -max_target_seqs 1 -outfmt 6 -out tmpfile -evalue 1e-150。
- 因此,它在每个数据集内创建了EPSPS蛋白序列的数据集。来自ATGC数据库的密切相关的EPSPS蛋白序列的预先计算数据集可用29。
- 将ATGC的整个数据库从链接31 和COG0128的所有蛋白质(数据库中与EPSPS蛋白质相对应的代码)32 下载到本地项目中。
4. 确定生物体对草甘膦潜在敏感性的算法(EPSPSClass Web服务器:输入、处理和输出)
注意:研究人员已经实现了一个易于使用的服务器,该服务器在 29 处免费提供,以确定EPSPS蛋白序列的类别12,35。服务器只需要以FASTA格式输入蛋白质序列,以确定每个EPSPS类别的同一性百分比及其对草甘膦的潜在敏感性。此外,用户可以利用Web服务器来测试自己的参考序列和氨基酸标记。首先,该算法(图5)使用多序列比对程序35 对齐查询序列和参考序列以确定氨基酸位置。然后,它搜索氨基酸标记物的存在以识别查询序列的EPSPS类(I,II,III或IV)。
- 将FASTA格式的EPSPS蛋白序列引入输入文本框以识别酶的类别(图6A),然后按 发送。
- 从服务器提供的输出评估查询序列对草甘膦的潜在敏感性(图6B-E):
输出1:查询序列中存在的氨基酸标记物(即同一性)的比例(I类,II类和IV类)和基序的数量(III类)。
输出 2:基于标记残留物的查询和参考序列的对齐。
输出 3:查询和引用序列的完全成对对齐。
产出4:EPSPS参考序列: 霍乱弧菌(vcEPSPS,I类), 贝氏柯克斯体(cbEPSPS,II类), 水疱性短链霉菌(bvEPSPS,III类), 达瓦氏链霉菌(sdEPSPS,IV类)。 - 在输出页面的末尾,找到指向外部工具(如 blastp 和保守域)的链接,以进一步分析查询 EPSPS 序列(图 6F)。
5. 从通用微生物标志物(16S rRNA 和 ITS )评估 EPSPS 类
注意:大多数微生物组研究都是基于对16S rRNA和/或ITS36的分析。在这种情况下,不可能对EPSPS序列进行直接分析。因此,有必要采用概率方法来估计生物体对草甘膦的潜在敏感性。该分析很简单,可以合理估计微生物组项目中EPSPS序列的类型。该过程分为 3 个步骤(图 7 和 图 8):
- 识别公共存储库中的 EPSPS 序列。代表序列综合数据集的EPSPS类已从PFAM37,GenBank38,COG39,PDB40,ATGC30编译并预先计算。从 EPSPSClass 服务器的主页访问这些数据集,其中包含分类信息和超过 50,000 个序列的 EPSPS 类(图 7)。
- 在生长季节每两周测量实验植物的高度,并在田间季节结束时称量植物的地上生物量,以比较植物在GBP和控制地块中的生长情况。
注意:来自现场实验的微生物群分析尚未得到充分分析。 - 使用电子表格将微生物组实验中的细菌 OTI(基于 16S rRNA 或 ITS)映射到预先计算的数据集中。
注意:以前的研究表明,EPSPS类(即对草甘膦的固有敏感性)在系统发育组14中高度保守。因此,可以相对安全地假设来自高度保守的分类单元的密切相关物种可能对草甘膦具有相似的EPSPS响应(图8)。 - 在同一电子表格中,根据概率评分(S = s / (s + r + u)计算对草甘膦的固有敏感性,其中S:敏感性评分;s:潜在敏感序列的数量;r:潜在耐药序列的数量;u:未分类序列的数量)根据公共数据库中已知的EPSPS序列计算。
注意:该评分范围从0(在分类单元中没有发现敏感的EPSPS序列)到1(分类单元中的所有序列都对草甘膦敏感)(图8)。此外,在两者之间有值,即具有敏感,耐药性或未知菌株的物种。
结果
该协议的目的是提供从现场实验到生物信息学分析的一般管道,以量化生物体对除草剂草甘膦的潜在敏感性。在实验2中,鹌鹑饲料中的平均草甘膦浓度为164毫克/千克,排泄物样品的平均草甘膦浓度(尿液和粪便物质合计)为199毫克/千克。从用受GBP污染的饲料喂养的鹌鹑中收集的垫料平均为158毫克/千克,对照垫料的草甘膦含量为0.17毫克/千克(表3)。在实地试验中,植物物种对土壤中的草甘膦残留物的反应不同(第1节)。与GBP处理的土壤相比,控制土壤中燕麦和萝卜油菜的生物量更高。然而,蚕豆和马铃薯似乎在第15个生长季节结束时受益于GBP处理。家禽粪便中的草甘膦降低了草(Festuca pratensis)和草莓(Fragaria x vescana)(第1节)的植物生长。来自现场实验的微生物群分析尚未得到充分分析,这里不作介绍(第2节)。当直接(如第3节和第4节所示)或间接(第5节)读取该协议的结果时,提供了数据集中潜在敏感和耐药生物体与草甘膦的比例的度量(图9)。使用该方法的使用与从公共存储库12获得的来自人类核心肠道微生物组微生物物种的EPSPS蛋白序列的集合进行测试。在这项研究中,使用EPSPSClass方法分析了来自101种最丰富的细菌物种的890株菌株,以量化敏感和耐药细菌的比例。结果显示,人类核心肠道微生物组中54%的物种对草甘膦12具有潜在敏感性。在大多数原核世界中也观察到这种趋势。此外,在真核生物(主要是植物和真菌)中,潜在敏感物种的比例甚至更高12。此外,我们已经利用这种方法在微观进化水平上量化EPSPS蛋白灵敏度的变化(图10)14。在所分析的32组密切相关的原核生物组中,有12组确定了敏感性状态的变化(表4)14。因此,连续使用GBPs可能会在植物,动物和土壤微生物组中产生微生物生态失调(即敏感和耐药细菌物种的不平衡)。此外,已经假设草甘膦耐药细菌的增加可能促进多药耐药微生物组14,41,42。因此,该协议阐明了所有这些场景的解释,因为EPSPS分类方法提供了微生物组对草甘膦的内在敏感性的直接估计。由于EPSPS蛋白对草甘膦的内在敏感性在系统发育上是保守的14,因此可以将现有数据集的结果外推到未知的微生物组中(图8)。
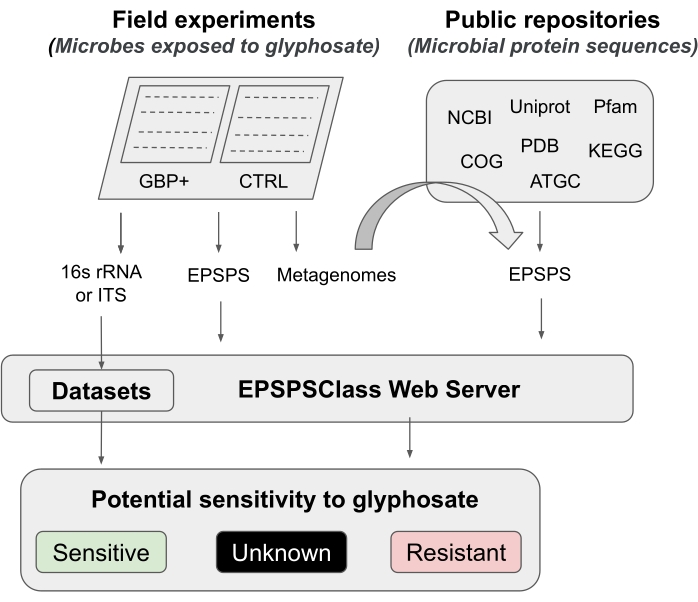
图1:通用管道 这是分析从现场实验到生物信息学分析对GBP敏感性的通用管道。 请点击此处查看此图的大图。
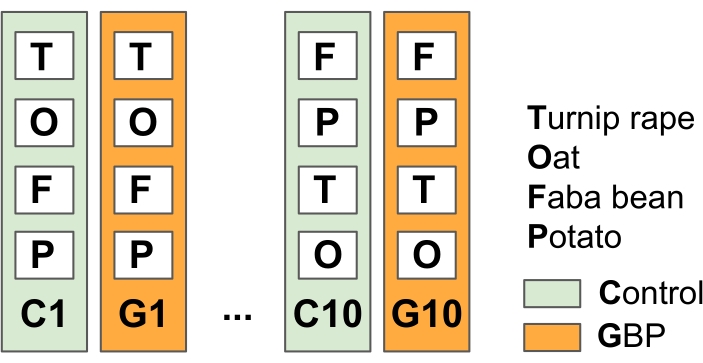
图2:田间实验1,以测试GBP残留物对作物植物相关微生物的影响。 实验场由交替的10个控制图和10个GBP处理图(23 m x 1.5 m)组成,图之间有1.5 m缓冲带。自2014年以来,每年2次对GBP样地进行商业GBP处理(每地块草甘膦浓度450 g L-1,施用率6.4 L ha-1/5 L自来水)和对照样地处理相同量自来水无草甘膦。使用手动操作的压力罐进行处理,该压力罐使用喷头中的塑料罩,以保护GBP不扩散到处理地块之外。在GBP施用后两周的安全期后,播种燕麦(Avena sativa),蚕豆(Vicia faba)和萝卜油菜(Brassica rapa subsp. oleifera),并在地块中种植土豆(Solanum tuberosum)。自2014年实验开始以来,从研究的作物植物,叶子和根部收集了多次微生物群样本。 请点击此处查看此图的大图。
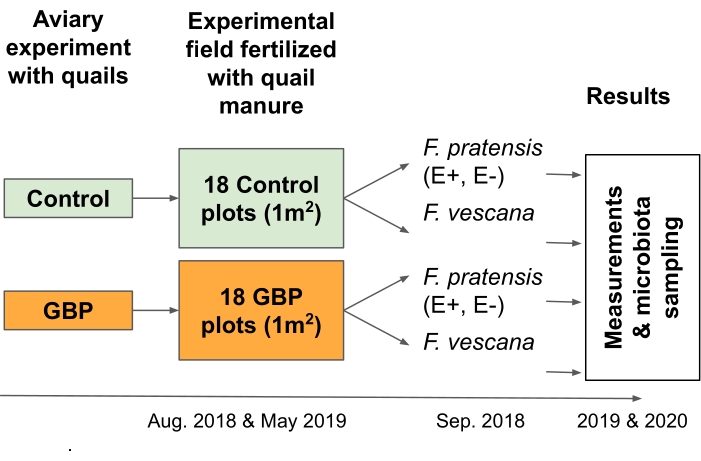
图3:田间实验2测试了两种多年生作物及其相关微生物群粪便肥料中GBP残留物的后果。 在田间实验中,从为期12个月的鸟舍实验中收集的垫料与对照或GBP污染的饲料喂养的日本鹌鹑一起使用,用作粪便肥料。实验场由18个控制和18个GBP图(1 m x 1 m)组成,排列在6 x 6棋盘网格中。床上用品在2018年8月和2019年5月两次在实验场上铺展(25 L /地块)。对照地块用从喂食对照饲料的鹌鹑中收集的垫料施肥,用用GBP污染的饲料喂养的鹌鹑的裂褒进行GBP地块的垫料施肥。对照垫料中的草甘膦残留量为0.17 mg/kg草甘膦,而GBP-垫层中草甘膦的残留量为158 mg/kg。2018年9月,在第一批垫料播种后约一个月,每地块种植了2株内生共生菌(E+)、2株无内生菌(E-) Festuca pratensis和2株 Fragaria x vescana 。在连续两个生长季节(2019年和2020年)进行了植物性能和适应性的测量以及根和叶相关微生物群的采样。 请点击此处查看此图的大图。
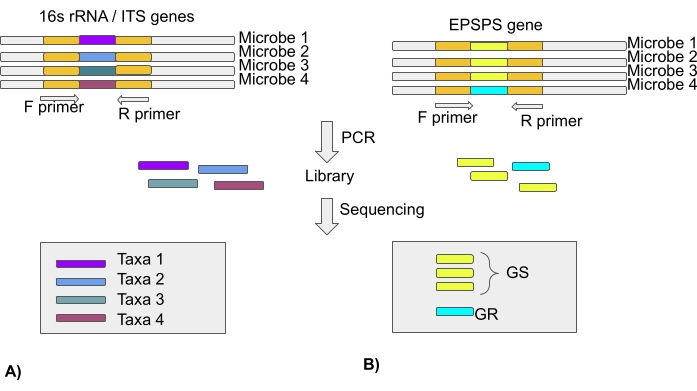
图4:使用16S rRNA基因/ ITS区域分析微生物分类群,并使用EPSPS基因分析微生物组对草甘膦的敏感性。(B)分析EPSPS序列以确定微生物对草甘膦的敏感性(GS-草甘膦敏感/GR-草甘膦抗性)请点击此处查看此图的大图。
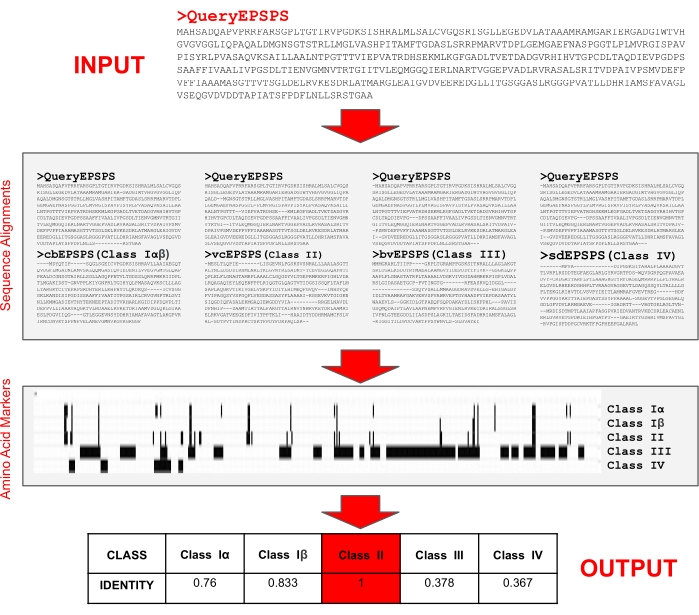
图5:鉴定EPSPS蛋白序列类别的算法。 输入是FASTA格式的EPSPS蛋白序列。该算法与参考蛋白质序列中的已知氨基酸标记物进行比较,以确定对草甘膦的潜在敏感性。该算法是在可自由访问的Web服务器EPSPSClass29上实现的。 请点击此处查看此图的大图。

图 6:EPSPSClass Web 服务器的基本输入和输出。 (A) 输入:FASTA 格式的 EPSPS 蛋白序列。(B) 产出1-同一性:查询序列中存在的氨基酸标记物(I-IV类)和基序(III类)的分数。(C) 输出 2 - 标识:查询序列和引用序列的对齐方式。(D) 输出 3 - 查询序列和引用序列的成对对齐。(E) 参考EPSPS序列: 霍乱弧菌 (vcEPSPS,I类), 贝氏柯克斯体 (cbEPSPS,II类), 水泡短尾单胞菌 (bvEPSPS,III类), 达瓦氏链霉菌 (sdEPSPS,IV类)。(F) 执行加法 blastp 搜索和识别保守域的链接 请单击此处查看此图的放大版本。
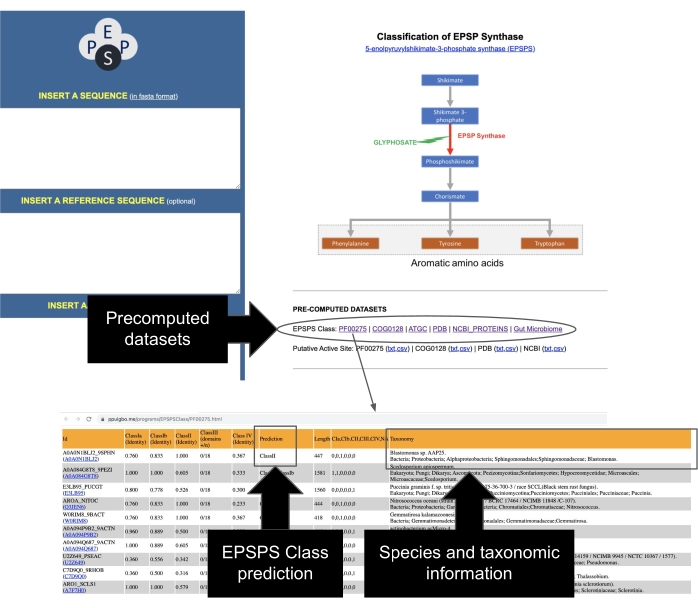
图 7:访问 EPSPS 序列的预计算数据集。 按照图中的指示访问 EPSPS 序列的预计算数据集。 请点击此处查看此图的大图。
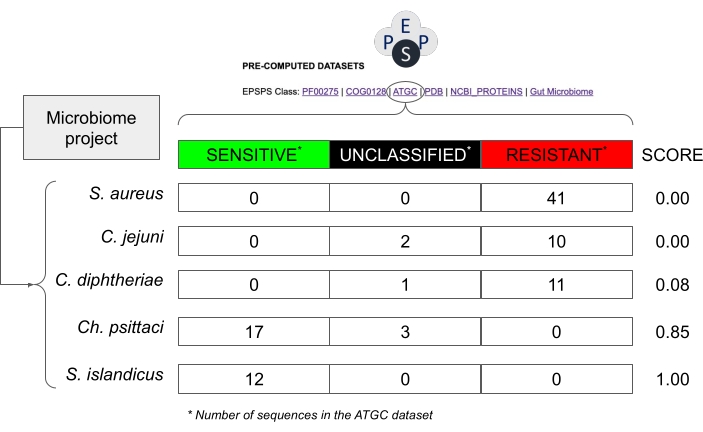
图8:如何在没有EPSPS序列的情况下估计微生物组项目中的潜在灵敏度的示例。 该示例使用可对齐紧密基因组簇30 数据库中的值,该数据库包含来自原核生物种的序列。来自微生物组项目的假设物种是 金黄色葡萄球菌, 白喉棒状杆菌, 空肠弯曲杆菌,鹦 鹉热衣原体 和 岛痣。草甘膦的敏感性评分计算为Number_Sensitive_Sequences/Total_Number_Of_Sequences。 请点击此处查看此图的大图。
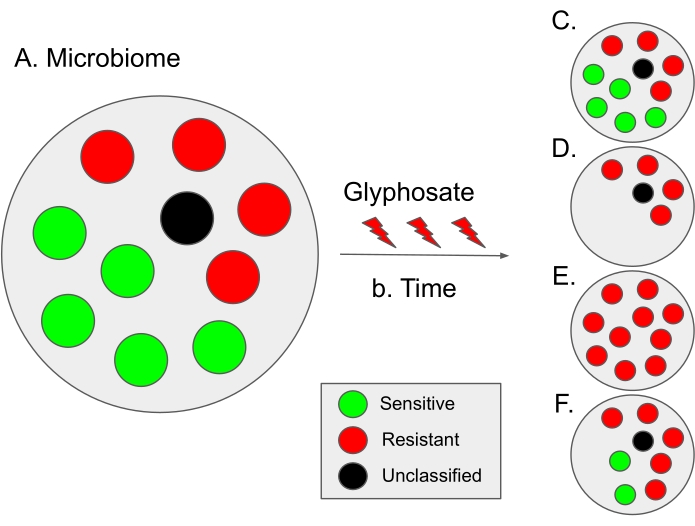
图9:该方案和假设进化情景的结果的解释方案。 (A)在微生物组中,潜在敏感性(绿色)和耐药性(红色)细菌的比例约为50:50。黑点表示未分类的微生物物种;因此,它们对草甘膦的敏感性是未知的。在一些微生物组中,敏感细菌的比例略高,如在人体肠道微生物组12中。(B)随着时间的推移,草甘膦的使用可能导致微生物生态失调(即敏感和耐药细菌比例的不平衡),从而导致不同的假设情景。(C) 假设情况1(无选择):草甘膦的使用不影响微生物组;因此,敏感和耐药细菌的比例保持不变。(D) 假设案例2:使用草甘膦从种群中去除对草甘膦敏感的细菌。我们推测这种情况可能是剂量依赖性的。(E)假设案例3:使用草甘膦产生的选择压力增强 了EPSPS 基因中的突变,从而改变了细菌的敏感性状态。因此,整个微生物群体对草甘膦产生抗性。此外,在这种情况下,耐多药细菌可能会增加。(F) 假设情况4:草甘膦的使用改变了某些细菌种类的组成,导致对耐药细菌的不平衡,而一些细菌种类保持不变,可能是由于额外的耐药机制,如外排泵或 EPSPS 基因13的过表达。这种情况还可能导致草甘膦耐药细菌的增加,以及细菌对其他抗生素的耐药性增加。 请点击此处查看此图的大图。
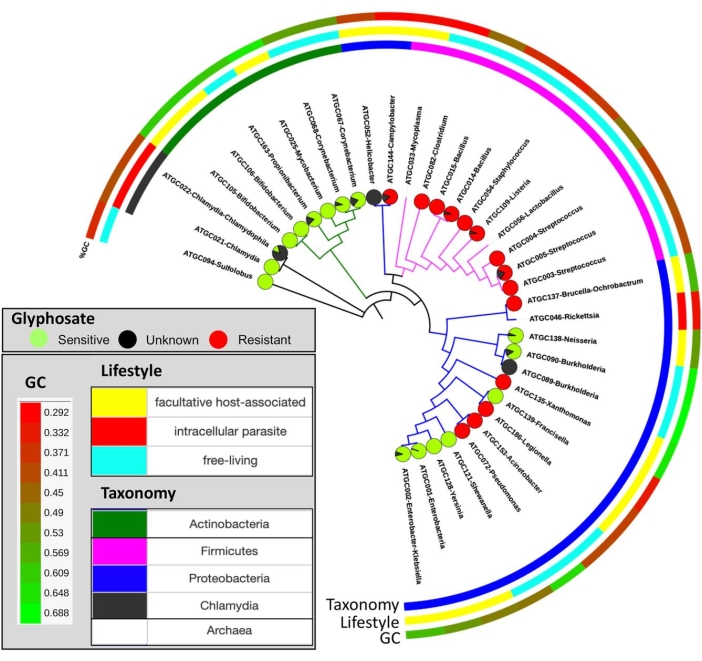
图10:预测的对草甘膦的敏感性在整个物种树上的分布。 饼图表示对草甘膦和未分类(黑色)的假定敏感(绿色)或抗性(红色)物种的比例。这个数字是在Rainio等人14的许可下改编的。 请点击此处查看此图的大图。
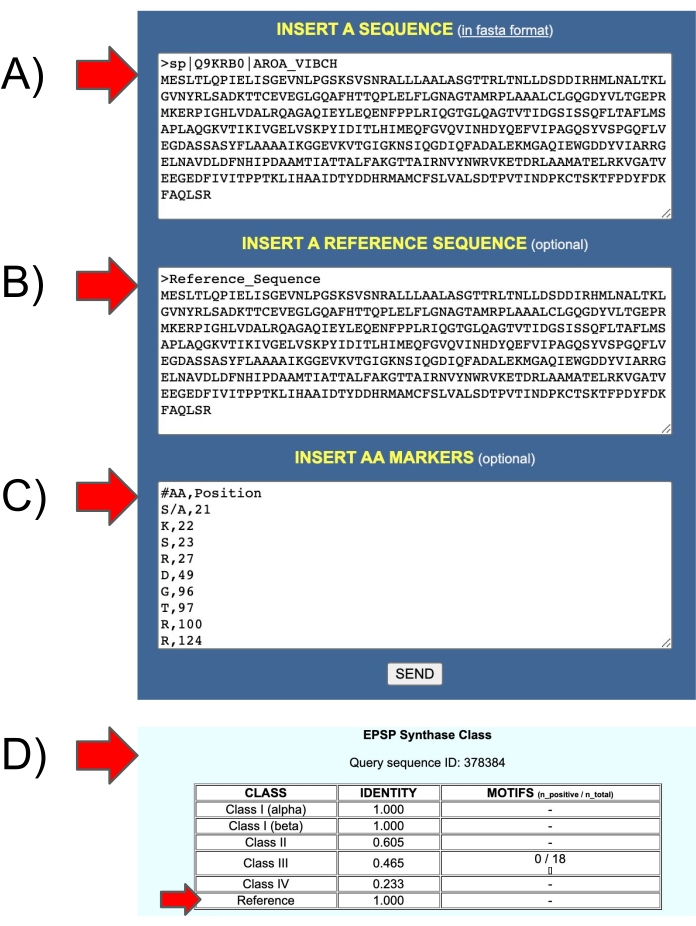
图 11:用于测试用户自己的引用序列的 EPSPSClass Web 服务器的输入和输出。 (A) 输入 1:查询序列。(B) 输入2:参考序列。(C)输入3:参考序列中的氨基酸标记物。(D)输出:同一性:查询序列中氨基酸标记物的比例(I-IV类和用户自己的参考序列)。 请点击此处查看此图的大图。
表1:微生物组分析中用于PCR扩增16S rRNA基因和ITS区的引物列表 请单击此处下载此表。
表2:不同数据库中5-烯醇丙酮基硫酸-3-磷酸合酶(EPSPS)的酶代码 请单击此处下载此表。
表3:草甘膦平均浓度 请点击这里下载此表。
表4:对草甘膦敏感/抗性的物种百分比总表。 此表已获得Rainio等人的许可进行了改编。请按此下载此表格。
表5:氨基酸标记物在参考序列中的位置 请点击此处下载此表。
讨论
该协议为如何根据EPSPS蛋白的分析量化GBP对微生物组的影响提供了一般指导。该协议有三个主要的关键步骤:(i)从微生物组数据中定量EPSPS蛋白。这一步至关重要,因为EPSPS是除草剂的直接靶酶。因此,具有 EPSPS 基因拷贝的物种可能会受到使用GBP的影响。然而,即使缺乏 EPSPS 基因拷贝的物种也可能通过替代的非靶机制受到除草剂的影响43,44。(ii)如果 EPSPS 基因的分析不包括在研究的设计中,则可以通过分析16S rRNA(细菌)或 ITS (真菌)来获得良好的估计。在这种情况下,必须依靠全面的参考表(例如,ATGC数据库提供了来自几个密切相关物种的EPSPS蛋白序列)。(iii)EPSPS蛋白根据EPSPS活性位点的某些氨基酸残基分为对草甘膦的潜在敏感性或抗性。然而,影响单个氨基酸的突变可能改变这种分类45 ,并且类之间的转变可能发生在相对较短的时间内14。
生物体对草甘膦的潜在敏感性可以通过参考基因组,氨基酸标记和序列比对来确定。(一) 参考基因组:EPSPS酶可根据氨基酸标记物和基序的存在(在III类的情况下)被分类为对草甘膦具有潜在敏感性(I类[α或β]46,47)或耐药性(II类48,49,III50和IV51)。这些氨基酸标志物和基序基于霍乱弧菌(vcEPSPS,I类)、贝氏柯克斯体(cbEPSPS,II类)、水泡短孢子虫(bvEPSPS,III类)和达瓦氏链霉菌(sdEPSPS,IV类)的EPSPS蛋白中氨基酸残基的位置。(ii)氨基酸标志物:草甘膦与EPSPS酶相互作用并与磷酸烯醇丙酮酸盐(PEP,EPSPS酶的第二底物)竞争52,53。在某些物种中,EPSPS序列中的小氨基酸变化为PEP提供了更高的亲和力和对草甘膦12,14,52,54,55的抗性。在其它序列中,草甘膦在非抑制性构象45中结合EPSPS序列。虽然已经描述了许多抗草甘膦的12,14,48,49,52,54,55和耐受性56,57 EPSP序列,但目前EPSPS的分类系统分为四大类(I-IV)12(表5).(iii)序列比对:为了对EPSPS酶进行分类,我们对每个参考序列(vcEPSPS,cbEPSPS,bvEPSPS和sdEPSPS)的查询序列进行了成对比对,使用多个序列比对程序默认参数35-进行。这些比对对于识别氨基酸标记物在查询序列中的位置是必要的。结果,根据氨基酸标记物的存在和基于III类基序标记物的存在,将酶分类为所述的12类I,II和/或IV。
该协议基于四种已知类型的EPSPS:一种类型是敏感的,另外三种是抵抗的)。然而,原核生物中大约10%的EPSPS序列尚未分类(古菌中为16%,细菌中为8%)12。因此,进一步的研究应分析这些序列以确定草甘膦的敏感性。EPSPSClass 服务器提供了一个测试新遗传标记的选项。EPSPS 的已知类别的标识非常简单,如第 4.4 节所示。和 图 5。此外,在那些用户想要比较自己的查询和参考蛋白的情况下,服务器提供了一个选项,以手动包括一个参考序列和一组氨基酸标记物(图11)。该选项可用于鉴定EPSPS的新类别,以及测试其他除草剂和靶序列。
EPSPS类的分析通过序列分析和氨基酸标志物的存在/不存在来确定。这是一个初步估计,可用于现场的假设检验。氨基酸标志物已在文献中基于经验和观察性研究46,47,48,49,50,51确定。然而,用于确定EPSPS类别的参考蛋白序列仅在有限数量的物种中进行了测试,并且可能偶尔无法解释对草甘膦的耐药性。代偿性突变和EPSPS相关结构域(主要在真菌中)的影响也可能影响对草甘膦58的敏感性。本文的分析基于四个EPSPS类。对人类肠道微生物组中细菌的调查显示,其中约30%是未分类的(即,来自这些物种的EPSPS蛋白不属于任何已知类别),需要进一步的研究来鉴定其他EPSPS类别。此外,应该注意的是,细菌和植物中的EPSPS蛋白序列是unidomain,而真菌EPSPS蛋白含有几个结构域59。因此,真菌中的蛋白质折叠可能导致EPSPS酶对草甘膦的不同反应。此外,不考虑其他非靶机制的抗性(例如,外排泵和EPSPS基因13的过表达)或对草甘膦的敏感性(例如,草甘膦对线粒体转运链12的影响)。
尽管GBPs自1974年以来一直作为除草剂存在,并且自1991年以来一直被广泛使用,但这是第一种确定生物体对草甘膦潜在敏感性的生物信息学方法。该方法基于靶序列中已知氨基酸残基的鉴定。因此,我们的方法提供了草甘膦对物种潜在影响的基线估计。在不久的将来,新的生物信息学方法应包括EPSPS蛋白的其他类别,以确定未分类序列对草甘膦的潜在敏感性12,54,55。此外,鉴于EPSPS酶的确切行为可能因单个氨基酸变化12,14,52,54,55而变化,进一步在计算机实验中应考虑EPSPS蛋白折叠的微小变化,以及EPSPS相关结构域对真菌中蛋白质结构的影响58.此外,已经表明对草甘膦的耐受性可能是通过EPSPS蛋白56,57的过表达产生的;因此,基于改进密码子使用60的生物信息学分析可用于鉴定最大化或最小化基因表达的新型EPSPS序列。
农民、政治家和决策者迫切需要彻底了解与大量使用杀虫剂相关的风险。因此,既有必要使用生物信息学工具揭示生物体对杀虫剂的潜在敏感性,也需要在不同环境中进行复制良好,随机化和现场现实的实验研究。所提出的旨在检查生物体对草甘膦敏感性的生物信息学方法可以调节其他杀虫剂。同样,实验生态学的方法可以应用于研究任何相关的生态问题。总之,这些方法可用于证明现场观察,基因组数据和农药使用之间的伤亡情况。所有介绍的方法在风险评估中都是无价的。例如,生物信息学方法可用于监测微生物对农用化学品的适应性,并提供一种定量方法来测试潜在的其他相关风险,例如病原体对农用化学品的耐药性增加,对综合虫害管理(IPM)中用作生物控制剂的微生物的负面影响,以及细菌中的抗生素耐药性。
披露声明
利益冲突:无。
致谢
这项工作由芬兰科学院资助(授予Marjo Helander的311077号)。
材料
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer Instrument | Agilent | G2939B | To check the concentration and quality of PCR products |
| dNTP mix (10 mM each) | ThermoFisher Scientific | R0192 | For PCR reactions |
| GoTaq G2 DNA Polymerase kit | Promega | M7848 | PCR buffer and DNA Polymerase for PCR amplification |
| Invisorb Spin Plant Mini Kit | INVITEK Molecular | 1037100300 | Genomic DNA extraction from plant tissues |
| Ion Chip Minifuge | ThermoFisher Scientific | 4479672 | For targeted sequencing of microbial PCR products |
| Ion PGM System | ThermoFisher Scientific | 4462921 | For targeted sequencing of microbial PCR products |
| Ion PGM Torrent Server | ThermoFisher Scientific | 4483643 | For targeted sequencing of microbial PCR products |
| Pippinprep | SageScience | PIP0001 | For size fractionation of PCR amplicons |
| Pressure tank | Berthoud | 102140 | For sprayin glyphosate based products in field |
| Primers | Sigma Aldrich | Custom-made | For PCR amplification |
| Rotary tiller | Grillo | 984511 | For tilling the soil in experimental plots |
| S1000 ThermalCycler | BIO-RAD | 1852196 | For PCR amplification |
参考文献
- Williams, G. M., Kroes, R., Munro, I. C. Safety evaluation and risk assessment of the herbicide Roundup and its active ingredient, glyphosate, for humans. Regulatory Toxicology and Pharmacology. 31, 117-165 (2000).
- Muola, A., et al. Risk in the circular food economy: Glyphosate-based herbicide residues in manure fertilizers decrease crop yield. The Science of the Total Environment. 750, 141422 (2021).
- Helander, M., Saloniemi, I., Saikkonen, K. Glyphosate in northern ecosystems. Trends in Plant Science. 17 (10), 569-574 (2012).
- Fuchs, B., Saikkonen, K., Helander, M. Glyphosate-Modulated Biosynthesis Driving Plant Defense and Species Interactions. Trends in Plant Science. 26 (4), 312-323 (2021).
- Helander, M., Saloniemi, I., Omacini, M., Druille, M., Salminen, J. -. P., Saikkonen, K. Glyphosate decreases mycorrhizal colonization and affects plant-soil feedback. The Science of the Total Environment. 642, 285-291 (2018).
- Bai, S. H., Ogbourne, S. M. Glyphosate: environmental contamination, toxicity and potential risks to human health via food contamination. Environmental Science and Pollution Research International. 23 (19), 18988-19001 (2016).
- Cuhra, M., Bøhn, T., Cuhra, P. Glyphosate: too much of a good thing. Frontiers in Environmental Science. 4, (2016).
- de Brito Rodrigues, L., et al. Impact of the glyphosate-based commercial herbicide, its components and its metabolite AMPA on non-target aquatic organisms. Mutation Research. 842, 94-101 (2019).
- Steinrücken, H. C., Amrhein, N. The herbicide glyphosate is a potent inhibitor of 5-enolpyruvylshikimic acid-3-phosphate synthase. Biochemical and Biophysical Research Communications. 94 (4), 1207-1212 (1980).
- Bentley, R. The shikimate pathway--a metabolic tree with many branches. Critical Reviews in Biochemistry and Molecular Biology. 25 (5), 307-384 (1990).
- Richards, T. A., et al. Evolutionary origins of the eukaryotic shikimate pathway: gene fusions, horizontal gene transfer, and endosymbiotic replacements. Eukaryotic Cell. 5 (9), 1517-1531 (2006).
- Leino, L., et al. Classification of the glyphosate target enzyme (5-enolpyruvylshikimate-3-phosphate synthase) for assessing sensitivity of organisms to the herbicide. Journal Of Hazardous Materials. 408, 124556 (2021).
- Rainio, M. J., Ruuskanen, S., Helander, M., Saikkonen, K., Saloniemi, I., Puigbò, P. Adaptation of bacteria to glyphosate: a microevolutionary perspective of the enzyme 5-enolpyruvylshikimate-3-phosphate (EPSP) synthase. BioRxiv. , (2020).
- Rainio, M. J., Ruuskanen, S., Helander, M., Saikkonen, K., Saloniemi, I., Puigbò, P. Adaptation of bacteria to glyphosate: a microevolutionary perspective of the enzyme 5-enolpyruvylshikimate-3-phosphate synthase. Environmental Microbiology Reports. 13 (3), 309-316 (2021).
- Helander, M., Pauna, A., Saikkonen, K., Saloniemi, I. Glyphosate residues in soil affect crop plant germination and growth. Scientific Reports. 9 (1), 19653 (2019).
- Ruuskanen, S., Rainio, M. J., Uusitalo, M., Saikkonen, K., Helander, M. Effects of parental exposure to glyphosate-based herbicides on embryonic development and oxidative status: a long-term experiment in a bird model. Scientific Reports. 10 (1), 6349 (2020).
- Ruuskanen, S., et al. female preference and adverse developmental effects of glyphosate-based herbicides on ecologically relevant traits in Japanese quails. Environmental Science & Technology. 54 (2), 1128-1135 (2020).
- Mäki, A., Rissanen, A. J., Tiirola, M. A practical method for barcoding and size-trimming PCR templates for amplicon sequencing. Biotechniques. 60 (2), 88-90 (2016).
- Chelius, M. K., Triplett, E. W. The diversity of archaea and bacteria in association with the roots of Zea mays L. Microbial Ecology. 41 (3), 252-263 (2001).
- Lane, D. J. 16S/23S rRNA sequencing. Nucleic acid techniques in bacterial systematics. , 115-175 (1991).
- Ghyselinck, J., Pfeiffer, S., Heylen, K., Sessitsch, A., De Vos, P. The effect of primer choice and short read sequences on the outcome of 16S rRNA gene based diversity studies. Plos One. 8 (8), 71360 (2013).
- Zheng, D., Alm, E. W., Stahl, D. A., Raskin, L. Characterization of universal small-subunit rRNA hybridization probes for quantitative molecular microbial ecology studies. Applied and Environmental Microbiology. 62 (12), 4504-4513 (1996).
- . JoVE Supplementary Material Available from: https://ppuigbo.me/programs/EPSPSClass/JOVE_SM (2021)
- . Alignments Conserved Positions Available from: https://ppuigbo.me/programs/primers (2021)
- . NCBI GenBank Available from: https://www.ncbi.nim.nih.gov/genbank (2021)
- . COG Database Available from: https://www.ncbi.nim.nih.gov/research/cog (2021)
- . Protein Data Bank Available from: https://www.rcsb.org (2021)
- . EPSPSClass Available from: https://ppuigbo.me/programs/EPSPSClass (2021)
- Kristensen, D. M., Wolf, Y. I., Koonin, E. V. ATGC database and ATGC-COGs: an updated resource for micro- and macro-evolutionary studies of prokaryotic genomes and protein family annotation. Nucleic Acids Research. 45, 210-218 (2017).
- . CSC - IT CENTER FOR SCIENCE LTD Available from: https://www.csc.fi (2021)
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Tkacz, A., Hortala, M., Poole, P. S. Absolute quantitation of microbiota abundance in environmental samples. Microbiome. 6 (1), 110 (2018).
- El-Gebali, S., et al. The Pfam protein families database in 2019. Nucleic Acids Research. 47, 427-432 (2019).
- Benson, D. A., et al. GenBank. Nucleic Acids Research. 41, 36-42 (2013).
- Galperin, M. Y., Makarova, K. S., Wolf, Y. I., Koonin, E. V. Expanded microbial genome coverage and improved protein family annotation in the COG database. Nucleic Acids Research. 43, 261-269 (2015).
- Burley, S. K., Berman, H. M., Kleywegt, G. J., Markley, J. L., Nakamura, H., Velankar, S. Protein data bank (PDB): the single global macromolecular structure archive. Methods in Molecular Biology. 1607, 627-641 (2017).
- Raoult, D., Hadjadj, L., Baron, S. A., Rolain, J. -. M. Role of glyphosate in the emergence of antimicrobial resistance in bacteria. The Journal of Antimicrobial Chemotherapy. 76 (7), 1655-1657 (2021).
- Liu, J., Gefen, O., Ronin, I., Bar-Meir, M., Balaban, N. Q. Effect of tolerance on the evolution of antibiotic resistance under drug combinations. Science. 367 (6474), 200-204 (2020).
- Peixoto, F. Comparative effects of the Roundup and glyphosate on mitochondrial oxidative phosphorylation. Chemosphere. 61 (8), 1115-1122 (2005).
- Peillex, C., Pelletier, M. The impact and toxicity of glyphosate and glyphosate-based herbicides on health and immunity. Journal of Immunotoxicology. 17 (1), 163-174 (2020).
- Funke, T., Han, H., Healy-Fried, M. L., Fischer, M., Schönbrunn, E. Molecular basis for the herbicide resistance of Roundup Ready crops. Proceedings of the National Academy of Sciences of the United States of America. 103 (35), 13010-13015 (2006).
- Light, S. H., Krishna, S. N., Minasov, G., Anderson, W. F. An unusual cation-binding site and distinct domain-domain interactions distinguish Class II Enolpyruvylshikimate-3-phosphate synthases. Biochemistry. 55 (8), 1239-1245 (2016).
- Firdous, S., Iqbal, S., Anwar, S., Jabeen, H. Identification and analysis of 5-enolpyruvylshikimate-3-phosphate synthase (EPSPS) gene from glyphosate-resistant Ochrobactrum intermedium Sq20. Pest Management Science. 74 (5), 1184-1196 (2018).
- Barry, G. F., Kishore, G. M., Padgette, S. R., Stallings, W. C. Glyphosate-tolerant 5-enolpyruvylshikimate-3-phosphate synthases. United States Patient. , (1997).
- Priestman, M. A., Funke, T., Singh, I. M., Crupper, S. S., Schönbrunn, E. 5-Enolpyruvylshikimate-3-phosphate synthase from Staphylococcus aureus is insensitive to glyphosate. FEBS Letters. 579, 728-732 (2005).
- Carozzi, N., Carr, B., Hammer, P. E. Identification of a new class of EPSP synthases. World Intellectual Property Organization Publ.of the Int.Appl. without Int.search. , (2006).
- Lira, J. M., Cicchillo, R. M., Nair, S. K. Novel class of glyphosate resistance genes. US Patent. , (2013).
- Funke, T., et al. Structural basis of glyphosate resistance resulting from the double mutation Thr97 -> Ile and Pro101 -> Ser in 5-enolpyruvylshikimate-3-phosphate synthase from Escherichia coli. The Journal of Biological Chemistry. 284 (15), 9854-9860 (2009).
- Schönbrunn, E., et al. Interaction of the herbicide glyphosate with its target enzyme 5-enolpyruvylshikimate 3-phosphate synthase in atomic detail. Proceedings of the National Academy of Sciences of the United States of America. 98 (4), 1376-1380 (2001).
- Stalker, D. M., Hiatt, W. R., Comai, L. A single amino acid substitution in the enzyme 5-enolpyruvylshikimate-3-phosphate synthase confers resistance to the herbicide glyphosate. The Journal of Biological Chemistry. 260 (8), 4724-4728 (1985).
- Eschenburg, S., Healy, M. L., Priestman, M. A., Lushington, G. H., Schönbrunn, E. How the mutation glycine96 to alanine confers glyphosate insensitivity to 5-enolpyruvyl shikimate-3-phosphate synthase from Escherichia coli. Planta. 216 (1), 129-135 (2002).
- Achary, V. M. M., et al. Overexpression of improved EPSPS gene results in field level glyphosate tolerance and higher grain yield in rice. Plant Biotechnology Journal. 18 (12), 2504-2519 (2020).
- Huang, Z., Liu, Y., Zhang, C., Jiang, C., Huang, H., Wei, S. Molecular basis of natural tolerance to glyphosate in Convolvulus arvensis. Scientific Reports. 9 (1), 8133 (2019).
- Tall, T. . A census analysis of the 5-enolpyruvylshikimate-3-phosphate (EPSP) synthase and EPSP-associated domains. , (2020).
- Tall, T., Puigbò, P. The glyphosate target enzyme 5-Enolpyruvyl Shikimate 3-Phosphate Synthase (EPSPS) contains several EPSPS-associated domains in fungi. ATLA Summary of Proceedings. 76 (1), 6 (2020).
- Puigbò, P., Guzmán, E., Romeu, A., Garcia-Vallvé, S. OPTIMIZER: a web server for optimizing the codon usage of DNA sequences. Nucleic Acids Research. 35, 126-131 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。