Method Article
グリホサートベースの製品がマイクロバイオームに及ぼす潜在的な影響の定量化
要約
グリホサートベースの製品(GBP)は、世界中で最も一般的な広域スペクトル除草剤です。本稿では、GBPがマイクロバイオームに与える影響を定量化するための一般的なガイドラインを、フィールド実験からバイオインフォマティクス解析まで紹介します。
要約
グリホサートベースの製品(GBP)は、世界中で最も一般的な広域スペクトル除草剤です。グリホサートの標的は、シキミ酸経路における酵素5-エノールピルビルシキミ酸-3-リン酸合成酵素(EPSPS)であり、これは植物において事実上普遍的である。酵素の阻害は、3つの必須アミノ酸の産生を停止します: フェニルアラニン, チロシン, トリプトファン.EPSPSは、古細菌や細菌などの真菌や原核生物にも存在します。したがって、GBPの使用は、土壌、植物、草食動物、および二次消費者のマイクロバイオーム組成に影響を与える可能性がある。この記事では、フィールド実験からバイオインフォマティクス分析までのマイクロバイオームに対するGBPの効果を評価するための一般的なガイドラインを提示し、いくつかの検証可能な仮説を提供することを目的としています。非標的生物でGBPを試験するために、2つの野外実験が提示される。まず、10の複製対照およびGBP処理プロットからの植物関連微生物をサンプリングし、無作作法をシミュレートし、分析する。第2の実験では、グリホサート残渣または未処理の対照肥料を含む家禽糞尿のいずれかによって受精された実験区画からサンプルが得られた。EPSPSタンパク質配列のバイオインフォマティクス解析は、グリホサートに対する微生物の潜在的な感受性を決定するために利用される。マイクロバイオームに対するGBPの効果を推定する最初のステップは、標的酵素(EPSPS)に対するGBPの潜在的な感受性を決定することです。微生物配列は、公共のリポジトリから、またはPCR増幅によって得ることができる。しかし、フィールド研究の大部分では、マイクロバイオーム組成は、16S rRNAや内部転写スペーサー(ITS)などの普遍的なDNAマーカーに基づいて決定されています。このような場合、グリホサートに対する感受性は、近縁種を用いたEPSPS配列の確率的分析によってのみ推定することができる。EPSPS酵素に基づくグリホサートに対する生物の潜在的な感受性の定量化は、標的および非標的耐性機構を研究するためのさらなる実験のための堅牢なアプローチを提供する。
概要
現代農業における農薬の大量使用は、明らかに生物多様性の低下の主な原因です1。この論文は、グリホサートベースの製品(GBP)がその効率と手頃な価格のために世界で最も広く使用されている農薬になっているため、グリホサートに焦点を当てています2,3。農業分野で雑草を殺すことに加えて、GBPは造林、都市環境、家庭菜園で一般的に使用されています。さらに、それらは製造業者の指示に従って使用された場合、非標的生物に対して無毒であると宣言されている。しかしながら、近年の研究の増加により、グリホサートおよびその分解産物の残留物が土壌中に保持および輸送され、それによって非標的生物にカスケード効果を有することが明らかになっている4、5、6、7、8。グリホサートの効果は植物だけに限定されず、シキミ酸経路は多くの真菌および原核生物にも存在する。グリホサートは、aroA9としても知られるシキミ酸経路における酵素5-エノールピルビルシキミ酸-3-リン酸合成酵素(EPSPS)を標的とする。この酵素は、3つの必須芳香族アミノ酸(フェニルアラニン、チロシン、およびトリプトファン)の合成におけるシキミ酸経路の中心にあり、ほとんどの原核生物、植物、および真菌に存在する10,11。いくつかの微生物種は、EPSPS配列の変異を含むいくつかのメカニズムによってグリホサートに対する部分的または絶対的な耐性を発達させている。したがって、GBPの使用は、ヒト腸内微生物叢を含む植物および動物の微生物叢に直接影響を及ぼし得ることが示唆されている12、13、14。それにもかかわらず、GBPの使用は、微生物および微生物促進プロセスに依存する事実上すべての生態系機能およびサービスに悪影響を及ぼす可能性がある。結果として生じる脅威は、生化学的土壌プロセス、受粉生物学、および動物および人間の幸福に関係している可能性があります。これは、グリホサートがシキミ酸経路およびグリホサートに対する微生物の感受性を評価する方法にどのように影響するかについてのより包括的な理解を必要とする。
このプロトコルでは、フィールド実験からバイオインフォマティクス分析まで、グリホサートとGBPがマイクロバイオームに及ぼす影響をテストするパイプラインを提示します。我々は、グリホサート12に対する生物の潜在的な感受性を決定するために使用することができる最近発表されたバイオインフォマティクス方法を詳細に説明する。研究者の知る限り、これはGBPの活性成分に対する酵素EPSPSの固有の感受性を評価するための最初でこれまでのところ唯一のバイオインフォマティクスツールです。このバイオインフォマティクス法は、グリホサート標的酵素(EPSPS)12中の既知のアミノ酸マーカーの検出に基づいている。パイプラインは、1)GBPの効果をテストするための2つのフィールド実験の簡単な紹介、2)マイクロバイオーム分析(16S rRNA、ITS、およびEPSPS遺伝子)の簡単な要約、3)公共リポジトリからのEPSPS配列の収集、4)グリホサートに対する生物の潜在的な感受性の決定、および5)普遍的な微生物マーカー(16S rRNAおよびITS)からのEPSPSクラスの評価の5つの主要な作業段階(図1)に分かれています。
プロトコル
1. GBPsの効果を試験するための2つの野外実験
注:このプロトコルは、植物関連微生物に対するGBPの効果をテストするためのフィールド実験計画の2つの例を示しています。どちらの実験も、フィンランドのトゥルク・ルイサロ植物園(北緯60度26分、東経22度10分)の除草剤や農業利用の歴史のない畑で実施されました。土壌は有機物の割合が高い砂質粘土です。
- 実験1
注:この実験は、雑草と戦うために成長期の前後にGBPを適用したノーティル農業の一般的な農業慣行をシミュレートするように設計されています。- 実験フィールドを10の複製対照およびGBP処理プロット(23 m x 1.5 m)に分割し、プロット間に植生のバッファーストリップ(2014年春のこの研究では15)(図2)。
- プロットが回転式耕運機で5cmの深さまで耕され、年に2回処理されていることを確認してください。ここでは、プロットは成長期の初め(5月)と終わり(10月)に処理されました。
- 対照プロットを水道水(5 L/プロット)で処理し、GBPプロットを市販のGBP(グリホサート濃度450 g·L−1、プロット当たり5Lの水道水における施用量6.4L・ha−1)を、農業慣行における最大許容グリホサート投与量(3kg・ha−1)を模倣する。
- 手動噴霧器を備えた手動圧力タンクで治療を適用します。GBP適用の2週間後、農業慣行に従って、オート麦(Avena sativa)、ファバ豆(Vicia faba)、カブレイプ(Brassica rapa subsp. oleifera)、および植物ジャガイモ(Solanum tuberosum)をプロットに播種する。
- 成長期には、対照区とGBP処理区で植物の競合と土壌構造をできるだけ類似させるために、区画を手で除草します。
- 実験植物から微生物叢をサンプリングする。この研究では、微生物叢は、2017年から2020年にかけて、GBP処理プロットと対照処理プロットの両方で、研究の過程で成長期に1回連続してサンプリングされました。
- 畑から植物サンプル(根と葉)の複製物を10個集め、すぐに氷の上に置き、セクション2.1で説明されているように、さらなる処理のために実験室に持って行きます。
- 実験2
注:この実験は、循環型食品経済に関連するリスクをテストするために設計されました。より正確には、作物植物2 に肥料として施用された肥料中のGBP残留物の影響を調べるように設計されました(図3)。- 12ヶ月の鳥小屋実験で、GBP汚染または対照飼料を与えられたウズラから、木材の削りくず、糞便、およびこぼれた飼料を含む寝具を採取する16,17。
注:GBP汚染飼料は、ニワトリを産むための有機飼料と160mgのグリホサート/ kgに相当するもので構成されており、これは成体の日本のウズラの体重1kgあたり12〜20mgのグリホサート1日摂取量に相当します16,17。 - 検証のために、サンプルを認定ラボに送って、6バッチの飼料でグリホサート濃度を測定します。
- さらに、曝露の12ヶ月後にウズラ排泄物サンプル中のグリホサート残基を測定する。対照群には、GBP添加なしの同じ有機飼料16、17を供給した。
- 鳥小屋の実験中は、寝具を隔週で交換してください。使用済みの寝具をGBP処理および対照から8〜12ヶ月の暴露から定期的に収集し、処理ごとにプールし、肥料として使用する前に6°Cの乾燥した暗い貯蔵室に密閉容器に保管する。
- 実験フィールドの6 x 6チェス盤グリッドの18 GBPと18のコントロールプロット(サイズ1 m x 1 m)のそれぞれに、2つの時点で12 Lの寝具を手動で広げます。この研究では、寝具は2018年8月と2019年5月に広がりました。
- 寝具サンプルは、グリホサート濃度測定が普及した直後に認定ラボに送付してください(2019年5月のこの研究では)。
- 各プロットに多年生草とイチゴの植物を植えて、それらの根と葉の微生物叢を研究します。
注:この研究では、4つの多年生草植物(Festuca pratensis)と2つのイチゴ植物(Fragaria x vescana)を各区画に植え、それらの根と葉の微生物叢について研究した。
- 12ヶ月の鳥小屋実験で、GBP汚染または対照飼料を与えられたウズラから、木材の削りくず、糞便、およびこぼれた飼料を含む寝具を採取する16,17。
2. マイクロバイオーム解析(16S rRNA、 ITS および EPSPS 遺伝子)
注:マイクロバイオーム研究のほとんどは、次世代シーケンシング技術を使用して、真菌群集の細菌および内部転写スペーサー(ITS)領域の16S rRNA遺伝子の解析に基づいています。したがって、この論文にはEPSPSの種類に関する情報がありません。数千種のEPSPS配列は、パブリックリポジトリ(プロトコルセクション3)で入手可能です(図4)。
- 16S rRNA遺伝子
- 上述の実験で採取した剥離した葉根サンプルから、内生微生物(すなわち、植物組織の内部に生息する微生物)を同定する。
- 植物サンプルを水道水で洗浄し、それらを滅菌して着生微生物(すなわち、植物組織の表面上の微生物)を除去する。3%漂白剤を用いて3分間殺菌し、続いて70%エタノール溶液を1分間、オートクレーブ処理した超純水でそれぞれ1分間3回洗浄した。
- ゲノムDNA抽出まで-80°Cでサンプルを凍結する。
- メーカーのプロトコールに従って市販の植物DNA抽出キットを用いてゲノムDNA抽出を行う。
- 細菌DNAに特異的に結合する識別プライマーを用いたネストアプローチを用いて抽出されたDNAサンプルから16S rRNA遺伝子の可変領域V6-V8を標的とし18、宿主植物DNAからの増幅を最小化する。
- ポリメラーゼ連鎖反応(PCR)を3回行った後、標的遺伝子にバーコード配列とアダプター配列をタグ付けし、シーケンシングのテンプレートとして準備します。PCR増幅のためのステップ2.1.7-2.1.11に従ってください
- 各反応の総容量が 30 μL、30 ng の DNA、1x PCR バッファー、0.2 mM dNTP、各プライマーの 0.3 μM、および 2000 U/mL DNA ポリメラーゼで構成されるように、必要なサンプル数の PCR マスターミックスを準備します。PCR の第 2 ラウンドと第 3 ラウンドで同じ手順に従います。
- PCRの第1ラウンドのために、プライマー799F19 および1492R(20から改変)を使用する(表1)。サーモサイクラーに増幅プロファイルを設定します(95°Cで3分間の初期変性、続いて95°Cで45秒間の変性を35サイクル、54°Cで45秒間アニーリング、72°Cで1分間伸長)。最終伸長を72°Cで5分間行う。
- 第 2 ラウンドの PCR のテンプレートとして、電気泳動 (1.5% アガロースゲル上の PCR 産物の 5 μL) によって増幅を確認し、PCR 産物の残り 25 μL をオートクレーブ処理された超純水で 1:10 の比率で希釈します。
- 希釈したPCRテンプレートをuni-1062F21およびuni-1390R22のプライマーを用いてPCRを繰り返す(表1)。サイクル数を 25 に減らす以外は、同じ PCR 反応条件と増幅プロファイルを維持します (ステップ 2.1.8)。
- 得られたPCR産物をオートクレーブ処理した超純水で1:1の比率で希釈する。3 回目の PCR ラウンドを実行して、ステップ 2.1.8 で説明したのと同じ PCR プロファイルの 8 サイクルでバーコードと P1-アダプター配列で製品にタグを付けます。
- ライブラリの準備
- バイオアナライザーでPCR産物の濃度と品質を検証し、各サンプルの30 ngのDNAを構成する容量を1.5 mLチューブにプールして等モルライブラリーを作製します。
- アガロースゲルカセット上の自動DNAサイズ選択システムを用いたサイズ分画によって、サイズ350〜550bpのアンプリコンを選択する。これはまた、ライブラリーから非特異的アンプリコンおよびPCR試薬を排除することに留意されたい。指定されたサイズのアンプリコンからなる溶出液をカセット内のバイアルに集め、精製された16S rRNA遺伝子ライブラリーを得た。
- 溶出液を1.5mLチューブにピペットで出し、バイオアナライザーで純度と濃度を検証した。オートクレーブ処理した超純水を用いてDNAライブラリーを終濃度26pMに希釈する;サンプルはシーケンシングの準備ができています。
- その
注:ITS特異的プライマー(表1)を用いてITS領域を増幅し、得られたPCR産物をバーコードおよびシーケンシング用のP1アダプター配列で標識する。- セクション 2.1 で説明したのと同じプロトコルに従って PCR マスターミックス を ITS プライマーと共に調製します。
- サーモサイクラー上の増幅プロファイルを95°Cで5分間の初期変性として設定し、その後、95°Cで30秒間、55°Cで30秒間、55°Cで30秒間、72°Cで1分間、最終伸長を72°Cで7分間、35サイクルの変性、アニーリング、および伸長の35サイクルを設定します。
- 1.5 % アガロースゲル上の 5 μL の PCR 産物を分析し、オートクレーブ処理された超純水を使用して残りの 25 μL を 1:10 に希釈します。希釈したPCR産物を第2ラウンドのPCRのテンプレートとして使用します。
- バーコードタグ付きフォワードプライマーおよびP1アダプタータグ付きリバースプライマーを使用して、必要なサンプル数のPCRマスターミックス(ステップ2.1.6を参照)を調製します。8サイクルを使用する以外は、ステップ2.3.2と同じ増幅プロファイルで増幅します。
- 得られたPCR産物を、セクション2.2に記載されているプロトコルに従ってシーケンシング用に準備します。
- EPSPS遺伝子
- 微生物の EPSPS 遺伝子の配列決定と解析を行います。
注:GBP曝露がコミュニティにおけるグリホサート感受性および耐性微生物の組成を変化させたかどうかを調べるためには、微生物の EPSPS 遺伝子を配列決定および解析する必要がある。したがって、アラインブル・タイトゲノム・クラスター(ATGC)データベース内の微生物分類群の幅広いコレクションから EPSPS 遺伝子の353配列が収集され、すべてのタンパク質配列がアラインメントされた22。これらのアラインメントは、ATGCデータベース23 で利用可能であり、保存された領域からプライマーを生成するために利用され得る。使いやすいバイオインフォマティクスツールは、複数の配列アライメントから保存領域を特定するように設計されており、これはPere Puigbo研究24ページで入手できます。ただし、この Web サーバーの詳細な説明を提供することは、本書の範囲外です。それにもかかわらず、グリホサートに対するマイクロバイオームの感受性を見出すために EPSPS 遺伝子の増幅のためにこれらのプライマーを利用するための前向きなプロトコールが 図4に提供される。
- 微生物の EPSPS 遺伝子の配列決定と解析を行います。
3. パブリックリポジトリからのEPSPSタンパク質配列の収集
- 大進化研究で使用されるEPSPS配列
- PFAM23(タンパク質ファミリー25のデータベース)、GenBank24(遺伝子、ゲノム、タンパク質のデータベース26)、COG25(Clusters of Orthologous Groups27; 古細菌からのオルソログタンパク質のデータベース)などの公的リポジトリからEPSPSタンパク質を収集する。PDB26(Protein Data Bank28; a database of protein structure)。
注:研究者らが実施した最近の研究では、これらのタンパク質が、シキミ酸経路を有する生物に対するグリホサートの潜在的な効果の微進化的および比較的分析を実行するために利用できることが示された12。著者らは、ヒト腸内微生物叢からのタンパク質の手動でキュレーションされたデータセットを含む、何万ものEPSPSタンパク質配列29に関する情報を収集するユーザーフレンドリーなウェブサイトを開発しました12。これらの事前に計算されたデータセットの情報には、推定感度およびグリホサート耐性への現在のEPSPS分類、種に関する分類学的情報、EPSPS活性部位の注釈、およびPDBおよびNCBIデータベースへのリンクが含まれる。さらに、Web サーバーには EPSPS の ID コードと複数の外部データベースへのリンクが含まれています (表 2)。
- PFAM23(タンパク質ファミリー25のデータベース)、GenBank24(遺伝子、ゲノム、タンパク質のデータベース26)、COG25(Clusters of Orthologous Groups27; 古細菌からのオルソログタンパク質のデータベース)などの公的リポジトリからEPSPSタンパク質を収集する。PDB26(Protein Data Bank28; a database of protein structure)。
- ATGCの微進化研究で使用されるEPSPS配列
注:タンパク質配列の一般的なリポジトリは、比較的遠い生物間の比較研究を行うのに有用である。しかし、グリホサートの潜在的な効果は、進化的観点からは比較的最近のものである。したがって、いくつかの研究では、グリホサート14の効果を決定するために、近縁種(例えば、同じ細菌種からの異なる株)を比較する必要がある。このような場合、密接に関連した古細菌ゲノムゲノムの包括的なリストを含むAlignable Tight Genomic Clusters(ATGC)30のデータベースは、より適切なリソースです。ATGCデータベースには、数百のクラスターに編成された数千のゲノムから数百万のタンパク質の情報が含まれています30。各ゲノムクラスターは整列可能(ゲノムは長さの≥85%以上のシンテニーを共有する)であり、タイトである(飽和以下の同義置換率を有する)。研究者らは、最近の研究でATGCデータセットを使用して、EPSPSタンパク質の微進化的変化を分析した14。ATGC中のEPSPSタンパク質配列を同定するには、以下のステップが必要である:- リンク31 からATGCのデータベース全体をダウンロードし、COG0128のすべてのタンパク質(データベース内のEPSPSタンパク質に対応するコード)32 をローカルプロジェクトにダウンロードします。
注:研究者/実験者がフィンランドに拠点を置く場合、CSC-IT Center for Science33 はストレージとソフトウェア設備を提供します。すべてのシーケンスを FASTA 形式で収集することが重要です。 - 原核生物の代表的な種のセットにおけるEPSPSタンパク質のオルソログを含むCOG0128の芽球データベースを構築した。CSC にはブラスト プログラム34 がプリインストールされており、 コマンド makeblastdb -in COG0128.fa -dbtype prot を使用して EPSPS シーケンスの参照データベースを作成できます。
- コマンド blastp - query [ATGC_X.fa] -db [COG0128.fa] -max_target_seqs 1 -outfmt 6 -out tmpfile -evalue 1e-150 を使用して反復ブラスト検索を使用して、ATGC データベースを COG0128.fa (EPSPS タンパク質) にマップします。
- その結果、それぞれにEPSPSタンパク質配列のデータセットが作成されます。ATGCデータベースから密接に関連したEPSPSタンパク質配列の事前計算されたデータセットが利用可能である29。
- リンク31 からATGCのデータベース全体をダウンロードし、COG0128のすべてのタンパク質(データベース内のEPSPSタンパク質に対応するコード)32 をローカルプロジェクトにダウンロードします。
4. グリホサートに対する生物の潜在的な感受性を決定するアルゴリズム(EPSPSClass Webサーバー:入力、処理、および出力)
注:研究者らは、EPSPSタンパク質配列12,35のクラスを決定するために、29で自由に利用できる使いやすいサーバーを実装しました。サーバーは、各EPSPSクラスに対する同一性パーセンテージおよびグリホサートに対するそれらの潜在的な感受性を決定するために、FASTAフォーマットのタンパク質配列の入力のみを必要とする。さらに、ユーザーはWebサーバーを利用して、独自の参照配列とアミノ酸マーカーをテストすることができます。まず、アルゴリズム(図5)は、アミノ酸位置を決定するために多重配列アラインメントプログラム35を用いてクエリ配列および参照配列をアラインメントする。次に、アミノ酸マーカーの存在を検索して、クエリ配列のEPSPSクラス(I、II、III、またはIV)を同定する。
- FASTA形式のEPSPSタンパク質配列を入力テキストボックスに導入して酵素のクラスを特定し(図6A)、 送信を押します。
- サーバー提供の出力からのグリホサート(図6B-E)に対するクエリ配列の潜在的な感度を評価します。
出力1:クエリ配列中に存在するアミノ酸マーカーの割合(すなわち、同一性)(クラスI、II、およびIV)、およびモチーフの数(クラスIII)。
出力2:マーカー残基に基づくクエリ配列と参照配列のアラインメント。
出力 3: クエリと参照シーケンスの完全なペアワイズ配置。
出力4:EPSPS参照配列:コレラ 菌(vcEPSPS、クラスI)、 コクシエラ・バーネティイ(cbEPSPS、クラスII)、 ブレブンジモナス・ベシキュラリス(bvEPSPS、クラスIII)、 ストレプトマイセス・ダバウェンシス(sdEPSPS、クラスIV)。 - 出力ページの最後に、blastp や保存ドメインなどの外部ツールへのリンクを見つけて、クエリ EPSPS シーケンスをさらに分析します (図 6F)。
5. 汎用微生物マーカー(16S rRNAおよび ITS )からのEPSPSクラスの評価
注:ほとんどのマイクロバイオーム研究は、16S rRNAおよび/またはITS36の分析に基づいています。このような場合、EPSPS配列の直接解析を行うことはできない。したがって、グリホサートに対する生物の潜在的な感受性を推定するための確率的アプローチが必要である。この分析は簡単で、マイクロバイオームプロジェクトにおけるEPSPS配列のタイプの合理的な推定値を提供します。このプロセスは、次の 3 つのステップに分かれています (図 7 と 図 8)。
- パブリックリポジトリからEPSPSシーケンスを識別します。代表的な配列の包括的なデータセットのEPSPSクラスは、PFAM 37、GenBank 38、COG 39、PDB 40、ATGC 30からコンパイルされ、事前計算されています。分類情報と 50,000 を超えるシーケンスの EPSPS クラスを含む EPSPSClass サーバーのメイン ページからこれらのデータセットにアクセスします (図 7)。
- 生育期に実験植物の高さを隔週で測定し、畑期の終わりに植物の地上バイオマスを計量して、GBPプロットと対照プロットで植物の成長を比較します。
注:野外実験による微生物叢分析は、まだ完全には分析されていません。 - スプレッドシートを使用して、マイクロバイオーム実験の細菌OTU(16S rRNAまたは ITSに基づく)を事前に計算されたデータセットにマッピングします。
注:以前の研究は、EPSPSクラス(すなわち、グリホサートに対する固有の感受性)が系統発生グループ内で高度に保存されていることを示している14。したがって、高度に保存された分類群からの近縁種がグリホサートに対して同様のEPSPS応答を有する可能性があると仮定することは比較的安全である(図8)。 - 同じスプレッドシートで、公共のデータベース内の既知のEPSPS配列から計算された確率的スコア(S = s / (s + r + u)に基づいて、グリホサートに対する固有の感受性を計算します(S:感度スコア;s:潜在的に感受性のある配列の数;r:潜在的に耐性のある配列の数;u:未分類の配列の数)。
注:このスコアの範囲は、0(タクソンに感受性EPSPS配列が見つからなかった)から1(タクソン内のすべての配列がグリホサートに感受性である)です(図8)。さらに、中間の値、すなわち、感受性、耐性または未知の株を有する種が存在する。
結果
このプロトコルの目的は、除草剤グリホサートに対する生物の潜在的な感受性を定量化する、野外実験からバイオインフォマティクス分析まで、一般的なパイプラインを提供することです。実験2では、ウズラ飼料中の平均グリホサート濃度は164mg/kgであり、排泄物サンプル(尿と糞便を合わせた)の平均グリホサート濃度は199mg/kgであった。GBP汚染飼料を与えられたウズラから採取された寝具は、平均して158mg / kgを有し、0.17mg / kgのグリホサートを測定する対照寝具を有していた(表3)。野外実験では、植物種は土壌中のグリホサート残基に対して異なる反応を示した(セクション1)。オート麦およびカブの菜種のバイオマスは、GBP処理土壌と比較して対照土壌において大きかった。しかし、ファバ豆とジャガイモは、栽培シーズンの終わりにGBP処理の恩恵を受けるように見えた15。家禽糞尿中のグリホサートは、草(Festuca pratensis)およびイチゴ(Fragaria x vescana)の植物成長を減少させた(セクション1)。野外実験による微生物叢分析はまだ完全には分析されておらず、ここでは紹介していません(セクション2)。このプロトコルの結果は、直接(セクション3および4に示すように)または間接的に(セクション5に示すように)読み取られた場合、データセット内のグリホサートに対する潜在的に感受性および耐性のある生物の割合の尺度を提供する(図9)。この方法の使用を、公共リポジトリ12から得られたコアヒト腸内微生物叢の微生物種からのEPSPSタンパク質配列の集合体を用いて試験した。この研究では、最も豊富な101種の細菌種から890株をEPSPSClass法で分析し、感受性および耐性細菌の割合を定量化しました。その結果、コアヒト腸内微生物叢の種の54%がグリホサートに潜在的に感受性であることが示された12。この傾向は、原核生物の世界のほとんどでも観察されています。さらに、真核生物(主に植物および真菌)では、潜在的に敏感な種の割合はさらに高い12。さらに、この方法を利用して、EPSPSタンパク質の感受性の変化を微進化レベルで定量化しました(図10)14。我々は、分析した原核生物の32の密接に関連したグループのうち12において感受性状態の変化を同定した(表4)14。したがって、GBPsの連続使用は、植物、動物、および土壌のマイクロバイオームにおいて微生物の叶攣異常(すなわち、感受性および耐性細菌種の不均衡)を生じ得る。さらに、グリホサート耐性細菌の増加が多剤耐性マイクロバイオームを促進する可能性があるという仮説が立てられている14、41、42。したがって、このプロトコルは、EPSPS分類法がグリホサートに対するマイクロバイオームの固有の感受性の直接的な推定を提供するため、これらすべてのシナリオの解釈に光を当てる。グリホサートに対するEPSPSタンパク質の固有の感受性は系統学的に保存されているため14、既存のデータセットから未知のマイクロバイオームに結果を外挿することが可能である(図8)。
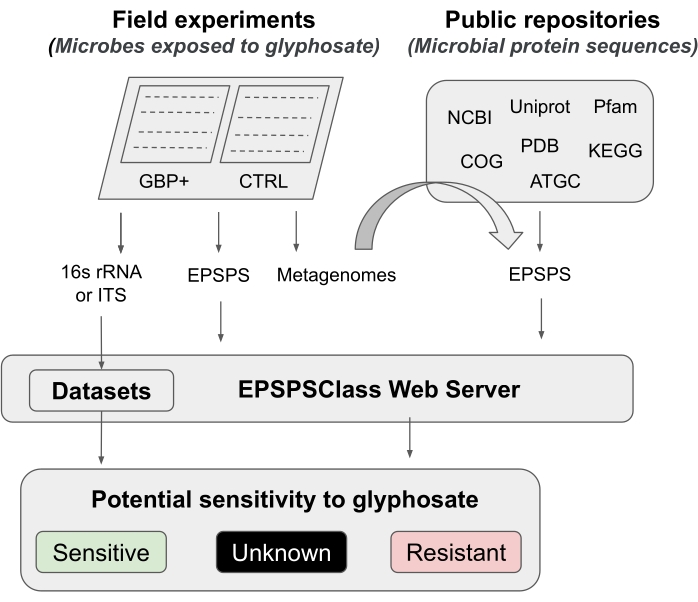
図1:一般 パイプライン 野外実験からバイオインフォマティクス解析まで、GBPに対する感度を解析するための汎用パイプラインです。 この図の拡大版を表示するには、ここをクリックしてください。
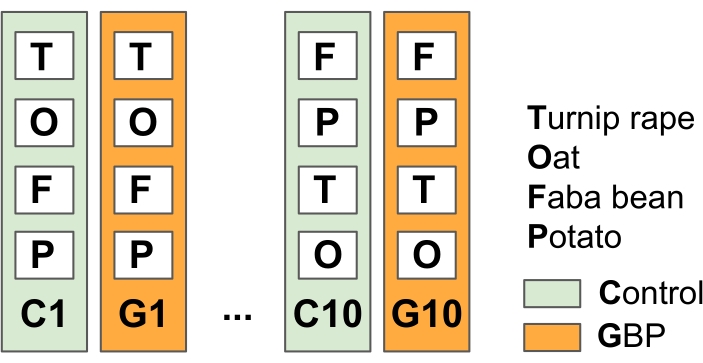
図2:作物植物関連微生物に対するGBP残留物の影響を試験するための野外実験1。 実験場は、10個の対照プロットと10個のGBP処理プロット(23 m x 1.5 m)を交互に並べ、プロット間に1.5 mのバッファストリップで構成されています。2014年から年に2回、GBPプロットは市販のGBP(グリホサート濃度450g L-1、1プロットあたり5Lの水道水中の施用量6.4L ha-1)で処理され、対照プロットはグリホサートを含まない同じ量の水道水で処理されました。処理は、GBPが処理区画の外に広がるのを防ぐために、スプリンクラー先端のプラスチックフードを使用して手動圧力タンクで適用されました。GBP施用後2週間の安全期間の後、オート麦(Avena sativa)、ファバ豆(Vicia faba)、カブレイプ(Brassica rapa subsp. oleifera)を播種し、ジャガイモ(Solanum tuberosum)をプロットに植えました。研究された作物の植物、葉および根からの微生物叢サンプルは、2014年の実験の開始以来数回収集された。 この図の拡大版を表示するには、ここをクリックしてください。
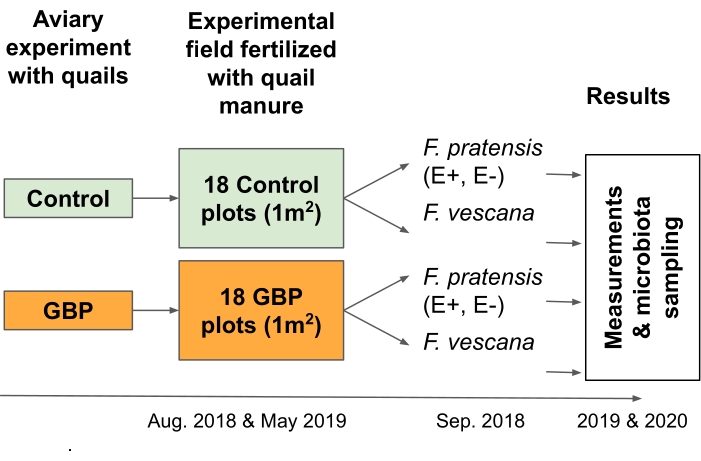
図3:野外実験2は、2つの多年生作物およびそれらに関連する微生物叢に対する肥料肥料中のGBP残留物の結果を試験した。 対照またはGBP汚染飼料を与えられた日本のウズラとの12ヶ月の鳥小屋実験から収集された寝具は、野外実験で肥料肥料として使用された。実験場は、6 x 6のチェス盤グリッドに配置された18のコントロールと18のGBPプロット(1 m x 1 m)で構成されていました。寝具は、2018年8月と2019年5月の2回(25L /プロット)実験場に広げられました。対照プロットは、対照飼料を与えられたウズラから採取された寝具で受精し、GBP汚染飼料を与えられたウズラからの寝具でGBPプロットを施肥した。対照寝具中のグリホサート残基は0.17mg / kgのグリホサートであり、GBP寝具では、その量は158mg / kgのグリホサートであった。2つの内生共生(E+)、2つの内生植物を含まない(E-) フェストゥカプラテンシス、および2つの フラガリア×ベスカナ が、最初の寝具の普及から約1ヶ月後の2018年9月にプロットごとに植えられました。植物のパフォーマンスと適応度の測定、ならびに根葉に関連する微生物叢のサンプリングは、2つの連続した成長期(2019年および2020年)に実施されました。 この図の拡大版を表示するには、ここをクリックしてください。
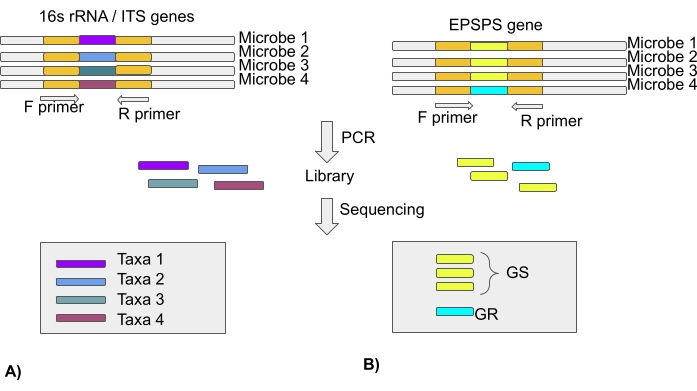
図4:16S rRNA遺伝子/ITS領域を用いた微生物分類群の解析と、EPSPS遺伝子を用いたグリホサートに対するマイクロバイオームの感受性 (A)微生物分類群を同定するための16S rRNAまたはITS配列の解析(B)グリホサートに対する微生物の感受性(GS-グリホサート感受性/GR-グリホサート耐性)を同定するためのEPSPS配列の解析この図の拡大版を表示するには、ここをクリックしてください。
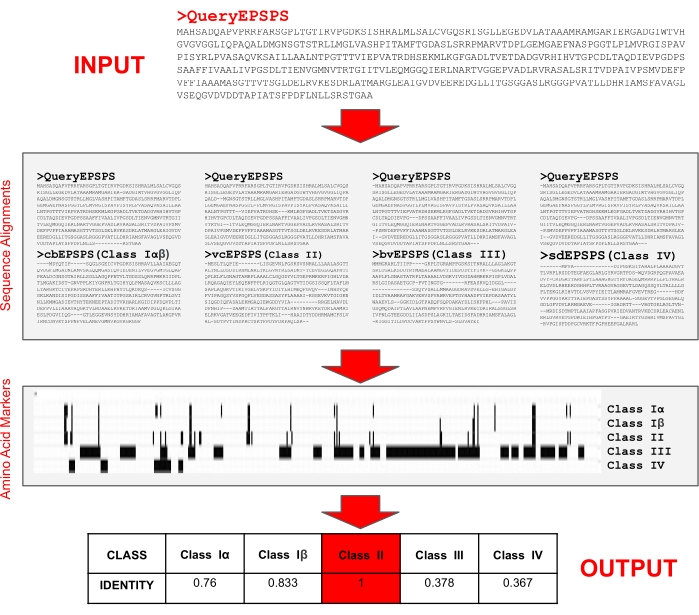
図5:EPSPSタンパク質配列のクラスを同定するアルゴリズム。 入力は、FASTA形式のEPSPSタンパク質配列である。このアルゴリズムは、グリホサートに対する潜在的な感受性を決定する参照タンパク質配列中の既知のアミノ酸マーカーとの比較を行う。このアルゴリズムは、自由にアクセスできるウェブサーバEPSPSClass29で実装された。 この図の拡大版を表示するには、ここをクリックしてください。

図 6: EPSPSClass Web サーバーの基本的な入出力 (A)入力:FASTA形式のEPSPSタンパク質配列。(b)出力1−同一性:クエリ配列(クラスI〜IV)およびモチーフ(クラスIII)中に存在するアミノ酸マーカーの割合。(C)出力2 - 同一性:クエリーおよび参照配列のアラインメント。(D)出力3 - クエリと参照シーケンスのペアワイズアラインメント。(e)参照EPSPS配列: コレラ菌 (vcEPSPS、クラスI)、 コクシエラ・バーネティイ (cbEPSPS、クラスII)、 ブレブンジモナス・ベシキュリス (bvEPSPS、クラスIII)、 ストレプトマイセス・ダバウェンシス (sdEPSPS、クラスIV)。(F) 追加のブラスト検索と保存されたドメインの識別を実行する ためのリンク この図の拡大版を表示するには、ここをクリックしてください。
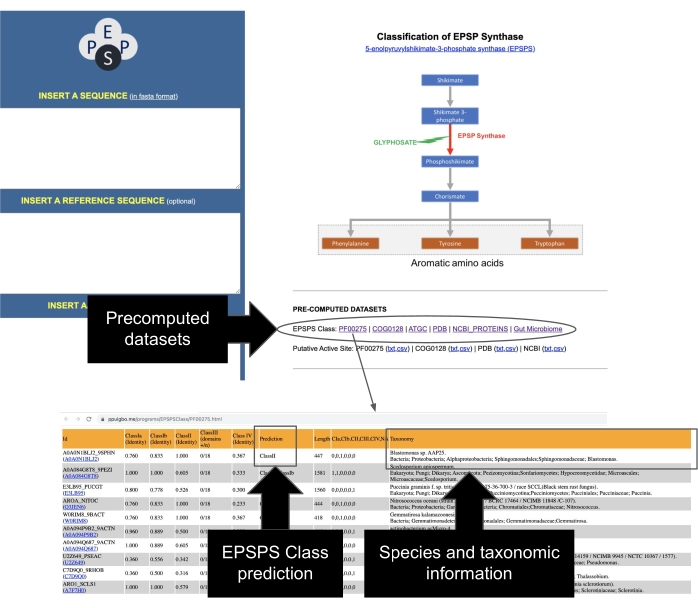
図 7: EPSPS シーケンスの事前計算データセットへのアクセス。 図の指示に従って、事前に計算された EPSPS シーケンスのデータセットにアクセスします。 この図の拡大版を表示するには、ここをクリックしてください。
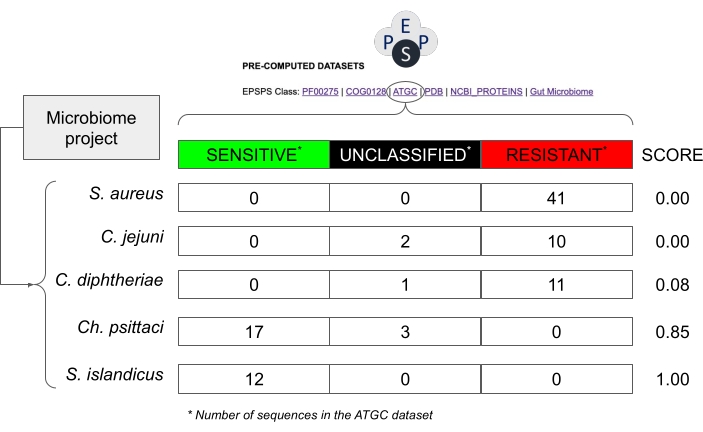
図8:EPSPS配列を含まないマイクロバイオームプロジェクトにおける潜在的な感度を推定する方法の例。 この例では、原核生物種からの配列を含むAlignable Tight Genomic Clusters30のデータベースからの値を使用しています。マイクロバイオームプロジェクトからの仮説的な種は、 黄色ブドウ球菌、 コリネバクテリウム・ジフテリア、 カンピロバクター・ジェジュニ、 クラミジア・プシッタキ 、 スルホロバス・アイランディクスである。グリホサートに対する感受性スコアは、Number_Sensitive_Sequences/Total_Number_Of_Sequencesとして計算される。 この図の拡大版を表示するには、ここをクリックしてください。
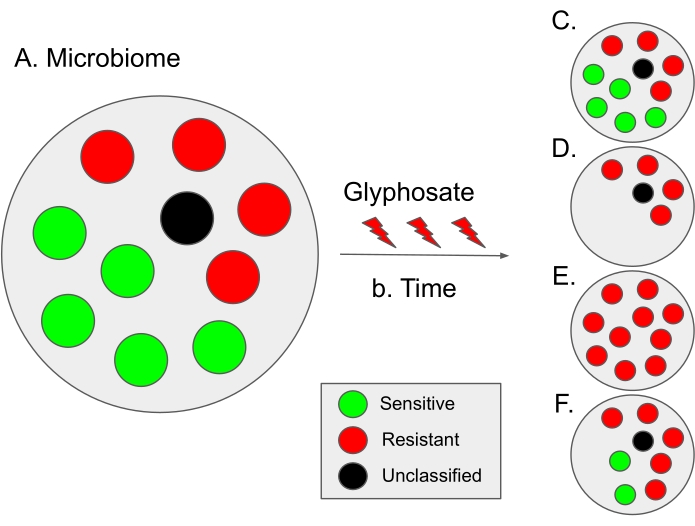
図9:このプロトコルと仮説的な進化シナリオの結果の解釈のスキーム。 (A)マイクロバイオームでは、潜在的な感受性(緑)と耐性(赤)の細菌の割合は約50:50です。黒い点は分類されていない微生物種を示す。したがって、グリホサートに対するそれらの感受性は不明である。いくつかのマイクロバイオームでは、感受性細菌の割合は、ヒト腸内マイクロバイオーム12のようにわずかに高い。(B)時間の経過とともに、グリホサートの使用は、微生物の嚥下障害(すなわち、感受性および耐性細菌の割合の不均衡)をもたらし、異なる仮説的なシナリオにつながる可能性がある。(C)仮説的なケース1(選択なし):グリホサートの使用はマイクロバイオームに影響を与えない。したがって、感受性および耐性細菌の割合は一定のままである。(D)仮説的なケース2:グリホサートの使用は、グリホサートに感受性の細菌を集団から除去する。我々は、このシナリオが用量依存性であるかもしれないと推測する。(E)仮説的なケース3:グリホサートの使用による選択圧力は、細菌の感受性状態を変化させる EPSPS 遺伝子の変異を増強する。したがって、微生物集団全体がグリホサートに対して耐性になる。さらに、このシナリオでは、多剤耐性菌が増加する可能性があります。(F)仮説的なケース4:グリホサートの使用は、特定の細菌種の組成を変化させ、耐性細菌に対する不均衡を生じさせるが、一部の細菌種は、おそらく流出ポンプなどの追加の耐性機構または EPSPS 遺伝子の過剰発現のために、変化しないままである13。このシナリオはまた、グリホサート耐性細菌の増加、ならびに追加の抗生物質に対する細菌耐性の増加をもたらし得る。 この図の拡大版を表示するには、ここをクリックしてください。
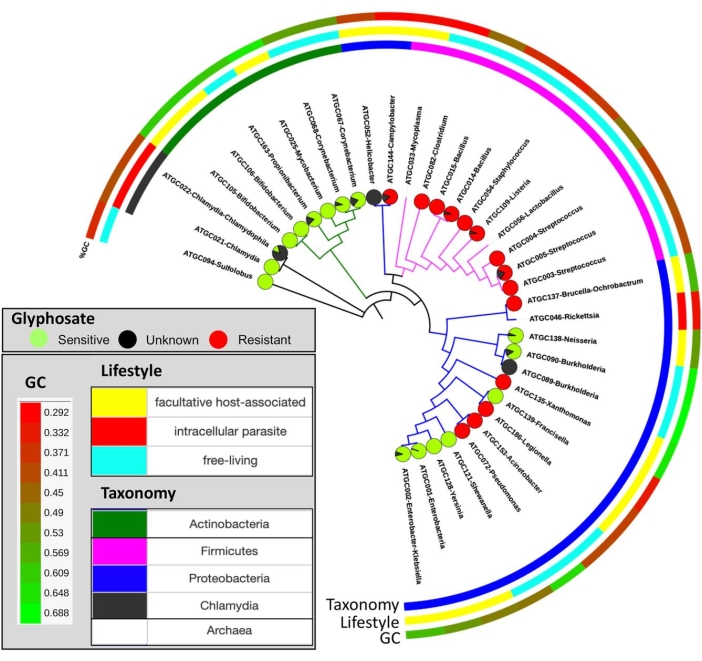
図10:種樹全体のグリホサートに対する予測感度の分布。 円グラフは、グリホサートに対して推定的に敏感(緑)または耐性(赤)であり、未分類(黒)の種の割合を示します。この図は、Rainioら14の許可を得て翻案されたものである。 この図の拡大版を表示するには、ここをクリックしてください。
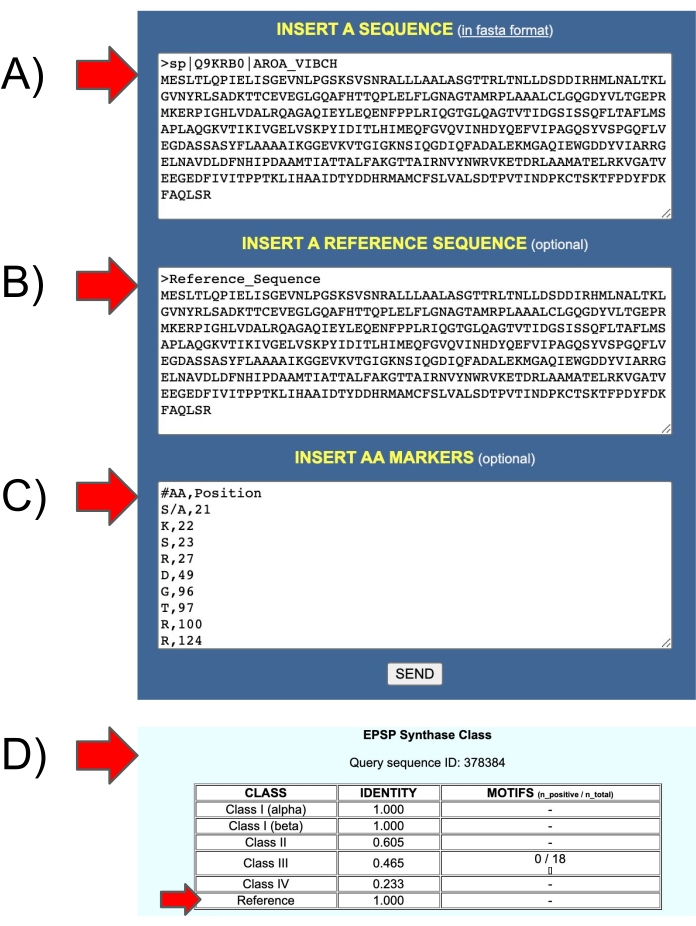
図 11: ユーザー自身の参照シーケンスをテストするための EPSPSClass Web サーバーの入出力。(A) 入力 1: クエリ シーケンス。(B)入力2:基準系列。(c)入力3:参照配列中のアミノ酸マーカー。(d)出力:同一性:クエリ配列(クラスI〜IVおよびユーザ自身の参照配列)中のアミノ酸マーカーの割合。この図の拡大版を表示するには、ここをクリックしてください。
表1:マイクロバイオーム解析における16S rRNA遺伝子およびITS領域のPCR増幅のためのプライマーのリスト この表をダウンロードするにはここをクリックしてください。
表2:異なるデータベース内の酵素5-エノールピルビルシキミ酸-3-リン酸合成酵素(EPSPS)のコード この表をダウンロードするにはここをクリックしてください。
表3:平均グリホサート濃度 この表をダウンロードするにはここをクリックしてください。
表4:グリホサートに敏感/耐性の種の割合の要約表。 この表は、Rainioら14の許可を得て翻案したものである。この表をダウンロードするには、ここをクリックしてください。
表5:参照配列中のアミノ酸マーカーの位置 この表をダウンロードするにはここをクリックしてください。
ディスカッション
このプロトコルは、EPSPSタンパク質の分析に基づいてマイクロバイオームに対するGBPの効果を定量する方法に関する一般的なガイダンスを提供します。このプロトコルには、(i)マイクロバイオームデータからのEPSPSタンパク質の定量化という3つの主要な重要なステップがあります。EPSPSは除草剤の直接の標的酵素であるため、このステップは重要です。したがって、 EPSPS 遺伝子のコピーを有する種は、GBPの使用によって影響を受ける可能性がある。それにもかかわらず、 EPSPS 遺伝子のコピーを欠いている種でさえ、代替の非標的機構を介して除草剤の影響を受ける可能性がある43、44。(ii) EPSPS 遺伝子の解析が研究の設計に含まれていない場合には、16S rRNA(細菌)または ITS (真菌)を分析することによって良好な推定値を得ることができる。この場合、包括的な参照表に頼ることが不可欠である(例えば、ATGCデータベースは、いくつかの近縁種由来のEPSPSタンパク質の配列を提供する)。(iii)EPSPSタンパク質は、EPSPSの活性部位の特定のアミノ酸残基に応じて、グリホサートに対して潜在的に感受性または耐性に分けられる。しかしながら、単一のアミノ酸に影響を及ぼす変異は、この分類45 を変化させる可能性があり、クラス間の遷移は比較的短期間に起こり得る14。
グリホサートに対する生物の潜在的な感受性は、参照ゲノム、アミノ酸マーカーおよび配列アラインメントによって決定することができる。(i)参照ゲノム:EPSPS酵素は、アミノ酸マーカーおよびモチーフの存在(クラスIIIの場合)に基づいて、グリホサートに対して潜在的に感受性(クラスIαまたはβ46,47)または耐性(クラスII 48,49、III50およびIV51)に分類され得る。これらのアミノ酸マーカーおよびモチーフは、コレラ菌(vcEPSPS、クラスI)、コクシエラ・バーネティイ(cbEPSPS、クラスII)、ブレブンジモナス・ベシキュリス(bvEPSPS、クラスIII)、およびストレプトマイセス・ダバウェンシス(sdEPSPS、クラスIV)のEPSPSタンパク質中のアミノ酸残基の位置に基づいている。(ii)アミノ酸マーカー:グリホサートはEPSPS酵素と相互作用し、ホスホエノールピルビン酸(PEP、EPSPS酵素の第2の基質)と競合する52,53。特定の種において、EPSPS配列中の小さなアミノ酸変化は、PEPに対するより高い親和性およびグリホサート12、14、52、54、55に対する耐性を提供する。他の配列において、グリホサートは、非阻害立体構造45においてEPSPS配列と結合する。グリホサートに対する耐性のある多くの12、14、48、49、52、54、55および耐性56、57のEPSP配列が記載されているが、EPSPSの現在の分類システムは、4つの主要なクラス(I−IV)12に分けられる(表5).(iii)配列アラインメント:EPSPS酵素を分類するために、我々は、参照配列(vcEPSPS、cbEPSPS、bvEPSPSおよびsdEPSPS)の各1つに対して、複数の配列アラインメントプログラムデフォルトパラメータ35-を用いて、ペアワイズアラインメントを実施した。これらのアラインメントは、クエリ配列中のアミノ酸マーカーの位置を同定するために必要である。その結果、酵素は、アミノ酸マーカーおよびクラスIIIベースのモチーフマーカーの存在に基づいて、12クラスI、IIおよび/またはIVに記載されるように分類される。
このプロトコルは、4つの既知のタイプのEPSPSに基づいています:1つのタイプは敏感で、他の3つは耐性です)。しかし、原核生物のEPSPS配列の約10%はまだ分類されていません(古細菌では16%、細菌では8%)12。したがって、さらなる研究は、グリホサート感受性を決定するためにこれらの配列を分析するべきである。EPSPSClass サーバーには、新しい遺伝子マーカーをテストするオプションが用意されています。EPSPS の既知のクラスの識別は、セクション 4.4 に示すように簡単です。および 図5.さらに、ユーザーが独自のクエリタンパク質と参照タンパク質を比較したい場合、サーバーは参照配列と一連のアミノ酸マーカーを手動で含めるオプションを提供します(図11)。このオプションは、EPSPSの新規クラスを同定し、他の除草剤および標的配列を試験するために利用することができる。
EPSPSクラスの解析は、配列解析とアミノ酸マーカーの有無によって決定されます。これは、現場での仮説検定に使用できる予備的な推定値です。アミノ酸マーカーは、実証研究および観察研究に基づいて文献46、47、48、49、50、51で決定されている。しかしながら、EPSPSクラスを決定するための参照タンパク質配列は、限られた数の種においてのみ試験されており、グリホサートに対する耐性を説明できない場合がある。代償性変異、およびEPSPS関連ドメイン(主に真菌)の効果も、グリホサート58に対する感受性に影響を及ぼし得る。このホワイト ペーパーの分析は、4 つの EPSPS クラスに基づいています。ヒト腸内微生物叢中の細菌の調査では、それらの約30%が未分類であることが示され(すなわち、これらの種のEPSPSタンパク質は既知のクラスのいずれにも属さない)、他のEPSPSクラスを同定するためにさらなる研究が必要である。また、細菌および植物におけるEPSPSタンパク質配列はユニドメインであるのに対し、真菌EPSPSタンパク質はいくつかのドメイン59を含むことに留意すべきである。したがって、真菌におけるタンパク質の折り畳みは、グリホサートに対するEPSPS酵素の異なる応答をもたらし得る。さらに、耐性のさらなる非標的機構(例えば、排出ポンプおよびEPSPS遺伝子13の過剰発現)またはグリホサートに対する感受性(例えば、ミトコンドリア輸送鎖12に対するグリホサートの効果)は考慮されない。
GBPは1974年以来除草剤として存在し、1991年以来広く利用されてきたが、これはグリホサートに対する生物の潜在的な感受性を決定する最初のバイオインフォマティクス法である。この方法は、標的配列中の既知のアミノ酸残基の同定に基づく。したがって、我々の方法は、種に対するグリホサートの潜在的な効果のベースライン推定値を提供する。近い将来、新規バイオインフォマティクス法は、未分類配列12、54、55のグリホサートに対する潜在的な感受性を決定するために、EPSPSタンパク質の追加のクラスを含むべきである。加えて、EPSPS酵素の正確な挙動が単一のアミノ酸変化によって変化し得ることを考えると12、14、52、54、55、さらにインシリコ実験では、EPSPSタンパク質の折り畳みにおける小さな変動、ならびに真菌におけるタンパク質構造に対するEPSPS関連ドメインの影響を考慮に入れるべきである58.さらに、グリホサートに対する耐性は、EPSPSタンパク質56、57の過剰発現によって産生され得ることが示されている。したがって、コドン使用頻度60の改善に基づくバイオインフォマティクス分析は、遺伝子発現を最大化または最小化する新規EPSPS配列を同定するために利用され得る。
農家、政治家、政策決定者は、農薬の大量使用に伴うリスクを徹底的に理解することが緊急に必要です。したがって、生物の農薬に対する潜在的な感受性を明らかにするバイオインフォマティクスツールと、異なる環境で実施されたよく複製された、無作為化された、フィールドリアリスティックな実験研究の両方が必要です。グリホサートに対する生物の感受性を調べるために設計された提示されたバイオインフォマティクス方法は、他の農薬に対して調節することができる。同様に、実験生態学の方法は、関連する生態学的問題を研究するために適用することができる。これらの方法を組み合わせることで、野外観察、ゲノムデータ、農薬使用の間の死傷者を実証することができます。提示されたすべての方法は、リスク評価において非常に貴重です。バイオインフォマティクス法は、例えば、農薬に対する微生物の適応をモニタリングし、農薬に対する病原体の耐性の増加、統合害虫管理(IPM)における生物学的防除剤として使用される微生物への悪影響、および細菌における抗生物質耐性など、潜在的な他の関連するリスクを試験するための定量的方法を提供するために使用することができる。
開示事項
利益相反:なし。
謝辞
この作品はフィンランドアカデミー(Marjo Helanderへの助成金番号311077)によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer Instrument | Agilent | G2939B | To check the concentration and quality of PCR products |
| dNTP mix (10 mM each) | ThermoFisher Scientific | R0192 | For PCR reactions |
| GoTaq G2 DNA Polymerase kit | Promega | M7848 | PCR buffer and DNA Polymerase for PCR amplification |
| Invisorb Spin Plant Mini Kit | INVITEK Molecular | 1037100300 | Genomic DNA extraction from plant tissues |
| Ion Chip Minifuge | ThermoFisher Scientific | 4479672 | For targeted sequencing of microbial PCR products |
| Ion PGM System | ThermoFisher Scientific | 4462921 | For targeted sequencing of microbial PCR products |
| Ion PGM Torrent Server | ThermoFisher Scientific | 4483643 | For targeted sequencing of microbial PCR products |
| Pippinprep | SageScience | PIP0001 | For size fractionation of PCR amplicons |
| Pressure tank | Berthoud | 102140 | For sprayin glyphosate based products in field |
| Primers | Sigma Aldrich | Custom-made | For PCR amplification |
| Rotary tiller | Grillo | 984511 | For tilling the soil in experimental plots |
| S1000 ThermalCycler | BIO-RAD | 1852196 | For PCR amplification |
参考文献
- Williams, G. M., Kroes, R., Munro, I. C. Safety evaluation and risk assessment of the herbicide Roundup and its active ingredient, glyphosate, for humans. Regulatory Toxicology and Pharmacology. 31, 117-165 (2000).
- Muola, A., et al. Risk in the circular food economy: Glyphosate-based herbicide residues in manure fertilizers decrease crop yield. The Science of the Total Environment. 750, 141422 (2021).
- Helander, M., Saloniemi, I., Saikkonen, K. Glyphosate in northern ecosystems. Trends in Plant Science. 17 (10), 569-574 (2012).
- Fuchs, B., Saikkonen, K., Helander, M. Glyphosate-Modulated Biosynthesis Driving Plant Defense and Species Interactions. Trends in Plant Science. 26 (4), 312-323 (2021).
- Helander, M., Saloniemi, I., Omacini, M., Druille, M., Salminen, J. -. P., Saikkonen, K. Glyphosate decreases mycorrhizal colonization and affects plant-soil feedback. The Science of the Total Environment. 642, 285-291 (2018).
- Bai, S. H., Ogbourne, S. M. Glyphosate: environmental contamination, toxicity and potential risks to human health via food contamination. Environmental Science and Pollution Research International. 23 (19), 18988-19001 (2016).
- Cuhra, M., Bøhn, T., Cuhra, P. Glyphosate: too much of a good thing. Frontiers in Environmental Science. 4, (2016).
- de Brito Rodrigues, L., et al. Impact of the glyphosate-based commercial herbicide, its components and its metabolite AMPA on non-target aquatic organisms. Mutation Research. 842, 94-101 (2019).
- Steinrücken, H. C., Amrhein, N. The herbicide glyphosate is a potent inhibitor of 5-enolpyruvylshikimic acid-3-phosphate synthase. Biochemical and Biophysical Research Communications. 94 (4), 1207-1212 (1980).
- Bentley, R. The shikimate pathway--a metabolic tree with many branches. Critical Reviews in Biochemistry and Molecular Biology. 25 (5), 307-384 (1990).
- Richards, T. A., et al. Evolutionary origins of the eukaryotic shikimate pathway: gene fusions, horizontal gene transfer, and endosymbiotic replacements. Eukaryotic Cell. 5 (9), 1517-1531 (2006).
- Leino, L., et al. Classification of the glyphosate target enzyme (5-enolpyruvylshikimate-3-phosphate synthase) for assessing sensitivity of organisms to the herbicide. Journal Of Hazardous Materials. 408, 124556 (2021).
- Rainio, M. J., Ruuskanen, S., Helander, M., Saikkonen, K., Saloniemi, I., Puigbò, P. Adaptation of bacteria to glyphosate: a microevolutionary perspective of the enzyme 5-enolpyruvylshikimate-3-phosphate (EPSP) synthase. BioRxiv. , (2020).
- Rainio, M. J., Ruuskanen, S., Helander, M., Saikkonen, K., Saloniemi, I., Puigbò, P. Adaptation of bacteria to glyphosate: a microevolutionary perspective of the enzyme 5-enolpyruvylshikimate-3-phosphate synthase. Environmental Microbiology Reports. 13 (3), 309-316 (2021).
- Helander, M., Pauna, A., Saikkonen, K., Saloniemi, I. Glyphosate residues in soil affect crop plant germination and growth. Scientific Reports. 9 (1), 19653 (2019).
- Ruuskanen, S., Rainio, M. J., Uusitalo, M., Saikkonen, K., Helander, M. Effects of parental exposure to glyphosate-based herbicides on embryonic development and oxidative status: a long-term experiment in a bird model. Scientific Reports. 10 (1), 6349 (2020).
- Ruuskanen, S., et al. female preference and adverse developmental effects of glyphosate-based herbicides on ecologically relevant traits in Japanese quails. Environmental Science & Technology. 54 (2), 1128-1135 (2020).
- Mäki, A., Rissanen, A. J., Tiirola, M. A practical method for barcoding and size-trimming PCR templates for amplicon sequencing. Biotechniques. 60 (2), 88-90 (2016).
- Chelius, M. K., Triplett, E. W. The diversity of archaea and bacteria in association with the roots of Zea mays L. Microbial Ecology. 41 (3), 252-263 (2001).
- Lane, D. J. 16S/23S rRNA sequencing. Nucleic acid techniques in bacterial systematics. , 115-175 (1991).
- Ghyselinck, J., Pfeiffer, S., Heylen, K., Sessitsch, A., De Vos, P. The effect of primer choice and short read sequences on the outcome of 16S rRNA gene based diversity studies. Plos One. 8 (8), 71360 (2013).
- Zheng, D., Alm, E. W., Stahl, D. A., Raskin, L. Characterization of universal small-subunit rRNA hybridization probes for quantitative molecular microbial ecology studies. Applied and Environmental Microbiology. 62 (12), 4504-4513 (1996).
- . JoVE Supplementary Material Available from: https://ppuigbo.me/programs/EPSPSClass/JOVE_SM (2021)
- . Alignments Conserved Positions Available from: https://ppuigbo.me/programs/primers (2021)
- . NCBI GenBank Available from: https://www.ncbi.nim.nih.gov/genbank (2021)
- . COG Database Available from: https://www.ncbi.nim.nih.gov/research/cog (2021)
- . Protein Data Bank Available from: https://www.rcsb.org (2021)
- . EPSPSClass Available from: https://ppuigbo.me/programs/EPSPSClass (2021)
- Kristensen, D. M., Wolf, Y. I., Koonin, E. V. ATGC database and ATGC-COGs: an updated resource for micro- and macro-evolutionary studies of prokaryotic genomes and protein family annotation. Nucleic Acids Research. 45, 210-218 (2017).
- . CSC - IT CENTER FOR SCIENCE LTD Available from: https://www.csc.fi (2021)
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Tkacz, A., Hortala, M., Poole, P. S. Absolute quantitation of microbiota abundance in environmental samples. Microbiome. 6 (1), 110 (2018).
- El-Gebali, S., et al. The Pfam protein families database in 2019. Nucleic Acids Research. 47, 427-432 (2019).
- Benson, D. A., et al. GenBank. Nucleic Acids Research. 41, 36-42 (2013).
- Galperin, M. Y., Makarova, K. S., Wolf, Y. I., Koonin, E. V. Expanded microbial genome coverage and improved protein family annotation in the COG database. Nucleic Acids Research. 43, 261-269 (2015).
- Burley, S. K., Berman, H. M., Kleywegt, G. J., Markley, J. L., Nakamura, H., Velankar, S. Protein data bank (PDB): the single global macromolecular structure archive. Methods in Molecular Biology. 1607, 627-641 (2017).
- Raoult, D., Hadjadj, L., Baron, S. A., Rolain, J. -. M. Role of glyphosate in the emergence of antimicrobial resistance in bacteria. The Journal of Antimicrobial Chemotherapy. 76 (7), 1655-1657 (2021).
- Liu, J., Gefen, O., Ronin, I., Bar-Meir, M., Balaban, N. Q. Effect of tolerance on the evolution of antibiotic resistance under drug combinations. Science. 367 (6474), 200-204 (2020).
- Peixoto, F. Comparative effects of the Roundup and glyphosate on mitochondrial oxidative phosphorylation. Chemosphere. 61 (8), 1115-1122 (2005).
- Peillex, C., Pelletier, M. The impact and toxicity of glyphosate and glyphosate-based herbicides on health and immunity. Journal of Immunotoxicology. 17 (1), 163-174 (2020).
- Funke, T., Han, H., Healy-Fried, M. L., Fischer, M., Schönbrunn, E. Molecular basis for the herbicide resistance of Roundup Ready crops. Proceedings of the National Academy of Sciences of the United States of America. 103 (35), 13010-13015 (2006).
- Light, S. H., Krishna, S. N., Minasov, G., Anderson, W. F. An unusual cation-binding site and distinct domain-domain interactions distinguish Class II Enolpyruvylshikimate-3-phosphate synthases. Biochemistry. 55 (8), 1239-1245 (2016).
- Firdous, S., Iqbal, S., Anwar, S., Jabeen, H. Identification and analysis of 5-enolpyruvylshikimate-3-phosphate synthase (EPSPS) gene from glyphosate-resistant Ochrobactrum intermedium Sq20. Pest Management Science. 74 (5), 1184-1196 (2018).
- Barry, G. F., Kishore, G. M., Padgette, S. R., Stallings, W. C. Glyphosate-tolerant 5-enolpyruvylshikimate-3-phosphate synthases. United States Patient. , (1997).
- Priestman, M. A., Funke, T., Singh, I. M., Crupper, S. S., Schönbrunn, E. 5-Enolpyruvylshikimate-3-phosphate synthase from Staphylococcus aureus is insensitive to glyphosate. FEBS Letters. 579, 728-732 (2005).
- Carozzi, N., Carr, B., Hammer, P. E. Identification of a new class of EPSP synthases. World Intellectual Property Organization Publ.of the Int.Appl. without Int.search. , (2006).
- Lira, J. M., Cicchillo, R. M., Nair, S. K. Novel class of glyphosate resistance genes. US Patent. , (2013).
- Funke, T., et al. Structural basis of glyphosate resistance resulting from the double mutation Thr97 -> Ile and Pro101 -> Ser in 5-enolpyruvylshikimate-3-phosphate synthase from Escherichia coli. The Journal of Biological Chemistry. 284 (15), 9854-9860 (2009).
- Schönbrunn, E., et al. Interaction of the herbicide glyphosate with its target enzyme 5-enolpyruvylshikimate 3-phosphate synthase in atomic detail. Proceedings of the National Academy of Sciences of the United States of America. 98 (4), 1376-1380 (2001).
- Stalker, D. M., Hiatt, W. R., Comai, L. A single amino acid substitution in the enzyme 5-enolpyruvylshikimate-3-phosphate synthase confers resistance to the herbicide glyphosate. The Journal of Biological Chemistry. 260 (8), 4724-4728 (1985).
- Eschenburg, S., Healy, M. L., Priestman, M. A., Lushington, G. H., Schönbrunn, E. How the mutation glycine96 to alanine confers glyphosate insensitivity to 5-enolpyruvyl shikimate-3-phosphate synthase from Escherichia coli. Planta. 216 (1), 129-135 (2002).
- Achary, V. M. M., et al. Overexpression of improved EPSPS gene results in field level glyphosate tolerance and higher grain yield in rice. Plant Biotechnology Journal. 18 (12), 2504-2519 (2020).
- Huang, Z., Liu, Y., Zhang, C., Jiang, C., Huang, H., Wei, S. Molecular basis of natural tolerance to glyphosate in Convolvulus arvensis. Scientific Reports. 9 (1), 8133 (2019).
- Tall, T. . A census analysis of the 5-enolpyruvylshikimate-3-phosphate (EPSP) synthase and EPSP-associated domains. , (2020).
- Tall, T., Puigbò, P. The glyphosate target enzyme 5-Enolpyruvyl Shikimate 3-Phosphate Synthase (EPSPS) contains several EPSPS-associated domains in fungi. ATLA Summary of Proceedings. 76 (1), 6 (2020).
- Puigbò, P., Guzmán, E., Romeu, A., Garcia-Vallvé, S. OPTIMIZER: a web server for optimizing the codon usage of DNA sequences. Nucleic Acids Research. 35, 126-131 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved