需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
水凝胶阵列可提高 3D 肿瘤模型中基质成分和治疗药物筛选效果的通量
摘要
本方案描述了一个实验平台,用于评估机械和生化线索对3D基质模拟培养物中患者来源的胶质母细胞瘤细胞的化疗反应的影响,使用定制的UV照明装置促进具有可调机械特征的水凝胶的高通量光交联。
摘要
细胞基质相互作用通过生化、机械和几何线索介导复杂的生理过程,影响病理变化和治疗反应。在药物开发管道的早期考虑基质效应预计将增加新疗法临床成功的可能性。基于生物材料的策略在3D细胞培养中重述特定的组织微环境,但将这些与主要用于药物筛选的2D培养方法相结合一直具有挑战性。因此,这里介绍的方案详细介绍了在多孔板格式的小型化生物材料基质中进行3D培养的方法开发,以促进与现有药物筛选管道和常规细胞活力测定的集成。由于对于在培养细胞中保存临床相关表型至关重要的基质特征具有高度的组织和疾病特异性,因此有必要对基质参数进行组合筛选,以确定特定应用的适当条件。这里描述的方法使用小型化培养形式来评估癌细胞对基质力学和配体呈递的正交变化的反应。具体而言,这项研究展示了使用该平台来研究基质参数对患者来源的胶质母细胞瘤(GBM)细胞对化疗反应的影响。
引言
开发新药的预期成本在过去十年中稳步上升,目前估计超过10亿美元1。这笔费用的一部分是进入临床试验的药物的高失败率。大约12%的候选药物最终在2019年获得美国食品和药物管理局(FDA)的批准。许多药物由于意想不到的毒性2而在I期失败,而其他通过安全性试验的药物可能由于缺乏疗效而失败3。由于无效而导致的这种损耗可以部分解释为,在药物开发过程中使用的癌症模型对临床疗效是出了名的不预测4。
体外和体内模型之间的功能差异可归因于从其天然微环境中去除癌细胞,包括非肿瘤细胞和物理ECM5,6。通常,研究小组使用市售的培养基质,例如Matrigel(一种来自小鼠肉瘤的蛋白质基底膜基质)为培养的肿瘤细胞提供3D基质微环境。与2D培养相比,膜基质中的3D培养提高了体外结果的临床相关性7,8。然而,来自去细胞化组织(包括膜基质)的培养生物材料通常表现出批次间的变异性,这可能影响重现性9。此外,衍生自具有不同组织起源的肿瘤的基质可能与所研究的那些基质10可能不提供适当的生理线索。最后,具有高度肿瘤内异质性的癌症具有微环境特征,这些特征在亚微米级尺度上变化,并且膜基质无法调整以重述11。
胶质母细胞瘤(GBM)是一种致死性均值的脑肿瘤,中位生存时间约为15个月,是一种治疗开发特别困难的癌症12,13。目前GBM的治疗标准包括原发性肿瘤切除术,然后放疗,然后使用替莫唑胺(TMZ)进行化疗14。然而,超过一半的临床GBM肿瘤通过各种机制表现出治疗耐药性15,16,17。预测治疗方案对个体患者的疗效是极其困难的。用于预测个体结果的标准临床前模型包括将患者来源的肿瘤细胞异位移植到免疫功能低下的小鼠中。虽然患者来源的异种移植物可以概括临床GBM肿瘤的许多方面,并且对于临床前模型18很有价值,但它们本质上是昂贵的,低通量的,耗时的,并且涉及伦理问题19。在2D塑料表面上或作为球体培养患者来源的细胞,大多避免了这些问题。虽然患者来源的细胞保留了遗传畸变,但它们在2D或悬浮球体中的培养物在很大程度上在啮齿动物和原始患者肿瘤中患者来源的异种移植物的代表性较差20。以前,我们和其他人已经证明,在模仿脑组织的机械和生化特性的3D ECM中培养的GBM细胞可以保留耐药表型10,21,22,23。
透明质酸(HA)是一种在脑ECM中丰富的多糖,在GBM肿瘤中过表达,其CD44受体调节体外耐药性的获得21,24,25,26,27之间的相互作用。例如,在柔软的3D培养物中加入HA增加了患者来源的GBM细胞获得治疗耐药性的能力。这种机械反应性取决于HA与GBM细胞21上的CD44受体的结合。此外,整合素与含RGD肽结合,并入3D培养基质中,以刚度依赖性方式扩增CD44介导的化学抗性21。除HA外,几种ECM蛋白的表达,其中许多含有RGD区域,在正常脑和GBM肿瘤28之间变化。例如,一项研究报告说,28种不同的ECM蛋白在GBM肿瘤中上调29。在这种复杂的肿瘤基质微环境中,癌细胞整合机械和生化线索以产生特定的抗性表型,这取决于Young的整合素结合肽的模量或密度28,29,30的相对较小的差异(例如,小于一个数量级)。
本方案描述了肿瘤细胞如何解释基质线索的独特组合,并鉴定促进治疗耐药性的复杂,患者特异性基质微环境(图1A)。用于生成用于3D培养的小型化,精确调谐的基质的光化学方法提供了一个大的正交可变空间。由微控制器运行的定制LED阵列被整合到384孔板格式中的光交联水凝胶中,以提高自动化和可重复性。暴露强度在井间变化,以改变所得水凝胶的微机械性质,如使用原子力显微镜(AFM)评估的那样。虽然本手稿并不侧重于构建照明阵列本身,但提供了电路图(图1B)和零件清单(材料表)作为器件再现的辅助工具。
该报告展示了在独特的3D微环境中培养的GBM细胞阵列的快速生成,其中Young的模量(单个数量级上的四个水平)和整合素结合肽含量(来自四种不同的ECM蛋白)正交变化。然后,该方法用于研究水凝胶力学和ECM特异性整合素参与对患者来源的GBM细胞的活力和增殖的相对贡献,因为它们对替莫唑胺(TMZ)化疗产生耐药性。
研究方案
患者来源的GBM细胞系(GS122和GS304)由David Nathanson教授(我们的合作者)提供,他根据加州大学洛杉矶分校机构审查委员会批准的方案(IRB# 10-000655)开发了这些细胞系。细胞被提供去识别化,以便细胞系不能与个体患者联系起来。
1. 水凝胶溶液的制备
- 通过将 HEPES 粉末以 20 mM 溶解在 Hank 的平衡盐溶液 (HBSS) 中来制备 HEPES 缓冲溶液。完全溶剂化后将pH值调节至7。
- 在HEPES缓冲溶液中,溶解硫醇化HA(700 kDa标称分子量,见 材料表),按照前一报告31制备,使每种葡萄糖醛酸上的6%-8%的羧酸残基用硫醇修饰,浓度为10mg / mL在缓冲溶液中。
注意:建议使用琥珀色小瓶,以防止硫醇被环境光氧化。- 在室温下使用磁力搅拌板(<1,000rpm)搅拌直至完全溶解,通常约为45分钟。
- 当HA溶解时,制备(1)100mg / mL的8臂PEG-降冰片烯(20 kDa),(2)100mg / mL的4臂PEG-PEG-硫醇(20 kDa),(3)4mM的半胱氨酸或含半胱氨酸肽(例如GCGYGRGDSPG)和(4)4mg / mL的LAP在微量离心管中的溶液(参见 材料表)。
- 在步骤1.1中制备的HEPES缓冲溶液中制备这四种溶液中的每一种。在执行步骤4之前,涡旋溶液以确保每种试剂完全溶解。
注意:如果测试多种不同的肽,则每种肽必须含有半胱氨酸或其他硫醇部分来源,用于这种偶联化学。 - 此时,准备所有肽的溶液(4mM可用硫醇)以在单个水凝胶中拴留。
注意:肽序列和ECM蛋白,它们从中衍生出来并用于本研究,列于 表1中。细胞不结合的N-乙酰半胱氨酸(见 材料表)可以被生物活性的、含硫醇的肽取代,以滴定粘附肽的浓度或作为阴性对照31。
- 在步骤1.1中制备的HEPES缓冲溶液中制备这四种溶液中的每一种。在执行步骤4之前,涡旋溶液以确保每种试剂完全溶解。
- 混合HA,PEG-降冰片烯,PEG-硫醇和半胱氨酸/硫醇含肽的单个溶液(参见 材料表),以达到 表2中列出的最终水凝胶基质的最终浓度。在磁力搅拌板上搅拌(<1,000rpm)至少30分钟以充分混合。
注:HA溶液粘度高,最好使用容积式移液器处理(见 材料表)。如果没有正置换式移液器,也可以使用标准微量移液管,使用宽孔吸头缓慢移液,从而省去粘性溶液。
2. 通过 LED 阵列对水凝胶进行照明和光交联
注意:佩戴紫外线防护眼镜,并用紫外线吸收材料覆盖照明区域。
注:此协议中描述的 LED 阵列由六组串联放置的八个 LED 组成,如提供的电路图所示(图 1A)。每组 LED 都可以独立供电,每次运行最多可产生六种不同的辐照度。 补充文件 1 包含与以下说明相对应的屏幕截图,以提供进一步的指导。
- 从 补充编码文件下载照明设备.zip文件。此目录包含以下文件:Arduino.zip(补充编码文件 1)、驱动程序.zip(补充编码文件 2)、GUI.zip(补充编码文件 3)和 Holder.zip(补充编码文件 4)。
注:3D 打印顶部和底部,以便将电路板固定到位(有关详细信息,请参见 补充编码文件 )。 - 下载并安装微控制器软件(请参见 材料表)。
- 下载并安装 GUI 软件(请参见 材料表)。有关软件操作说明,请参阅 补充文件 1 。
- 打开"处理"并通过单击"草图">"导入库">"添加库"来安装 controlIP5 库。然后,在库中搜索 controlIP5 并单击"安装"。这是第一次执行此操作。
- 使用 36 伏电源为照明设备供电(请参阅 材料表),并使用微型 USB 电缆将其连接到 PC。
注意:某些设备不会自动为各种Arduino纳米板安装驱动程序。设备 zip 文件中提供了一组驱动程序。 - 使用Arduino IDE打开位于Adruino.zip文件夹中的Arduino.ino文件。
- 通过单击复 选标记 按钮编译Arduino.ino文件。通过单击 箭头 按钮上传编译的代码。
- 使用"处理"打开位于 GUI.zip 文件夹中的 GUI.pde 文件。
- 单击处理程序中的 "运行 "以启动用于控制照明设备的图形用户界面。
- 在图形用户界面窗口中,单击" 强度 ",查找要交联的含有水凝胶前体溶液的色谱柱,并输入所需的强度。单击 "时间 "框并输入所需的时间。对于 表 2 中提供的解决方案,这将是 15 秒。
注意:最终用户需要使用辐射计将数字强度值校准为辐照度。 图2A提供了典型强度的示例。 - 将样品与照明设备(图2B)对齐,并在硅胶模具的单列(参见 材料表)或384孔板中每隔一个LED对齐。单击" 完成" 开始照明。根据需要重复此过程,以照亮384孔板的多个载玻片或其他孔。
注意:支架的设计使得384孔板在照明期间与内腔的一角齐平。- 照明后,当放置在一个角时,将孔板移动到下一个角并重复。要照亮板的另一半上的孔,请将板从支架中取出并旋转180°。
- 按照以下步骤生成具有不同力学原理的水凝胶,以进行机械表征。
- 使用胶带清洁载玻片和硅胶模具以清除碎屑。将硅胶模具粘在载玻片上,向下按压以确保良好的密封性,并置换任何气泡。
- 将步骤1.4中制备的80μL水凝胶前体溶液移液到载玻片上的每个硅胶模具中。
- 将载玻片放在照明设备上,该设备与单列中的所有其他LED对齐。如步骤2所述,将水凝胶前体暴露在紫外线下15秒,以光交联。
- 一旦照明停止,取回载玻片,并通过用细尖(10μL移液器吸头,30G针头等)跟踪模具的内圆周,从模具中松开凝胶。用镊子/镊子取下硅胶模具。
- 通过润湿刮刀并将其从载玻片上轻轻推开,将交联水凝胶移动到12孔板的各个孔中。在加入水凝胶之前,用2 mL DPBS(参见 材料表)填充每个孔。在室温下将凝胶在DPBS溶液中膨胀至少12小时(通常过夜)(用于第二天的机械表征)。
3. 原子力显微镜 (AFM) 测量
- 根据制造商的说明打开原子力显微镜(AFM)(参见 材料表)。该协议提供了使用仪器和相关软件的简要说明。
- 安装AFM探头(见 材料表)。
注:对于本研究,用球形2.5μm二氧化硅颗粒修饰了标称弹簧常数为0.01 N/m的三角形氮化硅悬臂。 - 安装后,将激光器对准三角形探头的顶点,然后调整反射镜和激光偏转以最大化信号总和(通常在1.5-2.2伏特之间)。
- 将探针浸入DPBS中并等待长达15分钟以获得热平衡。单击 "校准 "按钮,然后选择 "接触相关 校准"。单击" 收集热调谐 "按钮,然后收集数据,选择3 kHz左右的峰值进行校准。
注意:由于折射率的变化,浸入液体后可能需要对反射镜和激光导流板进行轻微的调整。 - 通过将接近 参数 设置为 恒定速度, 目标高度为 7.5μm,接近速度为 15μm / s,接近培养皿(塑料)的表面。 为进近启用每次运行的基线测量 ,以便进近连续运行,并且不会因偏转器漂移而提前停止。
- 接近时,将力映射的采集参数设置为4 nN周转,2 μm压痕距离,1 μm / s速度和0 s接触时间。按" 开始 "按钮开始收集塑料表面(例如,孔板)上的力曲线。
- 返回到校准窗口,选择与塑料接触和压痕相对应的力曲线部分。接受计算出的灵敏度和刚度值,以便探头完成校准。
- 校准后,升高AFM探针并放置水凝胶样品进行询问。按照步骤5中提供的设置接近水凝胶。
注意:在接近水凝胶表面的过程中,设备可能会错误地触发接近状态。要验证实际方法,请获得步骤4.6中的力曲线。如果生成的曲线未显示接触和导致的压痕,则重复接近过程。 - 表面接近成功后,切换到 力映射 模式,并将采集参数设置为 8 x 8 尺寸的映射,每轴长度为 40 μm。获取各个区域的力图,以评估刚度测量的均匀性。
- 使用软件程序JPK SPM数据处理通过赫兹/斯内登模型拟合(方程1和2,参见表3了解所有变量的定义)解释力曲线,并选择球面几何32,33,3 4。
 等式 132
等式 132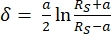 等式 232
等式 232
- 使用软件程序JPK SPM数据处理通过赫兹/斯内登模型拟合(方程1和2,参见表3了解所有变量的定义)解释力曲线,并选择球面几何32,33,3 4。
4. 3D基质嵌入培养物的设置和药物治疗
- 将所需细胞制备为单细胞溶液。
注意:不同的细胞类型可能需要不同的传代方法。参考文献31中报道了从T-75烧瓶传代GBM球体悬浮培养的典型方案。 - 从T-75烧瓶悬浮培养物中收集GBM球体(直径约150μm)到15mL锥形管中。用5 mL DPBS冲洗培养瓶以除去任何残留的细胞和培养基,并将该体积添加到锥形管中。
- 在室温下将含有细胞的锥形管以200× g 离心5分钟。离心后,用5mL血清学移液管除去上清液,注意不要干扰细胞沉淀,并重悬于5mL DPBS中。
- 在室温下以200× g 离心5分钟以洗涤细胞。用5mL血清学移液管吸出上清液,注意不要干扰细胞沉淀,然后将细胞重悬于2mL细胞解离试剂中(参见 材料表)。
- 在室温下孵育10-15分钟。加入3 mL完整培养基(见 材料表)并轻轻移液3-5次,将球体分解成单细胞悬浮液31。
- 将单细胞悬浮液在400× g 下离心5分钟以在室温下沉淀细胞(单细胞悬浮液可以更快地旋转以形成沉淀细胞)。用5mL血清学移液管吸出上清液,注意不要干扰细胞沉淀。将细胞重悬于1 mL完整培养基中。
注意:如果细胞在传代后保持在团块中,而不是作为悬浮液中的单个细胞,则细胞可以通过40μm细胞过滤器来实现单细胞悬浮液。 - 使用血细胞计数器取出一部分细胞进行计数。用台盼蓝稀释这部分两倍,其渗透细胞活力受损。只计算活的无色细胞。通常,每个烧瓶接种800,000个细胞的T-75在培养一周后产生2-300万个细胞。
- 确定封装所需的电池数量。将含有所需细胞总数的培养基体积转移到无菌的1.7 mL微量离心管中。在室温下以400× g 旋转5分钟。
注意:例如,至少需要将250万个细胞重悬于1 mL凝胶体积中,以250万个细胞/mL的速度封装细胞。1 mL 的凝胶体积允许用户分配 100 个凝胶滴,其中每个凝胶滴为 10 μL 体积。建议准备额外〜20%体积的悬浮在水凝胶溶液中的细胞,以解释移液器转移过程中的损失。因此,在本例中,将制备300万个细胞和1.2 mL水凝胶前体溶液。建议最小密度为50万个细胞/mL。 - 用微量移液管吸出上清液,注意不要干扰细胞沉淀。将细胞沉淀重悬于步骤1.4中制备的水凝胶前体溶液中,通过用1,000μL微量移液管上下移液4-5次充分混合。
- 将细胞装入重复移液器(参见 材料表)中,以分配10μL。为避免气泡和不均匀的分配,请将重复移液器再分配1-2次到废液容器中。
- 在384孔板的每个孔中,从重复移液器中分配10μL悬浮在水凝胶溶液中的细胞。使用LED阵列,以强度照亮每个含有细胞的孔(步骤2)15秒(图 2A 中的示例结果使用强度1.14,1.55,2.15,2.74 mW / cm2)以实现所需的机械性能。
注意:建议从每个实验条件的五次重复开始,并根据所需的通量和终点测定的方差进行放大或缩小。 - 向含有细胞的每个孔中加入40μL完整培养基。向凝胶周围的非实验性干燥孔中加入50μLDPBS,以最大限度地减少蒸发引起的损失。
- 对于GBM细胞,加入40μL含培养基的药物(例如,TMZ,参见 材料表)以达到最终所需的浓度(10μM-100μM在二甲基亚砜(DMSO)或载体(DMSO)中),相应地,从封装后3天开始。
5. CCK8增殖试验
- 向含有细胞的每个孔中加入10μLCCK8试剂(参见 材料表)。
注意:如果首次进行该测定,请在培养基中包括阴性对照孔,例如仅培养基或无细胞水凝胶。 - 根据制造商的说明孵育1-4小时。
注意:此时间可能因细胞类型和密度而异,因此需要针对每种应用测试孵育时间,以使吸光度值在线性范围内,这是应用Beer定律35的要求。 - 在450nm处读取孵育后所有孔的吸光度。
- 计算在步骤3中获得的450nm处对于每组的车辆状况的平均吸光度。将每个药物治疗良好的人除以每组车辆对照的平均值。
- 通过百分位方法36 生成引导分布 (N = 10,000) 来计算置信区间。
注意:通常,可以使用 95% 的置信区间,并将置信区间未超过 1 的条件解释为显著,并值得进一步研究。将置信区间设置为 95% 与设置 p = 0.05 的显著性截止值一致。对于结果中显示的数据,在区分促进或抑制基质介导的耐药性的条件方面是有用的,需要双侧分析。
结果
AFM测量结果证实,使用定制的Arduino控制LED阵列,在光交联过程中,水凝胶力学的精确控制是紫外辐照度(mW/cm2)的函数(图2A)。该方案中使用的水凝胶制剂可以在表2中找到。提供的模板上LED的间距与384孔板的所有其他孔的间距相匹配,从而允许在板内形成凝胶(图2B)。对单个水凝胶表面微米级区域的AFM询问表明,平均杨氏模量...
讨论
目前的工作提出了在基于HA的基质硬度和可用于整合素参与的肽中生成3D小型化培养物的方法。该技术能够系统地研究基质参数如何影响细胞表型(例如,暴露于化疗的癌细胞的活力),并提高通量。先前的方法,包括本文提出的方法,已经通过改变最终配方中总聚合物的百分比来调整水凝胶刚度,其中较硬的水凝胶具有更高的聚合物含量21,31。然而,...
披露声明
作者没有什么可透露的。
致谢
作者要特别感谢Carolyn Kim,Amelia Lao,Ryan Stoutamore和Itay Solomon对光凝胶化方案早期迭代的贡献。细胞系GS122和GS304由David Nathanson慷慨提供。所有数字都是用 BioRender.com 创建的。加州大学洛杉矶分校的核心设施,分子筛选共享资源以及纳米和Pico表征实验室对这项工作起到了重要作用。Chen Chia-Chun得到了加州大学洛杉矶分校Eli和Edythe Broad再生医学和干细胞研究培训计划中心的支持。Grigor Varuzhanyan得到了肿瘤细胞生物学培训计划NIH拨款(T32 CA 009056)的支持。
材料
| Name | Company | Catalog Number | Comments |
| 1.1 kOhm resistors, 6 W | Digikey | 35601k1ft | |
| 1.7 mL microcentrifuge tube | Genesse Scientific | 21-108 | |
| 15 mL conical tube | Fisher Scientific | 14-959-70C | |
| 365 nm LED | Digikey | ltpl-c034uvh365 | |
| 384 well plate | Bio Greiner One | 781090 | |
| 40 µm cell strainer | MTC bio | C4040 | |
| 4-Armed thiol terminated polyethlene glycol (20 kDa) | Laysan Bio | 4arm-PEG-SH-20K-1g | |
| 6 NPN BJTs | Digikey | 2n5550ta | |
| 80 Ohm resistors, 0.125 W | Digikey | erjj-6enf80r6v | |
| 8-Armed norbornene terminated polyethylene glycol (20 kDa) | Jenkem Technology | A7025-1 | |
| Accutase | Innovative Cell Technologies | AT104500 | cell dissociation reagent |
| AFM Probes | Novascan | 0.01 N/m Nominal spring constant, 2.5 µm SiO2 particle | |
| Arduino IDE | Arduino | 1.8.19 | |
| Arduino Nano | Makerfire | Mini Nano V3.0 ATmega328P Microcontroller Board | |
| bFGF | Peprotech | 100-18B | 20 ng/mL |
| CCK8 | Abcam | ab228554 | |
| Centrifuge | Thermoscientific | sorvall legend xtr | |
| CP100ST | Gilson | F148415 | Pipette tips for positive displacement pipette |
| Cubis Semi-Micro Balance | Sartorius | MSA225S100DI | |
| DMEM - F12 (50-50) | Life Technologies | 11330057 | 1x |
| DMSO | Fisher Scientific | BP231-100 | |
| DPBS Ca (-) Mg (-) | Genesse Scientific | 25-508 | |
| EGF | Peprotech | AF100-15 | 50 ng/mL |
| Ethanol, Anhydrous | Fisher Scientific | A405P | Add DI water to dilute to 70% |
| Fisherbrand Class B Amber Glass threaded vials | Fisher Scientific | 03-339-23C | |
| Fisherbrand Weighing Paper | Fisher Scientific | 09-898-12B | |
| G21 Supplement | Gemini Bio | 400-160 | 50x |
| Hanks Balanced Salt Solution | Thermo Fisher Scientific | 14175095 | |
| HCl, ACS, 12M | Sigma Aldrich | S25838A | Add DI water to dilute to 1 M |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H3149-100Ku | 25 µg/mL |
| HEPES | Sigma Aldrich | H7006-100G | |
| Hot Air Gun | Wagner | HT1000 | |
| Integrin-binding sialoprotein (IBSP) peptide | Genscript | Custom Order | GCGYGGGGNGEPRGDTYRAY |
| Lithium phenyl-2,4,6 trimethylbenzoylphosphinate (LAP) , >95% | Sigma Aldrich | 900889-1G | |
| Magnetic stir plate | Thermo Scientific | SP194715 | |
| Microcentrifuge | Thermo Scientific | Sorvall legend micro 21R | |
| Microman E single Channel Pipettor | Gilson | FD10004 | Positive displacement pipette |
| Micropipette Tips | Various Manufacturs | Various sizes | |
| mLine micropipette | Sartorious | ||
| N-acetyl Cysteine | Sigma Aldrich | A7250-10G | |
| Nanowizard 4 | Bruker | AFM microscope | |
| NaOH | Fisher Scientific | ss255-1 | Add DI water to dilute to 1 M |
| Normoicin | Invivogen | ant-nr-1 | 500x |
| Osteopontin Peptide | Genscript | Custom Order | GCGYGTVDVPDGRGDSLAYG |
| Pipet Aid | Drummond | 4000102 | |
| Plain Microscope Slides | Globe Scientific | 1301 | |
| Press-To-Seal silicone Isolator, 12-4.5mm diam x 2mm deep | Grace Bio Labs | 664201-A | Cut so that 8 individual molds are made from a single sheet |
| Processing | Processing | 3.5.4 | |
| Repeater M4 | Eppendorf | 4982000322 | |
| Repeater Pipette Tips | Sartorious | 30089430 | 1 mL sizes |
| RGD Peptide | Genscript | GCGYGRGDSPG | |
| Scoth Tape | |||
| Serological Pipettes | Genesse Scientific | 12-102,12-104 | 5,10 mL Pipettes |
| Solder Paste | Digikey | 315-NC191LT15T5-ND | |
| Solder Wire | |||
| Straight dissecting forceps | VWR Scientific | 82027-408 | |
| Synergy H1 Plate Reader | Biotek | ||
| T-75 Cell Culture Treated Flask | Genesee Scientific | 25-209 | |
| Temozolomide | Sigma Aldrich | T2577 | Typically used from 10 µM to 100 µM |
| Tenascin-C Peptide | Genscript | GCGYGRSTDLPGLKAATHYTITIR GV | |
| Thiolated Hyaluronic Acid (700 kDa), 6-8% modified | Lifecore Biomedical | HA700K5 | |
| VWR Spinbar, Flea Micro | VWR | 58948-375 |
参考文献
- Scannell, J. W., Blanckley, A., Boldon, H., Warrington, B. Diagnosing the decline in pharmaceutical R&D efficiency. Nature Reviews Drug Discovery. 11 (3), 191-200 (2012).
- Waring, M. J., et al. An analysis of the attrition of drug candidates from four major pharmaceutical companies. Nature Reviews Drug Discovery. 14 (7), 475-486 (2015).
- Khozin, S., Liu, K., Jarow, J. P., Pazdur, R. Why do oncology drugs fail to gain US regulatory approval. Nature Reviews Drug Discovery. 14 (7), 450-451 (2015).
- Booth, B., Ma, P., Glassman, R. Oncology's trials. Market indicators. Nature Reviews Drug Discovery. 2 (8), 609-610 (2003).
- Da Ros, M., et al. Glioblastoma chemoresistance: The double play by microenvironment and blood-brain barrier. International Journal of Molecular Sciences. 19 (10), 2879 (2018).
- Broekman, M. L., et al. Multidimensional communication in the microenvirons of glioblastoma. Nature Reviews Neurology. 14 (8), 482-495 (2018).
- Grundy, T. J., et al. Differential response of patient-derived primary glioblastoma cells to environmental stiffness. Scientific Reports. 6 (1), 1-10 (2016).
- Gomez-Roman, N., Stevenson, K., Gilmour, L., Hamilton, G., Chalmers, A. J. A novel 3D human glioblastoma cell culture system for modeling drug and radiation responses. Neuro-Oncology. 19 (2), 229-241 (2017).
- Simoni, R. D., et al. Basement membrane complexes with biological activity. Biochemistry. 25 (2), 312-318 (2002).
- Xiao, W., et al. Brain-mimetic 3D culture platforms allow investigation of cooperative effects of extracellular matrix features on therapeutic resistance in glioblastoma. Cancer Research. 78 (5), 1358-1370 (2018).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nature Reviews Materials. 5 (7), 539-551 (2020).
- Spinelli, C., et al. Molecular subtypes and differentiation programmes of glioma stem cells as determinants of extracellular vesicle profiles and endothelial cell-stimulating activities. Journal of Extracellular Vesicles. 7 (1), 1490144 (2018).
- Ostrom, Q. T., Cioffi, G., Waite, K., Kruchko, C., Barnholtz-Sloan, J. S. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2014-2018. Neuro-Oncology. 23, (2021).
- Stupp, R., et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma. New England Journal of Medicine. 352 (10), 987-996 (2005).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Tomczak, K., Czerwińska, P., Wiznerowicz, M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge. Contemporary oncology. 19, 68-77 (2015).
- Lee, S. Y. Temozolomide resistance in glioblastoma multiforme. Genes and Diseases. 3 (3), 198-210 (2016).
- Joo, K. M., et al. Patient-specific orthotopic glioblastoma xenograft models recapitulate the histopathology and biology of human glioblastomas in situ. Cell Reports. 3 (1), 260-273 (2013).
- Levy, N. The use of animal as models: Ethical considerations. International Journal of Stroke. 7 (5), 440-442 (2012).
- Phon, B. W. S., Kamarudin, M. N. A., Bhuvanendran, S., Radhakrishnan, A. K. Transitioning preclinical glioblastoma models to clinical settings with biomarkers identified in 3D cell-based models: A systematic scoping review. Biomedicine & Pharmacotherapy. 145, 112396 (2022).
- Xiao, W., et al. Bioengineered scaffolds for 3D culture demonstrate extracellular matrix-mediated mechanisms of chemotherapy resistance in glioblastoma. Matrix Biology. 85-86, 128-146 (2020).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Xu, X., Farach-Carson, M. C., Jia, X. Three-dimensional in vitro tumor models for cancer research and drug evaluation. Biotechnology Advances. 32 (7), 1256-1268 (2014).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived Glioblastoma Cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Preston, M. Digestion products of the PH20 hyaluronidase inhibit remyelination. Annals of Neurology. 73 (2), 266-280 (2013).
- Kim, Y., Kumar, S. CD44-mediated adhesion to hyaluronic acid contributes to mechanosensing and invasive motility. Molecular Cancer Research. 12 (10), 1416-1429 (2014).
- Pibuel, M. A., Poodts, D., Díaz, M., Hajos, S. E., Lompardía, S. L. The scrambled story between hyaluronan and glioblastoma. The Journal of Biological Chemistry. 296, 100549 (2021).
- Xiao, W., Sohrabi, A., Seidlits, S. K. Integrating the glioblastoma microenvironment into engineered experimental models. Future Science OA. 3 (3), (2017).
- Trombetta-Lima, M., et al. Extracellular matrix proteome remodeling in human glioblastoma and medulloblastoma. Journal of Proteome Research. 20 (10), 4693-4707 (2021).
- Schregel, K., et al. Characterization of glioblastoma in an orthotopic mouse model with magnetic resonance elastography. NMR in Biomedicine. 31 (10), 3840 (2018).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived glioblastoma cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Guz, N., Dokukin, M., Kalaparthi, V., Sokolov, I. If cell mechanics can be described by elastic modulus: Study of different models and probes used in indentation experiments. Biophysical Journal. 107 (3), 564-575 (2014).
- Sneddon, I. N. The relation between load and penetration in the axisymmetric boussinesq problem for a punch of arbitrary profile. International Journal of Engineering Science. 3 (1), 47-57 (1965).
- Soofi, S. S., Last, J. A., Liliensiek, S. J., Nealey, P. F., Murphy, C. J. The elastic modulus of MatrigelTM as determined by atomic force microscopy. Journal of Structural Biology. 167 (3), 216-219 (2009).
- Mayerhöfer, T. G., Popp, J. Beer's law - Why absorbance depends (almost) linearly on concentration. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 20 (4), 511-515 (2019).
- Puth, M. T., Neuhäuser, M., Ruxton, G. D. On the variety of methods for calculating confidence intervals by bootstrapping. Journal of Animal Ecology. 84 (4), 892-897 (2015).
- Lavrentieva, A. Gradient hydrogels. Advances in Biochemical Engineering/Biotechnology. 178, 227-251 (2020).
- Zhu, D., Trinh, P., Li, J., Grant, G. A., Yang, F. Gradient hydrogels for screening stiffness effects on patient-derived glioblastoma xenograft cellfates in 3D. Journal of Biomedical Materials Research. Part A. 109 (6), 1027-1035 (2021).
- da Hora, C. C., Schweiger, M. W., Wurdinger, T., Tannous, B. A. Patient-derived glioma models: From patients to dish to animals. Cells. 8 (10), 1177 (2019).
- Li, W., et al. Characterization and transplantation of enteric neural crest cells from human induced pluripotent stem cells. Molecular Psychiatry. 23 (3), 499-508 (2018).
- Scaringi, C., Minniti, G., Caporello, P., Enrici, R. M. Integrin inhibitor cilengitide for the treatment of glioblastoma: A brief overview of current clinical results. Anticancer Research. 32 (10), 4213-4224 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。