È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Gli array di idrogel consentono una maggiore produttività per gli effetti di screening dei componenti della matrice e delle terapie nei modelli tumorali 3D
In questo articolo
Riepilogo
Il presente protocollo descrive una piattaforma sperimentale per valutare gli effetti dei segnali meccanici e biochimici sulle risposte chemioterapiche delle cellule di glioblastoma derivate dal paziente in colture 3D matrix-mimetiche utilizzando un dispositivo di illuminazione UV su misura che facilita il fotocrosslinking ad alto rendimento di idrogel con caratteristiche meccaniche sintonizzabili.
Abstract
Le interazioni cellula-matrice mediano processi fisiologici complessi attraverso segnali biochimici, meccanici e geometrici, influenzando i cambiamenti patologici e le risposte terapeutiche. Si prevede che la contabilizzazione degli effetti della matrice nelle prime fasi della pipeline di sviluppo del farmaco aumenterà la probabilità di successo clinico di nuove terapie. Esistono strategie basate su biomateriali che ricapitolano specifici microambienti tissutali in colture cellulari 3D, ma integrarle con i metodi di coltura 2D utilizzati principalmente per lo screening dei farmaci è stata una sfida. Pertanto, il protocollo qui presentato descrive in dettaglio lo sviluppo di metodi per la coltura 3D all'interno di matrici di biomateriali miniaturizzati in un formato di piastra multi-pozzo per facilitare l'integrazione con le pipeline di screening dei farmaci esistenti e i saggi convenzionali per la vitalità cellulare. Poiché si prevede che le caratteristiche della matrice critiche per la conservazione dei fenotipi clinicamente rilevanti nelle cellule coltivate siano altamente specifiche per i tessuti e la malattia, sarà necessario lo screening combinatorio dei parametri della matrice per identificare le condizioni appropriate per applicazioni specifiche. I metodi qui descritti utilizzano un formato di coltura miniaturizzato per valutare le risposte delle cellule tumorali alla variazione ortogonale della meccanica della matrice e della presentazione del ligando. In particolare, questo studio dimostra l'uso di questa piattaforma per studiare gli effetti dei parametri della matrice sulle risposte delle cellule di glioblastoma derivate dal paziente (GBM) alla chemioterapia.
Introduzione
Il costo previsto per lo sviluppo di un nuovo farmaco è aumentato costantemente negli ultimi dieci anni, con oltre $ 1 miliardo nelle stime attuali1. Parte di questa spesa è l'alto tasso di fallimento dei farmaci che entrano negli studi clinici. Circa il 12% dei farmaci candidati alla fine ottiene l'approvazione dalla Food & Drug Administration (FDA) degli Stati Uniti (USA) nel 2019. Molti farmaci falliscono nella fase I a causa della tossicità imprevista2, mentre altri che superano gli studi di sicurezza possono fallire a causa della mancanza di efficacia3. Questo logoramento dovuto alla non efficacia può essere in parte spiegato dal fatto che i modelli di cancro utilizzati durante lo sviluppo del farmaco sono notoriamente non predittivi dell'efficacia clinica4.
Le disparità funzionali tra modelli in vitro e in vivo possono essere attribuite alla rimozione delle cellule tumorali dal loro microambiente nativo, comprese le cellule non tumorali e l'ECM fisico 5,6. Comunemente, i gruppi di ricerca utilizzano matrici di coltura disponibili in commercio, come Matrigel (una matrice di membrana basale proteica derivata da sarcomi di topo) per fornire alle cellule tumorali in coltura un microambiente a matrice 3D. Rispetto alla coltura 2D, la coltura 3D nella matrice di membrana ha migliorato la rilevanza clinica dei risultati in vitro 7,8. Tuttavia, i biomateriali di coltura da tessuti decellularizzati, compresa la matrice di membrana, in genere mostrano una variabilità da lotto a lotto che può compromettere la riproducibilità9. Inoltre, le matrici derivate da tumori con origini tissutali diverse da quelle studiate potrebbero non fornire gli opportuni spunti fisiologici10. Infine, i tumori con alti gradi di eterogeneità intratumorale hanno caratteristiche microambientali che variano su una scala di dimensioni submicroniche e che la matrice di membrana non può essere sintonizzata per ricapitolare11.
Il glioblastoma (GBM), un tumore cerebrale uniformemente letale con un tempo di sopravvivenza mediano di circa 15 mesi, è un tumore per il quale lo sviluppo del trattamento è stato particolarmente difficile12,13. L'attuale standard di cura per GBM consiste nella resezione primaria del tumore, seguita dalla radioterapia e quindi dalla chemioterapia con temozolomide (TMZ)14. Tuttavia, più della metà dei tumori GBM clinici mostra resistenza al trattamento attraverso vari meccanismi 15,16,17. Prevedere l'efficacia di un regime di trattamento per un singolo paziente è estremamente difficile. I modelli preclinici standard utilizzati per prevedere i risultati individuali sono costituiti da cellule tumorali derivate dal paziente che si sono deformate ortotopicamente in topi immunocompromessi. Mentre gli xenotrapianti derivati dal paziente possono ricapitolare molti aspetti dei tumori GBM clinici e sono preziosi per i modelli preclinici18, sono intrinsecamente costosi, a bassa produttività, richiedono molto tempo e comportano preoccupazioni etiche19. Le colture di cellule derivate dal paziente, su superfici di plastica 2D o come sferoidi, per lo più evitano questi problemi. Mentre le cellule derivate dal paziente conservano le aberrazioni genetiche, le loro colture in 2D o come sferoidi sospesi sono state in gran parte rappresentazioni scadenti di xenotrapianti derivati dal paziente nei roditori e nei tumori originali dei pazienti20. In precedenza, noi e altri abbiamo dimostrato che le cellule GBM coltivate in un ECM 3D che imita le proprietà meccaniche e biochimiche del tessuto cerebrale possono preservare i fenotipi di resistenza ai farmaci 10,21,22,23.
Le interazioni tra l'acido ialuronico (HA), un polisaccaride abbondante nell'ECM cerebrale e sovraespresso nei tumori GBM, e il suo recettore CD44 modulano l'acquisizione della resistenza ai farmaci in vitro 21,24,25,26,27. Ad esempio, l'inclusione di HA all'interno di colture 3D morbide ha aumentato la capacità delle cellule GBM derivate dal paziente di acquisire resistenza terapeutica. Questa meccano-responsività dipendeva dal legame dell'HA ai recettori CD44 sulle cellule GBM21. Inoltre, il legame dell'integrina ai peptidi portatori di RGD, incorporato in matrici di coltura 3D, ha amplificato la chemioresistenza mediata da CD44 in modo dipendente dalla rigidità21. Oltre all'HA, l'espressione di diverse proteine ECM, molte contenenti regioni RGD, varia tra il cervello normale e i tumori GBM28. Ad esempio, uno studio ha riportato che 28 distinte proteine ECM erano sovraregolate nei tumori GBM29. All'interno di questo complesso microambiente a matrice tumorale, le cellule tumorali integrano segnali meccanici e biochimici per produrre un particolare fenotipo di resistenza, che dipende da differenze relativamente piccole (ad esempio, meno di un ordine di grandezza) nel modulo di Young o densità di peptidi leganti l'integrina 28,29,30.
Il presente protocollo caratterizza il modo in cui le cellule tumorali interpretano combinazioni uniche di segnali matriciali e identificano microambienti a matrice complessi e specifici per il paziente che promuovono la resistenza al trattamento (Figura 1A). Un metodo fotochimico per generare matrici miniaturizzate e sintonizzate con precisione per la coltura 3D fornisce un ampio spazio variabile ortogonale. Un array di LED costruito su misura, gestito da un microcontrollore, è stato incorporato per fotocrosslink idrogel all'interno di un formato di piastra a 384 pozzetti per aumentare l'automazione e la riproducibilità. L'intensità dell'esposizione è stata variata in modo variabile per alterare le proprietà micromeccaniche degli idrogel risultanti, come valutato utilizzando la microscopia a forza atomica (AFM). Mentre questo manoscritto non si concentra sulla costruzione dell'array di illuminazione stesso, uno schema elettrico (Figura 1B) e un elenco di parti (Tabella dei materiali) sono forniti come ausili per la riproduzione del dispositivo.
Questo rapporto dimostra la rapida generazione di una serie di cellule GBM coltivate in microambienti 3D unici in cui il modulo di Young (quattro livelli in un singolo ordine di grandezza) e il contenuto peptidico legante l'integrina (derivato da quattro diverse proteine ECM) sono stati variati ortogonalmente. L'approccio è stato poi utilizzato per studiare i contributi relativi della meccanica dell'idrogel e dell'impegno dell'integrina specifica dell'ECM sulla vitalità e la proliferazione delle cellule GBM derivate dal paziente mentre acquisiscono resistenza alla chemioterapia temozolomide (TMZ).
Access restricted. Please log in or start a trial to view this content.
Protocollo
Le linee cellulari GBM derivate dal paziente (GS122 e GS304) sono state fornite dal professor David Nathanson (nostro collaboratore), che ha sviluppato queste linee secondo un protocollo approvato dall'UCLA Institutional Review Board (IRB # 10-000655). Le cellule sono state fornite de-identificate in modo che le linee cellulari non potessero essere ricollegate ai singoli pazienti.
1. Preparazione della soluzione di idrogel
- Preparare la soluzione tamponata con HEPES sciogliendo la polvere di HEPES a 20 mM nella soluzione salina bilanciata (HBSS) di Hank. Regolare il pH a 7 dopo la completa solvatazione.
- Nella soluzione tamponata con HEPES, sciogliere l'HA tiolato (peso molecolare nominale di 700 kDa, vedere Tabella dei materiali), preparato seguendo la precedente relazione31, in modo che il 6%-8% dei residui di acido carbossilico su ciascun acido glucuronico siano modificati con un tiolo, ad una concentrazione di 10 mg/mL in soluzione tampone.
NOTA: Si raccomanda un flaconcino ambrato per prevenire l'ossidazione del tiolo da parte della luce ambientale.- Mescolare usando una piastra magnetica (<1.000 giri / min) a temperatura ambiente fino a completa dissoluzione, in genere intorno ai 45 minuti.
- Durante la dissoluzione dell'HA, preparare soluzioni separate di (1) 100 mg/mL di PEG-Norbornene a 8 bracci (20 kDa), (2) 100 mg/mL di PEG-tiolo a 4 bracci (20 kDa), (3) 4 mM di peptide contenente cisteina o cisteina (ad esempio, GCGYGRGDSPG) e (4) 4 mg/mL di LAP in tubi di microcentrifuga (vedere Tabella dei materiali).
- Preparare ciascuna di queste quattro soluzioni nella soluzione con buffer HEPES preparata nel passaggio 1.1. Vortex le soluzioni per garantire la completa dissoluzione di ciascun reagente prima di eseguire la fase 4.
NOTA: Se si testano più peptidi diversi, ognuno deve contenere una cisteina o un'altra fonte di tiolo per questa chimica di coniugazione. - Preparare soluzioni (4 mM di tiolo disponibile) di tutti i peptidi da legare all'interno di un singolo idrogel a questo punto.
NOTA: Le sequenze peptidiche e le proteine ECM da cui sono state derivate e utilizzate in questo studio sono elencate nella Tabella 1. La N-acetil cisteina (vedi Tabella dei materiali), a cui le cellule non si legano, può essere sostituita da un peptide bioattivo contenente tiolo per titolare la concentrazione di un peptide adesivo o agire come controllo negativo31.
- Preparare ciascuna di queste quattro soluzioni nella soluzione con buffer HEPES preparata nel passaggio 1.1. Vortex le soluzioni per garantire la completa dissoluzione di ciascun reagente prima di eseguire la fase 4.
- Mescolare le singole soluzioni di HA, PEG-Norbornene, PEG-tiolo e peptidi contenenti cisteina/tiolo (vedere Tabella dei materiali) per ottenere le concentrazioni finali per le matrici finali di idrogel elencate nella Tabella 2. Mescolare (<1.000 giri /min) su una piastra magnetica per almeno 30 minuti per mescolare completamente.
NOTA: le soluzioni HA sono altamente viscose e meglio gestite utilizzando una pipetta a spostamento positivo (vedere Tabella dei materiali). Se una pipetta a spostamento positivo non è disponibile, le soluzioni viscose possono anche essere eliminate con una micropipetta standard mediante il pipettaggio lento utilizzando punte a orifizio largo.
2. Illuminazione e fotocrosslinking di idrogel tramite un array di LED
ATTENZIONE: Indossare occhiali protettivi UV e coprire il campo di illuminazione con materiale che assorbe i raggi UV.
NOTA: l'array di LED descritto in questo protocollo è costituito da sei set di otto LED disposti in serie, come illustrato dallo schema elettrico fornito (Figura 1A). Ogni set di LED può essere alimentato in modo indipendente, il che consente fino a sei diversi irraggiamenti per corsa. Il file 1 supplementare contiene schermate corrispondenti alle seguenti istruzioni per ulteriori indicazioni.
- Scaricare il file Illumination Device.zip dai file di codifica supplementari. Questa directory contiene i seguenti file: Arduino.zip (Supplementary Coding File 1), Drivers.zip (Supplementary Coding File 2), GUI.zip (Supplementary Coding File 3) e Holder.zip (Supplementary Coding File 4).
NOTA: stampa 3D delle parti superiore e inferiore per tenere in posizione il circuito stampato (vedere File di codifica supplementari per i dettagli). - Scaricare e installare il software del microcontrollore (vedere Tabella dei materiali).
- Scaricare e installare il software GUI (vedere Tabella dei materiali). Fare riferimento al file supplementare 1 per le istruzioni per l'uso del software.
- Apri Elaborazione e installa la libreria controlIP5 facendo clic su Sketch > Importa libreria > Aggiungi libreria. Quindi, cerca controlIP5 nelle librerie e fai clic su Installa. Eseguilo per la prima volta.
- Alimentare il dispositivo di illuminazione (vedere Tabella dei materiali) utilizzando l'alimentatore da 36 Volt e collegarlo a un PC tramite un cavo micro-USB.
NOTA: alcuni dispositivi non installeranno automaticamente i driver per varie schede nano Arduino. Un set di driver è fornito nel file zip del dispositivo. - Aprire il file Arduino.ino, che si trova nella cartella Adruino.zip, utilizzando Arduino IDE.
- Compila il file Arduino.ino cliccando sul pulsante Segno di spunta . Carica il codice compilato facendo clic sul pulsante Freccia .
- Aprire il file GUI.pde, che si trova nella cartella GUI.zip, utilizzando Elaborazione.
- Fare clic su Esegui nel programma di elaborazione per avviare l'interfaccia utente grafica per il controllo del dispositivo di illuminazione.
- Nella finestra dell'interfaccia utente grafica, fare clic su Intensità per la colonna contenente la soluzione precursore dell'idrogel da reticolare e inserire l'intensità desiderata. Fare clic sulla casella Ora e inserire l'ora desiderata. Per la soluzione fornita nella tabella 2, questo sarà 15 s.
NOTA: gli utenti finali devono calibrare i valori di intensità digitale in base all'irradianza utilizzando un radiometro. Esempi di intensità tipiche sono forniti nella Figura 2A. - Allineare i campioni con il dispositivo di illuminazione (Figura 2B) con ogni altro LED in una singola colonna degli stampi in silicone (vedere Tabella dei materiali) o piastra a 384 pozzetti. Fare clic su Fine per iniziare l'illuminazione. Ripetere questo processo se necessario per l'illuminazione di più diapositive o altri pozzetti di una piastra a 384 pozzetti.
NOTA: il supporto è progettato in modo tale che la piastra a 384 pozzetti si trovi a filo con un angolo della camera interna durante l'illuminazione.- Dopo l'illuminazione, quando viene posizionato in un angolo, spostare la piastra del pozzo nell'angolo successivo e ripetere. Per illuminare i pozzetti sull'altra metà della piastra, sollevare la piastra dal supporto e ruotare di 180°.
- Generare idrogel con meccanica variabile per la caratterizzazione meccanica seguendo i passaggi seguenti.
- Pulire le diapositive di vetro e gli stampi in silicone usando del nastro adesivo per rimuovere i detriti. Aderire gli stampi in silicone alla slitta di vetro, premere verso il basso per garantire una buona tenuta e spostare eventuali bolle d'aria.
- Pipettare 80 μL di soluzione precursore di idrogel, come preparato nella fase 1.4, in ogni stampo in silicone sul vetrino.
- Posizionare la slitta di vetro sul dispositivo di illuminazione allineato con ogni altro LED in una singola colonna. Esporre i precursori dell'idrogel alla luce UV per 15 s, come descritto nel passaggio 2, al photocrosslink.
- Una volta che l'illuminazione si è fermata, recuperare i vetrini e allentare i gel dagli stampi tracciando la circonferenza interna dello stampo con una punta fine (punta della pipetta da 10 μL, ago da 30 G, ecc.). Rimuovere gli stampi in silicone con pinzette / pinze.
- Spostare idrogel reticolati in singoli pozzetti di una piastra a 12 pozzetti bagnando una spatola e spingendoli delicatamente fuori dal vetrino. Riempire ogni pozzetto con 2 ml di DPBS (vedi Tabella dei materiali) prima di aggiungere l'idrogel. Gonfiare i gel in soluzione dpbs per almeno 12 ore (in genere durante la notte) a temperatura ambiente (per la caratterizzazione meccanica del giorno successivo).
3. Misure di microscopia a forza atomica (AFM)
- Accendere il microscopio a forza atomica (AFM) secondo le istruzioni del produttore (vedere Tabella dei materiali). Questo protocollo fornisce brevi istruzioni per l'utilizzo dello strumento e del relativo software.
- Installare la sonda AFM (vedere Tabella dei materiali).
NOTA: Per il presente studio, un cantilever triangolare in nitruro di silicio con una costante nominale della molla di 0,01 N/m è stato modificato con una particella sferica di biossido di silicio da 2,5 μm. - Dopo l'installazione, allineare il laser all'apice della sonda triangolare, quindi regolare la deflessione dello specchio e del laser per massimizzare la somma del segnale (in genere tra 1,5-2,2 Volt).
- Immergere la sonda in DPBS e attendere fino a 15 minuti per ottenere l'equilibrio termico. Fare clic sul pulsante Calibrazione e selezionare Calibrazione dipendente dal contatto. Fare clic sul pulsante Raccogli sintonizzazione termica e, dopo la raccolta dei dati, selezionare il picco intorno ai 3 kHz per la calibrazione.
NOTA: una leggera regolazione dello specchio e dei deflettori laser può essere necessaria dopo l'immersione in un liquido a causa di variazioni dell'indice di rifrazione. - Avvicinati alla superficie di una capsula di Petri (plastica) impostando i parametri di approccio su Velocità costante, un'altezza target di 7,5 μm e una velocità di avvicinamento di 15 μm/s. Abilitare la misurazione di base per run for approach in modo che l'approccio venga eseguito continuamente e non si fermi presto a causa della deriva nel deflettore.
- All'avvicinamento, impostare i parametri di acquisizione per la mappatura della forza su turnaround di 4 nN, distanza di rientro di 2 μm, velocità di 1 μm/s e tempo di contatto di 0 s. Premere il pulsante Start per iniziare a raccogliere una curva di forza sulla superficie di plastica (ad esempio, una piastra del pozzo).
- Tornate alla finestra di calibrazione e selezionate la porzione della curva di forza corrispondente al contatto e alla rientranza della plastica. Accettate i valori di sensibilità e rigidità calcolati affinché la sonda completi la calibrazione.
- Dopo la calibrazione, sollevare la sonda AFM e posizionare il campione di idrogel per l'interrogazione. Avvicinarsi all'idrogel seguendo le impostazioni fornite nel passaggio 5.
NOTA: durante la procedura di avvicinamento verso la superficie dell'idrogel, l'unità potrebbe erroneamente attivare lo stato avvicinato. Per verificare l'approccio effettivo, ottenere una curva di forza come nel passaggio 4.6. Ripetete la procedura di avvicinamento se la curva risultante non mostra il contatto e il rientro risultante. - Quando l'approccio superficiale ha esito positivo, passare alla modalità Force Mapping e impostare i parametri di acquisizione su una mappa di dimensioni 8 x 8 con una lunghezza di 40 μm per asse. Ottenere mappe di forza in varie regioni per valutare l'uniformità delle misurazioni di rigidità.
- Interpretare le curve di forza utilizzando il programma software JPK SPM Data Processing attraverso un adattamento del modello Hertz/Sneddon (equazioni 1 e 2, vedere tabella 3 per la definizione di tutte le variabili) con la geometria sferica selezionata 32,33,3 4.
 Equazione 132
Equazione 132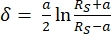 Equazione 232
Equazione 232
- Interpretare le curve di forza utilizzando il programma software JPK SPM Data Processing attraverso un adattamento del modello Hertz/Sneddon (equazioni 1 e 2, vedere tabella 3 per la definizione di tutte le variabili) con la geometria sferica selezionata 32,33,3 4.
4. Impostazione e trattamento farmacologico di colture 3D, matrix-embedded
- Preparare le cellule desiderate come soluzione a cella singola.
NOTA: diversi tipi di celle possono richiedere metodi di passaggio diversi. Un protocollo tipico per il passaggio di una coltura in sospensione di sferoidi GBM da un pallone T-75 è riportato nel riferimento31. - Raccogliere sferoidi GBM (circa 150 μm di diametro) da una coltura di sospensione del pallone T-75 in un tubo conico da 15 ml. Risciacquare il matraccio di coltura con 5 ml di DPBS per rimuovere eventuali cellule e mezzi residui e aggiungere questo volume al tubo conico.
- Centrifugare il tubo conico contenente le celle a 200 x g per 5 minuti a temperatura ambiente. Dopo la centrifugazione, rimuovere il surnatante con una pipetta sierologica da 5 ml, facendo attenzione a non disturbare il pellet cellulare, e risospendere in 5 ml di DPBS.
- Centrifugare a 200 x g per 5 minuti a temperatura ambiente per lavare le celle. Aspirare il surnatante con una pipetta sierologica da 5 ml, avendo cura di non disturbare il pellet cellulare, e quindi risospese le cellule in 2 mL di reagente di dissociazione cellulare (vedi Tabella dei materiali).
- Incubare a temperatura ambiente per 10-15 min. Aggiungere 3 ml di mezzo completo (vedere Tabella dei materiali) e pipettare delicatamente 3-5 volte per abbattere gli sferoidi in una sospensione a cella singola31.
- Centrifugare la sospensione monocellulare a 400 x g (le sospensioni monocellulari possono essere filate più velocemente per la formazione di pellet) per 5 minuti in celle a pellet a temperatura ambiente. Aspirare il surnatante con una pipetta sierologica da 5 ml, avendo cura di non disturbare il pellet cellulare. Sostituire le cellule in 1 mL di terreno completo.
NOTA: Se le cellule rimangono in grumi, piuttosto che come singole cellule in sospensione, dopo il passaggio, le cellule possono essere fatte passare attraverso un filtro cellulare da 40 μm per ottenere una sospensione a singola cellula. - Rimuovere una parte delle cellule per il conteggio utilizzando un emocitometro. Diluire questa porzione due volte con tripano blu, che permea le cellule con vitalità compromessa. Conta solo le cellule vive e incolori. Tipicamente, un T-75 seminato a 800.000 cellule per pallone produce 2-3 milioni di cellule dopo una settimana in coltura.
- Determinare il numero di celle necessarie per l'incapsulamento. Trasferire un volume di fluido contenente il numero totale di cellule necessarie in un tubo microcentrifuga sterile da 1,7 mL. Girare verso il basso a 400 x g per 5 minuti a temperatura ambiente.
NOTA: Ad esempio, è necessario un minimo di 2,5 milioni di cellule risospese in 1 mL di volume di gel per incapsulare le cellule a 2,5 milioni di cellule / mL. Un volume di gel di 1 mL consente agli utenti di erogare 100 gocce di gel, dove ogni goccia di gel è di 10 μL di volume. Si consiglia di preparare un volume extra di circa il 20% di celle sospese in soluzione di idrogel per tenere conto della perdita durante il trasferimento della pipetta. Quindi, si preparerebbero 3 milioni di cellule e 1,2 ml di soluzione precursore dell'idrogel in questo esempio. Si raccomanda una densità minima di 500 mila celle/ml. - Aspirare il surnatante con una micropipetta, avendo cura di non disturbare il pellet cellulare. Risospesso il pellet di cella nella soluzione precursore dell'idrogel, come preparato nella fase 1.4, mescolando bene pipettando su e giù con una micropipetta da 1.000 μL 4-5 volte.
- Caricare le celle in un pipettor ripetuto (vedere Tabella dei materiali) impostato per erogare 10 μL. Per evitare bolle e erogazione irregolare, innescare il pipettor ripetuto erogando altre 1-2 volte in un contenitore per rifiuti.
- In ogni pozzetto di una piastra da 384 pozzetti, erogare 10 μL di cellule sospese in soluzione di idrogel dal pipettor ripetuto. Utilizzando l'array di LED, illuminare ogni pozzo contenente celle (fase 2) per 15 s con intensità (esempio risultati in Figura 2A intensità utilizzate di 1,14, 1,55, 2,15, 2,74 mW / cm2) per ottenere le proprietà meccaniche desiderate.
NOTA: si consiglia di iniziare con cinque repliche per condizione sperimentale e scalare verso l'alto o verso il basso a seconda del throughput desiderato e della varianza del test dell'endpoint. - Aggiungere 40 μL di media completi a ciascun pozzo contenente le cellule. Aggiungere 50 μL di DPBS a pozzi asciutti non sperimentali che circondano i gel per ridurre al minimo le perdite dovute all'evaporazione.
- Per le cellule GBM, aggiungere 40 μL del farmaco contenente fluidi (ad esempio, TMZ, vedere Tabella dei materiali) per ottenere la concentrazione finale desiderata (10 μM-100 μM in dimetilsolfossido (DMSO) o veicolo (DMSO), di conseguenza, a partire da 3 giorni dopo l'incapsulamento.
5. Saggio di proliferazione CCK8
- Aggiungere 10 μL di reagente CCK8 (vedi Tabella dei materiali) a ciascun pozzetto contenente le cellule.
NOTA: se si esegue questo test per la prima volta, includere pozzetti di controllo negativi come solo media o idrogel privo di cellule nei media. - Incubare per 1-4 ore secondo le istruzioni del produttore.
NOTA: Questo tempo può variare in funzione del tipo di cellula e della densità, e quindi i tempi di incubazione devono essere testati per ogni applicazione in modo che i valori di assorbanza rientrino in un intervallo lineare, un requisito per l'applicazione della legge35 di Beer. - Leggere le assorbanze a 450 nm per tutti i pozzetti successivi all'incubazione.
- Calcolare l'assorbanza media a 450 nm ottenuta nella fase 3 per le condizioni del veicolo per ciascun gruppo. Dividere bene ogni farmaco trattato per la media del controllo del veicolo per gruppo.
- Calcola gli intervalli di confidenza generando distribuzioni bootstrap (N = 10.000) attraverso il metodo percentile36.
NOTA: Generalmente, si possono utilizzare intervalli di confidenza del 95% e interpretare le condizioni i cui intervalli di confidenza non attraversano 1 per essere significative e giustificare ulteriori indagini. L'impostazione degli intervalli di confidenza al 95% è congruente con l'impostazione di un limite di significatività di p = 0,05. Per i dati mostrati nei risultati, c'è utilità nel distinguere le condizioni che promuovono o inibiscono la resistenza ai farmaci mediata dalla matrice, richiedendo un'analisi a due lati.
Access restricted. Please log in or start a trial to view this content.
Risultati
Le misurazioni AFM hanno confermato il controllo preciso della meccanica dell'idrogel in funzione dell'irraggiamento UV (mW/cm2) durante la fotorelazione utilizzando un array di LED personalizzato e controllato da Arduino (Figura 2A). La formulazione in idrogel utilizzata in questo protocollo può essere trovata nella Tabella 2. La spaziatura dei LED sul modello fornito corrisponde alla spaziatura per ogni altro pozzo di una piastra a 384 pozzetti, consentendo la ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il lavoro attuale presenta metodi per generare colture miniaturizzate 3D all'interno di HA, alterando contemporaneamente la rigidità della matrice e i peptidi disponibili per l'impegno dell'integrina. Questa tecnica consente lo studio sistematico di come i parametri della matrice influenzano i fenotipi cellulari (ad esempio, la vitalità delle cellule tumorali esposte alla chemioterapia) con una maggiore produttività. Gli approcci precedenti, incluso quello qui presentato, hanno regolato la rigidità dell'idrogel varia...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori vorrebbero ringraziare specificamente Carolyn Kim, Amelia Lao, Ryan Stoutamore e Itay Solomon per i loro contributi alle precedenti iterazioni dello schema di fotogelazione. Le linee cellulari GS122 e GS304 sono state generosamente fornite da David Nathanson. Tutte le figure sono state create con BioRender.com. Le strutture principali dell'UCLA, le risorse condivise di screening molecolare e il laboratorio di caratterizzazione nano e pico sono stati fondamentali per il lavoro. Chen Chia-Chun è stato supportato dall'UCLA Eli and Edythe Broad Center of Regenerative Medicine and Stem Cell Research Training Program. Grigor Varuzhanyan è stato supportato da un tumor cell biology training program NIH Grant (T32 CA 009056).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.1 kOhm resistors, 6 W | Digikey | 35601k1ft | |
| 1.7 mL microcentrifuge tube | Genesse Scientific | 21-108 | |
| 15 mL conical tube | Fisher Scientific | 14-959-70C | |
| 365 nm LED | Digikey | ltpl-c034uvh365 | |
| 384 well plate | Bio Greiner One | 781090 | |
| 40 µm cell strainer | MTC bio | C4040 | |
| 4-Armed thiol terminated polyethlene glycol (20 kDa) | Laysan Bio | 4arm-PEG-SH-20K-1g | |
| 6 NPN BJTs | Digikey | 2n5550ta | |
| 80 Ohm resistors, 0.125 W | Digikey | erjj-6enf80r6v | |
| 8-Armed norbornene terminated polyethylene glycol (20 kDa) | Jenkem Technology | A7025-1 | |
| Accutase | Innovative Cell Technologies | AT104500 | cell dissociation reagent |
| AFM Probes | Novascan | 0.01 N/m Nominal spring constant, 2.5 µm SiO2 particle | |
| Arduino IDE | Arduino | 1.8.19 | |
| Arduino Nano | Makerfire | Mini Nano V3.0 ATmega328P Microcontroller Board | |
| bFGF | Peprotech | 100-18B | 20 ng/mL |
| CCK8 | Abcam | ab228554 | |
| Centrifuge | Thermoscientific | sorvall legend xtr | |
| CP100ST | Gilson | F148415 | Pipette tips for positive displacement pipette |
| Cubis Semi-Micro Balance | Sartorius | MSA225S100DI | |
| DMEM - F12 (50-50) | Life Technologies | 11330057 | 1x |
| DMSO | Fisher Scientific | BP231-100 | |
| DPBS Ca (-) Mg (-) | Genesse Scientific | 25-508 | |
| EGF | Peprotech | AF100-15 | 50 ng/mL |
| Ethanol, Anhydrous | Fisher Scientific | A405P | Add DI water to dilute to 70% |
| Fisherbrand Class B Amber Glass threaded vials | Fisher Scientific | 03-339-23C | |
| Fisherbrand Weighing Paper | Fisher Scientific | 09-898-12B | |
| G21 Supplement | Gemini Bio | 400-160 | 50x |
| Hanks Balanced Salt Solution | Thermo Fisher Scientific | 14175095 | |
| HCl, ACS, 12M | Sigma Aldrich | S25838A | Add DI water to dilute to 1 M |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H3149-100Ku | 25 µg/mL |
| HEPES | Sigma Aldrich | H7006-100G | |
| Hot Air Gun | Wagner | HT1000 | |
| Integrin-binding sialoprotein (IBSP) peptide | Genscript | Custom Order | GCGYGGGGNGEPRGDTYRAY |
| Lithium phenyl-2,4,6 trimethylbenzoylphosphinate (LAP) , >95% | Sigma Aldrich | 900889-1G | |
| Magnetic stir plate | Thermo Scientific | SP194715 | |
| Microcentrifuge | Thermo Scientific | Sorvall legend micro 21R | |
| Microman E single Channel Pipettor | Gilson | FD10004 | Positive displacement pipette |
| Micropipette Tips | Various Manufacturs | Various sizes | |
| mLine micropipette | Sartorious | ||
| N-acetyl Cysteine | Sigma Aldrich | A7250-10G | |
| Nanowizard 4 | Bruker | AFM microscope | |
| NaOH | Fisher Scientific | ss255-1 | Add DI water to dilute to 1 M |
| Normoicin | Invivogen | ant-nr-1 | 500x |
| Osteopontin Peptide | Genscript | Custom Order | GCGYGTVDVPDGRGDSLAYG |
| Pipet Aid | Drummond | 4000102 | |
| Plain Microscope Slides | Globe Scientific | 1301 | |
| Press-To-Seal silicone Isolator, 12-4.5mm diam x 2mm deep | Grace Bio Labs | 664201-A | Cut so that 8 individual molds are made from a single sheet |
| Processing | Processing | 3.5.4 | |
| Repeater M4 | Eppendorf | 4982000322 | |
| Repeater Pipette Tips | Sartorious | 30089430 | 1 mL sizes |
| RGD Peptide | Genscript | GCGYGRGDSPG | |
| Scoth Tape | |||
| Serological Pipettes | Genesse Scientific | 12-102,12-104 | 5,10 mL Pipettes |
| Solder Paste | Digikey | 315-NC191LT15T5-ND | |
| Solder Wire | |||
| Straight dissecting forceps | VWR Scientific | 82027-408 | |
| Synergy H1 Plate Reader | Biotek | ||
| T-75 Cell Culture Treated Flask | Genesee Scientific | 25-209 | |
| Temozolomide | Sigma Aldrich | T2577 | Typically used from 10 µM to 100 µM |
| Tenascin-C Peptide | Genscript | GCGYGRSTDLPGLKAATHYTITIR GV | |
| Thiolated Hyaluronic Acid (700 kDa), 6-8% modified | Lifecore Biomedical | HA700K5 | |
| VWR Spinbar, Flea Micro | VWR | 58948-375 |
Riferimenti
- Scannell, J. W., Blanckley, A., Boldon, H., Warrington, B. Diagnosing the decline in pharmaceutical R&D efficiency. Nature Reviews Drug Discovery. 11 (3), 191-200 (2012).
- Waring, M. J., et al. An analysis of the attrition of drug candidates from four major pharmaceutical companies. Nature Reviews Drug Discovery. 14 (7), 475-486 (2015).
- Khozin, S., Liu, K., Jarow, J. P., Pazdur, R. Why do oncology drugs fail to gain US regulatory approval. Nature Reviews Drug Discovery. 14 (7), 450-451 (2015).
- Booth, B., Ma, P., Glassman, R. Oncology's trials. Market indicators. Nature Reviews Drug Discovery. 2 (8), 609-610 (2003).
- Da Ros, M., et al. Glioblastoma chemoresistance: The double play by microenvironment and blood-brain barrier. International Journal of Molecular Sciences. 19 (10), 2879(2018).
- Broekman, M. L., et al. Multidimensional communication in the microenvirons of glioblastoma. Nature Reviews Neurology. 14 (8), 482-495 (2018).
- Grundy, T. J., et al. Differential response of patient-derived primary glioblastoma cells to environmental stiffness. Scientific Reports. 6 (1), 1-10 (2016).
- Gomez-Roman, N., Stevenson, K., Gilmour, L., Hamilton, G., Chalmers, A. J. A novel 3D human glioblastoma cell culture system for modeling drug and radiation responses. Neuro-Oncology. 19 (2), 229-241 (2017).
- Simoni, R. D., et al. Basement membrane complexes with biological activity. Biochemistry. 25 (2), 312-318 (2002).
- Xiao, W., et al. Brain-mimetic 3D culture platforms allow investigation of cooperative effects of extracellular matrix features on therapeutic resistance in glioblastoma. Cancer Research. 78 (5), 1358-1370 (2018).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nature Reviews Materials. 5 (7), 539-551 (2020).
- Spinelli, C., et al. Molecular subtypes and differentiation programmes of glioma stem cells as determinants of extracellular vesicle profiles and endothelial cell-stimulating activities. Journal of Extracellular Vesicles. 7 (1), 1490144(2018).
- Ostrom, Q. T., Cioffi, G., Waite, K., Kruchko, C., Barnholtz-Sloan, J. S. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2014-2018. Neuro-Oncology. 23, Supplement_3 (2021).
- Stupp, R., et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma. New England Journal of Medicine. 352 (10), 987-996 (2005).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Tomczak, K., Czerwińska, P., Wiznerowicz, M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge. Contemporary oncology. 19, Poznan, Poland. 68-77 (2015).
- Lee, S. Y. Temozolomide resistance in glioblastoma multiforme. Genes and Diseases. 3 (3), 198-210 (2016).
- Joo, K. M., et al. Patient-specific orthotopic glioblastoma xenograft models recapitulate the histopathology and biology of human glioblastomas in situ. Cell Reports. 3 (1), 260-273 (2013).
- Levy, N. The use of animal as models: Ethical considerations. International Journal of Stroke. 7 (5), 440-442 (2012).
- Phon, B. W. S., Kamarudin, M. N. A., Bhuvanendran, S., Radhakrishnan, A. K. Transitioning preclinical glioblastoma models to clinical settings with biomarkers identified in 3D cell-based models: A systematic scoping review. Biomedicine & Pharmacotherapy. 145, 112396(2022).
- Xiao, W., et al. Bioengineered scaffolds for 3D culture demonstrate extracellular matrix-mediated mechanisms of chemotherapy resistance in glioblastoma. Matrix Biology. 85-86, 128-146 (2020).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744(2020).
- Xu, X., Farach-Carson, M. C., Jia, X. Three-dimensional in vitro tumor models for cancer research and drug evaluation. Biotechnology Advances. 32 (7), 1256-1268 (2014).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived Glioblastoma Cells. Journal of Visualized Experiments: JoVE. (138), e58176(2018).
- Preston, M. Digestion products of the PH20 hyaluronidase inhibit remyelination. Annals of Neurology. 73 (2), 266-280 (2013).
- Kim, Y., Kumar, S. CD44-mediated adhesion to hyaluronic acid contributes to mechanosensing and invasive motility. Molecular Cancer Research. 12 (10), 1416-1429 (2014).
- Pibuel, M. A., Poodts, D., Díaz, M., Hajos, S. E., Lompardía, S. L. The scrambled story between hyaluronan and glioblastoma. The Journal of Biological Chemistry. 296, 100549(2021).
- Xiao, W., Sohrabi, A., Seidlits, S. K. Integrating the glioblastoma microenvironment into engineered experimental models. Future Science OA. 3 (3), (2017).
- Trombetta-Lima, M., et al. Extracellular matrix proteome remodeling in human glioblastoma and medulloblastoma. Journal of Proteome Research. 20 (10), 4693-4707 (2021).
- Schregel, K., et al. Characterization of glioblastoma in an orthotopic mouse model with magnetic resonance elastography. NMR in Biomedicine. 31 (10), 3840(2018).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived glioblastoma cells. Journal of Visualized Experiments: JoVE. (138), e58176(2018).
- Guz, N., Dokukin, M., Kalaparthi, V., Sokolov, I. If cell mechanics can be described by elastic modulus: Study of different models and probes used in indentation experiments. Biophysical Journal. 107 (3), 564-575 (2014).
- Sneddon, I. N. The relation between load and penetration in the axisymmetric boussinesq problem for a punch of arbitrary profile. International Journal of Engineering Science. 3 (1), 47-57 (1965).
- Soofi, S. S., Last, J. A., Liliensiek, S. J., Nealey, P. F., Murphy, C. J. The elastic modulus of MatrigelTM as determined by atomic force microscopy. Journal of Structural Biology. 167 (3), 216-219 (2009).
- Mayerhöfer, T. G., Popp, J. Beer's law - Why absorbance depends (almost) linearly on concentration. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 20 (4), 511-515 (2019).
- Puth, M. T., Neuhäuser, M., Ruxton, G. D. On the variety of methods for calculating confidence intervals by bootstrapping. Journal of Animal Ecology. 84 (4), 892-897 (2015).
- Lavrentieva, A. Gradient hydrogels. Advances in Biochemical Engineering/Biotechnology. 178, 227-251 (2020).
- Zhu, D., Trinh, P., Li, J., Grant, G. A., Yang, F. Gradient hydrogels for screening stiffness effects on patient-derived glioblastoma xenograft cellfates in 3D. Journal of Biomedical Materials Research. Part A. 109 (6), 1027-1035 (2021).
- da Hora, C. C., Schweiger, M. W., Wurdinger, T., Tannous, B. A. Patient-derived glioma models: From patients to dish to animals. Cells. 8 (10), 1177(2019).
- Li, W., et al. Characterization and transplantation of enteric neural crest cells from human induced pluripotent stem cells. Molecular Psychiatry. 23 (3), 499-508 (2018).
- Scaringi, C., Minniti, G., Caporello, P., Enrici, R. M. Integrin inhibitor cilengitide for the treatment of glioblastoma: A brief overview of current clinical results. Anticancer Research. 32 (10), 4213-4224 (2012).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon