このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ハイドロゲルアレイにより、3D腫瘍モデルにおけるマトリックス成分および治療薬の効果をスクリーニングするためのスループットの向上が可能
要約
本プロトコールは、調整可能な機械的特徴を有するヒドロゲルのハイスループット光架橋を容易にするカスタムメイドのUV照明装置を使用して、3Dマトリックス模倣培養における患者由来の膠芽腫細胞の化学療法応答に対する機械的および生化学的手がかりの効果を評価するための実験プラットフォームを記載する。
要約
細胞-マトリックス相互作用は、生化学的、機械的、幾何学的手がかりを通じて複雑な生理学的プロセスを媒介し、病理学的変化および治療応答に影響を与える。医薬品開発パイプラインの早い段階でマトリックス効果を考慮することで、新規治療薬の臨床的成功の可能性が高まると予想されます。3D細胞培養における特定の組織微小環境を再現する生体材料ベースの戦略が存在するが、これらを主に薬物スクリーニングに使用される2D培養方法と統合することは困難であった。したがって、ここで提示されたプロトコルは、既存の薬物スクリーニングパイプラインおよび細胞生存率のための従来のアッセイとの統合を容易にするために、マルチウェルプレートフォーマットの小型化された生体材料マトリックス内で3D培養のための方法の開発を詳述する。培養細胞における臨床的に関連する表現型を保存するために重要なマトリックス特徴は、高度に組織特異的および疾患特異的であると予想されるため、マトリックスパラメータの組み合わせスクリーニングは、特定の用途に適した条件を特定するために必要であろう。ここで説明する方法は、微細化された培養フォーマットを使用して、マトリックス力学およびリガンド提示の直交変動に対する癌細胞応答を評価する。具体的には、この研究は、化学療法に対する患者由来の膠芽腫(GBM)細胞の応答に対するマトリックスパラメータの効果を調査するために、このプラットフォームを使用することを実証する。
概要
新薬開発の予想コストは過去10年間で着実に上昇しており、現在の見積もりでは10億ドルを超えています1。この費用の一部は、臨床試験に入る薬の高い失敗率です。医薬品候補の約12%が、最終的に2019年に米国食品医薬品局(FDA)から承認を得ています。多くの薬剤は、予期せぬ毒性2のために第I相で失敗しますが、安全性試験に合格した他の薬剤は有効性の欠如3のために失敗する可能性があります。非有効性によるこの消耗は、医薬品開発中に使用される癌モデルが臨床的有効性の予測がないことで悪名高いという事実によって部分的に説明することができる4。
インビトロモデルとインビボモデルとの間の機能的差異は、非腫瘍細胞および物理的ECMを含むそれらの天然微小環境から癌細胞を除去することに起因し得る5,6。一般に、研究グループは、マトリゲル(マウス肉腫に由来するタンパク質性基底膜マトリックス)などの市販の培養マトリックスを使用して、培養腫瘍細胞に3Dマトリックス微小環境を提供する。2D培養と比較して、膜マトリックス中の3D培養は、in vitro結果の臨床的関連性を改善した7、8。しかしながら、膜マトリックスを含む脱細胞化組織からの培養生体材料は、典型的には、再現性を損なう可能性のあるバッチ間変動性を示す9。さらに、研究されたものとは異なる組織起源を有する腫瘍に由来するマトリックスは、適切な生理学的手がかり10を提供しない可能性がある。最後に、腫瘍内不均一性の高いがんは、サブミクロンサイズのスケールで変化し、膜マトリックスを反復するように調整することができない微小環境的特徴を有する11。
膠芽腫(GBM)は、生存期間の中央値が約15ヶ月の均一致死性の脳腫瘍であり、治療法の開発が特に困難であった癌である12,13。GBMの現在の標準治療は、原発性腫瘍切除、続いて放射線療法、次いでテモゾロミド(TMZ)を用いた化学療法からなる14。しかしながら、臨床的GBM腫瘍の半分以上が様々な機序を通じて治療抵抗性を示す15、16、17。個々の患者に対する治療レジメンの有効性を予測することは非常に困難である。個々の転帰を予測するために使用される標準的な前臨床モデルは、免疫不全マウスに同所性異所性に異所的に移植された患者由来の腫瘍細胞からなる。患者由来の異種移植片は、臨床GBM腫瘍の多くの側面を再現することができ、前臨床モデル18にとって価値があるが、本質的に高価で、スループットが低く、時間がかかり、倫理的懸念を伴う19。患者由来の細胞を、2Dプラスチック表面上で、または回転楕円体として培養すると、ほとんどの場合、これらの問題を回避します。患者由来の細胞は遺伝的異常を保持しているが、2Dまたは浮遊スフェロイドとしてのそれらの培養物は、げっ歯類および元の患者腫瘍における患者由来異種移植片の表現がほとんど不十分であった20。これまで、私たちや他の人々は、脳組織の機械的および生化学的特性を模倣する3D ECMで培養されたGBM細胞が薬剤耐性表現型を維持できることを示しました10,21,22,23。
脳ECMに豊富に存在し、GBM腫瘍において過剰発現される多糖であるヒアルロン酸(HA)とそのCD44受容体との間の相互作用は、インビトロでの薬物耐性の獲得を調節する21、24、25、26、27。例えば、軟質3D培養物内にHAを含めることで、患者由来のGBM細胞が治療抵抗性を獲得する能力が増加した。このメカノ応答性は、GBM細胞上のCD44受容体へのHA結合に依存していた21。さらに、RGD担持ペプチドへのインテグリン結合は、3D培養マトリックスに組み込まれ、CD44媒介性化学抵抗性を剛性依存的に増幅した21。HAを超えて、いくつかのECMタンパク質の発現は、多くはRGD領域を含み、正常な脳腫瘍とGBM腫瘍との間で変化する28。例えば、ある研究では、GBM腫瘍において28種類のECMタンパク質がアップレギュレートされたことが報告されています29。この複雑な腫瘍マトリックス微小環境内で、癌細胞は、機械的および生化学的手がかりを統合して、インテグリン結合ペプチドのヤング率または密度における比較的小さな差(例えば、一桁未満)に依存する特定の耐性表現型を生じる28、29、30。
本プロトコールは、腫瘍細胞がマトリックス合図のユニークな組み合わせを解釈し、治療抵抗性を促進する複雑で患者固有のマトリックス微小環境を特定する方法を特徴付けるものです(図1A)。3D培養用の小型で正確に調整された行列を生成するための光化学的方法は、大きくて直交する可変空間を提供する。マイクロコントローラによって実行されるカスタムメイドのLEDアレイは、自動化と再現性を高めるために、384ウェルプレートフォーマット内のヒドロゲルを光架橋するために組み込まれました。露光強度は、原子間力顕微鏡(AFM)を用いて評価されるように、得られたヒドロゲルのマイクロ機械的特性を変化させるためにウェルにわたって変化させた。本稿では照明アレイ自体の構築には焦点を当てていませんが、デバイスの再生の補助として回路図(図1B)と部品リスト(材料表)を提供しています。
この報告は、ヤング率(1桁の4つのレベル)とインテグリン結合ペプチド含量(4つの異なるECMタンパク質に由来する)が直交して変化したユニークな3D微小環境で培養された一連のGBM細胞の迅速な生成を実証している。次に、このアプローチを使用して、患者由来GBM細胞がテモゾロミド(TMZ)化学療法に対する耐性を獲得する際の生存率および増殖に対するヒドロゲル力学およびECM特異的インテグリン関与の相対的寄与を調査した。
プロトコル
患者由来のGBM細胞株(GS122およびGS304)は、UCLA治験審査委員会(IRB#10-000655)によって承認されたプロトコルの下でこれらの株を開発したDavid Nathanson教授(私たちの共同研究者)によって提供されました。細胞株を個々の患者に再び連結することができないように、細胞を非同定して提供した。
ヒドロゲル溶液の調製
- HEPES粉末をハンクス平衡塩溶液(HBSS)に20mMで溶解して、HEPES緩衝溶液を調製する。完全な溶媒和後にpHを7に調整します。
- HEPES 緩衝溶液に、前の報告31 に従って調製したチオール化 HA (700 kDa 公称分子量、材料表を参照) を溶解し、各グルクロン酸上のカルボン酸残基の 6% ~ 8% がチオールで修飾されるように、緩衝液に 10 mg/mL の濃度で溶解します。
注:周囲光によるチオール酸化を防ぐために、琥珀色のバイアルをお勧めします。- マグネチックスターリングプレート(<1,000rpm)を用いて、完全に溶解するまで室温で、典型的には約45分間撹拌する。
- HAが溶解している間に、(1)100mg/mLの8-アーム-PEG-ノルボルネン(20kDa)、(2)100mg/mLの4-アーム-PEG-チオール(20kDa)、(3)4mMのシステインまたはシステイン含有ペプチド(GCGYGRGDSPG)、および(4)4mg/mLのLAPの別々の溶液を微小遠心管で調製する( 材料表を参照)。
- ステップ1.1で調製したHEES緩衝溶液にこれら4つの溶液のそれぞれを調製する。溶液を渦巻き、ステップ4を実行する前に各試薬の完全な溶解を確実にする。
注:複数の異なるペプチドを試験する場合、各ペプチドは、このコンジュゲーション化学のためにシステインまたは他のチオール部分の供給源を含んでいなければならない。 - この時点で単一のヒドロゲル内で連結されるすべてのペプチドの溶液(4mM利用可能なチオール)を調製する。
注:ペプチド配列およびそれらが由来し、本研究で使用されたECMタンパク質を 表1に列挙する。細胞が結合しないN-アセチルシステイン( 材料表を参照)を生理活性チオール含有ペプチドに置換して、接着ペプチドの濃度を滴定するか、または陰性対照31として作用させることができる。
- ステップ1.1で調製したHEES緩衝溶液にこれら4つの溶液のそれぞれを調製する。溶液を渦巻き、ステップ4を実行する前に各試薬の完全な溶解を確実にする。
- HA、PEG-ノルボルネン、PEG-チオール、およびシステイン/チオール含有ペプチド( 材料表を参照)の個々の溶液を混合して、 表2にリストされている最終ヒドロゲルマトリックスの最終濃度を達成します。マグネチックスタープレート上で少なくとも30分間攪拌(<1,000rpm)して完全に混合する。
注:HA溶液は粘性が高く、容積ピペットを使用して取り扱うのが最適です( 材料表を参照)。正の変位ピペットが利用できない場合、粘性溶液は、ワイドオリフィスチップを使用してゆっくりとピペッティングすることによって、標準的なマイクロピペットで分配することもできます。
2. LEDアレイ を介した ヒドロゲルの照明と光架橋
警告: UV 保護メガネを着用し、照明フィールドを UV 吸収材で覆ってください。
メモ:このプロトコルで説明されているLEDアレイは、付属の回路図(図1A)に示すように、直列に配置された6組の8つのLEDで構成されています。各LEDセットは独立して給電できるため、1回のランで最大6つの異なる放射照度が可能です。 補足ファイル 1 には、詳細なガイダンスとして、次の指示に対応するスクリーンショットが含まれています。
- 補助コーディング ファイルから照明デバイス.zip ファイルをダウンロードします。このディレクトリには、Arduino.zip (補足コーディング ファイル 1)、ドライバー.zip (補足コーディング ファイル 2)、GUI.zip (補足コーディング ファイル 3)、およびホルダー.zip (補足コーディング ファイル 4) のファイルが含まれています。
メモ: 回路基板を所定の位置に保持するために、上部と下部を 3D 印刷します(詳細については、「 補足コーディングファイル 」を参照してください)。 - マイクロコントローラソフトウェアをダウンロードしてインストールします( 材料表を参照)。
- GUIソフトウェアをダウンロードしてインストールします( 資料表を参照)。ソフトウェアの操作手順については、 補足ファイル 1 を参照してください。
- 処理を開き、スケッチ>インポートライブラリ>ライブラリの追加をクリックしてcontrolIP5ライブラリをインストールします。次に、ライブラリでcontrolIP5を検索し、[インストール]をクリックします。これを初めて実行します。
- 36ボルト電源を使用して照明装置( 材料表を参照)に電力を供給し、マイクロUSBケーブルを使用してPCに接続します。
メモ:一部のデバイスでは、さまざまなArduinoナノボード用のドライバが自動的にインストールされません。ドライバーの 1 つのセットは、デバイスの zip ファイルで提供されます。 - Adruino.zip フォルダにある Arduino.ino ファイルを Arduino IDE を使用して開きます。
- Arduino.inoファイルをコンパイルするには、[ チェックマーク ]ボタンをクリックします。矢印 ボタンをクリックしてコンパイル 済みコードをアップロードします。
- GUI.zip フォルダーにある GUI.pde ファイルを「処理」を使用して開きます。
- 処理プログラムで[ 実行 ]をクリックして、照明デバイスを制御するためのグラフィカルユーザーインターフェイスを起動します。
- グラフィカルユーザーインターフェイスウィンドウで、架橋するヒドロゲル前駆体溶液を含むカラムの 強度 をクリックし、所望の強度を入力します。[時間]ボックスをクリックし、希望する 時間 を入力します。 表 2 に示すソリューションの場合、これは 15 秒になります。
メモ: エンドユーザーは、放射計を使用してデジタル強度値を放射照度に較正する必要があります。典型的な強度の例が 図2Aに提供される。 - サンプルを照明装置(図2B)と他のすべてのLEDとシリコーンモールド( 材料表を参照)または384ウェルプレートの1列に合わせます。 [完了] をクリックしてイルミネーションを開始します。384ウェルプレートの複数のスライドまたは他のウェルの照明のために、必要に応じてこのプロセスを繰り返します。
メモ:ホルダーは、照明中に384ウェルプレートが内室の片隅と同一平面に収まるように設計されています。- 照明に続いて、1つのコーナーに置かれたら、ウェルプレートを次のコーナーに移動して繰り返します。プレートのもう半分のウェルを照らすには、プレートをホルダーから持ち上げて180°回転させます。
- 以下の手順に従って、機械的特性評価のためのさまざまな力学を有するヒドロゲルを生成する。
- スライドガラスとシリコンモールドをテープで拭き取り、破片を取り除きます。シリコーンモールドをスライドガラスに接着し、押し下げて良好なシールを確保し、気泡を置換します。
- ステップ1.4で調製したヒドロゲル前駆体溶液80 μLをスライドガラス上の各シリコーンモールドにピペットで入れた。
- スライドガラスを、1つの列の他のすべてのLEDと整列した照明装置の上に置きます。ヒドロゲル前駆体を15秒間UV光に曝し、ステップ2で説明したように、光架橋させる。
- 照明が止まったらスライドを取り出し、金型の内周を細かい先端(10μLピペットチップ、30G針など)でなぞって金型からゲルをほぐします。ピンセット/鉗子でシリコーンモールドを取り除きます。
- 架橋ヒドロゲルをヘラを濡らし、スライドガラスから静かに押し出すことによって、12ウェルプレートの個々のウェルに移動する。ヒドロゲルを加える前に、各ウェルに2mLのDPBS( 材料表を参照)を充填する。DPBS溶液中のゲルを室温で少なくとも12時間(通常は一晩)膨潤させる(翌日の機械的特性評価のため)。
3. 原子間力顕微鏡(AFM)測定
- 製造元の指示に従って原子間力顕微鏡(AFM)の電源を入れます( 材料表を参照)。このプロトコルは、計測器および関連ソフトウェアの使用に関する簡単な手順を提供します。
- AFMプローブを取り付けます( 材料表を参照)。
注:本研究では、公称ばね定数0.01N/mの三角形窒化ケイ素片持ち梁を球状の2.5μm二酸化ケイ素粒子で修飾した。 - 取り付け後、レーザーを三角形プローブの頂点に合わせ、ミラーとレーザーのたわみを調整して信号の合計(通常は1.5~2.2ボルト)を最大にします。
- プローブをDPBSに浸し、熱平衡が得られるまで最大15分間待ちます。キャリブレーションボタンをクリックし、連絡先依存キャリブレーションを選択します。「サーマルチューニングを収集」ボタンをクリックし、データ収集に続いて、キャリブレーション用に約3kHzのピークを選択します。
注:屈折率の変化により、液体に浸漬した後、ミラーとレーザー偏向器のわずかな調整が必要な場合があります。 - アプローチパラメータを等速、目標高さ7.5μm、アプローチ速度15μm/sに設定して、シャーレ(プラスチック)の表面にアプローチします。アプローチの実行ごとのベースライン測定を有効にして、アプローチが継続的に実行され、ディフレクターのドリフトが原因で早期に停止しないようにします。
- アプローチ時に、フォースマッピング用の集録パラメータを4 nNターンアラウンド、2 μmのくぼみ距離、1 μm/sの速度、および0 sの接触時間に設定します。 スタート ボタンを押して、プラスチック表面(ウェルプレートなど)の力曲線の収集を開始します。
- キャリブレーションウィンドウに戻り、プラスチックの接触とくぼみに対応する力曲線の部分を選択します。計算された感度と剛性の値を受け入れて、プローブのキャリブレーションを完了します。
- 較正後、AFMプローブを持ち上げ、尋問のためにヒドロゲルサンプルを置く。ステップ5で提供された設定に従ってヒドロゲルにアプローチする。
注:ヒドロゲル表面へのアプローチ手順中に、ユニットが誤ってアプローチ状態をトリガーすることがあります。実際のアプローチを検証するには、手順 4.6 のように力曲線を取得します。結果のカーブに接触とインデントが表示されない場合は、アプローチ手順を繰り返します。 - サーフェスアプローチが成功したら、 フォースマッピング モードに切り替えて、集録パラメータを軸あたり40μmの長さで8 x 8サイズのマップに設定します。さまざまな領域の力マップを取得して、剛性測定の均一性を評価します。
- ソフトウェアプログラムJPK SPMデータ処理を使用して、球面形状を選択したヘルツ/スネドンモデルフィット(式1と式2、すべての変数の定義については表3を参照)を使用して力曲線を解釈します32,33,3 4。
 式 132
式 132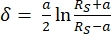 式 232
式 232
- ソフトウェアプログラムJPK SPMデータ処理を使用して、球面形状を選択したヘルツ/スネドンモデルフィット(式1と式2、すべての変数の定義については表3を参照)を使用して力曲線を解釈します32,33,3 4。
4. 3Dマトリックス埋め込み培養物のセットアップと薬物処理
- 所望の細胞を単一細胞溶液として調製する。
注: 細胞タイプが異なれば、異なる継代方法が必要になる場合があります。T−75フラスコからのGBMスフェロイドの浮遊培養物を継代するための典型的なプロトコールが参考文献31に報告されている。 - TBMスフェロイド(直径約150μm)をT-75フラスコ浮遊培養物から15mL円錐管に集める。培養フラスコを5mLのDPBSですすぎ、残留細胞および培地を除去し、この容量を円錐管に加える。
- 細胞を含む円錐管を室温で200 x g で5分間遠心分離する。遠心分離後、細胞ペレットを乱さないように注意しながら、5mL血清学的ピペットで上清を除去し、5mLのDPBSに再懸濁する。
- 200 x g で室温で5分間遠心分離し、細胞を洗浄する。細胞ペレットを乱さないように注意しながら、5 mLの血清学的ピペットで上清を吸引し、次いで細胞を2 mLの細胞解離試薬に再懸濁する( 材料表を参照)。
- 室温で10〜15分間インキュベートする。3mLの完全培地( 材料表を参照)および穏やかにピペットを3〜5回加えて、スフェロイドを単一細胞懸濁液31に分解する。
- 単一細胞懸濁液を400 x g で遠心分離し(単一細胞懸濁液はペレット形成のためにより速く紡糸され得る)、室温でペレット細胞に5分間遠心分離する。上清を5mL血清学的ピペットで吸引し、細胞ペレットを乱さないように注意してください。細胞を1mLの完全培地に再懸濁する。
注:細胞が懸濁液中の単一細胞としてではなく凝集塊に残っている場合、継代後、細胞を40μmの細胞ストレーナーに通して単一細胞懸濁液を達成することができます。 - 血球計数器を用いて計数のために細胞の一部を除去する。この部分をトリパンブルーで2倍に希釈し、生存率が損なわれた細胞に浸透させる。生きた無色の細胞だけを数えます。典型的には、フラスコ当たり800,000細胞で播種されたT−75は、培養において1週間後に200万〜300万個の細胞を生じる。
- カプセル化に必要なセルの数を決定します。必要な細胞の総数を含む量の培地を滅菌1.7mL微量遠心管に移す。室温で400 x g で5分間スピンダウンします。
注:例えば、250万細胞/mLで細胞を封入するには、1mLのゲル容量に再懸濁された最低250万個の細胞が必要である。1mLのゲル容量により、ユーザーは100個のゲル滴を分注することができ、各ゲル滴は10μLの体積である。ヒドロゲル溶液に懸濁された余分な〜20%容量の細胞を調製することは、ピペット移送中の損失を考慮するために推奨される。したがって、この例では、300万個の細胞および1.2mLのヒドロゲル前駆体溶液を調製するであろう。500,000細胞/mLの最小密度が推奨されます。 - 上清をマイクロピペットで吸引し、細胞ペレットを乱さないように注意する。ステップ1.4で調製したように、細胞ペレットをヒドロゲル前駆体溶液に再懸濁し、1,000 μLのマイクロピペットで上下に4〜5回ピペッティングしてよく混合する。
- セルを10 μLを分配するようにセットした繰り返しピペッター( 材料表を参照)にロードします。気泡や不均一な分配を避けるために、廃棄物容器にさらに1〜2回分注することによって、繰り返しピペッターをプライミングする。
- 384ウェルプレートの各ウェルに、ヒドロゲル溶液に懸濁した10μLの細胞を反復ピペッターから分注する。LEDアレイを使用して、所望の機械的特性を達成するために、細胞を含む各ウェルを15秒間強度で照明する(例えば、 図2A の結果は、1.14、1.55、2.15、2.74mW/cm2の強度を利用する)。
注: 実験条件ごとに 5 回の反復から開始し、エンドポイントアッセイの望ましいスループットと分散に応じてスケールアップまたはスケールダウンすることをお勧めします。 - 細胞を含む各ウェルに40 μLの完全培地を加える。50 μL の DPBS をゲルを囲む非実験的な乾燥ウェルに追加して、蒸発による損失を最小限に抑えます。
- GBM細胞の場合、40μLの培地含有薬物(例えば、TMZ、 材料表を参照)を添加して、最終所望の濃度(ジメチルスルホキシド(DMSO)またはビヒクル(DMSO)中の10μM〜100μM)を達成し、それに応じて、封入の3日後に開始する。
CCK8増殖アッセイ
- 細胞を含む各ウェルに10 μLのCCK8試薬( 材料表を参照)を加える。
注:このアッセイを初めて行う場合は、培地のみまたは無細胞ヒドロゲルなどの陰性対照ウェルを培地に含めてください。 - 製造業者の指示に従って1〜4時間インキュベートする。
注:この時間は細胞の種類と密度の関数として変化する可能性があるため、吸光度値がビールの法則35を適用するための要件である線形範囲内に収まるように、各アプリケーションでインキュベーション時間をテストする必要があります。 - インキュベーション後のすべてのウェルについて、450nmでの吸光度を読み取ります。
- 各群のビヒクル条件についてステップ3で得られた450nmにおける平均吸光度を算出する。ウェル処理した各薬物を、1群あたりのビヒクル対照の平均で割る。
- 百分位数法36を使用してブートストラップ分布(N = 10,000)を生成して信頼区間を計算します。
注: 一般に、95% 信頼区間を利用し、信頼区間が 1 を横切らない条件を有意であると解釈し、さらなる調査を正当化することができます。信頼区間を95%に設定することは、有意カットオフをp = 0.05に設定することと一致します。結果に示されたデータに対して、マトリックス媒介性薬物耐性を促進または阻害する条件を区別することに有用性があり、両面分析が必要である。
結果
AFM測定では、カスタムメイドのArduino制御LEDアレイを使用して、光架橋中のUV放射照度(mW/cm2)の関数としてヒドロゲル力学を正確に制御することが確認されました(図2A)。このプロトコールで使用されるヒドロゲル製剤を 表2に見出すことができる。提供されたテンプレート上のLEDの間隔は、384ウェルプレートの他のすべてのウェルの間隔と一致し、プ?...
ディスカッション
現在の研究は、HAベース内で3Dの小型化された培養物を生成すると同時に、マトリックスの剛性とインテグリンエンゲージメントに利用可能なペプチドを変更する方法を提示しています。この技術は、マトリックスパラメータが細胞表現型(例えば、化学療法に曝露された癌細胞の生存率)にどのように影響するかを、より高いスループットで体系的に研究することを可能にする。本明細書に提?...
開示事項
著者らは開示するものは何もありません。
謝辞
著者らは、Carolyn Kim、Amelia Lao、Ryan Stoutamore、Itay Solomonがフォトジェレーションスキームの初期の反復に貢献したことを特に認めたいと思います。細胞株GS122およびGS304は、David Nathansonによって寛大に提供された。すべての図は BioRender.com で作成されました。UCLAの中核施設、分子スクリーニング共有リソース、ナノおよびピコキャラクタリゼーション研究所は、この研究に役立った。Chen Chia-Chunは、UCLA Eli and Edythe Broad Center of Regenerative Medicine and Stem Cell Research Training Programの支援を受けました。Grigor Varuzhanyanは、Tumor Cell Biology Training Program NIH Grant(T32 CA 009056)の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 1.1 kOhm resistors, 6 W | Digikey | 35601k1ft | |
| 1.7 mL microcentrifuge tube | Genesse Scientific | 21-108 | |
| 15 mL conical tube | Fisher Scientific | 14-959-70C | |
| 365 nm LED | Digikey | ltpl-c034uvh365 | |
| 384 well plate | Bio Greiner One | 781090 | |
| 40 µm cell strainer | MTC bio | C4040 | |
| 4-Armed thiol terminated polyethlene glycol (20 kDa) | Laysan Bio | 4arm-PEG-SH-20K-1g | |
| 6 NPN BJTs | Digikey | 2n5550ta | |
| 80 Ohm resistors, 0.125 W | Digikey | erjj-6enf80r6v | |
| 8-Armed norbornene terminated polyethylene glycol (20 kDa) | Jenkem Technology | A7025-1 | |
| Accutase | Innovative Cell Technologies | AT104500 | cell dissociation reagent |
| AFM Probes | Novascan | 0.01 N/m Nominal spring constant, 2.5 µm SiO2 particle | |
| Arduino IDE | Arduino | 1.8.19 | |
| Arduino Nano | Makerfire | Mini Nano V3.0 ATmega328P Microcontroller Board | |
| bFGF | Peprotech | 100-18B | 20 ng/mL |
| CCK8 | Abcam | ab228554 | |
| Centrifuge | Thermoscientific | sorvall legend xtr | |
| CP100ST | Gilson | F148415 | Pipette tips for positive displacement pipette |
| Cubis Semi-Micro Balance | Sartorius | MSA225S100DI | |
| DMEM - F12 (50-50) | Life Technologies | 11330057 | 1x |
| DMSO | Fisher Scientific | BP231-100 | |
| DPBS Ca (-) Mg (-) | Genesse Scientific | 25-508 | |
| EGF | Peprotech | AF100-15 | 50 ng/mL |
| Ethanol, Anhydrous | Fisher Scientific | A405P | Add DI water to dilute to 70% |
| Fisherbrand Class B Amber Glass threaded vials | Fisher Scientific | 03-339-23C | |
| Fisherbrand Weighing Paper | Fisher Scientific | 09-898-12B | |
| G21 Supplement | Gemini Bio | 400-160 | 50x |
| Hanks Balanced Salt Solution | Thermo Fisher Scientific | 14175095 | |
| HCl, ACS, 12M | Sigma Aldrich | S25838A | Add DI water to dilute to 1 M |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H3149-100Ku | 25 µg/mL |
| HEPES | Sigma Aldrich | H7006-100G | |
| Hot Air Gun | Wagner | HT1000 | |
| Integrin-binding sialoprotein (IBSP) peptide | Genscript | Custom Order | GCGYGGGGNGEPRGDTYRAY |
| Lithium phenyl-2,4,6 trimethylbenzoylphosphinate (LAP) , >95% | Sigma Aldrich | 900889-1G | |
| Magnetic stir plate | Thermo Scientific | SP194715 | |
| Microcentrifuge | Thermo Scientific | Sorvall legend micro 21R | |
| Microman E single Channel Pipettor | Gilson | FD10004 | Positive displacement pipette |
| Micropipette Tips | Various Manufacturs | Various sizes | |
| mLine micropipette | Sartorious | ||
| N-acetyl Cysteine | Sigma Aldrich | A7250-10G | |
| Nanowizard 4 | Bruker | AFM microscope | |
| NaOH | Fisher Scientific | ss255-1 | Add DI water to dilute to 1 M |
| Normoicin | Invivogen | ant-nr-1 | 500x |
| Osteopontin Peptide | Genscript | Custom Order | GCGYGTVDVPDGRGDSLAYG |
| Pipet Aid | Drummond | 4000102 | |
| Plain Microscope Slides | Globe Scientific | 1301 | |
| Press-To-Seal silicone Isolator, 12-4.5mm diam x 2mm deep | Grace Bio Labs | 664201-A | Cut so that 8 individual molds are made from a single sheet |
| Processing | Processing | 3.5.4 | |
| Repeater M4 | Eppendorf | 4982000322 | |
| Repeater Pipette Tips | Sartorious | 30089430 | 1 mL sizes |
| RGD Peptide | Genscript | GCGYGRGDSPG | |
| Scoth Tape | |||
| Serological Pipettes | Genesse Scientific | 12-102,12-104 | 5,10 mL Pipettes |
| Solder Paste | Digikey | 315-NC191LT15T5-ND | |
| Solder Wire | |||
| Straight dissecting forceps | VWR Scientific | 82027-408 | |
| Synergy H1 Plate Reader | Biotek | ||
| T-75 Cell Culture Treated Flask | Genesee Scientific | 25-209 | |
| Temozolomide | Sigma Aldrich | T2577 | Typically used from 10 µM to 100 µM |
| Tenascin-C Peptide | Genscript | GCGYGRSTDLPGLKAATHYTITIR GV | |
| Thiolated Hyaluronic Acid (700 kDa), 6-8% modified | Lifecore Biomedical | HA700K5 | |
| VWR Spinbar, Flea Micro | VWR | 58948-375 |
参考文献
- Scannell, J. W., Blanckley, A., Boldon, H., Warrington, B. Diagnosing the decline in pharmaceutical R&D efficiency. Nature Reviews Drug Discovery. 11 (3), 191-200 (2012).
- Waring, M. J., et al. An analysis of the attrition of drug candidates from four major pharmaceutical companies. Nature Reviews Drug Discovery. 14 (7), 475-486 (2015).
- Khozin, S., Liu, K., Jarow, J. P., Pazdur, R. Why do oncology drugs fail to gain US regulatory approval. Nature Reviews Drug Discovery. 14 (7), 450-451 (2015).
- Booth, B., Ma, P., Glassman, R. Oncology's trials. Market indicators. Nature Reviews Drug Discovery. 2 (8), 609-610 (2003).
- Da Ros, M., et al. Glioblastoma chemoresistance: The double play by microenvironment and blood-brain barrier. International Journal of Molecular Sciences. 19 (10), 2879 (2018).
- Broekman, M. L., et al. Multidimensional communication in the microenvirons of glioblastoma. Nature Reviews Neurology. 14 (8), 482-495 (2018).
- Grundy, T. J., et al. Differential response of patient-derived primary glioblastoma cells to environmental stiffness. Scientific Reports. 6 (1), 1-10 (2016).
- Gomez-Roman, N., Stevenson, K., Gilmour, L., Hamilton, G., Chalmers, A. J. A novel 3D human glioblastoma cell culture system for modeling drug and radiation responses. Neuro-Oncology. 19 (2), 229-241 (2017).
- Simoni, R. D., et al. Basement membrane complexes with biological activity. Biochemistry. 25 (2), 312-318 (2002).
- Xiao, W., et al. Brain-mimetic 3D culture platforms allow investigation of cooperative effects of extracellular matrix features on therapeutic resistance in glioblastoma. Cancer Research. 78 (5), 1358-1370 (2018).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nature Reviews Materials. 5 (7), 539-551 (2020).
- Spinelli, C., et al. Molecular subtypes and differentiation programmes of glioma stem cells as determinants of extracellular vesicle profiles and endothelial cell-stimulating activities. Journal of Extracellular Vesicles. 7 (1), 1490144 (2018).
- Ostrom, Q. T., Cioffi, G., Waite, K., Kruchko, C., Barnholtz-Sloan, J. S. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2014-2018. Neuro-Oncology. 23, (2021).
- Stupp, R., et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma. New England Journal of Medicine. 352 (10), 987-996 (2005).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Tomczak, K., Czerwińska, P., Wiznerowicz, M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge. Contemporary oncology. 19, 68-77 (2015).
- Lee, S. Y. Temozolomide resistance in glioblastoma multiforme. Genes and Diseases. 3 (3), 198-210 (2016).
- Joo, K. M., et al. Patient-specific orthotopic glioblastoma xenograft models recapitulate the histopathology and biology of human glioblastomas in situ. Cell Reports. 3 (1), 260-273 (2013).
- Levy, N. The use of animal as models: Ethical considerations. International Journal of Stroke. 7 (5), 440-442 (2012).
- Phon, B. W. S., Kamarudin, M. N. A., Bhuvanendran, S., Radhakrishnan, A. K. Transitioning preclinical glioblastoma models to clinical settings with biomarkers identified in 3D cell-based models: A systematic scoping review. Biomedicine & Pharmacotherapy. 145, 112396 (2022).
- Xiao, W., et al. Bioengineered scaffolds for 3D culture demonstrate extracellular matrix-mediated mechanisms of chemotherapy resistance in glioblastoma. Matrix Biology. 85-86, 128-146 (2020).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Xu, X., Farach-Carson, M. C., Jia, X. Three-dimensional in vitro tumor models for cancer research and drug evaluation. Biotechnology Advances. 32 (7), 1256-1268 (2014).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived Glioblastoma Cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Preston, M. Digestion products of the PH20 hyaluronidase inhibit remyelination. Annals of Neurology. 73 (2), 266-280 (2013).
- Kim, Y., Kumar, S. CD44-mediated adhesion to hyaluronic acid contributes to mechanosensing and invasive motility. Molecular Cancer Research. 12 (10), 1416-1429 (2014).
- Pibuel, M. A., Poodts, D., Díaz, M., Hajos, S. E., Lompardía, S. L. The scrambled story between hyaluronan and glioblastoma. The Journal of Biological Chemistry. 296, 100549 (2021).
- Xiao, W., Sohrabi, A., Seidlits, S. K. Integrating the glioblastoma microenvironment into engineered experimental models. Future Science OA. 3 (3), (2017).
- Trombetta-Lima, M., et al. Extracellular matrix proteome remodeling in human glioblastoma and medulloblastoma. Journal of Proteome Research. 20 (10), 4693-4707 (2021).
- Schregel, K., et al. Characterization of glioblastoma in an orthotopic mouse model with magnetic resonance elastography. NMR in Biomedicine. 31 (10), 3840 (2018).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived glioblastoma cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Guz, N., Dokukin, M., Kalaparthi, V., Sokolov, I. If cell mechanics can be described by elastic modulus: Study of different models and probes used in indentation experiments. Biophysical Journal. 107 (3), 564-575 (2014).
- Sneddon, I. N. The relation between load and penetration in the axisymmetric boussinesq problem for a punch of arbitrary profile. International Journal of Engineering Science. 3 (1), 47-57 (1965).
- Soofi, S. S., Last, J. A., Liliensiek, S. J., Nealey, P. F., Murphy, C. J. The elastic modulus of MatrigelTM as determined by atomic force microscopy. Journal of Structural Biology. 167 (3), 216-219 (2009).
- Mayerhöfer, T. G., Popp, J. Beer's law - Why absorbance depends (almost) linearly on concentration. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 20 (4), 511-515 (2019).
- Puth, M. T., Neuhäuser, M., Ruxton, G. D. On the variety of methods for calculating confidence intervals by bootstrapping. Journal of Animal Ecology. 84 (4), 892-897 (2015).
- Lavrentieva, A. Gradient hydrogels. Advances in Biochemical Engineering/Biotechnology. 178, 227-251 (2020).
- Zhu, D., Trinh, P., Li, J., Grant, G. A., Yang, F. Gradient hydrogels for screening stiffness effects on patient-derived glioblastoma xenograft cellfates in 3D. Journal of Biomedical Materials Research. Part A. 109 (6), 1027-1035 (2021).
- da Hora, C. C., Schweiger, M. W., Wurdinger, T., Tannous, B. A. Patient-derived glioma models: From patients to dish to animals. Cells. 8 (10), 1177 (2019).
- Li, W., et al. Characterization and transplantation of enteric neural crest cells from human induced pluripotent stem cells. Molecular Psychiatry. 23 (3), 499-508 (2018).
- Scaringi, C., Minniti, G., Caporello, P., Enrici, R. M. Integrin inhibitor cilengitide for the treatment of glioblastoma: A brief overview of current clinical results. Anticancer Research. 32 (10), 4213-4224 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved