Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hydrogel-Arrays ermöglichen erhöhten Durchsatz für Screening-Effekte von Matrixkomponenten und Therapeutika in 3D-Tumormodellen
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt eine experimentelle Plattform zur Bewertung der Auswirkungen mechanischer und biochemischer Hinweise auf chemotherapeutische Reaktionen von patientenabgeleiteten Glioblastomzellen in 3D-matrixmimetischen Kulturen unter Verwendung eines maßgeschneiderten UV-Beleuchtungsgeräts, das die Hochdurchsatz-Photovernetzung von Hydrogelen mit abstimmbaren mechanischen Eigenschaften ermöglicht.
Zusammenfassung
Zell-Matrix-Interaktionen vermitteln komplexe physiologische Prozesse durch biochemische, mechanische und geometrische Hinweise und beeinflussen pathologische Veränderungen und therapeutische Reaktionen. Es wird erwartet, dass die Berücksichtigung von Matrixeffekten zu einem früheren Zeitpunkt in der Arzneimittelentwicklungspipeline die Wahrscheinlichkeit des klinischen Erfolgs neuartiger Therapeutika erhöhen wird. Es gibt auf Biomaterialien basierende Strategien, die spezifische Gewebemikroumgebungen in 3D-Zellkultur rekapitulieren, aber die Integration dieser mit den 2D-Kulturmethoden, die hauptsächlich für das Drogenscreening verwendet werden, war eine Herausforderung. Daher beschreibt das hier vorgestellte Protokoll die Entwicklung von Methoden für die 3D-Kultur in miniaturisierten Biomaterialmatrizen in einem Multi-Well-Plattenformat, um die Integration in bestehende Wirkstoff-Screening-Pipelines und konventionelle Assays für die Zelllebensfähigkeit zu erleichtern. Da von den Matrixmerkmalen, die für die Erhaltung klinisch relevanter Phänotypen in kultivierten Zellen entscheidend sind, ein hochgradig gewebe- und krankheitsspezifisches Screening erwartet wird, wird ein kombinatorisches Screening von Matrixparametern erforderlich sein, um geeignete Bedingungen für spezifische Anwendungen zu identifizieren. Die hier beschriebenen Methoden verwenden ein miniaturisiertes Kulturformat, um die Reaktionen von Krebszellen auf orthogonale Variation der Matrixmechanik und der Ligandenpräsentation zu bewerten. Insbesondere zeigt diese Studie die Verwendung dieser Plattform, um die Auswirkungen von Matrixparametern auf die Reaktionen von patientenabgeleiteten Glioblastomzellen (GBM) auf die Chemotherapie zu untersuchen.
Einleitung
Die erwarteten Kosten für die Entwicklung eines neuen Medikaments sind in den letzten zehn Jahren stetig gestiegen, mit über 1 Milliarde US-Dollar in aktuellen Schätzungen1. Ein Teil dieser Kosten ist die hohe Ausfallrate von Medikamenten, die in klinische Studien aufgenommen werden. Etwa 12% der Arzneimittelkandidaten erhalten schließlich im Jahr 2019 die Zulassung der US-amerikanischen Food & Drug Administration (FDA). Viele Medikamente versagen in Phase I aufgrund unvorhergesehener Toxizität2, während andere, die Sicherheitsstudien bestehen, aufgrund mangelnder Wirksamkeitscheitern können 3. Diese Fluktuation aufgrund der Nichtwirksamkeit kann teilweise durch die Tatsache erklärt werden, dass Krebsmodelle, die während der Arzneimittelentwicklung verwendet werden, notorisch nicht prädiktiv für die klinische Wirksamkeitsind 4.
Funktionelle Unterschiede zwischen In-vitro- und In-vivo-Modellen können auf die Entfernung von Krebszellen aus ihrer nativen Mikroumgebung zurückgeführt werden, einschließlich Nicht-Tumorzellen und der physikalischen ECM 5,6. Häufig verwenden Forschungsgruppen kommerziell erhältliche Kulturmatrizen wie Matrigel (eine proteinhaltige Basalmembranmatrix, die von Maussarkomen abgeleitet ist), um kultivierten Tumorzellen eine 3D-Matrix-Mikroumgebung zur Verfügung zu stellen. Im Vergleich zur 2D-Kultur hat die 3D-Kultur in der Membranmatrix die klinische Relevanz der In-vitro-Ergebnisse verbessert 7,8. Kulturbiomaterialien aus dezellularisierten Geweben, einschließlich der Membranmatrix, weisen jedoch typischerweise eine Variabilität von Charge zu Charge auf, die die Reproduzierbarkeit beeinträchtigenkann 9. Darüber hinaus liefern Matrizen, die von Tumoren stammen, deren Gewebeursprung sich von den untersuchten unterscheidet, möglicherweise nicht die geeigneten physiologischen Hinweise10. Schließlich weisen Krebsarten mit hohem Grad an intratumoraler Heterogenität mikroökologische Merkmale auf, die auf einer Submikrometer-Größenskala variieren und deren Rekapitulation der Membranmatrix nicht abgestimmt werdenkann 11.
Das Glioblastom (GBM), ein gleichmäßig tödlicher Hirntumor mit einer medianen Überlebenszeit von etwa 15 Monaten, ist eine Krebserkrankung, bei der die Behandlungsentwicklung besonders schwierig war12,13. Der derzeitige Behandlungsstandard für GBM besteht aus einer primären Tumorresektion, gefolgt von einer Strahlentherapie und einer anschließenden Chemotherapie mit Temozolomid (TMZ)14. Dennoch zeigen mehr als die Hälfte der klinischen GBM-Tumoren eine Behandlungsresistenz durch verschiedene Mechanismen15,16,17. Die Vorhersage der Wirksamkeit eines Behandlungsschemas für einen einzelnen Patienten ist äußerst schwierig. Präklinische Standardmodelle, die zur Vorhersage individueller Ergebnisse verwendet werden, bestehen aus von Patienten abgeleiteten Tumorzellen, die orthotopisch in immungeschwächte Mäuse xenotransplantiert werden. Während von Patienten abgeleitete Xenotransplantate viele Aspekte klinischer GBM-Tumoren rekapitulieren können und für präklinische Modellewertvoll sind 18, sind sie von Natur aus teuer, geringer Durchsatz, zeitaufwendig und beinhalten ethische Bedenken19. Kulturen von Patientenzellen, auf 2D-Kunststoffoberflächen oder als Sphäroide vermeiden diese Probleme meistens. Während patientenabgeleitete Zellen genetische Aberrationen bewahren, waren ihre Kulturen in 2D oder als suspendierte Sphäroide weitgehend schlechte Darstellungen von patientenabgeleiteten Xenotransplantaten in Nagetieren und ursprünglichen Patiententumoren20. Zuvor haben wir und andere gezeigt, dass GBM-Zellen, die in einem 3D-ECM kultiviert werden, das die mechanischen und biochemischen Eigenschaften des Hirngewebes nachahmt, die Phänotypen 10,21,22,23 der Arzneimittelresistenz erhalten können.
Wechselwirkungen zwischen Hyaluronsäure (HA), einem Polysaccharid, das im Gehirn-ECM reichlich vorhanden ist und in GBM-Tumoren überexprimiert wird, und seinem CD44-Rezeptor modulieren den Erwerb von Arzneimittelresistenzen in vitro 21,24,25,26,27. Zum Beispiel erhöhte die Einbeziehung von HA in weiche 3D-Kulturen die Fähigkeit von von Patienten abgeleiteten GBM-Zellen, therapeutische Resistenzen zu erlangen. Diese Mechano-Responsivität war abhängig von der HA-Bindung an CD44-Rezeptoren auf GBM-Zellen21. Zusätzlich verstärkte die Integrinbindung an RGD-tragende Peptide, die in 3D-Kulturmatrizen eingebaut wurden, CD44-vermittelte Chemoresistenz in einer steifigkeitsabhängigen Weise21. Über HA hinaus variiert die Expression mehrerer ECM-Proteine, von denen viele RGD-Regionen enthalten, zwischen normalen Hirn- und GBM-Tumoren28. Zum Beispiel berichtete eine Studie, dass 28 verschiedene ECM-Proteine in GBM-Tumoren hochreguliert wurden29. Innerhalb dieser komplexen Tumormatrix-Mikroumgebung integrieren Krebszellen mechanische und biochemische Hinweise, um einen bestimmten Resistenzphänotyp zu erhalten, der von relativ kleinen Unterschieden (z. B. weniger als einer Größenordnung) im Elastizitätsmodul oder der Dichte der integrinbindenden Peptide28,29,30 abhängt.
Das vorliegende Protokoll charakterisiert, wie Tumorzellen einzigartige Kombinationen von Matrixhinweisen interpretieren und komplexe, patientenspezifische Matrixmikroumgebungen identifizieren, die die Behandlungsresistenz fördern (Abbildung 1A). Eine photochemische Methode zur Erzeugung miniaturisierter, präzise abgestimmter Matrizen für die 3D-Kultur bietet einen großen, orthogonalen variablen Raum. Ein speziell angefertigtes Array von LEDs, die von einem Mikrocontroller betrieben werden, wurde integriert, um Hydrogele innerhalb eines 384-Well-Plattenformats zu vernetzen, um die Automatisierung und Reproduzierbarkeit zu erhöhen. Die Expositionsintensität wurde im gesamten Bohrloch variiert, um die mikromechanischen Eigenschaften der resultierenden Hydrogele zu verändern, wie sie mittels Rasterkraftmikroskopie (AFM) bewertet wurden. Während sich dieses Manuskript nicht auf die Konstruktion des Illuminationsarrays selbst konzentriert, werden ein Schaltplan (Abbildung 1B) und eine Stückliste (Table of Materials) als Hilfsmittel für die Gerätereproduktion bereitgestellt.
Dieser Bericht zeigt die schnelle Erzeugung einer Reihe von GBM-Zellen, die in einzigartigen 3D-Mikroumgebungen kultiviert wurden, in denen der Elastizitätsmodul (vier Ebenen über eine einzige Größenordnung) und der integrinbindende Peptidgehalt (abgeleitet von vier verschiedenen ECM-Proteinen) orthogonal variiert waren. Der Ansatz wurde dann verwendet, um die relativen Beiträge der Hydrogelmechanik und des ECM-spezifischen Integrin-Engagements auf die Lebensfähigkeit und Proliferation von patientenabgeleiteten GBM-Zellen zu untersuchen, wenn sie eine Resistenz gegen Temozolomid (TMZ) -Chemotherapie erwerben.
Protokoll
Von Patienten abgeleitete GBM-Zelllinien (GS122 und GS304) wurden von Professor David Nathanson (unserem Mitarbeiter) zur Verfügung gestellt, der diese Linien im Rahmen eines vom UCLA Institutional Review Board genehmigten Protokolls (IRB # 10-000655) entwickelte. Zellen wurden de-identifiziert zur Verfügung gestellt, so dass die Zelllinien nicht mit den einzelnen Patienten in Verbindung gebracht werden konnten.
1. Herstellung von Hydrogellösung
- Bereiten Sie HEPES-gepufferte Lösung vor, indem Sie HEPES-Pulver bei 20 mM in Hank's Balanced Salt Solution (HBSS) auflösen. Stellen Sie den pH-Wert nach vollständiger Solvatation auf 7 ein.
- In der HEPES-gepufferten Lösung wird thioliertes HA (700 kDa Nominalmolekulargewicht, siehe Materialtabelle), das nach dem vorherigen Bericht31 hergestellt wurde, so gelöst, dass 6%-8% der Carbonsäurereste auf jeder Glucuronsäure mit einem Thiol modifiziert werden, in einer Konzentration von 10 mg/ml in Pufferlösung.
HINWEIS: Eine bernsteinfarbene Durchstechflasche wird empfohlen, um die Oxidation von Thiol durch Umgebungslicht zu verhindern.- Mit einer magnetischen Rührplatte (<1.000 U/min) bei Raumtemperatur umrühren, bis sie sich vollständig aufgelöst hat, typischerweise etwa 45 min.
- Während sich HA auflöst, bereiten Sie separate Lösungen von (1) 100 mg/ml 8-arm-PEG-Norbornen (20 kDa), (2) 100 mg/ml 4-arm-PEG-Thiol (20 kDa), (3) 4 mM Cystein oder Cystein-haltigem Peptid (z. B. GCGYGRGDSPG) und (4) 4 mg/ml LAP in Mikrozentrifugenröhrchen vor (siehe Materialtabelle).
- Bereiten Sie jede dieser vier Lösungen in der HEPES-gepufferten Lösung vor, die in Schritt 1.1 vorbereitet wurde. Wirbeln Sie die Lösungen um, um eine vollständige Auflösung jedes Reagenzes sicherzustellen, bevor Sie Schritt 4 ausführen.
HINWEIS: Wenn Sie mehrere verschiedene Peptide testen, muss jedes eine Cystein- oder eine andere Quelle von Thiol-Einheit für diese Konjugationschemie enthalten. - Bereiten Sie Lösungen (4 mM verfügbares Thiol) aller Peptide vor, die an dieser Stelle in einem einzigen Hydrogel angebunden werden sollen.
HINWEIS: Peptidsequenzen und ECM-Proteine, von denen sie abgeleitet und in dieser Studie verwendet wurden, sind in Tabelle 1 aufgeführt. N-Acetylcystein (siehe Materialtabelle), an das Zellen nicht binden, kann durch ein bioaktives, thiolhaltiges Peptid ersetzt werden, um die Konzentration eines adhäsiven Peptids zu titrieren oder als Negativkontrolle zu wirken31.
- Bereiten Sie jede dieser vier Lösungen in der HEPES-gepufferten Lösung vor, die in Schritt 1.1 vorbereitet wurde. Wirbeln Sie die Lösungen um, um eine vollständige Auflösung jedes Reagenzes sicherzustellen, bevor Sie Schritt 4 ausführen.
- Mischen Sie die einzelnen Lösungen von HA, PEG-Norbornen, PEG-Thiol und Cystein/Thiol-haltigen Peptiden (siehe Materialtabelle), um die Endkonzentrationen für die in Tabelle 2 aufgeführten endgültigen Hydrogelmatrizen zu erreichen. Rühren Sie (<1.000 U / min) auf einer magnetischen Rührplatte für mindestens 30 Minuten, um vollständig zu mischen.
HINWEIS: HA-Lösungen sind hochviskos und werden am besten mit einer Verdrängerpipette gehandhabt (siehe Materialtabelle). Ist eine Verdrängerpipette nicht verfügbar, können viskose Lösungen auch durch langsames Pipettieren mit breitblendigen Spitzen auf eine Standard-Mikropipette verzichtet werden.
2. Beleuchtung und Photovernetzung von Hydrogelen über ein LED-Array
ACHTUNG: Tragen Sie UV-Schutzbrillen und bedecken Sie das Beleuchtungsfeld mit UV-absorbierendem Material.
HINWEIS: Das in diesem Protokoll beschriebene LED-Array besteht aus sechs Sätzen von acht LEDs, die in Reihe geschaltet sind, wie der mitgelieferte Schaltplan zeigt (Abbildung 1A). Jeder Satz LEDs kann unabhängig voneinander mit Strom versorgt werden, was bis zu sechs verschiedene Bestrahlungsstärken pro Durchlauf ermöglicht. Die Zusatzdatei 1 enthält Screenshots, die den folgenden Anweisungen für weitere Anleitungen entsprechen.
- Laden Sie die Datei Illumination Device.zip aus den Supplementary Coding Files herunter. Dieses Verzeichnis enthält die folgenden Dateien: Arduino.zip (Supplementary Coding File 1), Drivers.zip (Supplementary Coding File 2), GUI.zip (Supplementary Coding File 3) und Holder.zip (Supplementary Coding File 4).
HINWEIS: 3D-Druck des oberen und unteren Teils, um die Leiterplatte an Ort und Stelle zu halten (siehe Ergänzende Codierungsdateien für Details). - Laden Sie die Mikrocontroller-Software herunter und installieren Sie sie (siehe Materialverzeichnis).
- Laden Sie die GUI-Software herunter und installieren Sie sie (siehe Materialverzeichnis). Eine Bedienungsanleitung für Software finden Sie in der Zusatzdatei 1.
- Öffnen Sie Processing und installieren Sie die controlIP5-Bibliothek, indem Sie auf Sketch > Import Library > Add Library klicken. Suchen Sie dann in Bibliotheken nach controlIP5 und klicken Sie auf Installieren. Führen Sie dies zum ersten Mal durch.
- Versorgen Sie das Beleuchtungsgerät (siehe Materialtabelle) über das 36-Volt-Netzteil mit Strom und schließen Sie es über ein Micro-USB-Kabel an einen PC an.
HINWEIS: Einige Geräte installieren Treiber für verschiedene Arduino Nano-Boards nicht automatisch. Ein Satz von Treibern wird in der Zip-Datei des Geräts bereitgestellt. - Öffnen Sie die Datei Arduino.ino im Ordner Adruino.zip mit Arduino IDE.
- Kompilieren Sie die Datei Arduino.ino, indem Sie auf die Schaltfläche Häkchen klicken. Laden Sie den kompilierten Code hoch, indem Sie auf die Schaltfläche Pfeil klicken.
- Öffnen Sie die Datei GUI.pde, die sich im Ordner GUI.zip befindet, mithilfe von Processing.
- Klicken Sie im Verarbeitungsprogramm auf Ausführen , um die grafische Benutzeroberfläche zur Steuerung des Beleuchtungsgeräts zu starten.
- Klicken Sie im Fenster der grafischen Benutzeroberfläche auf Intensität, damit die Spalte mit der Hydrogel-Vorläuferlösung vernetzt werden soll, und geben Sie die gewünschte Intensität ein. Klicken Sie auf das Feld Zeit und geben Sie die gewünschte Zeit ein. Für die in Tabelle 2 bereitgestellte Lösung sind dies 15 s.
HINWEIS: Endbenutzer müssen digitale Intensitätswerte mit einem Radiometer auf Bestrahlungsstärke kalibrieren. Beispiele für typische Intensitäten sind in Abbildung 2A dargestellt. - Richten Sie die Proben mit der Beleuchtungsvorrichtung (Abbildung 2B) mit jeder anderen LED in einer einzigen Spalte der Silikonformen (siehe Materialtabelle) oder der 384-Well-Platte aus. Klicken Sie auf Fertig stellen , um die Beleuchtung zu starten. Wiederholen Sie diesen Vorgang nach Bedarf für die Beleuchtung mehrerer Dias oder anderer Vertiefungen einer 384-Well-Platte.
HINWEIS: Die Halterung ist so konzipiert, dass die 384-Well-Platte während der Beleuchtung bündig mit einer Ecke der Innenkammer sitzt.- Wenn Sie nach der Beleuchtung in einer Ecke platziert sind, bewegen Sie die Bohrlochplatte in die nächste Ecke und wiederholen Sie den Vorgang. Um Vertiefungen auf der anderen Hälfte der Platte zu beleuchten, heben Sie die Platte aus der Halterung und drehen Sie sie um 180°.
- Erzeugen Sie Hydrogele mit unterschiedlicher Mechanik für die mechanische Charakterisierung mit den folgenden Schritten.
- Reinigen Sie die Glasobjektträger und Silikonformen mit Klebeband, um Ablagerungen zu entfernen. Kleben Sie die Silikonformen auf den Glasschieber, drücken Sie sie nach unten, um eine gute Abdichtung zu gewährleisten, und verdrängen Sie Luftblasen.
- Pipette 80 μL Hydrogel-Vorläuferlösung, wie in Schritt 1.4 hergestellt, in jede Silikonform auf dem Glasobjektträger.
- Legen Sie den Glasschieber in einer einzigen Spalte auf das Beleuchtungsgerät, das mit jeder anderen LED ausgerichtet ist. Setzen Sie die Hydrogel-Vorstufen 15 s lang UV-Licht aus, wie in Schritt 2 beschrieben, der Photovernetzung.
- Sobald die Beleuchtung aufgehört hat, holen Sie die Objektträger zurück und lösen Sie die Gele aus den Formen, indem Sie den Innenumfang der Form mit einer feinen Spitze (10 μL Pipettenspitze, 30 G Nadel usw.) verfolgen. Entfernen Sie Silikonformen mit einer Pinzette/Pinzette.
- Bewegen Sie vernetzte Hydrogele in einzelne Vertiefungen einer 12-Well-Platte, indem Sie einen Spatel benetzen und vorsichtig vom Glasobjektträger drücken. Füllen Sie jede Vertiefung mit 2 ml DPBS (siehe Materialtabelle), bevor Sie das Hydrogel hinzufügen. Die Gele in DPBS-Lösung mindestens 12 h (typischerweise über Nacht) bei Raumtemperatur aufquellen lassen (für die mechanische Charakterisierung am nächsten Tag).
3. Messungen der Rasterkraftmikroskopie (AFM)
- Schalten Sie das Rasterkraftmikroskop (AFM) gemäß den Anweisungen des Herstellers ein (siehe Materialtabelle). Dieses Protokoll enthält kurze Anweisungen zur Verwendung des Geräts und der zugehörigen Software.
- Installieren Sie die AFM-Sonde (siehe Materialtabelle).
HINWEIS: Für die vorliegende Studie wurde ein dreieckiger Siliziumnitrid-Cantilever mit einer nominalen Federkonstante von 0,01 N/m mit einem sphärischen 2,5 μm Siliziumdioxidpartikel modifiziert. - Richten Sie den Laser nach der Installation an der Spitze der dreieckigen Sonde aus und passen Sie dann die Spiegel- und Laserablenkung an, um die Signalsumme zu maximieren (normalerweise zwischen 1,5 und 2,2 Volt).
- Tauchen Sie die Sonde in DPBS ein und warten Sie bis zu 15 Minuten, um ein thermisches Gleichgewicht zu erreichen. Klicken Sie auf die Schaltfläche Kalibrierung und wählen Sie Kontaktabhängige Kalibrierung. Klicken Sie auf die Schaltfläche Thermal Tuning sammeln, und wählen Sie nach der Datenerfassung den Peak um 3 kHz für die Kalibrierung aus.
HINWEIS: Nach dem Eintauchen in eine Flüssigkeit aufgrund von Brechungsindexänderungen kann eine leichte Anpassung des Spiegels und der Laserabweiser erforderlich sein. - Nähern Sie sich der Oberfläche einer Petrischale (Kunststoff), indem Sie die Annäherungsparameter auf konstante Geschwindigkeit, eine Zielhöhe von 7,5 μm und eine Annäherungsgeschwindigkeit von 15 μm/s einstellen. Aktivieren Sie die Baseline-Messung pro Lauf für Anflug, damit der Anflug kontinuierlich verläuft und nicht aufgrund von Drift im Deflektor vorzeitig anhält.
- Legen Sie bei der Annäherung die Erfassungsparameter für das Forcemapping auf 4 nN-Turnarounds, 2 μm Eindringabstand, 1 μm/s Geschwindigkeit und 0 s Kontaktzeit fest. Drücken Sie die Starttaste , um eine Kraftkurve auf der Kunststoffoberfläche (z. B. einer Bohrlochplatte) zu erfassen.
- Kehren Sie zum Kalibrierungsfenster zurück und wählen Sie den Teil der Kraftkurve aus, der dem Kontakt und der Einbuchtung des Kunststoffs entspricht. Akzeptieren Sie die berechneten Empfindlichkeits- und Steifigkeitswerte für die Sonde, um die Kalibrierung abzuschließen.
- Heben Sie nach der Kalibrierung die AFM-Sonde an und legen Sie die Hydrogelprobe zur Abfrage ein. Nähern Sie sich Hydrogel gemäß den Einstellungen in Schritt 5.
HINWEIS: Während des Annäherungsvorgangs auf die Hydrogeloberfläche kann das Gerät fälschlicherweise den angenäherten Zustand auslösen. Um den tatsächlichen Ansatz zu überprüfen, erhalten Sie eine Kraftkurve wie in Schritt 4.6. Wiederholen Sie den Annäherungsvorgang, wenn die resultierende Kurve keinen Kontakt und keine resultierende Einrückung aufweist. - Wenn der Oberflächenansatz erfolgreich ist, wechseln Sie in den Force-Mapping-Modus und legen Sie die Erfassungsparameter auf eine Karte von 8 x 8 Größe mit einer Länge von 40 μm pro Achse fest. Erhalten Sie Kraftkarten in verschiedenen Regionen, um die Gleichmäßigkeit der Steifigkeitsmessungen zu beurteilen.
- Interpretation von Kraftkurven mit dem Softwareprogramm JPK SPM Data Processing durch eine Hertz/Sneddon-Modellanpassung (Gleichungen 1 und 2, siehe Tabelle 3 zur Definition aller Variablen) mit ausgewähltersphärischer Geometrie 32,33,3 4.
 Gleichung 132
Gleichung 132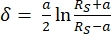 Gleichung 232
Gleichung 232
- Interpretation von Kraftkurven mit dem Softwareprogramm JPK SPM Data Processing durch eine Hertz/Sneddon-Modellanpassung (Gleichungen 1 und 2, siehe Tabelle 3 zur Definition aller Variablen) mit ausgewähltersphärischer Geometrie 32,33,3 4.
4. Aufbau und medikamentöse Behandlung von 3D-, Matrix-Embedded-Kulturen
- Bereiten Sie die gewünschten Zellen als Einzelzelllösung vor.
HINWEIS: Verschiedene Zelltypen erfordern möglicherweise unterschiedliche Passaging-Methoden. Ein typisches Protokoll für das Passieren einer Suspensionskultur von GBM-Sphäroiden aus einem T-75-Kolben wird in Referenz31 berichtet. - Sammeln Sie GBM-Sphäroide (etwa 150 μm Durchmesser) aus einer T-75-Kolbensuspensionskultur in ein 15 ml konisches Rohr. Spülen Sie den Kulturkolben mit 5 ml DPBS ab, um Restzellen und Medien zu entfernen, und fügen Sie dieses Volumen dem konischen Röhrchen hinzu.
- Das konische Röhrchen, das die Zellen enthält, bei 200 x g für 5 min bei Raumtemperatur zentrifugieren. Entfernen Sie nach der Zentrifugation den Überstand mit einer serologischen 5-ml-Pipette, achten Sie darauf, das Zellpellet nicht zu stören, und resuspendieren Sie in 5 ml DPBS.
- Zentrifen Sie bei 200 x g für 5 min bei Raumtemperatur, um Zellen zu waschen. Saugen Sie den Überstand mit einer serologischen 5-ml-Pipette ab, achten Sie darauf, das Zellpellet nicht zu stören, und resuspendieren Sie dann die Zellen in 2 ml Zelldissoziationsreagenz (siehe Materialtabelle).
- Bei Raumtemperatur für 10-15 min inkubieren. Fügen Sie 3 ml vollständiges Medium hinzu (siehe Materialtabelle) und pipettieren Sie vorsichtig 3-5 Mal, um die Sphäroide zu einer Einzelzellsuspension31 abzubauen.
- Zentrifugieren Sie die Einzelzellsuspension bei 400 x g (Einzelzellsuspensionen können für die Pelletbildung schneller gesponnen werden) für 5 Minuten auf Pelletzellen bei Raumtemperatur. Saugen Sie den Überstand mit einer 5 ml serologischen Pipette ab und achten Sie darauf, das Zellpellet nicht zu stören. Resuspendieren Sie Zellen in 1 ml vollständigem Medium.
HINWEIS: Wenn die Zellen nach dem Passieren in Klumpen und nicht als einzelne Zellen in Suspension verbleiben, können Zellen durch ein 40-μm-Zellsieb geleitet werden, um eine Einzelzellsuspension zu erreichen. - Entfernen Sie einen Teil der Zellen zum Zählen mit einem Hämozytometer. Verdünnen Sie diese Portion zweifach mit Trypanblau, das Zellen mit beeinträchtigter Lebensfähigkeit durchdringt. Zählen Sie nur die lebenden, farblosen Zellen. Typischerweise liefert ein T-75, der mit 800.000 Zellen pro Kolben ausgesät ist, nach einer Woche in Kultur 2-3 Millionen Zellen.
- Bestimmen Sie die Anzahl der Zellen, die für die Verkapselung erforderlich sind. Überführen Sie ein Medienvolumen mit der Gesamtzahl der benötigten Zellen in ein steriles 1,7-ml-Mikrozentrifugenröhrchen. Bei Raumtemperatur bei 400 x g für 5 min nach unten drehen.
HINWEIS: Zum Beispiel werden mindestens 2,5 Millionen Zellen, die in 1 ml Gelvolumen resuspendiert sind, benötigt, um Zellen mit 2,5 Millionen Zellen / ml zu verkapseln. Ein Gelvolumen von 1 ml ermöglicht es Benutzern, 100 Geltropfen abzugeben, wobei jeder Geltropfen ein Volumen von 10 μL hat. Es wird empfohlen, ein zusätzliches ~ 20% -Volumen von Zellen herzustellen, die in Hydrogellösung suspendiert sind, um den Verlust während des Pipettentransfers zu berücksichtigen. Somit würde man in diesem Beispiel 3 Millionen Zellen und 1,2 ml Hydrogel-Vorläuferlösung herstellen. Eine Mindestdichte von 500 Tausend Zellen / ml wird empfohlen. - Saugen Sie den Überstand mit einer Mikropipette ab und achten Sie darauf, das Zellpellet nicht zu stören. Resuspendiert das Zellpellet in der Hydrogel-Vorläuferlösung, wie in Schritt 1.4 hergestellt, und mischt gut, indem es 4-5 Mal mit einer 1.000-μL-Mikropipette auf und ab pipettiert wird.
- Laden Sie die Zellen in einen Wiederholungspipettierer (siehe Materialtabelle), der für die Abgabe von 10 μL eingestellt ist. Um Blasen und ungleichmäßiges Dosieren zu vermeiden, grundieren Sie den Wiederholungspipettierer, indem Sie zusätzlich 1-2 Mal in einen Abfallbehälter dosieren.
- Geben Sie in jeder Vertiefung einer 384-Well-Platte 10 μL Zellen ab, die in Hydrogellösung aus dem Wiederholungspipettor suspendiert sind. Beleuchten Sie mit dem LED-Array jede Vertiefung, die Zellen enthält (Schritt 2) für 15 s mit Intensitäten (Beispielergebnisse in Abbildung 2A verwendete Intensitäten von 1,14, 1,55, 2,15, 2,74 mW/cm2), um die gewünschten mechanischen Eigenschaften zu erreichen.
HINWEIS: Es wird empfohlen, mit fünf Replikaten pro experimenteller Bedingung zu beginnen und je nach gewünschtem Durchsatz und Varianz des Endpunktassays nach oben oder unten zu skalieren. - Fügen Sie 40 μL vollständiges Medium zu jeder Vertiefung hinzu, die die Zellen enthält. Fügen Sie 50 μL DPBS zu nicht-experimentellen, trockenen Vertiefungen hinzu, die die Gele umgeben, um Verluste durch Verdunstung zu minimieren.
- Für GBM-Zellen fügen Sie 40 μL des medienhaltigen Arzneimittels (z. B. TMZ, siehe Materialtabelle) hinzu, um die endgültige gewünschte Konzentration (10 μM-100 μM in Dimethylsulfoxid (DMSO) oder Vehikel (DMSO) zu erreichen, entsprechend beginnend 3 Tage nach der Verkapselung.
5. CCK8-Proliferationsassay
- Geben Sie 10 μL CCK8-Reagenz (siehe Materialtabelle) zu jeder Vertiefung, die die Zellen enthält.
HINWEIS: Wenn Sie diesen Assay zum ersten Mal durchführen, schließen Sie Negativkontrollquellen wie Medien oder zellfreies Hydrogel in Medien ein. - Inkubieren Sie für 1-4 h gemäß den Anweisungen des Herstellers.
HINWEIS: Diese Zeit kann je nach Zelltyp und Dichte variieren, und daher müssen die Inkubationszeiten für jede Anwendung getestet werden, so dass die Absorptionswerte in einen linearen Bereich fallen, eine Voraussetzung für die Anwendung des Biergesetzes35. - Ablesungsraten bei 450 nm für alle Vertiefungen nach der Inkubation.
- Berechnen Sie die durchschnittliche Absorption bei 450 nm, die in Schritt 3 für den Fahrzeugzustand für jede Gruppe erhalten wurde. Teilen Sie jedes gut behandelte Medikament durch den Durchschnitt der Fahrzeugkontrolle pro Gruppe.
- Berechnen Sie Konfidenzintervalle, indem Sie Bootstrap-Verteilungen (N = 10.000) mit der Perzentilmethode36 generieren.
HINWEIS: Im Allgemeinen kann man 95% Konfidenzintervalle verwenden und Bedingungen, deren Konfidenzintervalle nicht über 1 überschreiten, als signifikant interpretieren und weitere Untersuchungen rechtfertigen. Das Festlegen von Konfidenzintervallen auf 95 % ist deckungsgleich mit dem Festlegen eines Signifikanzgrenzwerts von p = 0,05. Für die in den Ergebnissen gezeigten Daten ist es nützlich, Bedingungen zu unterscheiden, die matrixvermittelte Arzneimittelresistenzen entweder fördern oder hemmen und eine beidseitige Analyse erfordern.
Ergebnisse
AFM-Messungen bestätigten die präzise Kontrolle der Hydrogelmechanik in Abhängigkeit von der UV-Bestrahlungsstärke (mW/cm2) während der Fotovernetzung mit einem speziell angefertigten, Arduino-gesteuerten LED-Array (Abbildung 2A). Die in diesem Protokoll verwendete Hydrogelformulierung ist in Tabelle 2 aufgeführt. Der Abstand der LEDs auf der mitgelieferten Schablone stimmt mit dem Abstand für jede andere Vertiefung einer 384-Well-Platte überein, wodurch d...
Diskussion
Die aktuelle Arbeit präsentiert Methoden zur Erzeugung von 3D-, miniaturisierten Kulturen innerhalb von HA-basierten bei gleichzeitiger Veränderung der Matrixsteifigkeit und der für das Integrin-Engagement verfügbaren Peptide. Diese Technik ermöglicht die systematische Untersuchung, wie Matrixparameter zelluläre Phänotypen (z. B. die Lebensfähigkeit von Krebszellen, die einer Chemotherapie ausgesetzt sind) mit erhöhtem Durchsatz beeinflussen. Frühere Ansätze, einschließlich der hier vorgestellten, haben die H...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren möchten Carolyn Kim, Amelia Lao, Ryan Stoutamore und Itay Solomon ausdrücklich für ihre Beiträge zu früheren Iterationen des Photogelationsschemas danken. Die Zelllinien GS122 und GS304 wurden großzügig von David Nathanson zur Verfügung gestellt. Alle Figuren wurden mit BioRender.com erstellt. Die Kerneinrichtungen der UCLA, die Molecular Screening Shared Resources und das Nano and Pico Characterization Laboratory waren maßgeblich an der Arbeit beteiligt. Chen Chia-Chun wurde von der UCLA Eli und dem Edythe Broad Center of Regenerative Medicine and Stem Cell Research Training Program unterstützt. Grigor Varuzhanyan wurde durch ein Tumor Cell Biology Training Program NIH Grant (T32 CA 009056) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.1 kOhm resistors, 6 W | Digikey | 35601k1ft | |
| 1.7 mL microcentrifuge tube | Genesse Scientific | 21-108 | |
| 15 mL conical tube | Fisher Scientific | 14-959-70C | |
| 365 nm LED | Digikey | ltpl-c034uvh365 | |
| 384 well plate | Bio Greiner One | 781090 | |
| 40 µm cell strainer | MTC bio | C4040 | |
| 4-Armed thiol terminated polyethlene glycol (20 kDa) | Laysan Bio | 4arm-PEG-SH-20K-1g | |
| 6 NPN BJTs | Digikey | 2n5550ta | |
| 80 Ohm resistors, 0.125 W | Digikey | erjj-6enf80r6v | |
| 8-Armed norbornene terminated polyethylene glycol (20 kDa) | Jenkem Technology | A7025-1 | |
| Accutase | Innovative Cell Technologies | AT104500 | cell dissociation reagent |
| AFM Probes | Novascan | 0.01 N/m Nominal spring constant, 2.5 µm SiO2 particle | |
| Arduino IDE | Arduino | 1.8.19 | |
| Arduino Nano | Makerfire | Mini Nano V3.0 ATmega328P Microcontroller Board | |
| bFGF | Peprotech | 100-18B | 20 ng/mL |
| CCK8 | Abcam | ab228554 | |
| Centrifuge | Thermoscientific | sorvall legend xtr | |
| CP100ST | Gilson | F148415 | Pipette tips for positive displacement pipette |
| Cubis Semi-Micro Balance | Sartorius | MSA225S100DI | |
| DMEM - F12 (50-50) | Life Technologies | 11330057 | 1x |
| DMSO | Fisher Scientific | BP231-100 | |
| DPBS Ca (-) Mg (-) | Genesse Scientific | 25-508 | |
| EGF | Peprotech | AF100-15 | 50 ng/mL |
| Ethanol, Anhydrous | Fisher Scientific | A405P | Add DI water to dilute to 70% |
| Fisherbrand Class B Amber Glass threaded vials | Fisher Scientific | 03-339-23C | |
| Fisherbrand Weighing Paper | Fisher Scientific | 09-898-12B | |
| G21 Supplement | Gemini Bio | 400-160 | 50x |
| Hanks Balanced Salt Solution | Thermo Fisher Scientific | 14175095 | |
| HCl, ACS, 12M | Sigma Aldrich | S25838A | Add DI water to dilute to 1 M |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H3149-100Ku | 25 µg/mL |
| HEPES | Sigma Aldrich | H7006-100G | |
| Hot Air Gun | Wagner | HT1000 | |
| Integrin-binding sialoprotein (IBSP) peptide | Genscript | Custom Order | GCGYGGGGNGEPRGDTYRAY |
| Lithium phenyl-2,4,6 trimethylbenzoylphosphinate (LAP) , >95% | Sigma Aldrich | 900889-1G | |
| Magnetic stir plate | Thermo Scientific | SP194715 | |
| Microcentrifuge | Thermo Scientific | Sorvall legend micro 21R | |
| Microman E single Channel Pipettor | Gilson | FD10004 | Positive displacement pipette |
| Micropipette Tips | Various Manufacturs | Various sizes | |
| mLine micropipette | Sartorious | ||
| N-acetyl Cysteine | Sigma Aldrich | A7250-10G | |
| Nanowizard 4 | Bruker | AFM microscope | |
| NaOH | Fisher Scientific | ss255-1 | Add DI water to dilute to 1 M |
| Normoicin | Invivogen | ant-nr-1 | 500x |
| Osteopontin Peptide | Genscript | Custom Order | GCGYGTVDVPDGRGDSLAYG |
| Pipet Aid | Drummond | 4000102 | |
| Plain Microscope Slides | Globe Scientific | 1301 | |
| Press-To-Seal silicone Isolator, 12-4.5mm diam x 2mm deep | Grace Bio Labs | 664201-A | Cut so that 8 individual molds are made from a single sheet |
| Processing | Processing | 3.5.4 | |
| Repeater M4 | Eppendorf | 4982000322 | |
| Repeater Pipette Tips | Sartorious | 30089430 | 1 mL sizes |
| RGD Peptide | Genscript | GCGYGRGDSPG | |
| Scoth Tape | |||
| Serological Pipettes | Genesse Scientific | 12-102,12-104 | 5,10 mL Pipettes |
| Solder Paste | Digikey | 315-NC191LT15T5-ND | |
| Solder Wire | |||
| Straight dissecting forceps | VWR Scientific | 82027-408 | |
| Synergy H1 Plate Reader | Biotek | ||
| T-75 Cell Culture Treated Flask | Genesee Scientific | 25-209 | |
| Temozolomide | Sigma Aldrich | T2577 | Typically used from 10 µM to 100 µM |
| Tenascin-C Peptide | Genscript | GCGYGRSTDLPGLKAATHYTITIR GV | |
| Thiolated Hyaluronic Acid (700 kDa), 6-8% modified | Lifecore Biomedical | HA700K5 | |
| VWR Spinbar, Flea Micro | VWR | 58948-375 |
Referenzen
- Scannell, J. W., Blanckley, A., Boldon, H., Warrington, B. Diagnosing the decline in pharmaceutical R&D efficiency. Nature Reviews Drug Discovery. 11 (3), 191-200 (2012).
- Waring, M. J., et al. An analysis of the attrition of drug candidates from four major pharmaceutical companies. Nature Reviews Drug Discovery. 14 (7), 475-486 (2015).
- Khozin, S., Liu, K., Jarow, J. P., Pazdur, R. Why do oncology drugs fail to gain US regulatory approval. Nature Reviews Drug Discovery. 14 (7), 450-451 (2015).
- Booth, B., Ma, P., Glassman, R. Oncology's trials. Market indicators. Nature Reviews Drug Discovery. 2 (8), 609-610 (2003).
- Da Ros, M., et al. Glioblastoma chemoresistance: The double play by microenvironment and blood-brain barrier. International Journal of Molecular Sciences. 19 (10), 2879 (2018).
- Broekman, M. L., et al. Multidimensional communication in the microenvirons of glioblastoma. Nature Reviews Neurology. 14 (8), 482-495 (2018).
- Grundy, T. J., et al. Differential response of patient-derived primary glioblastoma cells to environmental stiffness. Scientific Reports. 6 (1), 1-10 (2016).
- Gomez-Roman, N., Stevenson, K., Gilmour, L., Hamilton, G., Chalmers, A. J. A novel 3D human glioblastoma cell culture system for modeling drug and radiation responses. Neuro-Oncology. 19 (2), 229-241 (2017).
- Simoni, R. D., et al. Basement membrane complexes with biological activity. Biochemistry. 25 (2), 312-318 (2002).
- Xiao, W., et al. Brain-mimetic 3D culture platforms allow investigation of cooperative effects of extracellular matrix features on therapeutic resistance in glioblastoma. Cancer Research. 78 (5), 1358-1370 (2018).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nature Reviews Materials. 5 (7), 539-551 (2020).
- Spinelli, C., et al. Molecular subtypes and differentiation programmes of glioma stem cells as determinants of extracellular vesicle profiles and endothelial cell-stimulating activities. Journal of Extracellular Vesicles. 7 (1), 1490144 (2018).
- Ostrom, Q. T., Cioffi, G., Waite, K., Kruchko, C., Barnholtz-Sloan, J. S. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2014-2018. Neuro-Oncology. 23, (2021).
- Stupp, R., et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma. New England Journal of Medicine. 352 (10), 987-996 (2005).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Tomczak, K., Czerwińska, P., Wiznerowicz, M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge. Contemporary oncology. 19, 68-77 (2015).
- Lee, S. Y. Temozolomide resistance in glioblastoma multiforme. Genes and Diseases. 3 (3), 198-210 (2016).
- Joo, K. M., et al. Patient-specific orthotopic glioblastoma xenograft models recapitulate the histopathology and biology of human glioblastomas in situ. Cell Reports. 3 (1), 260-273 (2013).
- Levy, N. The use of animal as models: Ethical considerations. International Journal of Stroke. 7 (5), 440-442 (2012).
- Phon, B. W. S., Kamarudin, M. N. A., Bhuvanendran, S., Radhakrishnan, A. K. Transitioning preclinical glioblastoma models to clinical settings with biomarkers identified in 3D cell-based models: A systematic scoping review. Biomedicine & Pharmacotherapy. 145, 112396 (2022).
- Xiao, W., et al. Bioengineered scaffolds for 3D culture demonstrate extracellular matrix-mediated mechanisms of chemotherapy resistance in glioblastoma. Matrix Biology. 85-86, 128-146 (2020).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Xu, X., Farach-Carson, M. C., Jia, X. Three-dimensional in vitro tumor models for cancer research and drug evaluation. Biotechnology Advances. 32 (7), 1256-1268 (2014).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived Glioblastoma Cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Preston, M. Digestion products of the PH20 hyaluronidase inhibit remyelination. Annals of Neurology. 73 (2), 266-280 (2013).
- Kim, Y., Kumar, S. CD44-mediated adhesion to hyaluronic acid contributes to mechanosensing and invasive motility. Molecular Cancer Research. 12 (10), 1416-1429 (2014).
- Pibuel, M. A., Poodts, D., Díaz, M., Hajos, S. E., Lompardía, S. L. The scrambled story between hyaluronan and glioblastoma. The Journal of Biological Chemistry. 296, 100549 (2021).
- Xiao, W., Sohrabi, A., Seidlits, S. K. Integrating the glioblastoma microenvironment into engineered experimental models. Future Science OA. 3 (3), (2017).
- Trombetta-Lima, M., et al. Extracellular matrix proteome remodeling in human glioblastoma and medulloblastoma. Journal of Proteome Research. 20 (10), 4693-4707 (2021).
- Schregel, K., et al. Characterization of glioblastoma in an orthotopic mouse model with magnetic resonance elastography. NMR in Biomedicine. 31 (10), 3840 (2018).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived glioblastoma cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Guz, N., Dokukin, M., Kalaparthi, V., Sokolov, I. If cell mechanics can be described by elastic modulus: Study of different models and probes used in indentation experiments. Biophysical Journal. 107 (3), 564-575 (2014).
- Sneddon, I. N. The relation between load and penetration in the axisymmetric boussinesq problem for a punch of arbitrary profile. International Journal of Engineering Science. 3 (1), 47-57 (1965).
- Soofi, S. S., Last, J. A., Liliensiek, S. J., Nealey, P. F., Murphy, C. J. The elastic modulus of MatrigelTM as determined by atomic force microscopy. Journal of Structural Biology. 167 (3), 216-219 (2009).
- Mayerhöfer, T. G., Popp, J. Beer's law - Why absorbance depends (almost) linearly on concentration. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 20 (4), 511-515 (2019).
- Puth, M. T., Neuhäuser, M., Ruxton, G. D. On the variety of methods for calculating confidence intervals by bootstrapping. Journal of Animal Ecology. 84 (4), 892-897 (2015).
- Lavrentieva, A. Gradient hydrogels. Advances in Biochemical Engineering/Biotechnology. 178, 227-251 (2020).
- Zhu, D., Trinh, P., Li, J., Grant, G. A., Yang, F. Gradient hydrogels for screening stiffness effects on patient-derived glioblastoma xenograft cellfates in 3D. Journal of Biomedical Materials Research. Part A. 109 (6), 1027-1035 (2021).
- da Hora, C. C., Schweiger, M. W., Wurdinger, T., Tannous, B. A. Patient-derived glioma models: From patients to dish to animals. Cells. 8 (10), 1177 (2019).
- Li, W., et al. Characterization and transplantation of enteric neural crest cells from human induced pluripotent stem cells. Molecular Psychiatry. 23 (3), 499-508 (2018).
- Scaringi, C., Minniti, G., Caporello, P., Enrici, R. M. Integrin inhibitor cilengitide for the treatment of glioblastoma: A brief overview of current clinical results. Anticancer Research. 32 (10), 4213-4224 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten