Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Las matrices de hidrogel permiten un mayor rendimiento para los efectos de detección de los componentes de la matriz y la terapéutica en modelos tumorales 3D
En este artículo
Resumen
El presente protocolo describe una plataforma experimental para evaluar los efectos de las señales mecánicas y bioquímicas en las respuestas quimioterapéuticas de las células de glioblastoma derivadas de pacientes en cultivos miméticos de matriz 3D utilizando un dispositivo de iluminación UV hecho a medida que facilita la fotoreticulación de alto rendimiento de hidrogeles con características mecánicas sintonizables.
Resumen
Las interacciones célula-matriz median procesos fisiológicos complejos a través de señales bioquímicas, mecánicas y geométricas, influyendo en los cambios patológicos y las respuestas terapéuticas. Se espera que la contabilidad de los efectos de la matriz más temprano en la cartera de desarrollo de fármacos aumente la probabilidad de éxito clínico de las nuevas terapias. Existen estrategias basadas en biomateriales que recapitulan microambientes tisulares específicos en cultivos celulares 3D, pero la integración de estos con los métodos de cultivo 2D utilizados principalmente para la detección de fármacos ha sido un desafío. Por lo tanto, el protocolo presentado aquí detalla el desarrollo de métodos para el cultivo 3D dentro de matrices de biomateriales miniaturizados en un formato de placa de múltiples pocillos para facilitar la integración con las tuberías de detección de fármacos existentes y los ensayos convencionales para la viabilidad celular. Dado que se espera que las características de la matriz críticas para preservar fenotipos clínicamente relevantes en células cultivadas sean altamente específicas de tejidos y enfermedades, será necesario un cribado combinatorio de los parámetros de la matriz para identificar las condiciones apropiadas para aplicaciones específicas. Los métodos descritos aquí utilizan un formato de cultivo miniaturizado para evaluar las respuestas de las células cancerosas a la variación ortogonal de la mecánica de la matriz y la presentación del ligando. Específicamente, este estudio demuestra el uso de esta plataforma para investigar los efectos de los parámetros de la matriz en las respuestas de las células de glioblastoma derivado del paciente (GBM) a la quimioterapia.
Introducción
El costo esperado de desarrollar un nuevo medicamento ha aumentado constantemente en la última década, con más de $ 1 mil millones en estimaciones actuales1. Parte de este gasto es la alta tasa de fracaso de los medicamentos que ingresan a los ensayos clínicos. Aproximadamente el 12% de los candidatos a medicamentos finalmente obtienen la aprobación de la Administración de Alimentos y Medicamentos (FDA) de los Estados Unidos (EE. UU.) en 2019. Muchos medicamentos fallan en la Fase I debido a toxicidad imprevista2, mientras que otros que pasan los ensayos de seguridad pueden fallar debido a la falta de eficacia3. Este desgaste debido a la falta de eficacia puede explicarse en parte por el hecho de que los modelos de cáncer utilizados durante el desarrollo de fármacos son notoriamente no predictivos de la eficacia clínica4.
Las disparidades funcionales entre los modelos in vitro e in vivo pueden atribuirse a la eliminación de células cancerosas de su microambiente nativo, incluidas las células no tumorales y la ECM física 5,6. Comúnmente, los grupos de investigación utilizan matrices de cultivo disponibles comercialmente, como Matrigel (una matriz proteica de membrana basal derivada de sarcomas de ratón) para proporcionar a las células tumorales cultivadas un microambiente de matriz 3D. En comparación con el cultivo 2D, el cultivo 3D en matriz de membrana ha mejorado la relevancia clínica de los resultados in vitro 7,8. Sin embargo, los biomateriales de cultivo de tejidos descelularizados, incluida la matriz de membrana, generalmente exhiben variabilidad de lote a lote que puede comprometer la reproducibilidad9. Además, las matrices derivadas de tumores con orígenes tisulares diferentes a los estudiados pueden no proporcionar las señales fisiológicas adecuadas10. Finalmente, los cánceres con altos grados de heterogeneidad intratumoral tienen características microambientales que varían en una escala de tamaño submicrométrico y que la matriz de membrana no se puede ajustar para recapitular11.
El glioblastoma (GBM), un tumor cerebral uniformemente letal con una mediana de supervivencia de aproximadamente 15 meses, es un cáncer para el que el desarrollo del tratamiento ha sido particularmente difícil12,13. El estándar de atención actual para la GBM consiste en la resección primaria del tumor, seguida de radioterapia y luego quimioterapia con temozolomida (TMZ)14. Sin embargo, más de la mitad de los tumores clínicos de GBM exhiben resistencia al tratamiento a través de diversos mecanismos 15,16,17. Predecir la eficacia de un régimen de tratamiento para un paciente individual es extremadamente difícil. Los modelos preclínicos estándar utilizados para predecir los resultados individuales consisten en células tumorales derivadas del paciente xenoinjertadas ortotópicamente en ratones inmunocomprometidos. Si bien los xenoinjertos derivados del paciente pueden recapitular muchos aspectos de los tumores clínicos de GBM y son valiosos para los modelos preclínicos18, son intrínsecamente caros, de bajo rendimiento, requieren mucho tiempo e involucran preocupaciones éticas19. Los cultivos de células derivadas del paciente, en superficies plásticas 2D o como esferoides, evitan en su mayoría estos problemas. Si bien las células derivadas de pacientes conservan aberraciones genéticas, sus cultivos en 2D o como esferoides suspendidos han sido en gran medida representaciones pobres de xenoinjertos derivados de pacientes en roedores y tumores de pacientes originales20. Anteriormente, nosotros, y otros, hemos demostrado que las células GBM cultivadas en un ECM 3D que imita las propiedades mecánicas y bioquímicas del tejido cerebral pueden preservar los fenotipos de resistencia a los medicamentos 10,21,22,23.
Las interacciones entre el ácido hialurónico (HA), un polisacárido abundante en la ECM cerebral y sobreexpresado en los tumores GBM, y su receptor CD44 modulan la adquisición de resistencia a los fármacos in vitro 21,24,25,26,27. Por ejemplo, la inclusión de HA dentro de cultivos blandos en 3D aumentó la capacidad de las células GBM derivadas del paciente para adquirir resistencia terapéutica. Esta mecano-responsividad dependía de la unión de HA a los receptores CD44 en las células GBM21. Además, la unión de la integrina a péptidos portadores de RGD, incorporada a matrices de cultivo 3D, amplificó la quimiorresistencia mediada por CD44 de una manera dependiente de la rigidez21. Más allá de HA, la expresión de varias proteínas ECM, muchas de las cuales contienen regiones RGD, varían entre los tumores cerebrales normales y GBM28. Por ejemplo, un estudio informó que 28 proteínas ECM distintas estaban reguladas al alza en los tumores GBM29. Dentro de este complejo microambiente de matriz tumoral, las células cancerosas integran señales mecánicas y bioquímicas para producir un fenotipo de resistencia particular, que depende de diferencias relativamente pequeñas (por ejemplo, menos de un orden de magnitud) en el módulo de Young o la densidad de los péptidos de unión a la integrina 28,29,30.
El presente protocolo caracteriza cómo las células tumorales interpretan combinaciones únicas de señales de matriz e identifican microambientes de matriz complejos y específicos del paciente que promueven la resistencia al tratamiento (Figura 1A). Un método fotoquímico para generar matrices miniaturizadas y ajustadas con precisión para el cultivo 3D proporciona un gran espacio variable ortogonal. Se incorporó una matriz personalizada de LED, ejecutada por un microcontrolador, a los hidrogeles de enlace fotocrucedor dentro de un formato de placa de 384 pocillos para aumentar la automatización y la reproducibilidad. La intensidad de la exposición varió entre los pozos para alterar las propiedades micromecánicas de los hidrogeles resultantes, según se evaluó mediante microscopía de fuerza atómica (AFM). Si bien este manuscrito no se centra en la construcción de la matriz de iluminación en sí, se proporcionan un diagrama de circuito (Figura 1B) y una lista de piezas (Tabla de materiales) como ayudas para la reproducción del dispositivo.
Este informe demuestra la rápida generación de una serie de células GBM cultivadas en microambientes 3D únicos en los que el módulo de Young (cuatro niveles en un solo orden de magnitud) y el contenido de péptidos de unión a integrina (derivado de cuatro proteínas ECM diferentes) se variaron ortogonalmente. El enfoque se utilizó para investigar las contribuciones relativas de la mecánica del hidrogel y el compromiso de la integrina específica de ECM en la viabilidad y proliferación de las células GBM derivadas del paciente a medida que adquieren resistencia a la quimioterapia con temozolomida (TMZ).
Protocolo
Las líneas celulares GBM derivadas de pacientes (GS122 y GS304) fueron proporcionadas por el profesor David Nathanson (nuestro colaborador), quien desarrolló estas líneas bajo un protocolo aprobado por la Junta de Revisión Institucional de UCLA (IRB # 10-000655). Las células se proporcionaron desidentificadas para que las líneas celulares no pudieran vincularse de nuevo a los pacientes individuales.
1. Preparación de la solución de hidrogel
- Prepare la solución tamponada con HEPES disolviendo el polvo de HEPES a 20 mM en la solución salina equilibrada (HBSS) de Hank. Ajuste el pH a 7 después de la solvatación completa.
- En la solución tamponada con HEPES, disolver el HA tiolado (700 kDa de peso molecular nominal, ver Tabla de Materiales), preparado siguiendo el informe anterior31, de modo que el 6%-8% de los residuos de ácido carboxílico en cada ácido glucurónico se modifiquen con un tiol, a una concentración de 10 mg/ml en solución tampón.
NOTA: Se recomienda un vial de ámbar para prevenir la oxidación de tiol por la luz ambiental.- Revuelva con una placa de agitación magnética (<1.000 rpm) a temperatura ambiente hasta que se disuelva por completo, generalmente alrededor de 45 min.
- Mientras el HA se está disolviendo, prepare soluciones separadas de (1) 100 mg/mL de 8 brazos-PEG-Norborneno (20 kDa), (2) 100 mg/mL de 4 brazos-PEG-Tiol (20 kDa), (3) 4 mM de cisteína o péptido que contiene cisteína (por ejemplo, GCGYGRGDSPG) y (4) 4 mg/ml de LAP en tubos de microcentrífuga (ver Tabla de Materiales).
- Prepare cada una de estas cuatro soluciones en la solución con búfer HEPES preparada en el paso 1.1. Vórtice las soluciones para asegurar la disolución completa de cada reactivo antes de realizar el paso 4.
NOTA: Si se prueban múltiples péptidos diferentes, cada uno debe contener una cisteína u otra fuente de fracción de tiol para esta química de conjugación. - Preparar soluciones (4 mM de tiol disponibles) de todos los péptidos para ser atados dentro de un solo hidrogel en este punto.
NOTA: Las secuencias peptídicas y las proteínas ECM de las que se derivaron y utilizaron en este estudio se enumeran en la Tabla 1. La N-acetil cisteína (ver Tabla de Materiales), a la que las células no se unen, puede ser sustituida por un péptido bioactivo que contiene tiol para valorar la concentración de un péptido adhesivo o actuar como un control negativo31.
- Prepare cada una de estas cuatro soluciones en la solución con búfer HEPES preparada en el paso 1.1. Vórtice las soluciones para asegurar la disolución completa de cada reactivo antes de realizar el paso 4.
- Mezclar las soluciones individuales de HA, PEG-Norborneno, PEG-tiol y péptidos que contienen cisteína/tiol (ver Tabla de Materiales) para alcanzar las concentraciones finales para las matrices finales de hidrogel enumeradas en la Tabla 2. Revuelva (<1.000 rpm) en una placa de agitación magnética durante al menos 30 minutos para mezclar completamente.
NOTA: Las soluciones de ALTA disponibilidad son altamente viscosas y se manejan mejor utilizando una pipeta de desplazamiento positivo (consulte la Tabla de materiales). Si no se dispone de una pipeta de desplazamiento positivo, las soluciones viscosas también se pueden prescindir de una micropipeta estándar mediante el pipeteo lento con puntas de orificio ancho.
2. Iluminación y fotocrosslinking de hidrogeles a través de una matriz de LED
PRECAUCIÓN: Use gafas protectoras UV y cubra el campo de iluminación con material absorbente de UV.
NOTA: La matriz de LED descrita en este protocolo consta de seis conjuntos de ocho LED colocados en serie, como se ilustra en el diagrama de circuito proporcionado (Figura 1A). Cada conjunto de LED se puede alimentar de forma independiente, lo que permite hasta seis irradiancias diferentes por ejecución. El archivo complementario 1 contiene capturas de pantalla correspondientes a las siguientes instrucciones para obtener más orientación.
- Descargue el archivo .zip dispositivo de iluminación de los archivos de codificación suplementarios. Este directorio contiene los siguientes archivos: Arduino.zip (Supplementary Coding File 1), Drivers.zip (Supplementary Coding File 2), GUI.zip (Supplementary Coding File 3) y Holder.zip (Supplementary Coding File 4).
NOTA: Imprima en 3D las partes superior e inferior para mantener la placa de circuito en su lugar (consulte Archivos de codificación suplementarios para obtener más detalles). - Descargue e instale el software del microcontrolador (consulte la Tabla de materiales).
- Descargue e instale el software GUI (consulte la Tabla de materiales). Consulte el Archivo complementario 1 para obtener instrucciones de funcionamiento del software.
- Abra Procesamiento e instale la biblioteca controlIP5 haciendo clic en Sketch > Importar biblioteca > Agregar biblioteca. Luego, busque controlIP5 en bibliotecas y haga clic en Instalar. Realice esto por primera vez.
- Alimente el dispositivo de iluminación (consulte la Tabla de materiales) con la fuente de alimentación de 36 voltios y conéctelo a un PC mediante un cable micro-USB.
NOTA: Algunos dispositivos no instalarán controladores automáticamente para varias placas Arduino nano. Se proporciona un conjunto de controladores en el archivo zip del dispositivo. - Abra el archivo Arduino.ino, ubicado en la carpeta Adruino.zip, utilizando Arduino IDE.
- Compile el archivo Arduino.ino haciendo clic en el botón Marca de verificación . Cargue el código compilado haciendo clic en el botón Flecha .
- Abra el archivo GUI.pde, ubicado en la carpeta GUI.zip, mediante Processing.
- Haga clic en Ejecutar en el programa de procesamiento para iniciar la interfaz gráfica de usuario para controlar el dispositivo de iluminación.
- En la ventana de la interfaz gráfica de usuario, haga clic en Intensidad para que la columna que contiene la solución precursora de hidrogel se reticule e ingrese la intensidad deseada. Haga clic en el cuadro Hora e ingrese la hora deseada. Para la solución proporcionada en la Tabla 2, esto será de 15 s.
NOTA: Los usuarios finales deben calibrar los valores de intensidad digital a la irradiancia utilizando un radiómetro. En la Figura 2A se proporcionan ejemplos de intensidades típicas. - Alinee las muestras con el dispositivo de iluminación (Figura 2B) con cualquier otro LED en una sola columna de los moldes de silicona (consulte la Tabla de materiales) o en una placa de 384 pocillos. Haga clic en Finalizar para comenzar la iluminación. Repita este proceso según sea necesario para la iluminación de múltiples diapositivas u otros pozos de una placa de 384 pocillos.
NOTA: El soporte está diseñado de tal manera que la placa de 384 pocillos se encuentra al ras con una esquina de la cámara interior durante la iluminación.- Después de la iluminación, cuando se coloque en una esquina, mueva la placa del pozo a la siguiente esquina y repita. Para iluminar los pozos en la otra mitad de la placa, levante la placa del soporte y gire 180 °.
- Genere hidrogeles con mecánica variable para la caracterización mecánica siguiendo los pasos a continuación.
- Limpie los portaobjetos de vidrio y los moldes de silicona con cinta adhesiva para eliminar los escombros. Adhiera los moldes de silicona al portaobjetos de vidrio, presione hacia abajo para garantizar un buen sellado y desplace cualquier burbuja de aire.
- Pipetear 80 μL de solución precursora de hidrogel, según lo preparado en el paso 1.4, en cada molde de silicona en el portaobjetos de vidrio.
- Coloque la corredera de vidrio sobre el dispositivo de iluminación alineado con cualquier otro LED en una sola columna. Exponga los precursores de hidrogel a la luz UV durante 15 s, como se describe en el paso 2, al fotoenlace.
- Una vez que la iluminación se haya detenido, recupere las diapositivas y afloje los geles de los moldes trazando la circunferencia interna del molde con una punta fina (punta de pipeta de 10 μL, aguja de 30 G, etc.). Retire los moldes de silicona con pinzas/pinzas.
- Mueva los hidrogeles reticulados dentro de los pozos individuales de una placa de 12 pocillos humedeciendo una espátula y empujándolos suavemente fuera del portaobjetos de vidrio. Llene cada pozo con 2 ml de DPBS (ver Tabla de materiales) antes de agregar el hidrogel. Hinche los geles en la solución de DPBS durante al menos 12 h (generalmente durante la noche) a temperatura ambiente (para la caracterización mecánica del día siguiente).
3. Mediciones de microscopía de fuerza atómica (AFM)
- Encienda el microscopio de fuerza atómica (AFM) de acuerdo con las instrucciones del fabricante (consulte la Tabla de materiales). Este protocolo proporciona instrucciones breves para usar el instrumento y el software relacionado.
- Instale la sonda AFM (consulte la Tabla de materiales).
NOTA: Para el presente estudio, se modificó un voladizo triangular de nitruro de silicio con una constante nominal de resorte de 0,01 N/m con una partícula esférica de dióxido de silicio de 2,5 μm. - Después de la instalación, alinee el láser con el vértice de la sonda triangular y luego ajuste la deflexión del espejo y del láser para maximizar la suma de la señal (generalmente entre 1.5-2.2 voltios).
- Sumerja la sonda en DPBS y espere hasta 15 minutos para obtener el equilibrio térmico. Haga clic en el botón Calibración y seleccione Calibración dependiente del contacto . Haga clic en el botón Recopilar ajuste térmico y, después de la recopilación de datos, seleccione el pico alrededor de 3 kHz para la calibración.
NOTA: Puede ser necesario un ligero ajuste del espejo y los deflectores láser después de la inmersión en un líquido debido a cambios en el índice de refracción. - Acérquese a la superficie de una placa de Petri (plástico) estableciendo los parámetros de aproximación en velocidad constante, una altura objetivo de 7,5 μm y una velocidad de aproximación de 15 μm / s. Habilite la medición de línea base por ejecución para el enfoque para que el enfoque se ejecute continuamente y no se detenga antes de tiempo debido a la deriva en el deflector.
- Al acercarse, establezca parámetros de adquisición para el mapeo de fuerza a 4 nN turnarounds, 2 μm de distancia de indentación, 1 μm/ s de velocidad y 0 s de tiempo de contacto. Presione el botón Inicio para comenzar a recoger una curva de fuerza en la superficie de plástico (por ejemplo, una placa de pozo).
- Vuelva a la ventana de calibración y seleccione la parte de la curva de fuerza correspondiente al contacto y la sangría del plástico. Acepte los valores calculados de sensibilidad y rigidez para que la sonda complete la calibración.
- Después de la calibración, levante la sonda AFM y coloque la muestra de hidrogel para interrogarla. Acérquese al hidrogel siguiendo los ajustes proporcionados en el paso 5.
NOTA: Durante el procedimiento de aproximación hacia la superficie del hidrogel, la unidad puede desencadenar por error el estado aproximado. Para verificar el enfoque real, obtenga una curva de fuerza como en el paso 4.6. Repita el procedimiento de aproximación si la curva resultante no muestra el contacto y la sangría resultante. - Cuando el enfoque de superficie sea exitoso, cambie al modo de mapeo de fuerza y establezca los parámetros de adquisición en un mapa de tamaño 8 x 8 con una longitud de 40 μm por eje. Obtenga mapas de fuerza en varias regiones para evaluar la uniformidad de las mediciones de rigidez.
- Interpretar curvas de fuerza utilizando el programa de software JPK SPM Data Processing a través de un ajuste de modelo Hertz/Sneddon (Ecuaciones 1 y 2, ver Tabla 3 para la definición de todas las variables) con la geometría esférica seleccionada 32,33,3 4.
 Ecuación 132
Ecuación 132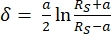 Ecuación 232
Ecuación 232
- Interpretar curvas de fuerza utilizando el programa de software JPK SPM Data Processing a través de un ajuste de modelo Hertz/Sneddon (Ecuaciones 1 y 2, ver Tabla 3 para la definición de todas las variables) con la geometría esférica seleccionada 32,33,3 4.
4. Configuración y tratamiento farmacológico de cultivos 3D, incrustados en matrices
- Prepare las células deseadas como una solución de una sola célula.
NOTA: Diferentes tipos de células pueden requerir diferentes métodos de pasaje. En la referencia31 se informa de un protocolo típico para pasar un cultivo en suspensión de esferoides GBM de un matraz T-75. - Recolecte esferoides GBM (aproximadamente 150 μm de diámetro) de un cultivo de suspensión de matraz T-75 en un tubo cónico de 15 ml. Enjuague el matraz de cultivo con 5 ml de DPBS para eliminar las células y medios residuales y agregue este volumen al tubo cónico.
- Centrifugar el tubo cónico que contiene células a 200 x g durante 5 min a temperatura ambiente. Después de la centrifugación, retire el sobrenadante con una pipeta serológica de 5 ml, teniendo cuidado de no perturbar el gránulo celular, y resuspenda en 5 ml de DPBS.
- Centrifugar a 200 x g durante 5 min a temperatura ambiente para lavar las celdas. Aspire el sobrenadante con una pipeta serológica de 5 ml, teniendo cuidado de no perturbar el gránulo celular, y luego resuspenda las células en 2 ml de reactivo de disociación celular (ver Tabla de materiales).
- Incubar a temperatura ambiente durante 10-15 min. Agregue 3 ml de medio completo (ver Tabla de materiales) y pipetee suavemente 3-5 veces para descomponer los esferoides a una suspensión de una sola celda31.
- Centrifugar la suspensión unicelular a 400 x g (las suspensiones monocelulares pueden girarse más rápido para la formación de pellets) durante 5 min para gletizar células a temperatura ambiente. Aspire el sobrenadante con una pipeta serológica de 5 ml, teniendo cuidado de no molestar el pellet celular. Resuspend células en 1 mL de medio completo.
NOTA: Si las células permanecen en grupos, en lugar de como células individuales en suspensión, después del pasaje, las células se pueden pasar a través de un colador de células de 40 μm para lograr una suspensión de una sola célula. - Extraiga una parte de las células para contar usando un hemocitómetro. Diluya esta porción dos veces con azul de tripano, que impregna las células con viabilidad comprometida. Cuente solo las células vivas e incoloras. Por lo general, un T-75 sembrado a 800,000 células por matraz produce 2-3 millones de células después de una semana en cultivo.
- Determine el número de celdas necesarias para la encapsulación. Transfiera un volumen de medio que contenga el número total de células necesarias en un tubo de microcentrífuga estéril de 1,7 ml. Girar hacia abajo a 400 x g durante 5 min a temperatura ambiente.
NOTA: Por ejemplo, se necesita un mínimo de 2,5 millones de células resuspendidas en 1 ml de volumen de gel para encapsular células a 2,5 millones de células/ml. Un volumen de gel de 1 ml permite a los usuarios dispensar 100 gotas de gel, donde cada gota de gel es de 10 μL de volumen. Se recomienda preparar un volumen adicional de ~ 20% de células suspendidas en solución de hidrogel para tener en cuenta la pérdida durante la transferencia de pipetas. Por lo tanto, se prepararían 3 millones de células y 1,2 ml de solución precursora de hidrogel en este ejemplo. Se recomienda una densidad mínima de 500 mil células/ml. - Aspire el sobrenadante con una micropipeta, teniendo cuidado de no molestar la bolita celular. Resuspend el pellet celular en la solución precursora de hidrogel, tal como se preparó en el paso 1.4, mezclando bien mediante pipeteo hacia arriba y hacia abajo con una micropipeta de 1.000 μL 4-5 veces.
- Cargue las celdas en un pipeteador repetido (consulte la Tabla de materiales) configurado para dispensar 10 μL. Para evitar burbujas y dispensación desigual, prepare el pipeteador repetido dispensando 1-2 veces más en un contenedor de residuos.
- En cada pocillo de una placa de 384 pocillos, dispense 10 μL de células suspendidas en solución de hidrogel del pipeteador repetido. Usando la matriz de LED, ilumine cada pozo que contenga celdas (paso 2) durante 15 s con intensidades (el ejemplo da como resultado que la Figura 2A utilice intensidades de 1.14, 1.55, 2.15, 2.74 mW / cm2) para lograr las propiedades mecánicas deseadas.
NOTA: Se sugiere comenzar con cinco réplicas por condición experimental y escalar hacia arriba o hacia abajo dependiendo del rendimiento deseado y la varianza del ensayo de punto final. - Añadir 40 μL de medio completo a cada pocillo que contenga las células. Agregue 50 μL de DPBS a los pozos secos no experimentales que rodean los geles para minimizar las pérdidas debidas a la evaporación.
- Para las células GBM, agregue 40 μL del fármaco que contiene el medio (por ejemplo, TMZ, consulte la Tabla de materiales) para lograr la concentración final deseada (10 μM-100 μM en dimetilsulfóxido (DMSO) o vehículo (DMSO), en consecuencia, a partir de 3 días después de la encapsulación.
5. Ensayo de proliferación de CCK8
- Añadir 10 μL de reactivo CCK8 (ver Tabla de Materiales) a cada pocillo que contenga las celdas.
NOTA: Si realiza este ensayo por primera vez, incluya pozos de control negativos, como solo medios o hidrogel libre de células en los medios. - Incubar durante 1-4 h de acuerdo con las instrucciones del fabricante.
NOTA: Este tiempo puede variar en función del tipo de célula y la densidad, por lo que los tiempos de incubación deben probarse para cada aplicación para que los valores de absorbancia caigan dentro de un rango lineal, un requisito para aplicar la Ley35 de Beer. - Lectura de absorbancias a 450 nm para todos los pocillos después de la incubación.
- Calcule la absorbancia media a 450 nm obtenida en el paso 3 para el estado del vehículo para cada grupo. Divida bien cada medicamento tratado por el promedio del vehículo de control por grupo.
- Calcule los intervalos de confianza generando distribuciones de arranque (N = 10.000) a través del método del percentil36.
NOTA: En general, se pueden utilizar intervalos de confianza del 95% e interpretar que las condiciones cuyos intervalos de confianza no superan 1 son significativas y justifican una mayor investigación. Establecer intervalos de confianza al 95% es congruente con establecer un punto de corte de significación de p = 0,05. Para los datos mostrados en los resultados, es útil distinguir las condiciones que promueven o inhiben la resistencia a los medicamentos mediada por la matriz, lo que requiere un análisis de dos lados.
Resultados
Las mediciones de AFM confirmaron el control preciso de la mecánica del hidrogel en función de la irradiancia UV (mW/cm2) durante la foto-reticulación utilizando una matriz led personalizada controlada por Arduino (Figura 2A). La formulación de hidrogel utilizada en este protocolo se puede encontrar en la Tabla 2. El espaciado de los LED en la plantilla proporcionada coincide con el espaciado para cada otro pozo de una placa de 384 pocillos, lo que permite la ...
Discusión
El trabajo actual presenta métodos para generar cultivos miniaturizados en 3D dentro de ha basados en HA, al tiempo que altera la rigidez de la matriz y los péptidos disponibles para el compromiso con la integrina. Esta técnica permite el estudio sistemático de cómo los parámetros de la matriz afectan a los fenotipos celulares (por ejemplo, la viabilidad de las células cancerosas expuestas a la quimioterapia) con un mayor rendimiento. Los enfoques anteriores, incluido el presentado en este documento, han ajustado ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
A los autores les gustaría agradecer específicamente a Carolyn Kim, Amelia Lao, Ryan Stoutamore e Itay Solomon por sus contribuciones a iteraciones anteriores del esquema de fotogelación. Las líneas celulares GS122 y GS304 fueron generosamente proporcionadas por David Nathanson. Todas las figuras fueron creadas con BioRender.com. Las instalaciones centrales de UCLA, los Recursos Compartidos de Detección Molecular y el Laboratorio de Caracterización Nano y Pico fueron fundamentales para el trabajo. Chen Chia-Chun fue apoyado por el UCLA Eli and Edythe Broad Center of Regenerative Medicine and Stem Cell Research Training Program. Grigor Varuzhanyan fue apoyado por una subvención del Programa de Capacitación en Biología celular tumoral de los NIH (T32 CA 009056).
Materiales
| Name | Company | Catalog Number | Comments |
| 1.1 kOhm resistors, 6 W | Digikey | 35601k1ft | |
| 1.7 mL microcentrifuge tube | Genesse Scientific | 21-108 | |
| 15 mL conical tube | Fisher Scientific | 14-959-70C | |
| 365 nm LED | Digikey | ltpl-c034uvh365 | |
| 384 well plate | Bio Greiner One | 781090 | |
| 40 µm cell strainer | MTC bio | C4040 | |
| 4-Armed thiol terminated polyethlene glycol (20 kDa) | Laysan Bio | 4arm-PEG-SH-20K-1g | |
| 6 NPN BJTs | Digikey | 2n5550ta | |
| 80 Ohm resistors, 0.125 W | Digikey | erjj-6enf80r6v | |
| 8-Armed norbornene terminated polyethylene glycol (20 kDa) | Jenkem Technology | A7025-1 | |
| Accutase | Innovative Cell Technologies | AT104500 | cell dissociation reagent |
| AFM Probes | Novascan | 0.01 N/m Nominal spring constant, 2.5 µm SiO2 particle | |
| Arduino IDE | Arduino | 1.8.19 | |
| Arduino Nano | Makerfire | Mini Nano V3.0 ATmega328P Microcontroller Board | |
| bFGF | Peprotech | 100-18B | 20 ng/mL |
| CCK8 | Abcam | ab228554 | |
| Centrifuge | Thermoscientific | sorvall legend xtr | |
| CP100ST | Gilson | F148415 | Pipette tips for positive displacement pipette |
| Cubis Semi-Micro Balance | Sartorius | MSA225S100DI | |
| DMEM - F12 (50-50) | Life Technologies | 11330057 | 1x |
| DMSO | Fisher Scientific | BP231-100 | |
| DPBS Ca (-) Mg (-) | Genesse Scientific | 25-508 | |
| EGF | Peprotech | AF100-15 | 50 ng/mL |
| Ethanol, Anhydrous | Fisher Scientific | A405P | Add DI water to dilute to 70% |
| Fisherbrand Class B Amber Glass threaded vials | Fisher Scientific | 03-339-23C | |
| Fisherbrand Weighing Paper | Fisher Scientific | 09-898-12B | |
| G21 Supplement | Gemini Bio | 400-160 | 50x |
| Hanks Balanced Salt Solution | Thermo Fisher Scientific | 14175095 | |
| HCl, ACS, 12M | Sigma Aldrich | S25838A | Add DI water to dilute to 1 M |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H3149-100Ku | 25 µg/mL |
| HEPES | Sigma Aldrich | H7006-100G | |
| Hot Air Gun | Wagner | HT1000 | |
| Integrin-binding sialoprotein (IBSP) peptide | Genscript | Custom Order | GCGYGGGGNGEPRGDTYRAY |
| Lithium phenyl-2,4,6 trimethylbenzoylphosphinate (LAP) , >95% | Sigma Aldrich | 900889-1G | |
| Magnetic stir plate | Thermo Scientific | SP194715 | |
| Microcentrifuge | Thermo Scientific | Sorvall legend micro 21R | |
| Microman E single Channel Pipettor | Gilson | FD10004 | Positive displacement pipette |
| Micropipette Tips | Various Manufacturs | Various sizes | |
| mLine micropipette | Sartorious | ||
| N-acetyl Cysteine | Sigma Aldrich | A7250-10G | |
| Nanowizard 4 | Bruker | AFM microscope | |
| NaOH | Fisher Scientific | ss255-1 | Add DI water to dilute to 1 M |
| Normoicin | Invivogen | ant-nr-1 | 500x |
| Osteopontin Peptide | Genscript | Custom Order | GCGYGTVDVPDGRGDSLAYG |
| Pipet Aid | Drummond | 4000102 | |
| Plain Microscope Slides | Globe Scientific | 1301 | |
| Press-To-Seal silicone Isolator, 12-4.5mm diam x 2mm deep | Grace Bio Labs | 664201-A | Cut so that 8 individual molds are made from a single sheet |
| Processing | Processing | 3.5.4 | |
| Repeater M4 | Eppendorf | 4982000322 | |
| Repeater Pipette Tips | Sartorious | 30089430 | 1 mL sizes |
| RGD Peptide | Genscript | GCGYGRGDSPG | |
| Scoth Tape | |||
| Serological Pipettes | Genesse Scientific | 12-102,12-104 | 5,10 mL Pipettes |
| Solder Paste | Digikey | 315-NC191LT15T5-ND | |
| Solder Wire | |||
| Straight dissecting forceps | VWR Scientific | 82027-408 | |
| Synergy H1 Plate Reader | Biotek | ||
| T-75 Cell Culture Treated Flask | Genesee Scientific | 25-209 | |
| Temozolomide | Sigma Aldrich | T2577 | Typically used from 10 µM to 100 µM |
| Tenascin-C Peptide | Genscript | GCGYGRSTDLPGLKAATHYTITIR GV | |
| Thiolated Hyaluronic Acid (700 kDa), 6-8% modified | Lifecore Biomedical | HA700K5 | |
| VWR Spinbar, Flea Micro | VWR | 58948-375 |
Referencias
- Scannell, J. W., Blanckley, A., Boldon, H., Warrington, B. Diagnosing the decline in pharmaceutical R&D efficiency. Nature Reviews Drug Discovery. 11 (3), 191-200 (2012).
- Waring, M. J., et al. An analysis of the attrition of drug candidates from four major pharmaceutical companies. Nature Reviews Drug Discovery. 14 (7), 475-486 (2015).
- Khozin, S., Liu, K., Jarow, J. P., Pazdur, R. Why do oncology drugs fail to gain US regulatory approval. Nature Reviews Drug Discovery. 14 (7), 450-451 (2015).
- Booth, B., Ma, P., Glassman, R. Oncology's trials. Market indicators. Nature Reviews Drug Discovery. 2 (8), 609-610 (2003).
- Da Ros, M., et al. Glioblastoma chemoresistance: The double play by microenvironment and blood-brain barrier. International Journal of Molecular Sciences. 19 (10), 2879 (2018).
- Broekman, M. L., et al. Multidimensional communication in the microenvirons of glioblastoma. Nature Reviews Neurology. 14 (8), 482-495 (2018).
- Grundy, T. J., et al. Differential response of patient-derived primary glioblastoma cells to environmental stiffness. Scientific Reports. 6 (1), 1-10 (2016).
- Gomez-Roman, N., Stevenson, K., Gilmour, L., Hamilton, G., Chalmers, A. J. A novel 3D human glioblastoma cell culture system for modeling drug and radiation responses. Neuro-Oncology. 19 (2), 229-241 (2017).
- Simoni, R. D., et al. Basement membrane complexes with biological activity. Biochemistry. 25 (2), 312-318 (2002).
- Xiao, W., et al. Brain-mimetic 3D culture platforms allow investigation of cooperative effects of extracellular matrix features on therapeutic resistance in glioblastoma. Cancer Research. 78 (5), 1358-1370 (2018).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nature Reviews Materials. 5 (7), 539-551 (2020).
- Spinelli, C., et al. Molecular subtypes and differentiation programmes of glioma stem cells as determinants of extracellular vesicle profiles and endothelial cell-stimulating activities. Journal of Extracellular Vesicles. 7 (1), 1490144 (2018).
- Ostrom, Q. T., Cioffi, G., Waite, K., Kruchko, C., Barnholtz-Sloan, J. S. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2014-2018. Neuro-Oncology. 23, (2021).
- Stupp, R., et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma. New England Journal of Medicine. 352 (10), 987-996 (2005).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Tomczak, K., Czerwińska, P., Wiznerowicz, M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge. Contemporary oncology. 19, 68-77 (2015).
- Lee, S. Y. Temozolomide resistance in glioblastoma multiforme. Genes and Diseases. 3 (3), 198-210 (2016).
- Joo, K. M., et al. Patient-specific orthotopic glioblastoma xenograft models recapitulate the histopathology and biology of human glioblastomas in situ. Cell Reports. 3 (1), 260-273 (2013).
- Levy, N. The use of animal as models: Ethical considerations. International Journal of Stroke. 7 (5), 440-442 (2012).
- Phon, B. W. S., Kamarudin, M. N. A., Bhuvanendran, S., Radhakrishnan, A. K. Transitioning preclinical glioblastoma models to clinical settings with biomarkers identified in 3D cell-based models: A systematic scoping review. Biomedicine & Pharmacotherapy. 145, 112396 (2022).
- Xiao, W., et al. Bioengineered scaffolds for 3D culture demonstrate extracellular matrix-mediated mechanisms of chemotherapy resistance in glioblastoma. Matrix Biology. 85-86, 128-146 (2020).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Xu, X., Farach-Carson, M. C., Jia, X. Three-dimensional in vitro tumor models for cancer research and drug evaluation. Biotechnology Advances. 32 (7), 1256-1268 (2014).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived Glioblastoma Cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Preston, M. Digestion products of the PH20 hyaluronidase inhibit remyelination. Annals of Neurology. 73 (2), 266-280 (2013).
- Kim, Y., Kumar, S. CD44-mediated adhesion to hyaluronic acid contributes to mechanosensing and invasive motility. Molecular Cancer Research. 12 (10), 1416-1429 (2014).
- Pibuel, M. A., Poodts, D., Díaz, M., Hajos, S. E., Lompardía, S. L. The scrambled story between hyaluronan and glioblastoma. The Journal of Biological Chemistry. 296, 100549 (2021).
- Xiao, W., Sohrabi, A., Seidlits, S. K. Integrating the glioblastoma microenvironment into engineered experimental models. Future Science OA. 3 (3), (2017).
- Trombetta-Lima, M., et al. Extracellular matrix proteome remodeling in human glioblastoma and medulloblastoma. Journal of Proteome Research. 20 (10), 4693-4707 (2021).
- Schregel, K., et al. Characterization of glioblastoma in an orthotopic mouse model with magnetic resonance elastography. NMR in Biomedicine. 31 (10), 3840 (2018).
- Xiao, W., Ehsanipour, A., Sohrabi, A., Seidlits, S. K. Hyaluronic-acid based hydrogels for 3-dimensional culture of patient-derived glioblastoma cells. Journal of Visualized Experiments: JoVE. (138), e58176 (2018).
- Guz, N., Dokukin, M., Kalaparthi, V., Sokolov, I. If cell mechanics can be described by elastic modulus: Study of different models and probes used in indentation experiments. Biophysical Journal. 107 (3), 564-575 (2014).
- Sneddon, I. N. The relation between load and penetration in the axisymmetric boussinesq problem for a punch of arbitrary profile. International Journal of Engineering Science. 3 (1), 47-57 (1965).
- Soofi, S. S., Last, J. A., Liliensiek, S. J., Nealey, P. F., Murphy, C. J. The elastic modulus of MatrigelTM as determined by atomic force microscopy. Journal of Structural Biology. 167 (3), 216-219 (2009).
- Mayerhöfer, T. G., Popp, J. Beer's law - Why absorbance depends (almost) linearly on concentration. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 20 (4), 511-515 (2019).
- Puth, M. T., Neuhäuser, M., Ruxton, G. D. On the variety of methods for calculating confidence intervals by bootstrapping. Journal of Animal Ecology. 84 (4), 892-897 (2015).
- Lavrentieva, A. Gradient hydrogels. Advances in Biochemical Engineering/Biotechnology. 178, 227-251 (2020).
- Zhu, D., Trinh, P., Li, J., Grant, G. A., Yang, F. Gradient hydrogels for screening stiffness effects on patient-derived glioblastoma xenograft cellfates in 3D. Journal of Biomedical Materials Research. Part A. 109 (6), 1027-1035 (2021).
- da Hora, C. C., Schweiger, M. W., Wurdinger, T., Tannous, B. A. Patient-derived glioma models: From patients to dish to animals. Cells. 8 (10), 1177 (2019).
- Li, W., et al. Characterization and transplantation of enteric neural crest cells from human induced pluripotent stem cells. Molecular Psychiatry. 23 (3), 499-508 (2018).
- Scaringi, C., Minniti, G., Caporello, P., Enrici, R. M. Integrin inhibitor cilengitide for the treatment of glioblastoma: A brief overview of current clinical results. Anticancer Research. 32 (10), 4213-4224 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados