需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
大豆共生结节多聚体纯化
摘要
该协议描述了从完整的大豆结节中纯化真核多体的方法。测序后,基因表达分析的标准管道可用于鉴定转录组和翻译组水平的差异表达基因。
摘要
该协议的目的是为研究大豆(甘氨酸max)共生结节的真核翻译组提供策略。本文描述了优化的方法,以分离植物来源的多核糖体及其相关的mRNA,以使用RNA测序进行分析。首先,通过在保存多小体和RNA的条件下从整个冷冻大豆结节中匀浆获得细胞质裂解物。然后,通过低速离心清除裂解物,并使用15%的上清液进行总RNA(TOTAL)分离。剩余的澄清裂解物用于通过两层蔗糖垫(12%和33.5%)超速离心分离多聚体。重悬后从多粒体沉淀中纯化多聚体相关mRNA(PAR)。TOTAL和PAR均通过高灵敏度毛细管电泳进行评估,以满足RNA-seq测序文库的质量标准。作为下游应用的一个例子,测序后,基因表达分析的标准管道可用于获得转录组和翻译组水平的差异表达基因。总之,该方法与RNA-seq相结合,可以研究复杂组织(例如共生结节)中真核mRNA的翻译调节。
引言
豆科植物,如大豆(Glycine max),可以与称为根瘤菌的特定土壤细菌建立共生关系。这种互惠关系导致植物根部形成新的器官,即共生结节。结节是寄存细菌的植物器官,由宿主细胞组成,其细胞质定植于一种称为拟杆菌的特殊形式的根瘤菌。这些拟杆菌催化大气氮(N 2)还原成氨,氨被转移到植物中以换取碳水化合物1,2。
尽管这种固氮共生是研究最充分的植物-微生物共生之一,但许多方面仍有待更好地理解,例如受到不同非生物胁迫条件的植物如何调节它们与共生伙伴的相互作用以及这如何影响结节代谢。通过分析结节翻译组(即主动翻译的信使RNA[mRNA]的子集),可以更好地理解这些过程。多核糖体或多核糖体是与mRNA相关的多个核糖体的复合物,通常用于研究翻译3。多聚体分析方法包括分析与多聚体相关的mRNA,并已成功用于研究控制不同生物过程中发生的基因表达的转录后机制4,5。
从历史上看,基因组表达分析主要集中在确定mRNA丰度6,7,8,9。然而,由于基因表达转录后调控的不同阶段,特别是翻译10,11,12,转录本和蛋白质水平之间缺乏相关性。此外,在转录组水平上的变化与在翻译组水平上发生的变化之间没有观察到依赖性13。对正在翻译的mRNA集的直接分析可以比仅分析mRNA水平时获得的细胞基因表达(其终点是蛋白质丰度)更准确和完整的测量14,15,16。
该协议描述了如何通过两层蔗糖垫的差异离心从完整的大豆结节中纯化植物来源的多聚体(图1)。然而,由于拟杆菌衍生的核糖体也存在于结节中,因此核糖体和RNA物种的混合物被纯化,即使真核糖体占主要部分(90%-95%)。还描述了随后的RNA分离、定量和质量控制(图1)。该协议与RNA-seq相结合,应提供有关复杂组织(例如共生结节)中真核mRNA的翻译调节的实验结果。
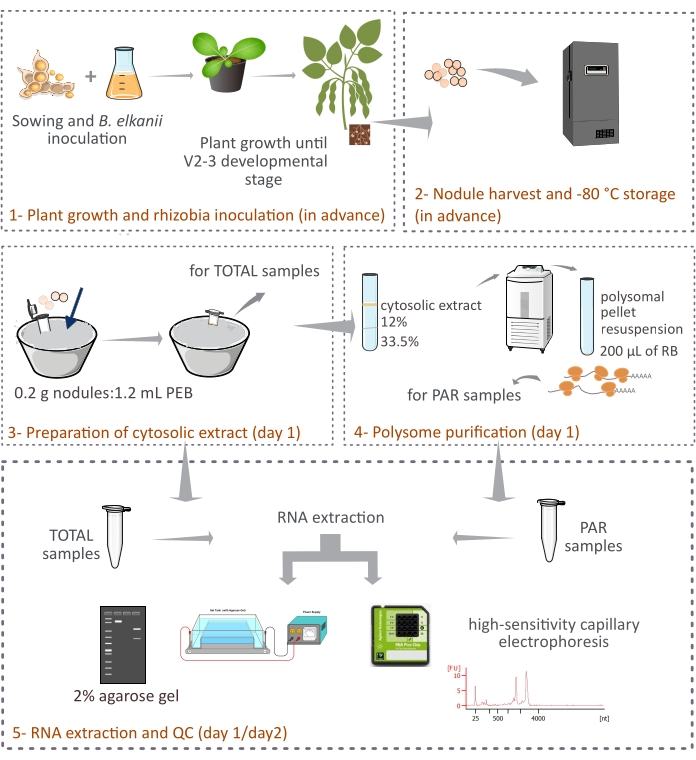
图1:从共生结节纯化真核多体的拟议方法的示意图概述。 该方案概述了方案中遵循的步骤,从(1)植物生长和(2)结节收获到(3)制备胞质提取物,(3)获得总样品和(4)PAR样品,以及(5)RNA提取和质量控制。缩写:PEB = 多聚体提取缓冲液;RB = 重悬缓冲液;总 = 总核糖核酸;PAR = 多聚体相关的 mRNA。 请点击此处查看此图的大图。
研究方案
1. 植物生长和根瘤菌接种
- 为了产生所需的结节,请在受控条件下将所选的大豆种子播种在生长室的选定基质中。
注意:在该协议中,种子播种在装有沙子:蛭石(1:1)混合物的0.5升塑料瓶中。生长室的昼/夜循环温度分别为28 °C / 20 °C,光/暗光周期分别为16 h/8 h。光合有效辐射强度为620 μmol·m−2·s−1. - 提前准备液体酵母提取物甘露醇(YEM)培养基(见 材料表)。高压灭菌介质。
- 在播种的同一天,用缓 根瘤菌 U1302 菌株接种一个含有 100 mL YEM 的烧瓶。将烧瓶在28°C的轨道振荡器上以100rpm孵育。
- 让根瘤菌生长 2 天,并用 2 mL 培养物接种幼苗。
2. 缺水处理(可选)
注意:该协议概述了大豆植物的缺水处理。这部分可以根据手头的实验问题完全更改或省略。
- 将大豆幼苗种植19天(V2-3发育阶段),不受水分限制。用补充有0.5 mM KNO3的B&D-培养基17保持基板的现场容量。
- 第20天,停止浇水。
注意:在这里,在生长和缺水期间,每天通过重量法(水重量含量)测量水分含量。 - 按照制造商的指示(见 材料表),每天在远轴叶表面上测量气孔导度,从第20天到缺水期结束。
注意:当气孔导度值约为播种后第20天获得的气孔导度值的50%时,为每个植物单独确定缺水期的结束。
3. 结核收获
- 将液氮收集在干净的容器中,并标记 15 mL 管(每个植物一个)。
注意:戴上冷冻手套、面罩和安全护目镜处理液氮。 - 单独检索每个植物。切割并丢弃地上部分。用水彻底清洗根部以去除任何残留的基质。
- 将每个结节从根部分离,并将它们收集在预冷的 15 mL 管中。快速冷冻试管并将其储存在-80°C。
4. 胞质提取物的制备
注意:该协议的最终目的是获得高质量的总RNA(TOTAL)和多聚体相关RNA(PAR)。因此,在防止RNA降解的条件下工作,始终将样品保持在4°C并使用不含RNase的实验室设备和溶液。除非另有说明,否则所有溶液均用无菌超纯水制备。
- 缓冲储备溶液的制备
- 制备 表1中列出的可高压灭菌储备溶液,高压灭菌15分钟,并保持在室温下。
- 制备 表1中列出的可过滤灭菌储备溶液,对其进行过滤灭菌,并将其保持在-20°C。
- 制备多聚体提取缓冲液(PEB,见 表1)并将其保存在冰上。
注意:给出的体积适用于六个样品。 - 将离心机冷却至4°C,并将2mL微量离心管置于冰上。
- 在称量培养皿中称取约 0.2 g 完整结节,并将它们转移到预冷的 2 mL 管中。
注意:快速执行此步骤以避免样品解冻。或者,可以使用估计的0.4-0.5mL结节体积代替0.2g。 - 加入 1.2 mL PEB,让样品解冻 2 分钟,并用组织研磨机匀浆,直到结节完全破碎和匀浆。
注意:解冻后一定要研磨结节,因为它们足够柔软,很容易被微量离心管中的组织研磨机(见 材料表)破坏。冷冻结节很难研磨。此外,建议将试管放置在台式冷却器(0°C)中,以便在保持样品冷却的同时进行适当的均质化。 - 将样品在冰上轻轻搅拌孵育10分钟或直到所有样品都已处理完毕。
- 在4°C下以16,000× g 离心15分钟以沉淀碎片。回收上清液并重复离心步骤。
- 小心回收澄清的胞质提取物,并将 200 μL 等分试样转移到干净的 1.5 mL 微量离心管中进行完全分离(总样品;参见第 7 节)。
5.蔗糖垫的制备
注意:本协议在13.2 mL超速离心管中使用两层蔗糖垫(12%和33.5%)(参见 材料表)。所有溶液均用无菌超纯水制备。
- 准备蔗糖和盐储备溶液。
- 制备100 mL的2 M蔗糖溶液(68.5%, 表1)。
- 准备10x盐溶液(表1)。
- 如 表2所述制备两层蔗糖垫。有关抗生素(CHX和CHL)储备溶液的组成,请参见 表1 。
注意:给出的体积适用于六个垫子。 - 将 4.5 mL 的 33.5% 蔗糖层倒入离心管中。然后,用 P1000 微量移液器小心缓慢地加入 4.5 mL 的 12% 层。将试管放在冰上,直到加入澄清的胞质提取物。
6. 多聚体纯化
- 小心地移液到管的侧壁上,将 1 mL 澄清的胞质提取物(步骤 4.8)加载到蔗糖垫的顶部。
- 将超速离心管转移到预冷桶中,并在4°C下以217,874 × g(参考转子中的35,000rpm [参见 材料表])离心2小时。
- 丢弃剩余的胞质提取物和蔗糖垫,并用200μL预冷RB(先前根据 表1制备)重悬多体沉淀。
- 在冰上孵育30分钟,然后将多体重悬转移到1.5mL预冷管中。继续进行RNA提取(PAR样品)。
7. RNA提取和质量控制
注意:此步骤对 TOTAL(步骤 4.8)和 PAR 样本(步骤 6.4)执行。
- 用无菌超纯水制备75%的EtOH,并将其冷却至-20°C。 此外,将氯仿和异丙醇冷却至-20°C。
- 用 750 μL RNA 分离试剂匀浆样品,并在室温下孵育 5 分钟。
- 加入 200 μL 冷氯仿并剧烈摇动试管 15 秒。在室温下孵育10分钟。
- 在4°C下以12,000× g 离心15分钟以进行相分离。在不干扰粉红色有机相的情况下将 500 μL 上层水相转移到干净的管中,并加入 375 μL 冷异丙醇和 0.5 μL 无 RNase 糖原(参见 材料表)。通过上下移液充分混合。
注意:糖原是一种载体,用作辅助沉淀剂,以增加酒精沉淀中的核酸回收率。 - 将混合物在4°C孵育10分钟,并以12,000× g 离心15分钟。弃去上清液。
- 用 1 mL 冷的 75% EtOH 洗涤 RNA 沉淀。通过短暂的涡旋混合。
注意:此时,样品可以在-20°C下储存过夜。 - 在4°C下以7,500× g 离心5分钟,用微量移液管小心地除去上清液,并风干RNA沉淀。
- 将沉淀溶解在50μL无RNA酶的水中,并在65°C下孵育5分钟。
- 使用高灵敏度毛细管电泳18(参见材料表)和/或在2%无RNA酶琼脂糖凝胶19上电泳(参见材料表)评估RNA浓度和完整性。
注意:如果RNA样品不打算立即使用或将被送到测序设施进行RNA-seq文库制备,建议使用EtOH沉淀它们。
8. 核糖核酸沉淀
- 将离心机和乙基醇冷却至4°C。
- 准备3 M乙酸钠。
- 用无菌超纯水制备70%的EtOH,并将其冷却至-20°C。
- 估计样品量。加入 0.1 体积的 3 M 乙酸钠、3 体积的冷 EtOH 和 0.5 μL 无 RNase 糖原。充分混合。
- 在-20°C下放置直到需要。
注意:RNA沉淀样品可以在-80°C下储存长达1年。
9. 基因表达分析的标准管道
- 平均执行 40M Illumina 配对端读取,读取长度>100 bp,以进行可靠的转录组和翻译组数据分析。
注意:标准RNA-seq数据分析包括以下步骤9.2-9.5。 - 使用 FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) 或多样本MultiQC 20 进行读取质量检测。
- 使用Trimmomatic21,BBDuk(http://jgi.doe.gov/data-and-tools/bb-tools/),cutadapt 22,镰刀23或Trim Galore(https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/)等去除适配器和低质量序列。
- 如果该物种有参考基因组,则使用Bowtie224,TopHat225或Salmon 26以及featureCounts 27以及其他开源工具对参考进行读取映射,然后进行丰度定量。
- 使用 edgeR28、DESeq229 或 limma30 等对差异表达基因进行统计分析。
注意:这些开源软件可以通过命令行在本地使用。或者,它们可以通过公共服务器上的Web浏览器进行操作,例如Galaxy31 或GEOexplorer32,它们提供图形用户界面,因此使用它们不需要命令行知识。
结果
使用上述程序纯化的TOTAL和PAR级分的数量和质量评估是决定其成功与否的关键,因为对于大多数下游应用,例如RNA测序,高质量样品是文库制备和测序的基础。此外,RNA分子的完整性允许在样品收集时捕获基因表达谱的快照18。在这种情况下,使用生物分析仪进行这些测量时会获得RNA完整性数(RIN)。RIN 用于以稳健、可靠且独立于用户的方式分配完整性值,范围从 10(完整)到 1...
讨论
在翻译水平上研究基因表达调控对于更好地理解不同的生物学过程至关重要,因为细胞基因表达的终点是蛋白质丰度13,14。这可以通过分析感兴趣的组织或生物体的翻译组来评估,其中应纯化多体部分并分析其相关的mRNA。
披露声明
作者没有利益冲突。
致谢
这项研究由CSIC I + D 2020年第282号资助,FVF 2017年第210号资助和PEDECIBA(玛丽亚·玛莎·塞恩斯)资助。
材料
| Name | Company | Catalog Number | Comments |
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
参考文献
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer's disease model mice. Journal of Alzheimer's Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。