Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Purification des polysomes à partir de nodules symbiotiques de soja
Dans cet article
Résumé
Ce protocole décrit une méthode de purification eucaryote des polysomes à partir de nodules de soja intacts. Après le séquençage, des pipelines standard pour l’analyse de l’expression génique peuvent être utilisés pour identifier les gènes exprimés différentiellement aux niveaux du transcriptome et du translatome.
Résumé
L’objectif de ce protocole est de fournir une stratégie pour étudier le translatome eucaryote du nodule symbiotique du soja (Glycine max). Cet article décrit des méthodes optimisées pour isoler les polyribosomes dérivés de plantes et leurs ARNm associés à analyser à l’aide du séquençage de l’ARN. Premièrement, les lysats cytoplasmiques sont obtenus par homogénéisation dans des conditions de préservation des polysomes et de l’ARN à partir de nodules de soja entiers congelés. Ensuite, les lysats sont éliminés par centrifugation à basse vitesse, et 15% du surnageant est utilisé pour l’isolement de l’ARN total (TOTAL). Le lysat éliminé restant est utilisé pour isoler les polysomes par ultracentrifugation à travers un coussin de saccharose à deux couches (12% et 33,5%). L’ARNm associé aux polysomes (PAR) est purifié à partir de pastilles polysomiques après remise en suspension. TOTAL et PAR sont évalués par électrophorèse capillaire très sensible pour répondre aux normes de qualité des bibliothèques de séquençage pour le séquençage de l’ARN-seq. À titre d’exemple d’application en aval, après séquençage, des pipelines standard pour l’analyse de l’expression génique peuvent être utilisés pour obtenir des gènes exprimés différentiellement aux niveaux du transcriptome et du translatome. En résumé, cette méthode, en combinaison avec le séquençage de l’ARN, permet l’étude de la régulation translationnelle des ARNm eucaryotes dans un tissu complexe tel que le nodule symbiotique.
Introduction
Les légumineuses, comme le soja (Glycine max), peuvent établir une symbiose avec des bactéries spécifiques du sol appelées rhizobiums. Cette relation mutualiste provoque la formation de nouveaux organes, les nodules symbiotiques, sur les racines des plantes. Les nodules sont les organes végétaux hébergeant la bactérie et sont constitués de cellules hôtes dont le cytoplasme est colonisé par une forme spécialisée de rhizobiums appelée bactéroïdes. Ces bactéroïdes catalysent la réduction de l’azote atmosphérique (N2) en ammoniac, qui est transféré à la plante en échange de glucides 1,2.
Bien que cette symbiose fixatrice d’azote soit l’une des symbioses plantes-microbes les mieux étudiées, de nombreux aspects restent à mieux comprendre, tels que la façon dont les plantes soumises à différentes conditions de stress abiotique modulent leur interaction avec leur partenaire symbiotique et comment cela affecte le métabolisme des nodules. Ces processus pourraient être mieux compris en analysant le translatome nodule (c’est-à-dire le sous-ensemble d’ARN messagers [ARNm] activement traduit). Les polyribosomes ou polysomes sont des complexes de ribosomes multiples associés à l’ARNm, couramment utilisés pour étudier la traduction3. La méthode de profilage des polysomes consiste en l’analyse des ARNm associés aux polysomes et a été utilisée avec succès pour étudier les mécanismes posttranscriptionnels contrôlant l’expression des gènes qui se produisent dans divers processus biologiques 4,5.
Historiquement, l’analyse de l’expression du génome s’est principalement concentrée sur la détermination de l’abondance de l’ARNm 6,7,8,9. Cependant, il y a un manque de corrélation entre les niveaux de transcrits et de protéines en raison des différentes étapes de la régulation post-transcriptionnelle de l’expression génique, en particulier la traduction10,11,12. De plus, aucune dépendance n’a été observée entre les changements au niveau du transcriptome et ceux qui se produisent au niveau du translatome13. L’analyse directe de l’ensemble des ARNm en cours de traduction permet une mesure plus précise et complète de l’expression génique cellulaire (dont le critère d’évaluation est l’abondance des protéines) que celle obtenue lorsque seuls les niveaux d’ARNm sont analysés14,15,16.
Ce protocole décrit comment les polysomes d’origine végétale sont purifiés à partir de nodules de soja intacts par centrifugation différentielle à travers un coussin de saccharose à deux couches (Figure 1). Cependant, comme les ribosomes dérivés de bactéroïdes sont également présents dans les nodules, un mélange d’espèces de ribosomes et d’ARN est purifié, même si les eucaryotes représentent la fraction principale (90%-95%). L’isolement, la quantification et le contrôle de la qualité de l’ARN qui s’ensuivent sont également décrits (figure 1). Ce protocole, en combinaison avec RNA-seq, devrait fournir des résultats expérimentaux sur la régulation translationnelle des ARNm eucaryotes dans un tissu complexe tel que le nodule symbiotique.
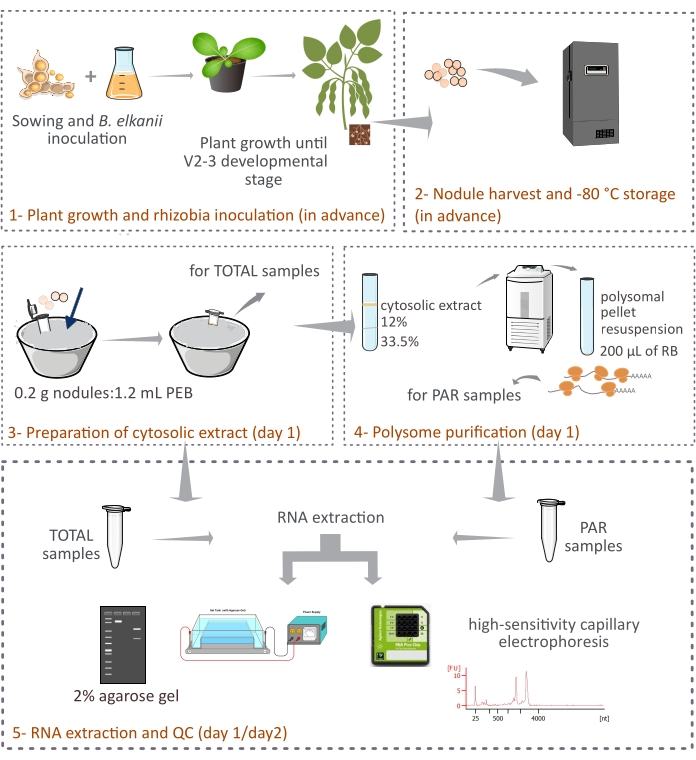
Figure 1 : Aperçu schématique de la méthodologie proposée pour la purification des polysomes eucaryotes à partir de nodules symbiotiques. Le schéma donne un aperçu des étapes suivies dans le protocole depuis (1) la croissance des plantes et (2) la récolte des nodules jusqu’à (3) la préparation des extraits cytosoliques, (3) l’obtention d’échantillons TOTAL et (4) d’échantillons PAR, et (5) l’extraction de l’ARN et le contrôle de la qualité. Abréviations : PEB = tampon d’extraction de polysomes; RB = tampon de remise en suspension; TOTAL = ARN total; PAR = ARNm associé aux polysomes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Croissance des plantes et inoculation des rhizobiums
- Pour générer les nodules requis, semez les graines de soja de votre choix dans le substrat sélectionné dans une chambre de croissance dans des conditions contrôlées.
NOTE: Dans ce protocole, les graines ont été semées dans une bouteille en plastique de 0,5 L remplie d’un mélange de sable:vermiculite (1:1). La chambre de croissance a été réglée avec une température du cycle jour/nuit de 28 °C / 20 ° C, respectivement, et une photopériode lumière/obscurité de 16 h / 8 h, respectivement. L’intensité du rayonnement photosynthétiquement actif était de 620 μmol·m−2·s−1. - Préparer à l’avance le milieu liquide d’extrait de levure de mannitol (YEM) (voir le tableau des matériaux). Autoclave le médium.
- Le jour même du semis, inoculer une fiole contenant 100 ml de YEM avec la souche Bradyrhizobium elkanii U1302 d’une fiole contenant 100 mL. Incuber la fiole à 28 °C sur un agitateur orbital à 100 tr/min.
- Laissez les rhizobiums pousser pendant 2 jours et inoculez les plantules avec 2 ml de culture.
2. Traitement du déficit hydrique (facultatif)
REMARQUE : Ce protocole décrit le traitement du déficit hydrique des plants de soya. Cette partie peut être modifiée ou omise entièrement en fonction de la question expérimentale en question.
- Cultivez les plants de soja pendant 19 jours (stade de développement V2-3) sans restriction d’eau. Maintenez le substrat à sa capacité sur le terrain avec B&D-medium17 complété par 0,5 mM KNO3.
- Le jour 20, retirez l’arrosage.
NOTE: Ici, la teneur en eau a été mesurée quotidiennement par gravimétrie (teneur gravimétrique en eau) pendant les périodes de croissance et de déficit hydrique. - Mesurer quotidiennement la conductance stomatique en trois exemplaires sur la surface foliaire abaxiale à l’aide d’un poromètre, selon les instructions du fabricant (voir le tableau des matériaux), du jour 20 jusqu’à la fin de la période de déficit hydrique.
NOTE : La fin de la période de déficit hydrique a été déterminée individuellement pour chaque plante lorsque la valeur de conductance stomatique était d’environ 50 % de celle obtenue au jour 20 du semis.
3. Récolte des nodules
- Prélever l’azote liquide dans un contenant propre et étiqueter des tubes de 15 mL (un pour chaque plante).
ATTENTION : Manipulez de l’azote liquide en portant des gants cryogéniques, des écrans faciaux et des lunettes de sécurité. - Récupérez chaque plante individuellement. Couper et jeter la partie aérienne. Lavez soigneusement la racine avec de l’eau pour enlever tout substrat restant.
- Détacher chaque nodule de la racine et les recueillir dans un tube prérefroidi de 15 mL. Congeler les tubes et les conserver à −80 °C.
4. Préparation d’extraits cytosoliques
NOTE: L’objectif final de ce protocole est d’obtenir un ARN total (TOTAL) et un ARN associé aux polysomes (PAR) de haute qualité. Par conséquent, travaillez dans des conditions qui empêchent la dégradation de l’ARN, en maintenant toujours les échantillons à 4 °C et en utilisant des équipements et des solutions de laboratoire exempts de RNase. Sauf indication contraire, toutes les solutions sont préparées avec de l’eau ultrapure stérile.
- Préparation de solutions de stock régulateur
- Préparer les solutions mères autoclavables énumérées dans le tableau 1, les autoclaver pendant 15 minutes et conserver à TA.
- Préparer les solutions mères stérilisables par filtre énumérées dans le tableau 1, les stériliser par filtre et les conserver à −20 °C.
- Préparez un tampon d’extraction de polysomes (PEB, voir le tableau 1) et conservez-le sur la glace.
NOTE: Les volumes donnés concernent six échantillons. - Refroidir la centrifugeuse à 4 °C et placer des tubes microcentrifugeux de 2 ml sur de la glace.
- Peser environ 0,2 g de nodules intacts dans une boîte de pesée et les transférer dans un tube prérefroidi de 2 mL.
REMARQUE: Effectuez cette étape rapidement pour éviter la décongélation des échantillons. Alternativement, un volume de nodule estimé de 0,4-0,5 mL peut être utilisé au lieu de 0,2 g. - Ajouter 1,2 mL de PEB, laisser l’échantillon décongeler pendant 2 minutes et homogénéiser avec un broyeur de tissus jusqu’à perturbation complète et homogénéisation des nodules.
REMARQUE : Assurez-vous de broyer les nodules une fois décongelés, car ils seront suffisamment mous pour être facilement perturbés avec des broyeurs de tissus (voir le tableau des matériaux) dans des tubes à microcentrifugeuses. Les nodules congelés sont difficiles à broyer. En outre, il est recommandé de placer les tubes dans une glacière de paillasse (0 ° C) pour une homogénéisation correcte tout en gardant les échantillons au frais. - Incuber les échantillons sur de la glace en agitant doucement pendant 10 minutes ou jusqu’à ce que tous les échantillons aient été traités.
- Centrifuger à 16 000 × g pendant 15 min à 4 °C pour pelleter les débris. Récupérez le surnageant et répétez l’étape de la centrifugeuse.
- Récupérer soigneusement l’extrait cytosolique clarifié et transférer une partie aliquote de 200 μL dans un tube microcentrifugé propre de 1,5 mL pour l’isolement TOTAL (échantillons TOTAL ; voir rubrique 7).
5. Préparation des coussins de saccharose
REMARQUE : Ce protocole utilise un coussin de saccharose à deux couches (12 % et 33,5 %) dans des tubes ultracentrifuges de 13,2 ml (voir le tableau des matériaux). Toutes les solutions sont préparées avec de l’eau stérile ultrapure.
- Préparer les solutions mères de saccharose et de sel.
- Préparer 100 mL de solution de saccharose 2 M (68,5 %, tableau 1).
- Préparer 10x solution saline (tableau 1).
- Préparer les deux couches du coussin de saccharose comme décrit dans le tableau 2. Voir le tableau 1 pour la composition des solutions mères d’antibiotiques (CHX et CHL).
NOTE: Les volumes donnés sont pour six coussins. - Verser 4,5 mL de la couche de saccharose à 33,5 % dans les tubes à centrifuger. Ensuite, ajoutez 4,5 ml de la couche à 12% avec précaution et lentement avec une micropipette P1000. Mettre les tubes sur de la glace jusqu’à l’ajout de l’extrait cytosolique clarifié.
6. Purification des polysomes
- Charger 1 mL de l’extrait cytosolique clarifié (étape 4.8) sur le coussin de saccharose en pipetant soigneusement sur la paroi latérale du tube.
- Transférer les tubes d’ultracentrifugation dans des godets prérefroidis et centrifuger à 217 874 × g (35 000 tr/min dans le rotor référencé [voir le tableau des matériaux]) pendant 2 h à 4 °C.
- Jeter l’extrait cytosolique restant et le coussin de saccharose, et remettre en suspension la pastille polysomale avec 200 μL de RB prérefroidi (préalablement préparé conformément au tableau 1).
- Incuber pendant 30 minutes sur de la glace, puis transférer la remise en suspension polysomale dans des tubes prérefroidis de 1,5 mL. Procéder à l’extraction de l’ARN (échantillons PAR).
7. Extraction de l’ARN et contrôle de la qualité
REMARQUE : Cette étape est effectuée pour les échantillons TOTAL (étape 4.8) et PAR (étape 6.4).
- Préparez 75% d’EtOH avec de l’eau ultrapure stérile et refroidissez-le à −20 °C. De plus, refroidir le chloroforme et l’isopropanol à −20 °C.
- Homogénéiser les échantillons avec 750 μL du réactif d’isolement d’ARN et incuber pendant 5 min à TA.
- Ajouter 200 μL de chloroforme froid et agiter vigoureusement les tubes pendant 15 s. Incuber à TA pendant 10 min.
- Centrifuger à 12 000 × g pendant 15 min à 4 °C pour la séparation des phases. Transférer 500 μL de la phase aqueuse supérieure dans un tube propre sans perturber la phase organique rose, et ajouter 375 μL d’isopropanol froid et 0,5 μL de glycogène sans RNase (voir le tableau des matières). Bien mélanger en tuyant de haut en bas.
REMARQUE: Le glycogène est un support utilisé comme co-précipitant pour augmenter la récupération des acides nucléiques de la précipitation de l’alcool. - Incuber le mélange pendant 10 min à 4 °C et centrifuger à 12 000 × g pendant 15 min. Jetez le surnageant.
- Laver le précipité d’ARN avec 1 mL d’EtOH froid à 75%. Mélanger par vortex bref.
REMARQUE : À ce stade, les échantillons peuvent être conservés à −20 °C pendant une nuit. - Centrifuger à 7 500 × g pendant 5 min à 4 °C, retirer délicatement le surnageant à l’aide d’une micropipette et sécher à l’air la pastille d’ARN.
- Dissoudre la pastille dans 50 μL d’eau exempte de RNase et incuber à 65 °C pendant 5 min.
- Évaluer la concentration et l’intégrité de l’ARN par électrophorèse capillaire18 très sensible (voir le tableau des matériaux) et/ou électrophorèse sur un gel d’agarose19 exempt de RNase à 2 % (voir le tableau des matériaux).
REMARQUE: Si les échantillons d’ARN ne vont pas être utilisés immédiatement ou vont être envoyés à l’installation de séquençage pour la préparation de la bibliothèque RNA-seq, il est recommandé d’utiliser EtOH pour les précipiter.
8. Précipitation de l’ARN
- Refroidir la centrifugeuse et l’EtOH à 4 °C.
- Préparer l’acétate de sodium 3 M.
- Préparez 70% d’EtOH avec de l’eau ultrapure stérile et refroidissez-le à −20 °C.
- Estimez le volume de l’échantillon. Ajouter 0,1 volume d’acétate de sodium 3 M, 3 volumes d’EtOH froid et 0,5 μL de glycogène sans RNase. Mélanger.
- Laisser à −20 °C jusqu’à ce que vous en ayez besoin.
REMARQUE : Les échantillons d’ARN précipités peuvent être conservés jusqu’à 1 an à −80 °C.
9. Pipeline standard pour l’analyse de l’expression génique
- Effectuez en moyenne 40 millions de lectures d’extrémités appariées Illumina, avec une longueur de lecture de >100 pb, pour une analyse fiable des données du transcriptome et du translatome.
REMARQUE : L’analyse standard des données RNA-seq comprend les étapes suivantes 9.2-9.5. - Effectuez un contrôle de qualité de lecture à l’aide de FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) ou du MultiQC20 multiéchantillon.
- Retirez l’adaptateur et les séquences de mauvaise qualité à l’aide de Trimmomatic21, BBDuk (http://jgi.doe.gov/data-and-tools/bb-tools/), cutadapt 22, sickle23 ou Trim Galore (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/), entre autres.
- Si un génome de référence est disponible pour l’espèce, effectuez une cartographie en lecture par rapport à la référence, suivie d’une quantification de l’abondance, en utilisant Bowtie224, TopHat2 25 ou Salmon 26, et featureCounts27, en plus d’autres outils open source.
- Effectuez une analyse statistique pour l’identification différentielle des gènes exprimés à l’aide de edgeR28, DESeq229 ou limma30, entre autres.
REMARQUE: Ces logiciels open-source peuvent être utilisés localement, via la ligne de commande. Alternativement, ils peuvent être exploités via un navigateur Web sur un serveur public, tel que Galaxy31 ou GEOexplorer32, qui offrent une interface utilisateur graphique, de sorte qu’aucune connaissance de la ligne de commande n’est requise pour leur utilisation.
Résultats
L’évaluation de la quantité et de la qualité des fractions TOTAL et PAR purifiées avec la procédure susmentionnée est essentielle pour déterminer son succès, car pour la plupart des applications en aval, telles que le séquençage de l’ARN, des échantillons de haute qualité sont fondamentaux pour la préparation et le séquençage des bibliothèques. De plus, l’intégrité des molécules d’ARN permet la capture d’un instantané du profil d’expression génique au moment du prélèvement de l’échan...
Discussion
L’étude de la régulation de l’expression génique au niveau translationnel est essentielle pour mieux comprendre les différents processus biologiques puisque le critère d’évaluation de l’expression génique cellulaire est l’abondance des protéines13,14. Ceci peut être évalué en analysant le translatome du tissu ou de l’organisme d’intérêt pour lequel la fraction polysomale doit être purifiée et ses ARNm associés...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts.
Remerciements
Cette recherche a été financée par la subvention 282 de la SCCI I+D 2020, la subvention FVF 2017 n° 210 et PEDECIBA (María Martha Sainz).
matériels
| Name | Company | Catalog Number | Comments |
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Références
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer's disease model mice. Journal of Alzheimer's Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon